PRISM -Tips 5. Fluorophore. Protokolle. Trennsysteme. Datenanalyse
|
|
|
- Gotthilf Huber
- vor 8 Jahren
- Abrufe
Transkript
1 PRISM -Tips 5 Informationen zur automatischen DNA-Sequenzierung und Fragmentanalyse mit den ABI PRISM -Systemen 310, 373 und 377 Fluorophore Protokolle Trennsysteme Datenanalyse April 99
2 Inhalt Seite 1 1. Einleitung 2 2. Fluoreszenz-Farbstoffe Überblick Fluoreszenz-Farbstoffe Auswahl von Farbstoffen für die Fragmentanalyse Tips zur Fragmentanalyse Matrixerstellung 5 3. Probenvorbereitung für die Sequenzanalyse Plasmid-DNA Präparation PCR-Produkte als Template Primer-Design und Qualität 6 4. Sequenzierungsreaktionen Protokolle für BigDyeTerminator Reaktionen Tips zur Sequenzierung Elektrophorese Zusätzlich benötigte Reagenzien und Materialien Elektrophorese Tips Standard Trennsysteme Spezial Trennsysteme Elektrophorese 377 / Zusätzlich benötigte Reagenzien und Materialien Stammlösungen Gel-Vorbereitung Wasserlade-Protokoll / 373 Elektrophorese Tips 16 SEQ Trennsysteme 17 SEQ Gel- und Ladeprotokolle 18 SEQ Moduleinstellungen 19 GS Trennsysteme 20 GS Gelprotokolle 20 GS Moduleinstellungen Gelsysteme Datenauswertung GS-Standards im Überblick Tips zum Sequence Navigator 24
3 1. Einleitung 2 Diese Broschüre enthält aktuelle Hinweise zur DNA-Sequenzierung und Fragmentanalyse mit den ABI PRISM -Systemen 310, 373 und 377. Ziel dieser Broschüre ist es, Anwendern den Einstieg in die neue Technologie zu erleichtern und erfahrene Anwender über neue Entwicklungen zu informieren. PE Biosystems als innovative Firma bietet kontinuierlich neue Produkte, Systemerweiterungen und Protokolloptimierungen an. Deshalb können Informationen, welche in dieser Broschüre enthalten sind, schon nach kurzer Zeit wieder überholt sein! Aktuelle Informationen erhalten Sie unter: Preise, Produktunterlagen, Angebote Verkauf Bestellungen, Reklamationen Auftragsbearbeitung Technische Fragen, Ersatzteile Service Hotline PCR-spezifische Fragen PCR Support Hotline Anwendungsspezifische Fragen GA Support Hotline Trainingskurse Anmeldung Oligosynthese Custom Synthesis PE Biosystems Österreich Support Homepage ( Literatur (GS+Seq Guide)
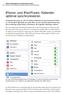
4 2. Fluoreszenz-Farbstoffe Überblick Fluoreszenz-Farbstoffe Filterset A Für jedes Farbstoff-Set muß eine spezifische Matrix erstellt werden! Sequenzanalyse Wellenlänge 535 nm 559 nm 586 nm 615 nm DyePrimer 5-FAM JOE TAMRA ROX (C) (A) (G) (T) DyeTerminator R110 R6G TAMRA ROX (G) (A) (T) (C) Filterset E Wellenlänge 540 nm 570 nm 595 nm 625 nm BigDye Primer dr110 BD dr6g BD dtamra BD drox BD (C) (A) (G) (T) drhodamin Terminator dr110 dr6g dtamra drox (G) (A) (C) (T) BigDye Terminator dr110 BD dr6g BD dtamra BD drox BD (G) (A) (T) (C) Fragmentanalyse (GeneScan) Filterset A Wellenlänge 535 nm 559 nm 586 nm 615 nm STR-Primer, Stockmarks Pferd 5-FAM JOE TAMRA ROX AFLP 6-FAM HEX TAMRA ROX FilterSet B/C Wellenlänge 532 nm 543 nm 557 nm 584 nm OLA 6-FAM TET HEX TAMRA Filterset D Wellenlänge 532 nm 559 nm 580 nm 615 nm Linkage Mapping Set 2 6-FAM HEX NED ROX Filterset F Wellenlänge 535 nm 559 nm 580 nm 615 nm Profiler, AFLP Microbial, 5-FAM JOE NED ROX Stockmarks Rind II
5 2.2 Auswahl von Farbstoffen für die Fragmentanalyse 4 Bei der Auswahl von Primermarkierungen für Multiplex-Analysen sollte die unterschiedliche Sensitivität und das unterschiedliche Laufverhalten der einzelnen Dyes berücksichtigt werden. Allgemein gilt: - PCR-Reaktionen, die hohe Ausbeuten liefern, sind mit einem Farbstoff geringer Empfindlichkeit zu markieren. - PCR-Reaktionen, die niedrige Ausbeuten liefern, sind mit einem Farbstoff hoher Empfindlichkeit zu markieren. - Sollen DNA-Fragmente gleicher Sequenz miteinander verglichen werden, müssen diese mit dem gleichen Farbstoff markiert werden, da die Farbstoffe wegen ihrer unterschiedlichen Größe ein voneinander abweichendes Laufverhalten zeigen. Abnehmende Empfindlichkeit > > > Farbstoff FAM JOE / TET HEX NED TAMRA ROX Sensitivität 100% 100% 50% 50% 25% 12% Faktor Farbstoff Abnehmende Laufgeschwindigkeit > > > FAM > HEX > JOE / TET > NED >> TAMRA >> ROX 2.3 Tips zur Fragmentanalyse Tips zur PCR PCR-Produkte mit +A Eine nachträgliche Behandlung des PCR-Ansatzes mit T4-Polymerase führt zur Entfernung überhängender ssdna und somit des zusätzlichen A s. Protokoll : 0,5 Units T4-Polymerase + 10 µl PCR-Ansatz 30 min bei 37 C inkubieren. Erzeugung von +A Für eine einfachere Interpretation von Mikrosatelliten-Daten empfiehlt es sich, die PCR- Bedingungen so zu wählen, daß die +A-Produkte mit Präferenz gebildet werden. Um das zu 3 erreichen, muß der reverse PCR-Primer am 5 -Ende um die Sequenz 5 GTTTCTT verlängert werden ( Pigtailing ). Außerdem sollte noch ein Zyklus von 60 C über 45 min an das Cycle-Protokoll angehängt werden. Poolen von PCR-Ansätzen Zur multifluorophoren Fragmentlängenbestimmung können auch mehere verschiedene PCR- Ansätze nach der Reaktion vereinigt werden. Der Pool muß dann jedoch bei RT mit 100% EtOH gefällt und anschließend bei RT mit 70% EtOH gut gewaschen werden. Nach dem Trocknen kann die Probe wieder in Wasser gelöst und zur Denaturierung in Formamid oder TSR eingesetzt werden. Tips zur Auswertung Signale brechen in anderen Farben durch Wenn zuviel DNA geladen wird, kann die systemeigene Matrix nicht mehr arbeiten (Überladen des Systems!). Eine überladene Probe zeigt an der Stelle der Hauptsignale an gleicher Position auch in anderen Farben Signale.
6 2.3 Matrixerstellung 5 1. Matrix Standard Läufe Matrix Standards getrennt voneinander auftrennen und individuelle Sample Files anlegen. 2. Rohdaten Analyse Matrix Standard Files öffnen und Rohdaten begutachten. Sind Signale vorhanden, gibt es unspezifische Peaks oder Fluoreszenzverunreinigungen, ist die Basislinie gerade? 3. Gute Peaks finden Je File einen Scanbereich (X-Achsenwert) definieren, der mindestens 5 eindeutig voneinander getrennte Peaks enthält, die von den Matrix Standard-Fragmenten stammen. Diese Peaks müssen mindestens 150 Fluoreszenzeinheiten (Y-Achsenwert) und nicht mehr als Fluoreszenzeinheiten hoch sein (von Basislinie bis Peakspitze). 4. Bereiche festlegen Scannummern der Anfangs- und Endpunkte (X-Achsenwerte) der Scanbereiche notieren. 5. Differenzen bilden Je Matrix-Standardfile die Differenz aus ermitteltem Anfangs- und Endpunkt bestimmen. Der kleinste Differenzwert wird bei der Erstellung der Matrix als Gesamtpunktzahl eingegeben. 6. Matrix erstellen Sequenz Matrix: A. DataUtility Programm öffnen > UTILITIES > MAKE MATRIX auswählen. B. Matrix-Standard Sample Files für C (blaue Farbe), A (grüne Farbe), G (gelbe Farbe), T (rote Farbe) importieren, bzw. Kitprotokoll beachten! C. Werte der jeweiligen Anfangspunkte bei Start At eingeben. D. Kleinsten Differenzwert oder bei Points eintragen. E. Matrix Art (Dye Primer oder Terminator) wählen. F. Matrix unter New File benennen und mit OK abspeichern. GeneScan Matrix: A. GS Analyse Programm öffnen > FILE > NEW > MATRIX auswählen. B. Matrix Standard Sample Files für B (blaue Farbe), G (grüne Farbe), Y (gelbe Farbe), R (rote Farbe) importieren. C. Werte der jeweiligen Anfangspunkte bei Start At eingeben. D. Kleinsten Differenzwert oder 1500 bei Points eintragen. E. Mit OK bestätigen und Matrix abspeichern. 7. Matrix überprüfen Sequenz Matrix: A. Im DataUtility Programm unter > FILE > Open Matrix Standard Files nacheinander auswählen und Datenfenster Analyzed betrachten. B. Bei einer guten Matrix sieht man nur Peaks in einer Farbe, während die Basislinien der anderen Farben gerade sind. Eine schlechte Matrix zeigt ebenfalls Peaks in anderen Farben an selber Stelle wie die Standardfragmente. GeneScan Matrix: A. Im GS Analyse Programm unter > FILE > NEW > PROJECT auswählen. B. Unter > PROJECT > ADD SAMPLE FILES Matrix Standard Files einladen. C. Spalte SAMPLE FILE markieren und Matrix unter > SAMPLE > INSTALL NEW MATRIX anhängen. D. Unter > SETTINGS > ANALYSIS PARAMETERS bei Analysis Range zuvor ermittelten Anfangspunkt bei Start und bei Stop eingeben. E. Spalten der Farben B, G, Y und R alle markieren und Analyze anklicken. F. Chromatogramme unter > WINDOWS > RESULTS CONTROL durch gleichzeitiges Aktivieren aller Farben pro Sample File betrachten. G. Bei einer guten Matrix sieht man nur Peaks in einer Farbe, während die Basislinien der anderen Farben gerade sind. Eine schlechte Matrix zeigt ebenfalls Peaks in anderen Farben an selber Stelle wie die Standardfragmente.
7 3. Probenvorbereitung für Sequenzanalysen Plasmid-DNA Präparation Alle Plasmid-Präparationsmethoden sind für eine anschließende DNA-Sequenzierung mit den ABI PRISM -Systemen geeignet. Beste Ergebnisse erhält man bei Beachtung folgender Punkte: RNAse-Behandlung Phenolextraktion Fällung, Zentrifugation, Waschen der DNA bei Raumtemperatur! Gründlich waschen bei Raumtemperatur! Fällung mit: 4 M Ammonium-Acetat Protokoll : 1 Vol DNA-Lösung + 1 Vol 4 M NH4OAc + 6 Vol 100% EtOH Lösen von DNA ausschließlich in Wasser Quantifizierung durch Aufnahme eines Spektrums nm 3.2 PCR-Produkte als Template Zwingende Voraussetzung für eine direkte Sequenzierung von PCR-Produkten ist ein einheitliches PCR-Produkt! Das beste PCR-Template erhält man aus einer optimierten PCR. Optimierungsschritte für PCR-Templates Primermenge 3-5 pmol, nicht mehr als 10 pmol einsetzen! Reaktionsvolumen < 25 µl Hot Start mit AmpliTaq Gold Verschiebung der Primersequenzen um 2-3 Basen in 5 -Richtung Bevor PCR-Produkte direkt sequenziert werden können, müssen unerwünschte Nebenprodukte (überschüssige Primer, nichteingebaute Nukleotide und Puffer-Salze) durch eine Ammonium-Acetat-Fällung (s.o.) oder Centricon 100 Säulenreinigung abgetrennt werden. Sind bei der PCR mehrere Produkte entstanden und möchte man eine Gelaufreinigung vermeiden Nested -Primer für die Sequenzierung verwenden. 3.3 Primer-Design und Qualität Ein PCR- oder Sequenzierprimer sollte möglichst nach folgenden Kriterien ausgewählt werden: Nukleotide lang Gleichmäßige Verteilung der Nukleotide (1:1:1:1) Base G oder C am 3 -Ende Keine Hairpin-loops (Komplementäre Rückfaltungen) Primer-Dimer Bildung sollte ausgeschlossen sein
8 Die Annealingtemperatur Ta des Primers kann nach folgender Näherungsformel abgeschätzt werden: Tm = 2 C x (A+T) + 4 C x (G+C) Ta = Tm - 5 C Der Primer sollte in Wasser gelöst sein und darf keine Nebenprodukte ( n-x Primer ) enthalten. Diese führen bei einer Sequenzierung mit DyeTerminatoren zu Sequenzüberlagerungen. 7 Sequenzüberlagerungen durch n-1 Primer Empfohlene Primersequenzen: Universal Primer -21M13: 5 ACG ACG TTG TAA AAC GAC GGC CAG 3 Reverse Primer M13RP1: 5 TTC ACA CAG GAA ACA GCT ATG ACC 3 4. Sequenzierungsreaktionen 4.1 Protokolle für BigDyeTerminator Reaktionen Auf den folgenden Seiten sind die wichtigsten Protokolle für eine Sequenzierung mit BigDyeTerminatoren auf je einer Seite zusammengefaßt. Angaben in diesen Protokollen können von Angaben in den mitgeschickten Kitprotokollen (englische Version) abweichen. Bitte führen Sie die Reaktionen unter den hier angegebenen Bedingungen durch!! Folgende Protokolle finden Sie in diesem Kapitel: - Standard Protokoll - Fast Protokoll - Large Template Protokoll Die Volumina des Reaktionsansatzes im Standardprotokoll können bei optimaler Templatequalität um den Faktor 4 reduziert werden, wobei es empfehlenswert ist, das Gesamtreaktionsvolumen nicht kleiner als 10 µl zu machen.
9 ABI PR SM Protokoll 8 AmpliTaq FS BigDyeTerminator Reaktionsansatz: Premix 8,0 µl DNA-Template ssdna 0,1 µg dsdna 0,2-0,5 µg PCR-Produkte (0,2-5 kb) ng Primer 10 pmol H 2 O ad 20 µl Nur bei TC 1 / 480 mit zwei Tropfen leichtem Mineralöl überschichten! Thermocycler Protokoll (25 Zyklen): Für Perkin Elmer Thermocycler! TC 1 / 480 TC 2400 / 9600 / C 30 sec 96 C 10 sec C 15 sec C 5 sec 60 C 4 min 60 C 4 min Aufreinigung Reaktionsansatz: (Centri Sep) (Ethanol Fällung) Säule mit 750 µl H 2 O 30 min vorquellen 80 µl H 2 O, 10 µl 3 M NaAc ph 4,6 Flüssigkeit ablaufen lassen und 250 µl 100% EtOH (RT) zugeben 2 min rpm zentrifugieren 15 min rpm zentrifugieren Reaktionsansatz (20 µl) zentral aufgeben EtOH entfernen 2 min rpm zentrifugieren mit 250 µl 70% EtOH (RT) waschen 5min rpm zentrifugieren bei Bedarf : EtOH entfernen Eluat 10 min in Speed-Vac trocknen Pellet 5 min in Speed-Vac trocknen Probenvorlage: µl Centri Sep Eluat + 20 µl TSR oder getrockneten Ansatz in 20 µl TSR aufnehmen 2 min bei 90 C denaturieren 377 Getrockneten Ansatz in 4 µl Formamid / 25 mm EDTA ph 8,0 (5:1) aufnehmen 2 min bei 90 C denaturieren und 1 µl auftragen 4 / 99
10 ABI PR SM Fast Protokoll 9 AmpliTaq FS BigDyeTerminator Reaktionsansatz: Premix 8,0 µl 4,0 µl Plasmid-DNA 1,5 µg - PCR-Produkte (0,2-5 kb) ng Primer (mind. 20mer) 10 pmol H 2 O ad 20 µl Thermocycler Protokoll (15 Zyklen): Für Perkin Elmer Thermocycler! TC 2400 / 9600 / C 10 sec 60 C 90 sec Aufreinigung Reaktionsansatz: (Centri Sep) (Ethanol Fällung) Säule mit 750 µl H 2 O 30 min vorquellen 80 µl H 2 O, 10 µl 3 M NaAc ph 4,6 Flüssigkeit ablaufen lassen und 250 µl 100% EtOH (RT) zugeben 2 min rpm zentrifugieren 15 min rpm zentrifugieren Reaktionsansatz (20 µl) zentral aufgeben EtOH entfernen 2 min rpm zentrifugieren mit 250 µl 70% EtOH (RT) waschen 5min rpm zentrifugieren bei Bedarf : EtOH entfernen Eluat 10 min in Speed-Vac trocknen Pellet 5 min in Speed-Vac trocknen Probenvorlage: µl Centri Sep Eluat + 20 µl TSR oder getrockneten Ansatz in 20 µl TSR aufnehmen 2 min bei 90 C denaturieren 377 Getrockneten Ansatz in 4 µl Formamid / 25 mm EDTA ph 8,0 (5:1) aufnehmen 2 min bei 90 C denaturieren und 1 µl auftragen 4 / 99
11 ABI PR SM Large Template Protokoll 10 AmpliTaq FS BigDyeTerminator Reaktionsansatz : Premix 8,0 µl DNA-Template Cosmide (bis 100 kb) 1,5 µg PACs ( kb) 3 µg BACs (bis 300 kb) 4-5 µg Primer 10 pmol DMSO 1 µl H 2 O ad 20 µl Thermocycler Protokoll (30 Zyklen): Für Perkin Elmer Thermocycler! Vorinkubation 5 min 96 C, dann Premix zugeben! Aufreinigung Reaktionsansatz : TC 2400 / 9600 / C 10 sec 50 C 5 sec 60 C 4 min (Centri Sep) (Ethanol Fällung) Säule mit 750 µl H 2 O 30 min vorquellen 80 µl H 2 O, 10 µl 3 M NaAc ph 4,6 Flüssigkeit ablaufen lassen und 250 µl 100% EtOH (RT) zugeben 2 min rpm zentrifugieren 15 min rpm zentrifugieren Reaktionsansatz (20 µl) zentral aufgeben EtOH entfernen 2 min rpm zentrifugieren mit 250 µl 70% EtOH (RT) waschen 5min rpm zentrifugieren EtOH entfernen Eluat 10 min in Speed-Vac trocknen Pellet 5 min in Speed-Vac trocknen Probenauftrag : 310 Getrockneten Ansatz in 20 µl TSR aufnehmen 2 min bei 90 C denaturieren 377 Getrockneten Ansatz in 4 µl Formamid / 25 mm EDTA ph 8,0 (5:1) aufnehmen 2 min bei 90 C denaturieren und 1 µl auftragen 4 / 99
12 4.2 Tips zur Sequenzierung 11 Reaktionsgefäße Sequenzierungsreaktionen nicht in farbigen Reaktionsgefäßen (Tubes) durchführen! BigDyeTerminator Sequenzierungs Tips GC-reiche DNA Zugabe von 1 µl DMSO zum 20 µl Reaktionsansatz (Endkonzentration 5%) oder Vorinkubation über 5 min bei 98 C vor Zugabe des Premixes und Cyceln. AT-reiche DNA Temperaturprofil 30 x (96 C 5 s, 50 C 4 min) oder Temperaturprofil 25 x (96 C 5 s, 60 C 90 s, 50 C 90 s) 4 µl Premix im 20 µl Ansatz Sekundärstrukturen 16 µl Premix und Temperaturprofil 25 x (98 C 20 s, 55 C 15 s, 60 C 4 min) oder Linearisierung des Templates oder die Generierung von ssdna z.b. über Magnetic Beads. Homopolymere Bereiche Temperaturprofil 30 x (96 C 5 s, 50 C 4 min) Neuer Sequenzierprimer, der 25 Basen aus dem Polymerbereich besitzt und am 3 -Ende die Base mit der die Sequenz weitergeht. Repeat-Strukturen Temperaturprofil 25 x (98 C 5 s, 60 C 90 s, 50 C 90 s) Niedrige Signalstärken trotz genügend DNA 16 µl Premix und 1 µl DMSO Primerannealing Primerbedingte Probleme, lassen sich in einigen Fällen lösen, indem man die Kettenverlängerung bei 55 C statt bei 60 C und 30 Zyklen anstelle von 25 durchführt. PCR-Produkte Sollen PCR-Produkte mit einer Länge bis zu 700 bp sequenziert werden, wird ein Ansatz mit halbem Premix = 4 µl im 20 µl Reaktionsansatz empfohlen. Fällung im 96er GeneAmp Format Folgendes Protokoll ermöglicht eine direkte Fällung nach der Sequenzierungsreaktion im Mikrotiterplatten-Format: 1. Zugabe von 74 µl 70% EtOH mit 0,5 mm MgCl 2 bei Raumtemperatur und gut mischen min bei Raumtemperatur stehen lassen min bei g zentrifugieren. 4. Mehrfach gefaltetes Papiertuch über Mikrotiterplatte legen und Platte umdrehen. 5. Umgekehrt 1 min bei 500 g zentrifugieren. 6. Papiertuch entfernen. Proben sind bereit und müssen nicht weiter getrocknet werden. Primer Tips (Dye)Primer Länge Für Primer mit niedrigen Schmelztemperaturen, wie z.b. die 17mere T3 / T7 und SP6, ist im Temperaturprofil eine Annealing-Temperatur von 45 C anstelle von 55 C auszuwählen. Probendenaturierung Denaturierungsbedingungen von markierter DNA: 2 min 90 C, nicht mehr!!!!
13 5. Elektrophorese Zusätzlich benötigte Reagenzien und Materialien Name Menge Anbieter Best.-Nr. Borsäure 1 kg Merck EDTA 100 g Sigma ED2SS Ethanol p.a. 2,5 l Merck Formamid Ultra 100 ml Sigma F5786 Glycerin p.a. 1l Merck Isopropanol, p.a. 2,5 l Merck Kimwipes Lite Stck Kimberly-Clark 7101 NaOH Plätzchen p.a. 1 kg Merck Tris 1 kg BIO-RAD Wasser (HPLC) 2,5 l Merck Zubehör für 96 Proben im Autosampler Genetic Analyzer Septa Strips PE Biosystems Genetic Analyzer Retainer Clips PE Biosystems ml MicroAmp Reaction Tubes PE Biosystems N MicroAmp Tray und Retainer PE Biosystems N MicroAmp Base PE Biosystems N Elektrophorese Tips Probeninjektion (Wasserladeprotokoll) Statt in TSR oder Formamid kann die DNA-Probe auch in Wasser vorgelegt und denaturiert werden. In diesem Fall muß die Injektionszeit verkürzt werden und zwar auf: 5-10 sec für Sequenzanalysen und auf 3-5 sec für Fragmentanalysen. Haltbarkeit von Elektrophorese-Reagenzien Geöffnete Reagenzien sollten spätestens nach 4 Wochen aufgebraucht sein. Ist Polymer auskristalisiert, kann es durch Erwärmen auf 37 C und gutes Mischen wieder in Lösung gebracht werden. Breite Peaks können entstehen, wenn Salze oder organische Verbindungen die Oberfläche der Kapillare besetzen. Reaktions-Ansätze bei RT fällen und gut waschen. Öl- und Ethanol-Reste führen ebenfalls zu Peakverbreiterungen. Probenladen Zur Denaturierung der PCR-Produkte, diese ausschließlich in hochreinem Formamid (Ultrapur) aufnehmen. Altes oder unreines Formamid führt zu einem abnormalen Laufverhalten der Fragmente. Außerdem wird damit weniger DNA geladen. Spannungsüberschläge Zur Vermeidung von Spannungsüberschlägen im Bereich des Autosamplers ist darauf zu achten, daß erstens die Puffergefäße nicht höher als bis zur Markierung gefüllt sind und zweitens keine Feuchtigkeit im Bereich des Septums oder an den Gefäßwänden kondensiert ist.
14 Standard Trennsysteme 13 Sequenzanalyse Anwendung: Ca. 450 Basen in 55 min Kapillare: Polymer: Puffer: Vorlage: Denaturierung: Modul: Seq POP6 RAPID (1 ml) 47 cm / 50 µm (grün) POP-6 1 x GA-Puffer mit EDTA Probe in 20 µl TSR lösen. 2 min bei 90 C und auf Eis abkühlen. Anwendung: Ca. 650 Basen in 2,5 h Kapillare: Polymer: Puffer: Vorlage: Denaturierung: Modul: Seq POP6 (1 ml) 61 cm / 50 µm (rot) POP-6 1 x GA-Puffer mit EDTA Probe in 20 µl TSR lösen. 2 min bei 90 C und auf Eis abkühlen. Denaturierende Fragmentanalyse Anwendung: 1 Basen-Auflösung bis 250 bp in 25 min Kapillare: Polymer: Puffer: Vorlage: Denaturierung: Modul: GS STR POP4 (1 ml) 47 cm / 50 µm (grün) POP-4 1 x GA mit EDTA 1 µl PCR-Ansatz / 0,5 µl Standard / 12 µl Formamid 2 min bei 90 C und auf Eis abkühlen. Anwendung: 1 Basen-Auflösung bis 350 bp in 55 min Kapillare: Polymer: Puffer: Vorlage: Denaturierung: Modul: Seq POP6 RAPID (1 ml) 47 cm / 50 µm (grün) POP-6 1 x GA mit EDTA 1 µl PCR-Ansatz / 0,5 µl Standard / 12 µl Formamid 2 min bei 90 C und auf Eis abkühlen.
15 Spezial Trennsysteme 14 Native Fragmentanalyse Kapillare Polymer I.D. 50 µm 47 cm (grün) GS Polymer (7%ig) Anwendung: SSCP Analyse Kapillare: Polymer: Puffer: Vorlage: Denaturierung: Modul: GS Template (1 ml) 47 cm / 50 µm (grün) 5% GS Polymer, 10% Glycerin, 1 x TBE 10% Glycerin, 1 x TBE 1 µl PCR-Ansatz / 0,5 µl Standard / 0,5 µl 0,3 N NaOH / 10,5 µl Formamid 2 min bei 90 C und auf Eis abkühlen. Einstellungen GS Template: Injection Time = 10 sec Syringe Pump Time = 180 sec Injection Voltage = 15 kv Pre-Injection EP = 0 sec Collection Time = 24 min EP Voltage 13 kv Heatplate Temperature = 30 C Anwendung: Fast Native mit 1% Basen-Auflösung bis 14 kb Modul: GS Template (1 ml) Kapillare: 47 cm / 50 µm (grün) Polymer: 2,5% GS Polymer, 1 x GA-Puffer mit EDTA Puffer: 1 x GA mit EDTA Vorlage: 1 µl PCR-Ansatz / 0,5 µl Standard / 12 µl Formamid Denaturierung: 2 min bei 90 C und auf Eis abkühlen. Einstellungen GS Template: Injection Time = 1 sec Syringe Pump Time = 30 sec Injection Voltage = 15 kv Pre-Injection EP = 0 sec Collection Time = 13 min EP Voltage 15 kv Heatplate Temperature = 60 C
16 6. Elektrophorese 377 / Zusätzlich benötigte Reagenzien und Materialien Name Menge Anbieter Best.-Nr. Acrylamid/Bisacrylamid (29:1) 30 g BIO-RAD Ammoniumperoxodisulfat 100 g Aldrich 24,861-4 Borsäure 1 kg Merck EDTA 100 g Sigma ED2SS Filtratflasche (250 ml) 1 Nalgene Formamid Ultra 100 ml Sigma F5786 Harnstoff 1 kg BIO-RAD Isopropanol p.a. 2,5 l Merck Kimwipes (Rolle) 1 Stck Kimberly-Clark 7201 Membranfilter RC58; 0.2 µm 1 Pckg Schleicher&Schuell NaOH Plätzchen p.a. 1 kg Merck PAGE-PLUS* 100 ml Biometra TAPS 1 kg Sigma T5130 TEMED 5 ml BIO-RAD Tris 1 kg BIO-RAD Wasser (HPLC) 2,5 l Merck Stammlösungen 30% PAA-Lösung* 10 x TBE-Puffer 10 x TTE-Puffer* 10% APS 58 g Acrylamid 108 g Tris g Tris 1 g APS 2 g Bisacrylamid 55 g Borsäure g TAPS 7.4 g Na 2 EDTA 3.72 g Na 2 EDTA 146 ml HPLC-H 2 O ad 1 l mit HPLC-H 2 O ad 1 l mit HPLC-H 2 O 10 ml HPLC-H 2 O * Diese Lösungen sind nach Öffnung nicht länger als 4 Wochen haltbar! 6.3 Gel-Vorbereitung Glasplatten mit 6 N NaOH behandeln, mit dest. Wasser spülen und mit Isopropanol p.a. / Wasser (9:1) nachbehandeln. Trocknen, Spacer auflegen und die Glasplatten zusammensetzen. - Gellösung filtrieren - APS und TEMED addieren - Lösung zwischen die vorbereiteten Platten gießen und 1 h polymerisieren lassen 6.4 Wasserlade-Protokoll - Obere Pufferkammer (max. 600 ml) mit deionisiertem Wasser füllen und Taschen spülen - DNA in Formamid / EDTA (10:1) aufnehmen und 2 min bei 90 C denaturieren, abkühlen - Ungerade Spuren beladen und DNA 2 min mit Pre-Run Modul einlaufen lassen - Gerade Spuren beladen und erneut 1-2 min einlaufen lassen - 10 x Puffer in obere Pufferkammer geben und mit dem Wasser gut mischen - Elektrophorese mit entsprechendem Runmodul starten 1 / 99
17 / 373 Elektrophorese Tips Haifischkamm einsetzen Setzen Sie den Kamm außerhalb des Gerätes ein, da dort die Lichtverhältnisse günstiger sind. Probenladen kann durch Zugabe von 1 µl einer konz. Dextran-Blau-Lösung zum Denaturierungs-Mix erleichtert werden. Für Fragmentanalysen (Square tooth comb) hilft eine Vorbeladung aller Taschen mit Dextranblau / Formamid, um die Taschen sichtbar zu machen. Nach Beladung 2 Minuten einwandern lassen und dann die Taschen spülen! Abnormales Laufverhalten Schlechte Resultate erhält man, wenn das Gel zu schnell läuft. Dies spiegelt sich in einem niedrigen Spacing-Wert, einer engen Peakfolge am Anfang und breiten Peaks ab Base 250 wieder. In diesem Fall bitte Wasser und Chemikalien überprüfen!! Schlechte Auflösung Zeigen insbesondere Sequenzierungsreaktionen einen plötzlichen Auflösungsverlust im Mittelteil der Sequenz, wobei die Signale immer breiter werden, befinden sich organische, nichtfluoresziernde Verbindungen auf den Glasplatten. Diese Verbindungen können durch Einlegen der Platten in 2 N NaOH über Nacht wieder entfernt werden. Auslöser können sein: Filtratflaschen, Handschuhe, Trockentücher, Spülmittel, Alkohol oder Detergenzien. Spuren verlassen den Fensterbereich Dieses Phänomen tritt auf, wenn entweder eine Verbindung zwischen oberer und unterer Pufferkammer besteht, indem Puffer in dem Spalt zwischen Spacer und Gel nach unten läuft, oder wenn die Taschen nach dem ersten Auftrag nicht gespült wurden. Da Formamid ein Isolator ist, ist es wichtig nach dem Laden und Einwandern der ersten Proben, alle Taschen nochmals gründlich zu spülen. Ansonsten fließt der Hauptstrom am linken und rechten Rand des Gels, was dazu führt, daß die Randspuren nach außen gezogen werden und eventuell das Gelfile-Fenster verlassen. Haltbarkeit von Elektrophorese-Lösungen Geöffnete Acrylamidlösungen sollten spätestens nach 4 Wochen aufgebraucht sein. TBE- Puffer sind zu verwerfen, wenn sich eine Trübung bzw. ein weißer Niederschlag zeigt. Taschen kollabieren (Bindesilanrezept) Bei Verwendung von Square-Tooth Kämmen entstehen Taschen aus Acrylamid, die beim Entfernen des Kammes leicht deformiert werden können. Um das zu verhindern, wird im Bereich dieser Taschen eine Glasplatte vor dem Gießen des Geles mit einer Bindesilan-Lösung behandelt. Bindesilan-Lösung: 1 ml EtOH µl 10%ige Essigsäure + 3 µl Bindesilan Reinigung von fluoreszierenden oder verunreinigten Glasplatten 1. Platten unter fließendem, warmen Wasser von Gel und Harnstoffresten befreien. 2. Platten über Nacht in 2 N NaOH p.a. einlegen. 2 N NaOH mit fluoreszenzfreiem, deionisierten Wasser ansetzen!! 3. Platten mit deionisiertem Wasser gut abspülen. 4. Platten trocknen. 5. Platten zusammensetzen und Gel gießen. Ladeschema für 96 Lane Gele rot 1 blau 2 schw 3 rot 4 blau 5 schw 6 rot 7 blau 8 schw 9 rot 10 blau 11 schw 12 A B C D E F G H
18 ABI PR SM SEQ Trennsysteme Leseweite (Basen) 4 h 8 h 9 h 10 h 12 h 18 h Datenaufnahme 36 cm 48 cm Glas- Platten Module* 200 B/h 100 B/h 90 B/h 4.5 % 5 % 4.8 % 4.25 % 5.25 % 4.25 % Gel- PAA PAA PAGE- PAA PAGE- PAGE- lösung (29:1) (29:1) PLUS (29:1) PLUS PLUS & TBE-Puffer TBE-Puffer TBE-Puffer TBE-Puffer TBE-Puffer TTE-Puffer Puffer * Veränderte Elektrophoreseparameter siehe Moduleinstellungen
19 ABI PR SM SEQ Gel- und Ladeprotokolle Für PAA-Gele (29:1) Lese- End- Laufpuffer weite konz. 30% Harnstoff 10 x 10x HPLC- TEMED 10% oben / (Basen) PAA TBE TTE Wasser APS unten % 7.5 ml 18 g 6 ml - 23 ml 20 µl 300 µl 1 x TBE % 8.4 ml 21 g 6 ml - 20 ml 20 µl 300 µl 1 x TBE % 7.1 ml 21 g 6 ml - 21 ml 20 µl 300 µl 1 x TBE Für PAGE-PLUS-Gele Lese- End- 40% Laufpuffer weite konz. PAGE- Harnstoff 10 x 10x HPLC- TEMED 10% oben / (Basen) Plus TBE TTE Wasser APS unten % 6 ml 18 g 5 ml - 23 ml 20 µl 300 µl 1 x TBE % 6.6 ml 18 g 5 ml - 23 ml 20 µl 300 µl 1 x TBE % 5.3 ml 18 g - 6 ml 21.5 ml 20 µl 300 µl 2 x TTE / 1 x TTE Ladeprotokolle Für alle Gelsysteme Wasserlade-Protokoll verwenden! Kamm 36 Lanes 48 Lanes 64 Lanes 96 Lanes max. Volumen 1-3 µl µl µl 0.5 µl 1 / 99
20 ABI PR SM SEQ Moduleinstellungen Für PAA-Gele (29:1) Modul (Filterset A oder E) Seq Run Für 550 Basen Electrophoresis Voltage 2700 V konstant Collection Time 4 Hours Electrophoresis Current 60 ma Gel Temperature 51 C Electrophoresis Power 200 W Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Modul (Filterset A oder E) Seq Run Für 650 Basen Electrophoresis Voltage 2500 V Collection Time 8 Hours Electrophoresis Current 50 ma Gel Temperature 51 C Electrophoresis Power 40 W konstant Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Modul (Filterset A oder E) Seq Run Für 750 Basen Electrophoresis Voltage 2400 V konstant Collection Time 10 Hours Electrophoresis Current 50 ma Gel Temperature 51 C Electrophoresis Power 200 W Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Für PAGE-PLUS-Gele Modul (Filterset A oder E) Seq Run Für 700 Basen Electrophoresis Voltage 1680 V konstant Collection Time 9 Hours Electrophoresis Current 50 ma Gel Temperature 51 C Electrophoresis Power 150 W Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Modul (Filterset A oder E) Seq Run Für 800 Basen Electrophoresis Voltage 2400 V konstant Collection Time 12 Hours Electrophoresis Current 50 ma Gel Temperature 51 C Electrophoresis Power 200 W Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Modul (Filterset A oder E) Seq Run Für 1000 Basen Electrophoresis Voltage 3000 V Collection Time 18 Hours Electrophoresis Current 60 ma Gel Temperature 45 C Electrophoresis Power 40 W konstant Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 * Bei 96 Lanes: CCD Offset = 0 CCD Gain = 4
21 ABI PR SM GS Trennsysteme 500 bp 800 bp 2500 bp Fragment- Länge Auflösung (Basen) 3 h 5 h 9 h Datenaufnahme 36 cm 12 cm Glas- Platten / 2400 Module* 4.5 % 5.0 % 4.0 % Gel- PAA PAA PAA lösung (29:1) (29:1) (29:1) & TBE-Puffer TBE-Puffer TBE-Puffer Puffer * Veränderte Elektrophoreseparameter siehe Moduleinstellungen GS Gelprotokolle Für PAA-Gele (29:1) Laufpuffer Fragment- End- 30% Harnstoff 10 x HPLC- TEMED 10% oben / länge konz. PAA TBE Wasser APS unten 500 bp 4.5 % 7.5 ml 18 g 6 ml 23 ml 20 µl 300 µl 1 x TBE 800 bp 5.0 % 8.4 ml 21 g 6 ml 20 ml 20 µl 300 µl 1 x TBE 2500 bp 4.0 % 6.7 ml 21 g 5 ml 21.3 ml 20 µl 300 µl 1 x TBE Ladeprotokolle Für alle Gelsysteme Wasserlade-Protokoll verwenden! Kamm 36 Lanes 48 Lanes 64 Lanes 96 Lanes max. Volumen 1-3 µl µl µl 0.5 µl 1 / 99
22 GS Moduleinstellungen 21 Für PAA-Gele (29:1) Modul (Filterset A oder E) GS Run Für 500 Basen Electrophoresis Voltage 3000 V konstant Collection Time 3 Hours Electrophoresis Current 60 ma Gel Temperature 51 C Electrophoresis Power 200 W Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Modul (Filterset A oder E) GS Run Für 800 Basen Electrophoresis Voltage 2500 V Collection Time 5 Hours Electrophoresis Current 50 ma Gel Temperature 51 C Electrophoresis Power 50 W konstant Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 Modul (Filterset A oder E) GS Run /2400 Für 2500 Basen Electrophoresis Voltage 750 V konstant Collection Time 9 Hours Electrophoresis Current 60 ma Gel Temperature 51 C Electrophoresis Power 200 W Laser Power 40 mw CCD Offset* 250 CCD Gain* 2 * Bei 96 Lanes: CCD Offset = 0 CCD Gain = 4
23 373 Gelsysteme 24/12cm WTR 6 %iges Polyacrylamidgel (60 ml): 30,0 g Harnstoff 12,0 ml 30 %ige Acrylamidlösung 6,0 ml 10 x TBE-Puffer 20,0 ml bidest. Wasser Laufbedingungen : 27 Watt, 14 h 34cm WTR 5 %iges Polyacrylamidgel (60 ml): 30,0 g Harnstoff 10,0 ml 30 %ige Acrylamidlösung 6,0 ml 10 x TBE-Puffer 22,0 ml bidest. Wasser Laufbedingungen : 27 Watt, 15 h 48cm WTR 5 %iges Polyacrylamidgel (60 ml): 30,0 g Harnstoff 10,0 ml 30 %ige Acrylamidlösung 6,0 ml 10 x TBE-Puffer 22,0 ml bidest. Wasser Laufbedingungen : 37 Watt, 18 h 24cm WTR (Basesprinter) 6 %iges Polyacrylamidgel (50 ml): 21,0 g Harnstoff 10,0 ml 30 %ige Acrylamidlösung 6,0 ml 10 x TBE-Puffer mit bidest. Wasser auf 50 ml auffüllen Laufbedingungen : 45 Watt, 8 h 34cm WTR (Basesprinter) 5 %iges Polyacrylamidgel (50 ml): 21,0 g Harnstoff 8,4 ml 30 %ige Acrylamidlösung 6,0 ml 10 x TBE-Puffer mit bidest. Wasser auf 50 ml auffüllen Laufbedingungen : 48 Watt, 10 h 48cm WTR (Basesprinter) 5,5 %iges Polyacrylamidgel (50 ml): 18,0 g Harnstoff 9,2 ml 30 %ige Acrylamidlösung 5,0 ml 10 x TBE-Puffer mit bidest. Wasser auf 50 ml auffüllen Laufbedingungen : 60 Watt, 15 h 22
24 7. Datenauswertung GS-Standards im Überblick HD GS 350 / GS * 233* 238* * * 536 Fragmente: 37, 94, 109, 116, 172, 186, 222*, 233*, 238*, 269, 286*, 361, 470, 490*, 536, 827, 1115, 1181, 1722, 2008, 2162, 2465, 2481, 2860, 4529, 4771, 5099, * Rechten Peak definieren Interne Standards für 310 geeignet: GS 350, HD 400, GS 500 Interne Standards für 37X geeignet: GS 350, HD 400, GS 500, GS 1000, GS 2500 Fragmentlängenbestimmung 310 Zuordnung GS 350 und GS 500 Die Fragmente mit den Längen 250 und 340 dürfen bei der Definition des Längenstandards nicht miteinbezogen werden, da sie unter den gegebenen Bedingungen ein abnormales Laufverhalten zeigen. Diese Fragmente erhalten die Größe = 0. GS 1000 und GS 2500 Diese Standards sind nicht zur Längenberechnung mit denaturierenden Polymerlösungen geeignet, weil die komplementären, markierten Stränge unter diesen Bedingungen ein unterschiedliches Laufverhalten zeigen.
25 7.2 Tips zum Sequence Navigator 24 Der Sequence Navigator ist ein Sequenzvergleichsprogramm für kleinere Sequenzierungsprojekte bis ca. 5 kb. Mit dem Programm kann man Sequenzvergleiche aber keine automatischen Alignments durchführen, d.h. die Sequenzen müssen in der richtigen Reihenfolge und auch in der richtigen Orientierung für das Alignment vorgelegt werden. Beim Arbeiten mit dem Navigator sollte man nach folgendem Schema vorgehen: 1. Sequence Navigator öffnen. 2. Gegebenenfalls neues Layout öffnen (File -> New Layout). 3. Sequenz Files laden (Sequences -> Import Sequence). 4. Alignment durchführen - Sollen 2 Sequenzen miteinander verglichen werden (Align -> Comparative) - Sollen mehr Sequenzen gleichzeitig miteinander verglichen werden, was einem Multiple- Alignment entspricht (Align -> Clustal). Nicht Multiple auswählen, diese Option ist ausschließlich für Aminosäure-Sequenzvergleiche gedacht! - Sollen überlappende Sequenzen verglichen werden, müssen zunächst die Sequenzen in der richtigen Reihenfolge vorgegeben werden, 1te Sequenz mit 2ter und nicht 2te mit 1ter (Align -> Overlap). Funktioniert diese Option nicht, weil der Überlappungsbereich zu groß ist, empfiehlt es sich, die Comparative Option auszuprobieren. 5. Unterschiede zwischen angepaßten Sequenzen anzeigen lassen (Sequences -> Create Shadow(s) -> Compare Two Sequences). 6. Chromatogramme vergleichen und Peakmuster beachten. Zu vergleichende Sequenzfiles durch Anklicken bei gehaltener Shift-Taste im linken Layout-Fenster markieren, und interessanten Sequenzbereich im Hauptlayout-Fenster markieren. Unter (Sequences -> Display Electropherograms) Chromatogramme anzeigen lassen. Sollen mehr als 3 Sequenzchromatogramme gleichzeitig angezeigt werden, kann der Bildausschnitt verkleinert werden, indem man die Alt- oder Option-Taste drückt bevor man Display Electropherograms unter Sequences auswählt. Wenn dann im folgenden Fenster unter Tile: half width ausgewählt wird, kann man bis zu 9 Chromatogramme gleichzeitig betrachten. Weitere Tips: Sequenzen Exportieren Sollen abgeleitete Proteinsequenzen (Sequences -> Create Shadow(s) -> Translate Codons to Amino Acids) oder Consensussequenzen (Sequences -> Create Shadow(s) -> Compute Consensus Sequence) bearbeitet oder exportiert werden, müssen diese Files zunächst aktiviert werden. Dazu wird das File im linken Layout- Fenster markiert und anschließend unter (Sequences -> Freeze Shadows) aktiviert.
SDS Polyacrylamidgelelektrophorese (SDS-PAGE) Mini-Gelträger nach Anleitung zusammenbauen. Saubere Handschuhe tragen!!!
 aktualisiert, 08.03.2011, Wera Roth1 SDS Polyacrylamidgelelektrophorese (SDS-PAGE) Anleitung für Minigele Mini-Gelträger nach Anleitung zusammenbauen. Saubere Handschuhe tragen!!! Glasscheiben ordentlich
aktualisiert, 08.03.2011, Wera Roth1 SDS Polyacrylamidgelelektrophorese (SDS-PAGE) Anleitung für Minigele Mini-Gelträger nach Anleitung zusammenbauen. Saubere Handschuhe tragen!!! Glasscheiben ordentlich
1. Adressen für den Serienversand (Briefe Katalogdruck Werbung/Anfrage ) auswählen. Die Auswahl kann gespeichert werden.
 Der Serienversand Was kann man mit der Maske Serienversand machen? 1. Adressen für den Serienversand (Briefe Katalogdruck Werbung/Anfrage ) auswählen. Die Auswahl kann gespeichert werden. 2. Adressen auswählen,
Der Serienversand Was kann man mit der Maske Serienversand machen? 1. Adressen für den Serienversand (Briefe Katalogdruck Werbung/Anfrage ) auswählen. Die Auswahl kann gespeichert werden. 2. Adressen auswählen,
Plasmidisolierung. Mit Plasmiden können Sie Gene in Organismen einschleusen und so deren Eigenschaften verändern.
 Plasmidisolierung Mit Plasmiden können Sie Gene in Organismen einschleusen und so deren Eigenschaften verändern. Was können Sie lernen? Sie lernen eine ringförmige DNA, ein Plasmid, zu isolieren. Mit diesem
Plasmidisolierung Mit Plasmiden können Sie Gene in Organismen einschleusen und so deren Eigenschaften verändern. Was können Sie lernen? Sie lernen eine ringförmige DNA, ein Plasmid, zu isolieren. Mit diesem
AGROPLUS Buchhaltung. Daten-Server und Sicherheitskopie. Version vom 21.10.2013b
 AGROPLUS Buchhaltung Daten-Server und Sicherheitskopie Version vom 21.10.2013b 3a) Der Daten-Server Modus und der Tresor Der Daten-Server ist eine Betriebsart welche dem Nutzer eine grosse Flexibilität
AGROPLUS Buchhaltung Daten-Server und Sicherheitskopie Version vom 21.10.2013b 3a) Der Daten-Server Modus und der Tresor Der Daten-Server ist eine Betriebsart welche dem Nutzer eine grosse Flexibilität
Handbuch. NAFI Online-Spezial. Kunden- / Datenverwaltung. 1. Auflage. (Stand: 24.09.2014)
 Handbuch NAFI Online-Spezial 1. Auflage (Stand: 24.09.2014) Copyright 2016 by NAFI GmbH Unerlaubte Vervielfältigungen sind untersagt! Inhaltsangabe Einleitung... 3 Kundenauswahl... 3 Kunde hinzufügen...
Handbuch NAFI Online-Spezial 1. Auflage (Stand: 24.09.2014) Copyright 2016 by NAFI GmbH Unerlaubte Vervielfältigungen sind untersagt! Inhaltsangabe Einleitung... 3 Kundenauswahl... 3 Kunde hinzufügen...
UMSTELLUNG DER RÖNTGEN-SCHNITTSTELLE DÜRR-DBSWIN AUF DÜRR-VDDS
 UMSTELLUNG DER RÖNTGEN-SCHNITTSTELLE DÜRR-DBSWIN AUF DÜRR-VDDS Pionier der Zahnarzt-Software. Seit 1986. 1 Seite 1/5 Diese Anleitung soll Ihnen dabei helfen, eine bestehende DBSWIN-Anbindung über den Patientendatenexport
UMSTELLUNG DER RÖNTGEN-SCHNITTSTELLE DÜRR-DBSWIN AUF DÜRR-VDDS Pionier der Zahnarzt-Software. Seit 1986. 1 Seite 1/5 Diese Anleitung soll Ihnen dabei helfen, eine bestehende DBSWIN-Anbindung über den Patientendatenexport
Elexis-BlueEvidence-Connector
 Elexis-BlueEvidence-Connector Gerry Weirich 26. Oktober 2012 1 Einführung Dieses Plugin dient dazu, den Status Hausarztpatient zwischen der BlueEvidence- Anwendung und Elexis abzugleichen. Das Plugin markiert
Elexis-BlueEvidence-Connector Gerry Weirich 26. Oktober 2012 1 Einführung Dieses Plugin dient dazu, den Status Hausarztpatient zwischen der BlueEvidence- Anwendung und Elexis abzugleichen. Das Plugin markiert
ecall sms & fax-portal
 ecall sms & fax-portal Beschreibung des Imports und Exports von Adressen Dateiname Beschreibung_-_eCall_Import_und_Export_von_Adressen_2015.10.20 Version 1.1 Datum 20.10.2015 Dolphin Systems AG Informieren
ecall sms & fax-portal Beschreibung des Imports und Exports von Adressen Dateiname Beschreibung_-_eCall_Import_und_Export_von_Adressen_2015.10.20 Version 1.1 Datum 20.10.2015 Dolphin Systems AG Informieren
Pfötchenhoffung e.v. Tier Manager
 Pfötchenhoffung e.v. Tier Manager A.Ennenbach 01.08.2015 Tier Manager Inhalt Administrationsbereich Tiere auflisten & suchen Tier hinzufügen Tier bearbeiten (Status ändern, etc.) Administrationsbereich
Pfötchenhoffung e.v. Tier Manager A.Ennenbach 01.08.2015 Tier Manager Inhalt Administrationsbereich Tiere auflisten & suchen Tier hinzufügen Tier bearbeiten (Status ändern, etc.) Administrationsbereich
2. Im Admin Bereich drücken Sie bitte auf den roten Button Webseite bearbeiten, sodass Sie in den Bearbeitungsbereich Ihrer Homepage gelangen.
 Bildergalerie einfügen Wenn Sie eine Vielzahl an Bildern zu einem Thema auf Ihre Homepage stellen möchten, steht Ihnen bei Schmetterling Quadra das Modul Bildergalerie zur Verfügung. Ihre Kunden können
Bildergalerie einfügen Wenn Sie eine Vielzahl an Bildern zu einem Thema auf Ihre Homepage stellen möchten, steht Ihnen bei Schmetterling Quadra das Modul Bildergalerie zur Verfügung. Ihre Kunden können
Erstellen einer Collage. Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu])
![Erstellen einer Collage. Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu]) Erstellen einer Collage. Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu])](/thumbs/29/13170670.jpg) 3.7 Erstellen einer Collage Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu]) Dann Größe des Dokuments festlegen beispielsweise A4 (weitere
3.7 Erstellen einer Collage Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu]) Dann Größe des Dokuments festlegen beispielsweise A4 (weitere
Text Formatierung in Excel
 Text Formatierung in Excel Das Aussehen des Textes einer oder mehrerer Zellen kann in Excel über verschiedene Knöpfe beeinflusst werden. Dazu zuerst die betroffenen Zelle(n) anwählen und danach den entsprechenden
Text Formatierung in Excel Das Aussehen des Textes einer oder mehrerer Zellen kann in Excel über verschiedene Knöpfe beeinflusst werden. Dazu zuerst die betroffenen Zelle(n) anwählen und danach den entsprechenden
V o r w o r t. A n l e i t u n g
 V o r w o r t In diesem Dokument wird erklärt, wie man mit Hilfe des Logitech Profiler Tastaturbefehle, welche von einem PC Spiel benutzt werden, auf die Tasten eines Eingabegeräts der Firma Logitech belegt.
V o r w o r t In diesem Dokument wird erklärt, wie man mit Hilfe des Logitech Profiler Tastaturbefehle, welche von einem PC Spiel benutzt werden, auf die Tasten eines Eingabegeräts der Firma Logitech belegt.
FH-SY Chapter 2.4 - Version 3 - FH-SY.NET - FAQ -
 FH-SY Chapter 2.4 - Version 3 - FH-SY.NET - FAQ - Version vom 02.02.2010 Inhaltsverzeichnis 1. KANN ICH BEI EINER EIGENEN LEKTION NACHTRÄGLICH NOCH NEUE LERNINHALTE ( WAS WURDE BEHANDELT? ) EINFÜGEN?...
FH-SY Chapter 2.4 - Version 3 - FH-SY.NET - FAQ - Version vom 02.02.2010 Inhaltsverzeichnis 1. KANN ICH BEI EINER EIGENEN LEKTION NACHTRÄGLICH NOCH NEUE LERNINHALTE ( WAS WURDE BEHANDELT? ) EINFÜGEN?...
Handbuch für Redakteure
 Handbuch für Redakteure Erste Schritte... 1 Artikel erstellen... 2 Artikelinhalt bearbeiten... 3 Artikel bearbeiten... 3 Grunddaten ändern... 5 Weitere Artikeleigenschaften... 5 Der WYSIWYG-Editor... 6
Handbuch für Redakteure Erste Schritte... 1 Artikel erstellen... 2 Artikelinhalt bearbeiten... 3 Artikel bearbeiten... 3 Grunddaten ändern... 5 Weitere Artikeleigenschaften... 5 Der WYSIWYG-Editor... 6
1 Einleitung. Lernziele. automatische Antworten bei Abwesenheit senden. Einstellungen für automatische Antworten Lerndauer. 4 Minuten.
 1 Einleitung Lernziele automatische Antworten bei Abwesenheit senden Einstellungen für automatische Antworten Lerndauer 4 Minuten Seite 1 von 18 2 Antworten bei Abwesenheit senden» Outlook kann während
1 Einleitung Lernziele automatische Antworten bei Abwesenheit senden Einstellungen für automatische Antworten Lerndauer 4 Minuten Seite 1 von 18 2 Antworten bei Abwesenheit senden» Outlook kann während
Stift-Karussell in M-Plot einrichten
 Einsteiger Fortgeschrittene Profis markus.meinl@m-quest.ch Version 1.0 Voraussetzungen für diesen Workshop Die M-Quest Suite 2005 oder höher ist auf einem Rechner installiert Das Produkt M-Plot Suite ist
Einsteiger Fortgeschrittene Profis markus.meinl@m-quest.ch Version 1.0 Voraussetzungen für diesen Workshop Die M-Quest Suite 2005 oder höher ist auf einem Rechner installiert Das Produkt M-Plot Suite ist
Mandant in den einzelnen Anwendungen löschen
 Mandant in den einzelnen Anwendungen löschen Bereich: ALLGEMEIN - Info für Anwender Nr. 6056 Inhaltsverzeichnis 1. Allgemein 2. FIBU/ANLAG/ZAHLUNG/BILANZ/LOHN/BELEGTRANSFER 3. DMS 4. STEUERN 5. FRISTEN
Mandant in den einzelnen Anwendungen löschen Bereich: ALLGEMEIN - Info für Anwender Nr. 6056 Inhaltsverzeichnis 1. Allgemein 2. FIBU/ANLAG/ZAHLUNG/BILANZ/LOHN/BELEGTRANSFER 3. DMS 4. STEUERN 5. FRISTEN
Erstellen von x-y-diagrammen in OpenOffice.calc
 Erstellen von x-y-diagrammen in OpenOffice.calc In dieser kleinen Anleitung geht es nur darum, aus einer bestehenden Tabelle ein x-y-diagramm zu erzeugen. D.h. es müssen in der Tabelle mindestens zwei
Erstellen von x-y-diagrammen in OpenOffice.calc In dieser kleinen Anleitung geht es nur darum, aus einer bestehenden Tabelle ein x-y-diagramm zu erzeugen. D.h. es müssen in der Tabelle mindestens zwei
Downloadfehler in DEHSt-VPSMail. Workaround zum Umgang mit einem Downloadfehler
 Downloadfehler in DEHSt-VPSMail Workaround zum Umgang mit einem Downloadfehler Downloadfehler bremen online services GmbH & Co. KG Seite 2 Inhaltsverzeichnis Vorwort...3 1 Fehlermeldung...4 2 Fehlerbeseitigung...5
Downloadfehler in DEHSt-VPSMail Workaround zum Umgang mit einem Downloadfehler Downloadfehler bremen online services GmbH & Co. KG Seite 2 Inhaltsverzeichnis Vorwort...3 1 Fehlermeldung...4 2 Fehlerbeseitigung...5
Lieferschein Dorfstrasse 143 CH - 8802 Kilchberg Telefon 01 / 716 10 00 Telefax 01 / 716 10 05 info@hp-engineering.com www.hp-engineering.
 Lieferschein Lieferscheine Seite 1 Lieferscheine Seite 2 Inhaltsverzeichnis 1. STARTEN DER LIEFERSCHEINE 4 2. ARBEITEN MIT DEN LIEFERSCHEINEN 4 2.1 ERFASSEN EINES NEUEN LIEFERSCHEINS 5 2.1.1 TEXTFELD FÜR
Lieferschein Lieferscheine Seite 1 Lieferscheine Seite 2 Inhaltsverzeichnis 1. STARTEN DER LIEFERSCHEINE 4 2. ARBEITEN MIT DEN LIEFERSCHEINEN 4 2.1 ERFASSEN EINES NEUEN LIEFERSCHEINS 5 2.1.1 TEXTFELD FÜR
AutoCAD 2007 - Dienstprogramm zur Lizenzübertragung
 AutoCAD 2007 - Dienstprogramm zur Lizenzübertragung Problem: Um AutoCAD abwechselnd auf mehreren Rechnern einsetzen zu können konnte man bis AutoCAD 2000 einfach den Dongle umstecken. Seit AutoCAD 2000i
AutoCAD 2007 - Dienstprogramm zur Lizenzübertragung Problem: Um AutoCAD abwechselnd auf mehreren Rechnern einsetzen zu können konnte man bis AutoCAD 2000 einfach den Dongle umstecken. Seit AutoCAD 2000i
Handbuch Fischertechnik-Einzelteiltabelle V3.7.3
 Handbuch Fischertechnik-Einzelteiltabelle V3.7.3 von Markus Mack Stand: Samstag, 17. April 2004 Inhaltsverzeichnis 1. Systemvorraussetzungen...3 2. Installation und Start...3 3. Anpassen der Tabelle...3
Handbuch Fischertechnik-Einzelteiltabelle V3.7.3 von Markus Mack Stand: Samstag, 17. April 2004 Inhaltsverzeichnis 1. Systemvorraussetzungen...3 2. Installation und Start...3 3. Anpassen der Tabelle...3
Praktikum: Nachweis einer gentechnischen Veränderung in Lebensmitteln durch PCR
 Praktikum: Nachweis einer gentechnischen Veränderung in Lebensmitteln durch PCR Vorbereitung Lehrer Für jede Arbeitsgruppe werden 550 µl InstaGene-Matrix aliquotiert! Das Tube wird mit IG beschriftet!
Praktikum: Nachweis einer gentechnischen Veränderung in Lebensmitteln durch PCR Vorbereitung Lehrer Für jede Arbeitsgruppe werden 550 µl InstaGene-Matrix aliquotiert! Das Tube wird mit IG beschriftet!
Bedienungsanleitung Anlassteilnehmer (Vereinslisten)
 Bedienungsanleitung Anlassteilnehmer Dieses Programm ist speziell für Vereine entworfen. Es ist lizenzfrei verwendbar und gratis. Das Programm ist mit Excel 2010 erstellt worden und enthält VBA Programmierungen,
Bedienungsanleitung Anlassteilnehmer Dieses Programm ist speziell für Vereine entworfen. Es ist lizenzfrei verwendbar und gratis. Das Programm ist mit Excel 2010 erstellt worden und enthält VBA Programmierungen,
Handbuch für Redakteure
 Handbuch für Redakteure Erste Schritte... 1 Artikel erstellen... 2 Artikelinhalt bearbeiten... 3 Artikel bearbeiten... 3 Trennen der Druck- und der Online-Version.. 4 Grunddaten ändern... 5 Weitere Artikel-eigenschaften...
Handbuch für Redakteure Erste Schritte... 1 Artikel erstellen... 2 Artikelinhalt bearbeiten... 3 Artikel bearbeiten... 3 Trennen der Druck- und der Online-Version.. 4 Grunddaten ändern... 5 Weitere Artikel-eigenschaften...
AutoTexte und AutoKorrektur unter Outlook verwenden
 AutoTexte und AutoKorrektur unter Outlook verwenden Die Hilfsmittel "AutoKorrektur" und "AutoTexte", die schon unter Microsoft Word das Arbeiten erleichtern, sind natürlich auch unter Outlook verfügbar.
AutoTexte und AutoKorrektur unter Outlook verwenden Die Hilfsmittel "AutoKorrektur" und "AutoTexte", die schon unter Microsoft Word das Arbeiten erleichtern, sind natürlich auch unter Outlook verfügbar.
Färbung mit AzurGel-K
 Arbeitsanleitung zur Färbung mit AzurGel-K für Gele im Format 10 x 10 x 0,1 cm Kat. Nr.: GF 10002 Ringstr. 4 64401 Gross-Bieberau Tel. ++49-6162-809840 Fax ++49-6162-8098420 www.anamed-gele.com Grundlage
Arbeitsanleitung zur Färbung mit AzurGel-K für Gele im Format 10 x 10 x 0,1 cm Kat. Nr.: GF 10002 Ringstr. 4 64401 Gross-Bieberau Tel. ++49-6162-809840 Fax ++49-6162-8098420 www.anamed-gele.com Grundlage
Handbuch B4000+ Preset Manager
 Handbuch B4000+ Preset Manager B4000+ authentic organ modeller Version 0.6 FERROFISH advanced audio applications Einleitung Mit der Software B4000+ Preset Manager können Sie Ihre in der B4000+ erstellten
Handbuch B4000+ Preset Manager B4000+ authentic organ modeller Version 0.6 FERROFISH advanced audio applications Einleitung Mit der Software B4000+ Preset Manager können Sie Ihre in der B4000+ erstellten
Zwischenablage (Bilder, Texte,...)
 Zwischenablage was ist das? Informationen über. die Bedeutung der Windows-Zwischenablage Kopieren und Einfügen mit der Zwischenablage Vermeiden von Fehlern beim Arbeiten mit der Zwischenablage Bei diesen
Zwischenablage was ist das? Informationen über. die Bedeutung der Windows-Zwischenablage Kopieren und Einfügen mit der Zwischenablage Vermeiden von Fehlern beim Arbeiten mit der Zwischenablage Bei diesen
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren
 Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
Sequenzierung. Sequenzierung. DNA in den letzten 50 Jahren. Warum Sequenzierung
 Sequenzierung von Thomas Grunwald Abteilung für Med. und Mol. Virologie Sequenzierung Hintergrund Allgemeiner Überblick Funktionsweise der Sequenzierung Sequenzierungsprotokoll Auswertung der Daten Probleme
Sequenzierung von Thomas Grunwald Abteilung für Med. und Mol. Virologie Sequenzierung Hintergrund Allgemeiner Überblick Funktionsweise der Sequenzierung Sequenzierungsprotokoll Auswertung der Daten Probleme
Teaser-Bilder erstellen mit GIMP. Bildbearbeitung mit GIMP 1
 Teaser-Bilder erstellen mit GIMP 08.08.2014 Bildbearbeitung mit GIMP 1 Auf den folgenden Seiten werden die wichtigsten Funktionen von GIMP gezeigt, welche zur Erstellung von Bildern für die Verwendung
Teaser-Bilder erstellen mit GIMP 08.08.2014 Bildbearbeitung mit GIMP 1 Auf den folgenden Seiten werden die wichtigsten Funktionen von GIMP gezeigt, welche zur Erstellung von Bildern für die Verwendung
Handbuch für Redakteure
 Handbuch für Redakteure Erste Schritte... 1 Artikel erstellen... 2 Artikelinhalt bearbeiten... 3 Artikel bearbeiten... 3 Grunddaten ändern... 5 Weitere Artikeleigenschaften... 5 Der WYSIWYG-Editor... 6
Handbuch für Redakteure Erste Schritte... 1 Artikel erstellen... 2 Artikelinhalt bearbeiten... 3 Artikel bearbeiten... 3 Grunddaten ändern... 5 Weitere Artikeleigenschaften... 5 Der WYSIWYG-Editor... 6
Enigmail Konfiguration
 Enigmail Konfiguration 11.06.2006 Steffen.Teubner@Arcor.de Enigmail ist in der Grundkonfiguration so eingestellt, dass alles funktioniert ohne weitere Einstellungen vornehmen zu müssen. Für alle, die es
Enigmail Konfiguration 11.06.2006 Steffen.Teubner@Arcor.de Enigmail ist in der Grundkonfiguration so eingestellt, dass alles funktioniert ohne weitere Einstellungen vornehmen zu müssen. Für alle, die es
Datenaufbereitung in SPSS. Daten zusammenfügen
 Daten zusammenfügen I. Fälle hinzufügen Diese Schritte müssen Sie unternehmen, wenn die Daten in unterschiedlichen Dateien sind; wenn also die Daten von unterschiedlichen Personen in unterschiedlichen
Daten zusammenfügen I. Fälle hinzufügen Diese Schritte müssen Sie unternehmen, wenn die Daten in unterschiedlichen Dateien sind; wenn also die Daten von unterschiedlichen Personen in unterschiedlichen
Historical Viewer. zu ETC5000 Benutzerhandbuch 312/15
 Historical Viewer zu ETC5000 Benutzerhandbuch 312/15 Inhaltsverzeichnis 1 Allgemeine Hinweise... 3 1.1 Dokumentation...3 2 Installation... 3 3 Exportieren der Logdatei aus dem ETC 5000... 3 4 Anlegen eines
Historical Viewer zu ETC5000 Benutzerhandbuch 312/15 Inhaltsverzeichnis 1 Allgemeine Hinweise... 3 1.1 Dokumentation...3 2 Installation... 3 3 Exportieren der Logdatei aus dem ETC 5000... 3 4 Anlegen eines
Protokoll zur Übung Ölanalyse
 Protokoll zur Übung Ölanalyse im Rahmen des Praktikums Betreuender Assistent Univ.Ass. Dipl.-Ing. Martin Schwentenwein Verfasser des Protokolls: Daniel Bomze 0726183 1 Theoretischer Hintergrund 1.1 Aufgabenstellung
Protokoll zur Übung Ölanalyse im Rahmen des Praktikums Betreuender Assistent Univ.Ass. Dipl.-Ing. Martin Schwentenwein Verfasser des Protokolls: Daniel Bomze 0726183 1 Theoretischer Hintergrund 1.1 Aufgabenstellung
3. Die tägliche E-Mail-Flut effizient verwalten
 3. Es ist wie im normalen Leben: Wenn man etwas vernünftig einsortiert, findet man es auch rasch wieder. In Outlook ist das ähnlich. Denn mit der Zeit sammeln sich sehr viele E-Mails an. Wer da keine logische
3. Es ist wie im normalen Leben: Wenn man etwas vernünftig einsortiert, findet man es auch rasch wieder. In Outlook ist das ähnlich. Denn mit der Zeit sammeln sich sehr viele E-Mails an. Wer da keine logische
Wo möchten Sie die MIZ-Dokumente (aufbereitete Medikamentenlisten) einsehen?
 Anleitung für Evident Seite 1 Anleitung für Evident-Anwender: Einbinden der MIZ-Dokumente in Evident. Wo möchten Sie die MIZ-Dokumente (aufbereitete Medikamentenlisten) einsehen? Zunächst müssen Sie entscheiden,
Anleitung für Evident Seite 1 Anleitung für Evident-Anwender: Einbinden der MIZ-Dokumente in Evident. Wo möchten Sie die MIZ-Dokumente (aufbereitete Medikamentenlisten) einsehen? Zunächst müssen Sie entscheiden,
Menü Macro. WinIBW2-Macros unter Windows7? Macros aufnehmen
 Menü Macro WinIBW2-Macros unter Windows7?... 1 Macros aufnehmen... 1 Menübefehle und Schaltflächen in Macros verwenden... 4 Macros bearbeiten... 4 Macros löschen... 5 Macro-Dateien... 5 Macros importieren...
Menü Macro WinIBW2-Macros unter Windows7?... 1 Macros aufnehmen... 1 Menübefehle und Schaltflächen in Macros verwenden... 4 Macros bearbeiten... 4 Macros löschen... 5 Macro-Dateien... 5 Macros importieren...
P&P Software - Adressexport an Outlook 05/29/16 14:44:26
 Adressexport an Outlook Wozu? Aus EASY können viele Daten im Excelformat ausgegeben werden. Diese Funktion kann zum Beispiel zum Export von Lieferantenadressen an Outlook genutzt werden. Hinweis Wir können
Adressexport an Outlook Wozu? Aus EASY können viele Daten im Excelformat ausgegeben werden. Diese Funktion kann zum Beispiel zum Export von Lieferantenadressen an Outlook genutzt werden. Hinweis Wir können
DAS HALLENBUCHUNGSPROGRAMM EBUSY
 DAS HALLENBUCHUNGSPROGRAMM EBUSY Das Buchungssystem kann von jedem Ort, jedem Computer mit Internet Anschluss und sogar vom Smartphone erreicht werden. Entweder rufen Sie die Seite direkt über die Internet
DAS HALLENBUCHUNGSPROGRAMM EBUSY Das Buchungssystem kann von jedem Ort, jedem Computer mit Internet Anschluss und sogar vom Smartphone erreicht werden. Entweder rufen Sie die Seite direkt über die Internet
Gebrauchsanleitung ChromoQuant QF-PCR Kit P/N 412.001-48
 Gebrauchsanleitung ChromoQuant QF-PCR Kit P/N 412.001-48 ChromoQuant Das ChromoQuant in vitro Diagnose-Set QF-PCR 1 zur Analyse von häufigen chromosomalen Erkrankungen der Chromosomen 13, 18 und 21 412.001-48u
Gebrauchsanleitung ChromoQuant QF-PCR Kit P/N 412.001-48 ChromoQuant Das ChromoQuant in vitro Diagnose-Set QF-PCR 1 zur Analyse von häufigen chromosomalen Erkrankungen der Chromosomen 13, 18 und 21 412.001-48u
Windows 7 Winbuilder USB Stick
 Windows 7 Winbuilder USB Stick Benötigt wird das Programm: Winbuilder: http://www.mediafire.com/?qqch6hrqpbem8ha Windows 7 DVD Der Download wird in Form einer gepackten Datei (7z) angeboten. Extrahieren
Windows 7 Winbuilder USB Stick Benötigt wird das Programm: Winbuilder: http://www.mediafire.com/?qqch6hrqpbem8ha Windows 7 DVD Der Download wird in Form einer gepackten Datei (7z) angeboten. Extrahieren
Anwendertreffen 20./21. Juni
 Anwendertreffen Verbindungsmittelachsen VBA Allgemein Die Verbindungsmittelachsen werden nun langsam erwachsen. Nach zwei Jahren Einführungszeit haben wir bereits viele Rückmeldungen mit Ergänzungswünschen
Anwendertreffen Verbindungsmittelachsen VBA Allgemein Die Verbindungsmittelachsen werden nun langsam erwachsen. Nach zwei Jahren Einführungszeit haben wir bereits viele Rückmeldungen mit Ergänzungswünschen
Anleitung zum LPI ATP Portal www.lpi-training.eu
 Anleitung zum LPI ATP Portal www.lpi-training.eu Version 1.0 vom 01.09.2013 Beschreibung des Anmeldevorgangs und Erklärung der einzelnen Menüpunkte. Anmeldevorgang: 1. Gehen Sie auf die Seite http://www.lpi-training.eu/.
Anleitung zum LPI ATP Portal www.lpi-training.eu Version 1.0 vom 01.09.2013 Beschreibung des Anmeldevorgangs und Erklärung der einzelnen Menüpunkte. Anmeldevorgang: 1. Gehen Sie auf die Seite http://www.lpi-training.eu/.
Handbuch ECDL 2003 Modul 2: Computermanagement und Dateiverwaltung Der Task-Manager
 Handbuch ECDL 2003 Modul 2: Computermanagement und Dateiverwaltung Der Task-Manager Dateiname: ecdl2_03_05_documentation Speicherdatum: 22.11.2004 ECDL 2003 Modul 2 Computermanagement und Dateiverwaltung
Handbuch ECDL 2003 Modul 2: Computermanagement und Dateiverwaltung Der Task-Manager Dateiname: ecdl2_03_05_documentation Speicherdatum: 22.11.2004 ECDL 2003 Modul 2 Computermanagement und Dateiverwaltung
Anleitung zur Erstellung einer Gefährdungsbeurteilung
 Anleitung zur Erstellung einer Gefährdungsbeurteilung 1 Vorbereiten der Gefährdungsbeurteilung 1.1 Richten Sie mit Hilfe des Windows-Explorers (oder des Windows-Arbeitsplatzes) einen neuen Ordner ein,
Anleitung zur Erstellung einer Gefährdungsbeurteilung 1 Vorbereiten der Gefährdungsbeurteilung 1.1 Richten Sie mit Hilfe des Windows-Explorers (oder des Windows-Arbeitsplatzes) einen neuen Ordner ein,
Wie man Registrationen und Styles von Style/Registration Floppy Disketten auf die TYROS-Festplatte kopieren kann.
 Wie man Registrationen und Styles von Style/Registration Floppy Disketten auf die TYROS-Festplatte kopieren kann. Einleitung Es kommt vor, dass im Handel Disketten angeboten werden, die Styles und Registrationen
Wie man Registrationen und Styles von Style/Registration Floppy Disketten auf die TYROS-Festplatte kopieren kann. Einleitung Es kommt vor, dass im Handel Disketten angeboten werden, die Styles und Registrationen
In der Combobox der Art der Übermittlung (steht derzeit auf 0 für keine Definition), können Sie folgende Einstellungen vornehmen:
 1. Firmendaten: Klicken Sie auf den Bereich Firma oder gehen Sie über das Menü Bearbeiten Firma Firmendaten in die Firmenanlage und über die Schaltfläche weiter > auf die 2. Seite um den nachfolgenden
1. Firmendaten: Klicken Sie auf den Bereich Firma oder gehen Sie über das Menü Bearbeiten Firma Firmendaten in die Firmenanlage und über die Schaltfläche weiter > auf die 2. Seite um den nachfolgenden
2. Im Admin Bereich drücken Sie bitte auf den roten Button Webseite bearbeiten, sodass Sie in den Bearbeitungsbereich Ihrer Homepage gelangen.
 Deeplink einfügen Zur Angebotserstellung über Ihre Buchungsmaschine können Sie mit Schmetterling Quadra Deeplinks durch einen speziellen Deeplink Generator auf Ihre Homepage einfügen. Abhängig von Ihrer
Deeplink einfügen Zur Angebotserstellung über Ihre Buchungsmaschine können Sie mit Schmetterling Quadra Deeplinks durch einen speziellen Deeplink Generator auf Ihre Homepage einfügen. Abhängig von Ihrer
Stundenerfassung Version 1.8
 Stundenerfassung Version 1.8 Anleitung Überstunden Ein Modul der Plusversion 2008 netcadservice GmbH netcadservice GmbH Augustinerstraße 3 D-83395 Freilassing Dieses Programm ist urheberrechtlich geschützt.
Stundenerfassung Version 1.8 Anleitung Überstunden Ein Modul der Plusversion 2008 netcadservice GmbH netcadservice GmbH Augustinerstraße 3 D-83395 Freilassing Dieses Programm ist urheberrechtlich geschützt.
Leica 3D Disto Veranda und Wintergarten
 Leica 3D Disto Veranda und Wintergarten Worauf kommt es an? Was ist zu messen? 1) Position der Veranda 2) Höhe und Breite an der Fassade 3) Länge 4) Unebenheiten an der Fassade 5) Zustand des Untergrunds
Leica 3D Disto Veranda und Wintergarten Worauf kommt es an? Was ist zu messen? 1) Position der Veranda 2) Höhe und Breite an der Fassade 3) Länge 4) Unebenheiten an der Fassade 5) Zustand des Untergrunds
Anleitung über den Umgang mit Schildern
 Anleitung über den Umgang mit Schildern -Vorwort -Wo bekommt man Schilder? -Wo und wie speichert man die Schilder? -Wie füge ich die Schilder in meinen Track ein? -Welche Bauteile kann man noch für Schilder
Anleitung über den Umgang mit Schildern -Vorwort -Wo bekommt man Schilder? -Wo und wie speichert man die Schilder? -Wie füge ich die Schilder in meinen Track ein? -Welche Bauteile kann man noch für Schilder
Um eine Person in Magnolia zu erfassen, gehen Sie wie folgt vor:
 Personendaten verwalten mit Magnolia Sie können ganz einfach und schnell alle Personendaten, die Sie auf Ihrer Webseite publizieren möchten, mit Magnolia verwalten. In der Applikation Adressbuch können
Personendaten verwalten mit Magnolia Sie können ganz einfach und schnell alle Personendaten, die Sie auf Ihrer Webseite publizieren möchten, mit Magnolia verwalten. In der Applikation Adressbuch können
Die Textvorlagen in Microsoft WORD und LibreOffice Writer
 Die Textvorlagen in Microsoft WORD und LibreOffice Writer Liebe Teilnehmer(-innen) am Landeswettbewerb Deutsche Sprache und Literatur Baden- Württemberg, Diese Anleitung soll Ihnen helfen Ihren Wettbewerbsbeitrag
Die Textvorlagen in Microsoft WORD und LibreOffice Writer Liebe Teilnehmer(-innen) am Landeswettbewerb Deutsche Sprache und Literatur Baden- Württemberg, Diese Anleitung soll Ihnen helfen Ihren Wettbewerbsbeitrag
PCR (polymerase chain reaction)
 PCR (polymerase chain reaction) Ist ein in vitro Verfahren zur selektiven Anreicherung von Nukleinsäure-Bereichen definierter Länge und definierter Sequenz aus einem Gemisch von Nukleinsäure-Molekülen
PCR (polymerase chain reaction) Ist ein in vitro Verfahren zur selektiven Anreicherung von Nukleinsäure-Bereichen definierter Länge und definierter Sequenz aus einem Gemisch von Nukleinsäure-Molekülen
email-passwort ändern
 email-passwort ändern Bei der Einrichtung von email-konten durch einen EDV-Kollegen wird zunächst ein Standard- Passwort verwendet. Aus Gründen des Datenschutzes und der Datensicherheit wird dringend angeraten,
email-passwort ändern Bei der Einrichtung von email-konten durch einen EDV-Kollegen wird zunächst ein Standard- Passwort verwendet. Aus Gründen des Datenschutzes und der Datensicherheit wird dringend angeraten,
Flyer, Sharepics usw. mit LibreOffice oder OpenOffice erstellen
 Flyer, Sharepics usw. mit LibreOffice oder OpenOffice erstellen Wir wollen, dass ihr einfach für eure Ideen und Vorschläge werben könnt. Egal ob in ausgedruckten Flyern, oder in sozialen Netzwerken und
Flyer, Sharepics usw. mit LibreOffice oder OpenOffice erstellen Wir wollen, dass ihr einfach für eure Ideen und Vorschläge werben könnt. Egal ob in ausgedruckten Flyern, oder in sozialen Netzwerken und
Menü auf zwei Module verteilt (Joomla 3.4.0)
 Menü auf zwei Module verteilt (Joomla 3.4.0) Oft wird bei Joomla das Menü in einem Modul dargestellt, wenn Sie aber z.b. ein horizontales Hauptmenü mit einem vertikalen Untermenü machen möchten, dann finden
Menü auf zwei Module verteilt (Joomla 3.4.0) Oft wird bei Joomla das Menü in einem Modul dargestellt, wenn Sie aber z.b. ein horizontales Hauptmenü mit einem vertikalen Untermenü machen möchten, dann finden
Anleitung zum GUI Version 2.x
 GUI (Graphical User Interface) für PC-DMIS V3.x 4.x Seite 1 von 7 Anleitung zum GUI Version 2.x Einsatzziel des Programms: Über ein grafisches Menü PC-DMIS Messprogramme starten. Empfehlenswert für Anwender
GUI (Graphical User Interface) für PC-DMIS V3.x 4.x Seite 1 von 7 Anleitung zum GUI Version 2.x Einsatzziel des Programms: Über ein grafisches Menü PC-DMIS Messprogramme starten. Empfehlenswert für Anwender
iphone- und ipad-praxis: Kalender optimal synchronisieren
 42 iphone- und ipad-praxis: Kalender optimal synchronisieren Die Synchronisierung von ios mit anderen Kalendern ist eine elementare Funktion. Die Standard-App bildet eine gute Basis, für eine optimale
42 iphone- und ipad-praxis: Kalender optimal synchronisieren Die Synchronisierung von ios mit anderen Kalendern ist eine elementare Funktion. Die Standard-App bildet eine gute Basis, für eine optimale
Skript zum Praktikum Instrumentelle Analytik der Universität Heidelberg im 4. Fachsemester Pharmazie
 HPLC 1 Skript zum Praktikum Instrumentelle Analytik der Universität Heidelberg im 4. Fachsemester Pharmazie 1. Aufgabe In diesem Versuch werden die Retentionszeiten der Substanzen Acetylsalicylsäure (ASS)
HPLC 1 Skript zum Praktikum Instrumentelle Analytik der Universität Heidelberg im 4. Fachsemester Pharmazie 1. Aufgabe In diesem Versuch werden die Retentionszeiten der Substanzen Acetylsalicylsäure (ASS)
Arbeiten mit UMLed und Delphi
 Arbeiten mit UMLed und Delphi Diese Anleitung soll zeigen, wie man Klassen mit dem UML ( Unified Modeling Language ) Editor UMLed erstellt, in Delphi exportiert und dort so einbindet, dass diese (bis auf
Arbeiten mit UMLed und Delphi Diese Anleitung soll zeigen, wie man Klassen mit dem UML ( Unified Modeling Language ) Editor UMLed erstellt, in Delphi exportiert und dort so einbindet, dass diese (bis auf
Shopz Zugang Neuanmeldung
 Web Sales, IBM Deutschland Shopz Zugang Neuanmeldung IBM ID einrichten Shopz-Zugang anmelden Shopz Login User ID Management IBM ID und Shopz Zugang im Überblick Überblick ibm.com/software/de/websolutions/
Web Sales, IBM Deutschland Shopz Zugang Neuanmeldung IBM ID einrichten Shopz-Zugang anmelden Shopz Login User ID Management IBM ID und Shopz Zugang im Überblick Überblick ibm.com/software/de/websolutions/
Stundenerfassung Version 1.8 Anleitung Arbeiten mit Replikaten
 Stundenerfassung Version 1.8 Anleitung Arbeiten mit Replikaten 2008 netcadservice GmbH netcadservice GmbH Augustinerstraße 3 D-83395 Freilassing Dieses Programm ist urheberrechtlich geschützt. Eine Weitergabe
Stundenerfassung Version 1.8 Anleitung Arbeiten mit Replikaten 2008 netcadservice GmbH netcadservice GmbH Augustinerstraße 3 D-83395 Freilassing Dieses Programm ist urheberrechtlich geschützt. Eine Weitergabe
Der digitale. Workflow Workflow. [PDF-Erstellung aus CorelDraw für Windows]
![Der digitale. Workflow Workflow. [PDF-Erstellung aus CorelDraw für Windows] Der digitale. Workflow Workflow. [PDF-Erstellung aus CorelDraw für Windows]](/thumbs/28/12880006.jpg) Der digitale Workflow Workflow [PDF-Erstellung aus CorelDraw für Windows] 2 Der Digitale Workflow schnell und wirtschaftlich bei höchster Qualität Inhalt Der digitale Workflow gibt die optimale Voraussetzung
Der digitale Workflow Workflow [PDF-Erstellung aus CorelDraw für Windows] 2 Der Digitale Workflow schnell und wirtschaftlich bei höchster Qualität Inhalt Der digitale Workflow gibt die optimale Voraussetzung
Terminabgleich mit Mobiltelefonen
 Terminabgleich mit Mobiltelefonen Sie können Termine- und Aufgaben aus unserem Kalender, sowie die Adressdaten aus dem Hauptprogramm mit Ihrem Mobiltelefon abgleichen. MS Outlook dient dabei als Schnittstelle
Terminabgleich mit Mobiltelefonen Sie können Termine- und Aufgaben aus unserem Kalender, sowie die Adressdaten aus dem Hauptprogramm mit Ihrem Mobiltelefon abgleichen. MS Outlook dient dabei als Schnittstelle
Mit der Maus im Menü links auf den Menüpunkt 'Seiten' gehen und auf 'Erstellen klicken.
 Seite erstellen Mit der Maus im Menü links auf den Menüpunkt 'Seiten' gehen und auf 'Erstellen klicken. Es öffnet sich die Eingabe Seite um eine neue Seite zu erstellen. Seiten Titel festlegen Den neuen
Seite erstellen Mit der Maus im Menü links auf den Menüpunkt 'Seiten' gehen und auf 'Erstellen klicken. Es öffnet sich die Eingabe Seite um eine neue Seite zu erstellen. Seiten Titel festlegen Den neuen
Wie Sie mit Mastern arbeiten
 Wie Sie mit Mastern arbeiten Was ist ein Master? Einer der großen Vorteile von EDV besteht darin, dass Ihnen der Rechner Arbeit abnimmt. Diesen Vorteil sollten sie nutzen, wo immer es geht. In PowerPoint
Wie Sie mit Mastern arbeiten Was ist ein Master? Einer der großen Vorteile von EDV besteht darin, dass Ihnen der Rechner Arbeit abnimmt. Diesen Vorteil sollten sie nutzen, wo immer es geht. In PowerPoint
Stammdatenanlage über den Einrichtungsassistenten
 Stammdatenanlage über den Einrichtungsassistenten Schritt für Schritt zur fertig eingerichteten Hotelverwaltung mit dem Einrichtungsassistenten Bitte bereiten Sie sich, bevor Sie starten, mit der Checkliste
Stammdatenanlage über den Einrichtungsassistenten Schritt für Schritt zur fertig eingerichteten Hotelverwaltung mit dem Einrichtungsassistenten Bitte bereiten Sie sich, bevor Sie starten, mit der Checkliste
Erstellen eines Beitrags auf der Homepage Schachverband Schleswig-Holstein - Stand vom 01.06.2015
 Erstellen eines Beitrags auf der Homepage Einleitung... 3 01 Startseite aufrufen... 4 02 Anmeldedaten eingeben... 5 03 Anmelden... 6 04 Anmeldung erfolgreich... 7 05 Neuen Beitrag anlegen... 8 06 Titel
Erstellen eines Beitrags auf der Homepage Einleitung... 3 01 Startseite aufrufen... 4 02 Anmeldedaten eingeben... 5 03 Anmelden... 6 04 Anmeldung erfolgreich... 7 05 Neuen Beitrag anlegen... 8 06 Titel
Google Places Karte in die Homepage integrieren. Was ist Google Places?
 Google Places Karte in die Homepage integrieren Was ist Google Places? Google Places ist ein Unternehmensverzeichnis regionaler Anbieter, das sich mit anderen Diensten von Google, wie Google Maps und Google
Google Places Karte in die Homepage integrieren Was ist Google Places? Google Places ist ein Unternehmensverzeichnis regionaler Anbieter, das sich mit anderen Diensten von Google, wie Google Maps und Google
Lehrer: Einschreibemethoden
 Lehrer: Einschreibemethoden Einschreibemethoden Für die Einschreibung in Ihren Kurs gibt es unterschiedliche Methoden. Sie können die Schüler über die Liste eingeschriebene Nutzer Ihrem Kurs zuweisen oder
Lehrer: Einschreibemethoden Einschreibemethoden Für die Einschreibung in Ihren Kurs gibt es unterschiedliche Methoden. Sie können die Schüler über die Liste eingeschriebene Nutzer Ihrem Kurs zuweisen oder
Anleitung für die Formularbearbeitung
 1 Allgemeines Anleitung für die Formularbearbeitung Die hier hinterlegten Formulare sind mit der Version Adobe Acrobat 7.0 erstellt worden und im Adobe-PDF Format angelegt. Damit alle hinterlegten Funktionen
1 Allgemeines Anleitung für die Formularbearbeitung Die hier hinterlegten Formulare sind mit der Version Adobe Acrobat 7.0 erstellt worden und im Adobe-PDF Format angelegt. Damit alle hinterlegten Funktionen
Zahlen auf einen Blick
 Zahlen auf einen Blick Nicht ohne Grund heißt es: Ein Bild sagt mehr als 1000 Worte. Die meisten Menschen nehmen Informationen schneller auf und behalten diese eher, wenn sie als Schaubild dargeboten werden.
Zahlen auf einen Blick Nicht ohne Grund heißt es: Ein Bild sagt mehr als 1000 Worte. Die meisten Menschen nehmen Informationen schneller auf und behalten diese eher, wenn sie als Schaubild dargeboten werden.
Der neue persönliche Bereich/die CommSy-Leiste
 Der neue persönliche Bereich/die CommSy-Leiste Mit der neue CommSy-Version wurde auch der persönliche Bereich umstrukturiert. Sie finden all Ihre persönlichen Dokumente jetzt in Ihrer CommSy-Leiste. Ein
Der neue persönliche Bereich/die CommSy-Leiste Mit der neue CommSy-Version wurde auch der persönliche Bereich umstrukturiert. Sie finden all Ihre persönlichen Dokumente jetzt in Ihrer CommSy-Leiste. Ein
Konvertieren von Settingsdateien
 Konvertieren von Settingsdateien Mit SetEdit können sie jedes der von diesem Programm unterstützten Settingsformate in jedes andere unterstützte Format konvertieren, sofern Sie das passende Modul (in Form
Konvertieren von Settingsdateien Mit SetEdit können sie jedes der von diesem Programm unterstützten Settingsformate in jedes andere unterstützte Format konvertieren, sofern Sie das passende Modul (in Form
Hohe Kontraste zwischen Himmel und Landschaft abmildern
 PhotoLine-Bildbearbeitung Erstellt mit Version 16.11 In diesem Beispiel möchte ich zeigen, wie ich zur Zeit Landschaftsbilder mit hohen Kontrasten bearbeite. "Zur Zeit" deshalb, weil sich das natürlich
PhotoLine-Bildbearbeitung Erstellt mit Version 16.11 In diesem Beispiel möchte ich zeigen, wie ich zur Zeit Landschaftsbilder mit hohen Kontrasten bearbeite. "Zur Zeit" deshalb, weil sich das natürlich
NODELOCKED LIZENZ generieren (ab ST4)
 NODELOCKED LIZENZ generieren () Besuchen Sie folgende Webseite support.ugs.com/global/de ( ohne www oder http:// ) Klicken Sie auf Lizenz Verwaltung und dann auf aktuelle Lizenz 1 1. Geben Sie Ihren Webkey
NODELOCKED LIZENZ generieren () Besuchen Sie folgende Webseite support.ugs.com/global/de ( ohne www oder http:// ) Klicken Sie auf Lizenz Verwaltung und dann auf aktuelle Lizenz 1 1. Geben Sie Ihren Webkey
Nutzung von GiS BasePac 8 im Netzwerk
 Allgemeines Grundsätzlich kann das GiS BasePac Programm in allen Netzwerken eingesetzt werden, die Verbindungen als Laufwerk zu lassen (alle WINDOWS Versionen). Die GiS Software unterstützt nur den Zugriff
Allgemeines Grundsätzlich kann das GiS BasePac Programm in allen Netzwerken eingesetzt werden, die Verbindungen als Laufwerk zu lassen (alle WINDOWS Versionen). Die GiS Software unterstützt nur den Zugriff
Professionelle Seminare im Bereich MS-Office
 Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Anleitung zum Importieren, Durchführen und Auswerten von Umfragen in Blackboard
 Center für Digitale Systeme (CeDiS) E-Learning, E-Research, Multimedia evaluation@cedis.fu-berlin.de Januar 2015 Anleitung zum Importieren, Durchführen und Auswerten von Umfragen in Blackboard Kontakte
Center für Digitale Systeme (CeDiS) E-Learning, E-Research, Multimedia evaluation@cedis.fu-berlin.de Januar 2015 Anleitung zum Importieren, Durchführen und Auswerten von Umfragen in Blackboard Kontakte
PDAP7 FMEA Erste Schritte Stand März 2010
 PDAP7 FMEA Erste Schritte Stand März 2010 Dieses Modul bietet Ihnen eine einfache Möglichkeit Ihre Fehler-Möglichkeits- und Einflussanalysen zu visualisieren, dokumentieren und deren Termine zu überwachen.
PDAP7 FMEA Erste Schritte Stand März 2010 Dieses Modul bietet Ihnen eine einfache Möglichkeit Ihre Fehler-Möglichkeits- und Einflussanalysen zu visualisieren, dokumentieren und deren Termine zu überwachen.
S/W mit PhotoLine. Inhaltsverzeichnis. PhotoLine
 PhotoLine S/W mit PhotoLine Erstellt mit Version 16.11 Ich liebe Schwarzweiß-Bilder und schaue mir neidisch die Meisterwerke an, die andere Fotografen zustande bringen. Schon lange versuche ich, auch so
PhotoLine S/W mit PhotoLine Erstellt mit Version 16.11 Ich liebe Schwarzweiß-Bilder und schaue mir neidisch die Meisterwerke an, die andere Fotografen zustande bringen. Schon lange versuche ich, auch so
Ich möchte meine Beitragsnachweise nach dem vereinfachten Schätzverfahren erstellen.
 Das vereinfachte Schätzverfahren Ich möchte meine Beitragsnachweise nach dem vereinfachten Schätzverfahren erstellen. Wie muss ich vorgehen? 1 Einstieg Wenn Sie in den Firmenstammdaten auf der Seite Sozialversicherung
Das vereinfachte Schätzverfahren Ich möchte meine Beitragsnachweise nach dem vereinfachten Schätzverfahren erstellen. Wie muss ich vorgehen? 1 Einstieg Wenn Sie in den Firmenstammdaten auf der Seite Sozialversicherung
ECO-Manager - Funktionsbeschreibung
 ECO-Manager - Funktionsbeschreibung Version Autor Datum Kommentare 1.0 A. Sterzenbach 24.03.2006 - Generell Das Einarbeiten und das damit verbundene Aktualisieren eines großen Zusammenbaus (z.b. Werkzeugaufbau)
ECO-Manager - Funktionsbeschreibung Version Autor Datum Kommentare 1.0 A. Sterzenbach 24.03.2006 - Generell Das Einarbeiten und das damit verbundene Aktualisieren eines großen Zusammenbaus (z.b. Werkzeugaufbau)
Electronic Systems GmbH & Co. KG
 Anleitung zum Anlegen eines POP3 E-Mail Kontos Bevor die detaillierte Anleitung für die Erstellung eines POP3 E-Mail Kontos folgt, soll zuerst eingestellt werden, wie oft E-Mail abgerufen werden sollen.
Anleitung zum Anlegen eines POP3 E-Mail Kontos Bevor die detaillierte Anleitung für die Erstellung eines POP3 E-Mail Kontos folgt, soll zuerst eingestellt werden, wie oft E-Mail abgerufen werden sollen.
Ein PDF erstellen. aus Adobe InDesign CC. Langner Marketing Unternehmensplanung Metzgerstraße 59 72764 Reutlingen
 aus Adobe InDesign CC Langner Marketing Unternehmensplanung Metzgerstraße 59 72764 Reutlingen T 0 71 21 / 2 03 89-0 F 0 71 21 / 2 03 89-20 www.langner-beratung.de info@langner-beratung.de Über Datei >
aus Adobe InDesign CC Langner Marketing Unternehmensplanung Metzgerstraße 59 72764 Reutlingen T 0 71 21 / 2 03 89-0 F 0 71 21 / 2 03 89-20 www.langner-beratung.de info@langner-beratung.de Über Datei >
ID VisitControl. Dokumentation Administration. 2015 Equitania Software GmbH cmc Gruppe Seite 1
 ID VisitControl Dokumentation Administration 2015 Equitania Software GmbH cmc Gruppe Seite 1 Inhalt 1. Anmeldung... 3 2. Benutzer anlegen oder bearbeiten... 4 2.1. Benutzer aus LDAP Anbindung importieren/updaten...
ID VisitControl Dokumentation Administration 2015 Equitania Software GmbH cmc Gruppe Seite 1 Inhalt 1. Anmeldung... 3 2. Benutzer anlegen oder bearbeiten... 4 2.1. Benutzer aus LDAP Anbindung importieren/updaten...
Einführungskurs MOODLE Themen:
 Einführungskurs MOODLE Themen: Grundlegende Einstellungen Teilnehmer in einen Kurs einschreiben Konfiguration der Arbeitsunterlagen Konfiguration der Lernaktivitäten Die Einstellungen für einen Kurs erreichst
Einführungskurs MOODLE Themen: Grundlegende Einstellungen Teilnehmer in einen Kurs einschreiben Konfiguration der Arbeitsunterlagen Konfiguration der Lernaktivitäten Die Einstellungen für einen Kurs erreichst
Fotostammtisch-Schaumburg
 Der Anfang zur Benutzung der Web Seite! Alles ums Anmelden und Registrieren 1. Startseite 2. Registrieren 2.1 Registrieren als Mitglied unser Stammtischseite Wie im Bild markiert jetzt auf das Rote Register
Der Anfang zur Benutzung der Web Seite! Alles ums Anmelden und Registrieren 1. Startseite 2. Registrieren 2.1 Registrieren als Mitglied unser Stammtischseite Wie im Bild markiert jetzt auf das Rote Register
PDF-Dateien erstellen mit edocprinter PDF Pro
 AT.014, Version 1.4 02.04.2013 Kurzanleitung PDF-Dateien erstellen mit edocprinter PDF Pro Zur einfachen Erstellung von PDF-Dateien steht den Mitarbeitenden der kantonalen Verwaltung das Programm edocprinter
AT.014, Version 1.4 02.04.2013 Kurzanleitung PDF-Dateien erstellen mit edocprinter PDF Pro Zur einfachen Erstellung von PDF-Dateien steht den Mitarbeitenden der kantonalen Verwaltung das Programm edocprinter
Programme im Griff Was bringt Ihnen dieses Kapitel?
 3-8272-5838-3 Windows Me 2 Programme im Griff Was bringt Ihnen dieses Kapitel? Wenn Sie unter Windows arbeiten (z.b. einen Brief schreiben, etwas ausdrucken oder ein Fenster öffnen), steckt letztendlich
3-8272-5838-3 Windows Me 2 Programme im Griff Was bringt Ihnen dieses Kapitel? Wenn Sie unter Windows arbeiten (z.b. einen Brief schreiben, etwas ausdrucken oder ein Fenster öffnen), steckt letztendlich
Mediator 9 - Lernprogramm
 Mediator 9 - Lernprogramm Ein Lernprogramm mit Mediator erstellen Mediator 9 bietet viele Möglichkeiten, CBT-Module (Computer Based Training = Computerunterstütztes Lernen) zu erstellen, z. B. Drag & Drop
Mediator 9 - Lernprogramm Ein Lernprogramm mit Mediator erstellen Mediator 9 bietet viele Möglichkeiten, CBT-Module (Computer Based Training = Computerunterstütztes Lernen) zu erstellen, z. B. Drag & Drop
Bedienungsanleitung Albumdesigner. Neues Projekt: Bestehendes Projekt öffnen:
 Bedienungsanleitung Albumdesigner Hier wählen Sie aus ob Sie mit einem neuen Album beginnen - Neues Projekt erstellen oder Sie arbeiten an einem bestehenden weiter - Bestehendes Projekt öffnen. Neues Projekt:
Bedienungsanleitung Albumdesigner Hier wählen Sie aus ob Sie mit einem neuen Album beginnen - Neues Projekt erstellen oder Sie arbeiten an einem bestehenden weiter - Bestehendes Projekt öffnen. Neues Projekt:
Access 2010. Grundlagen für Anwender. Andrea Weikert 1. Ausgabe, 1. Aktualisierung, Juli 2012. inkl. zusätzlichem Übungsanhang ACC2010-UA
 Andrea Weikert 1. Ausgabe, 1. Aktualisierung, Juli 2012 Access 2010 Grundlagen für Anwender inkl. zusätzlichem Übungsanhang ACC2010-UA 3 Access 2010 - Grundlagen für Anwender 3 Daten in Formularen bearbeiten
Andrea Weikert 1. Ausgabe, 1. Aktualisierung, Juli 2012 Access 2010 Grundlagen für Anwender inkl. zusätzlichem Übungsanhang ACC2010-UA 3 Access 2010 - Grundlagen für Anwender 3 Daten in Formularen bearbeiten
Kapitel 2. Rechnungs-Management
 Kapitel 2 Notwendige Voraussetzungen für diese Schulung: Internetfähiger Computer mit aktuellem Browser (z.b. Firefox, Internet Explorer) Internet-Domain mit aufgeschaltetem Worldsoft SalesPower-Paket.
Kapitel 2 Notwendige Voraussetzungen für diese Schulung: Internetfähiger Computer mit aktuellem Browser (z.b. Firefox, Internet Explorer) Internet-Domain mit aufgeschaltetem Worldsoft SalesPower-Paket.
