Regulation von Segmentierungs- und Hox- Genen durch Gap-Gene im Kurzkeim- Embryo von Tribolium castaneum
|
|
|
- Lennart Morgenstern
- vor 6 Jahren
- Abrufe
Transkript
1 Regulation von Segmentierungs- und Hox- Genen durch Gap-Gene im Kurzkeim- Embryo von Tribolium castaneum Den Naturwissenschaftlichen Fakultäten der Friedrich-Alexander-Universität Erlangen-Nürnberg zur Erlangung des Doktorgrads der Naturwissenschaften vorgelegt von Alexander Christian Cerny aus München
2 Als Dissertation genehmigt von den Naturwissenschaftlichen Fakultäten der Universität Erlangen-Nürnberg Tag der mündlichen Prüfung: Vorsitzender der Promotionskomission: Prof. Dr. E. Bänsch Erstberichterstatter: Zweitberichterstatter: Prof. Dr. M. Klingler Prof. Dr. J. Büning
3 Danksagung Herrn Prof. M. Klingler danke ich für die Vergabe des interessanten Themas und für die Betreuung dieser Arbeit. Dr. Michael Schoppmeier danke ich herzlich für die wertvollen Tipps und Kommentare bei der Erstellung dieser Arbeit. Ein Dankeschön geht auch an die zahlreichen Münchner und Erlanger Mitarbeiter für die gute Zusammenarbeit während der letzten Jahre, besonders an: Dr. Gregor Bucher für die Einführung in das Thema und zahlreiche enthusiastische wissenschaftliche Diskussionen. Dr. Monica Ronco für eine lustige Zeit in unserer WG. Daniel Bäumer für die gute Atmosphäre im Labor und für zahlreiche Ausflüge zur Bäckerei. Prof. Dr. J. Büning für seine Grillpartys. Markus Weber für stets einen guten Spruch zur rechten Zeit und für die moralische Unterstützung während der gemeinsam durchlittenen Begutachtung unserer Werke. Ganz herzlich möchte ich mich auch bei Sue und Jim Brown sowie den Mitgliedern des Brown-Lab für die tolle Gastfreundschaft in Kansas/USA bedanken. Bei Olga Alexandrova, Christian Lauterbach und Ruth Stadler möchte ich mich für die Einweisung in die Confocalmikroskopie bedanken. Kea Edler-Cerny und Prof. Dr. Gerhard Cerny verdienen ein dickes Dankeschön für das Korrekturlesen. Meinen Eltern danke ich für die tolle Unterstützung meiner bisherigen Ausbildung. Dem Sportzentrum der Universität Erlangen gebührt tiefer Dank für die Veranstaltung des Volleyball-Anfängerkurses im WS 03/04, stimmt s Kea? Meiner Frau Kea, meiner Familie und meinen Freunden danke ich für die moralische Unterstützung in schlechten Zeiten und für viele schöne Momente außerhalb des Labors.
4
5 Lebenslauf Name: Alexander Christian Cerny Geburtsdatum: Geburtsort: München Familienstand: verheiratet, 1 Kind Staatsangehörigkeit: deutsch : Besuch der Grundschule in München : Besuch des Gymnasiums in München; Abschluss Abitur mit Note 1, : Zivildienst in der individuellen Schwerbehindertenbetreuung : Weiterbeschäftigung an der Zivildienststelle : Beginn des Studiums Lehramt Gymnasium Biologie / Chemie an der LMU in München : Zwischenprüfung im Fach Chemie; Note 1, : Zwischenprüfung im Fach Biologie; Note 1, : Wechsel zum Studienfach Biologie an der LMU : Vordiplom im Fach Biologie; Note 1, : Beginn der Diplomarbeit: "Funktionelle Charakterisierung des Gap-Gens Krüppel in Tribolium castaneum" im Labor von Prof. Klingler : Abschluss des Diplom Studiengangs Biologie mit der Note 1, : wiss. Hilfskraft im Labor von Prof. Klingler : Beginn der Promotion bei Prof. Klingler; im Frühjahr 2003 gemeinsamer Wechsel zur FAU in Erlangen. Einmonatiger Gastaufenthalt an der Kansas State University im April wiss. Angestellter ( Postdoc ) am Institut für Genetik an der Universität Stuttgart-Hohenheim.
6
7 Publikationen: Cerny, A.C., Bucher, G., Schröder, R. and Klingler, M. (2005). Breakdown of abdominal patterning in the Tribolium Krüppel mutant jaws. Development 132, Cerny, A.C., Bucher, G. and Klingler, M. The single Tribolium ortholog of knirps/knirpsrelated is required for proper segmentation of the head but not the abdomen. In preparation. Cerny, A.C. and Klingler, M. Regulation of Tribolium gap and pair-rule genes by hunchback, Krüppel and giant. In preparation. Vorträge: 18 th European Drosophila Research Conference (Evo-Devo Workshop). Göttingen, Horizons in molecular Biology: 2 nd international PhD student symposium. Göttingen, th Annual Drosophila Research Conference. San Diego,
8 Inhaltsverzeichnis Zusammenfassung...I Summary... III Abbildungsverzeichnis... V Abkürzungsverzeichnis... VIII 1. Einleitung Die Entwicklung des Langkeim-Insekts Drosophila melanogaster Die Entwicklung des Kurzkeim-Insekts Tribolium castaneum Die Segmentierung in anderen Insekten Die Segmentierung in anderen Arthropoden Die Segmentierung in Anneliden und Vertebraten Ziele dieser Arbeit Material und Methoden Haltung und Kreuzung von mutanten Tribolium-Stämmen Kartierung der Mutante krusty Klonierung von Tc knirps Klonierung von Tc domeless, Tc STAT 92E und Tc SOCS 36E Parentale RNAi Embryonale RNAi und embryonale Morpholino-Injektionen Erstellung und Photographie von Kutikulapräparaten Confocalaufnahmen von Kutikulapräparaten Fixierung und in situ-färbungen Ergebnisse Die Klonierung und Analyse von Tc knirps Klonierung und Sequenzanalyse Expression von Tc knirps Expression von Tc knirps in Relation zu anderen Segmentierungsgenen Phänotyp von Tc knirps-rnai-larven Regulation von Paarregel- und Segmentpolaritätsgenen durch Tc knirps Die funktionelle Analyse von Tc Krüppel Kutikulaphänotypen von Tc Krüppel-RNAi und Tc Kr jaws Regulation von Gap-Genen durch Tc Krüppel Interaktionen von Tc Krüppel und Tc giant Expression von Tc knirps in Tc Kr jaws -Embryonen Expression von Tc hunchback in Tc Kr jaws - und Tc Krüppel-RNAi- Embryonen Regulation von Paarregel- und Segmentpolaritätsgenen durch Tc Krüppel Expression von Tc runt und Tc wingless in Tc Kr jaws -Keimstreifen Expression von Tc hairy und Tc engrailed in Tc Kr jaws -Keimstreifen Expression von Tc even-skipped und Tc wingless in Tc Krüppel-RNAi- Embryonen Regulation von Hox-Genen durch Tc Krüppel Expression von Tc Dfd und Tc Ubx in Tc Kr jaws -Keimstreifen Expression von Tc Scr und Tc Antp in Tc Kr jaws - und Tc Krüppel-RNAi- Keimstreifen Die Effekte von Tc abd-b-rnai und Tc wurm auf die posteriore Entwicklung von Tc Kr jaws -mutanten Larven... 81
9 3.3. Die funktionelle Analyse von Tc giant Kutikulaphänotypen von Tc giant-rnai-larven Regulation von Gap-Genen durch Tc giant Expression von Tc hunchback in Tc giant-rnai-embryonen Autoregulation von Tc Krüppel und Tc giant Regulation von Paarregel- und Segmentpolaritätsgenen durch Tc giant Expression von Tc runt in Tc giant-rnai-embryonen Expression von Tc even-skipped in Tc giant-rnai-embryonen Expression von Tc engrailed und Tc wingless in Tc giant-rnai-embryonen Regulation von Hox-Genen durch Tc giant Die funktionelle Analyse von Tc hunchback Kutikulaphänotypen von Tc hunchback-rnai Regulation von Gap-Genen durch Tc hunchback Regulation von Paarregel-und Segmentpolaritätsgenen durch Tc hunchback Expression von Tc eve und Tc runt in frühen Tc hunchback-rnai- Embryonen Expression von Tc eve, Tc runt und Tc wingless in späten Tc hunchback- RNAi-Keimstreifen Regulation von Hox-Genen durch Tc hunchback Injektion von Tc hunchback-dsrna in ältere Embryonen Die Phänotypen von allen Kombinationen an doppelten Gap-Gen Inaktivierungen Die funktionelle Analyse von Tc orthodenticle Die funktionelle Analyse der Mutante krusty Kutikulaphänotyp der Mutante krusty Expression von Tc runt und Tc wingless in der Mutante krusty Expression von Gap-Genen in der Mutante krusty Kartierung der Mutante krusty Die funktionelle Analyse weiterer potentieller Segmentierungsgene Kutikulaphänotypen von Tc Retinsäurerezeptor RNAi Funktionelle Analyse des JAK/STAT-Signalwegs Interpretation der Phänotypen von Tc domeless- und Tc STAT 92E-RNAi Diskussion Die anteriore Expression von Tc knirps ist zu Dm knirps konserviert Die Expression der posterioren Gap-Gen-Domänen unterscheidet sich zwischen Tribolium und Drosophila Die Gap-Gen-Interaktionen in Tribolium sind von anterior nach posterior gerichtet Die Regulation von Gap-Genen durch maternale Gradienten in Kurzkeim-Insekten Interpretation der Phänotypen von Gap-Gen-Orthologen in Tribolium Tc knirps wirkt als Gap-Gen in der Kopfsegmentierung Tc Krüppel limitiert die gnathale Identität und ist essentiell für die Bildung der posterioren abdominalen Segmente Tc giant begrenzt die thorakale Identität und hat eine komplexe Funktion in der Segmentierung Tc hunchback begrenzt die abdominale Identität und ist notwendig für die posteriore Segmentierung Die Gap-Gen-Orthologen in Tribolium spielen eine zentrale Rolle in der Regulation der Hox-Gene Regulation von Hox-Genen durch die Orthologen von Krüppel Regulation von Hox-Genen durch die Orthologen von giant Regulation von Hox-Genen durch die Orthologen von hunchback
10 4.7. Die Segmentierungsfunktion der Gap-Gen-Orthologen ist in Tribolium nicht konserviert Es gibt kein Gap-Gen-System im Blastoderm von Tribolium Die Gap-Gen-Orthologen in Tribolium üben während des Keimstreifwachstums eine Funktion in der posterioren Segmentierung aus Die Mutante krusty deutet auf eine erweiterte Segmentierungsfunktion der primären Paarregelgene in Tribolium hin Modelle zur Segmentierung in Tribolium Ausblick Literaturverzeichnis Anhang
11 Zusammenfassung Im Langkeim-Insekt Drosophila melanogaster fungieren die Gap-Gene als erste zygotische Regulatoren der Segmentierung und bilden im syncytialen Blastoderm kurzreichweitige Morphogengradienten, welche die Expression einzelner Paarregelstreifen kontrollieren. Im Kurzkeim-Insekt Tribolium castaneum werden hingegen nur die anteriorsten Segmente im Blastoderm angelegt, während die posterioren Segmente aus einer zellulären Wachstumszone entstehen. Die Orthologen der Gap-Gene knirps, Krüppel, giant und hunchback sind dabei sowohl im Blastoderm als auch im wachsenden Keimstreif exprimiert und wurden im Rahmen dieser Arbeit eingehend funktionell analysiert. Bei der Klonierung von Genen der knirps-familie konnte in Tribolium nur ein Ortholog des Dm knirps/dm knirps-related-genpaares gefunden werden, das während der Segmentierung zwei Expressionsdomänen zeigt. Dabei deckt die anteriore Domäne von Tc knirps den Bereich der Kopfanlage bis einschließlich des mandibulären Segments ab, während die posteriore Domäne einen schmalen Streifen in der Anlage des ersten abdominalen Segments bildet. Die Inaktivierung von Tc knirps mittels parentaler RNAi erzeugt mit dem Verlust der antennalen und mandibulären Segmente zwar einen interessanten Kopf-Gap-Gen -Phänotyp, hat aber keinen Effekt auf die posteriore Segmentierung. Bei der funktionellen Inaktivierung von Tc Krüppel, Tc giant und Tc hunchback treten komplexe Phänotypen auf, die durch anteriore homeotische Transformationen und posteriore Segmentierungsdefekte gekennzeichnet sind. Wie schon an Hand der Phänotypen ersichtlich, besteht ein gemeinsames Merkmal dieser drei Gap-Gene in der Regulation der homeotischen Gene. Dabei wirkt Tc Krüppel als posteriorer Repressor von Tc Deformed und Tc Sex combs reduced, Tc giant als anteriorer Repressor von Tc Antennapedia sowie Tc hunchback als anteriorer Repressor von Tc abdominal-a. In ihren Segmentierungsfunktionen unterscheiden sich die einzelnen Gap-Gene deutlich voneinander. Der Ausfall von Tc Krüppel in Tc Kr jaws führt über einen vorzeitigen Segmentierungsabbruch zur Deletion der posteriorsten sechs Segmente. Die einzige Gap-Gen- Domäne mit einer zu Drosophila vergleichbaren Funktion ist die anteriore Tc giant-domäne, deren Inaktivierung den Verlust des zweiten Tc runt-streifens bewirkt. Der Ausfall der posterioren Domäne von Tc giant führt hingegen zum Ausfall der Paarregelstreifen 6-8, was schließlich eine sehr unregelmäßige Expression der abdominalen Segmentpolaritätsgene zur Folge hat. Im Falle von Tc hunchback konnte die publizierte Interpretation des Phänotyps als anteriore Deletion (Schröder 2003) nicht bestätigt werden, da mit molekularen Markern keine I
12 gravierenden Defekte in der anterioren Musterbildung gefunden werden konnten. Stattdessen führt die fehlende Aktivierung von Tc Krüppel zu einem posterioren Segmentierungsabbruch, der aber in den stärksten Phänotypen aufgrund einer späten Segmentierungsfunktion von Tc hunchback stärker als in Tc Kr jaws ausfällt. Neben den Funktionen der Gap-Gen-Orthologen haben sich auch deren Interaktionen in Tribolium verändert. Einhergehend mit der von anterior nach posterior voranschreitenden Segmentierung, finden auch die Interaktionen von Tc Krüppel, Tc giant und Tc hunchback fast ausschließlich von anterior nach posterior statt, wobei es mit der Repression der posterioren Tc hunchback-domäne durch Tc Krüppel zu einer weit reichenden Interaktion im wachsenden Keimstreif kommt. Abschließend lässt sich für die Gap-Gen-Orthologen in Tribolium eine zu Drosophila prinzipiell konservierte Rolle in der Regulation der Hox-Gene finden, aber mit Ausnahme der anterioren Domäne von Tc giant beobachtet man keine offensichtlich direkten Auswirkungen auf spezifische Streifen der Paarregelgene. Vielmehr scheinen die Gap-Gen-Orthologen eher eine permissive Funktion für die Expression weiter posterior gelegener Paarregelstreifen zu erfüllen. Eine unmittelbare Dereprimierung der Paarregelstreifen konnte hingegen in der Mutante krusty gefunden werden, die mit hoher Wahrscheinlichkeit auf einem Defekt im Paarregelgen Tc odd-skipped beruht. Außerdem zeigt die funktionelle Analyse weiterer Paarregelgene, dass die Regulation der Paarregelstreifen in Tribolium im Wesentlichen durch einen Paarregel- Oszillationsmechanismus geschieht (Choe et al. 2006). II
13 Summary In the long-germ insect Drosophila melanogaster, the gap-genes are the first zygotic segmentation genes to become activated. In the syncytial blastoderm, their gene products are forming short-range morphogen-gradients that govern the expression of single pair-rule stripes. In the short-germ insect Tribolium castaneum however, only the anteriormost segments arise during the blastoderm stage whereas the posterior segments are set up by a cellular growth zone. The orthologues of the gap-genes knirps, Krüppel, giant and hunchback are expressed both in the blastoderm and in the growing germband. In this thesis, a functional analysis of theses gap-gene orthologues was performed. By cloning genes of the knirps-family, only a single orthologue of the Dm knirps/dm knirpsrelated gene-pair could be identified in Tribolium. Tc knirps is expressed in two domains: the anterior one covers the head anlagen and the mandibulary segment, while the posterior domain forms a narrow stripe in the primordia of the first abdominal segment. The inactivation of Tc knirps via parental RNAi results in a "head gap" phenotype, affecting the antennal and mandibulary segments, but has no effect on posterior segmentation. The functional inactivation of Tc Krüppel, Tc giant and Tc hunchback leads to complex phenotypes, combining anterior homeotic transformations and posterior segmentation defects. As evident from their phenotypes, a common feature of these three gap-gene orthologues is a role in the regulation of Hox-genes: Tc Krüppel acts as a posterior repressor of the gnathal Hox-genes Tc Deformed and Tc Sex combs reduced, Tc giant as anterior repressor of Tc Antennapedia and Tc hunchback as anterior repressor of Tc abdominal-a. Concerning their segmentation function, there are differences between these gap-genes. The lack of Tc Krüppel function in Tc Kr jaws -mutant embryos leads to a segmentation breakdown, resulting in the loss of the six posteriormost segments. The anterior Tc giant domain is the only gap-gene domain with a function comparable to the gap-genes of Drosophila, as its inactivation causes a specific deletion of the Tc runt2-stripe. In the absence of the posterior Tc giant activity, however, the pair-rule stripes 6-8 fail to be activated. As a consequence, an irregular expression of abdominal segment-polarity genes occurs. In the case of Tc hunchback, the already published interpretation of the phenotype as an anterior deletion (Schröder 2003) could not be verified, as molecular markers do not detect significant anterior patterning defects. There is a posterior segmentation breakdown caused by the failure of Tc Krüppel activation instead, which is enhanced by a late segmentation function of Tc hunchback in the strongest phenotypes. III
14 Beside their functions, also the interactions among the gap-gene orthologues have changed. Corresponding to the anterior towards posterior proceeding segmentation, the interactions of Tc Krüppel, Tc giant and Tc hunchback also are mostly directed from anterior to posterior. Interestingly, the repression of the posterior Tc hunchback domain by Tc Krüppel represents an interaction over a long distance in the growing germband. In summary, the regulation of Hox-genes by the gap-gene orthologues of Tribolium is mainly conserved to Drosophila. Concerning segmentation however, they do not appear to exert a direct function in the regulation of pair-rule stripes except for the anterior Tc giant domain, but rather may play a permissive role for the expression of more posterior pair-rule stripes. In the mutant krusty which is most likely caused by a defect in the pair-rule gene Tc oddskipped, an immediate derepression of pair-rule stripes can be observed. In addition, the functional analysis of further pair-rule genes suggests a pair-rule oscillation to regulate pairrule stripes in Tribolium (Choe et al. 2006).. IV
15 Abbildungsverzeichnis: Abbildung 1: Larvale Morphologie von Tribolium und Drosophia...9 Abbildung 2: Kurzkeimentwicklung in Tribolium castaneum...11 Abbildung 3: Alignment der Aminosäuresequenz von Tc Knirps/Knirps-related mit anderen Proteinen aus der knirps-famile...36 Abbildung 4: Phylogramm der knirps-familie Proteine...38 Abbildung 5: Die Struktur der Tc kni/knrl-, Dm knirps- und Dm knirps-related-gene...39 Abbildung 6: Expression von Tc knirps im Blastoderm...43 Abbildung 7: Expression von Tc knirps im Keimstreifstadium...45 Abbildung 8: Expression von Tc knirps, Tc giant und Tc eve im Blastoderm...47 Abbildung 9: Expression von Tc knirps, Tc Krüppel, Tc giant und Tc eve im späten Blastoderm und in frühen Keimstreifen...49 Abbildung 10: Der Kopfdefekt in Tc knirps-rnai-larven...51 Abbildung 11: Expression von Tc eve und Tc wg in Wildtypen und in Tc knirps- RNAi-Blastodermembryonen...53 Abbildung 12: Expression von Tc eve und Tc wg in Wildtypen und in Tc knirps- RNAi-Keimstreifen...55 Abbildung 13: Phänotypische Serie von Tc Krüppel...58 Abbildung 14: Interaktionen zwischen Tc Krüppel und Tc giant...61 Abbildung 15: Expression von Tc knirps in Wildtyp- und Tc Kr jaws -Keimstreifen...62 Abbildung 16: Expression von Tc hunchback und Tc giant in Wildtyp-, Tc Krüppel- RNAi- und Tc Kr jaws -Embryonen...65 Abbildung 17: Expression von Tc runt und Tc wingless in Wildtypen und in Tc Kr jaws - Keimstreifen...69 Abbildung 18: Expression von Tc hairy und Tc engrailed in Wildtypen und in Tc Kr jaws -Keimstreifen...71 Abbildung 19: Expression von Tc hairy und Tc engrailed in späten Tc Kr jaws -Keimstreifen...72 Abbildung 20: Expression von Tc eve und Tc wingless in späten Tc Krüppel- RNAi-Keimstreifen...75 V
16 Abbildung 21: Expression von Hox-Genen in Wildtypen und in Tc Kr jaws -Keimstreifen...79 Abbildung 22: Expression von Tc Scr und Tc Antennapedia in Tc Krüppel- RNAi-Keimstreifen...80 Abbildung 23: Die Mutante Tc wurm und Tc abd-b RNAi in Tc Kr jaws - mutantem Hintergrund...83 Abbildung 24: Tc giant-rnai-phänotypen...87 Abbildung 25: Expression von Tc hunchback und Tc wg in Tc giant-rnai-embryonen..89 Abbildung 26: Autoregulation von Tc Krüppel und Tc giant...91 Abbildung 27: Expression von Tc runt in Wildtypen und in Tc giant-rnai-embryonen..95 Abbildung 28: Expression von Tc eve in Wildtypen und in Tc giant-rnai-embryonen...97 Abbildung 29: Expression von Paarregel- und Segmentpolaritätsgenen in späten Tc giant-rnai-keimstreifen...99 Abbildung 30: Expression von Hox-Genen in Wildtyp- und Tc giant-rnai-embryonen.101 Abbildung 31: Tc hunchback-rnai-phänotypen Abbildung 32: Expression von Tc Krüppel und Tc giant in Wildtypen und in Tc hunchback-rnai-embryonen Abbildung 33: Expression von Tc eve und Tc runt in Wildtyp- und Tc hunchback- RNAi-Blastodermembryonen Abbildung 34: Expression von Tc eve und Tc runt in Wildtyp- und Tc hunchback- RNAi-Keimstreifen Abbildung 35: Expression von Tc eve, Tc runt und Tc en/tc wg in späten Tc Kr jaws - und Tc hunchback-rnai-keimstreifen Abbildung 36: Expression von Tc Deformed und Tc abd-a in Wildtypen und Tc hunchback-rnai-keimstreifen Abbildung 37: Kutikulaphänotypen aus später embryonaler Tc hunchback-rnai Abbildung 38: Phänotypen von Larven mit jeweils zwei inaktivierten Gap-Gen- Orthologen Abbildung 39: Expression von Tc eve und Tc knirps- in Wildtyp- und Tc otd-rnai-blastodermembryonen Abbildung 40: Der Phänotyp der Mutante krusty VI
17 Abbildung 41: Expression von Tc runt und Tc wingless in Wildtypen und krustymutanten Keimstreifen Abbildung 42: Expression von Tc wingless in späten Wildtyp-, krusty- und scratchy-keimstreifen Abbildung 43: Expression der Gap-Gene in Wildtypen und in krustymutanten Keimstreifen Abbildung 44: Rekombination von krusty bezüglich des Markers 16.G01s Abbildung 45: Expression und Funktion des Retinsäurerezeptors in Tribolium Abbildung 46: Die Phänotypen von Tc domeless- und Tc STAT 92E-RNAi Abbildung 47: Interaktionen der Gap-Gene in Tribolium und Drosophila Abbildung 48: Regulation der Hox-Gene durch Gap-Gene in Tribolium VII
18 Abbkürzungsverzeichnis Abb abd-a Abd-B Ag Antp AP As β-gal bp cdna cm Dfd Dm dsrna en EST eve FGF ftz Gb gt hb Hox kb kni knrl Kr mrna Nv Of otd pb PCR PS RACE RNAi Scr Su(H) Tc Ubx UTR wg Wnt zen ZNS Abbildung abdominal A Abdominal B Anopheles gambia Antennapedia Alkalische Phosphatase Aminosäure beta-galaktosidase Basenpaare copy DNA (durch reverse Transkription aus mrna) centi-morgan (% Rekombinationsfrequenz) Deformed Drosophila melanogaster doppelsträngige RNA engrailed expressed sequence tag even-skipped Fibroblast Growth Factor fushi-tarazu Gryllus bimaculatus giant hunchback Homeobox Kilobasen knirps knirps-related Krüppel messenger RNA Nasonia vitripennis Oncopeltus fasciatus orthodenticle proboscipedia polymerase chain reaction Parasegment rapid amplification of cdna ends RNA-Interferenz Sex combs reduced Suppressor of Hairless Tribolium castaneum Ultrabithorax untranslated region (auf der mrna) wingless Wg/int ähnliche Signalmoleküle zerknüllt Zentralnervensystem VIII
19 1. Einleitung Die Ontogenese von der Zygote zu vielzelligen und komplexen Organismen mit zahlreichen Geweben stellt einen äußerst komplizierten Prozess dar, der von einer Vielzahl räumlich und zeitlich exakt definierter molekularer Signale gesteuert wird. Während bis zu den 80er Jahren durch Gewebetransplantationen allenfalls die Quelle instruierender Signale bestimmt werden konnte (Spemann und Mangold 1924), ermöglichte die Anwendung eines Saturierungs- Mutagenesescreens auf die Embryonalentwicklung der Taufliege Drosophila melanogaster erstmals die Isolierung zahlreicher Entwicklungsgene (Nüsslein-Volhard und Wieschaus 1980; Jürgens et al. 1984). Die funktionelle Charakterisierung dieser Gene führte, zusammen mit neuen histologischen Techniken zur Detektion von mrnas (Tautz und Pfeifle 1989) und Proteinen, schließlich zu einem weitgehenden molekularen Verständnis der frühen Embryonalentwicklung in Drosophila melanogaster (Klingler und Tautz 1999; Rivera-Pomar und Jäckle 1996; St Johnston und Nüsslein-Volhard 1992). Im Laufe der nächsten Jahre wurden dann anhand von Drosophila melanogaster, sowie weiteren Modellorganismen wie Caenorhabditis elegans (Nematoden) oder Danio rerio (Zebrafisch), zahlreiche Kenntnisse über Entwicklungsvorgänge in den verschiedensten Tierstämmen gewonnen. Die evolutionäre Entwicklungsbiologie (engl. evolution of development, kurz evo-devo) vergleicht Entwicklungsvorgänge in verschiedenen Spezies hinsichtlich ihrer Homologie und nutzt die daraus gewonnenen Erkenntnisse als ein weiteres Merkmal zur Rekonstruktion des Stammbaums. Eine der zentralen Fragen der evolutionären Entwicklungsbiologie betrifft den Ursprung der Segmentierung, die bei Anneliden, Arthropoden und Vertebraten auftritt (Tautz 2004). Phylogenetische Analysen zeigen jedoch, dass diese Phyla zu drei verschiedenen taxonomischen Großgruppen gehören (Lophotrochozoa, Ecdysozoa, Deuterostomia), wobei die meisten anderen Phyla in jeder dieser Großgruppen nicht segmentiert sind. Dieser Befund lässt nun 3 verschiedene Interpretationen zu: 1. Der gemeinsame Vorläufer von Anneliden, Arthropoden und Vertebraten ( Urbilateria ) zeigte bereits eine segmentale Unterteilung der anterior-posterioren Achse, die unabhängig voneinander in zahlreichen Spezies wieder sekundär reduziert wurde. 2. Der gemeinsame Vorläufer von Anneliden, Arthropoden und Vertebraten war noch nicht segmentiert und die Segmentierung ist in allen drei Phyla unabhängig entstanden. 3. Es sind jedoch auch "gemischte" Szenarien wie z.b. ein homologer Segmentierungsmechanismus zwischen Arthropoden und Vertebraten und eine unabhängige Entwicklung der Segmentierung in Anneliden denkbar. 1
20 Zur Klärung dieser Frage muss das im Wesentlichen auf Drosophila beruhende Wissen über die Segmentierung bei Insekten, welche zahlenmäßig die größte Gruppe der Arthropoden darstellen, auf eine deutlich breitere Basis gestellt werden. Da die Erzeugung und Identifizierung von Mutanten in neuen Arthropodenspezies sehr zeitaufwendig ist, werden in diesen Organismen häufig funktionelle Untersuchungen mittels der erstmals in C. elegans (Fire et al. 1998) angewandten Methode der RNA-Interferenz (kurz RNAi) durchgeführt (Brown et al. 1999a; Hughes und Kaufman 2000). Dabei führt die Injektion von Doppelstrang-RNA(dsRNA) zur Bildung von 21nt langen short interfering RNAs (sirnas), die schließlich im RNA-induced silencing complex (RISC) den Abbau ( Knockdown ) der in ihrer Sequenz komplementären mrna bewirken und somit zu einer Gen-spezifischen transkriptionellen Inaktivierung führen (Mello und Conte 2004; Novina und Sharp 2004). In einigen Insekten wie Tribolium castaneum, Gryllus bimaculatus oder Oncopeltus fasciatus tritt auch systemische RNAi auf, d.h. eine Injektion von dsrna in pupale bzw. adulte weibliche Tiere führt zur "Vererbung" der RNAi auf deren zahlreiche Nachkommen, wodurch in diesen Embryonen sowohl maternale als auch zygotische Entwicklungsgene inaktiviert werden können (Bucher et al. 2002; Liu und Kaufman 2004a; Mito et al. 2005). Allerdings kommt es dabei in der Praxis häufig vor, dass bestimmte dsrnas gar keine oder aber sehr variable Phänotypen erzeugen. Im Vergleich dazu gestaltet sich die Analyse einer Genfunktion mit einer klar definierten Nullmutante wesentlich einfacher. Außerdem birgt die RNAi als Methode die Gefahr, dass nur schon bekannte Entwicklungsgene untersucht werden, während mittels Mutagenesescreens in anderen Organismen auch neue Entwicklungsgene entdeckt werden können. Dies kann jedoch auch teilweise durch RNAi-Screening einer großen Anzahl von codierenden EST-Sequenzen erreicht werden. Neben Drosophila wurden bisher nur in der Wespe Nasonia vitripennis (Pultz et al. 2000) und im roten Reismehlkäfer Tribolium castaneum (Maderspacher et al. 1998; Sulston und Anderson 1996) Mutagenesescreens durchgeführt. Da Tribolium mittlerweile über ein voll sequenziertes Genom mit zahlreichen genetischen Markern verfügt (Lorenzen et al. 2005), sich gut für RNAi eignet (Bucher et al. 2002) und für die Expression transgener Konstrukte zugänglich ist (Berghammer et al. 1999a), ist diese Spezies auf dem Weg, neben Drosophila zu einem weiteren Modellorganismus für die Entwicklung von Insekten aufzusteigen. Die Entwicklung der meisten Insektenembryonen erfolgt über ein syncytiales Blastodermstadium, d.h. die ersten Kernteilungen erfolgen noch ohne Zellteilungen und die etwa 6000 an der Oberfläche angeordneten Zellkerne können durch per Diffusion verbreitete Transkriptionsfaktoren miteinander interagieren. In Kurzkeim-Insekten wie Tribolium werden 2
21 in diesem Stadium neben dem Kopf allerdings nur die gnathalen und teilweise auch die thorakalen Segmente angelegt (Sander 1976), während die restlichen Segmente nach einer morphogenetischen Umwandlung (Handel et al. 2000) im Stadium der Keimstreifverlängerung aus einer Wachstumszone im posterioren Bereich des Keimstreifs gebildet werden. Dieses zeitlich gestaffelte Hinzufügen neuer posteriorer Segmente stellt eine völlig neue Situation dar, da es, im Gegensatz zum früheren Blastodermstadium, in einer zellularisierten Umgebung stattfindet. Die Kurzkeimentwicklung tritt bei den meisten Insektenspezies auf und wird auch als ursprüngliches Merkmal der Insektenentwicklung angesehen (Davis und Patel 2002; Tautz et al. 1994). Die Dipteren einschließlich des Modellorganismus Drosophila zeigen jedoch den abgeleiteten Modus der Langkeimentwicklung, d.h. es werden bereits im syncytialen Blastoderm alle Segmente gleichzeitig angelegt. Ein weiterer Unterschied zwischen diesen Entwicklungstypen besteht außerdem in der Position der Serosaanlage, welche in Kurzkeim- Insekten am anterioren Pol liegt und in den Dipteren an den dorsalen Rand des Blastoderms gewandert ist (Schmidt-Ott 2000) Die Entwicklung des Langkeim-Insekts Drosophila melanogaster Die Segmentierung im syncytialen Drosophila-Embryo erfolgt kaskadenartig, wobei aufeinander abfolgend die maternalen Gene, Gap-Gene und Paarregelgene, sowie nach der Zellularisierung die Segmentpolaritätsgene aktiv werden (Klingler und Tautz 1999; Rivera- Pomar und Jäckle 1996; St Johnston und Nüsslein-Volhard 1992). Die Gene dieser Klassen führen dabei Schritt für Schritt zu einer immer feineren Unterteilung des Embryos. Den Ausgangspunkt stellen vier maternale Gradienten dar, die schon vor der Befruchtung für eine Polarisation der Eizelle sorgen. Dabei wird die bicoid-mrna anterior und die nanos-mrna posterior über Adapterproteine und das Mikrotubuli-Cytoskelett an die jeweiligen Pole lokalisiert (Riechmann und Ephrussi 2001). Im befruchteten Ei beginnt dann die Translation der lokalisierten mrnas, und aufgrund freier Diffusion können sich Gradienten in der Bicoid- (Driever und Nüsslein-Volhard 1988a und 1988b) und Nanos-Proteinkonzentration (Wang und Lehmann 1991) ausbilden. Der posteriore Nanos-Gradient wirkt dabei zusammen mit dem Cofaktor Pumilio als translationaler Repressor der ubiquitär vorhandenen maternalen hunchback-mrna und erzeugt auf diese Weise einen sekundären anterioren Hunchback- Proteingradienten (Irish et al. 1989a; Murata und Wharton 1995). Der anteriore Bicoid- 3
22 Gradient wirkt als transkriptioneller Aktivator der zygotischen Expression von hunchback und definiert dabei über die synergistischen Wechselwirkungen mehrerer Bicoid-Bindungsstellen im hunchback-promotor eine scharfe posteriore Grenze der zygotischen hunchback- Expression (Driever und Nüsslein-Volhard 1989; Ma et al. 1996; Struhl et al. 1989). Zusätzlich inhibiert Bicoid auch noch die Translation der ubiquitären maternalen caudalmrna und erzeugt so einen sekundären posterioren Caudal-Proteingradienten (Mlodzik und Gehring 1987). In späteren Stadien wird caudal dann auch noch zygotisch exprimiert und fungiert dabei als Aktivator für die posterioren Gap-Gene knirps und giant (Rivera-Pomar et al. 1995; Schulz und Tautz 1995). Neben den maternalen Proteingradienten existiert auch noch ein maternales terminales Signal, welches über den Transmembranrezeptor torso übertragen wird (Casanova und Struhl 1989; Klingler et al. 1988) und zur Aktivierung der terminalen Gap-Gene tailless und huckebein führt (Weigel et al. 1990). Außerdem wird auch die dorso-ventrale Achse durch maternale Faktoren bestimmt, welche zu einer von dorsal nach ventral zunehmenden, nukleären Lokalisation des Transkriptionsfaktors dorsal führen (Klingler und Tautz 1999; St Johnston und Nüsslein-Volhard 1992). Die Gap-Gene sind die ersten zygotischen Transkriptionsfaktoren in der Etablierung der anterior-posterioren Achse und ihre Genprodukte bilden wiederum per Diffusion Morphogengradienten, die allerdings im Vergleich zu den maternalen Gradienten eine deutlich geringe Reichweite aufweisen. Ihre Transkription wird in der Regel durch maternale Faktoren aktiviert und durch andere Gap-Gene reprimiert (Hülskamp und Tautz 1991; Rivera- Pomar und Jäckle 1996). Ein Beispiel dafür ist die Reprimierung von Krüppel, knirps und giant durch den Hunchback-Proteingradienten, wobei eine von Krüppel über knirps nach giant zunehmende Sensitivität gegenüber dem Repressor Hunchback für eine posteriore Verlagerung der jeweiligen Domänen sorgt (Hülskamp et al. 1990; Kraut und Levine 1991a). Interessanterweise wirkt Hunchback in niedriger Konzentration aber auch als Aktivator für Krüppel (Hülskamp et al. 1990), während knirps und die posteriore giant-domäne durch Caudal aktiviert werden (Schulz und Tautz 1995). Zusätzlich führt eine gegenseitige Inhibierung zwischen Krüppel und den beiden giant-domänen zu einer weiteren Präzisierung der jeweiligen Expression (Kraut und Levine 1991b). Die posteriorsten Gap-Gene tailless und huckebein werden ausschließlich durch das terminale Signal aktiviert (Bronner und Jäckle 1991; Weigel et al. 1990). Das terminale Gap-Gen tailless fungiert wiederum sowohl als Aktivator der posterioren hunchback-domäne (Margolis et al. 1995) als auch als Repressor- Gradient, der die posterioren Grenzen von knirps und giant definiert (Bronner und Jäckle 1991; Eldon und Pirrotta 1991). 4
23 Die nächstfolgende Ebene in der Hierarchie der Segmentierungskaskade besteht aus der Klasse der Paarregelgene. Diese Gene werden in jeweils sieben Streifen exprimiert, deren Breite sowie Abstand zueinander je etwa einem Segment entspricht, wobei die Lage der Streifen bei den verschiedenen Paarregelgenen relativ zueinander verschoben ist (Carroll 1990). Die Paarregelstreifen werden durch die Gradienten der Gap-Gene reguliert (Klingler und Gergen 1993; Langeland et al. 1994; Pankratz et al. 1990; Reinitz und Sharp 1995). Durch die Analyse der Enhancer in den verschiedenen Paarregelgenen konnte außerdem gezeigt werden, dass bei den primären Paarregelgenen eve, runt, hairy und odd-skipped die frühe Expression der Streifen in den meisten Fällen jeweils von verschiedenen, streifenspezifischen Enhancern gebildet werden (Goto et al. 1989; Klingler et al. 1996; Riddihough und Ish-Horowicz 1991; Schroeder et al. 2004). Eine wesentliche Aufgabe der Gap-Gen-Gradienten besteht also darin, die Positionsinformation für die Expression der einzelnen Paarregelstreifen zu liefern, wobei in jedem Enhancer der Input mehrerer Gap-Gen- Gradienten integriert wird (Klingler et al und Langeland et al. 1994). Das am besten untersuchte Beispiel hierzu stellt der Enhancer für den eve2-streifen dar, der sowohl Bindungsstellen für die Aktivatoren Bicoid und Hunchback als auch für die Repressoren Giant und Krüppel enthält (Small et al und Stanojevic et al. 1991). In giant-mutanten Embryonen fehlt demzufolge der anteriore Repressor und der eve2-streifen expandiert nach anterior, während in Krüppel-mutanten Embryonen der posteriore Repressor fehlt und der eve2-streifen in Richtung posterior expandiert. Interessanterweise gibt es aber mit den eve3+7- und eve4+6-enhancern auch regulatorische Elemente, welche die Expression von je zwei verschiedenen eve-streifen kontrollieren (Small et al. 1996). Dies wird durch die symmetrische Lage der knirps-domäne in der Mitte zwischen den beiden hunchback- Domänen ermöglicht, wodurch die gleichen Positionswerte aus den knirps- und hunchback- Repressorgradienten für die eve-streifen 3 und 7 einerseits, sowie für eve4 und eve6 andererseits genutzt werden können (Clyde et al. 2003). Neben der primären Regulation durch Gap-Gene gibt es auch bei den primären Paarregelgenen wechselseitige Interaktionen, die zur Präzisierung und Stabilisierung der jeweiligen Expressionsmuster essentiell sind (z.b. Hartmann et al. 1994; Klingler und Gergen 1993; Manoukian und Krause 1992). Bei den so genannten sekundären Paarregelgenen wie z.b. paired wird das Streifenmuster sogar ausschließlich durch die Interaktionen mit anderen Paarregelgenen erzeugt, während die Gap- Gene allenfalls noch als allgemeine Aktivatoren fungieren (Gutjahr et al und 1994). Die sieben Streifen der verschiedenen Paarregelgene sind gegeneinander verschoben. Dieses Muster an unterschiedlichen Überlappungen der Paarregelstreifen definiert schließlich, bei 5
24 beginnender Zellularisierung, die Expression der Segmentpolaritätsgene in 14 segmentalen Streifen (Ingham et al. 1988; Swantek und Gergen 2004). Diese Klasse von Genen bewirkt letztendlich die gegenseitige Aktivierung der Wingless- und Hedgehog-Signalwege, welche für die Erhaltung der Segmentgrenzen und für die intrasegmentale Musterbildung sorgen (Klingler und Tautz 1999; Lawrence und Sampedro 1993; Martinez-Arias et al. 1988). Dabei exprimieren die Zellen an der posterioren Grenze eines jeden Parasegments das Signalprotein Wingless. An der benachbarten anterioren Grenze des folgenden Parasegments wird der Ligand Wingless vom Transmembranrezeptor Frizzled gebunden und das Signal über den Wingless-Signaltransduktionsweg in den Kern weitergeleitet, wo neben dem Transkriptionsfaktor engrailed(en) auch noch hedgehog transkribiert wird. Das Signalprotein Hedgehog wird wiederum an der benachbarten posterioren Grenze des vorigen Parasegments vom Transmembranrezeptor Patched gebunden, desses Signal durch den Hedgehog- Signaltransduktionsweg in den Kern übertragen wird, wo sich schließlich durch die Transkription von wingless(wg) der Kreis wieder schließt. Im Gegensatz zu den nur vorübergehend exprimierten Transkriptionsfaktoren der früheren Segmentierungskaskade, werden die Segmentpolaritätsgene der Wingless- und Hedgehog-Signalwege von nun an permanent transkribiert. Dabei stimmen die später gebildeten Segmentgrenzen allerdings nicht mit den zwischen den wingless- und engrailed-streifen verlaufenden Grenzen der embryonalen Parasegmente überein, sondern werden neu hinter den jeweiligen engrailed- Streifen ausgebildet. Ursprünglich erfolgte die Einteilung der Segmentierungsgene anhand der Phänotypen der jeweiligen Mutanten, die für jede Klasse der Segmentierungskaskade charakteristische Merkmale aufweisen. Mutante Segmentpolaritätsgene resultieren typischerweise in Larven, bei denen etwa die Hälfte eines jeden Parasegments fehlt und durch eine spiegelbildliche Duplikation der anderen Hälfte ersetzt wird (Martinez-Arias und Ingham 1985). Bei defekten Paarregelgenen kommt es hingegen im Phänotyp zum periodischen Ausfall eines jeden zweiten Segments, wobei je nach dem Expressionsraster des jeweiligen Paarregelgens geradzahlige Parasegmente (even-skipped), ungeradzahlige Parasegmente (odd-skipped) oder Abschnitte über die Parasegmentgrenzen hinweg (hairy) fehlen (Nüsslein-Volhard und Wieschaus 1980). Da jede Gap-Gen-Domäne die Expression einiger benachbarter Paarregelstreifen steuert, kommt es bei Gap-Gen-Mutanten zur Deletion mehrerer benachbarter Segmente, während weiter anterior oder posterior gebildete Segmente nicht betroffen sind. Auf diese Art entstehen Larven mit einer Lücke in der Segmentierung (Lehmann und Nüsslein-Volhard 1987; Nauber et al. 1988; Petschek et al und 6
25 Wieschaus et al. 1984), wobei die Lage der Lücke mit der Position der Expressionsdomäne im Anlagenplan korreliert (Bender et al. 1988; Knipple et al. 1985; Mohler et al und Nauber et al. 1988). Anders als bei den zygotischen Gap-, Paarregel- und Segmentpolaritätsmutanten sind es im Falle der beiden maternalen Faktoren bicoid und nanos die homozygot-mutanten Mütter, welche Nachkommen mit großräumigen anterioren bzw. posterioren Segmentierungsdefekten produzieren (Nüsslein-Volhard et al. 1987). Die 14 über die Segmentierungskaskade erzeugten Segmentanlagen differenzieren sich im Laufe der weiteren Entwicklung zu den mandibulären, maxillären und labialen Kopfsegmenten, den drei thorakalen Segmenten sowie den abdominalen Segmenten A1-A8. Die Differenzierung der ursprünglich identischen Segmentanlagen zu den verschiedensten segmentalen Identitäten wird dabei von der Klasse der Hox-Gene kontrolliert (Lawrence und Morata 1994; Mc Ginnis und Krumlauf 1992). Diese Homeobox-Transkriptionsfaktoren sind im gesamten Tierreich konserviert und ursprünglich in einem Cluster angeordnet, in dem die Anordnung der Hox-Gene ihrer anterior-posterioren Abfolge in der Expression entspricht. Während es in Vertebraten durch Genomduplikationen vier oder mehr Hox-Komplexe gibt (Holland und Garcia-Fernandez 1996; Krumlauf 1994), erfolgte in Drosophila eine Aufspaltung des ursprünglichen Clusters in den Antennapedia- und den Bithorax-Komplex. Das Expressionsmuster der Hox-Gene wird primär durch Gap- (Casares und Sanchez-Herrero 1995; Harding und Levine 1988; Irish et al. 1989b; Jack und McGinnis 1990; Qian et al. 1991; Reinitz und Levine 1990; Riley et al. 1987; Shimell et al und 2000) und Paarregelgene (Ingham und Martinez-Arias 1986; Jack und McGinnis 1990) erzeugt und dann mit Hilfe von Genen der Polycomb- und der Trithorax-Gruppe dauerhaft stabilisiert (Dejardin and Cavalli 2005). Allerdings treten die Missregulationen der Hox-Gene in den Phänotypen von Gap- oder Paarregel-Mutanten nicht in Erscheinung, da die jeweils betroffenen Segmente in der Regel auch von der Deletion erfasst werden. Neben den 14 über die Segmentierungskaskade erzeugten en- bzw. wg-streifen erscheinen auch in der Kopfanlage weiter en- und wg-expressionsdomänen. Diese Domänen werden als frühe Marker des Labrums sowie des okularen, antennalen und intercalaren Segments angesehen, wobei es aber im Detail verschiedene Interpretationen gibt (Haas et al. 2001; Rogers und Kaufman 1997). Die Bildung dieser Segmente wird u. a. von den drei sogenannten Kopf-Gap-Genen orthodenticle(otd), empty spiracles(ems) und buttonhead(btd) reguliert, deren Expressionsdomänen wiederum durch den Bicoid-Gradienten aktiviert werden (Cohen und Jürgens 1990). 7
26 1.2. Die Entwicklung des Kurzkeim-Insekts Tribolium castaneum Die Entwicklung von Drosophila-Embryonen verläuft im syncytialen Blastoderm nach dem abgeleiteten Modus der Langkeimentwicklung, doch inwiefern ist die aus Drosophila bekannte Segmentierungskaskade in Kurzkeim-Insekten von Bedeutung? Zur Klärung dieser Frage ist das funktionelle Verständnis der orthologen Segmentierungsgene in Kurzkeim- Insekten notwendig. Da der rote Mehlkäfer Tribolium castaneum als Vorratsschädling schon sehr gut beschrieben worden ist, sich leicht im Labor kultivieren lässt und für genetische Analysen zugänglich ist (Beeman et al und Sokoloff 1974), wurde er als erstes Modellsystem zur funktionellen Analyse der Kurzkeimsegmentierung ausgewählt. Neben dem Segmentierungsmodus weist aber auch das erste Larvenstadium einige Unterschiede zwischen Tribolium und Drosophila auf (Abbildung 1). Anders als die stark abgeleitete, beinlose Drosophila Larve mit invaginiertem Kopf, lässt sich die Tribolium Larve klar in einen Kopf mit den Extremitäten der drei gnathalen Segmente (Mandibeln, Maxillen und das Labium), drei thorakale Segmente mit larvalen Beinen und das Abdomen untergliedern. Allerdings lassen sich in Tribolium die abdominalen Segmente A2-A8 in Kutikulapräparaten aber leider nicht voneinander unterscheiden. Die letzten beiden der insgesamt zehn embryonal angelegten abdominalen Segmente fusionieren im Laufe der Entwicklung mit dem Telson und bilden schließlich die terminalen Strukturen der Urogomphi (A9) und der Pygopodien (A10). 8
27 Abbildung 1: Larvale Morphologie von Tribolium und Drosophila Kutikulapräparate vom ersten Larvenstadium von Tribolium und Drosophila in lateraler Ansicht (Photos von Martin Klingler). Die Larve von Tribolium ist klar in Kopf, drei thorakale Segmente mit Beinpaaren (T1-T3), acht abdominale Segmente (A1-A8) und in die terminalen Strukturen mit den Urogomphi (aus A9) und Pygopodien (aus A10) untergliedert. Die thorakalen Segmente lassen sich in Kutikulapräparaten unter anderem durch die nur in T2 vorkommenden Stigmaöffnungen voneinander unterscheiden, während zwischen den abdominalen Segmenten A2-A8 keine morphologischen Unterschiede erkennbar sind. Die in ihrer Morphologie stark abgeleitete Larve von Drosophila zeigt eine Invagination der Kopfsegmente sowie drei beinlose thorakale Segmente. Die acht abdominalen Segmente lassen sich an geringen Unterschieden in dem ventralen Zähnchenband eindeutig identifizieren. Anterior ist links und ventral ist unten. Abkürzungen: Py = Pygopodien; U = Urogomphi 9
28 Bei der mittels degenerierten Primern durchgeführten Suche nach orthologen Segmentierungsgenen in Tribolium konnten neben dem Hox-Komplex unter anderem die Segmentpolaritätsgene Tc engrailed (Brown et al. 1994a) und Tc wingless (Nagy und Caroll 1994), sowie die Orthologen der Paarregelgene Tc even-skipped(tc eve) (Brown et al. 1997), Tc runt (Brown und Denell 1996), Tc hairy (Sommer und Tautz 1993) und Tc ftz (Brown et al. 1994b) kloniert werden. Durch in situ-färbungen konnte außerdem gezeigt werden, dass die Expression der jeweiligen Paarregel- und Segmentpolaritätsstreifen in Relation zu den Parasegmenten in Tribolium weitgehend konserviert ist (Brown und Denell 1996). Anders als in Drosophila, sind in Tribolium im Blastodermstadium aber nur zwei Tc runt- bzw. drei Tc eve- und Tc hairy-streifen exprimiert, was einem für Kurzkeim-Insekten typischen Anlageplan entspricht, in dem die abdominalen Segmente noch nicht im Blastodermstadium gebildet werden (Abbildung 2A, E). Die restlichen der in Tribolium insgesamt je acht Paarregelstreifen entstehen später, Streifen für Streifen, aus der Wachstumszone des sich verlängernden Keimstreifs (Abb. 2B-D, F-H). Während posterior neue Streifen hinzugefügt werden, verschwindet zunehmend die Expression der anterioreren Streifen, so dass zu jedem Zeitpunkt der Keimstreifverlängerung maximal drei bis vier Streifen eines Paarregelgens gleichzeitig exprimiert werden. Im Falle von Tc eve zeigen dabei die älteren Tc eve-streifen eine Aufspaltung in zwei sekundäre, segmentale Streifen (eve2a und eve2b in Abb. 2F; eve4a und eve4b in Abb. 2G), die in ihrer späten Expression mit dem unmittelbar posterior von Tc wingless exprimierten Segmentpolaritätsgen Tc engrailed überlappen. Die ersten beiden segmentalen engrailed- und wingless-streifen treten in Tribolium im frühen Keimrudiment zeitgleich mit dem Verschwinden der ersten Paarregelstreifen auf. Im Zuge der weiteren Keimstreifverlängerung kommt es dann parallel mit dem Verschwinden weiterer Paarregelstreifen zur von anterior nach posterior fortschreitenden Expression neuer en- und wg-streifen (Abb. 2F-H). Dadurch kann man mittels in situ-doppelfärbungen und dem Zählen der segmentalen Streifen auch noch in späten Keimstreifstadien die Identität der posterioren Paarregelstreifen eindeutig bestimmen. Am Ende der Keimstreifverlängerung erlischt schließlich die Expression der achten und letzten Paarregelstreifen, und es bilden sich die letzten beiden der insgesamt 16 regulären en/wg-streifen, während, vergleichbar mit Drosophila, ein komplexes wg-muster in der Kopfsegmentierung erkennbar wird (Abb. 2H). 10
29 Abbildung 2: Kurzkeimentwicklung in Tribolium castaneum Die Abbildungen (A-D) zeigen eine alkoholische Fuchsin-Färbung der Zellkerne in fixierten Tribolium-Embryonen in lateraler Ansicht (Bilder von Martin Klingler). In diesen Fuchsin- Färbungen erkennt man den Übergang vom Blastodermstadium (A) über das Keimrudiment mit einem ventral gelegenen Serosafenster (B) und den posterior wachsenden Keimstreif (C) bis hin zum Stadium des vollständig ausgewachsenen Keimstreifs (D). In Abbildung (E) erkennt man drei Streifen des Paarregelgens Tc eve in einem späten Blastodermstadium mit beginnender posteriorer Invagination. Die Abbildungen (F-H) zeigen eine ventrale Aufsicht auf präparierte Keimstreifen in Stadien vergleichbar zu (B-D), die gegen das Paarregelgen Tc eve (braune Alkalische-Phosphatase-Färbung) und das Segmentpolaritätsgen Tc wingless (blaue β-galaktosidase-färbung) gefärbt wurden. Man erkennt dabei, dass im wachsenden Keimstreif (F, G) stets nur drei eve-streifen gleichzeitig exprimiert werden, wobei sich die späten eve-streifen segmental aufspalten (F, G). Die Expression von Tc wg schreitet ebenfalls von anterior nach posterior voran (F, G), ehe im vollständig ausgewachsenen Keimstreifen schließlich alle 16 Segmente auf der Ebene der Segmentpolaritätsgene angelegt sind (H). Daneben erkennt man noch ein weiteres Tc wg Muster in der Kopfanlage (F-H) sowie in den terminalen Anlagen posterior der Wachstumszone (G, H). Anterior ist immer links, in den Abbildungen (A-E) ist ventral unten. Abkürzungen: wg = wingless, Md = Mandibuläres Segment, Mx = Maxilläres Segment, Lb = Labiales Segment 11
30 Die im Vergleich zu Drosophila zusätzlichen Segmentpolaritätsstreifen lassen sich dadurch erklären, dass ursprünglichere Insekten einschließlich Tribolium noch 10 abdominale Segmente anlegen, während es in Drosophila zu einer sekundären Reduktion der Anlage für die posteriorsten abdominalen Segmente A9 und A10 kommt. Zusammenfassend kann man also sagen, dass in Tribolium zwar die räumliche Expression der Paarregel- und Segmentpolaritätsgene weitgehend konserviert ist, es aber, im Gegensatz zur synchronen Segmentierung in Drosophila, zu einer von anterior nach posterior gestaffelten zeitlichen Abfolge der Segmentierung kommt. Zur weiteren Untersuchung der Segmentierung wurden in Tribolium auch die Orthologen der Gap-Gene hunchback (Wolff et al. 1995), Krüppel (Sommer und Tautz 1993), giant (Bucher und Klingler 2004) und tailless (Schröder et al. 2000) kloniert und analysiert. Dabei zeigte eine in situ-färbung von Tc hunchback neben einer Expression in der Serosa zwei Domänen im Embryo, die mit den Gap-Gen-Domänen in Drosophila homologisierbar sind (Wolff et al. 1995). Die vordere dieser beiden Domänen liegt im posterioren Blastoderm im Bereich der Anlagen der gnathalen Segmente. Aufgrund der unterschiedlichen Anlagepläne der Blastodermstadien in Kurzkeim- und Langkeim-Insekten, umfasst die im posterioren Blastoderm gelegene Tc hunchback-domäne in Tribolium jedoch die gleichen Segmentanlagen wie die im anterioren Blastoderm gelegene Dm hunchback-domäne, und ist daher als homolog zu ihr anzusehen. In späteren Stadien der Keimstreifverlängerung tritt in der Wachstumszone die posteriore Tc hunchback-domäne auf, die sich schließlich über die Segmentanlagen A6-A10 erstreckt (Wolff et al. 1995). Anders als Tc hunchback, wird in Tribolium aber das Ortholog von tailless, welches in Drosophila die posteriore Dm hunchback Domäne aktiviert, schon zu einem deutlich früheren Zeitpunkt am posterioren Pol des Blastoderms exprimiert (Schröder et al. 2000). Überlappend mit Tc tailless, wird auch Tc Krüppel anfänglich im posterioren Blastoderm exprimiert (Sommer und Tautz 1993). Später bildet sich dann in frühen Keimstreifstadien eine posteriore Grenze der Tc Krüppel-Domäne aus, und ein breiter Streifen mit Tc Krüppel- Expression umfasst schließlich die Anlagen der drei thorakalen Segmente (Cerny et al. 2005). Im Vergleich zu Drosophila, wo die Dm Krüppel-Domäne die Anlagen der Segmente T2-A3 umfasst (Gaul et al. 1987), kommt es in Tribolium also zu einer leichten Verschiebung der Krüppel-Domäne in Richtung anterior. In Drosophila liegt Dm Krüppel zwischen der anterioren und der posterioren Dm giant- Domäne und auch in Tribolium ist das Ortholog von giant in zwei Domänen exprimiert (Bucher und Klingler 2004). Die anteriore Tc giant-expression umfasst dabei in frühen 12
31 Blastodermstadien ursprünglich den gesamten Embryo mit Ausnahme der am anterioren Pol gelegenen Serosaanlage. Im Laufe der weiteren Entwicklung zieht sich die Tc giant- Expression aber vom posterioren Pol zurück, und die anteriore Domäne beschränkt sich schließlich auf die Kopfanlagen, wobei ein Streifen kräftigerer Expression das maxilläre Segment markiert. Die posteriore Domäne von Tc giant erscheint erstmals kurz vor Beginn der Gastrulationsbewegung ("posterior pit"-stadium) und spaltet sich im Keimstreifstadium in zwei Streifen auf, die sich mit den Segmentanlagen T3 und A2 überlappen. Während also die anteriore Tc giant-domäne bezüglich ihrer Lage zu Drosophila weitgehend konserviert ist, kommt es bei der posterioren Domäne gegenüber der in Drosophila von A5 bis A7 reichenden Expression (Mohler et al. 1989) zu einer deutlichen Verschiebung nach anterior. Neben ihrer Expression wurde auch die Funktion einiger Gap-Gen Orthologen in Tribolium mittels parentaler RNAi (Bucher et al. 2002) untersucht. In Tc hunchback-rnai -Phänotypen folgen die abdominalen Segmente direkt auf das mandibuläre Segment, weshalb diese Phänotypen als eine Deletion vom maxillären Segment bis einschließlich des dritten thorakalen Segments beschrieben wurden, und man eine zur anterioren Dm hunchback Domäne konservierte Funktion annahm (Schröder 2003). Die Inaktivierung von Tc Krüppel führt zu einer phänotypischen Serie, die den Phänotyp der Mutante jaws (Sulston und Anderson 1996) beinhaltet und inzwischen konnte jaws durch eine Kopplungsanalyse sowie durch Sequenzieren des mutanten Allels auch als eine Tc Krüppel- Nullmutante identifiziert werden (Cerny et al. 2005). In der daraufhin Tc Kr jaws genannten Mutante ist der Kopf einschließlich der drei gnathalen Segmente normal ausgebildet, aber anstelle der Segmente T1-A1 treten ektopische Mundwerkzeuge auf. Außerdem ist das Abdomen auf ein Segment verkürzt, während die beiden terminalen Segmente wiederum nicht beeinträchtigt scheinen (siehe Abb. 13F). Eine Antikörperfärbung gegen Proboscipedia ergab, dass die ektopischen Mundwerkzeuge mit einer zusätzlichen Expression dieses homeotischen Gens in den Extremitäten der jeweiligen Segmente einhergehen. In einer Engrailed-Antikörperfärbung von späten Keimstreifen zeigte sich eine unregelmäßige Expression des siebten und des achten Streifens, worauf auf einen Gap-Phänotyp mit abdominaler Deletion geschlossen wurde (Sulston und Anderson 1998). Interessanterweise führt der Knockdown von Tc giant zur homeotischen Transformation der maxillären und labialen Segmente in thorakale Segmente, womit sich genau der gegensätzliche Phänotyp wie in Tc Kr jaws einstellt (Bucher und Klingler 2004). Daneben konnten mittels Tc engrailed in situ-färbungen auch noch sehr unregelmäßige und großräumig von T1-A9 reichende Segmentierungsdefeke nachgewiesen werden (Bucher und 13
32 Klingler 2004). Somit unterscheiden sich die Segmentierungsdefekte in Tc giant-rnai und Tc Kr jaws mutanten Larven also deutlich von den klassischen Gap-Phänotypen der jeweiligen Mutanten in Drosophila. Über maternale Gradienten ist in Tribolium noch vergleichsweise wenig bekannt. Das aus einer zen/hox3-duplikation (Brown et al und Stauber et al. 1999) entstandene anteriore Morphogen bicoid scheint jedoch nur in höheren Dipteren zu existieren, da es weder im Genom von Anopheles gambia, noch in anderen basalen Dipteren (Stauber et al. 2002) gefunden werden konnte. Dafür ist in Tribolium das Ortholog von orthodenticle(otd), welches in Drosophila als ein durch bicoid reguliertes Kopf-Gap-Gen wirkt, schon maternal exprimiert (Li et al. 1996), und Knockdown -Experimente mittels RNAi zeigen eine komplette anteriore Deletion bis einschließlich T1 (Schröder 2003). Allerdings ist die Tc otdmrna nicht anterior lokalisiert, obwohl die Lokalisation der maternalen Transkripte von Tc eagle und Tc pangolin am anterioren Pol auf die Existenz eines solchen Mechanismus in Tribolium hinweist (Bucher et al. 2005). Im posterioren Blastoderm bildet sich in Tribolium ein mit Drosophila vergleichbarer Tc Caudal-Proteingradient aus, der sich später auf die Wachstumszone zurückzieht und als zygotische Expression im sich verlängernden Keimstreif weiter besteht (Schulz et al. 1998). Da im Kurzkeimer Tribolium die Anlagen der gnathalen und thorakalen Segmente im posterioren Blastoderm liegen, kommt es in Tc caudal- Knockdown-Phänotypen dementsprechend - zusätzlich zum Verlust der Wachstumszone - auch zur Deletion der gnathalen und thorakalen Segmente, so dass nur die weiter anterior gelegene Kopfstrukturen erhalten bleiben (Copf et al. 2004). Einen Verlust aller posterioren Segmente ab dem dritten thorakalen Segment erzeugt auch der Knockdown der an der terminalen Signalkaskade beteiligten Gene Tc torso (Rezeptor) und Tc torso-like (Prozessierung des Ligands) (Schoppmeier und Schröder 2005). Bei der Inaktivierung des torso-signals kommt es nicht zur Ausbildung einer Wachstumszone, wodurch nur die im Blastoderm spezifizierten Segmente (bis einschließlich T2) gebildet werden können (Schoppmeier und Schröder 2005). Tc nanos konnte erst kürzlich im Genom identifiziert werden (C. Schmitt, pers. Mitteilung) und wird momentan funktionell analysiert. Die Existenz von Nanos-Bindungsstellen in den 3 UTRs von Tc otd und Tc hb (Schröder 2003) sowie die fehlende Translation der maternalen hb-mrna am posterioren Pol (Wolff et al. 1995) deuten dabei auf eine konservierte Funktion hin. Parallel zur funktionellen Analyse von Orthologen bekannter Segmentierungsgene aus Drosophila wurden in Tribolium auch erste Mutagenesescreens durchgeführt, die aus 14
33 Kapazitätsgründen allerdings nicht das Niveau eines Sättigungsscreens erreichten (Sulston und Anderson 1996; Maderspacher et al. 1998). Dabei konnten neben der schon erwähnten Krüppel-Mutante Tc Kr jaws mit bollig und krusty zwei weitere Mutanten mit einem Gap- Phänotyp isoliert werden, wobei in krusty-mutanten Keimstreifen das Expressionsmuster von Tc eve die für Drosophila-Gap-Mutanten typische Verbreiterung von Paarregelstreifen zeigt (Maderspacher et al. 1998). Mit itchy und scratchy konnten außerdem zwei klassische Paarregelmutanten isoliert werden (Maderspacher et al. 1998), die aber keine Kopplung mit den primären Paarregelgenen Tc eve, Tc runt und Tc hairy aufweisen (Bucher 2002). Vielmehr konnten durch RNAi-Knockdowns die sekundären Paarregelgene Tc sloppy-paired (itchy) bzw. Tc paired (scratchy) als Kandidatengene identifiziert werden (C. P. Choe, pers. Mitteilung), während der Knockdown der primären Paarregelgene Tc eve und Tc runt zu einem völligen Verlust der Segmentierung führt (Choe et al. 2006). Neben der Segmentierung wurden in Tribolium auch die anders als in Drosophila (Antennapedia- und Bithorax-Komplex) in nur einem Komplex angeordneten Hox-Gene (Beeman et al und Brown et al. 2002a) analysiert. Entfernt man in Tribolium den Hox- Komplex in einer homozygoten Deletionsmutante, so bleiben aufgrund einer fehlenden Segmentierungsfunktion von Tc fushi-tarazu alle Segmente erhalten, und es tritt eine Transformation aller Segmente zu einer antennalen Identität auf (Brown et al. 2002b und Stuart et al. 1991). Daraus kann geschlossen werden, dass die antennale Identität den Grundzustand darstellt, der in den weiter posterior gelegenen Segmenten durch die Hox-Gene modifiziert wird. Im Einzelnen führt die Inaktivierung von Tc Deformed zur Transformation des mandibulären Segments in ein antennales Segment (Brown et al. 2000), die Inaktivierung von Tc Sex combs reduced zur Umwandlung des labialen Segments in ein antennales Segment (Curtis et al. 2001) und erst die kombinierte Inaktivierung beider Gene zur Transformation des maxillären Segments in ein antennales Segment (Brown et al. 2002b). Im Gegensatz dazu zeigt sich der Phänotyp einer Tc proboscipedia-mutante in einer Transformation der maxillären und labialen Extremitäten in Beinextremitäten (Shippy et al. 2000) und deutet eine im Vergleich zu den anderen anterioren Hox-Genen veränderte Funktion von Tc proboscipedia an. Die Inaktivierung von Tc Antennapedia transformiert wiederum die thorakalen Segmente zu antennalen Segmenten (D. Bäumer, pers. Mitteilung), während die weiter posterior gelegenen Hox-Gene Tc Ultrabithorax und Tc abdominal-a erst bei gleichzeitiger Inaktivierung eine Transformation von abdominalen Segmenten zu thorakalen Segmenten bewirken (Lewis et al. 2000). 15
34 1.3. Die Segmentierung in anderen Insekten Zusätzlich zu den am intensivsten untersuchten Modellorganismen Drosophila und Tribolium wird die Segmentierung der Insekten noch in weiteren Kurzkeim- und Langkeim-Spezies studiert. Die ersten Unterschiede werden dabei schon innerhalb der Dipteren sichtbar, denn obwohl alle Dipteren Langkeim-Insekten sind, existiert das aus einer Hox3/zen-Duplikation entstandene maternale Morphogen bicoid nur bei den höheren Fliegen (Cyclorrhaphae) (Stauber et al. 2002). Bei der Malariamücke Anopheles gambia treten zusätzlich noch Unterschiede in der Expression der posterioren Gap-Gene Ag giant und Ag tailless auf, ohne dass sich das Muster der Ag eve Paarregel-Streifen ändert (Goltsev et al. 2004). Das bedeutet, dass sich die Enhancer einzelner Paarregelstreifen im Laufe der Evolution offenbar an verschobene Gap-Gen-Gradienten anpassen können. Neben den Dipteren gibt es auch bei den Hymenopteren, deren genaue Position in der Phylogenie der Insekten gegenwärtig noch umstritten ist, Arten mit Langkeimentwicklung. Eine davon ist die Wespe Nasonia vitripennis, in der ein Mutagenesescreen für zygotische Entwicklungsgene durchgeführt wurde und einige mit Drosophila vergleichbare Segmentierungsmutanten generierte (Pultz et al. 1999). So trat unter anderem die durch eine Deletion anteriorer Segmente gekennzeichnete Mutante headless auf, die schließlich als mutantes Nv hunchback-allel identifiziert werden konnte (Pultz et al. 2005). Daneben weist Nv otd eine gegenüber Dm otd deutlich erweiterte Funktion in der anterioren Segmentierung auf und zeigt völlig unerwartet sogar eine Funktion in der posterioren Segmentierung (J. Lynch, 46th Annual Drosophila Conference). Unter den Kurzkeim-Insekten wird die Segmentierung gegenwärtig auch noch in der Wanze Oncopeltus fasciatus (Hemiptera) sowie in der Mittelmeergrille Gryllus bimaculatus (Orthoptera) funktionell untersucht. Diese beiden Spezies gehören zu den hemimetabolen Insekten und nehmen gegenüber Tribolium eine ursprünglichere Position in der Phylogenie der Insekten ein. Die Entwicklung von Oncopeltus verläuft ähnlich wie in Tribolium über ein Blasotdermstadium, das den Anlageplan bis einschließlich T3 enthält. Daneben ist auch die Expression von Of Krüppel und Of hunchback im Vergleich zu den Orthologen in Tribolium weitgehend konserviert (Liu und Kaufman 2004a und 2004b). Im Gegensatz zu Tribolium führt der Knockdown des Krüppel-Orthologs in Oncopeltus aber zu einem Segmentierungsdefekt im Bereich von T2-A6 und schließt somit Segmente innerhalb der Of Krüppel-Domäne ein (Liu und Kaufman 2004b). Homeotische Transformationen treten in Of Krüppel-RNAi nicht auf, in den Keimstreifen zeigt sich jedoch eine Missregulation des gnathalen Hox-Gens Of Dfd, die aber im Bereich der durch Of Krüppel-RNAi 16
35 hervorgerufenen Deletion liegt (Liu und Kaufman 2004b). Somit wird wie in Drosophila Gap-Gen-Mutanten die homeotische Missregulation durch den an gleicher Stelle auftretenden Segmentierungsdefekt im Phänotyp überdeckt. Die funktionelle Inaktivierung von Of hunchback führt zu keiner Störung der anterioren Segmentierung, es kommt jedoch mit Ausnahme des mandibulären und des dritten thorakalen Segments zu einer Transformation von gnathalen und thorakalen Segmenten zu Segmenten mit abdominaler Identität (Liu und Kaufman 2004a). Das eigentliche Abdomen zeigt hingegen in einer in situ-färbung gegen Of engrailed eine deutlich gestörte Segmentierung (Liu und Kaufman 2004a). Die Embryogenese der Mittelmeergrille Gryllus bimaculatus verläuft nach einem extremen Kurzkeim-Mechanismus: Ein eigentliches syncytiales Blastoderm gibt es bei Orthopteren wahrscheinlich nicht, vielmehr kondensieren Zellen im posterioren Bereich des sehr langen Eies direkt zum Keimrudiment, das in diesem Stadium allerdings nur etwa die Anlagen für den Kopf einschließlich der gnathalen Segmente enthält (Sarashina et al. 2005). Im wachsenden Keimstreif tritt eine, die drei thorakalen Segmente umfassende, Gb Krüppel- Gap-Domäne auf, deren Inaktivierung mittels RNAi die Hox-Gen-Expression nicht beeinflusst, aber zu weit reichenden Segmentierungsdefekten von T1 bis einschließlich A10 führt (Mito et al. 2006). Wie bei Of hunchback führt auch der Knockdown von Gb hunchback zu einer anterioren Expansion der Gb abd-a-domäne (Mito et al. 2005), während der ab T1 auftretende Segmentierungsdefekt (Mito et al. 2005) u. a. auf die fehlende Aktivierung von Gb Krüppel in Gb hunchback RNAi zurückzuführen ist (Mito et al. 2006). Die Expression des Paarregelgens eve unterscheidet sich interessanterweise innerhalb der Orthoptera deutlich: Während in der Mittelmeergrille Gryllus bimaculatus eine segmentale und im frühen Keimstreif auch vorübergehend eine Paarregel-ähnliche Expression von Gb eve auftritt (Mito et al. 2005), kann man im Grashüpfer Schistocerca americana eine Expression des Sa eve- Orthologs lediglich in der Wachstumszone des Keimstreifs feststellen (Patel et al. 1992). Dennoch verfügt auch Schistocera americana über einen Paarregel-Mechanismus, da zumindest das paired-ortholog Sa pairberry-1 in doppelsegmentalen Streifen exprimiert wird (Davis et al. 2001). 17
36 1.4. Die Segmentierung in anderen Arthropoden Um ein umfassendes Bild über die Evolution der Segmentierung innerhalb der Arthropoden zu erhalten, bedarf es auch der funktionellen Untersuchung von Spezies außerhalb der Klasse der Insekten (Peel et al. 2005). Insgesamt ist dabei die Analyse der Segmentierung in der Kammspinne Cupiennius salei am weitesten fortgeschritten. In diesem Cheliceraten konnten mit Cs hairy, Cs eve, Cs runt, Cs pairberry-3 (paired-ortholog), Cs odd-paired, Cs oddskipped-related-1 und Cs sloppy-paired eine ganze Reihe von Paaregelgenen kloniert werden, allerdings zeigten detaillierte in situ-färbungen eine Abspaltung von segmentalen Streifen in der Wachstumszone, und es konnten keine Hinweise auf einen Paarregel-Mechanismus gefunden werden (Damen et al und 2005; Schoppmeier und Damen 2005a). Von den Gap-Genen konnte lediglich ein Ortholog von Krüppel, Cs Krüppel-1 identifiziert werden, das während der Segmentierung in der Wachstumszone exprimiert ist und für die Erhaltung der Wachstumszone nach der Bildung des vierten opisthosomalen Segments gebraucht wird (Schoppmeier 2003). Überraschenderweise spielt, zusätzlich zu den Orthologen der aus der Drosophila-Segmentierungskaskade bekannten Paarregel- und Segmentpolaritätsgenen, auch der in Vertebraten für die Somitogenese notwendige Notch/Delta-Signaltransduktionsweg (siehe Kapitel 1.5.) eine entscheidende Rolle in der Segmentierung von Cupiennius (Schoppmeier und Damen 2005b; Stollewerk et al. 2003). Der zelluläre Ligand Cs Delta-1 ist ähnlich den "Paarregelgenen" in einem dynamischen, segmentalen Muster exprimiert, und Cs Delta-1-RNAi führt - ebenso wie die Inaktivierung von Cs Notch - zu einer unregelmäßigen posterioren Cs engrailed-expression und zu damit einhergehenden Segmentierungsdefekten. Inwiefern die Übermittlung des Signals aus dem Notch/Delta- Signalweg auf die Segmentierung über die Paarregelgene" geschieht ist noch nicht endgültig geklärt, da nur Cs hairy (das Bindungsstellen für den Transkriptionsfaktor des Notch- Signalwegs, Su(H) enthält) durch den Knockdown des Notch/Delta-Signalwegs beeinträchtigt wird (Schoppmeier 2003). Da auch in Tribolium die posteriore Segmentierung in einer zellularisierten Wachstumszone, vergleichbar mit der Situation in Cupiennius salei, stattfindet, wurde der Notch/Delta- Signalweg auch in Tribolium funktionell untersucht. Dabei konnte jedoch nur eine Funktion in der Beinentwicklung, nicht aber in der Segmentierung nachgewiesen werden (M. Aranda und D. Tautz, pers. Mitteilung). In anderen Arthropoden außerhalb der Insekten wurden noch weitere Expressionsanalysen von Orthologen der Paarregelgene durchgeführt (Davis und Patel 2003), es konnte aber 18
37 lediglich in dem Hundertfüßler Strigamia maritima eine doppelsegmentale Expression eines zu odd-skipped orthologen Gens beobachtet werden (Chipman et al. 2004). Daneben deutet auch das Tu pax3/7-gen (paired-ähnliches Pax3-Gen) in der Milbe Tetranychus urticae einen möglichen Paarregel-Mechanismus an, da ein verzögertes Erscheinen der segmentalen Streifen für die Laufbeinsegmente L1 und L3 beobachtet werden konnte (Dearden et al. 2002). In den Crustaceen, die als unmittelbare Schwestergruppe der Insekten gelten, wird mit der funktionellen Analyse von Segmentierungsgenen gerade erst begonnen. In Artemia franciscana zeigt sich zumindest eine konservierte Funktion des Af caudal-orthologs in der posterioren Elongation (Copf et al. 2004). Neben Artemia könnte sich in der Zukunft möglicherweise auch Parhyale hawaiensis, dessen Entwicklung detailliert beschrieben wurde (Browne et al. 2005) und der für transgene Untersuchungen zugänglich ist (Pavlopoulos und Averof 2005), als weiterer Modellorganismus für die Segmentierung in den Crustaceen etablieren Die Segmentierung in Anneliden und Vertebraten Aufgrund ihrer gemeinsamen Segmentierung wurden die Anneliden ursprünglich gemeinsam mit den Arthropoden zu den Artikulata zusammengefasst. Neuere molekulare Analysen zeigen jedoch eine große phylogenetische Distanz zwischen diesen beiden Phyla (Adoutte et al. 2000), die sich auch in der Sequenz der in beiden Gruppen existierenden Hox-Gene äußert (Balavoine et al. 2002). In dem als Modellorganismus dienenden Polychaeten Platynereis dumerilii wird momentan die Expression von aus Drosophila bekannten Segmentierungsgenen untersucht, und es wurde eine konservierte Expression der Orthologen von wingless und engrailed in jeweils benachbarten Streifen entlang der Segmentgrenzen festgestellt (Prud homme et al. 2003). Daneben erfolgt die posteriore Addition von Segmenten aus einer Wachstumszone, in der die Orthologen von caudal und even-skipped jeweils in einem schmalen Streifen exprimiert werden (de Rosa et al. 2005). Im Gegensatz zu Platynereis dumerilii geht im Blutegel Helobdella robusta die Segmentierung von morphologisch klar unterscheidbaren einzelnen posterioren Stammzellen (Teloblasten) aus, welche nach einem strikten Muster Zelllinien generieren. Dabei kommt es in den Teloblasten zu einer zyklischen Expression von Hro hes (hairy/enhancer of split-ortholog) (Song et al. 2004) und zur Anlagerung der Hro eve-transkripte an mitotisches Chromatin (Song et al. 2002). Die Inaktivierung von Hro eve mittels Injektion eines Antisense-Morpholino- 19
38 Oligonukleotides in einzelne Teloblasten führt zu einer gestörten Teilungsaktivität, und als Folge davon auch zu unregelmäßigen Segmentierungsdefekten, welche aber keiner Paarregel- Periodizität gehorchen (Song et al. 2002). Die Segmentierung der Vertebraten erfolgt nicht im Ektoderm, sondern im Mesoderm und führt zur Unterteilung des präsomitischen Mesoderms in die Somiten, welche die Vorläufer der Wirbel, der Rippen und der segmentalen Skelettmuskulatur darstellen (Pourquié 2001a; Saga und Takeda 2001). Die molekulare Analyse dieses Prozesses ergab, dass in Vertebraten eine Reihe von Genen im präsomitischen Mesoderm durch eine oszillierende Expression gekennzeichnet sind (Pourquié 2001b; Saga und Takeda 2001). Ausgehend von großflächigen Expressionsdomänen im präsomitischen Mesoderm, kommt es bei der Somitogenese zu einer von posterior nach anterior fortschreitenden Verschiebung bei gleichzeitiger Verschmälerung der Expression ( Wanderwelle ), bis schließlich ein weiterer Somit definiert wird und der ganze Zyklus mit einer neuen breiten Expressionsdomäne von Neuem beginnt (Palmeirim et al. 1997). Durch funktionelle Analysen in der Maus, im Zebrafisch und im Huhn konnte ein durch den Notch/Delta-Signalweg vermittelter, negativer Autoregulationsmechanismus als wichtige Komponente für diesen Prozess identifiziert werden (Dale et al. 2003; Dubrulle und Pourduié 2004a; Rida et al. 2004). Diese Funktion des Notch/Delta-Signalwegs ist in Drosophila nicht konserviert, wo dieser Signaltransduktionsweg zwar wichtige Funktionen in der Neurogenese und in der Etablierung von Grenzen in der Bein- und Flügelentwicklung hat, aber keinerlei Einfluss auf die Segmentierungskaskade ausübt (Davis und Patel 1999; Tautz 2004). In Wirbeltieren hingegen spielt der Notch/Delta-Signalweg, dessen Komponenten zum Teil selbst eine oszillierende Expression zeigen (Pourquié 2001b; Saga und Takeda 2001), eine zentrale Rolle in der Regulation des molekularen Oszillators im präsomitischen Mesoderm. Dementsprechend führt der Ausfall dieses Signalwegs zu einer wesentlichen Störung der oszillierenden Genexpression (Dale et al. 2003; Holley et al. 2000; Jiang et al und Jouve et al. 2000), was wiederum die Bildung von deformierten Somiten zur Folge hat (Saga und Takeda 2001). Außerdem zeigen die durch den Notch/Delta-Signalwegs regulierten und zur hairy/enhancer of split-genfamilie in Drosophila verwandten her/hes Gene ebenfalls eine zyklische Expression (Holley et al und Palmeirim et al. 1997) und spielen eine entscheidende Rolle in der zeitlich verzögerten negativen Autoregulation des Oszillators (Bessho et al. 2003; Holley et al. 2002; Oates und Ho 2002). Zusätzlich zur Notch/Delta-Signalkaskade sind auch noch die Wnt- und FGF-Signalwege an der Somitogenese beteiligt. In der Maus ist Wnt-3a als Gradient mit maximaler Expression in 20
39 der Nähe des Hensen schen Knotens exprimiert und für die Aktivität des Notch-Oszillators notwendig (Aulehla et al. 2003). Zusätzlich konnte kürzlich, ebenfalls in der Maus, der Wnt- Transkriptionsfaktor LEF1 als Aktivator für die Expression von Delta-like 1 identifiziert werden (Galceran et al. 2004). Der FGF-8-Gradient spielt hingegen eher eine allgemeine Rolle in der Spezifizierung und Aktivierung der posterioren Oszillationszone sowie deren Abgrenzung von dem anterior davon gelegenen Bereich der Somitendifferenzierung (Dubrulle et al. 2001; Dubrulle und Pourquié 2004b; Kawamura et al. 2005). Außerdem sorgt er, in Zusammenarbeit mit dem molekularen Oszillator, auch noch für die korrekte Expression der Hox-Gene, welche bei den Vertebraten wie bei den Arthropoden die Segmentidentitäten definieren (Deschamps und van Nes 2005; Dubrulle et al. 2001; Dubrulle und Pourquié 2002) Ziele dieser Arbeit Eines der mittelfristigen Ziele der evolutionären Entwicklungsbiologie besteht darin, aus dem Vergleich funktioneller Analysen in verschiedenen Spezies den "Urtyp" der Kurzkeimentwicklung bei Insekten zu definieren und diesen innerhalb der Entwicklungsmechanismen der Arthropoden einzuordnen. Das Ziel dieser Arbeit bestand darin, im Modellorganismus Tribolium castaneum ein umfassendes Bild über die Funktion der Gap-Gen-Orthologen zu gewinnen. Hierzu wurden die Interaktionen zwischen den Gap-Gen-Domänen ermittelt und schon begonnene funktionelle Untersuchungen von Tc Krüppel, Tc giant und Tc hunchback im Hinblick auf deren Funktion in der Regulation von Hox-, Paarregel- und Segmentpolaritätsgenen weiter vertieft. Daneben sollte mit Tc knirps das letzte, noch fehlende Gap-Gen-Ortholog kloniert und analysiert werden. Neben der Analyse von Gap-Gen-Orthologen aus Drosophila war es ein weiteres Ziel, die Gap-Mutante krusty (Maderspacher et al. 1998) detaillierter zu untersuchen und in Zusammenarbeit mit dem Labor von Prof. Sue Brown an der Kansas State University im Hinblick auf die Identifizierung von Kandidatengenen zu kartieren. Außerdem sollte noch der JAK/STAT Signalweg hinsichtlich einer potentiellen Funktion in der zellulären Wachstumszone untersucht werden. 21
40 2. Material und Methoden 2.1. Haltung und Kreuzung von mutanten Tribolium-Stämmen Durch Mutagenesescreens (Maderspacher et al. 1998; Sulston und Anderson 1996) konnten in Tribolium rezessiv letale Entwicklungsmutanten generiert werden, deren Haltung es mangels Balancer-Chromosomen erfordert, alle paar Generationen aufs Neue heterozygote Männchen zu identifizieren (Berghammer et al. 1999b). Die Analyse der Mutanten Tc Kr jaws und Tc krusty mittels in situ-färbungen erfordert den Aufbau einer größeren Population an heterozygoten Elterntieren. Um einen zeitaufwendigen Großansatz an Einzelkreuzungen zu umgehen, wurden zuerst nur 40 heterozygote Käfer mittels Einzelkreuzungen identifiziert. Als nächstes wurden deren zahlreiche Nachkommen im Puppenstadium dem Geschlecht nach sortiert. Die auf diese Art gewonnen Jungfrauen, die statistisch zu 2/3 heterozygot für die Mutation sind, wurden anschließend mit ihren als heterozygot identifizierten Vätern gepaart und liefern in ihren Nachkommen in 1/6 aller Fälle (1/2 x 1/2 x 2/3) homozygote Mutanten. Zur Erzeugung von Tc Kr jaws Tc wurm Doppelmutanten wurden die aus den Nachkommen einer Tc wurm-einzelkreuzung gewonnenen Jungfrauen (zu 2/3 Tc wurm/+) mit Tc Kr jaws heterozygoten Männchen (Tc Kr jaws /+) verpaart. Die F1-Nachkommen dieser Ausgangskreuzung wurden daraufhin im Puppenstadium nach ihrem Geschlecht sortiert. Durch Rückkreuzung mit sowohl Tc Kr jaws - als auch Tc wurm-heterozygoten Weibchen wurden dann Tc Kr jaws -Tc wurm doppelheterozygote Männchen (Tc Kr jaws /+; Tc wurm/+) identifiziert. Diese wurden im nächsten Schritt mit ihren jungfräulichen Schwestern gekreuzt, ehe diese für eine Ablage vereinzelt wurden. Treten in der Ablage eines einzelnen F1- Weibchens dann sowohl Tc Kr jaws - als auch Tc wurm-mutanten auf, so kann das jeweilige Weibchen ebenfalls als doppelheterozygot (Tc Kr jaws /+; Tc wurm/+) identifiziert werden. Abschließend werden die doppelheterozygoten Männchen und Weibchen miteinander verpaart und liefern in 1/16 aller Fälle Doppelmutanten. 22
41 2.2. Kartierung der Mutante krusty Die Segmentierungsmutante krusty wurde im SB-Stamm induziert. Zur Bestimmung des krusty-lokus wurden zuerst von Gregor Bucher männliche, krusty-heterozygote SB-Käfer mit Wildtyp-Weibchen des Tiw1-Stammes gekreuzt (siehe Bucher 2002). Die F1-Nachkommen sind dann an allen Loci SB/Tiw1-heterozygot, und mittels Rückkreuzung mit krustyheterozygoten Weibchen wurden diejenigen Männchen bestimmt, die noch die krusty- Mutation tragen. Diese Männchen wurden nun erneut mit Wildtyp-Weibchen des Tiw1- Stamms gekreuzt, bevor mittels Einzelkreuzungen wieder die krusty-heterozygoten (krusty/+) F2-Käfer identifiziert wurden. Diese F2-Käfer sind nun an allen nicht mit krusty gekoppelten Loci statistisch zu je 50% SB/Tiw1 heterozygot bzw. Tiw1/Tiw1 homozygot. An gekoppelten Loci erwartet man hingegen bis zu 100% SB/Tiw1-heterozygote Käfer, da ja auf das im SB- Chromosom induzierte krusty-allel selektiert wurde. Zur weiteren Untersuchung der (krusty/+) F2-Käfer mit molekularen Markern war die Isolierung von deren DNA erforderlich. Hierzu wurden die einzelnen Käfer bei 20 C eingefroren und dann im gefrorenen Zustand in 200µl Pufferlösung (80mM EDTA ph 8, 100mM Tris ph 8, 0,5% SDS, 100µg/ml Protease K) homogenisiert. Nach 1h Inkubation bei 55 C wurden 200µl 5M NaCl zugesetzt und eine Chloroform-Extraktion durchgeführt. Abschließend wurde die DNA-Lösung noch mittels Microcon-Filter (Millipore) gereinigt und mit 200µl TE eluiert. Im Rahmen einer früheren Kopplungsanalyse konnte Gregor Bucher bereits einen Abstand von 16cM zwischen krusty und einem Marker im zweiten Intron des Tc runt-gens finden (Bucher 2002). Die inzwischen im Labor von Dick Beeman erstellten genetischen Marker (Lorenzen et al. 2005) wurden in anderen Stämmen erstellt, so dass sich davon nur der Marker 50.L10t/ 50.4F05t für die weitere Kartierung von krusty verwenden ließ. Daraufhin wurden neue Primerpaare erstellt, und es konnte nahe dem Marker 16.G01s (15,6 cm von Tc runt enfernt) das Primerpaar ctg 1606f2/r2 für einen PCR-Längenpolymorphismus zwischen den SB- und Tiw1-Stämmen gefunden werden (siehe Kapitel ). 23
42 Die Amplifikation der genomischen DNA mit diesem Primerpaar erfolgte folgendermaßen: 1 µl Ziel-DNA (aus einzelnen Käfern gewonnen) 0,8 µl Primer ctg 1606f2 5 - TGA AAA TCA AAG CAA GAG CAG TG -3 (25pmol/µl) 0,8 µl Primer ctg 1606r2 5 - CAG GTT TTG CCT CAT TTC ATT G -3 (25pmol/µl) 0,8 µl 5mM dntps 1,6 µl 25mM MgCl2 2 µl 10X Puffer 12,85 µl Wasser 0,15 µl Taq Polymerase 20 µl PCR-Zyklus: 94 C 4 min 94 C 30sec 55 C 30sec 35 Zyklen 72 C 30sec 72 C 5 min 2.3. Klonierung von Tc knirps Aus einem Alignment mehrerer Knirps- und Eagle-Proteine in verschiedenen Spezies wurden von Gregor Bucher folgende degenerierte Primer erstellt (Bucher 2002 und Bucher et al. 2005): Tc redup 1: TGY AAR RTN TGY GGN GA (Aminosäuren CKVCGE im ersten Zinkfinger) Tc redup 2: ACN TGY GAR GGN TGY AA (Aminosäuren TCEGCK im ersten Zinkfinger) Tc redlow 6: AR RCA RTG DAT YTT RAA CCA (Aminosäuren WFKIHCL in der kni-box) In einer ersten PCR-Reaktion wurde 3µl cdna, die mittels reverser Transkription aus RNA von 0-24h (bei 32 C) alten Embryonen gewonnen wurde (Bucher 2002), in einer touch-down- PCR mit den äußeren Primern Tc redup 1 und Tc redlow 6 amplifiziert. 94 C 2 min 94 C 30sec 55 C 30sec (Zyklen 1-5), 53 C 30sec (Zyklen 6-10), 50 C 30sec (Zyklen 11-30) 72 C 30sec 72 C 2 min 24
43 0,5µl des PCR-Produkts wurden dann in einer nested-pcr mit den Primern Tc redup 2 und Tc redlow 6 unter denselben Bedingungen amplifiziert. Das 209bp lange PCR-Fragment wurde aus dem Gel ausgeschnitten, mittels Jetsorb (Genomed) aufgereinigt und erneut mit den Primern Tc redup 2 und Tc redlow 6 unter den folgenden Bedingungen amplifiziert. 94 C 2 min 94 C 30sec 45 C 40sec 20 Zyklen 72 C 50sec 72 C 2 min Nach erfolgter blunt-end-ligation mit dem Bluescript-Vektor von Invitrogen (linearisiert mit EcoRV) wurden die transformierten E.coli-Kolonien in 30µl TE-Puffer homogenisiert und 3min bei 94 C inkubiert. Nach Zentrifugation der unlöslichen Bestandteile, wurde 1µl des Überstandes mit der Plasmid-DNA für eine Colony-PCR mit den Vektorprimern T3 und T7 eingesetzt. 94 C 2 min 94 C 30sec 45 C 40sec 30 Zyklen 72 C 50sec 72 C 2 min Da mit den gleichen degenerierten Primern zuvor bereits Tc eagle kloniert worden war (Bucher et al. 2005), wurden 3µl der PCR-Produkte mit der erwarteten Länge (209bp + 168bp Vektorsequenz) zuerst mit Bcl-1 verdaut. Die Bcl-1-Schnittstelle TGATCA liegt in einem nicht konservierten Bereich der Tc eagle-sequenz und ist daher in weiteren Genen der knirps-familie vermutlich nicht konserviert. Nicht geschnittene PCR-Produkte wurden anschließend mit Sepharose-G50-Säulchen gereinigt und von den Primern T3 und T7 ausgehend bei der Firma Seqlab sequenziert, wobei insgesamt 16 mal die Sequenz von Tc knirps/knirps-related identifiziert wurde. Im nächsten Schritt wurde in der Tc knirps/knirps-related-sequenz nach Bereichen mit geringer Konservierung zur Tc eagle Sequenz gesucht und in diesen die Primer für die Klonierung des 5 -Endes mittels RACE (rapid amplification of cdna ends) erstellt. Dabei weisen die vergleichsweise langen 5 -RACE-Primer in Richtung des 5 -Endes des sense- Stranges, wo ein an die cdna ligierter Adapter das Template für die Adapter-Primer (AP) 25
44 darstellt. Die auf diese Art ausgeführte Amplifikation der 5 -Sequenzen gestaltet sich nicht einfach und wird zur Erhöhung der Spezifität in Form von zwei aufeinander folgenden Amplifikationen mit verschachtelten Primern (nested-pcr) unter der Verwendung spezieller Reagenzien (Clontech Advantage 2 Polymerase Mix) durchgeführt: 1. Amplifikation: AP-1: 5 - CCA TCC TAA TAC GAC TCA CTA TAG GGC-3 knirps 5 -RACE: 5 - CCA GTT TGA TCT TCG GCC GTA TCT TGA G-3 94 C 2 min 94 C 30sec 72 C (Zyklus 1-6), 70 C (Zyklus 7-12), 68 C (Zyklus 13-28), 66 C (Zyklus 29-39) je 3 min In dieser Reaktion wird die Hybridisierung der Primer an die zu amplifizierende DNA und die Elongation zu einem Schritt zusammengefasst. 2. Amplifikation: Danach wurde 1µl einer 1:20 Verdünnung der ersten Amplifikation als Ziel-DNA für die zweite Amplifikation (Reaktionsvolumen 20µl) mit den weiter innen liegenden ( nested ) Primern eingesetzt, wobei die Reaktionsbedingungen gegenüber der ersten Amplifikation nicht verändert wurden. AP-2 (nested): 5 - ACT CAC TAT AGG GCT CGA GCG GC-3 knirps 5 -RACE nested: 5 - CAC TTT TTG ACA TAC CGA CCA TCA GAC ATT TC-3 Das auf diese Art gewonnene, knapp 650bp lange, 5 -RACE-Fragment wurde abschließend in den Bluescript-Vektor einkloniert, sequenziert und als Template für die Synthese von antisense-sonden und dsrna verwendet. Eine Amplifikation des 3 -Endes von Tc knirps/knirps-related mittels 3 -RACE war (wie auch bei Tc eagle) nicht erfolgreich. 26
45 Nach Veröffentlichung der Genomsequenz konnten schließlich spezifische Primer zur Amplifizierung des vermuteten 3 -Endes erstellt werden. knirps out up: 5 -CCG AAG CTC GAG ACA TGT G-3 knirps in up (nested): 5 -AGT GTC GCG TGT GTC ACA G-3 knirps out low: 5 -CAA TAC TCG AAT GGA ACA CAG-3 94 C 2 min 94 C 30sec 47 C 40sec 30 Zyklen 72 C 90sec 72 C 2 min Das mit diesen Primern amplifizierte, etwa 1,1kb große, PCR-Fragment wurde ebenfalls in den Bluescript-Vektor (Invitrogen) einkloniert. Die Sequenzierung des Klons ergab, dass dieses ab der knirps-box beginnende Fragment des Tc knirps-gens tatsächlich den gesamten 3 -Bereich einschließlich des Stoppcodons enthält. Mit diesem Template erstellte Sonden und dsrnas bestätigten in einer in situ-färbung und in einer parentalen RNAi exakt die unter Verwendung des 5 -RACE-Klons erzielten Ergebnisse Klonierung von Tc domeless, Tc STAT 92E und Tc SOCS 36E Nach Veröffentlichung der Genomsequenz wurde im Tribolium-BLAST-Server ( die Aminosäuresequenz der Domeless-, STAT 92E- und SOCS 36E-Proteine von Drosophila melanogaster und Anopheles gambia eingegeben und mit Hilfe der Funktion tblastn (diese übersetzt DNA-Sequenzen in der Datenbank in allen Leserastern in Aminosäuresequenzen und vergleicht sie mit einer eingegebenen Aminosäuresequenz) nach Orthologen in Tribolium gesucht. Im Falle von Domeless und STAT 92E konnten so aufgrund von mehreren, konservierten Domänen direkt große Teile der jeweiligen Gene in Tribolium identifiziert werden. SOCS 36E ist hingegen nur in einer kleinen C-terminalen Domäne konserviert. Deshalb wurde mit Hilfe des Programms Gene-JockeyII (Biosoft) nach potentiellen Leserahmen in 5 -Richtung der 27
46 konservierten Domäne gesucht und in einem ca. 550bp langen Leserahmen das Exon für den N-terminalen Teil von Tc SOCS 36E vermutet. Im nächsten Schritt wurden in den vermuteten Exons spezifische Primer erstellt und die jeweiligen Gene mittels (nested)-pcr (Clontech Advantage 2 Polymerase Mix) von cdna amplifiziert. SOCS in up: 5 -AAT GTT CCC CCA AAG ATA AG-3 SOCS in low: 5 -TCA AAC CTC TCC ACT CTC AC-3 STAT out up: 5 -GTT GCC CCC CGA GAG TCT C-3 STAT out low: 5 -CCA CTG ATG GGG TGT CTC TTG-3 STAT in low (nested): 5 -ATC AGC CAG CGA CCT TAT TG-3 domeless out up: 5 -CGG TAC CCC AAG GAG ACA TC-3 domeless in up (nested): 5 -GCG TGC CCT CAT ATT GAC TA-3 domeless out low: 5 -GGG ACG AGG TCA CAC TCT CA-3 domeless in low (nested): 5 -GGG GGA GTT TTA CCT CAA CA-3 94 C 2 min 94 C 30sec 47 C 40sec 30 Zyklen 72 C 3 min 72 C 2 min Das ca. 1,2kb große Tc SOCS 36E-Fragment konnte blunt mit dem Bluescript-Vektor ligiert werden (siehe Klonierung von Tc knirps). Für die circa 2kb großen Tc STAT 92E- und Tc domeless-fragmente wurden hingegen BamHI- (in den up Primern für das 5 -Ende des sense-strangs) und NotI-Schnittstellen (in den low Primern für das 3 -Ende des sense- Strangs) angehängt (in den obigen Primersequenzen weggelassen). 28
47 2.5. Parentale RNAi Zur Synthese von Doppelstrang-RNA wird ein PCR-Produkt von codierenden Bereichen des jeweiligen Gens erstellt, in dem durch einen 5 -Überhang in den Primern die Startsequenz für die T7-RNA-Polymerase an das 5 -Ende beider Stränge eingefügt wird. Im nächsten Schritt wird das PCR-Produkt gereinigt (Fällung oder Mini-Elute-PCR-Purification-Kit von Qiagen) und circa ng davon als Template in eine in vitro-transkription mit einer T7-RNA- Polymerase (T7-Megascript-Kit von Ambion) eingesetzt. Nach einer LiCl-Fällung der beiden RNA Stränge werden diese in DEPC(Diethylpyrocarbonat)-behandeltem Wasser gelöst, bei 94 C denaturiert und durch langsames Abkühlen auf Raumtemperatur (etwa 1h) zur Doppelstrang-RNA hybridisiert. Die Injektion der dsrna (Konzentration: 1-4µg/µl) in die mit dem posterioren Ende aufgeklebten weiblichen Puppen erfolgt ventrolateral an der ersten Segmentgrenze posterior der Flügelspitze (Bucher et al. 2002). Die aus einer rein heterozygot mutanten Elternpopulation hervorgehenden weiblichen F1- Puppen sind zu 2/3 ebenfalls heterozygot. Injiziert man diese Puppen mit dsrna und verpaart die daraus schlüpfenden Käfer mit ihren als heterozygot identifizierten Vätern, so erhält man, neben den reinen RNAi-Phänotypen, in 1/6 aller Fälle auch einen RNAi-Knockdown in einem mutanten Hintergrund. Daneben wurden noch Versuche zur Verstärkung der RNAi-Phänotypen mit Hilfe eines lipophilen Transfektionsreagenz (GenePORTER von Peqlab) durchgeführt. Dabei führte die Injektion eines 1:1-Gemisches aus 0,2µg/µl Tc Krüppel-dsRNA und Transfektionsreagenz (Endkonzentration der dsrna: 0,1µg/µl) zu deutlich stärkeren Phänotypen als die Injektion von 0,1µg/µl Tc Krüppel-dsRNA, nicht aber zu stärkeren Phänotypen als bei der Injektion höherer Konzentrationen (2-4µg/µl) an Tc Krüppel-dsRNA. Die Verwendung höherer Konzentrationen an dsrna in Kombination mit dem für Zellkulturen entwickelten Lipofektionsreagenz GenePORTER wurde vom Hersteller nicht empfohlen und führte zur Bildung von schwerlöslichen Partikeln, welche die Injektionsnadel verstopften. Die zeitlich getrennte Injektion (1h Abstand) von 2µg/µl Tc Krüppel-dsRNA und reinem GenePORTER- Reagenz war zwar technisch problemlos möglich, führte aber zu keiner weiteren Verstärkung der RNAi. Aus diesen Gründen wurden für weitere prnai-experimente keine Transfektionsreagenzien verwendet. Zur Injektion adulter weiblicher Käfer werden diese mit CO 2 betäubt und dann mit Fixogum an den Elytren auf einen Objektträger geklebt. Die Injektion von etwa 0,1-0,15µl dsrna der Konzentration 1µg/µl (bei höheren Konzentrationen kommt es häufig zum Verstopfen der 29
48 Injektionsnadel) erfolgt dann von posterior durch den unter CO 2 -Betäubung geöffneten Chitinpanzer in das hintere Ende des Abdomens. Nach wenigen Stunden bei 32 C lösen sich die Käfer vom Objektträger und ab dem zweiten Tag bei 32 C können Eier mit RNAi- Knockdown-Phänotypen geerntet werden Embryonale RNAi und embryonale Morpholino-Injektionen Die Wildtyp-Eier werden nach einer einstündigen Ablage zuerst eine weitere Stunde bei 32 C inkubiert, bevor sie mit 25% Klorix-Lösung zwei Minuten dechorionisiert und dann mit einem feuchten Pinsel auf einen Objektträger aufgereiht werden. Nach lateraler Injektion an der Luft, werden die Objektträger anschließend bei 32 C in einer feuchten Kammer über einer 15% NaCl-Lösung inkubiert und nach vier Tagen in Hoyers-Milchsäure eingebettet. Zur Untersuchung der Autoregulation von Tc giant wurden 0,3mM des giant-morpholino 5 -CCA TCG CAA ATT CTG CTT TTT CCA T-3 embryonal injiziert (Bucher und Klingler 2004). Für den RNAi-Knockdown von Tc domeless wurden 0,5µg/µl dsrna eines 450bp langen Fragments injiziert, während für die späte, embryonale Injektion der Tc hunchback-dsrna eine Konzentration von 1µg/µl verwendet wurde Erstellung und Photographie von Kutikulapräparaten Die vier Tage bei 32 C gereiften Larven werden mit Klorix vom Mehl befreit, auf einen Objektträger in einen Tropfen mit einem 1:1 Gemisch aus Hoyers-Medium und Milchsäure transferiert und mit einem Deckglas überschichtet. Danach werden die Larven durch vorsichtiges Drücken auf das Deckglas aus den Eihüllen gequetscht. Schließlich werden die Objektträger über Nacht bei 60 C inkubiert, wodurch sich das Gewebe bis auf die Kutikula auflöst, die dann im Dunkelfeld mit dem 10fach Objektiv analysiert und photographiert werden kann. Daneben kann auch die Autofluoreszenz der Kutikula ausgenutzt werden, um die Präparate im TRITC/Cy3-Kanal darzustellen. Dabei können mit Hilfe der Funktion "Z- Stapel Aufnehmen" im Programm Analysis^D auch mehrere Fokusebenen aufgenommen 30
49 werden, aus denen sich durch die Funktion "Projektion" "maximale Intensität" eine Abbildung des Objekts mit großer Tiefenschärfe erzielen lässt (Bäumer 2006) Confocalaufnahmen von Kutikulapräparaten Die gereiften Larven werden in ein Blockschälchen mit Milchsäure/10% Ethanol gegeben und über Nacht bei 60 C inkubiert. Danach wird eine einzelne Kutikula mit einer abgeschnittenen gelben Spitze zum Waschen in ein weiteres Blockschälchen mit Milchsäure/10% Ethanol übertragen und schließlich in einen sich auf einem Objektträger befindenden Tropfen Milchsäure transferiert. Im nächsten Schritt wird nun die Vitellinmembran entfernt und die Kutikula mit der ventralen Seite nach oben ausgerichtet. Abschließend wird ein mit Plastilinfüßchen versehenes Deckglas aufgesetzt und die Kutikula durch sanften Druck auf das Deckglas vorsichtig zwischen Objektträger und Deckglas eingeklemmt. Durch seitliches Verschieben des Deckglases lässt sich die Kutikula noch in die optimale Position rollen. Bei der Verwendung von Wasserobjektiven (höhere Auflösung als Luftobjektive) wird das Protokoll wie folgt verändert: Zuerst wird ein doppelseitiger, nicht fluoreszierender Tesafilm auf einen Objektträger aufgeklebt. Dann wird ein Tropfen Milchsäure auf den Tesafilm aufgetragen, in den nun die geklärte Larve pipettiert wird. Als nächstes wird die Eihülle vorsichtig auf den Tesafilm geklebt, die Kutikula herauspräpariert und an einer anderen Stelle mit der ventralen Seite nach oben auf dem Tesafilm fixiert. Abschließend ersetzt man die Milchsäure durch Wasser, indem man mehrfach Teile des Tropfens absaugt und frisches Wasser hinzufügt. Folgende Einstellungen des Confocalmikroskops wurden verwendet: Objektiv: 10X oder 20X Anregungswellenlänge: 488nm Detektionsspektrum: nm Anzahl der Ebenen: je nach Präparat zwischen 50 und 80 Bildverarbeitung: maximal projection (verwendet aus allen Fokusebenen für jeden Bildpunkt nur das jeweils stärkste Signal) 31
50 2.9. Fixierung und in situ-färbungen Die Embryonen für in situ-färbungen werden nach zweifachem, je dreiminütigem Dechorionisieren in unverdünntem Klorix in 2ml PEMS (0,1M Pipes, 2mM MgSO 4, 1mM EDTA; ph 6,9), 6ml Heptan und 300µl Formaldehyd für 40min fixiert. Anschließend wird die PEMS-Phase durch 8ml Methanol ersetzt und mittels sofortigem, etwa 10-sekündigem und heftigem Schütteln die frühen Embryonalstadien devitellinisiert, während spätere Stadien zur Devitellinisierung mehrmals durch eine Spritze (Nadeldurchmesser 1,1mm) gedrückt werden müssen. Diese sehr schnelle Methode führt allerdings zur Zerstörung der meisten Embryonen in der späten Segmentierungsphase (15-20h bei 32 C) und funktioniert auch nicht bei zuvor durch eine embryonale Injektion angestochenen Eiern. Deshalb wurden die mit dem Tc giant-morpholino injizierten Eier (siehe Abb. 26E, F) sowie die späten Tc Krüppel-pRNAi Keimstreifen für die Tc eve/tc wingless-doppelfärbung (siehe Abb. 20B-D) wie folgt manuell präpariert: Die Embryonen werden in Ethanol auf einen doppelseitigen Klebestreifen überführt und mit ihrer Vitellinmembran daran befestigt. Anschließend wird das Ethanol durch Wasser ersetzt, wodurch sich die Adhäsion der Vitellinmembran an den Klebestreifen soweit erhöht, dass die Embryonen mit Hilfe einer Insektennadel frei präpariert werden können. Abschließend überführt man die Embryonen zügig in Methanol und lagert sie darin bei -20 C. Die in situ-detektion der Genexpression erfolgt mit Digoxigenin- bzw. Fluoresceinmarkierten antisense-sonden und Alkalischer-Phosphatase- bzw. beta-galaktosidasekonjugierten Antikörpern (Tautz und Pfeifle 1989; Prpic et al. 2001). Dabei wird das Signal für die beta-galaktosidase-färbung zuvor mittels des TSA-Biotin-Systems von Perkin-Elmer verstärkt. 32
51 3. Ergebnisse 3.1. Die Klonierung und Analyse von Tc knirps Die zu Beginn dieser Arbeit bereits publizierten Expressionsdomänen der Gap-Gen- Orthologen Tc Krüppel (Sommer und Tautz 1993), Tc giant (Bucher und Klingler 2004), Tc hunchback (Wolff et al. 1995) und Tc tailless (Schröder et al. 2000) lassen zwischen der posterioren Tc giant-domäne (A2) und der posterioren Tc hunchback-domäne (A6) eine Lücke frei. Deshalb sollte aus Tribolium noch das Ortholog des in Drosophila von A2-A6 exprimierten Gap-Gens knirps als mögliches abdominales Gap-Gen kloniert werden Klonierung und Sequenzanalyse Das Gap-Gen Dm knirps gehört zu den Zinkfinger-Transkriptionsfaktoren und bildet zusammen mit Dm knirps-related sowie dem in der Keimbahn exprimierten Dm eagle-gen die knirps-familie, deren DNA-Bindungsdomäne Ähnlichkeiten mit den nukleären Hormon- Rezeptoren von Vertebraten aufweist (Oro et al und Rothe et al. 1989). Die Proteinsequenzen von Eagle einerseits und Knirps/Knirps-related andererseits unterscheiden sich innerhalb der stark konservierten Zinkfinger- und knirps-box-domänen in verschiedenen Spezies nur unwesentlich (Abb. 3). Deshalb wurden zur Klonierung von Tc knirps/knirpsrelated dieselben degenerierten Primer verwendet, die bereits Gregor Bucher für die Klonierung des anterior lokalisierten Tc eagle-gens verwendet hatte (Bucher et al. 2005; siehe Material und Methoden). Dabei wurden unter Plasmiden mit einklonierten PCR- Fragmenten zuerst mittels eines Bcl-I-Verdaus die Tc eagle-klone ermittelt, um die Sequenzierung weiterer Tc eagle-fragmente zu vermeiden (siehe Material und Methoden). Unter den restlichen Klonen trat insgesamt 16mal eine gegenüber Tc eagle leicht veränderte Sequenz auf, aus der schließlich die RACE-Primer zur Verlängerung der Sequenz konstruiert wurden (siehe Material und Methoden). Dabei lieferte die 5 -RACE-Amplifikation ein 650bp langes Fragment, während die 3 -RACE Amplifikation (wie schon bei Tc eagle) vermutlich wegen sehr GC-reicher Sequenzabschnitte keine verwertbaren Ergebnisse produzierte. Die funktionelle Analyse von Tc knirps/knirps-related (Tc kni/knrl) erfolgte zunächst mit dem 650bp langen 5 -RACE-Fragment, wobei ein nach Erscheinen der genomischen Sequenz mit spezifischen Primern erstelltes 1,1kb-Fragment des 3 -Endes bei in situ-färbungen und RNAi-Knockdowns exakt die gleichen Ergebnisse lieferte. 33
52 Nachdem sich das klonierte Gen in der abdominalen Segmentierung von Tribolium als funktionslos herausstellte, wurden noch weitere Klone sequenziert, wobei allerdings weder unter den Bcl-I geschnittenen noch unter den Bcl-I ungeschnittenen Klonen ein weiteres Gen der knirps-familie erschien (insgesamt 16 Tc kni/knrl-klone (Bcl-I ungeschnitten) und 27 Tc eagle-klone (Bcl-I geschnitten)). Nach Erscheinen der genomischen Sequenz von Tribolium wurde außerdem eine tblastn-analyse durchgeführt (Genomsequenzen aus der Datenbank werden in allen Leserastern in Aminosäuresequenzen übersetzt und mit einer eingegebenen Aminosäuresequenz verglichen), die neben Tc eagle und Tc kni/knrl keine weiteren Gene aus der knirps-familie identifizieren konnte. Auch im Genom der Malariamücke Anopheles gambia resultiert eine tblastn-analyse in nur zwei Genen der knirps-familie (Ag eagle und Ag kni/knrl), so dass die Duplikation zu knirps und knirpsrelated wohl erst nach der Abspaltung der höheren Dipteren entstanden ist. Das neu klonierte Gen ist damit als gemeinsames Ortholog von knirps und knirps-related anzusehen und wird daher beim Vergleich der Gene als Tc knirps/knirps-related (Tc kni/knrl) bezeichnet, während später bei der funktionellen Analyse vereinfachend nur von Tc knirps gesprochen wird. In Abb. 3 wird die gesamte Sequenz des Tc Knirps/Knirps-related-Proteins mit dem Kni/Knrl-Protein in Anopheles gambia, dem Knirps-related- und dem Knirps-Protein in Drosophila melanogaster sowie mit den Eagle-Proteinen in Tribolium und Drosophila verglichen. Das Dm Knirps-related-Protein weist neun zusätzliche, N-terminale Aminosäuren auf, so dass die Aminosäuren den As 1-96 der anderen Proteine homolog sind. Es zeigt sich insgesamt eine sehr hohe Konservierung der aus zwei Cys-Cys/Cys-Cys- Zinkfingern sowie der knirps-box bestehenden DNA-Bindungsdomäne (Abb. 3). Eine genauere Betrachtung der DNA-Bindungsdomäne zeigt aber, dass es einzelne, für die Eagle- Proteine spezifische Aminosäuren gibt, und dass die Kni/Knrl-Proteine von Tribolium und Anopheles dem Dm Knirps-related-Protein ähneln, wohingegen die Aminosäuresequenz von Dm Knirps doch einige Abweichungen zeigt. Diese Einschätzung bestätigt sich in einer mit dem Programm "Phylip erstellten molekularen Stammbaumanalyse der konservierten Sequenz der DNA-Bindungsdomäne (Abb. 4). Außerhalb der DNA-Bindungsdomäne können noch vier konservierte Motive identifiziert werden, wobei die Übereinstimmung mit der Aminosäuresequenz von Tc Knirps am geringsten ist (Abb. 3). Damit zeigt sich, dass das knirps-related-gen wohl das evolutionär ursprünglichere ist, während sich das nur in den höheren Dipteren vorkommende und aus einer Duplikation entstandene knirps-gen divergent evolvieren konnte. 34
53 Im Dm Knirps-Protein konnte mit Hilfe einer Serie von mutanten Allelen und transgenen Konstrukten gezeigt werden, dass die Region zwischen FQLPP (Mitte Motiv 2) und VESQN (Ende Motiv 3) wichtig für die Funktion dieses Proteins als transkriptioneller Repressor ist (Gerwin et al. 1994). Diese Region kann auch in den anderen Proteinen identifiziert werden, wobei die Sequenz von Tc Knirps die größten Abweichungen zeigt (Abb. 3). Im Gegensatz zu dieser Region ist die biochemische Funktion der konservierten Motive eins und vier noch unklar. Ein weiterer Unterschied zwischen Dm knirps und Dm knirps-related besteht in der mit 21kb recht großen Länge des Primärtranskripts von Dm knrl, wodurch die Transkription dieses Gens zu langsam für die schnell verlaufende Musterbildung im syncytialen Blastoderm erfolgt (Rothe et al. 1992). Das Tc knirps/knirps-related-gen besitzt mit 13,7kb und 8,1kb Länge ebenfalls zwei relativ große Introns, deren Lage zu den beiden kurzen Introns im Dm knirps- Gen konserviert ist (Abb. 5). Im Dm knirps-related-gen treten sogar drei Introns auf (3,3kb, 15,1kb und 2,2kb), wobei die Lage des letzten Introns mit den anderen Genen übereinstimmt (Abb. 5). Daneben stimmt auch das 3 -Ende des zweiten Introns von Dm knrl mit dem 3 - Ende des ersten Introns von Tc kni/knrl und Dm knirps überein (Abb. 5). Somit ist im Dm knrl-gen vermutlich im ursprünglich ersten Intron ein weiteres Exon entstanden, welches für die zusätzlichen, N-terminalen Aminosäuren codiert. Insgesamt ist das Tc Knirps/Knirps-related-Protein mit 330 Aminosäuren deutlich kürzer als die 647 bzw. 429 Aminosäuren umfassenden Dm Knirps-related- und Dm Knirps-Proteine. Damit bestätigt sich die schon bei zahlreichen anderen Transkriptionsfaktoren beobachtete Tendenz zu deutlich längeren Genprodukten in Drosophila im Vergleich zu Tribolium (Schmid und Tautz 1999). 35
54
55
56 Abbildung 4: Phylogramm der knirps-familie-proteine Mit ClustalW-Phylip erstellte Stammbaumanalyse aus den ersten 96 Aminosäuren (in Dm Knirps-related: As ) der in Abb. 3 verglichenen Proteinsequenzen. Man erkennt, dass die Knirps/Knirps-related-Proteinsequenzen aus Tribolium und Anopheles eine deutlich nähere molekulare Verwandschaft zum Dm Knirps-related- als zum Dm Knirps-Protein aufweisen. Die Eagle-Proteine von Tribolium und Drosophila bilden eine eigene Gruppe, was auf eine phylogenetische alte Aufspaltung zwischen den knirps-related- und eagle-genen hindeutet. Abbildung 5: Die Struktur der Tc kni/knrl-, Dm knirps- und Dm knirps-related-gene Schematische Darstellung der drei Exons von Tc kni/knrl und Dm knirps sowie der vier Exons im Dm knirps-related-gen. Oberhalb der als gerade Linien dargestellten Exons ist die Nukleotidsequenz angeordnet. Darüber sind die von den jeweiligen Tripletts codierten Aminosäuren und ihre Position im Protein dargestellt. Die Lage des ersten Introns ist zwischen Tc kni/knrl und Dm knirps konserviert. Man beachte, dass das Dm Knirps-related- Protein am N-terminalen Ende neun zusätzliche Aminosäuren enthält (Abb. 3). Dadurch ist das Codon für das erste Methionin in Tc kni/knrl und Dm knirps homolog zu dem Codon für das Methionin an der 10. Position in Dm knirps-related. Somit entspricht das dritte Exon in Dm knirps-related dem zweiten Exon in Tc kni/knrl und Dm knirps. Die Lage des letzten Introns ist in allen drei Genen konserviert. Die Länge der Introns ist im Dm knirps-gen gegenüber Tc kni/knrl und Dm knirps-related deutlich verkürzt. 38
57
58 40
59 Expression von Tc knirps In Drosophila sind Dm knirps und Dm knirps-related nahezu identisch exprimiert und zeigen während der frühen Entwicklung eine Aktivität in der Kopfanlage und in den Anlagen der Segmente A2 bis A6 (Nauber et al und Rothe et al. 1994). In Tribolium ist Tc knirps/knirps-related (im Folgenden nur noch Tc knirps genannt) im Verlauf der Segmentierung ebenfalls in zwei Domänen exprimiert (Abb. 6, 11A-C, 13A-D). Von den beiden Domänen entsteht, wie schon bei Tc giant und Tc hunchback, eine anteriore im Blastodermstadium (Abb. 6) und eine posteriore während des Keimstreifwachstums (Abb. 7A-C). Anders als bei Tc giant und Tc hunchback, ist die früheste anteriore Tc knirps- Expression aber nicht ubiquitär. Vielmehr bleiben beide Pole von Anfang an frei von Tc knirps-transkripten (Abb. 6A). Im weiteren Verlauf verschwindet die Expression von Tc knirps aus dem posterioren Bereich des Blastoderms, und es bildet sich eine scharfe, zur anterior-posterioren Achse senkrecht stehende posteriore Grenze bei etwa 40-45% (von anterior aus betrachtet) der Eilänge (Abb. 6C, E, G). Parallel dazu präzisiert sich auch die anteriore Expressionsgrenze, die nebenher im dorsalen Bereich gleichzeitig nach posterior kippt, so dass sich schließlich eine in der lateralen Ansicht dreieckige, ventral breitere, anteriore Tc knirps-domäne bildet (Abb. 6G). Ein ähnliches Kippen der anterioren Expressionsgrenze tritt auch bei anderen, im anterioren Blastoderm exprimierten Genen wie Tc giant und Tc otd (Abb. 26A, B und 32A, B) auf und spiegelt die Verlagerung der Serosaanlage von anterior nach anterior-dorsal wider. Allerdings zeigen die Höchst Färbungen der jeweiligen Embryonen (Abb. 6B, D, F, H) zwar die beginnende posteriore Invagination der spätesten Embryonen (Abb. 6G, H), aber noch keine Unterschiede zwischen der Serosa und dem embryonalen Gewebe. Möglicherweise stellt die anteriore Grenze der Tc knirps-domäne im Blastoderm also einen frühen Marker für die Grenze zwischen der Serosa- und der Embryoanlage dar, der in seiner Expression der morphologischen Differenzierung dieser Gewebe in noch späteren Blastodermstadien vorausgeht. In späten Blastodermstadien mit deutlicher posteriorer Invagination schrumpft die anteriore Tc knirps- Domäne mit Ausnahme des ganz ventralen Bereichs zu einem schmalen Streifen zusammen (Abb. 8G, H und 13A, E, I, M), während die posteriore Tc knirps-domäne ähnlich wie die posteriore Tc giant-domäne erstmals im posterior pit erscheint (Abb. 9A). In frühen Keimstreifstadien setzt sich die Expression der anterioren Tc knirps-domäne noch vorübergehend in Form eines leicht gebogenen, schmalen Streifens posterior der Kopflappen fort (Abb. 7A-C und 13A-D). 41
60 Die posteriore Tc knirps-expression erscheint in präparierten und gequetschten Keimstreifen als eine Domäne, aus der sich etwas später ein schmaler Streifen herausbildet (Abb. 9B-D). Eine genaue Betrachtung der unpräparierten und nicht gequetschten Keimstreifen zeigt jedoch, dass sich diese Domäne in Wirklichkeit aus zwei Expressionsmustern zusammensetzt (Abb. 7A-C): einem schmalen Streifen im Ektoderm (schwarze Pfeilspitzen in Abb. 7A-C) sowie einer vorübergehenden Expression im posterioren Bereich des invaginierenden Mesoderms (Pfeile in Abb. 7A-C). In früheren Keimstreifstadien liegen beide Tc knirps-expressionsbereiche in etwa auf gleicher Höhe (bzgl. der anterior-posterioren Achse) und überlagern sich somit (Abb. 7A, B und 9B, C). In etwas späteren Keimstreifstadien kommt es hingegen zur deutlichen Trennung der beiden Expressionen, da die Aktivität im posterioren Mesoderm dem wachsenden Keimstreif folgt, wohingegen die Lage des ektodermalen Tc knirps-streifens mehr oder weniger unverändert bleibt (Abb. 7C und 9D). Kurz darauf verschwinden sowohl die ektodermale als auch die mesodermale Expression, womit die posteriore Tc knirps-expression (Abb. 7A-C) deutlich kurzlebiger als die im selben Bereich exprimierte posteriore Tc giant-domäne ist. Die späte Expression von Tc knirps um die Invagination des Stomodeums (Abb. 7D), am terminalen Ende des Keimstreifs (Abb. 7D, E) und im sich entwickelnden Tracheensystem (Abb. 7E, F) weist wiederum große Ähnlichkeit mit der späten Expression von Dm knirps und Dm knirps-related auf (Chen et al. 1998; González-Gaitán et al. 1994). Abbildung 6: Expression von Tc knirps im Blastoderm In situ-ap-färbung gegen Tc knirps in Blastodermembryonen (A, C, E, G), die in B, D, F, H mit Höchst gegengefärbt wurden. Ausgehend von einer breiten Expression in der Mitte des Blastoderms (A), zieht sich die Expression von Tc knirps in die anteriore Hälfte zurück (C). Im weiteren Verlauf der Blastodermentwicklung werden schärfere Grenzen der Tc knirps Domäne ausgebildet (E, G). Dabei ist in späten Stadien die anteriore, nicht aber die posteriore Grenze leicht in Richtung der dorso-ventrale Achse geneigt (G). Im Gegensatz zur Dynamik in der Expression von Tc knirps, zeigt sich in den dazugehörigen Höchst Färbungen keine größere Veränderung in der Zellmorphologie zwischen Serosa- und Embryoanlage (B, D, F, H). Die in situ-färbungen (A, C, E, G) wurden als optische Schnitte durch die Mittelachse photographiert, bei den dazugehörigen Höchst Färbungen (B, D, F, H) wurde dagegen auf die Oberfläche fokussiert. Anterior ist immer links und ventral ist immer unten. 42
61
62 Abbildung 7: Expression von Tc knirps im Keimstreifstadium In situ AP-Färbung gegen Tc knirps in frühen, unpräparierten (A-C) sowie in älteren, präparierten Keimstreifen (D-F). Die anteriore Tc knirps-expression besteht in frühen Keimstreifen als schmaler Streifen posterior der Kopflappen fort (graue Pfeilspitzen in A-C). Im posterioren Bereich tritt in jungen Keimstreifen eine Überlagerung zwischen einer ektodermalen streifenförmigen Tc knirps-expression (schwarze Pfeilspitze in A) und einer (bzgl. der anterior-posterioren Achse) auf gleicher Höhe liegenden Expression im invaginierenden Mesoderm (Pfeil in A) auf. In etwas späteren Stadien bleibt der posteriore Tc knirps-streifen (schwarze Pfeilspitzen in B, C) in etwa an seiner Position, während die Expression im posterioren Mesoderm (Pfeile in B, C) dem weiter wachsenden Keimstreifen folgt und damit hinter den posterioren Streifen wandert. Während der weiteren Keimstreifverlängerung verschwindet die posteriore Expression. In ausgewachsenen Keimstreifen tritt um das Stomodeum (Pfeilspitzen in D) sowie am posterioren Ende des Keimstreifs (Pfeil in D und E) eine neue Tc knirps-expression auf. Mit beginnender Keimstreifverkürzung erscheint eine weitere Expression von Tc knirps in den Plakoden des Tracheensystems (Pfeilspitze in E), die im weiteren Verlauf der Tracheenentwicklung ein komplexes Muster annimmt (F). Anterior ist immer links; sowohl die unpräparierten als auch die präparierten Keimstreifen wurden in ventraler Aufsicht photographiert. 44
63
64 Expression von Tc knirps in Relation zu anderen Segmentierungsgenen Zur genaueren Lokalisierung der beiden während der Segmentierung exprimierten Domänen von Tc knirps wurden Doppelfärbungen mit weiteren Segmentierungsgenen durchgeführt. In der Expression der anterioren Domäne von Tc knirps zeigt sich dabei eine vergleichbare Dynamik wie in der Expression der anterioren Domäne von Tc giant (Abb. 8A, C, E, G). Beide Domänen sind anfänglich überlappend exprimiert und ziehen sich in etwa synchron aus dem posterioren Blastoderm zurück (Abb. 8A, C). In späten Blastodermstadien bildet sich aus der anterioren Tc giant-domäne ein kräftiger Streifen im maxillären Segment, der unmittelbar posterior des späten, anterioren Tc knirps-streifens zu liegen kommt (Abb. 8E, G und 9I). In einer Doppelfärbung mit dem Paarregelgen Tc eve zeigt sich, dass die anteriore Tc knirps- Domäne stets unmittelbar anterior der ursprünglich breiten Tc eve-domäne (Abb. 8B) bzw. des daraus hervorgehenden ersten Tc eve-streifens liegt (Abb. 8D, F, H). Nach Aufspaltung von eve1 in die segmentalen Streifen eve1a und 1b liegt der anteriore Tc knirps-streifen unmittelbar vor dem eve1a-streifen (Abb. 9M, N), der die anteriore Grenze des ersten Parasegments und das posteriore Ende des mandibulären Segments markiert. Damit deckt die anteriore Tc knirps-domäne im Laufe der Entwicklung die Anlagen für alle Kopfsegmente vor dem posterioren Teil des mandibulären Segments ab. Abbildung 8: Expression von Tc knirps, Tc giant und Tc eve im Blastoderm In-situ-Doppelfärbungen gegen Tc knirps (braune AP-Färbung) und Tc giant (blaue β-gal- Färbung) (A, C, E, G) sowie gegen Tc knirps (braune AP-Färbung) und Tc eve (blaue β-gal- Färbung) (B, D, F, H). Die anteriore Tc knirps-domäne überlappt mit der anterioren Tc giant- Domäne (A, C, E; G), während das posteriore Ende der Tc knirps-domäne genau an das anteriore Ende der frühen Tc eve-domäne bzw. des ersten Tc eve-streifens angrenzt (B, D, F, H). In früheren Blastodermstadien (A, B) sind Tc eve und Tc giant in breiten Domänen im posterioren Bereich exprimiert, wobei Tc giant, nicht aber Tc eve, noch mit Tc knirps überlappt. Etwas später zieht sich die Expression von Tc giant und Tc eve in etwa synchron vom posterioren Pol zurück (C, D). Während sich später die anteriore Grenze von Tc knirps neigt und die Expressionsdomäne schmäler wird (E, G, bzw. F, H), bildet sich innerhalb der anterioren Tc giant-domäne der kräftigere Streifen im maxillären Segment heraus (Pfeilspitze in G), der schließlich posterior an die Tc knirps-domäne angrenzt (G). Im Falle von Tc eve liegt die anteriore Grenze des reifenden ersten eve-streifens genau am posterioren Ende der Tc knirps-domäne (F, H), während weiter posterior der zweite eve-streifen entsteht. Anterior ist immer links und ventral ist unten. e1, e2 = eve-streifen 1,2. 46
65
66 Die posteriore Tc knirps-aktivität wird erstmals in der posterioren Invagination ( posterior pit ) sichtbar (Abb. 9A, E, I, M). In frühen, prärarierten Keimstreifen überlagert sich die Expression des schmalen, ektodermalen Tc knirps-streifens mit der Expression im invaginierenden posterioren Mesoderm (Abb. 9B, C; siehe auch Abb. 7A, B), so dass erst in etwas späteren Keimstreifstadien die Lage des ektodermalen Tc knirps-streifens gut erkennbar wird (Abb. 9D; siehe auch Abb. 7C). Dieser Streifen liegt stets unmittelbar posterior der Gap-Domäne von Tc Krüppel (Abb. 9E-H), welche die Anlagen bis einschließlich des dritten thorakalen Segments umfasst. Eine funktionelle Untersuchung der Regulation von Tc knirps durch Tc Krüppel ergab außerdem, dass nur die ektodermale Expression von Tc Krüppel aktiviert wird (siehe Abb. 15 in Kapitel ), so dass von einer unterschiedlichen Regulation der beiden posterioren Tc knirps-domänen ausgegangen werden kann. In mit Tc giant-doppelgefärbten Embryonen ist die Lage des posterioren Tc knirps-streifens innerhalb der posterioren Tc giant-domäne in frühen Keimstreifstadien schwer erkennbar (Abb. 9I-K), bevor sich in späteren Stadien eine Überlappung mit etwa 50% des vorderen Tc giant-streifens zeigt (Abb. 9L). Abbildung 9: Expression von Tc knirps, Tc Krüppel, Tc giant und Tc eve im späten Blastoderm und in frühen Keimstreifen In situ-ap-färbung gegen Tc knirps (A-D) sowie in situ-doppelfärbungen gegen Tc knirps (braune AP-Färbung in E-P) und Tc Krüppel (blaue β-gal-färbung in E-H), Tc giant (blaue β-gal-färbung in I-L) sowie Tc eve (blaue β-gal-färbung in M-P). Die anteriore Tc knirps- Domäne verschmälert sich in späten Blastodermstadien zu einem Streifen (A), der in frühen Keimstreifen schließlich unmittelbar posterior der Kopflappen liegt (B-D). Die posteriore Tc knirps-expression taucht erstmals im so genannten posterioren pit auf (A) und bildet im Keimstreif eine aus mesodermaler und ektodermaler Expression zusammengesetzte Färbung (B, C), die sich in späteren Stadien in den ektodermalen Streifen und die mesodermale Expression auftrennt (D, siehe auch Abb. 7C). Im späten Blastoderm überlappen die Expressionen von Tc Krüppel und Tc knirps im posterioren Pol (E), bevor in den Keimstreifstadien die posteriore Tc knirps-domäne exakt posterior der Tc Krüppel-Domäne zu liegen kommt (F-H). Der späte, anteriore Tc knirps-streifen (schwarze Pfeilspitze in I) liegt unmittelbar anterior des kräftigen Streifens in der anterioren Tc giant-domäne (graue Pfeilspitze in I). Die posteriore Tc knirps-expression befindet sich anfänglich innerhalb der sich aufspaltenden posterioren Tc giant-domäne (J, K), während der ektodermale Tc knirps- Streifen später etwas mit dem posterioren Teil des vorderen Tc giant-streifen überlappt (L). Nach Aufspaltung des ersten Tc eve-streifens liegt der segmentale eve1a-streifen unmittelbar posterior der späten anterioren Tc knirps-domäne (M-O). Die posteriore Tc knirps- Expression bildet sich hinter dem eve3-streifen aus (N, O), und der ektodermale Streifen wird schließlich in der Lücke zwischen eve3 und eve4 exprimiert (schwarze Pfeilspitze in P). Anterior ist immer oben; in den Blastodermstadien (A, E, I, M) ist ventral rechts. 48
67
68 Das Paarregelgen Tc eve bildet seinen dritten Streifen vor der posterioren Tc knirps- Expression aus (Abb. 9N-P), während der eve4-streifen unmittelbar hinter dem schmalen, ektodermalen Tc knirps-streifen erscheint (Abb. 9P). Somit weist der posteriore Tc knirps- Streifen eine Breite von etwa einer Segmentanlage auf. Da die Expression des posterioren Tc knirps-streifens rasch wieder verschwindet, kommt es nicht zur Koexpression mit den sekundären, segmentalen Tc eve-streifen, die üblicherweise zur Kartierung von Gap-Gen- Domänen in Tribolium verwendet werden. Dennoch kann der posteriore Tc knirps-streifen indirekt auf die Anlage des ersten abdominalen Segments kartiert werden, da er unmittelbar an das hintere Ende der Tc Krüppel-Domäne angrenzt, welche wiederum dem posterioren Ende der Anlage für das dritte thorakale Segment entspricht (Cerny et al. 2005). Demzufolge müsste der posteriore Tc knirps-streifen eigentlich in der Lücke zwischen den Streifen der posterioren Tc giant-domäne liegen, welche in ihrer späten Expression anhand der sekundären Tc eve-streifen auf die Segmentanlagen T3 und A2 kartiert wurden (Bucher und Klingler 2004). Im Zuge der Segmentierung beobachtet man jedoch eine in Relation zu Tc eve leichte anteriore Verschiebung der posterioren Tc giant-streifen, weshalb der in früheren Stadien minimal posteriorer gelegene vordere Tc giant-streifen partiell mit dem posterioren Tc knirps-streifen überlappt. Mit der Expressionsanalyse von Tc knirps konnte ein weiteres Gap-Gen-Ortholog in Tribolium beschrieben werden, wodurch die Liste der bekannten Gap-Gen-Orthologen in Tribolium vervollständigt wurde (siehe Abbildung 47 im Kapitel 4.2.). Insgesamt weist die späte Expression der im Blastoderm von Tribolium gebildeten anterioren Domänen von hunchback, knirps und giant (sowie die ebenfalls während des Blastodermstadiums definierte anteriore Grenze von Krüppel) eine große Ähnlichkeit zu den jeweiligen Domänen in Drosophila auf. Im Gegensatz dazu zeigen die während des zellulären Keimstreifwachstums definierten posterioren Domänen von Tc knirps und Tc giant (sowie die posteriore Grenze von Tc Krüppel) eine deutliche Verschiebung nach anterior gegenüber den im Langkeim- Blastoderm gebildeten Domänen ihrer Orthologe in Drosophila. Einen interessanten Aspekt in der Expression von Gap-Genen in Tribolium stellt der von keinem Gap-Gen-Ortholog abgedeckte Bereich der Segmentanlagen A2-A5 dar (Abb. 47). In Drosophila sorgen überlappende Diffusionsgradienten von Gap-Gen-Transkriptionsfaktoren über die gesamte Eilänge für die Positionsinformation zur Regulation der Paarregelgene. Im zellulär wachsenden Keimstreif von Tribolium erscheint das Konzept der weiträumig diffundierenden Transkriptionsfaktoren aber recht unwahrscheinlich, so dass entweder die nach anterior verschobenen Gap-Gen-Domänen über einen neuen Mechanismus die weiter posterior 50
69 gelegenen Segmente spezifizieren, oder aber neue, noch unbekannte Gene an der Segmentierung beteiligt sein könnten. Zur Klärung dieser Frage wurden im Rahmen dieser Arbeit die Gap-Gen-Orthologe Tc knirps, Tc Krüppel, Tc giant und Tc hunchback sowie die Gap-Mutante krusty funktionell untersucht bzw. schon begonnene Untersuchungen weiter vertieft. Für das Tribolium-Ortholog des terminalen Gap-Gens tailless konnte keine Segmentierungsfunktion gefunden werden (R. Schröder, pers. Mitteilung), weshalb dieses Gen im Rahmen dieser Arbeit nicht weiter untersucht wurde Phänotyp von Tc knirps-rnai-larven Die funktionelle Analyse von Tc knirps erfolgte mittels parentaler RNAi (siehe Material und Methoden). Dabei konnte in den Tc knirps-rnai-larven eine Deletion der Antennen sowie der Mandibeln beobachtet werden, wohingegen sowohl das weiter anterior gelegene Labrum als auch die weiter posterior gelegenen maxillären und labialen Segmente nicht von der Deletion betroffen sind (Abb. 10). Abbildung 10: Der Kopfdefekt in Tc knirps-rnai-larven Confocalaufnahmen von Kutikulapräparaten einer Wildtyp-Larve (A) und einer Tc knirps- RNAi-Larve (B). Im Kopf der Wildtyp-Larve (A) erkennt man von anterior nach posterior das Labrum (Lr), die Antennen (Ant), die klauenförmigen Mandibeln (Md), die Maxillen (Mx) und das aus einer ventralen Fusion zweier Palpen gebildete Labium (Lb). In der Tc knirps- RNAi-Larve (B) fehlen die Antennen und die Mandibeln, während das anterior der Deletion gelegene Labrum (Lr) sowie die posterior der Deletion gelegenen Maxillen (Mx) und Labialpalpen (Lb) unverändert sind. Anterior ist in beiden Aufnahmen oben. 51
70 Während diese Phänotypen gut mit der anterioren Tc knirps-domäne, welche die Kopfanlage bis einschließlich dem anterioren Teil des mandibulären Segments umfasst (Abb. 8 und 13M- O), korrelieren, konnten auch nach Injektion hoher Konzentrationen (bis 4µg/µl) keine Veränderungen im Bereich der posterioren Expression oder der späteren Aktivität beobachtet werden. Dies könnte möglicherweise daran liegen, dass die injizierte Tc knirps-dsrna zwar zu einer funktionellen Inaktivierung der früh im syncythialen Blastoderm exprimierten anterioren Tc knirps-domäne in der Lage ist, aber die Inaktivierung von später exprimierten Domänen aufgrund der Verdünnung durch Zellteilungen oder auch infolge eines Abbaus der dsrna im wachsenden Keimstreif, nicht mehr in ausreichendem Maße erfolgt. In der Tat zeigt sich bei RNAi-Knockdowns anderer Gene wie Tc giant (siehe Kapitel ) und Tc hunchback (siehe Kapitel ) eine offenbar höhere Sensitivität der anterioren Domänen. Andererseits können bei diesen beiden Genen durch genügend hohe Konzentrationen an dsrna auch die posterioren Domänen zuverlässig ausgeschaltet werden, und im Falle von Tc spalt lässt sich selbst die Entwicklung des Tracheensystems noch wirkungsvoll mittels RNAi beeinflussen (Berghammer 2003). In einer in situ-färbung gegen Tc knirps in Tc knirps-rnai-keimstreifen konnte keine Aktivität der posterioren Domäne mehr detektiert werden (Daten nicht gezeigt). Obwohl diese Methode nicht empfindlich genug ist, um eine evtl. noch vorhandene funktionelle Restaktivität gänzlich auszuschließen, besteht Grund zu der Annahme, dass Tc knirps in der Tat keine posteriore Segmentierungsfunktion hat. Letztendlich kann der endgültige Beweis einer Nicht-Funktion der posterioren Tc knirps-domäne in der Segmentierung aber nur durch eine Tc knirps-nullmutante erbracht werden Regulation von Paarregel- und Segmentpolaritätsgenen durch Tc knirps Zur Analyse des anterioren Segmentierungsphänotyps von Tc knirps wurde die Expression des Paarregelgens Tc even-skipped und des Segmentpolaritätsgens Tc wingless während der Segmentierung verfolgt (Abb. 11 und 12). Im Wildtyp-Blastoderm grenzt das posteriore Ende der anterioren Tc knirps-domäne unmittelbar an den anterioren Rand des ersten Tc eve- Streifens (Abb. 8) bzw. des daraus entstehenden, segmentalen Tc eve1a-streifens (Abb. 9N, O). In Tc knirps-rnai-blastodermen tritt weder eine anteriore Expansion des eve1- Paarregelstreifens (Abb. 11D, E) noch des segmentalen eve1a-streifens (Abb. 11F) auf, so dass eine direkte anteriore Repression von Tc eve1 durch Tc knirps wohl ausgeschlossen 52
71 werden kann. Auch die weiter posterior gelegenen Tc eve-streifen sind in Tc knirps-rnai- Embryonen nicht verändert (Abb. 11 und 12B, D, F; Daten nicht gezeigt), so dass anscheinend weder die anteriore noch die posteriore Tc knirps-domäne einen direkten Einfluss auf die Expression der Paarregelgene auszuüben scheint. Das Segmentpolaritätsgen Tc wingless ist im Blastodermstadium in einem anterior-ventralen Streifen exprimiert, der später Teile der Augenanlage markiert (Abb. 11). Diese Domäne ist in Tc knirps-rnai- Blastodermembryonen ebenfalls exprimiert, aber der deutlich verkürzte Abstand zwischen diesem Streifen und dem ersten Tc eve-streifen (schwarze Balken in Abb. 11 D-F) zeigt, dass die Anlagen zwischen diesen Markern bereits im Blastoderm fehlen bzw. verkleinert sind. Abbildung 11: Expression von Tc eve und Tc wg in Wildtypen und in Tc knirps-rnai- Blastodermembryonen In situ-doppelfärbung gegen Tc eve (braune AP-Färbung) und Tc wg (blaue β-gal-färbung) in Wildtypen (A-C) und Tc knirps-rnai-blastodermembryonen (D-F). Im Wildtyp sind mit beginnender (A) bzw. deutlich sichtbarer posteriorer Invagination (B, C) drei eve-streifen ausgebildet, wobei sich eve1 gerade in die segmentalen Streifen 1a und 1b aufspaltet (A-C). In vergleichbaren Tc knirps-rnai-embryonen ist das Muster der eve-streifen nicht wesentlich verändert (D-F), lediglich die Aufspaltung von eve1 in die segmentalen Streifen eve1a und eve1b erscheint minimal verzögert. In späten Blastodermstadien tritt eine schmale, anterior-ventrale Tc wg-domäne im Bereich der frühen Augenanlage auf, deren Abstand zur anterioren Grenze von eve1 bzw. eve1a in Tc knirps-rnai-blastodermembryonen deutlich verringert ist (schwarze Balken in A-C bzw. D-F). Orientierung: Anterior ist links und ventral ist unten. In frühen Keimrudimenten (Abb. 12A, B) ist das Fehlen von Teilen der Kopfanlage ebenfalls als Verkürzung des Abstands zwischen den segmentalen Streifen eve1a und eve1b erkennbar. 53
72 Während in Wildtyp-Keimstreifen unmittelbar anterior von eve1a (eve1a überlappt mit den Anlagen des ersten Tc engrailed-streifen) der erste (mandibuläre) Tc wingless-streifen gebildet wird (Abb. 12C, E), taucht dieser Streifen in Tc knirps-rnai-keimstreifen nur fragmentär auf (Abb. 12D), um kurz darauf wieder zu verschwinden (Abb. 12F). Im Gegensatz zum ersten Tc wingless-streifen sind der darauf folgende, zweite Tc wg-streifen sowie alle weiteren wg-streifen nicht mehr von der Deletion betroffen und bleiben während des gesamten Keimstreifwachstums normal exprimiert (Abb. 12F, H, J, L; Daten nicht gezeigt). Die beiden Tc wg-streifen in den antennalen Segmentanlagen sind um etwa 45 Grad schräg gestellt und werden im Wildtyp erst deutlich nach den ocularen und gnathalen Domänen exprimiert (Abb. 12G). Dem larvalen Phänotyp entsprechend, tauchen diese antennalen Tc wg-streifen in Tc knirps-rnai-keimstreifen nicht oder nur als temporär exprimierte Fragmente auf (Abb. 12H). Ansonsten normalisiert sich im weiteren Verlauf der Entwicklung die Morphologie der Kopflappen wieder deutlich, und der oculare Tc wg- Streifen spaltet sich in mehrere Domänen auf (Abb. 12I, J). Zusätzlich treten in Wildtypen wie in Tc knirps-rnai-keimstreifen neue Tc wg-expressionsdomänen im Labrum, Stomodeum und im interkalaren Segment auf (Abb. 12I, J). Noch etwas später wird mit beginnendem Extremitätenwachstum das Fehlen der mandibulären und antennalen Tc wg- Streifen in Tc knirps-rnai-keimstreifen nochmals deutlich erkennbar (Abb. 12L). Abbildung 12: Expression von Tc eve und Tc wg in Wildtypen und in Tc knirps-rnai- Keimstreifen In situ-doppelfärbung gegen Tc eve (braune AP-Färbung) und Tc wg (blaue β-gal-färbung) in Wildtypen (A, C, E, G, I, K) und Tc knirps-rnai-keimstreifen (B, D, F, H, J, L). Der Tc eve1a-streifen wird in Tc knirps-rnai-embryonen angelegt. In frühen Keimrudimenten sind, wie im Wildtyp, noch Reste der eve1a-expression in der Mitte erkennbar (A, B), wobei der Abstand zwischen eve1a und eve1b in Tc knirps-rnai-keimrudimenten verkürzt erscheint. Auch der Abstand zwischen dem eve1a-streifen und der Tc wg-domäne in der Augenanlage ist in Tc knirps-rnai-keimstreifen deutlich verringert (A, B). Später wird in Tc knirps-rnai-keimstreifen kein mandibulärer wg-streifen (wg1) gebildet (A-F), und es treten höchstens einige wg-flecken anterior des zweiten wg-streifens auf (B, D, F). Posterior davon werden alle weiteren Tc eve- und Tc wg-streifen normal ausgebildet (F, H, L). Die in den Kopflappen älterer Wildtyp-Keimstreifen auftretenden antennalen wg-streifen (Pfeile in G) tauchen in Tc knirps-rnai-keimstreifen nicht auf (H), wohingegen das noch später auftretende interkalare Segment auch in Tc knirps-rnai-keimstreifen deutlich sichtbar ist (Pfeilspitzen in I, J). In vollständig ausgewachsenen Keimstreifen mit beginnender Extremitätenbildung erkennt man deutlich den durch Tc knirps-rnai erzeugten Verlust der antennalen und mandibulären Extremitätenknospen, während die zum Labrum und zur Augenanlage gehörenden Tc wg-domänen im Kopf nicht betroffen sind (K, L). Anterior ist bei allen Keimstreifen oben; e = eve(n-skipped), wg = wingless. 54
73
74 3.2. Die funktionelle Analyse von Tc Krüppel Kutikulaphänotypen von Tc Krüppel-RNAi und Tc Kr jaws Die erstmals von R. Schröder durchgeführte posttranskriptionelle Inaktivierung von Tc Krüppel mittels RNA-Interferenz (RNAi) erzeugt ein ganzes Spektrum von Phänokopien (exakter Begriff für Phänotypen, die nicht durch eine genetische Mutation, sondern durch RNAi erzeugt werden), die zum Teil in den Abbildungen 13C-E dargestellt sind. Die stärksten Phänokopien (nicht gezeigt) gleichen dabei sogar dem Phänotyp der Mutante Tc jaws (Abb. 13F). Gregor Bucher hatte durch eine Segregationsanalyse eine äußerst enge Kopplung zwischen der Mutation Tc jaws und einem Polymorphismus im 3 UTR des Tc Krüppel-Lokus zeigen können (Bucher 2002), und im Rahmen der eigenen Diplomarbeit (Cerny 2002) konnte in Tc jaws eine Mutation in der codierenden Sequenz von Tc Krüppel gefunden werden (Cerny et al. 2005). Dabei handelt es sich um eine, durch einen Basenaustausch hervorgerufene, missense -Mutation im ersten DNA-bindenden Zinkfinger, die das zweite Histidin des essentiellen Cys-Cys-His-His-Motivs zu einem Tyrosin abändert. Diese Mutation in der DNA-Bindungsdomäne erzeugt einen kompletten Funktionsverlust, da der Phänotyp der daraufhin Tc Kr jaws genannten Mutante nicht durch Tc Krüppel-RNAi in Tc Kr jaws - mutantem Hintergrund weiter verstärkt werden kann (Cerny et al. 2005). Im Gegensatz zu Dm Krüppel-Mutanten, weist der Phänotyp von Tc Kr jaws aber keine zur Tc Krüppel-Expression passende Lücke innerhalb des segmentierten Bereichs auf und entspricht daher nicht einem klassischen Gap-Gen-Phänotyp. Vielmehr wird die Segmentzahl von 16 im Wildtyp (Abb. 13A; siehe auch Abb. 2H) auf nur noch 10 in Tc Kr jaws (Abb. 13F) reduziert, wobei außerdem die Segmente T1-A1 zu je zwei ektopischen, alternierend angeordneten maxillären und labialen Segmenten transformiert werden. Während die ektopischen Labialpalpen mit Ausnahme der fehlenden ventralen Fusion gegenüber dem ersten Labium unverändert sind, kommt es bei den ektopischen Maxillen zum Verlust der Enditen (Mala) und beim dritten Maxillenpaar zu einer Tendenz in Richtung Beinmorphologie. Posterior des dritten labialen Segments folgt noch ein abdominales Segment unbekannter Identität sowie die terminalen Segmente mit den Urogomphi und Pygopodien (Abb. 13F). Da die terminalen Segmente am posterioren Ende von Tc Kr jaws - mutanten Larven gegenüber Wildtypen (Abb. 13A) nicht verändert sind, lässt der Kutikulaphänotyp von Tc Kr jaws eine Deletion von abdominalen Segmenten vermuten 56
75 (Anderson and Sulston 1998), die aber nicht direkt mit der von T1-T3 reichenden Tc Krüppel- Domäne in Einklang zu bringen ist. Starke Tc Krüppel-RNAi-Phänokopien, die aber nicht ganz den Phänotyp von Tc Kr jaws erreichen, weisen den gleichen Segmentierungsdefekt wie die Nullmutante auf, allerdings ist die Morphologie der ektopischen Maxillen deutlich stärker in Richtung Beinextremität ausgeprägt (Abb. 13E). In mittelstarken Tc Krüppel-RNAi-Phänokopien ist hingegen der Segmentierungsdefekt im Vergleich zu Tc Kr jaws etwas abgeschwächt, und es kommt zur Ausbildung zusätzlicher Segmente (Abb. 13D). Dabei bleibt das aus Tc Kr jaws bekannte Muster alternierender homeotischer Transformationen erhalten, und es wird ein viertes labiales Segment anstatt des dritten abdominalen Segments (A3) ausgebildet. Im Gegensatz zu den ektopischen labialen Segmenten werden die ektopischen Maxillen mit abnehmender Intensität der Tc Krüppel-RNAi zunehmend in Beinextremitäten umgewandelt. So zeigen in mittelstarken Tc Krüppel-RNAi-Phänokopien nur noch die Extremitäten im ersten thorakalen Segment partiell maxillären Charakter, während die Extremitäten des dritten thorakalen Segments (T3) schon keine Anzeichen einer Transformation in Richtung Maxillen mehr zeigen (Abb. 13D). Im Abdomen kommt es bei mittelstarken Tc Krüppel-RNAi-Phänokopien schließlich zu einer alternierenden Transformation in labiale (A1 und A3) und thorakale (A2 und A4) Segmente (Abb. 13D). Dies deutet darauf hin, dass in Tc Krüppel-RNAi für Transformationen in ektopische Maxillen einerseits und für Transformationen in ektopische Labialpalpen andererseits möglicherweise unterschiedliche Schwellenwerte an Tc Krüppel- Aktivität unterschritten werden müssen. In schwachen Tc Krüppel-RNAi-Phänokopien ist einzig das zweite thorakale Segment in ein zweites Labium transformiert und reagiert somit am sensibelsten auf eine abfallende Tc Krüppel-Aktivität (Abb. 13C). Ansonsten lässt sich diese Klasse an Phänokopien klar in gnathale, thorakale und abdominale Segmente untergliedern, wobei die abdominalen Segmente gelegentlich noch einige unregelmäßige Anhänge aufweisen (Abb. 13C). Aufgrund des geringeren Segmentierungsdefekts in schwachen Tc Krüppel-RNAi-Phänokopien zeigen diese Larven ein nur leicht verkürztes Abdomen von sechs Segmenten (Abb. 13C), an das sich, wie auch in stärkeren Tc Krüppel- RNAi-Phänokopien, die terminalen Segmente anschließen. Wie schon in Tc Kr jaws, lässt sich auch in schwachen und mittelstarken Tc Krüppel-RNAi-Kutikulae zwar eine reduzierte Anzahl an Segmenten, aber keine klar erkennbare Lücke in der Segmentierung feststellen. 57
76 Abbildung 13: Phänotypische Serie von Tc Krüppel (aus Cerny et al. 2005) Confocalbilder von Kutikulapräparaten einer phänotypischen Serie von Tc Krüppel (ventrale Aufsicht, anterior ist immer links): (A, B) Wildtyp-Larve (Bilder von Hilde Wohlform), (C-E) Tc Krüppel-pRNAi-Larven ansteigender phänotypischer Stärke und (F) eine Larve der Tc Krüppel-Nullmutante Tc Kr jaws (vergrößerte Darstellung gegenüber A und C-E). Die gnathalen Extremitäten der Wildtyp-Larve in A sind in Abbildung B vergrößert dargestellt, und durch eine weiße Umrandung wird eine Maxille (mx) sowie das Labium (lb) hervorgehoben. In den Tc Krüppel-defizienten Larven (C-F) ist die Anzahl an Segmenten reduziert, und es treten ektopische gnathale Extremitäten auf, die durch Pfeilspitzen (Labiumähnliche Extremitäten) bzw. durch Pfeile (Extremitäten mit Maxillen-ähnlichem Charakter) markiert sind. Im Wildtyp (A) und in den schwächeren Tc Krüppel-pRNAi-Phänokopien (C, D) sind außerdem einzelne thorakale und abdominale Segmente gekennzeichnet. Die terminalen Strukturen der Pygopodien (Py) und Urogomphi (U) sind stets am posterioren Ende von Tc Krüppel-RNAi- und Tc Kr jaws -Larven vorhanden. 58
77 Regulation von Gap-Genen durch Tc Krüppel Interaktionen von Tc Krüppel und Tc giant Die in Tc Kr jaws homeotisch transformierten Segmente T1-A1 korrelieren mit der von T1 bis T3 reichenden Expressionsdomäne von Tc Krüppel, wohingegen der abdominale Segmentierungsdefekt posterior der Tc Krüppel-Domäne auftritt. Diese Beobachtung könnte darauf hindeuten, dass die homeotische Identität direkt von Tc Krüppel reguliert wird, der abdominale Segmentierungsdefekt aber indirekt durch die Missregulation anderer, weiter posterior gelegener Gap-Gene erzeugt wird. Daher wurde im Rahmen der eigenen Diplomarbeit die Expression von Tc giant (Abb. 14A, C; siehe auch Bucher und Klingler 2004) in Tc Kr jaws -mutanten Embryonen analysiert. Es stellte sich heraus, dass die anteriore Tc giant- Domäne unverändert bleibt, während die posteriore Tc giant-domäne in Tc Kr jaws -mutanten Keimrudimenten nicht oder nur schwach aktiviert wird (Abb. 14A, B). In etwas späteren Tc Kr jaws -mutanten Keimstreifen taucht weiter anterior ein schwacher Tc giant-streifen auf (Abb. 13D). Da dieser Streifen aber erst deutlich später als die posteriore Tc giant-domäne im Wildtyp auftritt und auch in seiner Intensität wesentlich geringer ist, kann dieser als ektopische Expression und nicht als nach anterior verschobene posteriore Tc giant-domäne interpretiert werden. Mittels in situ-dreifachfärbungen gegen Tc eve (AP) und Tc giant / Tc engrailed (beides β-gal-färbungen) lässt sich Position des ektopischen Tc giant-streifens ungefähr zwischen die sekundären eve-streifen 2a und 2b (Abb. 35E) bzw. den daraus entstehenden engrailed-streifen 3 und 4 lokalisieren (Abb. 35I). Außerdem konnte in dieser Färbung in späten Tc Kr jaws -mutanten Keimstreifen ein weiterer Tc giant-streifen detektiert werden, der sich zwischen eve3a und eve3b befindet (Abb. 35I). Damit entspricht er in seiner Lage in etwa dem vorderen Streifen der posterioren Tc giant-domäne im Wildtyp (Bucher und Klingler 2004), aufgrund des deutlich späteren und nochmals schwächeren Erscheinens wird er aber ebenfalls als ektopischer Streifen interpretiert. Neben der Aktivierung der posterioren Tc giant-domäne durch Tc Krüppel, kommt es in Tribolium auch zu einer Repression der Tc Krüppel-Expression durch die anteriore Tc giant- Domäne. Daher zeigen Tc giant-rnai-embryonen eine anteriore Expansion der im Wildtyp (Abb. 14E) hinter dem segmentalen eve2a-streifen beginnenden Tc Krüppel-Expression bis an die hintere Grenze des segmentalen eve1b-streifens (Abb. 14F). Die posteriore Grenze der Tc Krüppel-Expression lässt sich in Tc giant-rnai-embryonen nicht an Hand der Tc eve- Streifen bestimmen, da die funktionelle Inaktivierung von Tc giant auch eine Störung des Paarregel-Musters von Tc eve bewirkt (Abb. 28). Es kann jedoch ein zum Wildtyp 59
78 vergleichbares Zurückziehen der Tc Krüppel-Expression aus dem Bereich der Wachstumszone beobachtet werden (Abb. 14E, F), so dass von einer im Wesentlichen unveränderten posterioren Grenze der Tc Krüppel-Domäne in Tc giant-rnai-keimstreifen ausgegangen werden kann. Insgesamt unterscheidet sich die Regulation von Tc giant durch Tc Krüppel also grundlegend von der Situation in Drosophila, wo Dm Krüppel als Repressor für sowohl die anteriore, als auch die posteriore Dm giant-domäne fungiert (Kraut und Levine 1991b). Außerdem ist in Drosophila die Interaktion zwischen diesen beiden Gap-Genen symmetrisch, da auch die beiden Dm giant-domänen als Repressoren gegenüber der in ihrer Mitte gelegenen Dm Krüppel-Domäne fungieren (Kraut und Levine 1991b). Abbildung 14: Interaktionen zwischen Tc Krüppel und Tc giant In situ-färbung gegen Tc giant in Wildtypen (A, C) und in Tc Kr jaws -mutanten Keimstreifen (B, D) sowie in situ-doppelfärbung gegen Tc even-skipped (braune AP-Färbung) und Tc Krüppel (blaue β-gal-färbung) in einem Wildtyp- (E) und einem Tc giant-rnai- Keimstreif (F). Während in Tc Kr jaws -mutanten Keimrudimenten die anteriore Tc giant- Expression unverändert bleibt, tritt ein nahezu vollständiger Verlust der posterioren Tc giant- Domäne auf (A-D). In späteren Stadien von Tc Kr jaws -mutanten Keimstreifen kommt es dann noch zu einer streifenförmigen, schwachen, ektopischen Expression anterior der eigentlichen posterioren Domäne (Pfeilspitze in D). Die anteriore Grenze der Tc Krüppel-Domäne wird im Wildtyp durch den posterioren Rand des Tc eve2a-streifens markiert (E). In Tc giant-rnai- Keimstreifen expandiert die Tc Krüppel-Domäne in anteriorer Richtung bis zum posterioren Rand des Tc eve1b-streifens, während die posteriore Grenze nicht beeinflusst wird (F). Alle Embryonen sind anterior nach oben ausgerichtet; e = eve(n-skipped). Die Abbildungen A-D sind aus der eigenen Diplomarbeit (Cerny 2002) entnommen. 60
79
80 Expression von Tc knirps in Tc Kr jaws -Embryonen Da die posteriore Tc knirps-expression innerhalb der durch Tc Krüppel aktivierten, posterioren Tc giant-domäne liegt (Abb. 9I-L), wurde auch eine mögliche Aktivierung der posterioren Tc knirps-expression durch Tc Krüppel untersucht. Die posteriore Tc knirps- Expression unterteilt sich eigentlich in zwei Domänen: einen schmalen, ektodermalen Streifen in der Anlage von A1 sowie eine temporäre Expression im posterior invaginierenden Mesoderm (Abb. 15A, B; siehe auch Abb. 7A-C und 9B-D). Es zeigte sich, dass in eindeutig identifizierten Tc Kr jaws -Keimstreifen (als Marker dient die fehlende posteriore Tc giant- Domäne) der ektodermale Tc knirps-streifen fehlt, wohingegen die mesodermale Tc knirps- Expression erhalten bleibt (Abb. 15C), was eine unabhängige Regulation dieser beiden Domänen spricht. Die in Tc Kr jaws ebenfalls fehlende posteriore Tc giant-domäne spielt dabei keine Rolle in der Aktivierung der ektodermalen Tc knirps-expression, da Tc knirps in Tc giant-rnai-keimstreifen normal ausgeprägt ist (Daten nicht gezeigt). Abbildung 15: Expression von Tc knirps in Wildtyp- und Tc Kr jaws -Keimstreifen In situ-färbung gegen Tc knirps (braune AP-Färbung in A-C) in einem unpräparierten Wildtyp-Keimstreif (A, gleicher Embryo wie in Abb. 7A), einem präparierten Wildtyp- Keimstreif (B) sowie in einem präparierten Tc Kr jaws -Keimstreifen (C) mit zusätzlicher Färbung gegen Tc giant (blaue β-gal-färbung). Der posteriore, ektodermale Tc knirps- Streifen (Pfeilspitzen in A, B) fehlt in Tc Kr jaws -mutanten Keimstreifen (C), während der anteriore Tc knirps-streifen sowie die posteriore Expression im sich invaginierenden Mesoderm nicht durch die Tc Kr jaws -Mutation beeinträchtigt werden (A-C). Anterior ist immer oben. 62
81 Expression von Tc hunchback in Tc Kr jaws - und Tc Krüppel-RNAi-Embryonen Ähnlich wie Tc giant und Tc knirps, wird auch Tc hunchback während der Segmentierung in zwei Domänen exprimiert (Abb. 16A1-A4; siehe auch Wolff et al. 1995). Die anteriore Tc hunchback-domäne taucht schon im Blastodermstadium auf und besteht noch bis ins frühe Keimstreifstadium fort, wo sie partiell mit der anterioren, nicht aber mit der posterioren Tc giant-domäne überlappt (Abb. 16A1). Die posteriore Tc hunchback-domäne erscheint während des Keimstreifwachstums erst zu einem vergleichsweise späten Zeitpunkt, zu dem der vordere Streifen der posterioren Tc giant-domäne sich schon aufzulösen beginnt (Abb. 16A3). Anfänglich weist die deutlich hinter den posterioren Tc giant-streifen gelegene, posteriore Tc hunchback-domäne eine scharfe anteriore Grenze auf (Abb. 16A3), die aber im Laufe des weiteren Keimstreifwachstums verloren geht (Abb. 16A4). In mittelstarken Tc Krüppel-RNAi-Keimstreifen ist in frühen Stadien gegenüber dem Wildtyp keine Veränderung der anterioren Tc hunchback-expression sichtbar (Daten nicht gezeigt), während in späten Keimstreifen eine leichte anteriore Verschiebung der posterioren Tc hunchback-aktivität erkennbar wird (Abb. 16B3). Dabei tritt unmittelbar vor der posterioren Tc hunchback-domäne ein kräftiger Tc giant-streifen auf (Abb. 16B3), der allerdings weder eindeutig als ein Teil der posterioren Tc giant-domäne (Abb. 16A2), noch als ein ektopischer Streifen wie in Tc Kr jaws -Keimstreifen (Abb. 16C3) eingeordnet werden kann. Durch die Verwendung der posterioren Tc giant-domäne als Marker kann in frühen, eindeutig identifizierten Tc Kr jaws -Keimrudimenten eine unveränderte Expression der anterioren Tc hunchback-domäne gezeigt werden (Abb. 16C1). Im Gegensatz zu den frühen Keimrudimenten, zeigen etwas ältere Tc Kr jaws -Keimstreifen aber eine durchgehende, von den gnathalen Segmentanlagen bis zur Wachstumszone reichende Tc hunchback-expression (Abb. 16C2). Im Laufe der weiteren Entwicklung von Tc Kr jaws -Keimstreifen verschwindet das anteriore Tc hunchback-muster und posterior des ektopischen Tc giant-streifens tritt eine Tc hunchback-expression auf (Abb. 16C3), welche der späten, etwas diffusen posterioren Tc hunchback-aktivität in sehr viel älteren Wildtypen ähnelt (Abb. 16A4). Die Expansion der Tc hunchback-expression in jungen Tc Kr jaws -Keimstreifen könnte auf eine späte posteriore Repression der anterioren Tc hunchback-domäne durch Tc Krüppel hinweisen. Allerdings zeigt sich in mittelstarken Tc Krüppel-RNAi-Keimstreifen keine abgeschwächte Missregulation der anterioren Tc hunchback-domäne, sondern vielmehr eine schwache anteriore Verschiebung der posterioren Tc hunchback-domäne. Dadurch kann man von einer in Tc Kr jaws gegenüber Tc Krüppel-RNAi verstärkten anterioren Verschiebung der posterioren Tc hunchback-domäne ausgehen, die, aufgrund der im Keimstreif von anterior 63
82 nach posterior voranschreitenden Segmentierung, auch mit einer zeitlich verfrühten Aktivierung der posterioren Tc hunchback-domäne einhergeht. Dies führt in Tc Kr jaws - Keimstreifen zu einer zeitlichen Überlappung der anterioren und der posterioren Tc hunchback-aktivität, die schließlich vorübergehend eine durchgehende Tc hunchback- Expression zur Folge hat (Abb. 16C2). In mittelstarken Tc Krüppel-RNAi-Keimstreifen tritt hingegen, aufgrund der im Vergleich zu Tc Kr jaws geringeren phänotypischen Stärke, keine Überlappung mit der anterioren Tc hunchback-domäne mehr auf und so wird die klare anteriore Grenze der misslokalisierten posterioren Tc hunchback-domäne erkennbar (Abb. 16B3). Neben Tc hunchback wird auch die posteriore Tc giant-domäne durch Tc Krüppel beeinflusst (Abb. 14). Eine Funktion von Tc giant als Übermittler der anterioren Repression von Tc hunchback durch Tc Krüppel kann aber ausgeschlossen werden, da Tc giant-rnai die posteriore Tc hunchback-domäne nicht beeinflusst (Abb. 25). Abbildung 16: Expression von Tc hunchback und Tc giant in Wildtypen, Tc Krüppel- RNAi- und Tc Kr jaws -Embryonen In situ-doppelfärbung gegen Tc hunchback (braune AP-Färbung) und Tc giant (blaue β-gal- Färbung) in Wildtypen (A1-A4), einem Tc Krüppel-RNAi-Keimstreif (B3) und in Tc Kr jaws - mutanten Keimstreifen (C1-C3). In Wildtyp-Embryonen verschwindet die anteriore Tc hunchback-domäne während des Keimstreifwachstums (A1, A2), und die posteriore Tc hb-expression erscheint deutlich später und weit hinter der posterioren Tc giant-domäne (A3). Anfänglich ist dabei eine scharfe anteriore Grenze der posterioren Tc hb-domäne erkennbar (A3), bevor in späteren Stadien eine eher diffuse Expression auftritt (A4). In diesen späten Keimstreifstadien ist auch schon die neurogene Tc hb-expression in weiter anterior gelegenen Segmenten erkennbar (A4). In mittelstarken Tc Krüppel-RNAi-Embryonen kommt es zu einer leichten anterioren Verschiebung der posterioren Tc hb-domäne, die sich posterior eines kräftigen Tc giant-streifens befindet (B3). In Tc Kr jaws -mutanten Keimrudimenten ist die anteriore Tc hb-domäne nicht verändert (C1; die bogenförmige Expression posterior des Serosafensters entsteht durch die Tc hb-aktivität in den nicht vollständig abpräparierten Serosazellen). In etwas späteren Tc Kr jaws -Keimstreifstadien ist hingegen eine durchgehende Tc hb-expression zu beobachten (C2). Im Laufe der weiteren Entwicklung taucht der ektopische Tc giant-streifen auf (Pfeilspitze in C3), während posterior davon eine diffuse Tc hb-expression erscheint (C3), die der späten posterioren Tc hunchback-expression im Wildtyp ähnelt (A4). Anterior ist bei allen Keimstreifen oben. 64
83
84 Regulation von Paarregel- und Segmentpolaritätsgenen durch Tc Krüppel Im Gegensatz zur ursprünglichen Interpretation des jaws-phänotyps (Anderson und Sulston 1996 und 1998), zeigte eine erste, während der eigenen Diplomarbeit (Cerny 2002) durchgeführte Tc eve-färbung in Tc Kr jaws -mutanten Keimstreifen keine Störung des Paarregel- Musters bis zu einem vorzeitigen Segmentierungsabbruch nach dem primären eve5-streifen. Auch die Expression der Tc wingless-streifen erscheint, von einer vorübergehenden Störung abgesehen, bis zum 10. Streifen weitgehend normal (Abb. 35A, E, I, M; siehe auch Cerny et al. 2005). Dies führte zu der ketzerischen These, dass die Paarregelgenstreifen 1-5 in Tc Kr jaws -mutanten Keimstreifen, trotz des Ausfalls von Tc Krüppel und der posterioren Tc giant-domäne sowie der Missregulation der posterioren Tc hunchback-domäne, normal ausgebildet werden können. Zur Überprüfung dieser These wurden im Rahmen dieser Arbeit mit Tc runt und Tc hairy zwei weitere Paarregelgene in Tc Kr jaws -mutanten Keimstreifen analysiert sowie die Expression von Tc eve in mittelstarken Tc Krüppel-RNAi-Keimstreifen untersucht Expression von Tc runt und Tc wingless in Tc Kr jaws -Keimstreifen Das Paarregelgen Tc runt wird in seiner frühen Aktivität nach dem Verlassen der Wachstumszone in doppelsegmentalen Streifen exprimiert, die sich etwa ein halbes Segment posterior der Tc eve-paarregelstreifen befinden (Choe et al. 2006). Ähnlich wie bei Tc eve, verschwindet auch bei Tc runt mit zunehmendem Keimstreifwachstum die Expression weiter anterior gelegener Streifen, so dass maximal drei Streifen gleichzeitig exprimiert werden (Abb. 17). Die anterioren Tc runt-streifen verringern dabei ihre Ausdehnung bis auf die Größe von Segmentpolaritätsstreifen (runt1 in Abb. 17A, B, runt2 in Abb. 17C, D), ohne sich aber wie Tc eve in zwei segmentale Streifen aufzuspalten. Zur Identifizierung der posterioren Streifen wurde in Wildtyp-Keimstreifen (Abb. 17A, C, E, G, I) eine Tc runt/tc wingless-doppelfärbung durchgeführt, während in Tc Kr jaws -mutanten Keimstreifen (Abb. 17B, D, F, H, J) zur Identifizierung von mutanten Embryonen in frühen Stadien noch zusätzlich gegen den Marker Tc giant gefärbt wurde. Es zeigte sich, dass die ersten vier Streifen von Tc runt in Tc Kr jaws -Keimstreifen gegenüber vergleichbaren Wildtyp- Keimstreifen keine Veränderungen in der Expression zeigen (Abb. 17A-F), obwohl es im Bereich der Streifen 2-4 zum Verlust der Tc Krüppel-Domäne sowie der posterioren Tc giant- Aktivität kommt. Im Laufe der weiteren Entwicklung von Tc Kr jaws -mutanten Keimstreifen 66
85 wird zwar noch sehr nahe an der (durch eine Tc wg-expression markierten) Proctodeum- Anlage der runt5-streifen gebildet, welcher aber von keinen weiteren Streifen mehr gefolgt wird (Abb. 17H, J). Schließlich verblasst in Tc Kr jaws -mutanten Keimstreifen die Paarregel- Expression von Tc runt mit dem fünften Streifen (Abb. 17J), während in vergleichbaren (Tc wg-muster in den Kopflappen als Marker) Wildtypen der runt5-streifen ebenfalls verschwindet, die Tc runt-expression aber in den Streifen 6 und 7 fortbesteht (Abb. 17G, I). Insgesamt werden in Tc Kr jaws -mutanten Keimstreifen also auch die Tc runt-streifen 6-8 nicht ausgebildet, was der aus den fehlenden posterioren Tc eve-streifen abgeleiteten Theorie eines vorzeitigen Abbruchs der Segmentierung entspricht. Die segmentale Tc wingless-expression bestätigt dabei den posterioren Segmentierungsabbruch von Tc Kr jaws -Keimstreifen, und es werden nur die ersten sieben wg-streifen völlig normal ausgebildet (Abb. 17G, H). Anstelle der weiteren wg-streifen 8-11 (Abb. 17I) beobachtet man in späten Tc Kr jaws -Keimstreifen ein vorübergehend irreguläres wg-muster hinter dem 7. Streifen (Abb. 17J), das sich aber später in vollständig ausgewachsenen Tc Kr jaws -Keimstreifen letztendlich doch zu 10 intakten wg- Streifen organisiert (Cerny et al. 2005). Diese 10 wg-streifen entsprechen exakt dem auf 10 Segmente (7 gnathale, 1 abdominales und 2 terminale Segmente) reduzierten Phänotyp von Tc Kr jaws (Abb. 13F). Merkwürdigerweise ist in späten, Tc Kr jaws -mutanten Keimstreifen gelegentlich auch noch eine leichte Störung in der anterioren Segmentierung zu beobachten, und es kommt, anders als in früheren Stadien, mitunter zu einem sekundär etwas vergrößerten Abstand zwischen den Segmentpolaritätsstreifen 3 und 4 (Abb. 17H, J). Dieser Effekt könnte durch die Missregulation weiterer Paarregelgene, oder aber durch den in diesem Bereich exprimierten, ektopischen Tc giant-streifen (Abb. 35D) hervorgerufen werden. 67
86 Abbildung 17: Expression von Tc runt und Tc wingless in Wildtypen und in Tc Kr jaws - Keimstreifen In situ-doppelfärbung gegen Tc runt (braune AP-Färbung) und Tc wg (blaue β-gal-färbung) in Wildtypen (A, C, E, G, I) und Tc Kr jaws -Keimstreifen (B, D, F, H, J). Die Wildtyp- Keimstreifen in G, I stammen aus einer anderen Färbung als die früheren Stadien in A, C und E. Die Tc Kr jaws -mutanten Keimstreifen stammen wiederum aus einer anderen Färbung und wurden zur Identifizierung noch zusätzlich mit β-gal gegen Tc giant gefärbt. Die runt- Streifen 1-4 sind in Tc Kr jaws -mutanten Keimstreifen identisch zum Wildtyp exprimiert (vergl. B, D, F mit A, C, E) und auch die wg-streifen 1-7 werden in Tc Kr jaws -Keimstreifen regulär gebildet (vergl. F, H mit E, G). Nach dem runt5-streifen stoppt in Tc Kr jaws -mutanten Keimstreifen die Segmentierung (H), während vergleichbare Wildtyp-Keimstreifen bereits runt6 in der Wachstumszone exprimieren (G). In späteren, Tc Kr jaws -mutanten Keimstreifen wird die runt5-expression diffuser und schwächer (J). Posterior von wg7 treten unregelmäßige wg-streifen auf (J), während in vergleichbaren Wildtyp-Keimstreifen die abdominale Segmentierung bis zur Expression von runt7 bzw. wg11 voranschreitet (I). Neben den Segmentpolaritätsstreifen ist Tc wg auch noch in einem dynamischen Muster in den Kopflappen sowie in einer terminalen Domäne posterior der Wachstumszone exprimiert (A- J). Die dynamische Kopfexpression von Tc wg dient in älteren Tc Kr jaws -Keimstreifen als Marker für die Bestimmung der Stadien, wobei in Tc Kr jaws -Keimstreifen eine Überlagerung mit der späten, neurogenen Expression von Tc giant auftritt. Anterior ist immer oben; r = runt, wg = wingless. 68
87
88 Expression von Tc hairy und Tc engrailed in Tc Kr jaws -Keimstreifen Da in Drosophila die Streifen der verschiedenen Paarregelgene unterschiedlich reguliert werden, wurde mit Tc hairy ein weiteres Paarregelgen in Tc Kr jaws -mutantem Hintergrund analysiert. Die Tc hairy-streifen liegen jeweils etwa ein halbes Segment anterior der Tc eve- Streifen (Brown und Denell 1996), und wie schon bei Tc runt, kann man auch bei Tc hairy in Tc Kr jaws -mutanten Keimstreifen keine Änderung im Expressionsmuster der ersten fünf Streifen erkennen (Abb. 18A-H). Später tritt in Tc Kr jaws -Keimstreifen hinter dem Tc hairy5- Streifen eine weitere, streifenförmige Tc hairy-expression auf (h? in Abb. 18J), die sich im weiteren Verlauf allerdings nicht zu einem distinkten Streifen verschmälert, sondern im Proctodeum fortbesteht (Abb. 19). Somit entspricht diese posteriore Tc hairy-expression nicht dem Tc hairy6-streifen in Wildtypen (Abb. 18I), so dass auch Tc hairy den Verlust der posterioren Streifen 6-8 zeigt. Abbildung 18: Expression von Tc hairy und Tc engrailed in Wildtypen und in Tc Kr jaws - Keimstreifen In situ-dreifach-färbung gegen Tc hairy (braune AP-Färbung) sowie gegen Tc giant und Tc engrailed (beides blaue β-gal-färbungen) in Wildtypen (A, C, E, G, I) und Tc Kr jaws - mutanten Keimstreifen (B, D, F, H, J). Die Wildtypen und die Tc Kr jaws -mutanten Keimstreifen stammen aus der gleichen Färbung, wobei Tc giant als Marker zur Identifizierung der mutanten Embryonen dient. Die Expression der hairy-streifen 1-5 ist in Tc Kr jaws -mutanten Keimstreifen gegenüber vergleichbaren Wildtyp-Stadien nicht verändert (A-H). In späten, Tc Kr jaws -mutanten Keimstreifen taucht eine hinter dem hairy5-streifen gelegene, bogenförmige hairy-expression im Proctodeum auf (J). Diese als h? bezeichnete Expression entspricht nicht dem in vergleichbaren Wildtypen gebildeten hairy6-streifen (I). Die Streifen der posterioren Tc giant-domäne werden in Wildtypen zwischen den hairy- Streifen 3 und 4 bzw. 4 und 5 exprimiert (C, E, G). In Tc Kr jaws -mutanten Keimstreifen tritt der erste ektopische Tc giant-streifen anterior von hairy3 auf (Pfeilspitze in H). In späteren Stadien sind außerdem noch die anterioren Tc engrailed-streifen 1-6 zu erkennen (J). Anterior ist immer oben; h = hairy, en = engrailed. 70
89
90 Das Segmentpolaritätsgen Tc engrailed wird unmittelbar posterior der jeweiligen Tc wingless-streifen exprimiert. Dabei decken die Tc engrailed-streifen die gesamte Breite des Keimstreifs ab (Abb. 18I, J und 19), während die Tc wingless-streifen nur im mittleren Bereich und mit einer ventralen Lücke exprimiert werden (Abb. 17). Bei der Expression von Tc en in späten Tc Kr jaws -Keimstreifen zeigte sich mit der korrekten Ausbildung der ersten sechs bis sieben Tc en-streifen (Abb. 18J und 24A), gefolgt von einem unregelmäßigen Tc en Muster (Pfeilspitzen in Abb. 19A, B), ein ähnliches Bild wie für Tc wg (Abb. 35I, M; siehe auch Cerny et al. 2005). Schließlich kommt es aber (vermutlich aufgrund von stabilisierenden Interaktionen zwischen den Segmentpolaritätsgenen) in späten Tc Kr jaws -Keimstreifen letztendlich zur Etablierung von 10 korrekten Tc en- (und Tc wg-) Streifen (Abb. 19C), was mit den 10 im Kutikulaphänotyp gebildeten Segmenten übereinstimmt (Abb. 13F). Abbildung 19: Expression von Tc hairy und Tc engrailed in späten Tc Kr jaws - Keimstreifen In situ-färbung gegen Tc engrailed, Tc giant (beides β-gal-färbungen in blau) und Tc hairy (AP-Färbung in braun) in späten Tc Kr jaws -mutanten Keimstreifen (Embryonen aus der gleichen Färbung wie Abb. 18). Die posteriorsten Tc engrailed-streifen entstehen aus einer zunächst sehr unregelmäßigen Expression. In jungen Embryonen erkennt man sechs intakte engrailed-streifen (A), und einige, punktförmige engrailed-flecken (Pfeilspitzen in A). Ein anderer Embryo (B) zeigt acht intakte engrailed-streifen und nur eine punktförmige Expression an der Position des entstehenden, neunten engrailed-streifens (Pfeilspitze in B). Letztendlich zeigen vollständig ausgewachsenen Keimstreifen dann aber doch meist 10 regelmäßig angeordnete engrailed-streifen (C). In diesen Embryonen ist auch die nach dem Verschwinden der Paarregelstreifen andauernde hairy-expression am posterioren Ende des Keimstreifs zu erkennen (A-C). Anterior ist immer oben; en = engrailed. 72
91 73
92 Expression von Tc even-skipped und Tc wingless in Tc Krüppel-RNAi-Embryonen Während in der Nullmutante Tc Kr jaws meistens exakt 10 statt 16 Segmente gebildet werden (Abb. 13A, F), treten in schwachen und mittelstarken Tc Krüppel-RNAi-Phänokopien intermediäre Segmentierungsdefekte auf (14 Segmente in Abb. 13C, 12 Segmente in Abb. 13D). Um eventuell in der Nullmutante verdeckte Aspekte analysieren zu können, wurde auch eine molekulare Analyse der intermediären Segmentierungsdefekte in mittelstarken Tc Krüppel-RNAi-Phänokopien durchgeführt. Nach der Injektion von dsrna in weibliche Puppen kommt es bei den daraus geschlüpften Käfern im Laufe der Zeit zu einem Abklingen der parentalen RNAi, wobei deren genauer Verlauf von dem ausgeschalteten Gen abhängt und bis zu einem gewissen Grad auch experimentellen Schwankungen unterliegt. Deshalb wurden zur Gewinnung von mittelstarken Tc Krüppel-RNAi-Keimstreifen täglich ein Gelege von 8-20h (bei 32 C) alten Embryonen fixiert, wobei zur Kontrolle der phänotypischen Stärke zweimal pro Woche ein Teil des Geleges bis zur Bildung des ersten Larvenstadiums inkubiert wurde. Anschließend wurde nach Gelegen mit einer hohen Frequenz an mittelstarken Phänokopien wie in den Abb. 13C und 13D gesucht und die in demselben Zeitraum fixierten Keimstreifen für die in situ- Färbung eingesetzt. Eine erste Tc eve- und Tc wg- Färbung dieser Keimstreifen zeigte, wie schon in der Nullmutante Tc Kr jaws, keine Veränderung in der anterioren Segmentierung (Daten nicht gezeigt). Allerdings traten als Folge des harschen Entfernens der Vitellinmembran mittels Drücken durch eine Spritze meistens nur noch Bruchstücke der späten Keimstreifen auf. Deshalb wurden in einem erneuten Experiment in 15-20h Gelegen bei 32 C speziell ältere Keimstreifen fixiert und schonender manuell devitellinisiert (siehe Material und Methoden). Dabei zeigen späte, mittelstarke Tc Krüppel-RNAi-Keimstreifen ebenfalls einen vorzeitigen Segmentierungsabbruch, der allerdings im Vergleich zu Tc Kr jaws -mutanten Keimstreifen etwas weiter posterior auftritt (Abb. 20). Im Gegensatz zur normalen Expression der posterioren Tc eve-streifen im Wildtyp (Abb. 20A), treten in vergleichbaren Tc Krüppel- RNAi-Keimstreifen je nach phänotypischer Stärke 9 (Abb. 20B) bzw. 11 (Abb. 20C, D) intakte wg-streifen auf, an die sich posterior eine irreguläre Expression von Tc eve (Abb. 20B, C) bzw. Tc wg (Abb. 20D) anschließt. Somit zeigt sich auch in mittelstarken Tc Krüppel-RNAi-Keimstreifen ein Segmentierungsabbruch als Ursache für den Verlust von posterioren Segmenten, wobei sich das Einsetzen des vorzeitigen Segmentierungsabbruchs mit schwächer werdendem Phänotyp nach posterior verschiebt. 74
93 Abbildung 20: Expression von Tc eve und Tc wingless in späten Tc Krüppel-RNAi- Keimstreifen In situ-doppelfärbung gegen Tc eve (braune AP-Färbung) und Tc wg (blaue β-gal-färbung) im Wildtypen (A) und mittelstarken Tc Krüppel-RNAi-Keimstreifen (B-D; als Marker für vergleichbare Stadien dient die Tc wg-expression in den Kopflappen). Der Wildtyp- Keimstreif in A zeigt die segmentalen eve-streifen 6b, 7a und 7b sowie den eve8- Paarregelstreifen in der Wachstumszone. Die Expression des Segmentpolaritätsgens Tc wg ist in diesem Embryo bis zum 11. Streifen vorangeschritten. In mittelstarken Tc Krüppel-RNAi- Keimstreifen zeigt sich gegenüber Tc Kr jaws -Keimstreifen ein etwas nach posterior verschobener Segmentierungsabbruch (B-D). Der Tc Krüppel-RNAi-Embryo in B zeigt neun intakte wg-streifen, gefolgt von zwei weiteren, unregelmäßig geformten wg-streifen sowie einer irregulären eve-expression. Die etwas schwächeren Tc Krüppel-RNAi-Keimstreifen in C und D weisen 11 intakte wg-streifen auf. Posterior davon folgt eine irreguläre Expression von Tc eve (C) bzw. ein unregelmäßiges Streifenmuster von Tc wg (D). Anterior ist immer oben. 75
94 Regulation von Hox-Genen durch Tc Krüppel Neben der Deletion von Segmenten, beobachtet man in Tc Kr jaws -mutanten Larven auch eine veränderte Identität der Segmente T1-A1 (Abb. 13F). Wie schon in der Einleitung erwähnt, wird die Identität der Segmente durch die Klasse der innerhalb der Insekten funktionell stark konservierten Hox-Gene spezifiziert (Angelini et al. 2005; Hughes and Kaufman 2002). Es war bereits gezeigt worden, dass in Tc Kr jaws -Keimstreifen die zusätzlichen maxillären und labialen Segmente im Bereich von T1-A1 mit der ektopischen Expression von Tc proboscipedia einhergehen (Anderson und Sulston 1998). Allerdings ist Tc pb erst relativ spät exprimiert und nur für die Spezifizierung der Extremitäten verantwortlich (Shippy et al. 2000), weshalb im Rahmen dieser Arbeit weitere, früher exprimierte Hox-Gene analysiert wurden Expression von Tc Dfd und Tc Ubx in Tc Kr jaws -Keimstreifen Das Hox-Gen Tc Deformed (Tc Dfd) ist in frühen Wildtyp-Keimstreifen in einem breiten anterioren Streifen exprimiert, der im Laufe der weiteren Entwicklung fortbesteht und die Anlagen des mandibulären und des maxillären Segments abdeckt (Brown et al. 1999b; Abb. 21A, C). Dieser Streifen ist in Tc Kr jaws -mutanten Keimstreifen nicht verändert, es tritt aber schon in relativ frühen Keimstreifen ein deutlich sichtbarer, weiterer Tc Dfd-Streifen auf (Abb. 21B), während ein dritter Streifen gerade im Ansatz erkennbar wird (Pfeilspitze in Abb. 21B). In vollständig ausgewachsenen Tc Kr jaws -Keimstreifen (Abb. 21D) sind schließlich drei starke Tc Dfd-Streifen sowie ein weiterer Streifen mit deutlich schwächerer Tc Dfd- Expression (Pfeilspitze in Abb. 21D) erkennbar. Im Gegensatz zu dem, auch im Wildtyp exprimierten und zwei Segmentanlagen umfassenden, anteriorsten Tc Dfd-Streifen, erstrecken sich die, jeweils durch eine Segmentanlage getrennten, ektopischen Tc Dfd-Streifen nur über die Länge von etwa einem Segment (Abb. 21D). Dabei entsprechen die zweiten und dritten Tc Dfd-Streifen den beiden ektopischen maxillären Segmenten in den Anlagen für T1 und T3, wohingegen die Intensität des letzten, schwachen Tc Dfd-Streifens offenbar zu gering für die Spezifizierung eines weiteren, maxillären Segments ist. Das Hox-Gen Tc Ultrabithorax (Tc Ubx) ist im Wildtyp in älteren Keimstreifen wie in Drosophila ab dem posterioren Teil des dritten thorakalen Segment exprimiert (Bennett et al. 1999; Abb. 21A, C). In Tc Kr jaws -Keimstreifen ist die Expression von Tc Ubx in früheren Keimstreifen nicht wesentlich verändert (Abb. 21B), in späteren Keimstreifen aber sichtbar 76
95 nach posterior verschoben und auf einen kleinen Bereich am hinteren Ende des verkürzten Keimstreifens beschränkt (Abb. 21D) Expression von Tc Scr und Tc Antp in Tc Kr jaws - und Tc Krüppel-RNAi-Keimstreifen Tc Sex combs reduced (Tc Scr) ist in Wildtyp-Keimstreifen in einem anterioren Streifen exprimiert, der den posterioren Teil der maxillären Segmentanlage sowie die gesamte labiale Segmentanlage abdeckt (Curtis et al. 2001; Abb. 21E, G). Ähnlich wie Tc Dfd, zeigt auch Tc Scr in Tc Kr jaws -Keimstreifen zwei starke, jeweils durch eine Segmentanlage voneinander getrennte, ektopische Streifen (Abb. 21F, H), während die schwache Tc Scr-Expression zwischen den ektopischen Streifen (Abb. 21H) der mesodermalen Expression von Tc Scr im ersten thorakalen Segment von Wildtyp-Keimstreifen zu entsprechen scheint (Curtis et al. 2001; Abb. 21E, G). Die ektopischen Streifen von Tc Scr sind weitgehend komplementär zu den ektopischen Tc Dfd-Streifen exprimiert (Abb. 21D, H), was exakt dem Phänotyp von Tc Kr jaws mit alternierenden ektopischen maxillären und labialen Segmenten entspricht. Die Expression von Tc Antennapedia (Tc Antp) beginnt ab der anterioren Grenze des ersten thorakalen Segments (Brown et al. 2002b) und ist somit im Vergleich zu Drosophila und Oncopeltus fasciatus konserviert (Angelini et al. 2005). Dies zeigt sich auch in der in situ- Doppelfärbung mit Tc Scr, bei der die Expression von Tc Antp in Wildtyp-Keimstreifen unmittelbar an die posteriore Grenze des ektodermalen Tc Scr-Streifens in der labialen Segmentanlage angrenzt (Abb. 21E, G). Im Gegensatz zu Tc Ubx (Abb. 21C, D), ist die anteriore Grenze von Tc Antp in Tc Kr jaws -Keimstreifen nicht verändert und liegt, wie in Wildtyp-Keimstreifen, direkt posterior des ersten Tc Scr-Streifens (Abb. 21F, H). Dadurch kommt es in Tc Kr jaws -mutanten Keimstreifen zu einer Überlagerung der ektopischen Domänen von Tc Dfd und Tc Scr mit Tc Antp (Abb. 21F, H), was den gnathal-thorakal gemischten Charakter der ektopischen maxillären Segmente in Tc Kr jaws - und Tc Krüppel- RNAi-Larven erklären könnte. In mittelstarken Tc Krüppel-RNAi-Larven tritt in der Anlage von A3 sogar ein viertes labiales Segment auf (Abb. 13D und 22A). Tatsächlich kann man in Keimstreifen der entsprechenden phänotypischen Stärke einen stark exprimierten vierten Tc Scr-Streifen beobachten (Abb. 22C). In schwachen Tc Krüppel-RNAi-Keimstreifen erscheint hingegen nur noch ein einziger ektopischer Tc Scr-Streifen (Abb. 22D), was wiederum exakt dem Erscheinungsbild schwacher Tc Krüppel-RNAi-Larven entspricht (Abb. 13C und 22B). 77
96 Abbildung 21: Expression von Hox-Genen in Wildtypen und in Tc Kr jaws -Keimstreifen In situ-doppelfärbungen gegen Tc Dfd (braune AP-Färbung) und Tc Ubx (blaue β-gal- Färbung) (A-D) sowie gegen Tc Scr (braune AP-Färbung) und Tc Antp (blaue β-gal- Färbung) (E-H) in Wildtypen (A, C, E, G) und Tc Kr jaws -mutanten-keimstreifen (B, D, F, H). In Wildtypen tritt Tc Dfd in einem gnathalen Streifen auf, welcher die Anlagen der mandibulären und maxillären Segmente beinhaltet (A, C). Dieser Streifen ist in Tc Kr jaws - mutanten-keimstreifen nicht verändert (B, D), allerdings erscheint schon in frühen Keimstreifen posterior davon zusätzlich ein zweiter Tc Dfd-Streifen, während ein dritter Streifen gerade in der Entstehung begriffen ist (Pfeilspitze in B). In ausgewachsenen Tc Kr jaws -Keimstreifen (D) erkennt man schließlich drei kräftige Tc Dfd-Streifen sowie einen deutlich schwächeren vierten Streifen (Pfeilspitze in D). Die Expression von Tc Ubx ist in frühen Stadien zwischen Wildtyp- und Tc Kr jaws -Keimstreifen nahezu identisch (A, B), in späteren Stadien kommt es aber in Tc Kr jaws zur posterioren Verschiebung der anterioren Tc Ubx-Grenze (C, D). Die geringere Länge von älteren, Tc Kr jaws -mutanten Keimstreifen resultiert aus dem posterioren Segmentierungsdefekt (siehe Abb ). Tc Scr ist im labialen Segment sowie im Mesoderm des ersten thorakalen Segmentes exprimiert (E, G). Auch bei Tc Scr treten zwei ektopische Streifen in Tc Kr jaws -Keimstreifen auf (F, H). In jüngeren Stadien (F) ist dabei der zweite Tc Scr-Streifen schon deutlich ausgebildet, während der dritte Streifen gerade erst entsteht (Pfeilspitze in F). Die Tc Scr-Expression zwischen den drei Streifen bzw. hinter dem dritten Streifen ähnelt dabei der mesodermalen Expression im ersten thorakalen Segment von Wildtyp-Keimstreifen (G, H). Die Expression von Tc Antp ist in Tc Kr jaws -Keimstreifen nicht verändert (E H). Anterior ist immer oben. 78
97
98 Abbildung 22: Expression von Tc Scr und Tc Antp in Tc Krüppel-RNAi-Keimstreifen Confocalaufnahmen von Kutikulapräparaten mittelstarker (A, siehe auch Abb. 13D) und schwacher (B, siehe auch Abb. 13C) Tc Krüppel-RNAi-Phänokopien, sowie eine in situ- Doppelfärbung gegen Tc Scr (braune AP-Färbung) und Tc Antp (blaue β-gal-färbung) in Keimstreifen mit entsprechender phänotypischer Stärke (C, D). In mittelstarken Tc Krüppel- RNAi-Keimstreifen tritt noch ein vierter Tc Scr-Streifen auf (C), und demzufolge entwickeln aus solchen Embryonen hervorgehende Larven auch vier Paar labiale Palpen (Pfeilspitzen in A). In schwachen Tc Krüppel-RNAi-Keimstreifen erscheinen hingegen nur noch zwei Tc Scr- Streifen (D). Die entsprechenden Phänokopien (B) sind schon deutlich in Kopf, Thorax und Abdomen untergliedert, zeigen aber eine, dem zweiten Tc Scr-Streifen entsprechende, Transformation von T2 zu einem Segment mit labialer Identität (Pfeilspitze in B). Anterior ist jeweils links, die Kutikulae sind in ventrolateraler Aufsicht abgebildet. 80
99 Die Effekte von Tc abd-b-rnai und Tc wurm auf die posteriore Entwicklung von Tc Kr jaws -mutanten Larven Um zu verstehen, wie in Tc Kr jaws -Larven der Segmentierungsabbruch und die homeotische Spezifizierung der posterioren Segmente zustande kommt, wurde in Tc Kr jaws -mutantem Hintergrund die Mutante Tc wurm eingekreuzt bzw. das posteriorste Hox-Gen Tc abdominal- B mittels RNAi inaktiviert (siehe Material und Methoden). Die Mutante Tc wurm wurde ursprünglich als homeotische Mutante klassifiziert, da mutante Larven eine Transformation der terminalen Segmente A9 und A10 (siehe Abb. 1) zu Segmenten mit abdominaler Morphologie aufweisen (Abb. 23A). Eine genauere Analyse zeigte außerdem, dass häufig sogar ein partielles oder ganzes 11. abdominales Segment auftritt (Abb. 23A), und dass es gelegentlich im Bereich von A7/A8 zu leichten Segmentierungsdefekten kommt (Berghammer 2003). Die funktionelle Inaktivierung von Tc abd-b führt hingegen weder zu einem 11. abdominalen Segment, noch zu einer Beeinflussung der Segmentierung. Vielmehr erkennt man nur eine Transformation der terminalen Segmente zu Segmenten mit abdominaler Identität, die aber, im Gegensatz zu der Mutante wurm, nach posterior hin in ihrem Durchmesser abnehmen (Abb. 23B). Somit zeigt der Phänotyp der Mutante wurm zusätzliche Aspekte gegenüber der Inaktivierung von Tc abd-b, welches auch durch eine Kopplungsanalyse als Kandidatengen für die Mutante wurm ausgeschlossen werden konnte (Berghammer 2003). Interessanterweise kommt es in Tc Kr jaws /Tc wurm-doppelmutanten zu einer teilweisen Rettung des Segmentierungsphänotyps von Tc Kr jaws, und es können statt 10 Segmenten (Abb. 13F) Segmente identifiziert werden (Abb. 23C, D). Da eine genaue Zählung der Segmente aufgrund der häufig ungünstigen Anordnung der gnathalen Segmente nur selten möglich war, konnten letztendlich nur acht doppelmutante Larven genauer analysiert werden, wovon zwei 12, vier 13 und zwei 14 Segmente aufweisen. In einigen Tc Kr jaws /Tc wurmdoppelmutanten Larven sind keine Segmentierungsdefekte erkennbar, und es können maximal neun gnathale und fünf abdominale Segmente gezählt werden (Abb. 23C). Wie in Tc Kr jaws alleine (Abb. 13F), tritt auch in den Tc Kr jaws /Tc wurm-doppelmutanten ein direkter Übergang von gnathalen zu abdominalen Segmenten auf; erstaunlicherweise aber erst hinter einem zusätzlichem vierten Paar von maxillären und labialen Segmenten (Abb. 23C). In einigen anderen Tc Kr jaws /Tc wurm-doppelmutanten Larven kann man aber auch Segmentierungsdefekte im Übergang zwischen den gnathalen und abdominalen Segmenten beobachten, wodurch sich die Anzahl der intakten Segmente auf acht gnathale und vier abdominale 81
100 Segmente verringert (Abb. 23D). Im Gegensatz zu den komplexen Phänotypen der Tc Kr jaws /Tc wurm-doppelmutanten, führt die Inaktivierung von Tc abd-b in Tc Kr jaws - mutantem Hintergrund zu einer einfachen Addition beider Effekte, und es treten Tc Kr jaws - ähnliche Larven mit drei abdominalen Segmenten anstelle von einem abdominalen und zwei terminalen Segmenten auf (vergl. Abb. 13F und 28E). Diese Experimente zeigen, dass die fehlende Differenzierung der letzten beiden abdominalen Segmente in terminale Strukturen nur einen Teilaspekt des Phänotyps von Tc wurm darstellt, und dass Tc wurm den posterioren Segmentierungsdefekt von Tc Kr jaws signifikant abschwächt. Abbildung 23: Die Mutante Tc wurm und Tc abd-b-rnai in Tc Kr jaws -mutantem Hintergrund Phasenkontrastaufnahme einer Tc wurm-mutanten Kutikula (A; Photo aus Berghammer 2003) sowie Confocalaufnahmen von folgenden Kutikulae: Tc abd-b-rnai (B), Tc Kr jaws /Tc wurm -Doppelmutanten (C, D) und Tc abd-b-rnai in Tc Kr jaws -mutantem Hintergrund (E). Die Kutikulae in C-E sind gegenüber denen in A und B vergrößert dargestellt. In Tc wurmmutanten Larven (A) werden 10 Segmente mit abdominaler Identität gebildet, und häufig tritt auch noch ein (partielles) 11. abdominales Segment mit Stigmaöffnungen auf. In Tc abd-b- RNAi-Phänotypen (B) erkennt man ebenfalls 10 abdominale Segmente, allerdings erscheint keine 11. Stigmenöffnung und im Gegensatz zu Tc wurm nimmt der Durchmesser der letzten beiden abdominalen Segmente ab. In Tc'Kr jaws /Tc wurm-doppelmutanten werden bis zu 14 Segmente gebildet (C). Dabei kommt es im anterioren Bereich zur alternierenden Ausbildung von vier maxillären (Pfeile in der unteren Bildhälfte) und vier labialen Segmenten (Pfeilspitzen), wobei die dritten und vierten Maxillen einen zunehmenden Übergang in Richtung Beinmorphologie aufweisen. Im posterioren Bereich sind fünf abdominale Segmente an ihren Stigmaöffnungen erkennbar (Pfeile in der oberen Bildhälfte), und es erscheinen, wie in Tc wurm alleine (A), keine terminalen Segmente. In einigen Tc Kr jaws /Tc wurm-doppelmutanten treten am Übergang von gnathalen zu abdominalen Segmenten Segmentierungsdefekte auf (D). Dabei sind nur noch die drei ersten maxillären (Pfeile) und labialen Segmente (die Pfeilspitze zeigt auf das hinter den Maxillen liegende dritte Labium, die ersten beiden sind verdeckt) normal ausgebildet. Posterior davon folgt eine Zone mit unregelmäßiger Segmentierung, wobei gelegentlich irreguläre Anhänge auftreten (* in D). Im posterioren Bereich sind noch vier abdominale Segmente an ihren Stigmaöffnungen erkennbar (Pfeile in der oberen Bildhälfte). Die Inaktivierung von Tc abd-b in Tc Kr jaws - mutantem Hintergrund bewirkt keine Ausbildung zusätzlicher Segmente (E). Während die drei maxillären (Pfeile) und labialen Segmente (Pfeilspitzen) gegenüber Tc Kr jaws nicht verändert sind, kommt es, wie in Tc abd-b-rnai alleine, zur Transformation der terminalen Segmente in abdominale Segmente (Pfeile in E markieren die Stigmaöffnungen). Anterior ist immer links; die Kutikulae sind von ventrolateral aufgenommen. 82
101
102 84
103 3.3. Die funktionelle Analyse von Tc giant Kutikulaphänotypen von Tc giant-rnai-larven Die Serie der Kutikulaphänotypen mit unterschiedlich starker funktioneller Inaktivierung von Tc giant wurde bereits Gregor Bucher ausführlich beschrieben (Bucher 2002; Bucher und Klingler 2004). Dabei zeigte sich, ähnlich wie bei der Inaktivierung von Tc Krüppel (Abb. 13), eine komplexe Überlagerung von homeotischen Transformationen und Defekten in der Segmentierung (Abb. 24). Interessanterweise stellt der durch eine Transformation gnathaler Segmente zu thorakaler Identität gekennzeichnete homeotische Phänotyp von Tc giant (Abb. 24) genau das Gegenteil zu dem Phänotyp der Nullmutante Tc Kr jaws dar (thorakale Segmente mit gnathaler Identität; siehe Abb. 13). In Tc giant-rnai spricht die Regulation der homeotischen Identität am sensitivsten auf eine Verringerung der Tc giant-aktivität an, da in den schwächsten Tc giant-rnai-phänotypen eine Transformation der maxillären und labialen Segmente zu T1 und T2, aber kein Segmentierungsdefekt beobachtet werden kann (Abb. 24A). Mit ansteigender phänotypischer Stärke (Abb. 24B-D) kommt es zu einer zunehmenden Deletion der abdominalen Segmente, wobei sich aber keine schwerpunktmäßig betroffene Region ermitteln ließ (Bucher und Klingler 2004). Im anterioren Bereich werden die ersten drei thorakalen Segmente (von denen die ersten beiden eigentlich transformierte gnathale Segmente sind) scheinbar normal ausgebildet, während die vierten und fünften Segmente mit thorakaler Identität zunehmend von der posterioren Deletion erfasst werden (Abb. 24B-D). Das Expressionsmuster des Segmentpolaritätsgen Tc engrailed zeigte jedoch eine zusätzliche, in den Kutikulaphänotypen verborgene, Defektzone zwischen dem zweiten und dritten Segment mit thorakaler Identität (Bucher und Klingler 2004). Neben der von Gregor Bucher beschriebenen phänotypischen Serie konnte eine weitere Klasse von Phänotypen beobachtet werden, in der mehr als fünf Beinpaare erkennbar sind (Abb. 24E-G). Die in ihrer Stärke zwischen der rein homeotischen Transformation (Abb. 24A) und dem deutlichen Auftreten abdominaler Segmentierungsdefekte (Abb. 24B) liegenden Phänotypen konnten in drei bis fünf Wochen nach der Injektion gewonnenen Gelegen bei 17 von 374 Larven beobachtet werden (4,5%). Dabei konnten in 13 Fällen sechs Beine (Abb. 23E) und in 4 Fällen sogar sieben Beine (Abb. 24F, G) auf einer Körperseite gezählt werden. Ähnlich wie in etwas stärkeren Phänotypen (Abb. 24B), ist das vorletzte Beinpaar dabei häufig degeneriert, während das letzte Beinpaar wiederum deutlicher ausgeprägt ist (Abb. 24F, G). Daneben kommt es bei dieser Klasse von Phänotypen auch 85
104 häufig zu Asymmetrien bezüglich der Anzahl an Beinen in den Körperhälften, wobei die Unregelmäßigkeiten stets im Bereich des dritten oder vierten thorakalen Segments liegen (Abb. 24F, G). Die Interpretation des gesamten Spektrums an Tc giant-rnai-phänotypen erfolgt nach der Analyse der molekularen Marker im Rahmen der Diskussion (Kapitel ). Abbildung 24: Tc giant-rnai-phänotypen (A-D) Dunkelfeldaufnahmen von Tc giant-rnai-kutikulae mit zunehmender phänotypischer Stärke (A, B, D aus Bucher und Klingler 2004, C aus der eigenen Diplomarbeit (Cerny 2002)), sowie (E-G) Autofluoreszenzaufnahmen (E, G mit Maximalprojektion eines Z-Stapels (siehe Material und Methoden) von seltenen Tc giant-rnai-kutikulae mit mehr als fünf Beinpaaren (vergrößerte Darstellung gegenüber A-D). In schwachen Tc giant-rnai- Phänotypen (A) sind die maxillären und labialen Segmente zu thorakalen Segmenten transformiert. Die eigentlichen thorakalen Segmente nehmen die Identität von T3 an, sie sind aber, ebenso wie die abdominalen und terminalen Segmente, nicht von Segmentierungsdefekten betroffen. In mittelstarken Tc giant-rnai-phänotypen (B) tritt eine Deletion von abdominalen Segmenten auf, die aufgrund der einheitlichen Morphologie der abdominalen Segmente nicht näher charakterisiert werden kann. Zusätzlich sind die vierten (Pfeilspitze in B) und fünften thorakalen Segmente und ihre Extremitäten oft unregelmäßig ausgebildet. In starken Tc giant-rnai-phänotypen (C, D) verschwinden diese Segmente, und es kommt zur zunehmenden Deletion von abdominalen Segmenten, bis im Extremfall nur noch ein abdominales Segment ausgebildet wird (Pfeilspitze in D). In einigen Larven mit schwächerem Tc giant-knockdown treten sechs (E, F) oder sieben (G) Beine pro Körperseite auf. Dabei kommt es häufig im Bereich des dritten und vierten Beinpaars zu Asymmetrien bezüglich der Anzahl an Beinen pro Körperseite (in E erkennt man auf der Vorderseite mit 3v und 4v zwei Beine, während die Hinterseite mit 4h nur ein Bein aufweist; analog in F mit 3l, 4l (links) und 3r (rechts)). Die Beinextremitäten im vorletzten thorakalen Segment sind öfters nur partiell ausgebildet (G) oder vollständig verkümmert (F), wohingegen sie im letzten thorakalen Segment wieder deutlicher ausgeprägt sind (F, G, siehe auch B). In den Larven mit mehr als fünf Beinpaaren (E-G) treten nur schwache Segmentierungsdefekte auf, so dass eine Zählung der in F vollzählig gebildeten Segmente eine homeotische Transformation von A1 und A2 zu thorakalen Segmenten nahe legt. Anterior ist immer links; U = Urogomphi, P = Pygopodien. 86
105
106 Regulation von Gap-Genen durch Tc giant Wie bereits im Kapitel beschrieben, fungiert die anteriore Tc giant-domäne als Repressor für Tc Krüppel, wodurch die Tc Krüppel-Domäne in Tc giant-rnai-keimstreifen um ein Segment nach anterior expandiert (Abb. 14E, F). Die posteriore Tc knirps-expression liegt innerhalb der posterioren Tc giant-domäne (Abb. 9J-L); es konnten aber keine Interaktionen zwischen diesen beiden Genen gefunden werden (Daten nicht gezeigt) Expression von Tc hunchback in Tc giant-rnai-embryonen Es konnte bereits gezeigt werden, dass der Verlust der Tc Krüppel-Aktivität zur fehlenden Aktivierung der posterioren Tc giant-domäne sowie zu einer drastischen anterioren Verschiebung der posterioren Tc hunchback-domäne führt (Abb. 14A-D und 16). Zur Klärung der Frage, ob die Reprimierung der posterioren Tc hunchback-domäne direkt durch Tc Krüppel oder indirekt über die posteriore Tc giant-domäne vermittelt wird, wurde die Expression von Tc hunchback in Tc giant-rnai-keimstreifen untersucht. Der Knockdown von Tc giant (und anderen Genen) mittels parentaler RNAi führt aber, im Gegensatz zum einheitlichen Phänotyp der genetischen Nullmutante Tc Kr jaws, zu deutlichen experimentellen Schwankungen in der Stärke der erzielten Phänotypen. Deshalb empfiehlt sich in RNAi-Embryonen eine zusätzliche Färbung gegen ein weiteres Gen, das in Embryonen mit starken Knockdown eine deutliche und spezifische Veränderung in seinem Expressionsmuster zeigt. In diesem Experiment wurde daher eine Tc hunchback/tc wingless- Doppelfärbung durchgeführt, um an Hand der gestörten Tc wg-expression (siehe Abb. 29) die stark betroffenen Keimstreifen identifizieren zu können. Es zeigte sich, dass weder die anteriore noch die posteriore Tc hunchback-domäne durch Tc giant reguliert wird (Abb. 25). In frühen Stadien ist in Tc giant-rnai-keimstreifen die anteriore Tc hunchback-domäne nicht wesentlich gegenüber vergleichbaren Wildtyp- Keimstreifen verändert (Abb. 16A1 und 25A). In Tc giant-rnai-keimstreifen mittleren Alters (Abb. 25B) wird der thorakale Segmentierungsdefekt erstmals erkennbar (Pfeilspitzen in Abb. 25B, siehe auch Abb. 29), und es ist, wie in vergleichbaren Wildtyp-Keimstreifen (Abb. 16A2), noch keine posteriore Tc hunchback-expression erkennbar. In späteren Tc giant-rnai-keimstreifen tritt schließlich die posteriore Tc hunchback-domäne zuerst mit einer relativ scharfen anterioren Grenze (Abb. 25C) und dann mit einem diffuseren Muster auf (Abb. 25D). Obwohl die späten Tc giant-rnai-keimstreifen aufgrund ihrer defekten 88
107 Segmentierung in ihrer Morphologie gestört sind und auch eine geringere Länge als späte Wildtyp-Keimstreifen aufweisen (siehe Abb ), lässt sich die posteriore Tc hunchback- Expression in Tc giant-rnai-keimstreifen (Abb. 25C, D) doch gut mit der in späten Wildtyp-Keimstreifen (Abb. 16A3, A4) vergleichen und unterscheidet sich deutlich von der nach anterior verschobenen Expression in Tc Kr jaws -Keimstreifen (Abb. 16C2, C3). Tc giant übt also keinen Einfluss auf die posteriore Tc hunchback-domäne aus und ist daher auch nicht an der Vermittlung der anterioren Repression durch Tc Krüppel beteiligt. Abbildung 25: Expression von Tc hunchback und Tc wg in Tc giant-rnai-embryonen In situ-doppelfärbung gegen Tc hunchback (braune AP-Färbung) und Tc wingless (blaue β- Gal-Färbung) in zunehmend älteren Tc giant-rnai-keimstreifen (A-D). Die anteriore Tc hunchback-domäne ist gegenüber dem Wildtyp nicht verändert (A). In Tc giant-rnai- Keimstreifen mittleren Alters ist noch keine Expression der posterioren Tc hunchback- Domäne erkennbar (B). Die fusionierenden wg-streifen von T2 und T3 (Pfeilspitzen in B, siehe auch Abb. 29) kennzeichnen dabei einen starken Knockdown von Tc giant in diesem Keimstreif. In späteren Keimstreifen taucht zu einem ähnlichen Zeitpunkt wie im Wildtyp (siehe Abb. 16A3) die posteriore Tc hunchback-expression in der Wachstumszone auf (C). Aufgrund des posterioren Segmentierungsdefekts sind die Tc giant-rnai-keimstreifen allerdings gegenüber Wildtyp-Keimstreifen verkürzt (C, D). Beim Abschluss des Keimstreifwachstums nimmt die posteriore Tc hunchback-domäne, ähnlich wie im Wildtyp (Abb. 16A4), eine diffuse Form an, und in den anterioren Segmenten erscheint die neuronale Expression von Tc hunchback (D). Anterior ist bei allen Keimstreifen oben. 89
108 Autoregulation von Tc Krüppel und Tc giant Bei der Analyse von Tc Kr jaws im Rahmen der eigenen Diplomarbeit (Cerny 2002) stellte sich heraus, dass die Expression von Tc Krüppel in Tc Kr jaws -mutanten Embryonen sowohl im späten Blastoderm als auch in frühen Keimstreifstadien drastisch reduziert ist (Abb. 26A-D). Da die Mutation in Tc Kr jaws nicht im Promotorbereich, sondern in der DNA-bindenden Zinkfinger-Domäne liegt (Cerny et al. 2005), lässt die verminderte Aktivität von Tc Krüppel auf eine starke Aktivierung der eigenen Transkription durch das Tc Krüppel-Protein schließen. Da es für Tc giant leider keine Mutante gibt, und da RNAi über die spezifische Zerstörung der jeweiligen mrna wirkt, wurde zur Analyse einer möglicherweise vergleichbaren Autoaktivierung von Tc giant auf die embryonale Injektion eines antisense-morpholino- Oligonucleotides zurückgegriffen (siehe Material und Methoden; Bucher 2002 sowie Bucher und Klingler 2004). In den zuvor injizierten Embryonen ließ sich jedoch keine signifikante Änderung der Transkriptionsintensität von Tc giant feststellen (Abb. 26E, F), obwohl der aus Tc giant-rnai-experimenten bekannte Verlust des runt2-streifens (Abb. 27D) eine starke translationale Inaktivierung von Tc giant in dem betroffenen Keimstreif zeigt. Bei der Interpretation dieses Experiments gilt es jedoch zu bedenken, dass gebundene Morpholino- Oligonukleotide den Abbau der jeweiligen mrna behindern und somit das in situ-signal erhöhen können. Daher sollte man eigentlich Intronsequenzen (die es aber in Tc giant nicht gibt) in frisch synthetisierten Transkripten detektieren. Trotz dieses möglichen Einwands erscheinen die Daten aber aussagekräftig genug, um eine starke, mit Tc Krüppel vergleichbare Autoaktivierung für Tc giant mit hoher Wahrscheinlichkeit ausschließen zu können. 90
109 Abbildung 26: Autoregulation von Tc Krüppel und Tc giant In situ-ap-färbung gegen Tc otd und Tc Krüppel (A-D; aus Diplomarbeit A. Cerny) in Wildtyp- (A, C) und Tc'Kr jaws -Embryonen (B, D) sowie in situ-doppelfärbung gegen Tc giant (braune AP-Färbung) und Tc runt (blaue β-gal-färbung) in einem Wildtyp-Embryo (E) bzw. in einem Tc giant-morpholino injizierten Embryo (F). Im späten Wildtyp-Blastoderm ist Tc Krüppel im posterioren Bereich exprimiert (A), bevor sich im frühen Keimstreifstadium ein breiter Streifen in den thorakalen Segmentanlagen bildet (C). In Tc Kr jaws -mutanten Embryonen aus der gleichen Färbereaktion ist die Expression von Tc Krüppel drastisch reduziert (B, D), wobei Tc otd als interner Marker für eine vergleichbare AP-Färbung dient (A-D). Die etwas lateral verbreiterte Tc otd-expression im Tc Kr jaws -Blastoderm (B) ergibt sich durch eine leichte Stauchung des Embryos. Die Expression von Tc giant ist in Morpholino-injizierten Embryonen weder in der anterioren noch in der posterioren Domäne verändert (E, F; Details siehe Text). Der Verlust des runt2-streifens im Tc giant-morpholino injizierten Keimstreif (F) ist ein spezifischer Effekt des Verlusts an Tc giant-aktivität (siehe Abb. 27) und zeigt so eine funktionelle Inaktivierung von Tc giant in diesem Keimstreifen an. Anterior ist immer oben, in den Blastodermstadien (A, B) ist ventral links; r = runt, gt = giant. 91
110 Regulation von Paarregel- und Segmentpolaritätsgenen durch Tc giant Die funktionelle Inaktivierung von Tc giant führt bei starken Phänotypen (Abb. 24C, D) zu einem komplexen Defekt in der Segmentierung, der als weiträumige Störung der Tc engrailed-expression in den Segmentanlagen von T1 bis A9 beschrieben wurde (Bucher und Klingler 2004). Da Gap-Gene in Drosophila ihre unmittelbare Segmentierungsfunktion aber durch die Regulation der Paarregelgene ausüben, wurde zur detaillierteren Analyse die Expression der Paarregelgene Tc runt und Tc eve in Kombination mit den Segmentmarkern Tc en und Tc wg über alle Stadien der Segmentierung in Tc giant-rnai-embryonen verfolgt Expression von Tc runt in Tc giant-rnai-embryonen In späten Wildtyp Blastodermembryonen ist Tc runt in zwei Streifen exprimiert (Abb. 27A, siehe auch Abb. 33I, J), von denen der zweite etwa zeitgleich mit der posterioren Invagination gebildet wird. In Tc giant-rnai-blastodermembryonen erkennt man hingegen selbst bei deutlich ausgeprägter posteriorer Invagination keinen runt2-streifen (Abb. 27B), womit der früheste Segmentierungsdefekt also schon in späten Blastodermstadien beobachtet werden kann. Kurz darauf taucht in jungen Tc giant-rnai-keimstreifen ein weiterer runt-streifen nahe der Wachstumszone auf (Abb. 27D), der jedoch vom Zeitpunkt seines Erscheinens eher dem runt3-streifen entspricht, obwohl er im Vergleich zu Wildtyp-Keimstreifen deutlich näher an runt1 liegt (Abb. 27C, D). Diese Interpretation wird auch durch feine runt2- Zwischenstreifen unterstützt, die in Keimstreifen mit schwächerem Tc giant-knockdown erscheinen (Abb. 27D). Nach runt3 wird auch der runt4-streifen in wachsenden Tc giant- RNAi-Keimstreifen wie im Wildtyp exprimiert, auch wenn die Embryonen aufgrund des anterioren Segmentierungsdefektes nun bereits deutlich verkürzt sind (Abb. 27E, F). Im Gegensatz zur anfänglich normalen Expression (Abb. 27D, F), verliert der dritte runt-streifen in Tc giant-rnai-keimstreifen aber im weiteren Verlauf seine korrekte Form (Abb. 27H) und es kommt zu einer in diesem Bereich auftretenden Wachstumsstörung und Verbreiterung der Keimstreifen (Abb. 27H, J). In diesen Keimstreifen wird außerdem auch der runt5- Streifen gebildet, allerdings werden die runt-streifen 4 und 5 nur noch unzureichend voneinander getrennt (Abb. 27H, J). Posterior des runt5-streifens werden schließlich keine neuen runt-streifen mehr abgespalten, und es kommt zum allmählichen Erlöschen der runt- Expression im Bereich der Wachstumszone (Abb. 29A-D). 92
111 Expression von Tc even-skipped in Tc giant-rnai-embryonen Die früheste Missregulation von Tc eve in Tc giant-rnai-embryonen kann erst in jungen Keimrudimenten beobachtet werden, wenn posterior der schon segmental aufgespaltenen Streifen eve1a und eve1b eine großflächige und einheitliche Tc eve-expression auftritt, die als Fusion der eve2- und eve3-streifen interpretiert werden kann (Abb. 28A, B). Eine möglicherweise schon früher im Blastoderm bestehende Fusion von eve2 und eve3 konnte leider nicht nachgewiesen werden, da diese Streifen auch im Wildtyp aus einer zunächst einheitlichen Domäne entstehen (Daten nicht gezeigt). Die uniforme posteriore Expression von Tc eve bleibt auch in etwas späteren Tc giant-rnai-keimstreifstadien erhalten (Abb. 28C, D), wobei die Expression von Tc eve bis ans posteriore Ende des Keimstreifs reicht und somit über das hintere Ende des eve3-streifens in vergleichbaren Wildtyp-Keimstreifen hinausragt (die Altersbestimmung erfolgte über den Abstand eve1a- und eve1b-streifen sowie über die Morphologie der Kopflappen). Im Laufe des weiteren Wachstums von Tc giant- RNAi-Keimstreifen kristallisiert sich der eve2a-streifen am anterioren Rand der großflächigen Tc eve-expression heraus (Abb. 28E-H) und es bildet sich ein Bereich geringerer Tc eve-expression innerhalb der großflächigen Tc eve-domäne (Abb. 28H). Noch etwas später verschwinden in Tc giant-rnai-keimstreifen die segmentalen Streifen eve1a und eve1b, während posterior des eindeutig identifizierbaren eve2a-streifens zwei schmale eve-streifen erscheinen (Pfeilspitzen in Abb. 28J). Diese Streifen ähneln in ihrem Abstand den eve-streifen 3a und 3b in vergleichbaren Wildtyp-Keimstreifen (Abb. 28I, J), eine eindeutige Zuordung ist aber wegen der zu stark beeinträchtigten Expression der primären eve-paarregelstreifen nicht möglich. Ähnlich wie bei Tc runt, werden auch bei Tc eve keine neuen posterioren Streifen während der späteren Segmentierung von Tc giant-rnai- Keimstreifen abgespalten (Abb. 29E-G). 93
112 Abbildung 27: Expression von Tc runt in Wildtypen und in Tc giant-rnai-embryonen In situ-färbung gegen Tc runt (braune AP-Färbung in A-J) und Tc wingless (blaue β-gal- Färbung in C, E, G, I) bzw. Tc engrailed (blaue β-gal-färbung in H, J) in Wildtypen (A, C, E, G, I) und in Tc giant-rnai-embryonen (B, D, F, H, J). In späten Blastodermstadien mit beginnender posteriorer Invagination beobachtet man in Tc giant-rnai-embryonen den Verlust des zweiten runt-streifens (A, B), der auch in den darauf folgenden frühen Keimstreifstadien fehlt (C-F). Die runt-streifen 3 und 4 werden hingegen in Tc giant-rnai- Keimstreifen primär normal ausgebildet (D, F). In Wildtypen bleiben die runt3- und runt4- Streifen im weiteren Verlauf relativ schmal, während an der Wachstumszone der runt5- Streifen gebildet wird (G, I). In vergleichbaren Tc giant-rnai-embryonen erkennt man in diesem Stadium hingegen eine sekundären Verbreiterung von runt3, einen deutlich verringerten Abstand zwischen den runt4- und runt5-streifen sowie eine in diesem Bereich gestörte Morphologie der Keimstreifen (H, J). Außerdem werden in Tc giant-rnai- Keimstreifen als Folge der anterioren Deletion nur drei anstelle von fünf Segmentpolariätsstreifen vor dem runt3-streifen angelegt (G-J; der wg6-streifen in I überlappt schon mit runt3). Anterior ist immer oben, in den Blastodermembryonen (A, B) ist dorsal rechts; r = runt, wg = wingless, en = engrailed. 94
113
114 Abbildung 28: Expression von Tc eve in Wildtypen und in Tc giant-rnai-embryonen In situ-färbung gegen Tc eve (braune AP-Färbung) in Wildtypen (A, C, E, G, I) und in Tc giant-rnai-keimstreifen (B, D, F, H, J). Der älteste Wildtyp-Keimstreif in I wurde noch zusätzlich gegen Tc engrailed (blaue β-gal-färbung) gefärbt. Die Expression der sekundären eve1a- und eve1b-streifen ist in Tc giant-rnai-embryonen nicht verändert (A, B, die Pfeilspitze in B zeigt auf eve1b). In frühen Tc giant-rnai-keimrudimenten tritt im posterioren Bereich anstatt distinkter Streifen eine größere Tc eve-domäne auf, die man als Fusion der eve2- und eve3-streifen interpretieren kann (B). Dieses Muster bleibt auch noch in etwas späteren Stadien erhalten (C, D), bevor sich am anterioren Rand der flächigen Expression ein Streifen bildet, der dem, in vergleichbaren Wildtypen mittlerweile gebildeten, sekundären eve2a-streifen entspricht (E, F). Während der eve2a-streifen im Laufe der weiteren Entwicklung von Tc giant-rnai-keimstreifen deutlich hervortritt (H), ist posterior davon kein den eve3- und eve4-streifen entsprechendes Expressionsmuster erkennbar (G, H). Erst später treten posterior von eve2a zwei segmentale eve-streifen auf (Pfeilspitzen in J), die den in vergleichbaren Wildtypen zu diesem Zeitpunkt gebildeten, segmentalen eve3a- und eve3b-streifen ähneln (I, J). Anterior ist immer oben. 96
115
116 Expression von Tc engrailed und Tc wingless in Tc giant-rnai-embryonen Die ersten drei Streifen der Segmentpolaritätsgene Tc engrailed und Tc wingless werden in Tc giant-rnai-keimstreifen in Übereinstimmung mit dem ungestörten Muster der segmentalen eve-streifen 1a, 1b und 2a normal exprimiert (Abb. 27H, J und 29A-G). Posterior davon treten hingegen zwei unregelmäßige Tc en- (Pfeilspitzen in Abb. 29A) bzw. Tc wg-streifen (Pfeilspitzen in Abb. 29E, siehe auch Abb. 25B) in deutlich verringertem Abstand auf. Diese Streifen entstehen aus dem Bereich, in dem die runt2- (Abb. 27) und hairy2-streifen (M. Aranda, pers. Mitteilung) fehlen, sowie die eve2- und eve3-streifen miteinander fusioniert sind (Abb. 28). Da die zweiten Streifen von runt und hairy die Anlagen für das erste thorakale Segment abdecken, kommt es in Tc giant-rnai-embryonen aufgrund der fehlenden Aktivierung dieser Streifen vermutlich schon in früheren Stadien zur Deletion der Segmentanlage von T1. Demzufolge fehlt in Tc giant-rnai-keimstreifen vermutlich auch der sekundäre eve2b-streifen, und die zwei später auftretenden, segmentalen eve- Streifen (Abb. 28J) würden den eve-streifen 3a und 3b entsprechen. Aus diesen Streifen gehen später vermutlich die zwei Segmentpolaritätsstreifen mit sehr geringem Abstand zueinander hervor (Pfeilspitzen in Abb. 29A, E), die später miteinander zu fusionieren beginnen (Pfeilspitzen in Abb. 29B, siehe auch Abb. 25C). Dabei entsteht schließlich ein, häufig verbreiterter, Segmentpolaritätsstreifen mit zwischenzeitlich erhöhtem Abstand zum anterior davon gelegenen dritten Streifen (Pfeilspitzen in Abb. 29C, F). Gegen Ende des Keimstreifwachstums reguliert sich der Abstand zwischen dem vermutlich T2/T3 entsprechenden Fusionsstreifen und dem dritten gnathalen Streifen auf ein normales Maß (Pfeilspitzen in Abb. 29D, G; siehe auch Abb. 25D), wobei gelegentlich dazwischen noch vereinzelte Zellen eine Expression der Segmentpolaritätsgene zeigen (Abb. 29C, D). Posterior davon beginnt dann eine, wenn auch etwas verkürzte, abdominale Segmentanlage auszuwachsen (Abb. 29A-G). Dabei werden keine neuen Paarregelstreifen mehr von der Wachstumszone abgespalten, während die Expression der Paarregelgene Tc runt (Abb. 29A- D) und Tc eve (Abb. 29E-G) im Bereich der Wachstumszone langsam erlischt. Ohne den Input von Paarregelstreifen ist die Expression der Segmentpolaritätsgene im gesamten Abdomen völlig irregulär, ehe, vermutlich durch Interaktionen auf Ebene der Segmentpolaritätsgene, einzelne segmentale Streifen entstehen. (Abb. 29 und Abb. 25C, D). 98
117 Abbildung 29: Expression von Paarregel- und Segmentpolaritätsgenen in späten Tc giant-rnai-keimstreifen (A-D) In situ-doppelfärbungen gegen Tc runt (braune AP-Färbung) und Tc engrailed (blaue β-gal-färbung) bzw. (E-G) gegen Tc eve (braune AP-Färbung) und Tc wingless (blaue β- Gal-Färbung) in späten Tc giant-rnai-keimstreifen. In diesen Keimstreifen werden keine Paarregelstreifen mehr von der Wachstumszone abgespalten, während die Expression von Tc runt (A-D) und Tc eve (E-G) in der Wachstumszone selbst allmählich erlischt. Die ersten drei Streifen von Tc en (A-D) und Tc wg (E-G) werden in allen Stadien normal ausgebildet, doch posterior davon treten bei beiden Segmentpolaritätsgenen zunächst zwei nah beieinander gelegene, thorakale Streifen auf (Pfeilspitzen in A und E). Im weiteren Verlauf fusionieren diese beiden Streifen (Pfeilspitzen in B), wobei sich häufig vorübergehend ein vergrößerter Abstand zwischen den jeweils dritten en- bzw. wg-streifen und dem Fusionsstreifen (Pfeilspitzen in C und D bzw. in F und G) ergibt. Posterior der verdickten thorakalen Segmentanlagen entsteht in den abdominalen Segmentanlagen ein irreguläres Muster der Tc en- (B-D) bzw. Tc wg-expression (F, G). Anterior ist immer oben; r = runt, en = engrailed, wg = wingless. 99
118 Regulation von Hox-Genen durch Tc giant Da die homeotische Transformation gnathaler Segmente in Tc giant-rnai-larven (Abb. 24A) die Missregulation von Hox-Genen vermuten lässt, wurde die Expression von Tc Sex combs reduced (Tc Scr), Tc Antennapedia (Tc Antp) und Tc Ultrabithorax (Tc Ubx) in Tc giant-rnai-keimstreifen untersucht. In Wildtyp-Keimstreifen ist Tc Scr in einem kräftigen Streifen in der labialen Segmentanlage (PS-2) exprimiert, während die Expression von Tc Antp unmittelbar posterior davon (PS-3) im anterioren Bereich der ersten thorakalen Segmentanlage beginnt (Abb. 30A). In Übereinstimmung mit dem Phänotyp (Abb. 24), expandiert in Tc giant-rnai-keimstreifen die vordere Grenze der Tc Antp-Domäne um zwei Segmente in Richtung anterior, während im Falle von Tc Scr die ektodermale Expression in der labialen Segmentanlage verschwindet (Abb. 30B). Die mesodermale Expression von Tc Scr (im Wildtyp in T1) bleibt hingegen auch in Tc giant-rnai-keimstreifen erhalten, allerdings folgt sie dort der anterioren Expansion von Tc Antp und tritt in der zweiten Segmentanlage auf (Abb. 30A, B), die in Tc giant-rnai aber die Identität von T1 annimmt. In starken Tc giant-rnai-keimstreifen kommt es scheinbar zu einer anterioren Expansion des in Wildtypen ab dem posterioren Teil des dritten thorakalen Segments exprimierten Hox- Gens Tc Ubx (Abb. 30C, D). In Wirklichkeit sind in starken Tc giant-rnai-keimstreifen aber die ersten beiden thorakalen Segmente deletiert (Abb ), so dass letztendlich die Kombination aus einer um zwei Segmente nach anterior expandierten Tc Antp-Domäne und einer Deletion der ersten beiden thorakalen Segmente für einen natürlichen Abstand von zwei Segmenten zwischen den anterioren Grenzen von Tc Antp und Tc Ubx sorgt (Abb. 30D). In schwachen Tc giant-rnai-phänotypen zeigen die vierten und fünften Segmente die Identität von T3 (Abb. 24A). Ob es in diesem Hintergrund zu einer Missregulation von Tc Ubx kommt, wurde im Rahmen dieser Arbeit allerdings nicht untersucht. 100
119 Abbildung 30: Expression von Hox-Genen in Wildtyp- und Tc giant-rnai-embryonen (A, B) In situ-doppelfärbungen gegen Tc Scr (braune AP-Färbung) und Tc Antp (blaue β- Gal-Färbung) sowie (C, D) gegen Tc Antp (braune AP-Färbung) und Tc Ubx (blaue β-gal- Färbung) in Wildtypen (A, C) und Tc giant-rnai-keimstreifen (B, D). In Wildtypen ist Tc Scr im labialen Segment exprimiert, während die Tc Antp-Domäne mit dem ersten thorakalen Segment beginnt (A, gleicher Embryo wie in Abb. 21G). In Tc giant-rnai- Keimstreifen verschwindet der ektodermale Streifen von Tc Scr (die schwache Expression im zweiten Parasegment entspricht der mesodermalen Expression im ersten thorakalen Segment von Wildtyp-Keimstreifen), und es kommt zu einer anterioren Expansion der Tc Antp- Domäne um zwei Segmente (B). Die Expression von Tc Ubx beginnt in Wildtyp- Keimstreifen ab dem dritten thorakalen Segment und somit zwei Segmente hinter der von Tc Antp (C). In starken Tc giant-rnai-keimstreifen beginnt die Expression von Tc Ubx ab dem durch die thorakalen Segmentierungsdefekte verdickten Bereich des Keimstreifens (D). Anterior ist immer oben, gt = giant. 101
120 102
121 3.4. Die funktionelle Analyse von Tc hunchback Die Funktion von Tc hunchback wurde bereits in der Literatur beschrieben, wobei aus Kutikulaphänotypen wie in Abb. 31C auf eine Deletion der maxillären, labialen und thorakalen Segmente geschlossen wurde (Schröder 2003). Auf dieser Basis wurde daraufhin eine konservierte Funktion der anterioren hunchback-domänen in Drosophila und Tribolium postuliert (Schröder 2003). Später wurden die Orthologen von hunchback auch in den Kurzkeim-Insekten Oncopeltus fasciatus und Gryllus bimaculatus funktionell untersucht. Dabei entstanden vergleichbare Phänotypen, die aber mit Hilfe von molekularen Markern als Folge einer homeotischen Transformation der gnathalen und thorakalen Segmente identifiziert werden konnten (Liu und Kaufman 2004a; Mito et al. 2005). Da in Tribolium schwächere Tc hunchback-rnai-phänotypen ebenfalls homeotische Transformationen in den anterioren Segmenten aufweisen und nicht etwa eine partielle Deletion zeigen (Abb. 31A), wurde die publizierte Interpretation des Tc hunchback-phänotyps in Frage gestellt und die Funktion von Tc hunchback einer erneuten Untersuchung unterzogen Kutikulaphänotypen von Tc hunchback-rnai In den Kutikulaphänotypen von Tc hunchback-rnai kommt bei einer schwachen funktionellen Inaktivierung, zunächst unter Beibehaltung aller Segmente, zu einer partiellen homeotischen Transformation der Segmente G2 (maxilläres Segment) bis T3 in Richtung abdominale Identität (Abb. 31A). In stärkeren Phänotypen sind dann alle Segmente hinter dem maxillären Segment als abdominale Segmente ausgebildet, wobei deren Anzahl je nach phänotypischer Stärke variiert (Abb. 31B-E). In mittelstarken Phänotypen beobachtet man mitunter deutlich mehr Segmente mit abdominaler Identität als im Wildtyp (11 in Abb. 31B; die Zählweise in den Abb. 31B-E bezieht sich auf die Segmente mit abdominaler Morphologie und nicht auf den segmentalen Ursprung). Sehr häufig erscheinen dann allerdings Phänotypen mit genau acht abdominalen Segmenten posterior des mandibulären Segments (Abb. 31C), die aber noch nicht die stärksten Phänotypen darstellen. In diesen treten posterior des vierten abdominalen Segments Defekte in der Musterbildung auf (Abb. 31D), so dass in manchen Larven nur noch vier abdominale Segmente übrig bleiben (Abb. 31E). Neben der zunehmenden Deletion abdominaler Segmente ist in stärkeren Phänotypen auch der Verlust der terminalen Segmente zu beobachten (Abb. 31C-E), welche in den 103
122 schwächeren Phänotypen noch normal ausgebildet sind (Abb. 31A, B). Außerdem erkennt man in den stärkeren Phänotypen eine (nur in Fluoreszenzbildern gut sichtbare) Verdickung in dem evertierten Enddarm (Abb. 31E). Neben seiner Expression im eigentlichen Embryo ist Tc hunchback auch in der Serosaanlage exprimiert, wobei jedoch in Höchst gefärbten Tc hunchback-rnai-embryonen keine Beeinträchtigung dieses Gewebes festgestellt werden konnte (Daten nicht gezeigt). Abbildung 31: Tc hunchback-rnai-phänotypen Kutikulapräparate von Tc hunchback-rnai-larven zunehmender phänotypischer Stärke (A, B: Confocalaufnahmen; C: Dunkelfeldaufnahme; E, F: Epifluoreszenzaufnahme). In den schwächsten Phänotypen beobachtet man posterior des mandibulären Segments eine partielle homeotische Transformation der gnathalen und thorakalen Segmente in Richtung abdominale Identität (A). Mit zunehmender Inaktivierung von Tc hunchback erscheinen posterior vom mandibulären Segment nur noch Segmente abdominaler Identität (B-E, die Zählweise der mit A bezeichneten Segmente bezieht sich auf Segmente mit abdominaler Morphologie). Mit zunehmender Stärke des Tc hunchback-rnai-knockdowns verringert sich die Anzahl der Segmente mit abdominaler Identität von 11 auf 4 (B-E), und es tritt außerdem ein Verlust der terminalen Strukturen auf (C-E). Zusätzlich beobachtet man in starken Tc hunchback-rnai- Phänotypen häufig einen nach außen gerichteten und stark verdickten Enddarm (E). Anterior ist immer links, die Larve in B ist in ventrolateraler Ansicht abgebildet, alle anderen Larven sind in ventraler Aufsicht photographiert. Mx = maxilläres Segment. 104
123
124 Regulation von Gap-Genen durch Tc hunchback In Drosophila liefert der durch die anteriore Dm hunchback-domäne erzeugte Proteingradient ein wichtiges Signal zur Regulation der posterior davon gelegenen Gap-Gen-Domänen. Dabei wird Dm Krüppel durch Dm hunchback in niedriger Konzentration aktiviert und in hoher Konzentration inhibiert, während Dm knirps und Dm giant durch Dm hunchback nur inhibiert werden (Hülskamp et al. 1990; Kraut und Levine 1991a). Zur Untersuchung einer möglicherweise vergleichbaren Funktion der im posterioren Blastoderm gelegenen Gap-Gen- Domäne von Tc hunchback, wurden daher die Expressionsmuster der Gap-Gen-Orthologen Tc Krüppel, Tc knirps und Tc giant in Tc hunchback-rnai-embryonen analysiert. Die Gap-Gen-Domäne von Tc Krüppel umfasst im Wildtyp den posterioren Bereich des Blastoderms, und bildet in jungen Keimstreifen eine breite Domäne in den thorakalen Segmentanlagen aus (Abb. 32A, C). In Tc hunchback-rnai-embryonen fehlt die Tc Krüppel-Domäne sowohl im Blastoderm (Abb. 32B) als auch im frühen Keimstreifstadium (Abb. 32D), was auf eine Aktivierung von Tc Krüppel durch Tc hunchback schließen lässt. Die Expression von Tc otd ist hingegen in Tc hunchback-rnai-embryonen nicht verändert (Abb. 32A-D; die leicht verbreiterte Domäne in Abb. 32B ist nicht repräsentativ) und dient, wie im Falle der Autoregulation von Tc Krüppel, als interne Kontrolle für die Färbeintensität. Neben Tc Krüppel fehlen in Tc hunchback-rnai-embryonen auch die posterioren Domänen von Tc knirps (nicht gezeigt) und Tc giant (Abb. 32E-H). Da diese beiden Domänen aber auch in Tc Kr jaws -mutanten Keimstreifen nicht aktiviert werden (Abb. 14A-D und 15), handelt es sich hierbei möglicherweise um einen indirekten Effekt durch die fehlende Tc Krüppel- Domäne. Eine vermutlich direkte Rolle von Tc hunchback auf die Expression von Tc giant zeigt sich hingegen in den schwachen ektopischen Tc giant-streifen. Diese kommen in Tc Kr jaws -mutanten Keimstreifen in der Gegenwart von Tc hunchback (Abb. 14D, 16C3 und 35E, I), nicht aber in Tc hunchback-rnai-keimstreifen vor (Abb. 32H). Außerdem ist in Tc hunchback-rnai-keimstreifen auch die späte, anteriore Tc giant-expression im Bereich der vorderen gnathalen Segmente etwas verändert (Abb. 32G, H). 106
125 Abbildung 32: Expression von Tc Krüppel und Tc giant in Wildtypen und in Tc hunchback-rnai-embryonen In situ-ap-färbungen gegen Tc otd und Tc Krüppel (A-D) sowie gegen Tc giant (E-H) in Wildtypen (A, C, E, G) und Tc hunchback-rnai-embryonen (B, D, F, H). In Tc hunchback- RNAi-Embryonen wird Tc Krüppel weder im späten Blastoderm (B) noch in frühen Keimstreifen (D) exprimiert. Die unveränderte Tc otd-färbung dient dabei als interne Kontrolle für eine gleiche Färbungsintensität zwischen den aus einer Färbereaktion stammenden Wildtyp- (A, C) und Tc hunchback-rnai-embryonen (B, D). Die posteriore Tc giant-domäne wird in Tc hunchback-rnai-keimstreifen nicht aktiviert, während die anteriore Tc giant-expression in frühen Stadien nicht verändert ist (E, F). In späteren Stadien taucht in Tc hunchback-rnai-keimstreifen kein ektopischer Tc giant-streifen auf (H). Die späte anteriore Tc giant-expression im Bereich der maxillären Segmentanlagen (Pfeilspitzen in G) ist in Tc hunchback-rnai-keimstreifen auf das Mesoderm beschränkt (Pfeilspitzen in H), während die Tc giant-expression in den Kopflappen nicht verändert ist (G, H). Anterior ist immer oben, in den Blastodermembryonen (A, B) ist ventral links. 107
126 Regulation von Paarregel-und Segmentpolaritätsgenen durch Tc hunchback Der Verlust der Tc Krüppel- sowie der posterioren Tc giant-aktivität stellt ein gemeinsames Merkmal von Tc Kr jaws -mutanten und Tc hunchback-rnai-embryonen dar. Deshalb sollten die Segmentierungsdefekte in Tc hunchback-rnai-embryonen, neben zusätzlichen Aspekten durch die Inaktivierung von Tc hunchback, auch den posterioren Segmentierungsabbruch von Tc Kr jaws -mutanten Keimstreifen (Abb ) beinhalten Expression von Tc eve und Tc runt in frühen Tc hunchback-rnai-embryonen Der Tc hunchback-rnai-phänotyp wurde als eine Deletion der maxillären, labialen und thorakalen Segmente beschrieben (Schröder 2003). Demnach müssten bei einer frühen Deletion dieser Segmente die entsprechenden Paarregelstreifen eve1-3 sowie runt1-3 fehlen oder zumindest missexprimiert sein (siehe Abb. 2). Allerdings zeigt sich in frühen Tc hunchback-rnai-keimstreifen keine Missregulation dieser Paarregelstreifen (Abb. 34C- J). Im Gegensatz zum frühen Keimstreifstadium, kann jedoch während der Segmentierung im Blastodermstadium eine gegenüber dem Wildtyp verringerte Anzahl an Paarregelstreifen beobachtet werden (Abb. 33). Dabei erkennt man in Tc hunchback-rnai- Blastodermembryonen einen unveränderten Abstand zwischen den ersten Paarregelstreifen und der Tc wingless-domäne in der Augenanlage bzw. zwischen dieser Domäne und dem anterioren Pol (Abb. 33C, F-J), wohingegen der Abstand zwischen den jeweils ersten eve- und runt-streifen und der posterioren Invagination verringert ist (Abb. 33). Dies erkennt man auch an der Lage des zweiten runt-streifens, der in Tc hunchback-rnai-blastodermembryonen erst später erscheint und deutlich näher an der posterioren Invagination liegt (Abb. 33H, J). Im Falle von Tc eve tritt in Tc hunchback-rnai-blastodermembryonen hinter dem ersten Streifen eine Expression in der posterioren Invagination auf (Abb. 33E, F), die der Bildung des dritten eve-streifens in Wildtyp-Blastodermembryonen ähnelt (Abb. 33B, C). Unter Berücksichtigung der verzögerten Bildung von runt2 (Abb. 33G-J) sowie des in späteren Stadien korrekten eve-musters (Abb. 34C, D, G, H) erscheint es allerdings auch möglich, dass es sich bei der posterioren Expression von Tc eve in Tc hunchback-rnai-blastodermembryonen um einen etwas verzögert gebildeten zweiten eve-streifen handelt, der, ähnlich wie runt2, bis an die posteriore Invagination rückt (Abb. 33A-F). Bei der Expression von Tc runt in frühen Tc hunchback-rnai-keimrudimenten zeigt sich ein ähnliches Bild wie in späten Blastodermstadien, und es werden die ersten beiden Tc runt- 108
127 Streifen korrekt exprimiert, während der in vergleichbaren Wildtyp-Keimrudimenten bereits gebildete dritte Tc runt-streifen fehlt (Abb. 34A, B). Außerdem nehmen in frühen Keimrudimenten die schon in späten Blastodermstadien leicht geneigten Paarregelstreifen (siehe Abb. 11C, F) in der ventralen Aufsicht eine elliptische Form an (Abb. 34A, B), da sich in diesem Stadium im Zuge der morphogenetischen Umwandlung das Amnion gerade über den Keimstreif zu stülpen beginnt. Mit der Schließung des Serosafensters (siehe Abb. 2B) ist in späteren Stadien die Bildung des Amnions abgeschlossen und die Expression der Paarregelstreifen im Amnion deckt sich in ventraler Aufsicht wieder mit der Expression der jeweiligen Streifen im Keimstreifembryo. Im Laufe des weiteren Wachstums von Tc hunchback-rnai-keimstreifen wird nun auch der dritte Streifen der Paarregelgene Tc eve (Abb. 34D, H) und Tc runt (Abb. 34E, I) gebildet, und das Muster der Paarregelgene ähnelt in diesem Stadium stark dem von vergleichbaren Wildtyp-Keimstreifen (Abb. 34C, F, G, J). Allerdings erreichen die Tc hunchback-rnai- Keimstreifen nicht die Länge von vergleichbaren Wildtyp-Keimstreifen (Abb. 34C-J). Anscheinend weist die Steuerung der Paarregelgene aber ein gewisses Maß an Toleranz gegenüber einer Verkürzung der Keimstreifen auf und so erscheint das Paarregelmuster in Tc hunchback-rnai-keimstreifen lediglich etwas gestaucht (Abb. 34C-J). Möglicherweise tritt die im Tc hunchback-rnai-hintergrund beobachtete Verkürzung der Segmentanlage auch schon im Blastoderm auf und sorgt so für den verringerten Abstand zwischen den Paarregelstreifen und der posterioren Invagination (Abb. 33). Demzufolge könnte es einem Musterbildungsmechanismus im posterioren Blastoderm an Platz fehlen, wodurch es zu der verzögerten Bildung der Paarregelstreifen kommt. Daneben kann aber auch ein direkter Einfluss der anterioren Tc hunchback-domäne auf die Positionierung der distinkten Paarregelstreifen nicht ausgeschlossen werden. Auf alle Fälle scheint die frühe Segmentierung in Tribolium noch ein gewisses Regenerierungspotential zu besitzen, da der Verlust von Paarregelstreifen im Blastodermstadium im Laufe des Keimstreifwachstums ausgeglichen werden kann. 109
128 Abbildung 33: Expression von Tc eve und Tc runt in Wildtyp- und Tc hunchback-rnai- Blastodermembryonen In situ-ap-färbung gegen Tc eve (A-F) und Tc runt (G-J) in Wildtyp- (A-C, I, J) bzw. Tc hunchback-rnai-blastodermembryonen (D-H). Die Embryonen in C-J sind zusätzlich gegen Tc wingless gefärbt (blaue β-gal-färbung). In Wildtyp-Blastodermstadien mittleren Alters sind bereits die ersten beiden eve-streifen erkennbar (A) bevor später im Bereich der posterioren Invagination der dritte eve-streifen ausgebildet wird (B, C). In vergleichbaren Tc hunchback-rnai-blastodermembryonen (Morphologie des posterior pit als Marker) beobachtet man eine Verkürzung der posterior gelegenen Segmentanlagen, wobei zunächst erstmal nur ein eve-streifen auftritt (D). Mit fortschreitender Entwicklung erscheint in den späten Tc hunchback-rnai-blastodermembryonen (E, F) in der posterioren Invagination eine weitere Tc eve-expression (über die Interpretation der eve-streifen wird im Text detaillierter eingegangen). Vom Paarregelgen Tc runt sind die ersten beiden Streifen in mittleren (I), wie auch in späten (J) Wildtyp-Blastodermembryonen gut zu sehen. In den Tc hunchback-rnai- Blastodermembryonen taucht der zweite runt-streifen verspätet auf und befindet sich im Bereich der posterioren Invagination (Pfeilspitze in H), wohingegen der entsprechende Streifen im Wildtyp deutlich vor dieser liegt (J). Im anterioren Bereich von Tc hunchback- RNAi-Blastodermembryonen ist die Position der Tc wg-domäne in der Augenanlage sowie deren Abstand zu den ersten eve- bzw. runt-streifen nicht verändert (C, F, H, J). Anterior ist immer links und ventral ist immer unten. 110
129
130 Abbildung 34: Expression von Tc eve und Tc runt in Wildtyp- und Tc hunchback-rnai- Keimstreifen In situ-ap-färbung gegen Tc eve (C, D, G, H) und Tc runt (A, B, E, F, I, J) in Wildtyp- (B, C, F, G, J) bzw. Tc hunchback-rnai-keimstreifen (A, D, E, H, I). Die Embryonen in A und C-J wurden zusätzlich gegen Tc wingless gefärbt (blaue β-gal-färbung). In frühen Keimrudimenten erscheinen die Streifen von Tc runt aufgrund der morphogenetischen Umbildung im Embryo und im Amnion in der Aufsicht ellipsenförmig (A, B). In diesem Stadium werden in Tc hunchback-rnai-embryonen (A) nur die ersten beiden Tc runt- Streifen gebildet, während vergleichbare Wildtyp-Embryonen (B) schon den dritten Tc runt- Streifen zu exprimieren beginnen. Im weiteren Verlauf zeigen Tc hunchback-rnai- Keimstreifen eine zu den Wildtypen vergleichbare Expression der ersten drei Streifen von Tc eve (C, D, G, H) und Tc runt (E, F, I, J), auch wenn die Tc hunchback-rnai-keimstreifen (D, E, H, I) kürzer als vergleichbare Wildtyp-Keimstreifen (C, F, G, J) ausfallen. Anterior ist immer oben, alle Keimstreifen sind in ventraler Ansicht aufgenommen. Das Tc hunchback- RNAi-Keimrudiment in A wurde, im Gegensatz zu allen anderen Keimstreifen, nicht vom Dotter befreit und flach gepresst. e = eve(n-skipped); r = runt. 112
131
132 Expression von Tc eve, Tc runt und Tc wingless in späten Tc hunchback-rnai- Keimstreifen Bei der Analyse der späten Segmentierung in Tc hunchback-rnai-keimstreifen gilt es zu berücksichtigen, dass es durch die Inaktivierung der anterioren Tc hunchback-domäne zu einem Verlust der Tc Krüppel-Expression kommt (Abb. 32A-D). Das führt wiederum zu einer drastischen anterioren Verschiebung der posterioren Tc hunchback-domäne (Abb. 16), die sich vorübergehend nicht mehr klar von der anterioren Tc hunchback-domäne trennen lässt (Abb. 16C2). Daher muss man in starken Tc hunchback-rnai-keimstreifen von einem, durch die Inaktivierung von Tc Krüppel verursachten, posterioren Segmentierungsabbruch ausgehen (Abb ), in dessen Rahmen die nach anterior verschobene, posteriore Tc hunchback-domäne im Vergleich zu Tc Kr jaws noch zusätzlich mittels RNAi inaktiviert wird. Aus diesem Grund werden bei der Analyse der späten Segmentierung die Tc hunchback-rnai-keimstreifen (aus Gelegen mit starken Phänotypen wie in Abb. 35D, E) auch mit Tc Kr jaws -mutanten Keimstreifen verglichen. In Tc Kr jaws -Keimstreifen sind die ersten fünf Streifen der Paarregelgene Tc eve (Abb. 35A, E, I, M) und Tc runt (Abb. 35D, H, L, P; siehe auch Abb. 17) normal ausgebildet, bevor es zu einem posterioren Abbruch der Segmentierung kommt. Als Folge dessen werden keine weiteren Paarregelstreifen gebildet, und aus den fünf anteriorsten Paarregelstreifen entstehen in weiteren Verlauf 10 Segmente (Abb im Kapitel ). In Tc hunchback-rnai-keimstreifen mittleren Alters macht sich noch immer der Verlust der anterioren Tc hunchback-domäne in der etwas geringeren Länge der Keimstreifen bemerkbar (Abb. 34 und 35A-D), wodurch die eindeutig voneinander unterscheidbaren Streifen 2-4 der Paarregelgene Tc eve (Abb. 35B) und Tc runt (Abb. 35C) etwas kompakter angeordnet sind. Anders als in Tc Kr jaws -Keimstreifen, bleiben in Tc hunchback-rnai-keimstreifen allerdings in etwas späteren Stadien die Streifen eve3, eve4, runt3 und runt4 nicht bestehen (Abb. 35E- L). Stattdessen löst sich die Expression dieser Gene bis auf eine noch weiter andauernde Aktivität in der Wachstumszone auf (Abb. 35J, K, N, O). Auf der Ebene der Segmentpolaritätsgene beobachtet man in Tc hunchback-rnai-keimstreifen mittleren Alters zunächst eine in Relation zu den Kopflappen leichte anteriore Verschiebung der ersten beiden Tc wg-streifen (Abb. 35B, C). In etwas späteren Stadien wird der erste Tc wg-streifen häufig bis auf ein paar kleine Flecken reduziert (Abb. 35F, G), ehe die ersten beiden Tc wg-streifen schließlich an der normalen Position erscheinen (Abb. 35J-P). Posterior davon werden in den stärksten Tc hunchback-rnai-keimstreifen nur noch die wg-streifen 3-5 gebildet (Abb. 35F, 114
133 G, J, K, N, O), an die sich in manchen Keimstreifen noch eine kurze Zone mit unregelmäßiger Tc wg-expression anschließt (Abb. 35N). Verfolgt man in Tc hunchback-rnai-embryonen die molekularen Marker Tc eve, Tc runt und Tc wingless durchgehend während der Segmentierung (Abb ), so erkennt man bei den Paarregelstreifen im Blastoderm sowie dem ersten Tc wg-streifen vorübergehend einige leichte Abweichungen vom Wildtyp, die sich im weiteren Verlauf aber regenerieren. Insgesamt ergeben sich also keine Hinweise auf einen dauerhaft deletierten Bereich, wie man ihn etwa bei Tc giant-rnai-embryonen (Abb ) oder in der Mutante krusty (Abb. 41) findet. Es erscheint vielmehr so, dass die in den stärksten Tc hunchback-rnai-keimstreifen noch verbleibenden fünf Tc wg-streifen die anteriorsten Streifen darstellen, während dahinter keine korrekte Segmentierung mehr stattfindet. Somit ist der posteriore Segmentierungsdefekt in Tc hunchback-rnai gegenüber Tc Kr jaws nochmals deutlich verstärkt, wobei der Unterschied auf der Ebene der Segmentpolaritätsgene (5 statt 10 wg-streifen) deutlicher ausfällt als bei der frühen Expression der Paarregelstreifen. 115
134 Abbildung 35: Expression von Tc eve, Tc runt und Tc en/tc wg in späten Tc Kr jaws - und Tc hunchback-rnai-keimstreifen In situ-doppelfärbung gegen Tc eve (braune AP-Färbung in A, B, E, F, I, J, M, N) bzw. Tc runt (braune AP-Färbung in C, D, G, H, K, L, O, P) und Tc engrailed (blaue β-gal- Färbung in A, E, I) bzw. Tc wingless (blaue β-gal-färbung in B-D, F-H und J-P) in Tc Kr jaws - (A, D, E, H, I, L, M, P) und Tc hunchback-rnai-keimstreifen (B, C, F, G, J, K, N, O). Die Tc Kr jaws -Keimstreifen sind bis auf den in M zusätzlich gegen Tc giant (blaue β-gal-färbung) gefärbt (die Pfeilspitzen in A, E und I zeigen die ektopischen Streifen an). In Tc Kr jaws - Keimstreifen werden die ersten fünf Streifen von Tc eve normal ausgebildet (A, E, I). Die sekundäre Aufspaltung in die segmentalen eve-streifen erfolgt für den zweiten (A) und dritten (E) Streifen ebenfalls normal, während es beim vierten zu Unregelmäßigkeiten kommt und sich der fünfte eve-streifen gar nicht mehr aufspaltet (I). Tc runt bildet in Tc Kr jaws - Keimstreifen ebenfalls fünf Streifen, bevor es zum Segmentierungsabbruch kommt (D, H, L, P, siehe auch Abb. 17). Tc wingless ist in Tc Kr jaws -Keimstreifen bis zum siebten Streifen normal exprimiert, ehe sich ein vorübergehend unregelmäßiges posteriores Streifenmuster zu den Streifen 8-10 organisiert (L, M, P). In Tc hunchback-rnai-keimstreifen sind die ersten vier Streifen der Paarregelgene Tc eve (B) und Tc runt (C) normal ausgebildet, allerdings liegen diese Streifen deutlich näher aneinander als in vergleichbaren Stadien von Tc Kr jaws (A, D). Im weiteren Verlauf kommt es in Tc hunchback-rnai-embryonen zum Verlust des posterioren Streifenmusters bis auf die Expression in der Wachstumszone (F, G, J, K, N, O). Die ersten beiden Tc wg-streifen werden in Tc hunchback-rnai-keimstreifen deutlich näher an den Kopflappen exprimiert (B, C). Danach kommt es häufig zum vorübergehenden Verlust des ersten Tc wg-streifens (F, G), ehe er in späteren Stadien wieder an seiner normale Position auftritt (J, K, N, O). Insgesamt werden in starken Tc hunchback-rnai-embryonen nur noch die fünf anteriorsten Tc wg-streifen gebildet (J, K, N, O). Anterior ist immer oben; e = eve(n-skipped), r = runt. Die eve-färbung in Tc Kr jaws (A, E, I, M) stammt aus der eigenen Diplomarbeit (Cerny 2002). 116
135
136 Die Regulation von Hox-Genen durch Tc hunchback Die in den stärksten Tc hunchback-rnai-keimstreifen noch verbleibenden anteriorsten fünf wg-streifen (Abb. 35J, K, N, O) stimmen mit der Anzahl an Segmenten in den stärksten Kutikulaphänotypen überein, da diese posterior des mandibulären Segments nur noch vier abdominale Segmente zeigen (Abb. 31E). In schwächeren Tc hunchback-rnai-phänotypen zeigen sich hingegen weitere Segmente mit abdominaler Identität (Abb. 31B-D), die vermutlich aufgrund einer abgeschwächten posterioren Deletion gebildet werden. Da die anterioren Segmente in den stärksten Tc hunchback-rnai-phänotypen weitgehend normal gebildet werden (Abb ), lassen die Kutikulaphänotypen eher eine homeotische Transformation der Segmente hinter dem mandibulären Segment in Richtung abdominale Identität vermuten. Aus diesem Grund wurde in Tc hunchback-rnai-embryonen eine in situ- Doppelfärbung gegen Tc Deformed und Tc abdominal-a durchgeführt. Tc Deformed ist im Wildtyp in einem anterioren Streifen in den Anlagen der mandibulären und maxillären Segmente exprimiert (Abb. 36A, C, E siehe auch Abb. 21A, C), während Tc abd-a ab dem posterioren Kompartment des ersten abdominalen Segments exprimiert wird (Abb. 36C, E; Shippy et al und Stuart et al. 1993). Da die abdominalen Segmente erst während des Keimstreifwachstums gebildet werden, taucht die Expression von Tc abd-a im Wildtyp auch erst in Keimstreifstadien mittleren Alters auf (Abb. 36C). In Tc hunchback-rnai-keimstreifen ist die Tc Deformed-Domäne schmaler ausgeprägt und deckt nur die Anlagen des ersten gnathalen Segments ab (Abb. 36B, D, F), was sich im Phänotyp im Verlust der maxillären Identität des zweiten Segments widerspiegelt (Abb. 31). Die Tc abd-a-expression ist in Tc hunchback-rnai-keimstreifen wie erwartet stark nach anterior expandiert und grenzt unmittelbar an den Tc Deformed-Streifen an (Abb. 36B, D, F), wodurch sich in den Kutikulaphänotypen der unmittelbare Übergang vom mandibulären Segment zu Segmenten mit abdominaler Identität erklärt (Abb. 31). Neben der räumlichen Missregulation ergibt sich auch noch eine zeitliche Komponente, da Tc abd-a in Tc hunchback-rnai-embryonen schon zu einem deutlich früheren Zeitpunkt als im Wildtyp, nämlich in späten Blastodermstadien (Daten nicht gezeigt) und in frühen Keimstreifstadien (Abb. 36B, C) exprimiert wird. Dieser Zeitpunkt der anterioren Expansion von Tc abd-a korreliert mit der Expression der anterioren Tc hunchback-domäne in späten Blastoderm- und frühen Keimstreifembryonen und deutet eine möglicherweise unmittelbare Reprimierung von Tc abd-a durch Tc hunchback an. 118
137 Abbildung 36: Expression von Tc Deformed und Tc abdominal-a in Wildtypen und Tc hunchback-rnai-keimstreifen In situ-doppelfärbung gegen Tc Deformed (blaue β-gal-färbung) und Tc abdominal-a (braune AP-Färbung) in Wildtypen (A, C, E) bzw. Tc hunchback-rnai-keimstreifen (B, D, F). Die Expression des abdominalen Hox-Gens Tc abd-a ist in Tc hunchback-rnai- Keimstreifen deutlich nach anterior verschoben und grenzt in allen Stadien unmittelbar an die posteriore Grenze des gnathalen Hox-Gens Tc Dfd an (A-F). Einhergehend mit der anterioren Verschiebung, ist Tc abd-a in Tc hunchback-rnai-embryonen schon im Keimrudiment exprimiert (B), während es im Wildtyp erst später mit der Bildung der abdominalen Segmente aktiviert wird (C). Die gnathale Expression von Tc Dfd bedeckt im Wildtyp die Anlagen für das mandibuläre und das maxilläre Segment (A, C, E). In Tc hunchback-rnai-keimstreifen ist der anteriore Tc Dfd-Streifen schmaler ausgeprägt und nur noch in der mandibulären Segmentanlage exprimiert (B, D, F). Anterior ist immer oben. 119
138 Injektion von Tc hunchback-dsrna in ältere Embryonen Beim Knockdown von Tc hunchback mittels parentaler RNAi ist es nicht klar, inwiefern die Inaktivierung der posterioren Domäne zum Phänotyp beiträgt. Eine Möglichkeit die Funktion der beiden Domänen experimentell zu trennen, besteht in der Injektion von Tc hunchbackdsrna zum Zeitpunkt des späten Blastoderm- / frühen Keimrudimentstadiums (8-9h bei 32 C). Zu diesem Zeitpunkt hat die anteriore Tc hunchback-domäne bereits Tc Krüppel aktiviert, wodurch die posterioren Domänen von Tc giant und Tc hunchback in ihrer Expression nicht mehr verändert werden. Gleichzeitig sollte der Zeitpunkt der Injektion aber auch noch früh genug sein, um die posteriore Tc hunchback-domäne inaktivieren zu können. Die auf diese Art erzeugten Larven weisen noch, mehr oder weniger stark, die anteriore homeotische Transformation auf, zeigen aber keine Reduzierung der Anzahl abdominaler Segmente (Abb. 37A, D). Damit ähneln sie auf dem ersten Blick den durch schwache parentale RNAi erzeugten Phänotypen (Abb. 31A). Bei genauerer Betrachtung beobachtet man jedoch in den spät embryonal injizierten Tc hunchback-rnai-larven posterior des achten abdominalen Segments eine weitere Stigmaöffnung (Abb. 37A, C, D), die mit dem Verlust der terminalen Urogomphi einhergeht (Abb. 37C). Somit ist die Aktivität der posterioren Tc hunchback-domäne offenbar für die Differenzierung des neunten abdominalen Segments notwendig. Daneben tritt in den durch späte embryonale RNAi erzeugten Tc hunchback-phänotypen auch ein Defekt in der Bildung des Enddarms auf (Abb. 37B), der ebenfalls in starken parentalen Tc hunchback-rnai-phänotypen beobachtet werden kann (Abb. 31E). Daher unterscheiden sich die durch späte embryonale Injektion von Tc hunchback-dsrna generierten Larven doch klar von den durch schwache parentale RNAi erzeugten Phänotypen, in denen die terminalen Strukturen ungestört ausgebildet werden (Abb. 31A, B). In keiner dieser spät induzierten RNAi-Larven treten mit der scharfen anterioren Grenze der posterioren Tc hunchback-domäne korrelierende Defekte in den Segmenten A6-A8 auf; womöglich wird aber die spät injizierte dsrna nicht effizient in den zellulären Keimstreifembryo aufgenommen und führt daher nicht zu einer vollständigen Inaktivierung der posterioren Tc hunchback-domäne. 120
139 Abbildung 37: Kutikulaphänotypen aus später embryonaler Tc hunchback-rnai Kutikula-Autofluoreszenzbilder von Larven, die sich aus Eiern entwickelt haben, welche nach 8-9h Entwicklung bei 32 C mit Tc hunchback-dsrna injiziert wurden. In diesen Tc hunchback-rnai-larven erkennt man noch eine mehr (A) oder weniger (D) stark ausgeprägte homeotische Transformation der maxillären, labialen und thorakalen Segmente in Richtung abdominale Identität. Es tritt jedoch keine Deletion abdominaler Segmente auf (A, D), vielmehr lässt sich sogar eine zusätzliche Stigmaöffnung posterior des achten abdominalen Segments erkennen (Pfeil in A, C und D). Da in einer vergrößerten Darstellung einer anderen Larve (C) nur die Pygopodien (A10), nicht aber die Urogomphi (A9; siehe Abb. 1), sichtbar sind, fehlt in diesen Larven offenbar die Differenzierung des neunten abdominalen Segments zu einem terminalen Segment. Außerdem erkennt man in B (optischer Schnitt desselben Kutikulapräparates wie in A) einen, aus starken Tc hunchback-prnai- Phänotypen bekannten, Defekt im Enddarm. Anterior ist immer links; alle Larven sind in lateraler Ansicht abgebildet. Md = Mandibeln, Mx = Maxillen, Lb = Labium, Py = Pygopodien. 121
140 122
141 3.5. Die Phänotypen von allen Kombinationen an doppelten Gap-Gen Inaktivierungen Neben der detaillierten Analyse der einzelnen Gap-Gene wurden durch RNAi in Tc Kr jaws - mutantem Hintergrund (siehe Material und Methoden) bzw. durch Doppel-RNAi noch alle sechs möglichen Kombinationen einer funktionellen Inaktivierung von zwei Gap-Genen untersucht. Das Ziel dieser Experimente bestand darin, evtl. vorhandene Synergien zwischen den Gap-Genen aufdecken zu können. Die Inaktivierung von Tc knirps in Tc Kr jaws -mutantem Hintergrund führt nur zu einer Addition beider Effekte, und diese Larven zeigen den prinzipiellen Phänotyp von Tc Kr jaws (Abb. 13F), wobei ihnen zusätzlich die antennalen und mandibulären Segmente fehlen (Abb. 38A). Eine interessantere Aussage lässt sich aus dem Tc giant-rnai-knockdown in Tc Kr jaws -mutantem Hintergrund ziehen, da in diesen Larven (Abb. 38B) der homeotische Phänotyp von Tc Kr jaws (Abb. 13F) über den von Tc giant-rnai (Abb. 24) epistatisch ist und somit eine Rolle von Tc Krüppel in der Erzeugung der homeotischen Transformationen von Tc giant-rnai-phänotypen andeutet (siehe Kapitel ). Außerdem tritt im Vergleich zu Tc Kr jaws noch eine zusätzliche Deletion des dritten labialen sowie partiell des dritten maxillären Segments auf (Abb. 38B). Da die posteriore Tc giant-domäne in Tc Kr jaws - mutanten Keimstreifen nicht aktiviert wird (Abb. 14A-D), weist dieser zusätzliche Defekt auf eine Segmentierungsfunktion der anterioren Tc giant-domäne hin (siehe Kapitel ). Bei der funktionellen Inaktivierung von Tc hunchback in Tc Kr jaws -mutantem Hintergrund traten in den ersten Gelegen mit starkem Knockdown ausschließlich Tc hunchback-rnai- Phänotypen auf (Daten nicht gezeigt). Dies ist auch nicht weiter verwunderlich, da in Tc hunchback-rnai-keimstreifen die Tc Krüppel-Domäne nicht aktiviert wird (Abb. 32A- D) und die Tc hunchback-rnai-phänotypen somit den Tc Kr jaws -Phänotyp quasi schon als Teilaspekt beinhalten. In den Gelegen mit schwächer werdendem Tc hunchback-rnai- Knockdown (Abb. 31A, B) werden die Tc Kr jaws -mutanten Larven aufgrund des nun stärkeren Segmentierungsdefekts gegenüber dem Wildtyp-Hintergrund erkennbar. Dabei tritt in Tc Kr jaws -mutanten Larven mit schwacher funktioneller Inaktivierung von Tc hunchback auch der homeotische Phänotyp von Tc Kr jaws zutage und wird nur noch partiell von der Transformation in Richtung abdominale Identität überlagert (Abb. 38C). Die Tc hunchback/tc giant-doppel-rnai-larven weisen den homeotischen Phänotyp von Tc hunchback-rnai auf. Außerdem zeigen etliche Tc hunchback/tc giant-doppel-rnai- Kutikulae mit nur noch drei abdominalen Segmenten eine um ein Segment größere Deletion als die stärksten Tc hunchback-rnai-phänotypen (Abb. 31E und 38D). Da in Tc hunchback- 123
142 RNAi-Keimstreifen die posteriore Tc giant-domäne nicht aktiviert wird (Abb. 32E-H), deutet auch diese Verstärkung des Segmentierungsdefekts auf eine Segmentierungsfunktion der anterioren Tc giant-domäne hin. In Tc hunchback/tc knirps- und Tc giant/tc knirps-doppel-rnai-kutikulae (Abb. 38E, F) beobachtet man wiederum eine Addition der beiden Knockdowns, so dass im Wesentlichen die Effekte von Tc hunchback- (Abb. 31) bzw. Tc giant-rnai (Abb. 24) auftreten, wobei jeweils noch zusätzlich die durch Tc knirps-rnai (siehe Abb. 10) verursachte Deletion der antennalen- und mandibulären Segmente erfolgt. Abbildung 38: Phänotypen von Larven mit jeweils zwei inaktivierten Gap-Gen- Orthologen Tc Kr jaws -mutante Larven mit zusätzlichem Knockdown von Tc knirps (A), Tc giant (B) und Tc hunchback (C) sowie Tc hunchback/tc giant- (D), Tc hunchback/tc knirps- (E) und Tc giant/tc knirps-doppel-rnai-larven (F). Tc knirps-rnai in Tc Kr jaws -Hintergrund bewirkt den Phänotyp von Tc Kr jaws plus die durch Tc knirps-rnai verursachte Deletion der antennalen und mandibulären Segmente (A). Tc giant-rnai in Tc Kr jaws -Larven zeigen den homeotischen Phänotyp von Tc Kr jaws (Pfeile zeigen auf maxilläre, Pfeilspitzen auf labiale Extremitäten) sowie eine zusätzliche Deletion von Teilen des dritten maxillären sowie des gesamten dritten labialen Segments (B). Außerdem weist das erste Maxillenpaar in diesen Larven, anders als in Tc Kr jaws allein, keine Mala auf (erstes Pfeilpaar in B). Die schwache funktionelle Inaktivierung von Tc hunchback in Tc Kr jaws führt zu einer partiellen Umwandlung der ektopischen gnathalen Segmente in abdominale Segmente. So folgen auf je zwei maxilläre (kurze Pfeile in C) und labiale (Pfeilspitzen in C) Segmente statt einem drei, an ihren Stigmaöffnungen (lange Pfeile in C) erkennbare, abdominale Segmente. In starken Tc hunchback/tc giant-doppel-rnai-larven (D) treten nur noch drei Segmente mit abdominaler Identität hinter dem mandibulären Segment (Pfeil in D) auf. Die zusätzliche Inaktivierung von Tc knirps in Tc hunchback- (E) und Tc giant-rnai (F) bewirkt gegenüber dem einfachen Knockdown der jeweiligen Gene eine zusätzliche Deletion der antennalen und mandibulären Segmente. Anterior ist immer links, die Larven in C und F sind in ventrolateraler Ansicht dargestellt, alle anderen sind in ventraler Aufsicht abgebildet (A-C: Confocalaufnahmen, D: Epifluoreszenzaufnahme, E, F: Z-Stapel Maximalprojektion). U = Urogomphi, Py = Pygopodien. Die Aufnahme in B ist aus der eigenen Diplomarbeit entnommen (Cerny 2002); die Aufnahmen E und F wurden von N. Gerlitz / D. Hollfelder angefertigt. 124
143
144 3.6. Die funktionelle Analyse von Tc orthodenticle Während in Drosophila das zygotisch exprimierte Dm orthodenticle-gen nur eine Rolle als so genanntes Kopf-Gap-Gen spielt (Cohen und Jürgens 1990), hat sein maternal und zygotisch exprimiertes Ortholog in Tribolium eine sehr viel weit reichendere Funktion und führt in den stärksten Tc orthodenticle-rnai-phänotypen zum Verlust des Kopfes inklusive aller gnathalen Segmente sowie zur Deletion des ersten thorakalen Segments (Schröder 2003). Dieser Phänotyp deutet auf eine wichtige Rolle von Tc orthodenticle (Tc otd) in der anterioren Musterbildung hin, weshalb mit Tc knirps und Tc eve zwei Marker für die frühe Kopfanlage bzw. für die gnathale Segmentierung analysiert wurden. Von der anterioren Tc knirps-domäne in der Kopfanlage (Abbildung 39B) bleibt in Tc otd-rnai-blastodermembryonen nur noch ein schwacher nach anterior verschobener Ring übrig, der keine ausgeprägte dorso-ventrale Asymmetrie aufweist (Abb. 39E, H). Es ist hierbei allerdings nicht klar, welcher Teil der Tc knirps-domäne in Tc otd-rnai-embryonen deletiert wird. Das Paarregelgen Tc eve bildet in Wildtyp-Blastodermembryonen ausgehend von einer breiten Tc eve-domäne zuerst den Tc eve1-streifen, ehe später eve2 in einem anfänglich breiteren und später dann schmaleren Streifen exprimiert wird (Abb. 8B, D, F, H). In Tc otd-rnai- Embryonen ist der eve1-streifen zwar im Vergleich zum Wildtyp drastisch nach anterior verschoben, aber weiterhin als distinkter Streifen ohne expandierte Ränder exprimiert (Abb. 39A, D, G). Etwas später taucht posterior des ersten eve-streifens eine weitere Tc eve- Aktivität auf, die als anterior verschobene, frühe und breite eve2-expression interpretiert werden kann (Abb. 39G, siehe auch Abb. 8F). Somit werden in Tc otd-rnai- Blastodermembryonen die gnathalen Segmente anders als weite Teile der Kopfanlage offenbar primär angelegt, auch wenn es zu einer nach anterior gerichteten Verschiebung oder Streckung des Tc eve-expressionsmusters kommt. In späteren Blastodermstadien mit posteriorer Invagination kam es in den Experimenten zum Verlust des anterioren Gewebes, so dass nur noch der posteriorste Teil der Blastodermembryonen intakt war (Daten nicht gezeigt). Außerdem konnten nur sehr kurze und kopflose Keimrudimente beobachtet werden, die aber ein ganz normales posteriores Segmentierungsmuster zeigen (Daten nicht gezeigt). Der Verlust anterioren Gewebes in späten Tc otd-rnai-blastodermstadien kann auch als Artefakt durch die mechanischen Belastungen bei der Fixierung und der in situ-färbung gedeutet werden, jedoch geben die Höchst Färbungen (Abb. 39C, F, I) einen Hinweis auf eine evtl. reduzierte Stabilität der Tc otd-rnai-blastodermembryonen, da eine deutlich unregelmäßigere Verteilung der Zellkerne mit Bildung einzelner Zellkernklumpen 126
145 (Pfeilspitzen in Abb. 39F, I) beobachtet werden kann. Möglicherweise ist dieser Effekt letztendlich sogar für die Deletion der Segmente G1 bis T1 (mit)verantwortlich und muss getrennt von den früher auftretenden Musterbildungsdefekten in diesem Bereich betrachtet werden. Abbildung 39: Expression von Tc eve und Tc knirps in Wildtyp- und Tc otd-rnai- Blastodermembryonen In situ-ap-färbung gegen Tc eve (A, D, G) und Tc knirps (B, E, H; diese Embryonen sind in C, F, I zusätzlich mit Höchst gefärbt) in Wildtyp- (A-C) und Tc orthodenticle-rnai- Blastodermembryonen (D-I). In den Abbildungen D und G tritt noch eine Hintergrundfärbung aus einer β-gal-färbung gegen Tc wingless auf. Die Position des ersten Tc eve-streifens ist in Tc otd-rnai-embryonen deutlich nach anterior verschoben, ohne dass der Streifen selbst verbreitert erscheint (A, D). In einem etwas späteren Stadium (G) wird auch ein breiter und nach anterior verschobener eve2-streifen aktiv. Die anteriore Domäne von Tc knirps schrumpft in Tc otd-rnai-embryonen auf einen schmalen Streifen nahe dem anterioren Pol zusammen (E, H). Außerdem ist die anteriore Grenze dieser Expression (E, H) nicht wie im Wildtyp (B) deutlich von ventral nach dorsal geneigt. In den Höchst Färbungen erkennt man in den Tc otd-rnai-blastodermembryonen eine unregelmäßigere Verteilung der Zellkerne (C, F, I), wobei lokal begrenzt dichte Ansammlungen von Kernen auftreten (Pfeilspitzen in F, I). Anterior ist immer links und ventral ist immer unten. 127
146 3.7. Die funktionelle Analyse der Mutante krusty Die Analyse der Orthologen von aus Drosophila bekannten Segmentierungsgenen mittels parentaler RNAi birgt die Gefahr, sich ausschließlich auf im Langkeim-Insekt Drosophila relevante Gene zu konzentrieren und möglicherweise nur für Kurzkeim-Insekten spezifische Segmentierungsgene zu übersehen. Deshalb wurde auch die aus einem, im Klingler-Labor durchgeführten, Mutagenesescreen stammende Segmentierungsmutante krusty (Maderspacher et al. 1998) einer funktionellen Untersuchung mit molekularen Markern unterzogen. Zusätzlich sollten mit Hilfe einer Kopplungsanalyse mögliche Kandidatengene bestimmt werden Kutikulaphänotyp der Mutante krusty Die Segmentierungsmutante krusty lässt sich nur schwer einordnen, da sie sowohl Merkmale eines Gap- als auch eines Paarregel-Phänotyps aufweist (Maderspacher et al und Abb. 40). Der Verlust des labialen Segmentes (Abb. 40C), eines bis zweier thorakaler Segmente (Abb. 40A, B), vier (Abb. 40A, B) bis sechs (Maderspacher et al. 1998) abdominaler Segmente sowie der Urogomphi (A9) unter Erhaltung der Pygopodien (A10) (Abb. 40D) könnten auch als ein nicht ganz sauberer Paarregelphänotyp mit Deletionen in dem Segmentraster G3, T2, A1, A3, A5, A7 und A9 interpretiert werden. Das Vorkommen einer Stigmaöffnung im gelegentlich gebildeten zweiten thorakalen Segment (Abb. 40A) steht dem nicht entgegen, da in der Paarregelmutante scratchy die Segmentanlage für T2 auf molekularer Ebene eindeutig fehlt (Abb. 42F), das differenzierte T3 Segment aber letztendlich die Identität von T2 erhält (Maderspacher et al. 1998). Im Gegensatz zur Paarregelmutante scratchy, erscheint in krusty die Segmentierung im thorakalen und anterioren abdominalen Bereich aber deutlich gestörter, und es treten dort zusätzliche leichte Defekte in der Ausbildung des dorsalen Kutikulamusters auf (Daten nicht gezeigt). Eine in situ-färbung gegen das Paarregelgen Tc eve zeigte außerdem im Bereich der thorakalen Segmentanlagen eine weiträumig homogene Tc eve-expression bei einer gleichzeitigen Verbreiterung des Keimstreifs (Maderspacher et al. 1998). Deshalb wurde krusty als Gap-Mutante mit einer Deletion im Bereich von T2/T3 bis etwa A4 eingestuft, die zusätzlich noch weitere Deletionen im labialen Segment und in A9 (Urogomphi) aufweist (Maderspacher et al. 1998). Da Tc eve sowohl in einem frühen Paarregelmuster als auch in 128
147 einem späten segmentalen Muster exprimiert wird (Abb. 2), gestaltet sich die Interpretation der Tc eve-expression in mutantem Hintergrund mitunter schwierig (siehe auch Abb. 28). Deshalb wurde mit Tc runt ein weiteres Paarregelgen in krusty-mutanten Keimstreifen analysiert. Abbildung 40: Der Phänotyp der Mutante krusty Dunkelfeldaufnahmen von zwei verschiedenen Kutikulapräparaten der Mutante krusty in lateraler (A) und ventraler (B) Ansicht. Man erkennt eine Verkürzung des Thorax auf die ersten beiden Segmente (A) bzw. auf das erste thorakale Segment (B) sowie eine Reduktion des Abdomens auf vier Segmente (A, B). Eine vergrößerte Aufnahme der Mundwerkzeuge im Phasenkontrast (C) zeigt weiter eine Deletion des labialen Segments, während die anderen gnathalen Segmente nicht betroffen sind. In einer weiteren Phasenkontrastaufnahme des posterioren Endes sind die Pygopodien (A10) erkennbar, wohingegen die Urogomphi (A9) fehlen (D). In A, B ist anterior links, in C und D oben; Lr = Labrum, Md = Mandibel, Ant = Antenne, Mx = Maxille, Py = Pygopodien. Diese Abbildung ist aus der eigenen Diplomarbeit entnommen (Cerny 2002). 129
148 Expression von Tc runt und Tc wingless in der Mutante krusty In krusty-mutanten Keimstreifen ist die Expression von Tc runt ähnlich wie die von Tc eve verändert (Abb. 41; Maderspacher et al. 1998) und es tritt auch bei den Tc runt-streifen eine weiträumige Derepression auf (Abb. 41A, B). In älteren Embryonen werden allerdings, posterior der morphologisch klar erkennbaren Defektzone, wieder scharf begrenzte Tc runt- Streifen erkennbar (Abb. 41F, H). Diese Streifen scheinen den posterioren Tc runt-streifen 5-8 im Wildtyp zu entsprechen, wobei sich ihre genaue Identität aufgrund der anterior davon gelegenen Defektzone nicht eindeutig bestimmen lässt (Abb. 41E-H). Die ersten beiden Streifen des Segmentpolaritätsgens Tc wingless sind in allen Stadien regulär exprimiert (Abb. 41B, D, F, H, J) und markieren die Anlagen der nicht von der Deletion betroffenen mandibulären und maxillären Segmente (Abb. 40C). Posterior davon entsteht in der Defektzone ein weiterer häufig unregelmäßiger Tc wg-streifen, der letztendlich das erste thorakale Segment spezifiziert (Abb. 41D, F, H, J). Daneben bildet sich in der durch eine Verbreiterung des Keimstreifens gekennzeichneten Defektzone noch eine ungeordnete und zufällig erscheinende Tc wg-expression, aus der sich offenbar in einigen Embryonen ein zweites thorakales Segment organisiert. Im posterioren Abdomen werden hingegen wieder reguläre Tc wg-streifen mit deutlich vergrößertem Abstand ausgebildet (Abb. 41J und 42B- E). Abbildung 41: Expression von Tc runt und Tc wingless in Wildtypen und krustymutanten Keimstreifen In situ-doppelfärbung gegen Tc runt (A-H) bzw. Tc eve (I) (braune AP-Färbungen) und Tc wingless (blaue β-gal-färbung in A-J) in Wildtypen (A, C, E, G, I) und krusty-mutanten Keimstreifen (B, D, F, H, J). In krusty-mutanten Keimstreifen ist die Segmentierung vom labialen Segment bis zu den anterioren abdominalen Segmenten massiv gestört, was mit einer deutlichen Verbreiterung des Keimstreifs in diesem Bereich einhergeht (B, D, F, H, J). In frühen Stadien beobachtet man statt distinkter Streifen (A) eine großflächige Expression des Paaregelgens Tc runt (B), während die wg-streifen 1 und 2 korrekt gebildet werden (A, B). In späteren krusty-mutanten Keimstreifstadien erkennt man zunächst eine irreguläre Tc runt- Expression im Bereich der Wachstumszone (D). In noch späteren Stadien werden posterior des Segmentierungsdefektes wieder intakte Tc runt-streifen erkennbar (F, H). Ihre Identität lässt sich nicht eindeutig bestimmen; der Zeitpunkt ihres Erscheinens korreliert aber mit der Bildung der runt-streifen 5-8 in vergleichbaren Wildtyp-Keimstreifen (E, G). Im Bereich des thorakalen Segmentierungsdefekts tritt eine irreguläre Expression von Tc wg auf (F, H), aus der sich später ein bis zwei thorakale Segmente organisieren (J). Im posterioren Abdomen hinter der Defektzone werden wieder intakte Tc wg-streifen gebildet, deren Abstand zueinander aber erheblich größer als im Wildtyp ist (I, J). Anterior ist immer oben. 130
149
150 Der Vergleich zwischen den auf Höhe des posterioren Endes des Embryos angeordneten Wildtyp- (Abb. 42A) und krusty-keimstreifen (Abb. 42B-E) zeigt in letzteren einen annähernd doppelsegmentalen Abstand der posterioren Tc wg-streifen. In der Paarregelmutante scratchy kommt es auf der Ebene der Segmentpolaritätsgene prinzipiell zum Ausfall der geradzahligen Tc en- und Tc wg-streifen in den Segmenten G1, G3, T2, A1, A3, A5, A7 und A9, wobei aufgrund des nicht kompletten Paarregel-Phänotyps von scratchy jedoch auch Segmente in dem eigentlichen Deletionsraster gebildet werden (Abb. 42F aus Maderspacher et al. 1998). Vergleicht man nun krusty- (Abb. 42B-E) und scratchy-mutante Keimstreifen (Abb. 42F) miteinander, so entsprechen die krusty-mutanten Keimstreifen im Expressionsmuster der posterioren abdominalen Tc wg-streifen (ab A4) ziemlich gut dem (idealisierten) scratchy-paarregelphänotyp, weshalb die letzten vier Segmente in krusty-embryonen wahrscheinlich A4, A6, A8 und A10 entsprechen. Im Gegensatz dazu unterscheiden sich die beiden Mutanten in den anterioren Segmentierungsdefekten deutlich voneinander. Abbildung 42: Expression von Tc wingless in späten Wildtyp-, krusty- und scratchy- Keimstreifen In situ-ap- (A, F) und β-gal-färbung (B-E) gegen Tc wingless in einem Wildtyp-Keimstreif (A), sowie in krusty-mutanten (B-E) und einem scratchy-mutanten Keimstreifen (F). Die drei bis vier posteriorsten Tc wingless-streifen in krusty-keimstreifen weisen einen erhöhten Abstand auf, der etwa dem doppelten Segmentabstand im Wildtyp entspricht (gestrichelte Linien zwischen A und B). In der Paarregelmutante scratchy (F) fehlen prinzipiell die ungeradzahligen Segmente, wobei im posterioren Abdomen auch einzelne ungerade Segmente noch Tc wingless-streifen exprimieren (* in F). Im posterioren Abdomen ergibt sich in der Expression von Tc wg eine Ähnlichkeit zwischen krusty und dem (idealisierten) Paarregelphänotyp von scratchy (gestrichelte Linien zwischen E und F). Anterior ist immer oben. Die Abbildungen in A und F sind aus Maderspacher et al entnommen. 132
151 Expression von Gap-Genen in der Mutante krusty Obwohl im posterioren Abdomen von krusty-mutanten Keimstreifen ein Paarregel-artiger Ausfall von Tc wg-streifen beobachtet werden kann, unterscheidet sich die Defektzone im Bereich der thorakalen und anterioren abdominalen Segmentanlagen doch deutlich von der Paarregel-Mutante scratchy und weist vielmehr die Charakteristik eines Gap-Gen-Defekts auf (Abb. 42). Da in Wildtyp-Keimstreifen in diesem Bereich die Gap-Gene Tc hunchback, Tc giant, Tc Krüppel und Tc knirps lokalisiert sind (Abb. 47), wurden sie im Hinblick auf eine eventuelle Missexpression in krusty-mutanten Keimstreifen untersucht. Die Expression der anterioren Tc hunchback-domäne deckt im Wildtyp die gnathalen Segmentanlagen ab (Abb. 43A), womit sich die posteriore Grenze dieser Domäne schon in der, ab dem labialen Segment beginnenden, Defektzone der Gap-Mutante krusty befindet. Dennoch ist auch in krusty-mutanten Keimstreifen eine scharfe posteriore Grenze der anterioren Tc hunchback-domäne erkennbar (Abb. 43B), obwohl die Paarregelstreifen von Tc eve und Tc runt in diesem Bereich eine deutliche Derepression zeigen (Abb. 41B und Maderspacher et al. 1998). Die posteriore Tc giant-domäne spaltet sich im Wildtyp in zwei Streifen auf, welche in den Anlagen des dritten thorakalen und des zweiten abdominalen Segments lokalisiert sind (Abb. 47; Bucher und Klingler 2004). Obwohl diese Segmentanlagen in der Mutante krusty ebenfalls innerhalb der durch eine Verdickung des Keimstreifens gekennzeichneten Defektzone liegen (Abb. 41), kommt es zu keiner drastischen Veränderung der posterioren Tc giant-domäne (Abb. 43B). Die Lage ihrer klar definierten anterioren und posterioren Grenzen ist vielmehr mit der in Wildtyp-Keimstreifen vergleichbar, allerdings kann keine sekundäre Aufspaltung in zwei Streifen beobachtet werden (Abb. 43A, B). Die Expression der posterioren Tc hunchback-domäne beginnt im Wildtyp mit der Anlage des sechsten abdominalen Segments (Wolff et al. 1995), welche in der Gap-Mutante krusty hinter der Defektzone liegt (Abb. 42B-E). Da sich in krusty, anders als in der Mutante Tc Kr jaws (Abb. 16), keine Beeinflussung der posterioren Tc hunchback- Domäne durch die anterior davon gelegene Defektzone feststellen lässt, ist sie dementsprechend, wie im Wildtyp, hinter der morphologisch gut erkennbaren Defektzone exprimiert (Abb. 43C, D). Neben der posterioren Tc giant-domäne werden auch Tc Krüppel sowie die posteriore Tc knirps-domäne innerhalb der in krusty deletierten Segmentanlagen exprimiert. In krusty-mutanten Keimstreifen kann jedoch keine Veränderung in der Anordnung der Domänen von Tc Krüppel und Tc knirps, sowie in deren Expressionsstärke beobachtet werden (Abb. 43E-H). 133
152 Abbildung 43: Expression der Gap-Gene in Wildtypen und in krusty-mutanten Keimstreifen In situ-doppelfärbungen gegen Tc hunchback (braune AP-Färbung) und Tc giant (blaue β- Gal-Färbung) (A-D) sowie gegen Tc knirps (braune AP-Färbung) und Tc Krüppel (blaue β- Gal-Färbung) (E-H) in Wildtypen (A, C, E, G) und krusty-mutanten Embryonen (B, D, F, H). In krusty-mutanten Keimstreifen ist die Expression der Gap-Gene nicht wesentlich gestört (A- H). Die Expression der anterioren Tc hunchback-domäne ist in krusty-mutanten Keimstreifen nicht verändert (A, B). Auch die posteriore Tc hunchback-domäne erscheint, wie im Wildtyp, mit einer scharf definierten anterioren Grenze während der Bildung der posterioren abdominalen Segmente (C, D). Die posteriore Tc giant-domäne spaltet sich in krustymutanten Keimstreifen nicht wie im Wildtyp in zwei Streifen auf, ihre anteriore und posteriore Grenze ist aber gegenüber Wildtyp-Keimstreifen nicht wesentlich verändert (A, B). Tc Krüppel ist in krusty-keimstreifen ebenfalls wie im Wildtyp in einer breiten Domäne im Bereich der thorakalen Segmente exprimiert (E-H), und auch die Färbung gegen Tc knirps weist keine Unterschiede zum Wildtyp auf (E, F). In allen Abbildungen ist anterior oben. 134
153 Kartierung der Mutante krusty In der Mutante krusty ist die Expression der primären Paarregelgene Tc eve (Maderspacher et al. 1998) und Tc runt (Abb. 41) massiv gestört, während auf der Ebene der Gap-Gene nur minimale Veränderungen auftreten (Abb. 43). Deshalb könnte die Identifizierung des in krusty mutierten Gens einen Schlüssel für das Verständnis der Segmentierung in Kurzkeim- Insekten liefern. In einer von Gregor Bucher durchgeführten Segregationsanalyse wurde untersucht, ob der krusty-locus mit einem der damals bekannten Gap- und Paarregelgene gekoppelt ist (siehe Material und Methoden sowie Bucher 2002). Anders als im Fall von jaws und Tc Krüppel (Bucher 2002 und Cerny et al. 2005), konnte dabei aber keine direkte Kopplung zwischen krusty und einem dieser Kandidatengene gefunden werden (Bucher 2002). Es zeigte sich jedoch mit einer Rekombinationsfrequenz von 16cM eine signifikante Kopplung an einen Polymorphismus im zweiten Intron des Tc runt-gens auf dem achten Chromosom (LG 8; Position 34,8). Da im Nachhinein nicht mehr bestimmt werden konnte, welche krusty/+ F2- Käfer (Kreuzung siehe Material und Methoden) ein Rekombinationsereignis aufweisen, wurde als erstes die Analyse des Tc runt-polymorphismus in allen F2-Individuen wiederholt. Dabei zeigte sich auch mit 13,2cM (7/53) eine geringfügig abweichende Rekombinationsfrequenz. Im weiteren Verlauf sollten bei einem Aufenthalt im Labor von Sue Brown (Kansas State University) die im Labor von Dick Beeman erstellte, genetische Karte des Tribolium-Genoms (Lorenzen et al. 2005) für eine genauere Lokalisierung der krusty- Mutation verwendet werden. Diese Karte beruht auf SSCP- (Single Strand Conformation Polymorphism) Markern, wobei einzelne Basenaustausche in etwa 200bp langen PCR- Produkten allelspezifische Sekundärstrukturen der denaturierten Einzelstränge bewirken können, die als verschiedene Banden in Polyacrylamid-Gradientgelen sichtbar gemacht werden. Leider stellten sich diese zwischen den GA-2- und ab-stämmen erstellten Marker bis auf einen (50.L10t/50.4F05t auf LG 8, Position 52,6) bereits in Vorversuchen als ungeeignet heraus, da sie entweder zwischen den in unseren Kreuzungen verwendeten SB- und Tiw1- Stämmen nicht unterscheidbar waren, oder schon innerhalb des SB-Stamms verschiedene Allele aufwiesen. Der einzige funktionierende und 17,8cM von Tc runt entfernt liegende SSCP-Marker 50.L10t/50.4F05t ergab gegenüber krusty mit 28,3cM (13/46) eine deutlich höhere Rekombinationsfrequenz (Daten nicht gezeigt), die in etwa der Summe der Rekombinationsfrequenzen zwischen krusty und Tc runt (13,2cM) sowie zwischen Tc runt und dem SSCP-Marker (17,8cM) entspricht. Daraufhin wurden auf der anderen Seite von 135
154 Tc runt neue Marker erstellt, indem in der mittlerweile verfügbaren Genomsequenz in der Umgebung der, leider ungeeigneten, SSCP-Marker 16G01s (Position 19,2), 19.E03t (Position 20,6) und 36.B02t (Position 22,4) in nicht-codierenden und nicht-repetitiven Sequenzen auf gut Glück neue Primerpaare für potentielle SSCP-Marker erstellt wurden. Schließlich zeigte das nahe dem Marker 16.G01s gelegene Primerpaar ctg 1606f2/r2 (siehe Material und Methoden) sogar einen deutlich leichter zu identifizierenden PCR-Längenpolymorphismus, der in allen der jeweils 17 getesteten Individuen das SB- bzw. Tiw1-spezifische Allel zeigt (Abb. 44A). In den krusty/+ F2-Individuen weisen 52 von 54 Tieren noch das ursprüngliche SB-Allel auf (Abb. 44B). Die beiden bezüglich krusty und 16.G01s rekombinanten Individuen gehören dabei der Gruppe von sieben, zwischen krusty und Tc runt rekombinanten, Individuen an, so dass diese drei Loci auf dem Chromosom in der Reihenfolge krusty-16.g01s Tc runt angeordnet sind. Die höhere Rekombinationsfrequenz zwischen 16.G01s und Tc runt (15,6cM) gegenüber der von krusty und Tc runt (13,2cM) ist dabei kein Widerspruch, da die Rekombinationsfrequenzen in verschiedenen Hintergründen gemessen wurden und auch die Zahl der Rekombinationsereignisse zu gering für eine exakte Bestimmung dieser Werte ist. Für eine genauere Berechnung des auf knapp 4cM bestimmten Abstands zwischen krusty und 16.G01s war die Erstellung einer größeren Anzahl an krusty/+ F-2 Individuen geplant. In der Zwischenzeit konnte jedoch das Paarregelgen Tc odd-skipped aufgrund seiner RNAi- Phänotypen, sowie seiner genomischen Lokalisation nahe dem Marker 16.G01s als Kandidatengen identifiziert werden (S. Brown, pers. Mitteilung), wobei der endgültige Beweis durch die Bestimmung der odd-skipped-sequenz in krusty noch aussteht. 136
155 Abbildung 44: Rekombination von krusty bezüglich des Markers 16.G01s PCR-Längenpolymorphismus mit dem Primerpaar ctg 1606 f2/r2 (siehe Material und Methoden) nahe dem genetischen Marker 16.G01s in einem 2,5% Agarose-Gel (A, B). 8 von 8 (insgesamt 17 von 17) SB-Käfer sowie 9 von 9 (insgesamt 17 von 17) Tiw1-Käfer zeigen eine für den jeweiligen Stamm spezifische PCR-Bande, während Tiw1/SB heterozygote F1- Käfer beide Allele aufweisen (A). Nachdem die im SB-Hintergrund induzierte Mutation krusty zweimal mit Tiw1 ausgekreuzt wurde, weisen von den in diesem Gel gezeigten krusty/+ F2-Individuen 13 von 15 (insgesamt 52 von 54) Proben neben dem Tiw1-Allel noch das ursprüngliche SB-Allel auf (B). Die PCR für Spur 12 funktionierte in dieser Reaktion nicht; in einer erneuten Reaktion waren beide Allele erkennbar. 137
156 3.8. Die funktionelle Analyse weiterer potentieller Segmentierungsgene Die Kurzkeim-Segmentierung in einem zellulär wachsenden Keimstreif benötigt womöglich die Aktivität von Genen, deren Funktion im Segmentierungsprozess auf der evolutionären Linie zur Langkeim-Entwicklung in den höheren Dipteren verloren gegangen oder reduziert wurde. Deshalb erschien es in Tribolium aussichtsreich, auch Entwicklungsgene, die in Drosophila keine zentrale Rolle in der Segmentierung innehaben, auf ihre potentielle Segmentierungsfunktion zu untersuchen Kutikulaphänotypen von Tc Retinsäurerezeptor-RNAi Retinsäure spielt bei Vertebraten in allen drei Keimblättern eine wichtige Rolle in der Vermittlung der anterior-posterioren Identität (Yelon und Stainier 2002). Im Rahmen eines Laborpraktikums von Jana Burda wurde deshalb das von Olaf Voolstra (Freiburg) klonierte Tribolium-Ortholog des Retinsäurerezeptors (engl.: Retinoic Acid Receptor, kurz RAR) funktionell untersucht. In Tribolium ist das RAR-Gen in frühen Keimstreifen im posterioren Bereich einschließlich der Wachstumszone exprimiert, ohne dass eine scharfe anteriore Grenze auftritt (Abb. 45A, B). Diese wenig spezifische Expression ist für einen Rezeptor nicht überraschend, zumal in Vertebraten die lokale Erzeugung von Retinsäure durch die Retinaldehyd-Dehydrogenase oder der lokal verstärkte Abbau von Retinsäure durch ein Cytochrom-ähnliches Enzym als wahrscheinlicher Mechanismus zur Erzeugung eines Signalgradienten vermutet wird (Yelon and Stainier 2002). Die funktionelle Inaktivierung von Tc RAR mittels parentaler RNAi führt mit hoher Penetranz zu Defekten im dorsalen Rückenschluss (Abb. 45C) und zu fehlenden dorsalen Kutikulastrukturen (Abb. 45D, E). Da aber die Segmentierung und die homeotische Spezifizierung entlang der anterior-posterioren Achse nicht betroffen sind, wurde dieses Gen nicht mehr weiter untersucht. 138
157 Abbildung 45: Expression und Funktion des Retinsäurerezeptors in Tribolium In situ-ap-färbung gegen den Retinsäurerezeptor (Tc RAR) in jungen Keimstreifen (A, B) sowie Confocalaufnahmen von Tc RAR-RNAi-Kutikulae (C-E). In jungen Keimstreifen ist Tc RAR im posterioren Bereich des Keimstreifens exprimiert (A, B). In schwächeren Tc RAR- RNAi-Phänotypen fehlt der dorsale Rückenschluss (C), während in stärkeren Phänotypen (D, E) dorsales und laterales Gewebe im Bereich des Thorax und des Abdomens verloren geht. Die Morphologie des Kopfes sowie die Segmentierung entlang der anterior-posterioren Achse sind in Tc RAR-RNAi-Larven nicht gestört. Bei den in situ-färbungen (A, B) ist anterior oben, die Kutikulae (C-E) sind mit anterior nach links und ventral nach unten angeordnet. 139
158 Funktionelle Analyse des JAK/STAT-Signalwegs Der JAK/STAT-Signalweg ist in Drosophila an zahlreichen Entwicklungsvorgängen beteiligt, wie z.b. der Segmentierung, der Geschlechtsbestimmung, der Differenzierung im Follikelepithel, der Augenentwicklung sowie der Bildung von Blutzellen (Castelli-Gair Hombria und Brown 2002; Zeidler et al. 2000). Er besteht im Wesentlichen aus einer Familie von Liganden (Dm unpaired-1 bis 3), einem Transmembranrezeptor (Dm domeless), einer zur JAK-Kinase homologen Tyrosinkinase (Dm hopscotch) und schließlich dem Transkriptionsfaktor STAT (Dm STAT 92E) (Castelli-Gair Hombria und Brown 2002; Zeidler et al. 2000). In Drosophila wurde ein Segmentierungsphänotyp in den F1-Nachkommen von Müttern mit Dm hopscotchoder Dm STAT 92E-mutanter Keimbahn beobachtet, der durch leichte Segmentierungsdefekte im thorakalen Bereich sowie dem Verlust der Segmente A4/A5 gekennzeichnet ist (Hou et al. 1996; Yan et al. 1996). Durch in situ-färbungen von mutanten Embryonen konnte gezeigt werden, dass eine deutlich schwächere Expression der dritten und vor allem der fünften Dm runt- und Dm eve-streifen für diese Defekte verantwortlich ist (Hou et al. 1996; Yan et al. 1996). Der Ligand Dm unpaired-1 weist während der Segmentierung eine dynamische Expression auf, die von einer alle Segmentanlagen umfassenden Domäne über ein unsauberes Paarregelmuster schließlich in schmale, segmentale Streifen übergeht. Im Genom von Tribolium castaneum konnten (wie auch im Genom von Anopheles gambia) aufgrund des Fehlens konservierter Domänen keine Orthologe der unpaired-familie gefunden werden. In Vertebraten wiederum sind zu viele verschiedenartige Liganden des JAK/STAT-Signalwegs bekannt (Leaman et al. 1996), um für die Identifizierung potentieller Orthologe in Tribolium von Nutzen zu sein. In Drosophila wird die Aktivität von Dm unpaired-1 auch von der Expression des, im JAK/STAT-Signalweg als Negativregulator wirkenden, Dm SOCS 36E-Gens widergespiegelt (Karsten et al. 2002). Deshalb wurde das Tribolium-Ortholog von Dm SOCS 36E kloniert, welches aber lediglich eine ubiquitäre Expression ergab (Daten nicht gezeigt). Um das Aktivitätsmuster des JAK/STAT-Signalwegs in Tribolium sichtbar zu machen, ist für die Zukunft die Suche nach einem geeigneten Antikörper gegen die durch Phosphorylierung aktivierte Form des Tc STAT 92E-Proteins geplant. Zur Untersuchung der Funktion des JAK/STAT-Signalwegs in der Segmentierung wurden der Rezeptor Tc domeless sowie der Transkriptionsfaktor Tc STAT 92E kloniert (siehe Material und Methoden) und von diesen Sequenzen synthetisierte dsrna in weibliche Puppen 140
159 injiziert. Daraufhin trat unter fast allen geschlüpften weiblichen Käfern Sterilität auf und in einer, von Jochen Trauner durchgeführten, Untersuchung der Ovare dieser sterilen Käfer zeigte sich ein Defekt in der Entwicklung der Follikel. Um dennoch die Funktion von Tc domeless und Tc STAT 92E in der Segmentierung untersuchen zu können, wurden adulte weibliche Käfer injiziert. Diese legten dann ab dem zweiten Tag nach der Injektion, noch ehe sich der Ovarphänotyp aufbauen kann, für einige Tage Nachkommen mit einem embryonal wirksamen Knockdown der jeweiligen Gene. Da sich jedoch mit der Injektion von dsrna in adulte Käfer im Falle der Gap-Gen-Orthologen aus unbekannten Gründen nicht die stärksten Phänotypen erzielen ließen, wurde zusätzlich mit beiden Genen auch noch eine embryonale RNAi durchgeführt. Bei der Inaktivierung von Tc domeless konnte mit embryonaler und adulter RNAi ein vergleichbares Spektrum von Phänotypen erzeugt werden. Generell kommt es in Tc domeless- RNAi-Phänotypen zu einem dramatischen Zusammenschrumpfen der Larven (Abb. 46C), das sich bei einem schwächeren Knockdown von Tc domeless auf die gnathalen und thorakalen Segmente beschränkt (Abb. 46A, B). Eine erste in situ-färbung in Tc domeless-rnai- Embryonen zeigt keine Veränderung im Expressionsmuster des Paarregelgens Tc runt, und auch die frühe Expression des Segmentpolaritätsgens Tc wingless ist zunächst nicht gestört (Daten nicht gezeigt). Erst in späteren Stadien erfolgt eine mit dem Verlust der segmentalen Ordnung von Tc wg einhergehende Schrumpfung der Keimstreifen (Abb. 46D, E). Die funktionelle Inaktivierung von Tc STAT 92E wurde von N.Gerlitz und D. Hollfelder im Rahmen eines Laborpraktikums durchgeführt. Auch hier führten embryonale und adulte RNAi zu ähnlichen Phänotypen. Diese unterscheiden sich jedoch deutlich von den Tc domeless-rnai-phänotypen (Abb. 46A-C), da im anterioren Bereich der Larven bis einschließlich T2 keine Veränderungen auftreten und es erst ab T3 zu einer Schrumpfung der Larven kommt (Abb. 46F, G). Diese erreicht jedoch auch in den stärksten Phänotypen nicht das Ausmaß von starken Tc domeless-rnai-phänotypen (Abb. 46C, G). 141
160 Abbildung 46: Die Phänotypen von Tc domeless- und Tc STAT 92E-RNAi Dunkelfeld- (A, B, G) und Autofluoreszenz-Aufnahmen (Maximum-Projektion eines Z- Stapels in C, F) von Tc domeless- (A-C) und Tc STAT 92E-RNAi-Phänotypen (F, G) sowie eine in situ-färbung gegen Tc wg in Tc domeless-rnai-keimstreifen (D, E). In schwachen Tc domeless-rnai-phänotypen kommt es zu einer Deletion des Kopfes, der gnathalen und partiell auch der thorakalen Segmente (A). Mit zunehmender phänotypischer Stärke schrumpft auch das Abdomen zusammen (B, C), wobei zahlreiche abdominale Borsten als segmentale Marker sichtbar bleiben (C). Eine in situ-färbung gegen Tc wg zeigt einen sekundären Verlust der segmentalen Organisation (D, E). Die Tc STAT 92E-RNAi- Phänotypen weisen ab T3 eine, mit ansteigender phänotypischer Stärke zunehmende, Schrumpfung und Fusion der Segmente auf, während die anterioren Segmente bis einschließlich T2 keine Veränderungen zeigen. Anterior ist immer links. Die Tc domeless- RNAi-Kutikulae wurden in ventrolateraler Ansicht aufgenommen, wobei die stärkste Tc domeless-rnai-larve in C gegenüber A und B vergrößert dargestellt ist. Die Tc STAT 92E -RNAi-Kutikulae sind in lateraler Ansicht abgebildet. Die Abbildungen E und F wurden von N.Gerlitz / D. Hollfelder aufgenommen. 142
161
Teil I (Fischbach): Drosophila als Modellsystem der Entwicklungsgenetik
 Teil I (Fischbach): Drosophila als Modellsystem der Entwicklungsgenetik Termine 20.10. 2010 Reichweite der Entwicklungsgenetik 27.10. 2010 Die Festlegung der Körperachsen 03.11. 2010 Neurogenese 10.11.
Teil I (Fischbach): Drosophila als Modellsystem der Entwicklungsgenetik Termine 20.10. 2010 Reichweite der Entwicklungsgenetik 27.10. 2010 Die Festlegung der Körperachsen 03.11. 2010 Neurogenese 10.11.
Entwicklungsbiologie Master-Module. (bzw. 9 + 3)
 Entwicklungsbiologie Master-Module (bzw. 9 + 3) Entwicklungsbiologie Master-Module Block 3 Ebio I Evolution von Musterbildung und Gametogenese Mikroinjektion von Tribolium-Embryonen zur Proteinlokalisierung
Entwicklungsbiologie Master-Module (bzw. 9 + 3) Entwicklungsbiologie Master-Module Block 3 Ebio I Evolution von Musterbildung und Gametogenese Mikroinjektion von Tribolium-Embryonen zur Proteinlokalisierung
Übung 11 Genregulation bei Prokaryoten
 Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Expressionskontrolle in Eukaryonten
 Expressionskontrolle in Eukaryonten Warum muss Genexpression kontrolliert werden? 1. Gewebsspezifische Kontrolle - nicht jedes Genprodukt ist in allen Zellen erforderlich - manche Genprodukte werden ausschliesslich
Expressionskontrolle in Eukaryonten Warum muss Genexpression kontrolliert werden? 1. Gewebsspezifische Kontrolle - nicht jedes Genprodukt ist in allen Zellen erforderlich - manche Genprodukte werden ausschliesslich
Regulation der Genexpression: regulierbare Promotoren, Proteine und sirna
 Regulation der Genexpression: regulierbare Promotoren, Proteine und sirna Biochemie Praktikum Christian Brendel, AG Grez Ebenen der Genregulation in Eukaryoten Cytoplasma DNA Zellkern Introns Exons Chromatin
Regulation der Genexpression: regulierbare Promotoren, Proteine und sirna Biochemie Praktikum Christian Brendel, AG Grez Ebenen der Genregulation in Eukaryoten Cytoplasma DNA Zellkern Introns Exons Chromatin
Role of CXCR4 and Gab1 in the development of muscle precursor cells
 Role of CXCR4 and Gab1 in the development of muscle precursor cells Dissertation zur Erlangung des akademischen Grades des Doktors der Naturwissenschaften (Dr. rer. nat.) eingereicht im Fachbereich Biologie,
Role of CXCR4 and Gab1 in the development of muscle precursor cells Dissertation zur Erlangung des akademischen Grades des Doktors der Naturwissenschaften (Dr. rer. nat.) eingereicht im Fachbereich Biologie,
RNA-Regulationsmechanismen: RNA Interferenz
 RNA-Regulationsmechanismen: RNA Interferenz Vorlesung System-Biophysik 19. Dez. 2008 Literatur Martens: BIOspektrum 4/02 8. Jahrgang M. Kuhlmann: Biol. Unserer Zeit Nr.3 (2004), S. 142. Genregulation durch
RNA-Regulationsmechanismen: RNA Interferenz Vorlesung System-Biophysik 19. Dez. 2008 Literatur Martens: BIOspektrum 4/02 8. Jahrgang M. Kuhlmann: Biol. Unserer Zeit Nr.3 (2004), S. 142. Genregulation durch
Stammzellenmanipulation. Stammzellen können in Zellkultur manipuliert werden
 Stammzellenmanipulation Hämatopoietische Stammzellen können gebraucht werden um kranke Zellen mit gesunden zu ersetzen (siehe experiment bestrahlte Maus) Epidermale Stammzellpopulationen können in Kultur
Stammzellenmanipulation Hämatopoietische Stammzellen können gebraucht werden um kranke Zellen mit gesunden zu ersetzen (siehe experiment bestrahlte Maus) Epidermale Stammzellpopulationen können in Kultur
4. ERGEBNISSE. 4.2. Untersuchung des umgelagerten IgH Gens in Hodgkinzelllinien
 36 4. ERGEBNISSE 4.1. Immunglobulin Gentranskripte in Hodgkinzelllinien Mit Hilfe der RT PCR untersuchten wir die Expression umgelagerter Ig Gene in den Hodgkinzelllinien L1236, L428, L591 und KM-H2 sowie
36 4. ERGEBNISSE 4.1. Immunglobulin Gentranskripte in Hodgkinzelllinien Mit Hilfe der RT PCR untersuchten wir die Expression umgelagerter Ig Gene in den Hodgkinzelllinien L1236, L428, L591 und KM-H2 sowie
Das Paper von heute. Samuel Grimm & Jan Kemna
 Das Paper von heute Samuel Grimm & Jan Kemna Bisheriges Modell Was bereits bekannt war - TIR1 ist an Auxinantwort (Zellteilung, Elongation, Differenzierung) beteiligt, im selben Signalweg wie AXR1 - TIR1
Das Paper von heute Samuel Grimm & Jan Kemna Bisheriges Modell Was bereits bekannt war - TIR1 ist an Auxinantwort (Zellteilung, Elongation, Differenzierung) beteiligt, im selben Signalweg wie AXR1 - TIR1
THE CAENORHABDITIS ELEGANS MODEL
 THE CAENORHABDITIS ELEGANS MODEL C. elegans Der Name Caenorhabditis elegans stammt aus einer Mischung von griechischen und lateinischen Wörtern. Caeno (griechisch kaeno): neu, ungewöhnlich Rhabdos (griechisch):
THE CAENORHABDITIS ELEGANS MODEL C. elegans Der Name Caenorhabditis elegans stammt aus einer Mischung von griechischen und lateinischen Wörtern. Caeno (griechisch kaeno): neu, ungewöhnlich Rhabdos (griechisch):
Immunregulatorische Funktionen von T-Lymphozyten
 Immunregulatorische Funktionen von T-Lymphozyten DISSERTATION zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Christian-Albrechts-Universität zu Kiel vorgelegt von
Immunregulatorische Funktionen von T-Lymphozyten DISSERTATION zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Christian-Albrechts-Universität zu Kiel vorgelegt von
5.7 Blütenentwicklung als Beispiel für kombinatorische Genfunktion
 160 Entwicklung Infloreszenzmeristem: Sprossmeristem nach Blühinduktion. Bildet an den Flanken statt Blatt-Blütenprimordien. TERMINAL FLOWER (TFL): Gen, welches nötig ist für die Identität des Infloreszenzmeristems,
160 Entwicklung Infloreszenzmeristem: Sprossmeristem nach Blühinduktion. Bildet an den Flanken statt Blatt-Blütenprimordien. TERMINAL FLOWER (TFL): Gen, welches nötig ist für die Identität des Infloreszenzmeristems,
Evolution & Genetik (Beispiel Hämoglobin) Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de
 Evolution & Genetik (Beispiel Hämoglobin) Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de DNA (Desoxyribonukleinsäure) 5 3 CGATGTACATCG GCTACATGTAGC 3 5 Doppelhelix Basen: Adenin,
Evolution & Genetik (Beispiel Hämoglobin) Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de DNA (Desoxyribonukleinsäure) 5 3 CGATGTACATCG GCTACATGTAGC 3 5 Doppelhelix Basen: Adenin,
1. Definition und Mechanismen
 Zusammenfassung 1. Definition und Mechanismen Epigenetik (von griechisch epi- über ) bezeichnet erbliche Veränderungen in der Genexpression, die nicht von Veränderungen in der DNA Sequenz (Mutationen)
Zusammenfassung 1. Definition und Mechanismen Epigenetik (von griechisch epi- über ) bezeichnet erbliche Veränderungen in der Genexpression, die nicht von Veränderungen in der DNA Sequenz (Mutationen)
05_10_Genes_info.jpg
 Übertragung der Information von DNA auf RNA - Transkription von RNA auf Protein - Translation Übertragung der Information vom Gen auf Protein 05_10_Genes_info.jpg 1 Figure 6-2 Molecular Biology of the
Übertragung der Information von DNA auf RNA - Transkription von RNA auf Protein - Translation Übertragung der Information vom Gen auf Protein 05_10_Genes_info.jpg 1 Figure 6-2 Molecular Biology of the
Vorhersage von mirna targets in Säugetieren mirna target prediction am Beispiel von TargetScan
 VorhersagevonmiRNAtargetsinSäugetieren mirnatargetprediction ambeispielvontargetscan Christoph Theunert 8. Fachsemester Bioinformatik christoph.theunert@web.de Gliederung I mirna Eigenschaften II mirna
VorhersagevonmiRNAtargetsinSäugetieren mirnatargetprediction ambeispielvontargetscan Christoph Theunert 8. Fachsemester Bioinformatik christoph.theunert@web.de Gliederung I mirna Eigenschaften II mirna
The atypical kinase hrio1 is a trans- acting factor in cytoplasmic maturation of the 40S ribosomal subunit
 DISS. ETH NO. 19168 The atypical kinase hrio1 is a trans- acting factor in cytoplasmic maturation of the 40S ribosomal subunit A dissertation submitted to ETH Zurich for the degree of Doctor of Sciences
DISS. ETH NO. 19168 The atypical kinase hrio1 is a trans- acting factor in cytoplasmic maturation of the 40S ribosomal subunit A dissertation submitted to ETH Zurich for the degree of Doctor of Sciences
Zelldifferenzierung und Morphogenese
 Zelldifferenzierung und Morphogenese Fragestellung: Wie ist es möglich, daß sich aus einer einzigen Zelle ein hochkomplexes Lebewesen mit hohem Differenzierungsgrad entwickeln kann? weniger gut verstanden
Zelldifferenzierung und Morphogenese Fragestellung: Wie ist es möglich, daß sich aus einer einzigen Zelle ein hochkomplexes Lebewesen mit hohem Differenzierungsgrad entwickeln kann? weniger gut verstanden
carbohydrate binding site on laminin responsible for the L2/HNK-1 carbohydrate mediated neural cell adhesion
 Diss. ETH Nr. 10368 Identification and characterization of the L2/HNK-1 carbohydrate binding site on laminin responsible for the L2/HNK-1 carbohydrate mediated neural cell adhesion ABHANDLUNG Zur Erlangung
Diss. ETH Nr. 10368 Identification and characterization of the L2/HNK-1 carbohydrate binding site on laminin responsible for the L2/HNK-1 carbohydrate mediated neural cell adhesion ABHANDLUNG Zur Erlangung
β2-microglobulin deficient mice lack CD4-8+cytolytic T cells
 β2-microglobulin deficient mice lack CD4-8+cytolytic T cells Mäuse mit einem Knock-out bezüglich ß-Microglobulin sind nicht in der Lage CD4-8+ cytotoxische T-Zellen zu bilden Nature,Vol 344, 19. April
β2-microglobulin deficient mice lack CD4-8+cytolytic T cells Mäuse mit einem Knock-out bezüglich ß-Microglobulin sind nicht in der Lage CD4-8+ cytotoxische T-Zellen zu bilden Nature,Vol 344, 19. April
VORANGEGANGENE MODELLE
 VORANGEGANGENE MODELLE UNSER THEMA HEUTE Ziel dieses Papers: - Nähere Charakterisierung der AuxREs - Analyse eines zu den AuxREs gehörenden Transkriptionsfaktors WAS BEREITS BEKANNT WAR: - Auxin moduliert
VORANGEGANGENE MODELLE UNSER THEMA HEUTE Ziel dieses Papers: - Nähere Charakterisierung der AuxREs - Analyse eines zu den AuxREs gehörenden Transkriptionsfaktors WAS BEREITS BEKANNT WAR: - Auxin moduliert
DNA Replikation ist semikonservativ. Abb. aus Stryer (5th Ed.)
 DNA Replikation ist semikonservativ Entwindung der DNA-Doppelhelix durch eine Helikase Replikationsgabel Eltern-DNA Beide DNA-Stränge werden in 5 3 Richtung synthetisiert DNA-Polymerasen katalysieren die
DNA Replikation ist semikonservativ Entwindung der DNA-Doppelhelix durch eine Helikase Replikationsgabel Eltern-DNA Beide DNA-Stränge werden in 5 3 Richtung synthetisiert DNA-Polymerasen katalysieren die
Novel functions of the Nrf3 transcription factor in keratinocytes
 Diss. ETH No. 20506 Novel functions of the Nrf3 transcription factor in keratinocytes A dissertation submitted to the ETH ZURICH for the degree of Doctor of Sciences Presented by CLAUDIA DEFILA FLÜKIGER
Diss. ETH No. 20506 Novel functions of the Nrf3 transcription factor in keratinocytes A dissertation submitted to the ETH ZURICH for the degree of Doctor of Sciences Presented by CLAUDIA DEFILA FLÜKIGER
Expression der genetischen Information Skript: Kapitel 5
 Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Molekulare Mechanismen der Signaltransduktion
 Molekulare Mechanismen der Signaltransduktion 07 - Identifizierung von ARF1 + Hinweise für Vorträge Folien: http://tinyurl.com/modul-mms http://www.pnas.org/content/111/14/5427/f1.large.jpg neues Modell
Molekulare Mechanismen der Signaltransduktion 07 - Identifizierung von ARF1 + Hinweise für Vorträge Folien: http://tinyurl.com/modul-mms http://www.pnas.org/content/111/14/5427/f1.large.jpg neues Modell
Extended Ordered Paired Comparison Models An Application to the Data from Bundesliga Season 2013/14
 Etended Ordered Paired Comparison Models An Application to the Data from Bundesliga Season 2013/14 Gerhard Tutz & Gunther Schauberger Ludwig-Maimilians-Universität München Akademiestraße 1, 80799 München
Etended Ordered Paired Comparison Models An Application to the Data from Bundesliga Season 2013/14 Gerhard Tutz & Gunther Schauberger Ludwig-Maimilians-Universität München Akademiestraße 1, 80799 München
Aufbau und Funktion des Genoms: Von der Genstruktur zur Funktion
 Assoc. Prof. PD Mag. Dr. Aufbau und Funktion des Genoms: Von der Genstruktur zur Funktion Wien, 2013 Währinger Straße 10, A-1090 Wien helmut.dolznig@meduniwien.ac.at www.meduniwien.ac.at/medizinische-genetik
Assoc. Prof. PD Mag. Dr. Aufbau und Funktion des Genoms: Von der Genstruktur zur Funktion Wien, 2013 Währinger Straße 10, A-1090 Wien helmut.dolznig@meduniwien.ac.at www.meduniwien.ac.at/medizinische-genetik
Transgene Tiere: Genmodifikation in der Maus
 Transgene Tiere: Genmodifikation in der Maus Gentechnik und Genomics WiSe 2007/2008 Kristian M. Müller Institut für Biologie III Albert-Ludwigs-Universität Freiburg Nobelpreis Physiologie und Medizin 2007
Transgene Tiere: Genmodifikation in der Maus Gentechnik und Genomics WiSe 2007/2008 Kristian M. Müller Institut für Biologie III Albert-Ludwigs-Universität Freiburg Nobelpreis Physiologie und Medizin 2007
Das Human-Genom und seine sich abzeichnende Dynamik
 Das Human-Genom und seine sich abzeichnende Dynamik Matthias Platzer Genomanalyse Leibniz Institut für Altersforschung - Fritz-Lipmann Institut (FLI) Humangenom - Sequenzierung 1. Methode 2. Ergebnisse
Das Human-Genom und seine sich abzeichnende Dynamik Matthias Platzer Genomanalyse Leibniz Institut für Altersforschung - Fritz-Lipmann Institut (FLI) Humangenom - Sequenzierung 1. Methode 2. Ergebnisse
Klonierung von Säugern durch Zellkerntransfer
 Klonierung von Säugern durch Zellkerntransfer Gentechnik und Genomics WiSe 2007/2008 Kristian M. Müller Institut für Biologie III Albert-Ludwigs-Universität Freiburg Können differenzierte Zellen einen
Klonierung von Säugern durch Zellkerntransfer Gentechnik und Genomics WiSe 2007/2008 Kristian M. Müller Institut für Biologie III Albert-Ludwigs-Universität Freiburg Können differenzierte Zellen einen
Genexpressionsregulation
 Genexpressionsregulation Genexpressionsregulation Different tissue types 1 2 3 4 5 6 7 8 Taken from Caron et al., 2001 Verschiedene Ebenen der Genexpressionsregulation Epigenetic mechanisms Transkriptionskontrolle
Genexpressionsregulation Genexpressionsregulation Different tissue types 1 2 3 4 5 6 7 8 Taken from Caron et al., 2001 Verschiedene Ebenen der Genexpressionsregulation Epigenetic mechanisms Transkriptionskontrolle
Chelatisierende Koordination von Glucopyranose an ein Metallzentrum.
 Lars Jessen Dissertation 3 Zusammenfassung Ziel der vorliegenden Arbeit war, die Möglichkeit der Koordination von Kohlenhydratbausteinen, insbesondere von Glucopyranosen, als Diolatoliganden an (IV)- und
Lars Jessen Dissertation 3 Zusammenfassung Ziel der vorliegenden Arbeit war, die Möglichkeit der Koordination von Kohlenhydratbausteinen, insbesondere von Glucopyranosen, als Diolatoliganden an (IV)- und
Stand von letzter Woche
 RUB ECR1 AXR1 Stand von letzter Woche E2 U? E1-like AXR1 Repressor ARF1 Proteasom AuxRE Repressor wird sehr schnell abgebaut notwendig für Auxinantwort evtl. Substrat für SCF Identifikation des SCF-Ubiquitin
RUB ECR1 AXR1 Stand von letzter Woche E2 U? E1-like AXR1 Repressor ARF1 Proteasom AuxRE Repressor wird sehr schnell abgebaut notwendig für Auxinantwort evtl. Substrat für SCF Identifikation des SCF-Ubiquitin
Steuerung der Proteinproduktion durch kleine RNA-Moleküle
 Meister, Gunter Steuerung der Proteinproduktion durch kleine RNA-Moleküle Tätigkeitsbericht 2009/2010 Steuerung der Proteinproduktion durch kleine RNA-Moleküle Meister, Gunter Max-Planck-Institut für Biochemie,
Meister, Gunter Steuerung der Proteinproduktion durch kleine RNA-Moleküle Tätigkeitsbericht 2009/2010 Steuerung der Proteinproduktion durch kleine RNA-Moleküle Meister, Gunter Max-Planck-Institut für Biochemie,
^Stfir. Importance of the immunoglobulin-like domains and the fibronectin type III homologous repeats
 Diss.ETH
Diss.ETH
Rollen im Participant Portal
 Rollen im Participant Portal Stand Februar 2011 Inhaltsverzeichnis 1 Welche Aufteilung existiert grundsätzlich im PP?...3 1.1 Organisation Roles:...3 1.2 Project Roles:...4 1.2.1 1st level: Coordinator
Rollen im Participant Portal Stand Februar 2011 Inhaltsverzeichnis 1 Welche Aufteilung existiert grundsätzlich im PP?...3 1.1 Organisation Roles:...3 1.2 Project Roles:...4 1.2.1 1st level: Coordinator
Inhalte unseres Vortrages
 Inhalte unseres Vortrages Vorstellung der beiden paper: Germ line transmission of a disrupted ß2 mirkroglobulin gene produced by homologous recombination in embryonic stem cells ß2 Mikroglobulin deficient
Inhalte unseres Vortrages Vorstellung der beiden paper: Germ line transmission of a disrupted ß2 mirkroglobulin gene produced by homologous recombination in embryonic stem cells ß2 Mikroglobulin deficient
Field Librarianship in den USA
 Field Librarianship in den USA Bestandsaufnahme und Zukunftsperspektiven Vorschau subject librarians field librarians in den USA embedded librarians das amerikanische Hochschulwesen Zukunftsperspektiven
Field Librarianship in den USA Bestandsaufnahme und Zukunftsperspektiven Vorschau subject librarians field librarians in den USA embedded librarians das amerikanische Hochschulwesen Zukunftsperspektiven
Grundlegende Experimente der Molekulargenetik
 Übung 12 Wiederholung/Zusatzübung Inhalte: Mendelscher Erbgang Grundlegende Experimente der Molekulargenetik Transposons Methoden 1. Sie haben drei runde, gelbe Erbsen (A, B und C). Aus jeder der drei
Übung 12 Wiederholung/Zusatzübung Inhalte: Mendelscher Erbgang Grundlegende Experimente der Molekulargenetik Transposons Methoden 1. Sie haben drei runde, gelbe Erbsen (A, B und C). Aus jeder der drei
Draft. Draft Specimen 2018 Morning Time allowed: 2 hours
 SPECIMEN MATERIAL A-level GERMAN Paper 2 Writing Specimen 2018 Morning Time allowed: 2 hours Materials For this paper you must have: Answer book Instructions Use black ink or black ball-point pen. Answer
SPECIMEN MATERIAL A-level GERMAN Paper 2 Writing Specimen 2018 Morning Time allowed: 2 hours Materials For this paper you must have: Answer book Instructions Use black ink or black ball-point pen. Answer
DATENQUALITÄT IN GENOMDATENBANKEN
 DATENQUALITÄT IN GENOMDATENBANKEN Alexander Fehr 28. Januar 2004 Gliederung Motivation Biologische Grundkonzepte Genomdaten Datenproduktion und Fehler Data Cleansing 2 Motivation (1) Genomdatenbanken enthalten
DATENQUALITÄT IN GENOMDATENBANKEN Alexander Fehr 28. Januar 2004 Gliederung Motivation Biologische Grundkonzepte Genomdaten Datenproduktion und Fehler Data Cleansing 2 Motivation (1) Genomdatenbanken enthalten
Appendix 41: Die Entwicklung der Insektenflügel: Musterbildung durch morphogenetische Felder und direkte Zell-Zell-Kommunikation
 Appendix 41: Die Entwicklung der Insektenflügel: Musterbildung durch morphogenetische Felder und direkte Zell-Zell-Kommunikation Appendix 41: Die Entwicklung der Insektenflügel: Musterbildung durch morphogenetische
Appendix 41: Die Entwicklung der Insektenflügel: Musterbildung durch morphogenetische Felder und direkte Zell-Zell-Kommunikation Appendix 41: Die Entwicklung der Insektenflügel: Musterbildung durch morphogenetische
Inhalt 1 Modellorganismen
 Inhalt 1 Modellorganismen....................................... 1 1.1 Escherichia coli....................................... 1 1.1.1 Historisches...................................... 3 1.1.2 Lebenszyklus.....................................
Inhalt 1 Modellorganismen....................................... 1 1.1 Escherichia coli....................................... 1 1.1.1 Historisches...................................... 3 1.1.2 Lebenszyklus.....................................
Geometrie und Bedeutung: Kap 5
 : Kap 5 21. November 2011 Übersicht Der Begriff des Vektors Ähnlichkeits Distanzfunktionen für Vektoren Skalarprodukt Eukidische Distanz im R n What are vectors I Domininic: Maryl: Dollar Po Euro Yen 6
: Kap 5 21. November 2011 Übersicht Der Begriff des Vektors Ähnlichkeits Distanzfunktionen für Vektoren Skalarprodukt Eukidische Distanz im R n What are vectors I Domininic: Maryl: Dollar Po Euro Yen 6
PentaFluar S Begehrte Lichtpunkte
 Mikroskopie von Carl Zeiss PentaFluar S Begehrte Lichtpunkte Koaxiale Fluoreszenzeinrichtung für Stereomikroskope SteREO Discovery We make it visible. PentaFluar S Nachrüstbarer Fluoreszenzbaustein für
Mikroskopie von Carl Zeiss PentaFluar S Begehrte Lichtpunkte Koaxiale Fluoreszenzeinrichtung für Stereomikroskope SteREO Discovery We make it visible. PentaFluar S Nachrüstbarer Fluoreszenzbaustein für
Ergebnisse. I. Identifizierung intrazellulärer Interaktionspartner. Ergebnisse I. Identifizierung intrazellulärer Interaktionspartner
 Ergebnisse I. Identifizierung intrazellulärer Interaktionspartner Es sollten mithilfe des Yeast-Two-Hybrid-Systems Proteine identifiziert werden, die mit der zytoplasmatischen Domäne von CEACAM1-4L aus
Ergebnisse I. Identifizierung intrazellulärer Interaktionspartner Es sollten mithilfe des Yeast-Two-Hybrid-Systems Proteine identifiziert werden, die mit der zytoplasmatischen Domäne von CEACAM1-4L aus
Sind MADS-Box-Gene ein Schlüssel zum Verständnis der Entwicklung und Evolution der Gametophyten-Generation von Landpflanzen?
 P flanzenforschung Sind MADS-Box-Gene ein Schlüssel zum Verständnis der Entwicklung und Evolution der Gametophyten-Generation von Landpflanzen? Verelst, Wim; Münster, Thomas; Max-Planck-Institut für Züchtungsforschung,
P flanzenforschung Sind MADS-Box-Gene ein Schlüssel zum Verständnis der Entwicklung und Evolution der Gametophyten-Generation von Landpflanzen? Verelst, Wim; Münster, Thomas; Max-Planck-Institut für Züchtungsforschung,
Kapitel 8 Ò Chromosomen und Genregulation
 Kapitel 8 Ò Chromosomen und Genregulation 8.1 Struktur eukaryontischer Chromosomen Ein menschlicher Zellkern ist nur zehn Mikrometer gross und (10-9 ) hat zwei Meter DNA drin. Damit es da kein Durcheinander
Kapitel 8 Ò Chromosomen und Genregulation 8.1 Struktur eukaryontischer Chromosomen Ein menschlicher Zellkern ist nur zehn Mikrometer gross und (10-9 ) hat zwei Meter DNA drin. Damit es da kein Durcheinander
Nothing in Biology makes sense except in the light of evolution.
 Meine Referenz an Charles Darwin...ist ein Zitat von Theodosius Dobzhansky: Nothing in Biology makes sense except in the light of evolution. Collegium Generale, 18. März 2009 Genetik versus Epigenetik
Meine Referenz an Charles Darwin...ist ein Zitat von Theodosius Dobzhansky: Nothing in Biology makes sense except in the light of evolution. Collegium Generale, 18. März 2009 Genetik versus Epigenetik
Identification of microrna-mrna functional interactions in UVB-induced senescence of human diploid fibroblasts
 Identification of microrna-mrna functional interactions in UVB-induced senescence of human diploid fibroblasts Ruth Greussing, Matthias Hackl, Pornpimol Charoentong, Alexander Pauck, Rossella Monteforte,
Identification of microrna-mrna functional interactions in UVB-induced senescence of human diploid fibroblasts Ruth Greussing, Matthias Hackl, Pornpimol Charoentong, Alexander Pauck, Rossella Monteforte,
p^db=`oj===pìééçêíáåñçêã~íáçå=
 p^db=`oj===pìééçêíáåñçêã~íáçå= How to Disable User Account Control (UAC) in Windows Vista You are attempting to install or uninstall ACT! when Windows does not allow you access to needed files or folders.
p^db=`oj===pìééçêíáåñçêã~íáçå= How to Disable User Account Control (UAC) in Windows Vista You are attempting to install or uninstall ACT! when Windows does not allow you access to needed files or folders.
Arten der Intronen. Spliceosome Struktur des snrnp U1. Spliceosome. Vorlesung 3: Evolution des eukaryotischen Genoms 4/20/11
 Vorlesung 3: Evolution des eukaryotischen Genoms Struktur des eukaryotischen Gens Intronen und Exonen Genduplikation--Familien des Gens Eiweißdomänen (Protein domains) Domänen und Exonen: sind sie verwandt?
Vorlesung 3: Evolution des eukaryotischen Genoms Struktur des eukaryotischen Gens Intronen und Exonen Genduplikation--Familien des Gens Eiweißdomänen (Protein domains) Domänen und Exonen: sind sie verwandt?
Neue DNA Sequenzierungstechnologien im Überblick
 Neue DNA Sequenzierungstechnologien im Überblick Dr. Bernd Timmermann Next Generation Sequencing Core Facility Max Planck Institute for Molecular Genetics Berlin, Germany Max-Planck-Gesellschaft 80 Institute
Neue DNA Sequenzierungstechnologien im Überblick Dr. Bernd Timmermann Next Generation Sequencing Core Facility Max Planck Institute for Molecular Genetics Berlin, Germany Max-Planck-Gesellschaft 80 Institute
Molekulare Mechanismen der Signaltransduktion. 06 - Kartierung des AXR1 Gens + early auxin-induced genes Folien: http://tinyurl.
 Molekulare Mechanismen der Signaltransduktion 06 - Kartierung des AXR1 Gens + early auxin-induced genes Folien: http://tinyurl.com/modul-mms bisheriges Modell auxin auxin AXR1 auxin response AXR1 potentieller
Molekulare Mechanismen der Signaltransduktion 06 - Kartierung des AXR1 Gens + early auxin-induced genes Folien: http://tinyurl.com/modul-mms bisheriges Modell auxin auxin AXR1 auxin response AXR1 potentieller
RNAI SCREENING FOR GENES REQUIRED FOR VIRUS INFECTION AND ENDOCYTOSIS
 Diss. ETH N 18793 RNAI SCREENING FOR GENES REQUIRED FOR VIRUS INFECTION AND ENDOCYTOSIS A dissertation submitted to ETH ZURICH for the degree of Doctor of Sciences presented by RAPHAEL CHRISTIAN SACHER
Diss. ETH N 18793 RNAI SCREENING FOR GENES REQUIRED FOR VIRUS INFECTION AND ENDOCYTOSIS A dissertation submitted to ETH ZURICH for the degree of Doctor of Sciences presented by RAPHAEL CHRISTIAN SACHER
H.Schwab Genetik. Überblick
 Überblick Einleitung: Historisches Klassische - Mendel DNA, Aufbau, übergeordnete Strukturen, Konfigurationen, zelluläre Organisation Chromatin, Chromosomenaufbau, Genome Extrachromosomale Elemente, mobile
Überblick Einleitung: Historisches Klassische - Mendel DNA, Aufbau, übergeordnete Strukturen, Konfigurationen, zelluläre Organisation Chromatin, Chromosomenaufbau, Genome Extrachromosomale Elemente, mobile
In situ Hybridisierung
 In situ Hybridisierung eine Methode zum direkten und spezifischen Nachweis von Nukleinsäuren (DNA und RNA) in Gewebe, Zellen, Zellkompartimenten und Chromosomen Was kann damit erreicht werden? direkte
In situ Hybridisierung eine Methode zum direkten und spezifischen Nachweis von Nukleinsäuren (DNA und RNA) in Gewebe, Zellen, Zellkompartimenten und Chromosomen Was kann damit erreicht werden? direkte
3. BvD Transfer Pricing Day BEPS und die Auswirkungen auf das operationale Verrechnungspreis-Management
 3. BvD Transfer Pricing Day BEPS und die Auswirkungen auf das operationale Verrechnungspreis-Management Agenda Einführung Operationales Verrechnungspreis- Management Was bedeutet BEPS für Unternehmen?
3. BvD Transfer Pricing Day BEPS und die Auswirkungen auf das operationale Verrechnungspreis-Management Agenda Einführung Operationales Verrechnungspreis- Management Was bedeutet BEPS für Unternehmen?
Nachlese zu den Referaten
 Nachlese zu den Referaten Referate Die Themen Phylogenie Aminosäurematrizen für die Eukaryontenphylogenie Ansätze für die Prokaryontenphylogenie Vergleichende Sequenzierung Spezieswahl zur Informationsoptimierung
Nachlese zu den Referaten Referate Die Themen Phylogenie Aminosäurematrizen für die Eukaryontenphylogenie Ansätze für die Prokaryontenphylogenie Vergleichende Sequenzierung Spezieswahl zur Informationsoptimierung
Funktionale Sicherheit ISO 26262 Schwerpunkt Requirements Engineering,
 Funktionale Sicherheit ISO 26262 Schwerpunkt Requirements Engineering, Manfred Broy Lehrstuhl für Software & Systems Engineering Technische Universität München Institut für Informatik ISO 26262 Functional
Funktionale Sicherheit ISO 26262 Schwerpunkt Requirements Engineering, Manfred Broy Lehrstuhl für Software & Systems Engineering Technische Universität München Institut für Informatik ISO 26262 Functional
Evidence of strobilurine resistant isolates of A. solani and A. alternata in Germany
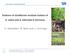 Technische Universität München Evidence of strobilurine resistant isolates of A. solani and A. alternata in Germany H. Hausladen, B. Adolf and J. Leiminger outline introduction material and methods results
Technische Universität München Evidence of strobilurine resistant isolates of A. solani and A. alternata in Germany H. Hausladen, B. Adolf and J. Leiminger outline introduction material and methods results
Parameter-Updatesoftware PF-12 Plus
 Parameter-Updatesoftware PF-12 Plus Mai / May 2015 Inhalt 1. Durchführung des Parameter-Updates... 2 2. Kontakt... 6 Content 1. Performance of the parameter-update... 4 2. Contact... 6 1. Durchführung
Parameter-Updatesoftware PF-12 Plus Mai / May 2015 Inhalt 1. Durchführung des Parameter-Updates... 2 2. Kontakt... 6 Content 1. Performance of the parameter-update... 4 2. Contact... 6 1. Durchführung
Musterlösung - Übung 5 Vorlesung Bio-Engineering Sommersemester 2008
 Aufgabe 1: Prinzipieller Ablauf der Proteinbiosynthese a) Erklären Sie folgende Begriffe möglichst in Ihren eigenen Worten (1 kurzer Satz): Gen Nukleotid RNA-Polymerase Promotor Codon Anti-Codon Stop-Codon
Aufgabe 1: Prinzipieller Ablauf der Proteinbiosynthese a) Erklären Sie folgende Begriffe möglichst in Ihren eigenen Worten (1 kurzer Satz): Gen Nukleotid RNA-Polymerase Promotor Codon Anti-Codon Stop-Codon
Alternative Methoden der RNA-Analyse
 Alternative Methoden der RNA-Analyse In diesem Versuch wurde die Northern Blot Hybridisierung zur Analyse isolierter mrna eingesetzt. Mit dieser Technik können Größe und Menge einer spezifischen RNA bestimmt
Alternative Methoden der RNA-Analyse In diesem Versuch wurde die Northern Blot Hybridisierung zur Analyse isolierter mrna eingesetzt. Mit dieser Technik können Größe und Menge einer spezifischen RNA bestimmt
Diplomarbeit. Entwurf eines generischen Prozessleitstandes für Change Request Systeme
 Fakultät für Mathematik, Informatik und Naturwissenschaften Forschungsgruppe Softwarekonstruktion Diplomarbeit Entwurf eines generischen Prozessleitstandes für Change Request Systeme Development of a Generic
Fakultät für Mathematik, Informatik und Naturwissenschaften Forschungsgruppe Softwarekonstruktion Diplomarbeit Entwurf eines generischen Prozessleitstandes für Change Request Systeme Development of a Generic
Überblick von DNA zu Protein. Biochemie-Seminar WS 04/05
 Überblick von DNA zu Protein Biochemie-Seminar WS 04/05 Replikationsapparat der Zelle Der gesamte Replikationsapparat umfasst über 20 Proteine z.b. DNA Polymerase: katalysiert Zusammenfügen einzelner Bausteine
Überblick von DNA zu Protein Biochemie-Seminar WS 04/05 Replikationsapparat der Zelle Der gesamte Replikationsapparat umfasst über 20 Proteine z.b. DNA Polymerase: katalysiert Zusammenfügen einzelner Bausteine
Employment and Salary Verification in the Internet (PA-PA-US)
 Employment and Salary Verification in the Internet (PA-PA-US) HELP.PYUS Release 4.6C Employment and Salary Verification in the Internet (PA-PA-US SAP AG Copyright Copyright 2001 SAP AG. Alle Rechte vorbehalten.
Employment and Salary Verification in the Internet (PA-PA-US) HELP.PYUS Release 4.6C Employment and Salary Verification in the Internet (PA-PA-US SAP AG Copyright Copyright 2001 SAP AG. Alle Rechte vorbehalten.
Ways and methods to secure customer satisfaction at the example of a building subcontractor
 Abstract The thesis on hand deals with customer satisfaction at the example of a building subcontractor. Due to the problems in the building branch, it is nowadays necessary to act customer oriented. Customer
Abstract The thesis on hand deals with customer satisfaction at the example of a building subcontractor. Due to the problems in the building branch, it is nowadays necessary to act customer oriented. Customer
Group and Session Management for Collaborative Applications
 Diss. ETH No. 12075 Group and Session Management for Collaborative Applications A dissertation submitted to the SWISS FEDERAL INSTITUTE OF TECHNOLOGY ZÜRICH for the degree of Doctor of Technical Seiences
Diss. ETH No. 12075 Group and Session Management for Collaborative Applications A dissertation submitted to the SWISS FEDERAL INSTITUTE OF TECHNOLOGY ZÜRICH for the degree of Doctor of Technical Seiences
Customer-specific software for autonomous driving and driver assistance (ADAS)
 This press release is approved for publication. Press Release Chemnitz, February 6 th, 2014 Customer-specific software for autonomous driving and driver assistance (ADAS) With the new product line Baselabs
This press release is approved for publication. Press Release Chemnitz, February 6 th, 2014 Customer-specific software for autonomous driving and driver assistance (ADAS) With the new product line Baselabs
Auftaktveranstaltung zum Vorhaben DendroMax
 Auftaktveranstaltung zum Vorhaben DendroMax Entwicklung der biotechnologischen Grundlagen und praxisnaher Anbauverfahren für die Steigerung der Dendromasseproduktion in Land- und Forstwirtschaft durch
Auftaktveranstaltung zum Vorhaben DendroMax Entwicklung der biotechnologischen Grundlagen und praxisnaher Anbauverfahren für die Steigerung der Dendromasseproduktion in Land- und Forstwirtschaft durch
Algorithms for graph visualization
 Algorithms for graph visualization Project - Orthogonal Grid Layout with Small Area W INTER SEMESTER 2013/2014 Martin No llenburg KIT Universita t des Landes Baden-Wu rttemberg und nationales Forschungszentrum
Algorithms for graph visualization Project - Orthogonal Grid Layout with Small Area W INTER SEMESTER 2013/2014 Martin No llenburg KIT Universita t des Landes Baden-Wu rttemberg und nationales Forschungszentrum
Informationsextraktion
 Informationsextraktion Bestimmte Anwendungen bei der semantischen Verarbeitung erfordern keine tiefe linguistische Analyse mit exakter Disambiguierung (= eine einzige und korrekte Lesart). Hierzu gehört
Informationsextraktion Bestimmte Anwendungen bei der semantischen Verarbeitung erfordern keine tiefe linguistische Analyse mit exakter Disambiguierung (= eine einzige und korrekte Lesart). Hierzu gehört
Genetische Regulation in Eukaryoten
 Genetische Regulation in Eukaryoten Definition: Im strengen Sinne: Regulierung der Transkription eines Genes zu RNAs Im weiteren Sinne: + Regulierung der Expression und Funktion von Genprodukten (RNAs,
Genetische Regulation in Eukaryoten Definition: Im strengen Sinne: Regulierung der Transkription eines Genes zu RNAs Im weiteren Sinne: + Regulierung der Expression und Funktion von Genprodukten (RNAs,
Homologie und Sequenzähnlichkeit. Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de
 Homologie und Sequenzähnlichkeit Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de Homologie Verwandtschaft aufgrund gleicher Abstammung basiert auf Speziation (Artbildung): aus einer
Homologie und Sequenzähnlichkeit Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de Homologie Verwandtschaft aufgrund gleicher Abstammung basiert auf Speziation (Artbildung): aus einer
Kursinhalte der Weiterbildung Molekulare Biotechnologie (MNr.: 237 / 0411 / 2010)
 Kursinhalte der Weiterbildung Molekulare Biotechnologie (MNr.: 237 / 0411 / 2010) Schwerpunkte im Bereich BIOANALYTIK Polyacrylamidelektrophorese von Nukleinsäuren im Vertikalsystem Agarosegelelektrophorese
Kursinhalte der Weiterbildung Molekulare Biotechnologie (MNr.: 237 / 0411 / 2010) Schwerpunkte im Bereich BIOANALYTIK Polyacrylamidelektrophorese von Nukleinsäuren im Vertikalsystem Agarosegelelektrophorese
ZUR ONTOGENESE DER KATZENKRALLE
 Aus dem Institut für Veterinär-Anatomie des Fachbereiches Veterinärmedizin der Freien Universität Berlin Laboratorium Prof. Dr. K.-D. Budras ZUR ONTOGENESE DER KATZENKRALLE INAUGURAL-DISSERTATION zur Erlangung
Aus dem Institut für Veterinär-Anatomie des Fachbereiches Veterinärmedizin der Freien Universität Berlin Laboratorium Prof. Dr. K.-D. Budras ZUR ONTOGENESE DER KATZENKRALLE INAUGURAL-DISSERTATION zur Erlangung
Biochemie. Arbeitsgruppen. AG Frederik Börnke. AG Jörg Hofmann. AG Christian Koch. AG Uwe Sonnewald. AG Sophia Sonnewald.
 Biochemie Arbeitsgruppen AG Frederik Börnke AG Jörg Hofmann AG Christian Koch AG Uwe Sonnewald AG Sophia Sonnewald AG Lars Voll http://www.biologie.uni-erlangen.de/bc/bchome.html Biochemie Forschungsprogramme
Biochemie Arbeitsgruppen AG Frederik Börnke AG Jörg Hofmann AG Christian Koch AG Uwe Sonnewald AG Sophia Sonnewald AG Lars Voll http://www.biologie.uni-erlangen.de/bc/bchome.html Biochemie Forschungsprogramme
Facetten von Designforschung Einblicke in den Stand der Dinge
 Hans Kaspar Hugentobler Designforschung: Vielfalt, Relevanz, Ideologie Facetten von Designforschung Einblicke in den Stand der Dinge Hans Kaspar Hugentobler Master of Design Diplom-Kommunikationswirt Bremen
Hans Kaspar Hugentobler Designforschung: Vielfalt, Relevanz, Ideologie Facetten von Designforschung Einblicke in den Stand der Dinge Hans Kaspar Hugentobler Master of Design Diplom-Kommunikationswirt Bremen
Gene-Silencing in transgenen Pflanzen
 Schmidt, Renate et al. Gen-Silencing in transgenen Pflanzen Tätigkeitsbericht 2006 Pflanzenforschung Gene-Silencing in transgenen Pflanzen Schmidt, Renate; Arlt, Matthias Max-Planck-Institut für molekulare
Schmidt, Renate et al. Gen-Silencing in transgenen Pflanzen Tätigkeitsbericht 2006 Pflanzenforschung Gene-Silencing in transgenen Pflanzen Schmidt, Renate; Arlt, Matthias Max-Planck-Institut für molekulare
Übung 8. 1. Zellkommunikation. Vorlesung Bio-Engineering Sommersemester 2008. Kapitel 4. 4
 Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 05. 05. 08
Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 05. 05. 08
DAS METABOLOM: ERNÄHRUNGSSTATUS, METABOLISMUS, METABOLITEN
 DAS METABOLOM: ERNÄHRUNGSSTATUS, METABOLISMUS, METABOLITEN Das Metabolom Extrinsiche Faktoren Ernährung (Nährstoffe und nicht nutritive Lebensmittelinhaltsstoffe) Arzneimittel Physische Aktivität Darmflora
DAS METABOLOM: ERNÄHRUNGSSTATUS, METABOLISMUS, METABOLITEN Das Metabolom Extrinsiche Faktoren Ernährung (Nährstoffe und nicht nutritive Lebensmittelinhaltsstoffe) Arzneimittel Physische Aktivität Darmflora
ABSTRACT. A large body of empirical research documents that different linguistic forms lead to a
 ABSTRACT The present research investigates the impact of gender-fair language on gender equality in the work context. A large body of empirical research documents that different linguistic forms lead to
ABSTRACT The present research investigates the impact of gender-fair language on gender equality in the work context. A large body of empirical research documents that different linguistic forms lead to
Spleissen Dezember 2009 Elmar Schiebel, ZMBH
 Spleissen Dezember 2009 Elmar Schiebel, ZMBH Bis 1970s: Eine Gen besteht aus einem Stück doppelsträngiger DNA. Dieses einfache Bild wurde 1977 durch die Entdeckung von Richard J. Roberts (Cold Spring Harbor
Spleissen Dezember 2009 Elmar Schiebel, ZMBH Bis 1970s: Eine Gen besteht aus einem Stück doppelsträngiger DNA. Dieses einfache Bild wurde 1977 durch die Entdeckung von Richard J. Roberts (Cold Spring Harbor
HISTONE MODIFICATIONS AND TRANSCRIPT PROCESSING DURING ARABIDOPSIS DEVELOPMENT
 DISS. ETH NO. 21256 HISTONE MODIFICATIONS AND TRANSCRIPT PROCESSING DURING ARABIDOPSIS DEVELOPMENT A dissertation submitted to ETH ZURICH for the degree of Doctor of Sciences presented by Walid Mahrez
DISS. ETH NO. 21256 HISTONE MODIFICATIONS AND TRANSCRIPT PROCESSING DURING ARABIDOPSIS DEVELOPMENT A dissertation submitted to ETH ZURICH for the degree of Doctor of Sciences presented by Walid Mahrez
In situ Abbildung magnetischer Domänen in dünnen Filmen mit magnetooptischer Rasternahfeldmikroskopie
 In situ Abbildung magnetischer Domänen in dünnen Filmen mit magnetooptischer Rasternahfeldmikroskopie von Gereon Meyer am Fachbereich Physik der Freien Universität Berlin eingereichte Dissertation Oktober
In situ Abbildung magnetischer Domänen in dünnen Filmen mit magnetooptischer Rasternahfeldmikroskopie von Gereon Meyer am Fachbereich Physik der Freien Universität Berlin eingereichte Dissertation Oktober
eurex rundschreiben 094/10
 eurex rundschreiben 094/10 Datum: Frankfurt, 21. Mai 2010 Empfänger: Alle Handelsteilnehmer der Eurex Deutschland und Eurex Zürich sowie Vendoren Autorisiert von: Jürg Spillmann Weitere Informationen zur
eurex rundschreiben 094/10 Datum: Frankfurt, 21. Mai 2010 Empfänger: Alle Handelsteilnehmer der Eurex Deutschland und Eurex Zürich sowie Vendoren Autorisiert von: Jürg Spillmann Weitere Informationen zur
Thema Gentechnologie. Erwin R. Schmidt Institut für Molekulargenetik Gentechnologische Sicherheitsforschung & Beratung
 Thema Gentechnologie Erwin R. Schmidt Institut für Molekulargenetik Gentechnologische Sicherheitsforschung & Beratung Die Genklonierung in Bakterien Vektor-DNA Spender-DNA Restriktionsenzym Rekombinante
Thema Gentechnologie Erwin R. Schmidt Institut für Molekulargenetik Gentechnologische Sicherheitsforschung & Beratung Die Genklonierung in Bakterien Vektor-DNA Spender-DNA Restriktionsenzym Rekombinante
Lsd1 weist Stammzellen im Mausembryo den richtigen Weg
 Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/lsd1-weist-stammzellen-immausembryo-den-richtigen-weg/ Lsd1 weist Stammzellen im Mausembryo den richtigen Weg Die
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/lsd1-weist-stammzellen-immausembryo-den-richtigen-weg/ Lsd1 weist Stammzellen im Mausembryo den richtigen Weg Die
Large-Scale Mining and Retrieval of Visual Data in a Multimodal Context
 Diss. ETH No. 18190 Large-Scale Mining and Retrieval of Visual Data in a Multimodal Context A dissertation submitted to the SWISS FEDERAL INSTITUTE OF TECHNOLOGY ZURICH for the degree of Doctor of Technical
Diss. ETH No. 18190 Large-Scale Mining and Retrieval of Visual Data in a Multimodal Context A dissertation submitted to the SWISS FEDERAL INSTITUTE OF TECHNOLOGY ZURICH for the degree of Doctor of Technical
Eine neue RNA-Welt. Uralte RNA-Welt Am Anfang der Entstehung des Lebens. Bekannte RNA-Welt Protein-Synthese. Neue RNA-Welt Regulatorische RNA-Moleküle
 RNAs Eine neue RNA-Welt 1. Uralte RNA-Welt Am Anfang der Entstehung des Lebens Bekannte RNA-Welt Protein-Synthese Neue RNA-Welt Regulatorische RNA-Moleküle 2. Eine neue RNA-Welt die Anzahl der nicht-kodierenden
RNAs Eine neue RNA-Welt 1. Uralte RNA-Welt Am Anfang der Entstehung des Lebens Bekannte RNA-Welt Protein-Synthese Neue RNA-Welt Regulatorische RNA-Moleküle 2. Eine neue RNA-Welt die Anzahl der nicht-kodierenden
ELBA2 ILIAS TOOLS AS SINGLE APPLICATIONS
 ELBA2 ILIAS TOOLS AS SINGLE APPLICATIONS An AAA/Switch cooperative project run by LET, ETH Zurich, and ilub, University of Bern Martin Studer, ilub, University of Bern Julia Kehl, LET, ETH Zurich 1 Contents
ELBA2 ILIAS TOOLS AS SINGLE APPLICATIONS An AAA/Switch cooperative project run by LET, ETH Zurich, and ilub, University of Bern Martin Studer, ilub, University of Bern Julia Kehl, LET, ETH Zurich 1 Contents
RS232-Verbindung, RXU10 Herstellen einer RS232-Verbindung zwischen PC und Messgerät oder Modem und Messgerät
 Betriebsanleitung RS232-Verbindung, RXU10 Herstellen einer RS232-Verbindung zwischen PC und Messgerät oder Modem und Messgerät ä 2 Operating Instructions RS232 Connection, RXU10 Setting up an RS232 connection
Betriebsanleitung RS232-Verbindung, RXU10 Herstellen einer RS232-Verbindung zwischen PC und Messgerät oder Modem und Messgerät ä 2 Operating Instructions RS232 Connection, RXU10 Setting up an RS232 connection
Franke & Bornberg award AachenMünchener private annuity insurance schemes top grades
 Franke & Bornberg award private annuity insurance schemes top grades Press Release, December 22, 2009 WUNSCHPOLICE STRATEGIE No. 1 gets best possible grade FFF ( Excellent ) WUNSCHPOLICE conventional annuity
Franke & Bornberg award private annuity insurance schemes top grades Press Release, December 22, 2009 WUNSCHPOLICE STRATEGIE No. 1 gets best possible grade FFF ( Excellent ) WUNSCHPOLICE conventional annuity
Extract of the Annotations used for Econ 5080 at the University of Utah, with study questions, akmk.pdf.
 1 The zip archives available at http://www.econ.utah.edu/ ~ ehrbar/l2co.zip or http: //marx.econ.utah.edu/das-kapital/ec5080.zip compiled August 26, 2010 have the following content. (they differ in their
1 The zip archives available at http://www.econ.utah.edu/ ~ ehrbar/l2co.zip or http: //marx.econ.utah.edu/das-kapital/ec5080.zip compiled August 26, 2010 have the following content. (they differ in their
Lehrstuhl für Allgemeine BWL Strategisches und Internationales Management Prof. Dr. Mike Geppert Carl-Zeiß-Str. 3 07743 Jena
 Lehrstuhl für Allgemeine BWL Strategisches und Internationales Management Prof. Dr. Mike Geppert Carl-Zeiß-Str. 3 07743 Jena http://www.im.uni-jena.de Contents I. Learning Objectives II. III. IV. Recap
Lehrstuhl für Allgemeine BWL Strategisches und Internationales Management Prof. Dr. Mike Geppert Carl-Zeiß-Str. 3 07743 Jena http://www.im.uni-jena.de Contents I. Learning Objectives II. III. IV. Recap
