Segmentierung der Knochenoberfläche einzelner Wirbel für die ultraschallbasierte Navigation an der Lendenwirbelsäule
|
|
|
- Heinz Auttenberg
- vor 5 Jahren
- Abrufe
Transkript
1 Segmentierung der Knochenoberfläche einzelner Wirbel für die ultraschallbasierte Navigation an der Lendenwirbelsäule Claudia Dekomien, Susanne Winter Institut für Neuroinformatik, Ruhr-Universität Bochum, Bochum, Zusammenfassung. Für die klinische Umsetzung computerunterstützter Verfahren ist es wichtig, die Datenverarbeitungsschritte so weit wie möglich zu automatisieren. Wir stellen eine Verarbeitungskette vor, die nach modellbasierter Segmentierung eines Wirbels, in CT-Daten, automatisch Knochenoberflächenpunkte einem einzelnen Wirbel zuordnet. Diese Knochenoberfläche dient im nächsten Schritt zur Registrierung der CT-Daten mit dreidimensionalen Ultraschalldaten. Zur Extraktion der Knochenoberflächenpunkte wurde in den CT-Daten ein Ultraschallscan simuliert, während zur Segmentierung einzelner Wirbel ein modifizierter Shape-based Level Set Ansatz eingesetzt wurde. Die Koppelung der beiden Verfahren lieferte die, für die Registrierung relevante, Knochenoberfläche in den CT-Daten, anhand derer die CT-Daten mit dreidimensionalen Ultraschalldaten registriert werden konnten. 1 Einleitung Die präzise und schnelle Registrierung präoperativer Bilddaten ist eines der Hauptprobleme in der bildbasierten navigierten Chirurgie [1,2]. Wir haben einen Oberflächen-Volumen-Registrierungsalgorithmus entwickelt, der intraoperativen, dreidimensionalen Ultraschall mit präoperativen CT- oder MRT-Daten registriert. Das Verfahren arbeitet robust [3] und die Prozedur kann jederzeit wiederholt werden. Für Details sei auf die Vorarbeiten verwiesen [4,5]. Die Knochenoberfläche wird aus den präoperativen Daten extrahiert und in die Ultraschalldaten projiziert. Mit einem Optimierungsverfahren wird diese Knochenoberfläche in den Ultraschalldaten an die richtige Position transformiert. Als Fitnesskriterium dient die Summe der Grauwerte im Ultraschallvolumen, die von den Punkten der Oberfläche abgedeckt werden. Die Basis für die rigide Registrierung bilden die Knochenoberflächenpunkte, die sich mit dem Ultraschall darstellen lassen. Da sich die einzelnen Wirbel relativ zueinander verschieben, ist es notwendig diese einzeln zu registrieren. Bisher wurde ein manuelles Verfahren verwendet um die einzelnen Knochen voneinander zu separieren. Für eine klinische Umsetzung der Methode ist es allerdings von großem Interesse die präoperativen Benutzeraktionen so gering wie möglich zu halten. Das Ziel unserer Arbeit ist eine möglichst vollständige Automatisierung der präoperativen Datenverarbeitung.
2 Ein Schritt der Verarbeitungskette besteht aus der Segmentierung der einzelnen Knochen. Schwellwert- oder Region Growing Verfahren haben hier den Nachteil, dass die Segmentierung häufig von einem Knochen in den anderen überläuft. Dieses Problem kann mit dem Einsatz modellbasierter Verfahren [6,7] gelöst werden. Die Segmentierung mittels eines Shape-based Level Set Ansatzes wurde mit einer schwellwertbasierten Extraktion von Knochenoberflächenpunkten gekoppelt. Damit kann die Oberflächenpunktmenge eines einzelnen Wirbels, die für die Registrierung notwendig ist, automatisch segmentiert werden. 2 Material und Methode Für die Versuche wurden Spiral-CT-Daten der Lendenwirbelsäule von 16 Patienten und einem Kunststoffphantom verwendet. Die Ultraschalldatensätze wurden mit einem Siemens Sonoline Omnia und einem 5 MHz curved-array Ultraschallwandler akquiriert. Die 3D-Rekonstruktion erfolgte durch magnetisches Tracking mit dem 3D Echotech System. Zur einfacheren Handhabbarkeit wurden alle Datensätze in isotrope Daten der Auflösung 0,5mmx0,5mmx0,5mm umgewandelt. Die automatische Segmentierung der Knochenoberfläche eines Wirbels besteht aus drei Schritten: Als erstes wird ein Ultraschallscan in den CT-Daten simuliert. Mit diesem Scan wird von einem Wirbelsäulenabschnitt der Teil der Knochenoberfläche extrahiert, der sich in einer solchen Ultraschallaufnahme abbilden würde. Anschließend wird ein einzelner Wirbel in den CT-Daten segmentiert. Im dritten Schritt werden die extrahierten Oberflächenpunkte dem segmentierten Wirbel zugeordnet. Das Ergebnis sind die Oberflächenpunkte eines einzelnen Wirbels, die sich in einer Ultraschallaufnahmen abbilden. Anhand dieser Punktmenge kann die Registrierung mit dem dreidimensionalen Ultraschall erfolgen. Die Simulation des Ultraschallscans ist ein halbautomatischer Prozess. Vom Benutzer werden einige Landmarken in den CT-Daten markiert, die den Scanpfad aufspannen, der sich dem intraoperativem Scanpfad annähert. Bei bekannter Schallrichtung werden anhand dieses Scanpfades die Schallstrahlen des Ultraschalls im CT simuliert. Mittels eines Schwellwertes werden diejenigen Knochenoberflächenpunkte extrahiert, die als erstes vom Schallstrahl getroffen werden (siehe Abbildung 1.a). Für die Segmentierung der einzelnen Wirbel wurde ein Shape-based Level Set Ansatz gewählt. Das Verfahren ist modellbasiert und kann durch seine Flexibilität komplexe Formen, wie die der Wirbelknochen, darstellen. Der in dieser Arbeit implementierte Algorithmus basiert auf dem von Tsai [6] vorgestellten Verfahren, welches in zwei Punkten modifiziert wurde [8]. Der Algorithmus besteht aus zwei Phasen, der Modellerstellung und der Segmentierungsphase. Dabei wird sowohl die Registrierung der verschiedenen Einzelwirbel zur Modellerstellung, als auch die Anpassung der Segmentierungsfunktion als Optimierungsproblem formuliert. Aufgrund der Robustheit und der großen Reichweite der
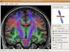
3 Abb. 1. Verarbeitungskette für die Registrierung einzelner Wirbel in CT- und Ultraschalldaten; a) Simulation eines Ultraschallscanpfades in CT-Daten; b) Ergebnis einer Shape-based Level Set Segmentierung; c) Die segmentierten Oberflächenpunkte eines Wirbels; d) Überlagerung von CT- und Ultraschalldaten nach der Registrierung mittels extrahierter Wirbeloberfläche (die Pfeile markieren die Oberfläche des registrierten Wirbels). CMA-Evolutionsstrategie [9] wurde die Lösung der Optimierungen mit diesem Verfahren umgesetzt. Zur Modellerstellung wurden 20 dreidimensionale Wirbel (Lendenwirbel 3 und 4) von 14 Patienten verwendet. Im dritten Schritt wurden aus den in Schritt 1 extrahierten Oberflächenpunkten diejenigen Punkte identifiziert, die eine Distanz von weniger als einem Pixel (kleiner 0,5 mm), zur Kontur des Segmentierungsergebnisses aus Schritt 2, aufwiesen. Die so gewonnene Oberfläche wurde dann zur Registrierung mit dreidimensionalen Ultraschalldaten genutzt. Die Ergebnisse der Registrierung anhand der automatisch segmentierten O- berfläche wurden mit den Ergebnissen einer Registrierung anhand einer manuell segmentierten Oberfläche verglichen. Als Fehlermaß diente der Target Registration Error, wobei die Targets definierten anatomischen Regionen entsprachen, die für eine Pedikelverschraubung wichtig sind (siehe Abbildung 2). In diesen Regionen wurden Punktsets definiert, für welche nach unterschiedlicher Registrierung die Distanzen bestimmt wurden. Wichtige Regionen der Pedikelverschraubung sind die Schraubeneintrittspunkte in den Wirbel, die Pedikel durch die die Schraube geführt werden und die Corpusvorderkante, die von der Schraube nicht durchbohrt werden darf. Die Evaluierung wurde mit drei Patienten- und zwei Phantomwirbeln durchgeführt, die nicht zur Erstellung des Modells verwendet wurden. 3 Ergebnisse Abbildung 1.a zeigt die Simulation des Ultraschallscans in einem Patientendatensatz. In Abbildung 1.b ist das Ergebnis der modellbasierten Segmentierung und in Abbildung 1.c die segmentierte Oberfläche eines einzelnen Wirbels dargestellt. Nach Zuordnung der Oberflächenpunkte zu dem segmentierten Wirbel
4 Abb.2. Punktsets zur Bestimmung des Target Registration Errors a) Pedikel links b) Schraubeneintrittspunkt c) Wirbelköpervorderkante d) Pedikel rechts Tabelle 1. Target Registration Error zwischen dem Registrierungsergebnis mit der manuell ausgeschnittenen Oberfläche und dem Registrierungsergebnis mit der automatisch segmentierten Oberfläche. Berechnet wurde für vier definierte anatomische Wirbelregionen der RMS, der mittlere und der maximale Fehler zwischen den beiden Registrierungen. Daten Pedikel links Pedikel rechts RMS Mean Max RMS Mean Max Patient1 L3 0,56 0,55 0,56 0,49 0,48 0,57 Patient1 L4 0,22 0,22 0,27 0,38 0,38 0,44 Patient2 L4 0,38 0,38 0,48 0,49 0,48 0,59 Phantom L3 0,00 0,00 0,00 0,00 0,00 0,00 Phantom L4 0,14 0,14 0,17 0,22 0,22 0,26 Daten Wirbelköpervorderkante Schraubeneintrittspunkt RMS Mean Max RMS Mean Max Patient1 L3 0,54 0,54 0,64 0,33 0,33 0,38 Patient1 L4 1,06 1,06 1,14 0,23 0,23 0,30 Patient2 L4 1,09 1,09 1,23 0,43 0,43 0,50 Phantom L3 0,00 0,00 0,00 0,00 0,00 0,00 Phantom L4 0,20 0,19 0,28 0,26 0,26 0,29 konnten die CT-Daten anhand der Oberfläche mit den Ultraschalldaten registriert werden (siehe Abbildung 1.d). Im Bereich der Schraubeneintrittspunkte zeigte sich bei Registrierung der Patientendaten ein RMS-Fehler von bis zu 0,43 mm. Die Zielpunkte im Bereich der Pedikel wiesen abweichende Registrierungen von bis zu 0,56 mm RMS-Fehler auf, während sich an der Wirbelkörpervorderkante RMS-Fehler von bis zu 1,09 mm ergaben. Die maximale Abweichung lag hier bei 1,23 mm. Die Phantomdaten konnten mit einem maximalen RMS-Fehler von 0,26 mm, bei maximaler Abweichung von 0,29 mm, im Bereich der Schraubeneintrittspunkte registriert werden. Die Einzelergebnisse aller Wirbel sind in Tabelle 1 aufgelistet. Dabei wurde für jede definierte anatomische Region der RMS-, der durschnittliche und der maximale Fehler berechnet. Die Rechenzeit für die Segmentierung betrug ca. 6 Minuten. Die nachfolgende Registrierung benötigte etwa 3 Sekunden.
5 4 Diskussion Zur klinischen Umsetzung computerunterstützter Verfahren ist es wünschenswert die Bearbeitungsschritte so weit wie möglich zu automatisieren. Der in dieser Arbeit vorgestellte Prozess beinhaltet einen Teil der Schritte, die zur präoperativen Vorbereitung von CT-Daten für die intraoperative Registrierung mittels Ultraschall notwendig sind. So ist es gelungen, nach einer halbautomatischen Extraktion der Oberflächenpunkte, die sich im Ultraschall darstellen lassen, automatisch die Punkte zu segmentieren, die zu einem einzelnen Wirbel gehören. Anhand dieser Punktmenge konnten die CT-Daten einzelner Wirbel mit dreidimensionalen Ultraschalldaten registriert werden. Das Verfahren war sowohl auf Phantomdaten, als auch auf Patientendaten erfolgreich. Der Target Registration Error der Schraubeneintrittspunkte und der Pedikel lag bei maximal 0,6mm Abweichung deutlich unter 1 mm, was in etwa der geforderten Genauigkeit eines Navigationssystems bei der Pedikelverschraubung entspricht. Die Abweichungen von bis zu 1,34mm im Bereich der Wirbelkörpervorderkante kam in erster Linie durch eine Winkelabweichung zustande. Diese Abweichung erfolgte somit nicht in der für eine Wirbelköperperforation kritischen Richtung bei der Pedikelverschraubung. Danksagung: Diese Arbeit ist im Rahmen des OrthoMIT-Projekts entstanden und wurde gefördert vom BMBF (Az. 01EQ0424). Literaturverzeichnis 1. Maintz, J.B.A., Viergever, M.A.: A survey of medical image registration. Medical Image Analysis 2 (1998) Langlotz, F., Nolte, L.P.: Technical approaches to computer assisted orthopaedic surgery. European Journal of Trauma 30 (2004) Winter, S., Brendel, B., et al.: Registration of CT and intraoperative 3D ultrasound images of the spine using evolutionary and gradient-based methods. IEEE Transactions on Evolutionary Computation (2007) (In Press). 4. Brendel, B., Winter, S., et al.: Registration of 3D CT- and ultrasound-datasets of the spine using bone structures. Computer Aided Surgery 7 (2002) Winter, S., Dekomien, C., et al.: Registrierung von intraoperativem 3D-Ultraschall mit präoperativen MRT-Daten für die computergestützte orthopädische Chirurgie. Zeitschrift für Orthopädie und Unfallchirurgie 145 (2007) Tsai, A., Yezzi, A., et al.: A shape-based approach to the segmentation of medical imagery using level sets. IEEE Transactions on Medical Images 22 (2003) Benameur, S., Mignotte, M., et al.: 3D/2D registration and segmentation of scoliotic vertebrae using statistical models. Computerized Medical Imaging and Graphics 27 (2003) Dekomien, C., Winter, S.: Segmentierung einzelner Wirbel in dreidimensionalen CT-Daten mit einem modelbasierten Level Set Ansatz. Biomedizinische Technik 52 (2007) 9. Hansen, N., Ostermeier., A.: Completely derandomized self-adaptation in evolution strategies. Evolutionary Computation 9 (2001)
Segmentierung der Knochenoberfläche einzelner Lendenwirbel für die ultraschallbasierte Navigation
 Segmentierung der Knochenoberfläche einzelner Lendenwirbel für die ultraschallbasierte Navigation Claudia Dekomien, Susanne Winter Institut für Neuroinformatik, Ruhr-Universität Bochum, 44780 Bochum Claudia.Dekomien@neuroinformatik.rub.de
Segmentierung der Knochenoberfläche einzelner Lendenwirbel für die ultraschallbasierte Navigation Claudia Dekomien, Susanne Winter Institut für Neuroinformatik, Ruhr-Universität Bochum, 44780 Bochum Claudia.Dekomien@neuroinformatik.rub.de
Segmentierung des Femurs aus MRT-Daten mit einem Shape-based Level-Set-Algorithmus
 Segmentierung des Femurs aus MRT-Daten mit einem Shape-based Level-Set-Algorithmus Claudia Dekomien 1, Martin Busch 2,Wolfram Teske 3, Susanne Winter 1 1 Institut für Neuroinformatik, Ruhr-Universität
Segmentierung des Femurs aus MRT-Daten mit einem Shape-based Level-Set-Algorithmus Claudia Dekomien 1, Martin Busch 2,Wolfram Teske 3, Susanne Winter 1 1 Institut für Neuroinformatik, Ruhr-Universität
Mathematische Symbole
 Abkürzungen Mathematische Symbole VII IX 1 Einleitung 1 1.1 Applikationen................................ 2 1.1.1 Pedikelverschraubung........................ 2 1.1.2 Kreuzbandersatz..........................
Abkürzungen Mathematische Symbole VII IX 1 Einleitung 1 1.1 Applikationen................................ 2 1.1.1 Pedikelverschraubung........................ 2 1.1.2 Kreuzbandersatz..........................
Kapitel 9. Zusammenfassung
 Kapitel 9 Zusammenfassung Medizinische Navigationssysteme verbessern die räumliche Orientierung von Chirurgen während Operationen. Eine Vielzahl von Navigationssystemen basieren auf der Registrierung von
Kapitel 9 Zusammenfassung Medizinische Navigationssysteme verbessern die räumliche Orientierung von Chirurgen während Operationen. Eine Vielzahl von Navigationssystemen basieren auf der Registrierung von
Parametrisierung evolutionärer Strategien für die Registrierung von Wirbelknochen in Ultraschall und CT-Daten
 Parametrisierung evolutionärer Strategien für die Registrierung von Wirbelknochen in Ultraschall und CT-Daten Susanne Winter 1, Bernhard Brendel 2, Bernd Illerhaus 3, Amir Al-Amin 3, Helmut Ermert 2 und
Parametrisierung evolutionärer Strategien für die Registrierung von Wirbelknochen in Ultraschall und CT-Daten Susanne Winter 1, Bernhard Brendel 2, Bernd Illerhaus 3, Amir Al-Amin 3, Helmut Ermert 2 und
Segmentierung des Femurs aus MRT-Daten mit Shape-Based Level-Sets
 Segmentierung des Femurs aus MRT-Daten mit Shape-Based Level-Sets Claudia Dekomien 1, Martin Busch 2, Wolfram Teske 3, Susanne Winter 1 1 Institut für Neuroinformatik, Ruhr-Universität Bochum, 44780 Bochum,
Segmentierung des Femurs aus MRT-Daten mit Shape-Based Level-Sets Claudia Dekomien 1, Martin Busch 2, Wolfram Teske 3, Susanne Winter 1 1 Institut für Neuroinformatik, Ruhr-Universität Bochum, 44780 Bochum,
Registrierung von Knochen in 3D-Ultraschallund CT-Daten: Vergleich verschiedener Optimierungsverfahren
 Registrierung von Knochen in 3D-Ultraschallund CT-Daten: Vergleich verschiedener Optimierungsverfahren Susanne Winter 1, Bernhard Brendel 2, Christian Igel 1 1 Institut für Neuroinformatik, Ruhr-Universität
Registrierung von Knochen in 3D-Ultraschallund CT-Daten: Vergleich verschiedener Optimierungsverfahren Susanne Winter 1, Bernhard Brendel 2, Christian Igel 1 1 Institut für Neuroinformatik, Ruhr-Universität
Koautoren: Bernhard Brendel, Andreas Rick, Martin Stockheim, Kirsten Schmieder, Helmut Ermert
 Autor: Susanne Winter Koautoren: Bernhard Brendel, Andreas Rick, Martin Stockheim, Kirsten Schmieder, Helmut Ermert Institute: Institut für Neuroinformatik, Institut für Hochfrequenztechnik, ZN Vision
Autor: Susanne Winter Koautoren: Bernhard Brendel, Andreas Rick, Martin Stockheim, Kirsten Schmieder, Helmut Ermert Institute: Institut für Neuroinformatik, Institut für Hochfrequenztechnik, ZN Vision
Einführung in die medizinische Bildverarbeitung WS 12/13
 Einführung in die medizinische Bildverarbeitung WS 12/13 Stephan Gimbel Kurze Wiederholung Prozess einer räumliche Transformation zu finden um ein Bild auf ein anderes Bild abzubilden Eingabe: Fixed und
Einführung in die medizinische Bildverarbeitung WS 12/13 Stephan Gimbel Kurze Wiederholung Prozess einer räumliche Transformation zu finden um ein Bild auf ein anderes Bild abzubilden Eingabe: Fixed und
Registrierung von 3D-Ultraschall mit MRT- und CT-Daten für die navigierte Chirurgie
 Registrierung von 3D-Ultraschall mit MRT- und CT-Daten für die navigierte Chirurgie Dissertation zur Erlangung des Grades eines Doktor-Ingenieurs der Fakultät für Elektrotechnik und Informationstechnik
Registrierung von 3D-Ultraschall mit MRT- und CT-Daten für die navigierte Chirurgie Dissertation zur Erlangung des Grades eines Doktor-Ingenieurs der Fakultät für Elektrotechnik und Informationstechnik
Vergleich von CT- mit C-Bogen-Segmentierungen für eine navigiert kontrollierte Fräse in der Neurochirurgie
 Vergleich von CT- mit C-Bogen-Segmentierungen für eine navigiert kontrollierte Fräse in der Neurochirurgie M. Dengl 1, R. Grunert 2, C. Trantakis 1,2, W. Korb 2, E. Jank 3, J. Krüger 3, T. Lueth 4, J.
Vergleich von CT- mit C-Bogen-Segmentierungen für eine navigiert kontrollierte Fräse in der Neurochirurgie M. Dengl 1, R. Grunert 2, C. Trantakis 1,2, W. Korb 2, E. Jank 3, J. Krüger 3, T. Lueth 4, J.
Automatic segmentation for dental operation planning. Diplomarbeit. Nguyen The Duy
 Automatic segmentation for dental operation planning Diplomarbeit Nguyen The Duy 24.02.2012 Motivation Quelle: bestbudapestdentist.com Aufgabenstellung Segmentierung des Oberkiefers (Maxilla) Detektion
Automatic segmentation for dental operation planning Diplomarbeit Nguyen The Duy 24.02.2012 Motivation Quelle: bestbudapestdentist.com Aufgabenstellung Segmentierung des Oberkiefers (Maxilla) Detektion
Optimization - Gradient- & Simplex- Algorithms
 Optimization - Gradient- & Simplex- Algorithms a.o. Univ.-Prof.DDr. Kurt Alexander Schicho, Dr. Michael Figl Dec 2010 Topics Optimization in Computer Assisted Surgery (CAS) Fields of applications Technical
Optimization - Gradient- & Simplex- Algorithms a.o. Univ.-Prof.DDr. Kurt Alexander Schicho, Dr. Michael Figl Dec 2010 Topics Optimization in Computer Assisted Surgery (CAS) Fields of applications Technical
Vollautomatische Segmentierung der Prostata aus 3D-Ultraschallbildern
 Vollautomatische Segmentierung der Prostata aus 3D-Ultraschallbildern Tobias Heimann, Tobias Simpfendörfer, Matthias Baumhauer, Hans-Peter Meinzer Abteilung für Medizinische und Biologische Informatik,
Vollautomatische Segmentierung der Prostata aus 3D-Ultraschallbildern Tobias Heimann, Tobias Simpfendörfer, Matthias Baumhauer, Hans-Peter Meinzer Abteilung für Medizinische und Biologische Informatik,
Bildregistrierung in der Medizin. Fethi KAYA - Yusuf ÖZBEK
 Bildregistrierung in der Medizin Fethi KAYA - Yusuf ÖZBEK 30.05.2011 1 Inhalt 1. Einführung zur Registrierung 2. Registrierungsalgorithmen 3. Transformationen 4. Mutual Information 5. Demo 2 1- Registrierung
Bildregistrierung in der Medizin Fethi KAYA - Yusuf ÖZBEK 30.05.2011 1 Inhalt 1. Einführung zur Registrierung 2. Registrierungsalgorithmen 3. Transformationen 4. Mutual Information 5. Demo 2 1- Registrierung
Erzeugung statistischer 3D-Formmodelle zur Segmentierung medizinischer Bilddaten
 Erzeugung statistischer 3D-Formmodelle zur Segmentierung medizinischer Bilddaten Hans Lamecker 1, Thomas Lange 2 und Martin Seebaß 1 1 Zuse Institut Berlin (ZIB), 14195 Berlin 2 Robert Rössle Klinik, Charité,
Erzeugung statistischer 3D-Formmodelle zur Segmentierung medizinischer Bilddaten Hans Lamecker 1, Thomas Lange 2 und Martin Seebaß 1 1 Zuse Institut Berlin (ZIB), 14195 Berlin 2 Robert Rössle Klinik, Charité,
Flächen- und Volumenmessung lokaler Objekte in DICOM-Bildern und Bildfolgen
 Flächen- und Volumenmessung lokaler Objekte in ICOM-Bildern und Bildfolgen Sergei Hludov, Christoph Meinel und Thomas Engel Institut für Telematik Bahnhofsstr. 30-3, 549 Trier Email: hludov@ti.fhg.de Zusammenfassung.
Flächen- und Volumenmessung lokaler Objekte in ICOM-Bildern und Bildfolgen Sergei Hludov, Christoph Meinel und Thomas Engel Institut für Telematik Bahnhofsstr. 30-3, 549 Trier Email: hludov@ti.fhg.de Zusammenfassung.
Ultraschallbasierte Navigation
 Ultraschallbasierte Navigation für die minimalinvasive onkologische Nieren- und Leberchirurgie vom Fachbereich Informatik der Technischen Universität Darmstadt genehmigte DISSERTATION zur Erlangung des
Ultraschallbasierte Navigation für die minimalinvasive onkologische Nieren- und Leberchirurgie vom Fachbereich Informatik der Technischen Universität Darmstadt genehmigte DISSERTATION zur Erlangung des
Automatische Segmentierung der Gewebegrenzen in 2D- Ultraschalldaten aus der Mund-, Kiefer- und Gesichtschirurgie
 Automatische Segmentierung der Gewebegrenzen in 2D- Ultraschalldaten aus der Mund-, Kiefer- und Gesichtschirurgie S. Göb 3, T. Maier 1, M. Benz 1, S. Lowitzsch 1, T. Wittenberg 2, W. Kullmann 3, E. Nkenke
Automatische Segmentierung der Gewebegrenzen in 2D- Ultraschalldaten aus der Mund-, Kiefer- und Gesichtschirurgie S. Göb 3, T. Maier 1, M. Benz 1, S. Lowitzsch 1, T. Wittenberg 2, W. Kullmann 3, E. Nkenke
Konsistente Parametrisierung von Flächen vom Geschlecht 1 zur Bildung eines statistischen Formmodells des Wirbels
 Konsistente Parametrisierung von Flächen vom Geschlecht 1 zur Bildung eines statistischen Formmodells des Wirbels Meike Becker 1, Matthias Kirschner 1, Stefan Wesarg 1 1 Graphisch-Interaktive Systeme,
Konsistente Parametrisierung von Flächen vom Geschlecht 1 zur Bildung eines statistischen Formmodells des Wirbels Meike Becker 1, Matthias Kirschner 1, Stefan Wesarg 1 1 Graphisch-Interaktive Systeme,
Graphbasierte Registrierung von Tubulären Strukturen
 Graphbasierte Registrierung von Tubulären Strukturen Thiago R. dos Santos, Ingmar Gergel, Sven Mersmann, Hans-Peter Meinzer, Lena Maier-Hein Abteilung für Medizinische und Biologische Informatik, DKFZ,
Graphbasierte Registrierung von Tubulären Strukturen Thiago R. dos Santos, Ingmar Gergel, Sven Mersmann, Hans-Peter Meinzer, Lena Maier-Hein Abteilung für Medizinische und Biologische Informatik, DKFZ,
Atlasbasierte 3D-Segmentierung medizinischer Bilddaten mit Fast-Marching-Methoden
 Atlasbasierte 3D-Segmentierung medizinischer Bilddaten mit Fast-Marching-Methoden Michael Schwenke 1,2, Matthias Färber 1, Jan Ehrhardt 1, Andreas Plaß 2, Heinz Handels 1 1 Institut für Medizinische Informatik,
Atlasbasierte 3D-Segmentierung medizinischer Bilddaten mit Fast-Marching-Methoden Michael Schwenke 1,2, Matthias Färber 1, Jan Ehrhardt 1, Andreas Plaß 2, Heinz Handels 1 1 Institut für Medizinische Informatik,
Roboterassistierte Reponierung von Oberschenkelfrakturen
 Roboterassistierte Reponierung von Oberschenkelfrakturen Ralf Westphal 1, Simon Winkelbach 1, Friedrich Wahl 1, Markus Oszwald 2, Thomas Gösling 2, Tobias Hüfner 2, Christian Krettek 2, 1 2 Inhalt Einleitung
Roboterassistierte Reponierung von Oberschenkelfrakturen Ralf Westphal 1, Simon Winkelbach 1, Friedrich Wahl 1, Markus Oszwald 2, Thomas Gösling 2, Tobias Hüfner 2, Christian Krettek 2, 1 2 Inhalt Einleitung
Computerunterstützte Prothesenkonstruktion mittels statistischen Formmodells bei Beckenresektionen
 Computerunterstützte Prothesenkonstruktion mittels statistischen Formmodells bei Beckenresektionen Thomas Lange 1, Per-Ulf Tunn 1, Hans Lamecker 2, Peter Scheinemann 3, Sebastian Eulenstein 1 und Peter-Michael
Computerunterstützte Prothesenkonstruktion mittels statistischen Formmodells bei Beckenresektionen Thomas Lange 1, Per-Ulf Tunn 1, Hans Lamecker 2, Peter Scheinemann 3, Sebastian Eulenstein 1 und Peter-Michael
Bildgestützte Quantifizierung von Aneurysmareperfusion zur Beurteilung des Behandlungserfolges von Coil-Therapien
 Bildgestützte Quantifizierung von Aneurysmareperfusion zur Beurteilung des Behandlungserfolges von Coil-Therapien Nils Daniel Forkert 1, Thorsten Ries 2, Jens Fiehler 2, Karl Wegscheider 3, Heinz Handels
Bildgestützte Quantifizierung von Aneurysmareperfusion zur Beurteilung des Behandlungserfolges von Coil-Therapien Nils Daniel Forkert 1, Thorsten Ries 2, Jens Fiehler 2, Karl Wegscheider 3, Heinz Handels
Automatische Initialisierung von Formmodellen mittels modellbasierter Registrierung
 Automatische Initialisierung von Formmodellen mittels modellbasierter Registrierung Matthias Kirschner, Stefan Wesarg Graphisch-Interaktive Systeme, TU Darmstadt matthias.kirschner@gris.tu-darmstadt.de
Automatische Initialisierung von Formmodellen mittels modellbasierter Registrierung Matthias Kirschner, Stefan Wesarg Graphisch-Interaktive Systeme, TU Darmstadt matthias.kirschner@gris.tu-darmstadt.de
Atlas-basierte 3D-Rekonstruktion des Beckens aus 2D-Projektionsbildern
 Atlas-basierte 3D-Rekonstruktion des Beckens aus 2D-Projektionsbildern Hans Lamecker 1, Thomas H. Wenckebach 1, Hans-Christian Hege 1, Georg N. Duda 2 und Markus O. Heller 2 1 Visualisierung und Datenanalyse,
Atlas-basierte 3D-Rekonstruktion des Beckens aus 2D-Projektionsbildern Hans Lamecker 1, Thomas H. Wenckebach 1, Hans-Christian Hege 1, Georg N. Duda 2 und Markus O. Heller 2 1 Visualisierung und Datenanalyse,
Segmentierung des Aterienbaums
 Segmentierung des Aterienbaums Christoph Schaefer Seminar: Bildverarbeitung für die Medizin Universität Koblenz-Landau 27.01.2007 1 Anwendungsgebiete 2 Segmentierung Überblick Deformierbare Modelle Snake
Segmentierung des Aterienbaums Christoph Schaefer Seminar: Bildverarbeitung für die Medizin Universität Koblenz-Landau 27.01.2007 1 Anwendungsgebiete 2 Segmentierung Überblick Deformierbare Modelle Snake
Ein Softwarepaket für die modellbasierte Segmentierung anatomischer Strukturen
 Ein Softwarepaket für die modellbasierte Segmentierung anatomischer Strukturen Thomas Lange 1, Hans Lamecker 2 und Martin Seebaß 2 1 Robert Rössle Klinik, Charité, 13125 Berlin 2 Zuse Institut Berlin (ZIB),
Ein Softwarepaket für die modellbasierte Segmentierung anatomischer Strukturen Thomas Lange 1, Hans Lamecker 2 und Martin Seebaß 2 1 Robert Rössle Klinik, Charité, 13125 Berlin 2 Zuse Institut Berlin (ZIB),
IGDT: Image Processing Advanced Teil 9
 Teil 9 Rainer Schubert Institut für Biomedizinische Bildanalyse Inhalt Teil 9 Registrierung Grauwert-basierte / Intensity-based Methoden Klassifikation von Registrierungsmethoden 1 Viele Klassifikationskriterien
Teil 9 Rainer Schubert Institut für Biomedizinische Bildanalyse Inhalt Teil 9 Registrierung Grauwert-basierte / Intensity-based Methoden Klassifikation von Registrierungsmethoden 1 Viele Klassifikationskriterien
DEUTSCHE GESELLSCHAFT FÜR KARDIOLOGIE HERZ- UND KREISLAUFFORSCHUNG e.v. German Cardiac Society
 Vergleich zwischen Flächen- und Perimeter-basierter Bestimmung der effektiven Anulusgröße mit der direkten intraoperativen Messung Dr. Won-Keun Kim, Bad Nauheim Einleitung: Die präzise Messung des Aortenanulus
Vergleich zwischen Flächen- und Perimeter-basierter Bestimmung der effektiven Anulusgröße mit der direkten intraoperativen Messung Dr. Won-Keun Kim, Bad Nauheim Einleitung: Die präzise Messung des Aortenanulus
Segmentierung des Knochens aus T1- und PD-gewichteten Kernspinbildern vom Kopf
 Segmentierung des Knochens aus T1- und PD-gewichteten Kernspinbildern vom Kopf Stefan Burkhardt, Dietmar Saupe, Frithjof Kruggel und Carsten Wolters Institut für Informatik Universität Leipzig, 04109 Leipzig
Segmentierung des Knochens aus T1- und PD-gewichteten Kernspinbildern vom Kopf Stefan Burkhardt, Dietmar Saupe, Frithjof Kruggel und Carsten Wolters Institut für Informatik Universität Leipzig, 04109 Leipzig
Simulation der Knochenverlagerung beim Frontal Orbital Advancement
 Simulation der Knochenverlagerung beim Frontal Orbital Advancement H. Grabowski*, S. Hassfeld**, J. Brief**, J. Münchenberg*, U. Rembold*, H. Wörn* *Institut für Prozeßrechentechnik, Automation und Robotik
Simulation der Knochenverlagerung beim Frontal Orbital Advancement H. Grabowski*, S. Hassfeld**, J. Brief**, J. Münchenberg*, U. Rembold*, H. Wörn* *Institut für Prozeßrechentechnik, Automation und Robotik
Tracking und Segmentierung baumförmiger, tubulärer Strukturen mit einem hybriden Verfahren
 Tracking und Segmentierung baumförmiger, tubulärer Strukturen mit einem hybriden Verfahren Philipp Wolber 1, Ingmar Wegner 1, Tobias Heimann 1, Michael Puderbach 2, Ivo Wolf 1, Hans-Peter Meinzer 1 1 Abt.
Tracking und Segmentierung baumförmiger, tubulärer Strukturen mit einem hybriden Verfahren Philipp Wolber 1, Ingmar Wegner 1, Tobias Heimann 1, Michael Puderbach 2, Ivo Wolf 1, Hans-Peter Meinzer 1 1 Abt.
4D-MRT-basierte Simulation der Lungenbewegung in statischen CT-Daten
 4D-MRT-basierte Simulation der Lungenbewegung in statischen CT-Daten Mirko Marx 1, Jan Ehrhardt 1, René Werner 1, Heinz-Peter Schlemmer 2, Heinz Handels 1 1 Institut für Medizinische Informatik, Universität
4D-MRT-basierte Simulation der Lungenbewegung in statischen CT-Daten Mirko Marx 1, Jan Ehrhardt 1, René Werner 1, Heinz-Peter Schlemmer 2, Heinz Handels 1 1 Institut für Medizinische Informatik, Universität
Erstellung eines 3D-Atlas ausgewählter Strukturen durch affine Transformation anatomischer Landmarken
 Kurzfassung der Masterthesis Erstellung eines 3D-Atlas ausgewählter Strukturen durch affine Transformation anatomischer Landmarken M.Sc. Soulimane Abdellaoui 1 Einleitung : In der vorliegenden wird ein
Kurzfassung der Masterthesis Erstellung eines 3D-Atlas ausgewählter Strukturen durch affine Transformation anatomischer Landmarken M.Sc. Soulimane Abdellaoui 1 Einleitung : In der vorliegenden wird ein
Robuste Verzweigungserkennung von Gefäßen in CTA-Datensätzen zur modellbasierten Extraktion der Centerline
 Robuste Verzweigungserkennung von Gefäßen in CTA-Datensätzen zur modellbasierten Extraktion der Centerline Thomas Beck 1,2, Dominik Fritz 2, Christina Biermann 2,3, Rüdiger Dillmann 1 1 Institut für Technische
Robuste Verzweigungserkennung von Gefäßen in CTA-Datensätzen zur modellbasierten Extraktion der Centerline Thomas Beck 1,2, Dominik Fritz 2, Christina Biermann 2,3, Rüdiger Dillmann 1 1 Institut für Technische
Klassifikation von Standardebenen in der 2D-Echokardiographie mittels 2D-3D-Bildregistrierung
 Klassifikation von Standardebenen in der 2D-Echokardiographie mittels 2D-3D-Bildregistrierung Christoph Bergmeir 1, Navneeth Subramanian 1 1 GE Global Research, Bangalore, India navneeth.s@ge.com Kurzfassung.
Klassifikation von Standardebenen in der 2D-Echokardiographie mittels 2D-3D-Bildregistrierung Christoph Bergmeir 1, Navneeth Subramanian 1 1 GE Global Research, Bangalore, India navneeth.s@ge.com Kurzfassung.
Automatische Detektion von Lymphknoten in CT-Datensätzen des Halses
 Automatische Detektion von Lymphknoten in CT-Datensätzen des Halses Lars Dornheim, Jana Dornheim Institut für Simulation und Graphik, Otto-von-Guericke-Universität Magdeburg lars@isg.cs.uni-magdeburg.de
Automatische Detektion von Lymphknoten in CT-Datensätzen des Halses Lars Dornheim, Jana Dornheim Institut für Simulation und Graphik, Otto-von-Guericke-Universität Magdeburg lars@isg.cs.uni-magdeburg.de
Seminar Ausgewählte Themen der medizinischen Visualisierung
 1 Ausgewählte Themen der medizinischen Visualisierung Institut für Informatik 08.07.2011 Arbeitsgruppe Visualisierung und Computergrafik (VisCG) Organisatorisches Veranstalter: Prof. Dr. Klaus Hinrichs,
1 Ausgewählte Themen der medizinischen Visualisierung Institut für Informatik 08.07.2011 Arbeitsgruppe Visualisierung und Computergrafik (VisCG) Organisatorisches Veranstalter: Prof. Dr. Klaus Hinrichs,
Modellfunktion zur Approximation von Ultraschallkontrastmittelkonzentration zur semi-quantitativen Gewebeperfusionsbestimmung
 Modellfunktion zur Approximation von Ultraschallkontrastmittelkonzentration zur semi-quantitativen Gewebeperfusionsbestimmung Kai Ritschel, Claudia Dekomien, Susanne Winter Institut für Neuroinformatik,
Modellfunktion zur Approximation von Ultraschallkontrastmittelkonzentration zur semi-quantitativen Gewebeperfusionsbestimmung Kai Ritschel, Claudia Dekomien, Susanne Winter Institut für Neuroinformatik,
Automatische Segmentierung der zerebralen Gefäße aus 3D-TOF-MRA-Bildsequenzen mittels Fuzzy-Methoden
 Automatische Segmentierung der zerebralen Gefäße aus 3D-TOF-MRA-Bildsequenzen mittels Fuzzy-Methoden Nils Daniel Forkert 1, Dennis Säring 1, Karolin Wenzel 2, Jens Fiehler 2, Till Illies 2, Dietmar Möller
Automatische Segmentierung der zerebralen Gefäße aus 3D-TOF-MRA-Bildsequenzen mittels Fuzzy-Methoden Nils Daniel Forkert 1, Dennis Säring 1, Karolin Wenzel 2, Jens Fiehler 2, Till Illies 2, Dietmar Möller
Automatische Segmentierung der Lungenflügel in CT-Daten
 Automatische Segmentierung der Lungenflügel in CT-Daten Matthias Wilms, Jan Ehrhardt, Heinz Handels Institut für Medizinische Informatik, Universität zu Lübeck matthias.wilms@gmail.com Kurzfassung. In
Automatische Segmentierung der Lungenflügel in CT-Daten Matthias Wilms, Jan Ehrhardt, Heinz Handels Institut für Medizinische Informatik, Universität zu Lübeck matthias.wilms@gmail.com Kurzfassung. In
Jan Putzer, Michael Teistler, Jochen Dormeier, Lars Mieth*, Tim Pohlemann*
 Computergestützte Segmentierung des frakturierten Acetabulums in CT-Aufnahmen mit Hilfe aktiver Konturen zur Klassifikation und Operationsplanung in der Unfallchirurgie Jan Putzer, Michael Teistler, Jochen
Computergestützte Segmentierung des frakturierten Acetabulums in CT-Aufnahmen mit Hilfe aktiver Konturen zur Klassifikation und Operationsplanung in der Unfallchirurgie Jan Putzer, Michael Teistler, Jochen
3D-Rekonstruktion aus Bildern
 Allgemeine Nachrichtentechnik Prof. Dr.-Ing. Udo Zölzer 3D-Rekonstruktion aus Bildern Dipl.-Ing. Christian Ruwwe 41. Treffen der ITG-Fachgruppe Algorithmen für die Signalverarbeitung HSU Hamburg 24. Februar
Allgemeine Nachrichtentechnik Prof. Dr.-Ing. Udo Zölzer 3D-Rekonstruktion aus Bildern Dipl.-Ing. Christian Ruwwe 41. Treffen der ITG-Fachgruppe Algorithmen für die Signalverarbeitung HSU Hamburg 24. Februar
Automatische Generierung dynamischer 3D-Modelle zur Segmentierung des linken Ventrikels in 3D-SPECT-Daten
 Automatische Generierung dynamischer 3D-Modelle zur Segmentierung des linken Ventrikels in 3D-SPECT-Daten Lars Dornheim 1 und Klaus D. Tönnies 1 1 Institut für Simulation und Graphik, Otto-von-Guericke-Universität
Automatische Generierung dynamischer 3D-Modelle zur Segmentierung des linken Ventrikels in 3D-SPECT-Daten Lars Dornheim 1 und Klaus D. Tönnies 1 1 Institut für Simulation und Graphik, Otto-von-Guericke-Universität
Aktive Sensoren: Kontextbasierte Filterung von Merkmalen zur modellbasierten Segmentierung
 Aktive Sensoren: Kontextbasierte Filterung von Merkmalen zur modellbasierten Segmentierung Lars Dornheim, Jana Dornheim, Heiko Seim, Klaus D. Tönnies 20.3.2006 Otto-von-Guericke-Universität Magdeburg,
Aktive Sensoren: Kontextbasierte Filterung von Merkmalen zur modellbasierten Segmentierung Lars Dornheim, Jana Dornheim, Heiko Seim, Klaus D. Tönnies 20.3.2006 Otto-von-Guericke-Universität Magdeburg,
Semi-automatische Segmentierung der Prostata mit Hilfe von 3D Ultraschallaufnahmen
 Semi-automatische Segmentierung der Prostata mit Hilfe von 3D Ultraschallaufnahmen Evelyn Firle Fraunhofer Institut Graphische Datenverarbeitung Rundeturmstr. 6, 64283 Darmstadt Email: efirle@igd.fhg.de
Semi-automatische Segmentierung der Prostata mit Hilfe von 3D Ultraschallaufnahmen Evelyn Firle Fraunhofer Institut Graphische Datenverarbeitung Rundeturmstr. 6, 64283 Darmstadt Email: efirle@igd.fhg.de
Formbasierte Segmentierung des Bronchialbaumes
 Formbasierte Segmentierung des Bronchialbaumes Florian Jäger 1, Joachim Hornegger 1, Eckhart Georg Hahn 2 1 Lehrstuhl für Mustererkennung, Friedrich-Alexander-Universität Erlangen-Nürnberg, {jaeger,hornegger}@informatik.uni-erlangen.de.
Formbasierte Segmentierung des Bronchialbaumes Florian Jäger 1, Joachim Hornegger 1, Eckhart Georg Hahn 2 1 Lehrstuhl für Mustererkennung, Friedrich-Alexander-Universität Erlangen-Nürnberg, {jaeger,hornegger}@informatik.uni-erlangen.de.
Variationeller Ansatz für eine integrierte Segmentierung und nicht-lineare Registrierung
 Variationeller Ansatz für eine integrierte Segmentierung und nicht-lineare Registrierung Alexander Schmidt-Richberg 1,2, Jan Ehrhardt 1, Heinz Handels 1 1 Institut für Medizinische Informatik, Universitätsklinikum
Variationeller Ansatz für eine integrierte Segmentierung und nicht-lineare Registrierung Alexander Schmidt-Richberg 1,2, Jan Ehrhardt 1, Heinz Handels 1 1 Institut für Medizinische Informatik, Universitätsklinikum
Simulation mammographischer Brustkompression zur Generierung von MRT-Projektionsbildern
 Simulation mammographischer Brustkompression zur Generierung von MRT-Projektionsbildern Julia Krüger 1, Jan Ehrhardt 1, Arpad Bischof 2, Heinz Handels 1 1 Institut für Medizinische Informatik, Universität
Simulation mammographischer Brustkompression zur Generierung von MRT-Projektionsbildern Julia Krüger 1, Jan Ehrhardt 1, Arpad Bischof 2, Heinz Handels 1 1 Institut für Medizinische Informatik, Universität
Volumenverarbeitung und Optimierung II
 Volumenverarbeitung und Optimierung II Praktikum Medizinische GPU Verfahren Susanne Fischer sanne@uni-koblenz.de Institut für Computervisualistik Universität Koblenz-Landau 9. Dezember 2006 Susanne Fischer
Volumenverarbeitung und Optimierung II Praktikum Medizinische GPU Verfahren Susanne Fischer sanne@uni-koblenz.de Institut für Computervisualistik Universität Koblenz-Landau 9. Dezember 2006 Susanne Fischer
Halbautomatische Segmentierung von Pulmonalgefäßen in CT Daten als Referenz zur Validierung automatischer Verfahren
 Halbautomatische Segmentierung von Pulmonalgefäßen in CT Daten als Referenz zur Validierung automatischer Verfahren Jens N. Kaftan 1,2, Annemarie Bakai 2, Florian Maier 3, Til Aach 1 1 Lehrstuhl für Bildverarbeitung,
Halbautomatische Segmentierung von Pulmonalgefäßen in CT Daten als Referenz zur Validierung automatischer Verfahren Jens N. Kaftan 1,2, Annemarie Bakai 2, Florian Maier 3, Til Aach 1 1 Lehrstuhl für Bildverarbeitung,
Praktikum Mathe und Medizin
 Segmentierung von Tumoren und der rechten Herzwand in CT Daten a.gripshoefer@gmx.de, SeWesti@gmx.de 08.07.2008 1 Problemstellung 2 3 4 Problemstellung Aufgrund des Partialvolumeneekts sind PET Aufnahmen
Segmentierung von Tumoren und der rechten Herzwand in CT Daten a.gripshoefer@gmx.de, SeWesti@gmx.de 08.07.2008 1 Problemstellung 2 3 4 Problemstellung Aufgrund des Partialvolumeneekts sind PET Aufnahmen
Marching Cubes - Erstellung von Polygonmodellen aus Voxelgittern
 Marching Cubes - Erstellung von Polygonmodellen aus Voxelgittern Matthias Kirschner Advanced Topics in Computer Graphics - WS06/07 Marching Cubes - Erstellung von Polygonmodellen aus Voxelgittern Matthias
Marching Cubes - Erstellung von Polygonmodellen aus Voxelgittern Matthias Kirschner Advanced Topics in Computer Graphics - WS06/07 Marching Cubes - Erstellung von Polygonmodellen aus Voxelgittern Matthias
Entwicklung von additiv gefertigten Phantomen für die Qualitätssicherung in der Strahlentherapie
 Entwicklung von additiv gefertigten Phantomen für die Qualitätssicherung in der Strahlentherapie 29. Mai 2018 In Kooperation mit der Klinik für Strahlentherapie und Radio-Onkologie Technische Universität
Entwicklung von additiv gefertigten Phantomen für die Qualitätssicherung in der Strahlentherapie 29. Mai 2018 In Kooperation mit der Klinik für Strahlentherapie und Radio-Onkologie Technische Universität
Tracking- und Navigationssysteme: Technik und Anwendungen. Dr.-Ing. Ralf Westphal,
 Tracking- und Navigationssysteme: Technik und Anwendungen Dr.-Ing. Ralf Westphal, 04.03.2013 Inhalt Chirurgische Navigation Trackingsysteme Optische Trackingsysteme Genauigkeit der Navigation Anwendungsbeispiele
Tracking- und Navigationssysteme: Technik und Anwendungen Dr.-Ing. Ralf Westphal, 04.03.2013 Inhalt Chirurgische Navigation Trackingsysteme Optische Trackingsysteme Genauigkeit der Navigation Anwendungsbeispiele
Beschleunigung von Bild-Segmentierungs- Algorithmen mit FPGAs
 Fakultät Informatik, Institut für Technische Informatik, Professur für VLSI-Entwurfssysteme, Diagnostik und Architektur Algorithmen mit FPGAs Vortrag von Jan Frenzel Dresden, Gliederung Was ist Bildsegmentierung?
Fakultät Informatik, Institut für Technische Informatik, Professur für VLSI-Entwurfssysteme, Diagnostik und Architektur Algorithmen mit FPGAs Vortrag von Jan Frenzel Dresden, Gliederung Was ist Bildsegmentierung?
Detektion von Lymphknoten in CT-Datensätzen des Halses
 Automatische Detektion von Lymphknoten in CT-Datensätzen des Halses Lars Dornheim, Jana Dornheim Institut für Simulation und Graphik, Otto-von-Guericke-Universität Magdeburg lars@isg.cs.uni-magdeburg.de
Automatische Detektion von Lymphknoten in CT-Datensätzen des Halses Lars Dornheim, Jana Dornheim Institut für Simulation und Graphik, Otto-von-Guericke-Universität Magdeburg lars@isg.cs.uni-magdeburg.de
Der Telemanipulator davinci als mechanisches Trackingsystem
 Der Telemanipulator davinci als mechanisches Trackingsystem Bestimmung von Präzision und Genauigkeit Johannes Kast 1, Jochen Neuhaus 1, Felix Nickel 2, Hannes Kenngott 2, Markus Engel 1, Elaine Short 1,
Der Telemanipulator davinci als mechanisches Trackingsystem Bestimmung von Präzision und Genauigkeit Johannes Kast 1, Jochen Neuhaus 1, Felix Nickel 2, Hannes Kenngott 2, Markus Engel 1, Elaine Short 1,
Wasserscheiden-Ansätze zur Bildsegmentierung I
 Seminar Bildsegmentierung und Computer Vision Wasserscheiden-Ansätze zur Bildsegmentierung I Stefan Sugg 19.12.2005 Gliederung 1. Einführung 2. Morphologische Grundlagen 3. Simulation durch Überflutung
Seminar Bildsegmentierung und Computer Vision Wasserscheiden-Ansätze zur Bildsegmentierung I Stefan Sugg 19.12.2005 Gliederung 1. Einführung 2. Morphologische Grundlagen 3. Simulation durch Überflutung
Messen auf Voxeldaten, Punktwolken, Polygonnetzen
 Messen auf Voxeldaten, Punktwolken, Polygonnetzen Martin Kronig, kronig@volumegraphics.com Volume Graphics GmbH Volume Graphics > Weltweit Anbieter für industrielle CT- Datenvisualisierung, -analyse und
Messen auf Voxeldaten, Punktwolken, Polygonnetzen Martin Kronig, kronig@volumegraphics.com Volume Graphics GmbH Volume Graphics > Weltweit Anbieter für industrielle CT- Datenvisualisierung, -analyse und
Erstellung eines anatomischen Templates zur Zielvolumendefinition der Zervixregion für die Bestrahlungsplanung
 Erstellung eines anatomischen Templates zur Zielvolumendefinition der Zervixregion für die Bestrahlungsplanung Detlef Richter 1, Soulimane Abdellaoui 1,2, Faisel Bekkaoui 1,2 Karsten Berthold 1,2 und Gerd
Erstellung eines anatomischen Templates zur Zielvolumendefinition der Zervixregion für die Bestrahlungsplanung Detlef Richter 1, Soulimane Abdellaoui 1,2, Faisel Bekkaoui 1,2 Karsten Berthold 1,2 und Gerd
Optimierung nicht-linearer Registrierung durch automatisch detektierte Landmarken
 Optimierung nicht-linearer Registrierung durch automatisch detektierte Landmarken Jan-Christoph Wolf, Alexander Schmidt-Richberg, Rene Werner, Jan Ehrhardt, Heinz Handels Institut für Medizinische Informatik,
Optimierung nicht-linearer Registrierung durch automatisch detektierte Landmarken Jan-Christoph Wolf, Alexander Schmidt-Richberg, Rene Werner, Jan Ehrhardt, Heinz Handels Institut für Medizinische Informatik,
Shape Gradient for Image and Video Segmentation
 Shape Gradient for Image and Video Segmentation [1] S. Jehan-Besson, A. Herbulot, M. Barlaud und G. Aubert 23. Mai 2007 Martin Schröttner und Michael Schneeberger Mat Vis-Gra SS07 1 Überblick Aktive Konturen
Shape Gradient for Image and Video Segmentation [1] S. Jehan-Besson, A. Herbulot, M. Barlaud und G. Aubert 23. Mai 2007 Martin Schröttner und Michael Schneeberger Mat Vis-Gra SS07 1 Überblick Aktive Konturen
Jürgen Salk Universitätsklinikum Ulm. Einsatzmöglichkeiten. Grundlagen der Virtuellen Simulation
 Grundlagen der Virtuellen Simulation Jürgen Salk Universitätsklinikum Ulm Was ist eine Virtuelle Simulation? Erzeugung Digital Rekonstruierter Röntgenbilder () Möglichkeiten zur Bildbearbeitung (Window-Level)
Grundlagen der Virtuellen Simulation Jürgen Salk Universitätsklinikum Ulm Was ist eine Virtuelle Simulation? Erzeugung Digital Rekonstruierter Röntgenbilder () Möglichkeiten zur Bildbearbeitung (Window-Level)
Detektion von Koronararterien: Das Beste aus zwei Welten
 Detektion von Koronararterien: Das Beste aus zwei Welten Andreas Grünauer 1, Sebastian Zambal 2, Katja Bühler 1 1 VRVis Zentrum für Virtual Reality und Visualisierung Forschungs-GmbH, Wien 2 Agfa Healthcare
Detektion von Koronararterien: Das Beste aus zwei Welten Andreas Grünauer 1, Sebastian Zambal 2, Katja Bühler 1 1 VRVis Zentrum für Virtual Reality und Visualisierung Forschungs-GmbH, Wien 2 Agfa Healthcare
Segmentierung von Aortenaneurysmen in CTA-Bildern mit dem statistischen Verfahren der Active Appearance Models
 Segmentierung von Aortenaneurysmen in CTA-Bildern mit dem statistischen Verfahren der Active Appearance Models Katharina Greiner 1,3, Jan Egger 2,3, Stefan Großkopf 3, Jens N. Kaftan 3,4, Ralf Dörner 1,
Segmentierung von Aortenaneurysmen in CTA-Bildern mit dem statistischen Verfahren der Active Appearance Models Katharina Greiner 1,3, Jan Egger 2,3, Stefan Großkopf 3, Jens N. Kaftan 3,4, Ralf Dörner 1,
4 Ergebnisbeschreibung 4.1 Patienten
 4 Ergebnisbeschreibung 4.1 Patienten In der Datenauswertung ergab sich für die Basischarakteristika der Patienten gesamt und bezogen auf die beiden Gruppen folgende Ergebnisse: 19 von 40 Patienten waren
4 Ergebnisbeschreibung 4.1 Patienten In der Datenauswertung ergab sich für die Basischarakteristika der Patienten gesamt und bezogen auf die beiden Gruppen folgende Ergebnisse: 19 von 40 Patienten waren
Halbautomatische Segmentierung von Pulmonalgef-aıen in CT Daten als Referenz zur Validierung automatischer Verfahren
 Lehrstuhl für Bildverarbeitung Institute of Imaging & Computer Vision Halbautomatische Segmentierung von Pulmonalgef-aıen in CT Daten als Referenz zur Validierung automatischer Verfahren Jens N. Kaftan
Lehrstuhl für Bildverarbeitung Institute of Imaging & Computer Vision Halbautomatische Segmentierung von Pulmonalgef-aıen in CT Daten als Referenz zur Validierung automatischer Verfahren Jens N. Kaftan
8.2 Nicht parametrische Tests Vergleich CT/2D/3D. Abb. 28 Mann-Whitney-U-Test
 41 8. Interpretationen der Studienergebnisse Im vorliegenden Kapitel werden die Studienergebnisse mit Hilfe des Mann-Whitney-U-Tests auf signifikante Unterschiede untersucht. Hierfür wurden die vorliegenden
41 8. Interpretationen der Studienergebnisse Im vorliegenden Kapitel werden die Studienergebnisse mit Hilfe des Mann-Whitney-U-Tests auf signifikante Unterschiede untersucht. Hierfür wurden die vorliegenden
Einführung in die medizinische Bildverarbeitung WS 12/13
 Einführung in die medizinische Bildverarbeitung WS 12/13 Stephan Gimbel Kurze Wiederholung Landmarkenbasierte anhand anatomischer Punkte interaktiv algorithmisch z.b. zur Navigation im OP Markierung von
Einführung in die medizinische Bildverarbeitung WS 12/13 Stephan Gimbel Kurze Wiederholung Landmarkenbasierte anhand anatomischer Punkte interaktiv algorithmisch z.b. zur Navigation im OP Markierung von
2 Die ILabMed-Workstation Die Basisplattform der ILabMed-Workstation wird seit 1993 am Centrum für Medizinische Diagnosesysteme und Visualisierung auf
 ILabMed-Workstation - Eine Entwicklungsumgebung für radiologische Anwendungen A. Schenk, J. Breitenborn, D. Selle, T. Schindewolf, D. Böhm, W. Spindler, H. Jürgens, H.-O. Peitgen MeVis - Centrum für Medizinische
ILabMed-Workstation - Eine Entwicklungsumgebung für radiologische Anwendungen A. Schenk, J. Breitenborn, D. Selle, T. Schindewolf, D. Böhm, W. Spindler, H. Jürgens, H.-O. Peitgen MeVis - Centrum für Medizinische
Modellbasierte Rekonstruktion von Organoberflächen auf der Basis von zweidimensionalen Schnittdaten
 Modellbasierte Rekonstruktion von Organoberflächen auf der Basis von zweidimensionalen Schnittdaten Sebastian von Klinski, Andreas Glausch, Claus Derz und Thomas Tolxdorff Institut für Medizinische Statistik,
Modellbasierte Rekonstruktion von Organoberflächen auf der Basis von zweidimensionalen Schnittdaten Sebastian von Klinski, Andreas Glausch, Claus Derz und Thomas Tolxdorff Institut für Medizinische Statistik,
Multimedia-Metadaten und ihre Anwendung
 Multimedia-Metadaten und ihre Anwendung 14.02.2006 Automatische Gesichtserkennung Dominikus Baur Gesichtserkennung in MPEG-7 MPEG-7 bietet Face Descriptor zur Beschreibung von Gesichtern Im Folgenden:
Multimedia-Metadaten und ihre Anwendung 14.02.2006 Automatische Gesichtserkennung Dominikus Baur Gesichtserkennung in MPEG-7 MPEG-7 bietet Face Descriptor zur Beschreibung von Gesichtern Im Folgenden:
Untersuchung von Registrierstrategien für die Anwendung des Navigationssystems FIAGON an der lateralen Schädelbasis
 Abstract: Untersuchung von Registrierstrategien für die Anwendung des Navigationssystems FIAGON an der lateralen Schädelbasis J. Kristin, D. Mucha, J. Schipper, T. Klenzner Universitätsklinikum Düsseldorf,
Abstract: Untersuchung von Registrierstrategien für die Anwendung des Navigationssystems FIAGON an der lateralen Schädelbasis J. Kristin, D. Mucha, J. Schipper, T. Klenzner Universitätsklinikum Düsseldorf,
MRT-basierte Perfusionsbildgebung in der Klinik und Forschung
 MRT-basierte Perfusionsbildgebung in der Klinik und Forschung Computer Assisted Clinical Medicine Medical Faculty Mannheim Heidelberg University Theodor-Kutzer-Ufer 1-3 www.umm.uni-heidelberg.de/inst/cbtm/ckm
MRT-basierte Perfusionsbildgebung in der Klinik und Forschung Computer Assisted Clinical Medicine Medical Faculty Mannheim Heidelberg University Theodor-Kutzer-Ufer 1-3 www.umm.uni-heidelberg.de/inst/cbtm/ckm
Segmentierung. Vorlesung FH-Hagenberg SEM
 Segmentierung Vorlesung FH-Hagenberg SEM Segmentierung: Definition Die Pixel eines Bildes A={a i }, i=1:n, mit N der Anzahl der Pixel, werden in Teilmengen S i unterteilt. Die Teilmengen sind disjunkt
Segmentierung Vorlesung FH-Hagenberg SEM Segmentierung: Definition Die Pixel eines Bildes A={a i }, i=1:n, mit N der Anzahl der Pixel, werden in Teilmengen S i unterteilt. Die Teilmengen sind disjunkt
CUDA Optimierung von nicht-linearer oberflächen- und intensitätsbasierter Registrierung
 CUDA Optimierung von nicht-linearer oberflächen- und intensitätsbasierter Registrierung Stefan Köhnen, Jan Ehrhardt, Alexander Schmidt-Richberg, Heinz Handels Institut für Medizinische Informatik, Universität
CUDA Optimierung von nicht-linearer oberflächen- und intensitätsbasierter Registrierung Stefan Köhnen, Jan Ehrhardt, Alexander Schmidt-Richberg, Heinz Handels Institut für Medizinische Informatik, Universität
Erweiterte Realität und 3-D Visualisierung für minimal-invasive Operationen durch Einsatz eines optischen Trackingsystems
 Erweiterte Realität und 3-D Visualisierung für minimal-invasive Operationen durch Einsatz eines optischen Trackingsystems F. Vogt 1, S. Krüger 2, M. Winter 3, H. Niemann 1, W. Hohenberger 2, G. Greiner
Erweiterte Realität und 3-D Visualisierung für minimal-invasive Operationen durch Einsatz eines optischen Trackingsystems F. Vogt 1, S. Krüger 2, M. Winter 3, H. Niemann 1, W. Hohenberger 2, G. Greiner
Multimodale Registrierung von Knochen-Szintigraphien und Röntgenbildern
 Multimodale Registrierung von Knochen-Szintigraphien und Röntgenbildern Nils Papenberg 1, Hanno Schumacher 2, Stefan Heldmann 1, Tobias Böhler 3, Dörte van Straaten 3, Stefan Wirtz 3 1 Fraunhofer MEVIS,
Multimodale Registrierung von Knochen-Szintigraphien und Röntgenbildern Nils Papenberg 1, Hanno Schumacher 2, Stefan Heldmann 1, Tobias Böhler 3, Dörte van Straaten 3, Stefan Wirtz 3 1 Fraunhofer MEVIS,
Dreidimensionales Computersehen SS 2016
 Dreidimensionales Computersehen SS 2016 Folien zur Vorlesung Dr.-Ing. Simon Winkelbach www.robotik-bs.de/teaching/courses/cs Dreidimensionales Computersehen Dr.-Ing. Simon Winkelbach http://www.robotik-bs.de/teaching/courses/cs
Dreidimensionales Computersehen SS 2016 Folien zur Vorlesung Dr.-Ing. Simon Winkelbach www.robotik-bs.de/teaching/courses/cs Dreidimensionales Computersehen Dr.-Ing. Simon Winkelbach http://www.robotik-bs.de/teaching/courses/cs
3D-Scans verbessern: MeshLab - Teil 2
 3D-Scans verbessern: MeshLab - Teil 2 Kategorien : Allgemein, Open Source Schlagwörter : 3D Scan, MeshLab Datum : 23. August 2017 Mit 3D Rekonstruktion wird das Erstellen von dreidimensionalen Modellen
3D-Scans verbessern: MeshLab - Teil 2 Kategorien : Allgemein, Open Source Schlagwörter : 3D Scan, MeshLab Datum : 23. August 2017 Mit 3D Rekonstruktion wird das Erstellen von dreidimensionalen Modellen
Gehirnsegmentierung mit der Wasserscheidentransformation. Xin Zhou
 Gehirnsegmentierung mit der Wasserscheidentransformation Xin Zhou 1 Gehirnsegmentierung mit der Wasserscheidentransformation (WST) Gliederung Sinn der Gehirnsegmentierung Struktur des Gehirns Wasserscheidentransformation
Gehirnsegmentierung mit der Wasserscheidentransformation Xin Zhou 1 Gehirnsegmentierung mit der Wasserscheidentransformation (WST) Gliederung Sinn der Gehirnsegmentierung Struktur des Gehirns Wasserscheidentransformation
IMRT und Patientenbewegung
 IMRT und Patientenbewegung Anne Richter Klinik und Poliklinik für Strahlentherapie Direktor: Prof. Dr. M. Flentje AK IMRT Neuruppin 27. 28.03.2008 Einleitung Einleitung Literatur Untersuchungen zum Einfluss
IMRT und Patientenbewegung Anne Richter Klinik und Poliklinik für Strahlentherapie Direktor: Prof. Dr. M. Flentje AK IMRT Neuruppin 27. 28.03.2008 Einleitung Einleitung Literatur Untersuchungen zum Einfluss
Bildanalyse und Visualisierung für die Planung von Nasennebenhöhlen-Operationen
 Bildanalyse und Visualisierung für die Planung von Nasennebenhöhlen-Operationen Dörte Apelt 1, Bernhard Preim 2, Horst K. Hahn 1, Gero Strauß 3 1 MeVis Centrum für Medizinische Diagnosesysteme und Visualisierung,
Bildanalyse und Visualisierung für die Planung von Nasennebenhöhlen-Operationen Dörte Apelt 1, Bernhard Preim 2, Horst K. Hahn 1, Gero Strauß 3 1 MeVis Centrum für Medizinische Diagnosesysteme und Visualisierung,
Ontologie Evolution. Konstantin Thierbach Problemseminar Ontologie Management Prof. Dr. E. Rahm Betreuer: M. Hartung
 Ontologie Evolution Konstantin Thierbach Problemseminar Ontologie Management Prof. Dr. E. Rahm Betreuer: M. Hartung 20.01.2009 Universität Leipzig, Abteilung Datenbanken Ontologie Evolution 1 / 32 Inhalt
Ontologie Evolution Konstantin Thierbach Problemseminar Ontologie Management Prof. Dr. E. Rahm Betreuer: M. Hartung 20.01.2009 Universität Leipzig, Abteilung Datenbanken Ontologie Evolution 1 / 32 Inhalt
Eine praktikable und genaue Methode zur Bestimmung von Wasser-Land-Grenzen aus Laser-Scanner-Daten
 Eine praktikable und genaue Methode zur Bestimmung von Wasser-Land-Grenzen aus Laser-Scanner-Daten Peter Dorninger pdo@ipf.tuwien.ac.at Institut für Photogrammetrie und Fernerkundung Technische Universität
Eine praktikable und genaue Methode zur Bestimmung von Wasser-Land-Grenzen aus Laser-Scanner-Daten Peter Dorninger pdo@ipf.tuwien.ac.at Institut für Photogrammetrie und Fernerkundung Technische Universität
3D-Rekonstruktion aus DSA-Projektionsdaten mittels diskreter Tomographie
 3D-Rekonstruktion aus DSA-Projektionsdaten mittels diskreter Tomographie Christoph Bodensteiner 1, Volker Martens 1, Stefan Schlichting 2, Norbert Binder 1, Rainer Burgkart 3, Achim Schweikard 1 1 Institut
3D-Rekonstruktion aus DSA-Projektionsdaten mittels diskreter Tomographie Christoph Bodensteiner 1, Volker Martens 1, Stefan Schlichting 2, Norbert Binder 1, Rainer Burgkart 3, Achim Schweikard 1 1 Institut
Echtzeitfähige Algorithmen für markerloses Tracking und Umfelderkennung , Dr.-Ing. Steffen Herbort, A.R.T. GmbH
 Echtzeitfähige Algorithmen für markerloses Tracking und Umfelderkennung 26.10.2016, TP 2: Arbeiten von A.R.T. TP2: Tracking und Umfelderkennung Markerloses Tracking texturierte Objekte Umfelderkennung
Echtzeitfähige Algorithmen für markerloses Tracking und Umfelderkennung 26.10.2016, TP 2: Arbeiten von A.R.T. TP2: Tracking und Umfelderkennung Markerloses Tracking texturierte Objekte Umfelderkennung
Bildsegmentierung mit Snakes und aktiven Konturen
 Bildsegmentierung mit Snakes und aktiven Konturen 5. Dezember 2005 Vortrag zum Seminar Bildsegmentierung und Computer Vision Übersicht 1 2 Definition der Snake Innere Energie S(c) 3 Statisches Optimierungsproblem
Bildsegmentierung mit Snakes und aktiven Konturen 5. Dezember 2005 Vortrag zum Seminar Bildsegmentierung und Computer Vision Übersicht 1 2 Definition der Snake Innere Energie S(c) 3 Statisches Optimierungsproblem
Implizite Modellierung zur Objekterkennung in der Fernerkundung
 Implizite Modellierung zur Objekterkennung in der Fernerkundung Mitarbeiterseminar 20.01.2011 (IPF) Fakultät für Bauingenieur-, Geo- und Umweltwissenschaften KIT Universität des Landes Baden-Württemberg
Implizite Modellierung zur Objekterkennung in der Fernerkundung Mitarbeiterseminar 20.01.2011 (IPF) Fakultät für Bauingenieur-, Geo- und Umweltwissenschaften KIT Universität des Landes Baden-Württemberg
Level-Set-Segmentierung von Rattenhirn MRTs
 Level-Set-Segmentierung von Rattenhirn MRTs Björn Eiben 1,2, Dietmar Kunz 2, Uwe Pietrzyk 1,3 und Christoph Palm 1,3 1 Institut für Neurowissenschaften und Biophysik, Jülich Forschungszentrum 2 Institut
Level-Set-Segmentierung von Rattenhirn MRTs Björn Eiben 1,2, Dietmar Kunz 2, Uwe Pietrzyk 1,3 und Christoph Palm 1,3 1 Institut für Neurowissenschaften und Biophysik, Jülich Forschungszentrum 2 Institut
2. Übung: Einführung in MeVisLab
 2. Übung: Einführung in MeVisLab MeVisLab Entwicklungsumgebung für die medizinische Bildverarbeitung und Visualisierung MeVisLab wird von der MeVis Medical Solutions AG und dem Fraunhofer Institut MEVIS
2. Übung: Einführung in MeVisLab MeVisLab Entwicklungsumgebung für die medizinische Bildverarbeitung und Visualisierung MeVisLab wird von der MeVis Medical Solutions AG und dem Fraunhofer Institut MEVIS
Ein effizienter geometrischer Ansatz zur Unterstützung der Trajektoriebestimmung bei der Tiefenhirnstimulation
 Ein effizienter geometrischer Ansatz zur Unterstützung der Trajektoriebestimmung bei der Tiefenhirnstimulation Jan Egger 1,2, Christoph Kappus 1, Bernd Freisleben 2, Christopher Nimsky 1 1 Klinik für Neurochirurgie,
Ein effizienter geometrischer Ansatz zur Unterstützung der Trajektoriebestimmung bei der Tiefenhirnstimulation Jan Egger 1,2, Christoph Kappus 1, Bernd Freisleben 2, Christopher Nimsky 1 1 Klinik für Neurochirurgie,
Gliederung: 1) Einleitung 2) Oberflächenvergleich 3) Objekterkennung
 Using Spin Images for Efficient Object Recogition in Cluttered 3D Scenes (Effiziente Objekterkennung von ungeordneten 3D Szenen mithilfe von Spin Images) Gliederung: 1) Einleitung 2) Oberflächenvergleich
Using Spin Images for Efficient Object Recogition in Cluttered 3D Scenes (Effiziente Objekterkennung von ungeordneten 3D Szenen mithilfe von Spin Images) Gliederung: 1) Einleitung 2) Oberflächenvergleich
Segmentierung der Prostata aus MRT-Bilddaten mittels eines statistischen Modells
 Segmentierung der Prostata aus MRT-Bilddaten mittels eines statistischen Modells Stefan Dänzer 1,5, Stefan Freitag 2, Dirk Beyersdorff 3, Markus Scholz 4, Oliver Burgert 1, Jens-Uwe Stolzenburg 5 1 ICCAS,
Segmentierung der Prostata aus MRT-Bilddaten mittels eines statistischen Modells Stefan Dänzer 1,5, Stefan Freitag 2, Dirk Beyersdorff 3, Markus Scholz 4, Oliver Burgert 1, Jens-Uwe Stolzenburg 5 1 ICCAS,
LiDAR Mobile Mapping
 Institut für Kartographie und Geoinformatik Leibniz Universität Hannover LiDAR Mobile Mapping Visualisierung, Vermessung, Fahrerassistenz Claus.Brenner@ikg.uni-hannover.de IKG Erfassungsfahrzeug Riegl
Institut für Kartographie und Geoinformatik Leibniz Universität Hannover LiDAR Mobile Mapping Visualisierung, Vermessung, Fahrerassistenz Claus.Brenner@ikg.uni-hannover.de IKG Erfassungsfahrzeug Riegl
