Bezeichnung Sequenz Verwendung
|
|
|
- Jasmin Matilde Diefenbach
- vor 6 Jahren
- Abrufe
Transkript
1 8 ANHANG i 8 ANHANG I Oligonukleotide Bezeichnung Sequenz Verwendung D ATGGCAGACGGTGAGGATATTCA-3 5 Aktin AC1 (neu) D GCCTTTGCAATCCACATCTGTTG-3 3 Aktin AC2 (neu) D98 5 -AT CC ATG GCA ATG TCA ATG GTG GTG TAC AAC-3 5 -MP17, AtSUC2-Prom. (NcoI) D C CAT GGC TCC GCG CTT GAT AAG TTT TGG-3 3 -MP17, AtSUC2-Prom. (NcoI) D GGA TCC TGT GCT TTA TTC TTT AGA AAC AAA AAA TAA-3` 3 -Tprom (BamHI) D GTC GAC GTT GAT GTT AAT ACA CGT TTC ACA ACT GAG-3 5 -Tprom_DL(-1645 (SalI) D AT GAG CTC TTA TTT GTA TAG TTC ATC CAT GC-3 3 -GFP f. TP -Fusion (SacI) D AT GGA TCC TCC GCG CTT GAT AAG TTT TGG-3 3 -MP17 f. TP -GUS (BamHI) KK8 5 -AGG TTT TAT TGC TTT TCA CA-3 5 ciw12, Chr. I, Genomk. KK9 5 -CTT TCA AAA GCA CAT CAC A -3 3 ciw12, Chr. I, Genomk. KK10 5 -TGT TTT TTA GGA CAA ATG GCG -3 5 nga111, Chr. I, Genomk. KK11 5 -CTC CAG TTG GAA GCT AAA GGG-3 3 nga111, Chr. I, Genomk. KK12 5 -CCC AAA AGT TAA TTA TAC TGT -3 5 ciw2, Chr. II, Genomk. KK13 5 -CCG GGT TAA TAA TAA ATG T-3 3 ciw2, Chr. II, Genomk. KK14 5 -GCA CAG TCC AAG TCA CAA CC-3 5 nga1126, Chr. II, Genomk. KK15 5 -CGC TAC GCT TTT CGG TAA AG-3 3 nga1126, Chr. II, Genomk. KK16 5 -CTC TGT CAC TCT TTT CCT CTG G-3 5 nga162, Chr. III, Genomk. KK17 5 -CAT GCA ATT TGC ATC TGA GG-3 3 nga162, Chr. III, Genomk. KK18 5 -ATG GAG AAG CTT ACA CTG ATC-3 5 nga6, Chr. III, Genomk. KK19 5 -TGG ATT TCT TCC TCT CTT CAC-3 3 nga6, Chr. III, Genomk. KK20 5 -GGT TAA AAA TTA GGG TTA CGA-3 5 ciw5, Chr. IV, Genomk. KK21 5 -AGA TTTACG TGG AAG CAA T-3 3 ciw5, Chr. IV, Genomk. KK22 5 -AAT TTG GAG ATT AGC TGG AAT-3 5 ciw7, Chr. IV, Genomk. KK23 5 -CCA TGT TGA TGA TAA GCA CAA-3 3 ciw7, Chr. IV, Genomk. KK24 5 -TAG TGA AAC CTT TCT CAG AT-3 5 ciw8, Chr. V, Genomk. KK25 5 -TTA TGT TTT CTTCAA TCA GTT-3 3 ciw8, Chr. V, Genomk. KK26 5 -CCA CATTTTCCT TCT TTC ATA-3 5 ciw10, Chr. V, Genomk. KK27 5 -CAA CAT TTA GCA AAT CAA CTT-3 3 ciw10, Chr. V, Genomk. KK28 5 -GGC TTT CTC GAA ATC TGT CC-3 5 F21M12, Chr. I, Genomk. KK29 5 -TTA CTT TTT GCC TCT TGT CAT TG-3 3 F21M12, Chr. I, Genomk. KK30 5 -ACA TTT TCT CAA TCC TTA CTC-3 5 ciw1, Chr. I, Genomk. KK31 5 -GAG AGC TTC TTT ATT TGT GAT-3 3 ciw1, Chr. I, Genomk.
2 8 ANHANG ii KK32 5 -CTG ATC TCA CGG ACA ATA GTG C-3 5 nga280, Chr. I, Genomk. KK33 5 -GGC TCC ATA AAA AGT GCA CC-3 5 ciw3, Chr. II, Genomk. KK34 5 -GAA ACT CAA TGA AAT CCA CTT-3 5 ciw3, Chr. II, Genomk. KK35 5 -TGA ACT TGT TGT GAG CTT TGA-3 3 ciw3, Chr. II, Genomk. KK36 5 -TCG TCT ACT GCA CTG CCG-3 5 nga168, Chr. II, Genomk. KK37 5 -GAG GAC ATG TAT AGG AGC CTC G-3 3 nga168, Chr. II, Genomk. KK38 5 -CCC CGA GTT GAG GTA TT-3 5 ciw11, Chr. III, Genomk. KK39 5 -GAA GAA ATT CCT AAA GCA TTC-3 3 ciw11, Chr. III, Genomk. KK40 5 -GTT CAT TAAACT TGC GTG TGT-3 5 ciw4, Chr. III, Genomk. KK41 5 -TAC GGT CAG ATT GAG TGA TTC-3 3 ciw4, Chr. III, Genomk. KK42 5 -CTC GTA GTG CAC TTT CAT CA-3 5 ciw6, Chr. IV, Genomk. KK43 5 -CAC ATG GTT AGG GAA ACA ATA -3 3 ciw6, Chr. IV, Genomk. KK44 5 -GCG AAA AAA CAA AAA AAT CCA-3 5 nga1107, Chr. IV, Genomk. KK45 5 -CGA CGA ATC GACAGA ATT AGG-3 3 nga1107, Chr. IV, Genomk. KK48 5 -CTC AGA GAA TTC CCA GAA AAA TCT-3 5 PHYC, Chr. V, Genomk. KK49 5 -AAA CTC GAG AGT TTT GTC TAG ATC-3 3 PHYC, Chr. V, Genomk. KK50 5 -CAG ACG TAT CAA ATG ACA AAT G-3 5 ciw9, Chr. V, Genomk. KK51 5 -GAC TAC TGC TCA AAC TAT TCG G-3 3 ciw9, Chr. V, Genomk. II Kohlenhydratgehalte in MP17:GFP-transgenen Arabidopsis Glukose Fruktose Saccharose Stärke Lösliche Zucker/Stärke Col-9 Col-14 Col-16 0,48 ± 6 0,52 ± 6 1,01 ± 0,15** 5,26 ± 0,54*** 0,92 ± 0,11 1,87 ± 0,36* 1,64 ± 6*** 2,11 ± 0,16*** 1,15 ± 7 0,99 ± 8 1,07 ± 7 2,65 ± 0,24*** 11,29 ± 0,46 10,43 ± 0,56 10,88 ± 0,43 23,10 ± 2,01*** 0,23 ± 1 0,32 ± 3* 0,34 ± 9,61e-3*** 0,44 ± *** Ler WT Ler-1 Ler-12 0,82 ± 0,10 5,52 ± 0,58 *** 3,49 ± 0,61*** 1,62 ± 0,13 4,72 ± 0,37*** 3,37 ± 0,29*** 1,21 ± 4 2,48 ± 0,15*** 2,02 ± 0,26** 12,04 ± 0,43 17,10 ± 1,26 ** 13,21 ± 1,43 0,30 ± 2 0,78 ± 9*** 0,76 ± 0,16* C24 WT C24-81 C24-84 C ,18 ± 0,15 0,92 ± 0,12 0,84 ± 7 1,10 ± 0,15 1,02 ± 0,18 0,93 ± 0,12 1,08 ± 9 1,20 ± 0,10 1,00 ± 6 0,78 ± 6* 0,79 ± 6* 1,09 ± 6 13,05 ± 0,66 12,05 ± 0,85 13,90 ± 0,93 14,76 ± 0,52 0,25 ± 3 0,22 ± 1 0,20 ± 1 0,20 ± 3 Angegeben sind die Mittelwerte mit Standardfehler der einzelnen Zuckermessungen aus je 10 Doppelproben von 5 Wochen alten source-blättern. Signifikante Unterschiede (* P < 5, ** P < 1, *** P < 01) im Vergleich zur jeweiligen Wildtyp-Kontrolle wurden mit Hilfe des Student s t-test ermittelt. Col-0: Columbia, Ler: Landsberg erecta.
3 8 ANHANG iii III Biomasseverteilung in konstitutiv MP17:GFP-exprimierenden Arabidopsis Anzucht Linie vegetative vegetative generative Samenertrag Anmerkungen Biomasse Biomasse Biomasse (Blattfrisch- (Blatttrocken- (Blattfrischgewicht) gewicht) gewicht) [g] [g] [g] [g] 1 2,026 ± 68 0,180 ± 05 0,152 ± 15 A Sept Col-0-9 2,532 ± 98 0,227 ± 08 0,140 ± 0 7 nach 6 Wochen Col ,948 ± 62 0,185 ± 09 0,199 ± 10 Kurztag Col ,361 ± ± 03 0,217 ± 12 Transfer unter Ler WT 2,381 ± 0,102 0,199 ± 07 0,128 ± 07 Langtag Ler-1 0,222 ± ± 01 0,215 ± ,158 ± 09 A Feb Col-0-9 0,132 ± 13 nach 2 Wochen Col ,174 ± 10 Kurztag Col ,188 ± 12 Transfer unter Langtag 3 2,106 ± 0,151 0,184 ± 10 0,120 ± 12 A Aug Col-0-9 2,242 ± 0,135 0,185 ± 09 0,165 ± 0 9 nach 6 Wochen Col ,774 ± 0,173 0,150 ± 11 0,138 ± 12 Kurztag Col ,255 ± ± 02 0,151 ± 60 Transfer unter Ler WT 1,972 ± 0,131 0,154 ± ± 09 Langtag Ler-1 0,219 ± ± 02 0,168 ± ,563 ± 0,106 0,1748 ± 084 A Dez Col-0-9 1,839 ± 44 0,177 ± 16 nach 6 Wochen Col ,343 ± 57 0,188 ± 12 Kurztag Col ,158 ± 12 0,197 ± 06 Transfer unter Ler WT 2,414 ± 0,136 0,124 ± 14 Langtag Ler-1 0,155 ± 17 0,185 ± 11 Ler-12 0,204 ± 24 0,156 ± ,739 ± 0,103 0,323 ± 28 0,139 ± 0, 005 A Aug Col-0-9 1,907 ± 57 0,411 ± 19 0,133 ± 08 nach 6 Wochen Col ,548 ± 78 0,327 ± 22 0,145 ± 0, 011 Kurztag Col ,367 ± 11 0,102 ± 03 0,163 ± 0, 005 Transfer unter Ler WT 2,167 ± 0,138 0,375 ± 22 0,125 ± 02 Langtag Ler-1 0,368 ± 36 0,147 ± 07 0,153 ± 08 Ler-12 0,464 ± 07 0,148 ± 09 0,151 ± 09 Die Daten repräsentieren die Verteilung der Biomasse in unterschiedlichen Entwicklungsstadien von 5 unabhängigen Anzuchten. Die vegetative Blattbiomasse wurde nach 6 Wochen Kurztag ermittelt, die generative Blattbiomasse zu Beginn der Sprossausbildung, nachdem die verbleibenden Pflanzen der
4 8 ANHANG iv vegetativen Biomassebestimmung unter Langtagbedingungen transferiert wurden. Nach vollständigem Abreifen der Pflanzen wurde der Samenertrag bestimmt. Pro Datenset wurden die Mittelwerte mit Standardfehler von 5-10 Pflanzen bestimmt. Angegeben sind die absoluten Werte in [g]. A: Aussaat, Col-0: Columbia, Ler: Landsberg erecta. IV Biomasseverteilung in Col-0-Arabidopsis mit Zell-spezifischer MP17:GFP-Expression Anzucht Linie vegetative Biomasse (Blattfrischgewicht) vegetative Biomasse Samenertrag Anmerkungen (Blattfrischgewicht) [g] [g] [g] 1 2,196 ± 0,154 0,218 ± 40 A Nov Col ,256 ± 13 0,234 ± 43 nach 6 Wochen L ,717 ± 87 0,228 ± 23 Kurztag L ,275 ± 21 0,226 ± 08 Transfer unter PEP-4 2,788 ± 0,237 0,204 ± 08 Langtag PEP-14 1,758 ± 0,176 0,210 ± 36 TP -6 2,496 ± 0,218 0,302 ± 0,300 TP -8 2,302 ± 0,13 0,288 ± 0 TP -16 2,240 ± 0,198 0,339 ± ,644 ± 0,101 0,346 ± 20 0,131 ± 1 3 A April 2006 Col ,251 ± 17 0,162 ± 10 0,166 ± 0 8 nach 6 Wochen L ,714 ± 48 0,229 ± 09 0,165 ± 10 Kurztag L ± 07 0,157 ± 10 0,173 ± 06 Transfer unter PEP-4 1,587 ± 78 0,291 ± 17 0,136 ± 11 Langtag PEP-14 1,696 ± 0,103 0,340 ± 18 0,128 ± 05 TP -6 1,388 ± 0,102 0,321 ± 19 0,190 ± 08 TP -8 1,731 ± 61 0,371 ± 15 0,191 ± 08 TP -16 1,937 ± 0,182 0,346 ± 17 0,195 ± ,739 ± 0,103 0,323 ± 28 0,139 ± 0 5 A Aug Col ,367 ± 11 0,102 ± 03 0,163 ± 0 5 nach 6 Wochen L ,904 ± 60 0,175 ± 08 0,150 ± 05 Kurztag L ,299 ± 28 0,100 ± 03 0,160 ± 09 Transfer unter PEP-4 1,618 ± 0,185 0,316 ± 25 0,128 ± 07 Langtag PEP-14 1,714 ± 0,136 0,337 ± 44 0,135 ± 13 TP -6 1,968 ± 0,215 0,372 ± 29 0,185 ± 08 TP -8 1,949 ± 26 0,376 ± 49 0,182 ± 15 TP -16 1,964 ± 37 0,371 ± 20 0,184 ± 08 In der Tabelle ist die Verteilung der Biomasse während unterschiedlicher Entwicklungsstadien von drei unabhängigen Anzuchten transgener Arabidopsis-Pflanzen zusammengefasst, die im Vergleich zur WT- und konstitutiven Col-0-16-Referenz MP17:GFP unter der Kontrolle eines Photosystem II- (L700), Geleitzell- (PEP) und Bündelscheiden-spezifischen (TP ) Promotors exprimieren. Die vegetative Blattbiomasse wurde nach 6 Wochen Kurztag ermittelt, die generative Blattbiomasse zu Beginn der Sprossausbildung, nachdem die verbleibenden Pflanzen der vegetativen Biomassebestimmung unter Langtagbedingungen transferiert wurden. Nach vollständigem Abreifen der Pflanzen wurde der
5 8 ANHANG v Samenertrag bestimmt. Pro Datenset wurden die Mittelwerte mit Standardfehler von 5-10 Pflanzen bestimmt. Angegeben sind die absoluten Werte in [g] sowie der prozentuale Anteil bezogen auf die WT-Referenz. A: Aussaat, Col-0: Columbia. V Kohlenhydratgehalte in Arabidopsis mit Zell-spezifischer MP17:GFP-Expression Glukose Fruktose Saccharose Stärke Lösliche Zucker/Stärke Col-16 0,72 ± 8 4,62 ± 0,30*** 0,80 ± 0,12 1,77 ± 0,21*** 0,70 ± 4 1,84 ± 0,18*** 13,00 ± 0,55 22,88 ± 2,64** 0,17 ± 1 0,41 ± 6** L L ,14 ± 0,57*** 5,67 ± 0,95*** 1,49 ± 0,11*** 2,04 ± 0,24*** 1,84 ± 0,13*** 2,15 ± 0,19*** 22,19 ± 2,16*** 24,99 ± 2,14*** 0,37 ± 5** 0,42 ± 6*** PEP-4 PEP-14 1,56 ± 0,26** 2,09 ± 0,40** 0,77 ± 0,13 0,67 ± 0,11 0,75 ± 4 0,72 ± 7 12,18 ± 0,45 12,62 ± 0,55 0,25 ± 2** 0,28 ± 4* TP -6 TP -8 TP -16 0,92 ± 0,25 0,69 ± 0,12 1,45 ± 0,20** 0,81 ± 0,19 1,09 ± 9 0,89 ± 7 0,45 ± 4** 0,69 ± 5 0,67 ± 5 10,73 ± 0,54* 11,54 ± 0,79 10,77 ± 1,28 0,21 ± 2 0,22 ± 2 0,32 ± 0,10 Angegeben sind die Mittelwerte mit Standardfehler der einzelnen Zuckermessungen aus je 10 Doppelproben der WT-, Col und L700-Linien und aus 4-6 Doppelproben der PEP- und TP - Linien von 5 Wochen alten source-blättern. Signifikante Unterschiede (* P < 5, ** P < 1, *** P < 01) im Vergleich zur Wildtyp-Kontrolle wurden mit Hilfe des Student s t-test ermittelt. Col-0: Columbia, L700: Konstrukt zur MP17:GFP-Expression in chloroplastenhaltigem Gewebe unter Kontrolle des ST-LS1-Promotors aus Solanum tuberosum, PEP: Konstrukt zur MP17:GFP-Expression in Geleitzellen unter Kontrolle des AtSUC2-Promotors aus Arabidopsis thaliana, TP : Konstrukt zur MP17:GFP-Expression in Bündelscheidenzellen unter Kontrolle des TP -Promotors des gdcst-gens aus Flaveria anomala. VI Ergänzende Bestimmung von Frisch- und Trockengewicht von MP17- und MP17:GFP transgenen Arabidopsis-Pflanzen mit konstitutiver und spezifischer Expression Zur Überprüfung der Biomasseverteilung während der vegetativen Wachstumsphase wurde neben dem Frischgewicht das Trockengewicht der konstitutiv MP17:GFPbzw. MP17-exprimierenden Arabidopsis-Linien im Col-0-, Ler- und C24-Hinterund sowie von Col-0-Linien, welche MP17:GFP unter der Kontrolle eines Geleitzell-, Bündelscheiden- und Chloroplasten-spezifischen Promotors exprimieren, ermittelt. Nach 6 Wochen unter Kurztagbedingungen wurde das Frischgewicht und nach
6 8 ANHANG vi weiteren zwei Tagen im Trockenschrank bei 50 C das Trockengewicht bestimmt (s. Abb.). (A) (B) 2,5 3,0 2,0 2,5 Frischgewicht [g] 1,5 1,0 Frischgewicht [g] 2,0 1,5 1,0 0,5 0,5 Trockengewicht [g] 0,3 0,2 0,1 Col-0-9 Col-0-14 Col-0-16 Ler WT Ler -1 Ler-12 C24 WT C24-81 C24-84 C24-74 C24-12 C24-14 C24-16 Trockengewicht [g] 0,3 0,2 0,1 Col-0-16 L L PEP-4 PEP-14 TP-6 TP-8 TP-16 Abbildung 8-1: Frisch- und Trockengewichte konstitutiv MP17:GFP- und MP17 sowie spezifisch MP17:GFP-exprimierender Arabidopsis-Linien während vegetativem Wachstumsstadium im Vergleich zur Ökotyp-spezifischen WT-Kontrolle Nach 6 Wochen Kurztagbedinungen wurden die Frischgewichte und nach weiteren zwei Tagen im Trockenschrank bei 50 C wurden die Trockengewichte bestimmt. Angegeben sind die Mittelwerte in Gramm mit Standardabweichung von 6-10 Pflanzen je Linie. Col-0: Columbia, Ler: Landsberg erecta, L700: Chloroplasten-spezifische Expression (ST LS-1-Promotor)), PEP: Geleitzell-spezifische Expression (At SUC2 SphI-Promotor), TP : Bündelscheiden-spezifische Expression (TP -Promotor).
Dendrogramm der Primaten
 Arbeitsblatt 1 Dargestellt ist ein Ausschnitt der DNA-Sequenz für das Enzym NAD- Polymerase, dass bei den Primatenarten Mensch(M), Schimpanse(S), Gorilla(G), Orang-Utan(O) und Gibbon(Gi) vorhanden ist.
Arbeitsblatt 1 Dargestellt ist ein Ausschnitt der DNA-Sequenz für das Enzym NAD- Polymerase, dass bei den Primatenarten Mensch(M), Schimpanse(S), Gorilla(G), Orang-Utan(O) und Gibbon(Gi) vorhanden ist.
Primer zum Nachweis eukaryoter DNA (Kontroll-PCR) Primer Sequenz Ziel-Gen Produktgröße T a
 7 Anhang 7.1 Primersequenzen Alle Oligonukleotide sind in 5-3 -Orientierung aufgelistet. 7.1.1.1.1 Primer zum Nachweis eukaryoter DNA (Kontroll-PCR) 258 s CCA TCC AAC ATC TCA GCA TGA TGA AA GCC CCT CAG
7 Anhang 7.1 Primersequenzen Alle Oligonukleotide sind in 5-3 -Orientierung aufgelistet. 7.1.1.1.1 Primer zum Nachweis eukaryoter DNA (Kontroll-PCR) 258 s CCA TCC AAC ATC TCA GCA TGA TGA AA GCC CCT CAG
Material MATERIAL 3.1 Oligonukleotide
 Material 13 3 MATERIAL 3.1 Oligonukleotide AGK34 LINE1 Ale1 Ale3 LIS1 LIS2 UR UL RA2 LS2 pbst7 sp6 DOP (6-MW) pcypac2-01 pcypac2-02 SD2 SA2 SA4 SD6 D11S2164 (CP5) forward D11S2164 (CP5) revers M16 forward
Material 13 3 MATERIAL 3.1 Oligonukleotide AGK34 LINE1 Ale1 Ale3 LIS1 LIS2 UR UL RA2 LS2 pbst7 sp6 DOP (6-MW) pcypac2-01 pcypac2-02 SD2 SA2 SA4 SD6 D11S2164 (CP5) forward D11S2164 (CP5) revers M16 forward
Sigma-Aldrich Chemie GmbH, München Merck Biosciences, Darmstadt. Thermo Hybaid GmbH, Ulm Roche Diagnostics GmbH, Mannheim
 3 Materialien 28 3 Materialien 3.1 Chemikalien Acrylamid Agar Agarose Ammoniumsulfat Ampicillin, Natriumsalz L-Arabinose Bradford-Reagenz Bromphenolblau Caseinhydrolysat Chloramphenicol Chloroform Coomassie-Brillant-Blau
3 Materialien 28 3 Materialien 3.1 Chemikalien Acrylamid Agar Agarose Ammoniumsulfat Ampicillin, Natriumsalz L-Arabinose Bradford-Reagenz Bromphenolblau Caseinhydrolysat Chloramphenicol Chloroform Coomassie-Brillant-Blau
Genannotation bei Prokaryoten
 Genannotation bei Prokaryoten Maike Tech Abt. Bioinformatik Institut für Mikrobiologie und Genetik (IMG) Universität Göttingen 28. November 2005 Genetik von Pro- und Eukaryoten Eukaryoten Prokaryoten Zellkern
Genannotation bei Prokaryoten Maike Tech Abt. Bioinformatik Institut für Mikrobiologie und Genetik (IMG) Universität Göttingen 28. November 2005 Genetik von Pro- und Eukaryoten Eukaryoten Prokaryoten Zellkern
Anhang. 1. Verwendete Abkürzungen
 A Anhang 1. Verwendete Abkürzungen ACF AM Ap r/s AS ATR ß-Gal BlaM BSA bp C-Quelle CAT CFTR cfu CI CT dntp ECA EDTA ELISA EPS HAP IS Km r/s LB LPS Mbp mcs Min MBK MHK MOI MOPS MSHA NAD OD x ORF accessory
A Anhang 1. Verwendete Abkürzungen ACF AM Ap r/s AS ATR ß-Gal BlaM BSA bp C-Quelle CAT CFTR cfu CI CT dntp ECA EDTA ELISA EPS HAP IS Km r/s LB LPS Mbp mcs Min MBK MHK MOI MOPS MSHA NAD OD x ORF accessory
MODUL 2.36 KLINISCHE CHEMIE UND LABORDIAGNOSTIK. TAG 6: HLA-Typisierung
 MODUL 2.36 KLINISCHE CHEMIE UND LABORDIAGNOSTIK TAG 6: HLA-Typisierung Diese Anleitung bitte ausdrucken, vor dem Praktikumstag durcharbeiten und mitbringen! Inhalte der Einführungsvorlesung und der Anleitung
MODUL 2.36 KLINISCHE CHEMIE UND LABORDIAGNOSTIK TAG 6: HLA-Typisierung Diese Anleitung bitte ausdrucken, vor dem Praktikumstag durcharbeiten und mitbringen! Inhalte der Einführungsvorlesung und der Anleitung
Anhang. Bedeutung Bezeichnung Rs + Nukleotidsequenz (5-3 ) Mutationsprimer Asp 29 Glu
 AHAG 115 Anhang Tab. 13: Konstruktion der Primer zur Generierung der EcIPDC-Mutanten. Ausgetauschte ukleotide sind unterstrichen. Das ukleotidtriplett, welches für die mutierte Aminosäure codiert, ist
AHAG 115 Anhang Tab. 13: Konstruktion der Primer zur Generierung der EcIPDC-Mutanten. Ausgetauschte ukleotide sind unterstrichen. Das ukleotidtriplett, welches für die mutierte Aminosäure codiert, ist
2. MATERIAL UND METHODEN
 2. MATERIAL UND METHODEN 2.1 Material 2.1.1 Proben zur DNA-Analytik Blutproben zur Gewinnung von DNA aus Leukozyten wurden uns freundlicherweise von Dr. Ramaekers, Universität Aachen, überlassen. Ein positives
2. MATERIAL UND METHODEN 2.1 Material 2.1.1 Proben zur DNA-Analytik Blutproben zur Gewinnung von DNA aus Leukozyten wurden uns freundlicherweise von Dr. Ramaekers, Universität Aachen, überlassen. Ein positives
Patente in der Biotechnologie. VPP-Düsseldorf. 2002-März-20
 Patente in der Biotechnologie VPP-Düsseldorf 2002-März-20 25 März, 2002 KB-RP / PL / PL4 / Biotechnology 1 Patente in der Biotechnologie Große Tiere, kleine Tiere, Gene, Proteine und Patente 25 März, 2002
Patente in der Biotechnologie VPP-Düsseldorf 2002-März-20 25 März, 2002 KB-RP / PL / PL4 / Biotechnology 1 Patente in der Biotechnologie Große Tiere, kleine Tiere, Gene, Proteine und Patente 25 März, 2002
TCC CCA GTG AAG ACC TCC TCA GAG TCC GAC TCT. 56 C 67 siehe oben siehe oben siehe oben 56 C 66 CCG GCG CCA TTC TAT CC CGA GAA TCT GAC GCA GG 56 C 738
 128 9. Anhang 9.1. Tabelle der verwendeten Primerpaare Nr. Name Sequenz 5 3 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 rcar. 981s rcar. 981s GAPDH. 682s GAPDH. 826a GAPDH. 381s GAPDH. 826a 18S-rRNA. 216s 18S-rRNA.
128 9. Anhang 9.1. Tabelle der verwendeten Primerpaare Nr. Name Sequenz 5 3 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 rcar. 981s rcar. 981s GAPDH. 682s GAPDH. 826a GAPDH. 381s GAPDH. 826a 18S-rRNA. 216s 18S-rRNA.
A- Zugabe von 180µl Pufferlösung und Proteinase K, Inkubation bei 56 C. B- Zugabe von 200µl einer zweiten Pufferlösung, Inkubation bei 70 C
 3 Methoden 3.1 Extraktion der DNA aus den Paraffinschnitten 3.1.1 Extraktions-Kit Das QIAamp DNA Mini Kit- System beruht auf dem Prinzip der Auflösung der Eiweiße mit Proteinase K, DNA Bindung an eine
3 Methoden 3.1 Extraktion der DNA aus den Paraffinschnitten 3.1.1 Extraktions-Kit Das QIAamp DNA Mini Kit- System beruht auf dem Prinzip der Auflösung der Eiweiße mit Proteinase K, DNA Bindung an eine
4.1. Physikalische Kartierung eines STS-Markers aus dem KPNA2-Gen
 Ergebnisse 51 4. Ergebnisse 4.1. Physikalische Kartierung eines STS-Markers aus dem KPNA2-Gen 4.1.1. Kartierung unter Verwendung somatischer Zellhybride Aufgrund der vollständigen Konkordanz der 18 somatischen
Ergebnisse 51 4. Ergebnisse 4.1. Physikalische Kartierung eines STS-Markers aus dem KPNA2-Gen 4.1.1. Kartierung unter Verwendung somatischer Zellhybride Aufgrund der vollständigen Konkordanz der 18 somatischen
HLA-READY GENE ABC Low
 LOT G953085 Arbeitsprotokoll / PCR Protocol 2006-12 Test-Durchführung / -Performance Datum / Date Unterschrift / Signature Bemerkungen / Comments Probe / Sample 1 2 3 4 5 Thermocycler 1 Thermocycler 2
LOT G953085 Arbeitsprotokoll / PCR Protocol 2006-12 Test-Durchführung / -Performance Datum / Date Unterschrift / Signature Bemerkungen / Comments Probe / Sample 1 2 3 4 5 Thermocycler 1 Thermocycler 2
Praktikum Molekularbiologie, 5. Semester, BT 12 Martin Sievers, Gottfried Dasen und Tobias Wermelinger RT 418
 Praktikum Molekularbiologie, 5. Semester, BT 12 Martin Sievers, Gottfried Dasen und Tobias Wermelinger RT 418 Datum Gruppe Thema 18.09.14 I Forensik: PCR und Restriktionsanalyse am Beispiel der ITS-Region
Praktikum Molekularbiologie, 5. Semester, BT 12 Martin Sievers, Gottfried Dasen und Tobias Wermelinger RT 418 Datum Gruppe Thema 18.09.14 I Forensik: PCR und Restriktionsanalyse am Beispiel der ITS-Region
3 Ergebnisse. 3.1 cdnas
 3 Ergebnisse 3.1 cdnas Das erste Screening einer Expressionsgenbank (λ-gt11, SCHARF et al., 1990) mit einem polyklonalen Kaninchenantiserum gegen HSP17 der Tomate ergab mehr als einhundert positive Klone.
3 Ergebnisse 3.1 cdnas Das erste Screening einer Expressionsgenbank (λ-gt11, SCHARF et al., 1990) mit einem polyklonalen Kaninchenantiserum gegen HSP17 der Tomate ergab mehr als einhundert positive Klone.
Konzept zur Untersuchung von Saatgut auf Anteile gentechnisch veränderter Pflanzen
 Konzept zur Untersuchung von Saatgut auf Anteile gentechnisch veränderter Pflanzen Unterausschuss Methodenentwicklung der Bund/Länder-Arbeitsgemeinschaft Gentechnik (LAG) (Stand: März 2006) Inhaltsverzeichnis
Konzept zur Untersuchung von Saatgut auf Anteile gentechnisch veränderter Pflanzen Unterausschuss Methodenentwicklung der Bund/Länder-Arbeitsgemeinschaft Gentechnik (LAG) (Stand: März 2006) Inhaltsverzeichnis
Material. 3 Material. 3.1 Reagenzien
 3 Material 3.1 Reagenzien Tabelle 1: verwendete Reagenzien Substrat Hersteller Firmensitz Agarose (NEEO, Ultra-Qualität) Carl Roth Karlsruhe BPB (Bromphenolblau) Merck Darmstadt DEPC (Diethylpyrocarbonat)
3 Material 3.1 Reagenzien Tabelle 1: verwendete Reagenzien Substrat Hersteller Firmensitz Agarose (NEEO, Ultra-Qualität) Carl Roth Karlsruhe BPB (Bromphenolblau) Merck Darmstadt DEPC (Diethylpyrocarbonat)
PROGRAMMHEFT TSV RUDOW BERLIN
 PROGRAMMHEFT TSV RUDOW BERLIN 17.6.2017 Meisterschaft der Landessieger Ü40 Fünf Mannschaften welches Bundesland holt sich den Titel? 1 AAT TGA CAC ACG TTC GCC GAT AAG GAA GTT ATG GAA CCT GGA CCA TCA CGA
PROGRAMMHEFT TSV RUDOW BERLIN 17.6.2017 Meisterschaft der Landessieger Ü40 Fünf Mannschaften welches Bundesland holt sich den Titel? 1 AAT TGA CAC ACG TTC GCC GAT AAG GAA GTT ATG GAA CCT GGA CCA TCA CGA
Sorbus-(Klein)arten aus genetischer Sicht
 Sorbus-(Klein)arten aus genetischer Sicht Ludger Leinemann, Bernhard Hosius, Karina Kahlert und Wolfgang Arenhövel Ilmenau 23.09.2016 Inhalt Einleitung Hypothesen über die Entstehung der Sorbus- (Klein)arten
Sorbus-(Klein)arten aus genetischer Sicht Ludger Leinemann, Bernhard Hosius, Karina Kahlert und Wolfgang Arenhövel Ilmenau 23.09.2016 Inhalt Einleitung Hypothesen über die Entstehung der Sorbus- (Klein)arten
4.1 Erstellung einer Mammakarzinom-spezifischen cdna- Expressionsbank in λ-phagen
 4 Ergebnisse 4.1 Erstellung einer Mammakarzinom-spezifischen cdna- Expressionsbank in λ-phagen 4.1.1 RNA-Isolierung aus den Mammakarzinom-Zellinien BT-474 und 8701-BC Von jeder Zellinie wurden 4 x 10 7
4 Ergebnisse 4.1 Erstellung einer Mammakarzinom-spezifischen cdna- Expressionsbank in λ-phagen 4.1.1 RNA-Isolierung aus den Mammakarzinom-Zellinien BT-474 und 8701-BC Von jeder Zellinie wurden 4 x 10 7
Erfassung und Bewertung von Genexpressionsmustern von Zebrabärblingen (Danio rerio) nach Belastung mit östrogenen Substanzen
 Lehrstuhl für Zellbiologie der Technischen Universität München Wissenschaftszentrum Weihenstephan Univ.-Prof. Dr. Bertold Hock Erfassung und Bewertung von Genexpressionsmustern von Zebrabärblingen (Danio
Lehrstuhl für Zellbiologie der Technischen Universität München Wissenschaftszentrum Weihenstephan Univ.-Prof. Dr. Bertold Hock Erfassung und Bewertung von Genexpressionsmustern von Zebrabärblingen (Danio
MtrA, ein bifunktionaler Antwortregulator
 MtrA, ein bifunktionaler Antwortregulator aus Corynebacterium glutamicum Inaugural-Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Heinrich-Heine-Universität
MtrA, ein bifunktionaler Antwortregulator aus Corynebacterium glutamicum Inaugural-Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Heinrich-Heine-Universität
2. Material und Methoden
 2. Material und Methoden 2.1. Material 2.1.1. Geräte BioPhotometer 6131 (Eppendorf, Hamburg, D.) Durchlichtmikroskop BX 60 (Olympus, Hamburg, D.) Durchlichtmikroskop MZ 12 (Leica, Bensheim, D.) Feinwaage
2. Material und Methoden 2.1. Material 2.1.1. Geräte BioPhotometer 6131 (Eppendorf, Hamburg, D.) Durchlichtmikroskop BX 60 (Olympus, Hamburg, D.) Durchlichtmikroskop MZ 12 (Leica, Bensheim, D.) Feinwaage
Zur Funktion des MPH1-Gens von Saccharomyces cerevisiae bei der rekombinativen Umgehung von replikationsarretierenden DNA-Schäden
 Zur Funktion des MPH1-Gens von Saccharomyces cerevisiae bei der rekombinativen Umgehung von replikationsarretierenden DNA-Schäden Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen
Zur Funktion des MPH1-Gens von Saccharomyces cerevisiae bei der rekombinativen Umgehung von replikationsarretierenden DNA-Schäden Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen
Charakterisierung von Proteinen der 5 -Prozessierung mitochondrialer Transkripte in Arabidopsis thaliana
 Charakterisierung von Proteinen der 5 -Prozessierung mitochondrialer Transkripte in Arabidopsis thaliana Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. der Fakultät für Naturwissenschaften der
Charakterisierung von Proteinen der 5 -Prozessierung mitochondrialer Transkripte in Arabidopsis thaliana Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. der Fakultät für Naturwissenschaften der
Protein-Protein-Interaktionen der U1snRNPKomponente Prp40 und deren FF-Domänen in Saccharomyces cerevisiae
 Forschungszentrum Karlsruhe in der Helmholtz-Gemeinschaft Wissenschaftliche Berichte FZKA 7304 Protein-Protein-Interaktionen der U1snRNPKomponente Prp40 und deren FF-Domänen in Saccharomyces cerevisiae
Forschungszentrum Karlsruhe in der Helmholtz-Gemeinschaft Wissenschaftliche Berichte FZKA 7304 Protein-Protein-Interaktionen der U1snRNPKomponente Prp40 und deren FF-Domänen in Saccharomyces cerevisiae
Posttranskriptionelle Regulation des Chemokins CCL5 (RANTES) durch den TAB1-p38 Signalweg
 Posttranskriptionelle Regulation des Chemokins CCL5 (RANTES) durch den TAB1-p38 Signalweg Inauguraldissertation zur Erlangung des Grades eines Doktors der Medizin des Fachbereichs Medizin der Justus-Liebig-Universität
Posttranskriptionelle Regulation des Chemokins CCL5 (RANTES) durch den TAB1-p38 Signalweg Inauguraldissertation zur Erlangung des Grades eines Doktors der Medizin des Fachbereichs Medizin der Justus-Liebig-Universität
7 Anhang. Tabelle I: Zusammensetzung der Restriktionsansätze.
 7 Anhang Tabelle I: Zusammensetzung der Restriktionsansätze. Reagenz Konzentration Volumen je Endkonzentration der Stammlösung Reaktionansatz Reaktionspuffer 10x NEB-Puffer 2 1.50 µl 1x MseI 4 U/µl 0.30
7 Anhang Tabelle I: Zusammensetzung der Restriktionsansätze. Reagenz Konzentration Volumen je Endkonzentration der Stammlösung Reaktionansatz Reaktionspuffer 10x NEB-Puffer 2 1.50 µl 1x MseI 4 U/µl 0.30
Funktionelle Analyse von ERF-Transkriptionsfaktoren aus N. tabacum und A. thaliana im Rahmen der Pathogenresistenz
 Funktionelle Analyse von ERF-Transkriptionsfaktoren aus N. tabacum und A. thaliana im Rahmen der Pathogenresistenz Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultäten
Funktionelle Analyse von ERF-Transkriptionsfaktoren aus N. tabacum und A. thaliana im Rahmen der Pathogenresistenz Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultäten
8 Anhang. 8.1 Vektorkarten und verwendete Sequenzen 8.1.1 Vektorkarten. Anhang 90
 Anhang 90 8 Anhang 8.1 Vektorkarten und verwendete Sequenzen 8.1.1 Vektorkarten 8.1.1.1 Expression in Sf9-Zellen durch Baculoviren pfastbac1 mit GST (Firma Invitrogen) Anhang 91 pbsv-8his mit Signalpeptid
Anhang 90 8 Anhang 8.1 Vektorkarten und verwendete Sequenzen 8.1.1 Vektorkarten 8.1.1.1 Expression in Sf9-Zellen durch Baculoviren pfastbac1 mit GST (Firma Invitrogen) Anhang 91 pbsv-8his mit Signalpeptid
HLA-READY GENE A Low SSP HLA-A low resolution
 IN VITRO DIAGNOSTICUM LOT G950085 Arbeitsprotokoll / PCR Protocol 2006-12 0123 Test-Durchführung / -Performance Datum / Date Unterschrift / Signature Bemerkungen / Comments Probe / Sample 1 5 9 13 17 2
IN VITRO DIAGNOSTICUM LOT G950085 Arbeitsprotokoll / PCR Protocol 2006-12 0123 Test-Durchführung / -Performance Datum / Date Unterschrift / Signature Bemerkungen / Comments Probe / Sample 1 5 9 13 17 2
Kartierung der Interaktionen peroxisomaler Membranproteine in Saccharomyces cerevisiae
 Kartierung der Interaktionen peroxisomaler Membranproteine in Saccharomyces cerevisiae Inaugural-Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Universität
Kartierung der Interaktionen peroxisomaler Membranproteine in Saccharomyces cerevisiae Inaugural-Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Universität
Material. Verwendungszweck WB: Western Blot FACS:Durchflusszytometrische Analyse IF: Immunfluoreszenz TZS: T-Zell Stimulation.
 2 Material 2.1 Antikörper Antikörper und Bindungsmoleküle Klon Verwendungszweck WB: Western Blot :Durchflusszytometrische Analyse IF: Immunfluoreszenz TZS: T-Zell Stimulation Lieferant anti-cd16/32 mak
2 Material 2.1 Antikörper Antikörper und Bindungsmoleküle Klon Verwendungszweck WB: Western Blot :Durchflusszytometrische Analyse IF: Immunfluoreszenz TZS: T-Zell Stimulation Lieferant anti-cd16/32 mak
Proteasenexpression einzelner disseminierter Tumorzellen aus Lymphknoten und Knochenmark von Patienten mit nichtkleinzelligem Bronchialkarzinom
 Aus dem Institut für Immunologie der Medizinischen Fakultät der Ludwig-Maximilians-Universität München Kommissarischer Vorstand: Prof. Dr. rer.nat. Thomas Brocker Proteasenexpression einzelner disseminierter
Aus dem Institut für Immunologie der Medizinischen Fakultät der Ludwig-Maximilians-Universität München Kommissarischer Vorstand: Prof. Dr. rer.nat. Thomas Brocker Proteasenexpression einzelner disseminierter
2. Material und Methoden
 2. Material und Methoden 2.1 Verwendete Materialien 2.1.1 Chemikalien und Enzyme Alle verwendeten Chemikalien wurden, sofern nicht anders angegeben, bei Sigma (München), Roth (Karlsruhe), Fluka (Buchs,
2. Material und Methoden 2.1 Verwendete Materialien 2.1.1 Chemikalien und Enzyme Alle verwendeten Chemikalien wurden, sofern nicht anders angegeben, bei Sigma (München), Roth (Karlsruhe), Fluka (Buchs,
Transformation und molekulargenetische Analyse der Diatomee Cylindrotheca fusiformis
 Biochemisches Grosspraktikum der Universität Regensburg Teil C: Molekularbiologie Transformation und molekulargenetische Analyse der Diatomee Cylindrotheca fusiformis Stand Dezember 07 Inhalt 1. Einleitung...1
Biochemisches Grosspraktikum der Universität Regensburg Teil C: Molekularbiologie Transformation und molekulargenetische Analyse der Diatomee Cylindrotheca fusiformis Stand Dezember 07 Inhalt 1. Einleitung...1
Epidemiologische und molekulare Studien zu PCV bei Wildschweinen deutscher Reviere
 Epidemiologische und molekulare Studien zu PCV bei Wildschweinen deutscher Reviere S. Knell, H. Willems, G. Reiner Professur für Schweinekrankheiten JLU Gießen Circoviren Taxonomie Familie: Circoviridae
Epidemiologische und molekulare Studien zu PCV bei Wildschweinen deutscher Reviere S. Knell, H. Willems, G. Reiner Professur für Schweinekrankheiten JLU Gießen Circoviren Taxonomie Familie: Circoviridae
Optimierte konstante Domänen für die effektive Expression von Fab-Fragmenten in E. coli
 Aus dem Institut für Biochemie der Medizinischen Fakultät Charité Universitätsmedizin Berlin DISSERTATION Optimierte konstante Domänen für die effektive Expression von Fab-Fragmenten in E. coli zur Erlangung
Aus dem Institut für Biochemie der Medizinischen Fakultät Charité Universitätsmedizin Berlin DISSERTATION Optimierte konstante Domänen für die effektive Expression von Fab-Fragmenten in E. coli zur Erlangung
1. Einleitung: 1. Einleitung. 1.1 Allgemeine Biologie der Echinokokken:
 1. Einleitung 1. Einleitung: 1.1 Allgemeine Biologie der Echinokokken: Der Genus Echinococcus gehört phylogenetisch dem Stamm der Plathelminthes (Plattwürmer), der Klasse der Cestoda (Bandwürmer) und der
1. Einleitung 1. Einleitung: 1.1 Allgemeine Biologie der Echinokokken: Der Genus Echinococcus gehört phylogenetisch dem Stamm der Plathelminthes (Plattwürmer), der Klasse der Cestoda (Bandwürmer) und der
E. coli B, F -, dcm, ompt, hsds(r B- m B- ), galλ (DE3) [Codon + ]
![E. coli B, F -, dcm, ompt, hsds(r B- m B- ), galλ (DE3) [Codon + ] E. coli B, F -, dcm, ompt, hsds(r B- m B- ), galλ (DE3) [Codon + ]](/thumbs/62/47608304.jpg) Material und Methoden 18 2 Material und Methoden 2.1 Material 2.1.1 Bakterienstämme Stamm Escherichia coli BL21(DE3) Codon + Escherichia coli XL1-blue Genotyp E. coli B, F -, dcm, ompt, hsds(r B- m B-
Material und Methoden 18 2 Material und Methoden 2.1 Material 2.1.1 Bakterienstämme Stamm Escherichia coli BL21(DE3) Codon + Escherichia coli XL1-blue Genotyp E. coli B, F -, dcm, ompt, hsds(r B- m B-
3. Methoden. 3.1 Chemikalien und Geräte
 3. Methoden Aufgabenstellung: Ziel der Arbeit war es eine eigene Multiplexreaktion zu erstellen, die im Rechtsmedizinischen Institut der Martin-Luther-Universität Halle-Wittenberg zur Durchführung von
3. Methoden Aufgabenstellung: Ziel der Arbeit war es eine eigene Multiplexreaktion zu erstellen, die im Rechtsmedizinischen Institut der Martin-Luther-Universität Halle-Wittenberg zur Durchführung von
Inaugural-Dissertation. zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Heinrich-Heine-Universität Düsseldorf
 Feinkartierung eines QTL (Quantitative Trait Locus) für Kühletoleranz auf Chromosom 4 in Mais und dessen molekularbiologische und phänotypische Charakterisierung. Inaugural-Dissertation zur Erlangung des
Feinkartierung eines QTL (Quantitative Trait Locus) für Kühletoleranz auf Chromosom 4 in Mais und dessen molekularbiologische und phänotypische Charakterisierung. Inaugural-Dissertation zur Erlangung des
Klonierung und Charakterisierung einer Adenylatcyclase aus Plasmodium falciparum
 Klonierung und Charakterisierung einer Adenylatcyclase aus Plasmodium falciparum Dissertation der Fakultät für Chemie und Pharmazie der Eberhard-Karls-Universität Tübingen zur Erlangung des Grades eines
Klonierung und Charakterisierung einer Adenylatcyclase aus Plasmodium falciparum Dissertation der Fakultät für Chemie und Pharmazie der Eberhard-Karls-Universität Tübingen zur Erlangung des Grades eines
Das GAF-Domänen-Protein NreA - ein neuartiger Nitratrezeptor aus Staphylococcus carnosus
 Das GAF-Domänen-Protein NreA - ein neuartiger Nitratrezeptor aus Staphylococcus carnosus DISSERTATION ZUR ERLANGUNG DES GRADES DOKTOR DER NATURWISSENSCHAFTEN Am Fachbereich Biologie der Johannes Gutenberg-Universität
Das GAF-Domänen-Protein NreA - ein neuartiger Nitratrezeptor aus Staphylococcus carnosus DISSERTATION ZUR ERLANGUNG DES GRADES DOKTOR DER NATURWISSENSCHAFTEN Am Fachbereich Biologie der Johannes Gutenberg-Universität
2 Materialien. Materialien 15. 2.1 Histondeacetylaseinhibitoren (HDIs) Sigma-Aldrich, Deutschland. 2.2 Zelllinien und Zellkultur.
 Materialien 15 2 Materialien 2.1 Histondeacetylaseinhibitoren (HDIs) Substanz Butyrat Trichostatin A (TSA) Valproat Hersteller Sigma-Aldrich, Sigma-Aldrich, Sigma-Aldrich, 2.2 Zelllinien und Zellkultur
Materialien 15 2 Materialien 2.1 Histondeacetylaseinhibitoren (HDIs) Substanz Butyrat Trichostatin A (TSA) Valproat Hersteller Sigma-Aldrich, Sigma-Aldrich, Sigma-Aldrich, 2.2 Zelllinien und Zellkultur
Bacteriocine in Streptococcus pneumoniae
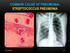 Bacteriocine in Streptococcus pneumoniae Vom Fachbereich Biologie der Technischen Universität Kaiserslautern zur Verleihung des akademischen Grades Doktor der Naturwissenschaften genehmigte Dissertation
Bacteriocine in Streptococcus pneumoniae Vom Fachbereich Biologie der Technischen Universität Kaiserslautern zur Verleihung des akademischen Grades Doktor der Naturwissenschaften genehmigte Dissertation
1. Zusammensetzung von Colletotrichum-Nährmedien
 VII Anhang -116- VII Anhang 1. Zusammensetzung von Colletotrichum-Nährmedien Haferflockenagar 50 g Haferflocken werden mit 500 ml H 2 O dest in einem Mixer sechsmal für 20 sec auf höchster Stufe zerkleinert.
VII Anhang -116- VII Anhang 1. Zusammensetzung von Colletotrichum-Nährmedien Haferflockenagar 50 g Haferflocken werden mit 500 ml H 2 O dest in einem Mixer sechsmal für 20 sec auf höchster Stufe zerkleinert.
Die funktionelle Rolle der Transkriptionsfaktoren bzip1 und bzip53 in der Arabidopsis thaliana-wurzel nach Salzstress
 Die funktionelle Rolle der Transkriptionsfaktoren bzip1 und bzip53 in der Arabidopsis thaliana-wurzel nach Salzstress Dissertation zur Erlangung des naturwissenschaftlichen Doktorgrades der Julius-Maximilians-Universität
Die funktionelle Rolle der Transkriptionsfaktoren bzip1 und bzip53 in der Arabidopsis thaliana-wurzel nach Salzstress Dissertation zur Erlangung des naturwissenschaftlichen Doktorgrades der Julius-Maximilians-Universität
Alle Primer wurden von der Firma Biotez GmbH (Berlin-Buch, BRD) bezogen. Die Darstellung der Primer erfolgt in 5 3 -Richtung.
 Material 28 2. Material 2.1. Synthetische Oligonukleotid-Primer Alle Primer wurden von der Firma Biotez GmbH (Berlin-Buch, BRD) bezogen. Die Darstellung der Primer erfolgt in 5 3 -Richtung. Primer für
Material 28 2. Material 2.1. Synthetische Oligonukleotid-Primer Alle Primer wurden von der Firma Biotez GmbH (Berlin-Buch, BRD) bezogen. Die Darstellung der Primer erfolgt in 5 3 -Richtung. Primer für
Gentechnik/Gentechnologie
 Gentechnik/Gentechnologie Bei zelltechnischen Verfahren werden Gene wenn überhaupt, dann nur im Gesamtverband, also als ganzes Genom verschoben oder kombiniert. Bei Gentechnologischen Verfahren wird in
Gentechnik/Gentechnologie Bei zelltechnischen Verfahren werden Gene wenn überhaupt, dann nur im Gesamtverband, also als ganzes Genom verschoben oder kombiniert. Bei Gentechnologischen Verfahren wird in
Universitätsklinikum Essen Medizinische Fakultät der Universität-Gesamthochschule Duisburg-Essen. Aus dem Institut für Humangenetik
 Universitätsklinikum Essen Medizinische Fakultät der Universität-Gesamthochschule Duisburg-Essen Aus dem Institut für Humangenetik Deletionen und Insertionen des RB1-Gens bei Patienten mit Retinoblastom:
Universitätsklinikum Essen Medizinische Fakultät der Universität-Gesamthochschule Duisburg-Essen Aus dem Institut für Humangenetik Deletionen und Insertionen des RB1-Gens bei Patienten mit Retinoblastom:
Nachweis und genetische Analyse der cag Pathogenitätsinsel. molekularbiologischen Methoden. Inaugural-Dissertation
 Aus dem Institut für Medizinische Mikrobiologie und Hygiene der Albert-Ludwigs-Universität Freiburg im Breisgau Nachweis und genetische Analyse der cag Pathogenitätsinsel von Helicobacter pylori mit neuen,
Aus dem Institut für Medizinische Mikrobiologie und Hygiene der Albert-Ludwigs-Universität Freiburg im Breisgau Nachweis und genetische Analyse der cag Pathogenitätsinsel von Helicobacter pylori mit neuen,
Generierung von Konsensusklonen für die Etablierung eines Replikonsystems nach Amplifikation klinisch charakterisierter Hepatitis C Virus Isolate
 Aus dem Fachbereich Medizin der Johann Wolfgang Goethe-Universität Frankfurt am Main Klinik für Innere Medizin II / Gastroenterologie und Hepatologie Direktor Prof. Dr. med. W. F. Caspary Generierung von
Aus dem Fachbereich Medizin der Johann Wolfgang Goethe-Universität Frankfurt am Main Klinik für Innere Medizin II / Gastroenterologie und Hepatologie Direktor Prof. Dr. med. W. F. Caspary Generierung von
Funktionelle Genomik. praktische Anwendungen
 Funktionelle Genomik praktische Anwendungen Schmelzpunkt der DNA Einzelsträngige DNA absorbiert UV-Licht effektiver SYBR Green fluoresziert nur wenn es an doppelsträngiger DNA bindet Fluoreszenz erhöht
Funktionelle Genomik praktische Anwendungen Schmelzpunkt der DNA Einzelsträngige DNA absorbiert UV-Licht effektiver SYBR Green fluoresziert nur wenn es an doppelsträngiger DNA bindet Fluoreszenz erhöht
Universitätsklinikum Hamburg-Eppendorf
 Universitätsklinikum Hamburg-Eppendorf Zentrum für Innere Medizin, III. Medizinische Klinik und Poliklinik Direktor: Prof. Dr. med. Rolf A. K. Stahl Häufigkeit von seltenen Varianten im Lipoproteinlipase
Universitätsklinikum Hamburg-Eppendorf Zentrum für Innere Medizin, III. Medizinische Klinik und Poliklinik Direktor: Prof. Dr. med. Rolf A. K. Stahl Häufigkeit von seltenen Varianten im Lipoproteinlipase
Methodensammlung der Bund/Länder-Arbeitsgemeinschaft Gentechnik. PCR- Nachweis von E. coli B- und BL21- Stämmen
 Methodensammlung der Bund/Länder-Arbeitsgemeinschaft Gentechnik PCR- Nachweis von E. coli B- und BL21- Stämmen Erstellt vom Unterausschuss Methodenentwicklung der LAG, März 2005 1. Zweck und Anwendungsbereich
Methodensammlung der Bund/Länder-Arbeitsgemeinschaft Gentechnik PCR- Nachweis von E. coli B- und BL21- Stämmen Erstellt vom Unterausschuss Methodenentwicklung der LAG, März 2005 1. Zweck und Anwendungsbereich
Aus dem Institut für Medizinische Virologie und Epidemiologie der Viruskrankheiten der Universität Tübingen Direktor: Professor Dr. G.
 Aus dem Institut für Medizinische Virologie und Epidemiologie der Viruskrankheiten der Universität Tübingen Direktor: Professor Dr. G. Jahn Vergleichende Sequenzanalyse der Tegumentproteine UL24, UL32,
Aus dem Institut für Medizinische Virologie und Epidemiologie der Viruskrankheiten der Universität Tübingen Direktor: Professor Dr. G. Jahn Vergleichende Sequenzanalyse der Tegumentproteine UL24, UL32,
Die Rolle der microrna-29 während der Leberfibrogenese
 Die Rolle der microrna-29 während der Leberfibrogenese Inaugural-Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Universität zu Köln vorgelegt von Monika
Die Rolle der microrna-29 während der Leberfibrogenese Inaugural-Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Universität zu Köln vorgelegt von Monika
Klinik und Poliklinik für Psychiatrie und Psychotherapie der Universität Würzburg Direktor: Professor Dr. med. H. Beckmann
 Klinik und Poliklinik für Psychiatrie und Psychotherapie der Universität Würzburg Direktor: Professor Dr. med. H. Beckmann Mutationsanalyse der Gene Connexin 36 (CX36) und Tyrosinkinase 3 (TYRO3) als Kandidatengene
Klinik und Poliklinik für Psychiatrie und Psychotherapie der Universität Würzburg Direktor: Professor Dr. med. H. Beckmann Mutationsanalyse der Gene Connexin 36 (CX36) und Tyrosinkinase 3 (TYRO3) als Kandidatengene
Charakterisierung des Chrysovirus FgV-ch9 aus Fusarium graminearum: Sequenzen, Synthese und Funktion der viralen Proteine
 Charakterisierung des Chrysovirus FgV-ch9 aus Fusarium graminearum: Sequenzen, Synthese und Funktion der viralen Proteine Dissertation zur Erlangung der Würde des Doktors der Naturwissenschaften an der
Charakterisierung des Chrysovirus FgV-ch9 aus Fusarium graminearum: Sequenzen, Synthese und Funktion der viralen Proteine Dissertation zur Erlangung der Würde des Doktors der Naturwissenschaften an der
Charakterisierung und Lokalisation der Toxoplasma gondii Katalase: Peroxisomen in Apicomplexa?
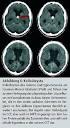 Aus dem Institut für Hygiene und Mikrobiologie der Universität Würzburg Vorstand: Prof. Dr. med. M. Frosch Charakterisierung und Lokalisation der Toxoplasma gondii Katalase: Peroxisomen in Apicomplexa?
Aus dem Institut für Hygiene und Mikrobiologie der Universität Würzburg Vorstand: Prof. Dr. med. M. Frosch Charakterisierung und Lokalisation der Toxoplasma gondii Katalase: Peroxisomen in Apicomplexa?
Die Rolle der Autoimmunreaktion gegen das SLA/LP - Molekül bei autoimmuner Hepatitis
 Die Rolle der Autoimmunreaktion gegen das SLA/LP - Molekül bei autoimmuner Hepatitis Dissertation zur Erlangung des Grades Doktor der Naturwissenschaften am Fachbereich Biologie der Johannes Gutenberg-Universität
Die Rolle der Autoimmunreaktion gegen das SLA/LP - Molekül bei autoimmuner Hepatitis Dissertation zur Erlangung des Grades Doktor der Naturwissenschaften am Fachbereich Biologie der Johannes Gutenberg-Universität
