Bioinformatik: Neue Paradigmen in der Forschung Sommerakademie der Studienstiftung
|
|
|
- Erica Weiß
- vor 7 Jahren
- Abrufe
Transkript
1 Bioinformatik: Neue Paradigmen in der Forschung Sommerakademie der Studienstiftung Thema 4: Paarweises und multiples (heuristisches) Alignment Tobias Thierer St. Johann, Zusammenfassung Eine der grundlegendsten und interessantesten Fragen in der modernen Biologie ist die, ob und auf welche Weise zwei oder mehrere Gene bzw. Proteine homolog, also im Laufe eines evolutionären Prozesses auseinander oder aus einem gemeinsamen Vorfahr hervorgegangen sind. Dabei interessiert nicht nur, wie groß der,,abstand zwischen den Nukleotid- oder Aminosäuresequenzen ist, sondern auch, welche Bereiche stärker und weniger stark konserviert wurden. Ein Alignment kann diese Fragen beantworten und ermöglicht Rückschlüsse auf Funktion und evolutionäre Entstehungsgeschichte von Genen und Proteinen. In dieser Arbeit werden erstens Bewertungsfunktionen für paarweise und multiple Sequenzalignments und zweitens Algorithmen vorgestellt, die bezüglich dieser Bewertungsfunktionen gute Alignments konstruieren. Im Falle des paarweisen Sequenzalignments wird eine Familie von Algorithmen vorgestellt, die mit dynamischer Programmierung das Alignmentproblem optimal lösen. Beim multiplen Alignment dagegen ist wegen den viel größeren Suchräumen ein Ausweichen auf Heuristiken wie zum Beispiel greedy Algorithmen, genetische Algorithmen oder Branch & Bound mit heuristischen Beschneidungen des Suchbaums nötig. 1
2 Inhaltsverzeichnis 1 Einleitung Formale Definitionen Motivation Paarweises Alignment Bewertungsfunktion Einleitung Grundansatz Zwei Modelle bzw. Hypothesen Übertragung auf die Bewertungsfunktion Bestimmung der Wahrscheinlichkeiten p ab ; PAM-Matrizen Exakte Lösung mit dynamischer Programmierung Globales Alignment (Needleman-Wunsch) Lokales Alignment (Smith-Waterman) Weitere Varianten Laufzeit und Speicherbedarf Multiples Alignment Progressiver versus globaler Ansatz Bewertungsfunktion Bewertungsfunktion für k-tupel von Zeichen SP-Score Coffee Maximum Weight Trace Progressive Verfahren CLUSTALW T-Coffee Andere Verfahren Dynamische Programmierung Branch&Bound für Maximum Weight Trace
3 1 Einleitung Im folgenden wollen wir uns mit dem Alignment von (z.b. Nukleotid- oder Aminosäuren-) Sequenzen beschäftigen. Ein Alignment ist das,,untereinanderschreiben zweier oder mehrere Sequenzen derart, dass möglichst wenige Lücken eingefügt werden müssen und meist nur gleiche Zeichen in einer Spalte stehen. Ein Alignment zweier kurzer Nukleotidsequenzen könnte z.b. so aussehen: 1.1 Formale Definitionen A-GTATAATTAC ACATATAAT--C Definition 1.1 (Alignment) Sei Σ ein endliches Alphabet und ˆΣ = Σ { }, wobei,, ein spezielles Symbol zur Darstellung von Lücken (gaps) ist. Die Eingabe für einen Alignierungsalgorithmus bestehe aus einer Menge S = {S 1, S 2,..., S k } von k endlichen Zeichenketten (Strings) über dem Alphabet Σ. Die Länge der Strings sei S i =: n i. Eine Menge Ŝ = {Ŝ1, Ŝ2,..., Ŝk} von Zeichenketten über dem Alphabet ˆΣ ist ein Alignment von S genau dann, wenn die folgenden beiden Bedingungen erfüllt sind: (1) Alle Strings in Ŝ haben die gleiche Länge. (2) Abgesehen von Lücken,, ist i: S i = Ŝi. Ist k = 2, so spricht man von einem paarweisen Alignment, für k > 2 von einem multiplen Alignment. Ein Alignment kann als eine Tabelle mit k Zeilen angesehen werden, die aus jeweils einem Ŝ i bestehen. Zwei Zeichen verschiedener S i heißen aligniert unter Ŝ, wenn sie in der gleichen Spalte liegen. Definition 1.2 (optimales Alignment) Ein optimales Alignment ist eines, das eine maximale Punktzahl bezüglich einer Bewertungsfunktion ergibt. Beim Alignieren von DNA- oder Proteinsequenzen erhöhen typischerweise identische oder ähnliche 1 alignierte Zeichen die Bewertung, Lücken und unterschiedliche alignierte Zeichen verringern sie. Das Problem der automatischen Konstruktion guter Alignments gliedert sich in zwei Teilprobleme: 1. Aufstellen einer geeigneten Kostenfunktion, die gute Alignments erkennbar macht. 2. Entwicklung und Implementierung eines Algorithmus, der zu einer Menge von Eingabesequenzen ein bezüglich der Kostenfunktion ganz oder nahezu optimales Alignment findet. 1.2 Motivation Eine der grundlegendsten Fragen in der Genetik und Molekularbiologie ist, ob zwei oder mehrere Sequenzen einen gemeinsamen evolutionären Ursprung haben (d.h. ob sie homolog sind) oder ob sie unabhängig entstanden. Ferner interessiert, welche Bereiche der Sequenzen besonders ähnlich sind 2 und in welchen Bereichen sie sich stärker unterscheiden. Optimale Alignments zweier oder mehrerer DNA- oder Aminosäurensequenzen 1 Ähnlichkeit kann sich bei Aminosäuren z.b. auf Hydrophobizität o.ä. beziehen. 2 Typischerweise sind dies Schlüsselpositionen bezüglich der Funktionalität des Gens bzw. Proteins. 3
4 erleichtern die Identifikation homologer DNA-/Proteinbereiche. geben über die Bewertung des Alignments ein Maß für den evolutionären Abstand der Sequenzen bzw. für die Wahrscheinlichkeit dafür, dass sie unabhängig voneinander entstanden sind. ermöglichen Rückschlüsse auf die Funktion des Gens/Proteins, denn eng verwandte Proteine haben oft auch ähnliche Funktionen. geben ein Maß dafür, wie stark bestimmte Positionen der Sequenzen variabel bzw. konserviert sind. Stark konservierte Bereiche sind tendenziell auch funktionell kritisch. 2 Paarweises Alignment In diesem Kapitel konzentrieren wir uns auf paarweises Sequenzalignment. Ziel ist also die Alignierung zweier Sequenzen x, y der Länge n bzw. m. Das Alphabet heiße nach wie vor Σ, und ˆΣ := Σ { } enthält zusätzlich das Symbol für Lücken. Zunächst wird ein Schema für eine Bewertungsfunktion vorgestellt. Anschließend wird erläutert, wie diese geeignet parametrisiert wird und auf welchem statistischen Modell sie beruht. Im zweiten Teil wird beschrieben, wie mit Hilfe dynamischer Programmierung das bezüglich dieser Bewertungsfunktion optimale globale und lokale Alignment gefunden wird und wie der Algorithmus für affine Lückenkosten angepasst werden kann. 2.1 Bewertungsfunktion Einleitung Jedes Alignment definiert eine Folge von Mutationen, die die beiden Eingabesequenzen ineinander überführen. Lücken im Alignment entsprechen dabei Insertionen bzw. Deletionen, alignierte, aber verschiedene Zeichen (mismatches) entsprechen einer Punktmutation. Eine gute Bewertungsfunktion sollte einem Alignment, das eine biologisch besonders plausible Erklärung für den evolutionären Zusammenhang zweier Sequenzen gibt also z.b. mit möglichst wenigen Mismatches und Lücken auskommt eine hohe Punktzahl zuordnen. In unserem Modell wird die Bewertung dem Logarithmus der relativen Wahrscheinlichkeit für einen evolutionären Zusammenhang zwischen den Sequenzen darstellen, im Verhältnis zur Wahrscheinlichkeit für eine unabhängige Entwicklung Grundansatz Damit das statistische Modell nicht zu kompliziert und der Suchraum nicht zu groß ist, werden wir eine vereinfachenden Annahme machen. Wir nehmen an, dass alle Positionen in den Sequenzen voneinander unabhängig evolvieren. Die Gesamtbewertung für ein Alignment ist dann die Summe der Bewertungen für die einzelnen Spalten, hier also für Paare von Zeichen. Wir benötigen also lediglich eine Funktion s: ˆΣ 2 Z, die jedem Paar von Zeichen eine Bewertung zuordnet. Die Gesamtbewertung eines Alignments ist dann: S(ˆx, ŷ) = i s(ˆx i, ŷ i ) Zwei Modelle bzw. Hypothesen Erinnern wir uns noch einmal an unsere ursprüngliche Motivation. Wir wollen entscheiden, ob ein evolutionärer Zusammenhang zwischen zwei Sequenzen besteht (sie also homolog sind) oder nicht. Wir müssen also entscheiden, welche der beiden Hypothesen 4
5 M Die beiden Sequenzen sind homolog. R Die beiden Sequenzen sind unabhängig entstanden, jede Übereinstimmung ist reiner Zufall. plausibler ist. Das Modell R (random) ist das einfachste. Dieses Modell nimmt an, dass jedes Zeichen einfach zufällig aus einem großen Vorrat herausgegriffen wird. Hier sind also nicht nur die einzelnen Spalten eines Alignments voneinander unabhängig, sondern auch alle Sequenzen. Um die Wahrscheinlichkeit des Auftretens einer bestimmten Zeichenpaarung unter diesem Modell zu bestimmen, müssen wir nur die relative Häufigkeit p x aller Zeichen x Σ kennen. Die Wahrscheinlichkeit P (a, b R), das Paar (a, b) in zufälligen untereinanderliegenden Sequenzen zu beobachten, ist dann einfach p a p b. Das Modell M (match) ist ein wenig komplizierter. Hier nehmen wir an, dass die beiden Sequenzen homolog ist und wollen für zwei gegebene Zeichen a und b die Wahrscheinlichkeit P (a, b M) =: p ab wissen, dass diese in homologen, alignierten Sequenzen untereinander auftreten. Die Bestimmung dieses Wertes werden wir noch ein wenig verschieben; nehmen wir also zunächst mal an, wir würden die Werte p ab bereits kennen Übertragung auf die Bewertungsfunktion Das Wahrscheinlichkeitsverhältnis (odds ratio) P (a, b M) P (a, b R) = p ab p a p b ist dann ein Maß dafür, wie viel wahrscheinlicher es ist, diese Alignierung bei evolutionär verwandten als bei unabhängig evolvierten Sequenzen zu beobachten. Für die Gesamtsequenz wäre dieses Verhältnis dann i p xi y i p xi p yi Um die Multiplikation vieler kleiner Zahlen zu vermeiden 3, addieren wir statt dessen die Logarithmen dieser Verhältnisse 4, um die Bewertung eines Alignments zu bestimmen: S(x, y) = log i p xi y i p xi p yi = i p x i y i log p xi p yi Ist p xi y i > p xi p yi (ein evolutionärer Zusammenhang ist also plausibler als die Unabhängigkeit), so ist der Logarithmus > 0, und es wird eine positive Punktzahl vergeben. Andernfalls sinkt der Wert des Alignments Bestimmung der Wahrscheinlichkeiten p ab ; PAM-Matrizen Zu bestimmen ist noch die Wahrscheinlichkeit, dass in einem Alignment homologer Zeichen die Buchstaben a und b übereinander liegen. Diesen Wert kann man empirisch aus manuellen Alignments ableiten. Man muss allerdings beachten, dass p ab von der evolutionären Distanz der betrachteten Sequenzen abhängt. Wenn die Sequenzen nur sehr kurz getrennt evolvierten, so wird p ab für a = b sehr nahe bei 1 und für a b sehr nahe bei 0 sein. Für sehr lang getrennt evolvierte Sequenzen wird dagegen kaum noch ein Unterschied zu unabhängigen Sequenzen feststellbar sein, dann ist also p ab p a p b. Zunächst definiert man sich also eine Zeiteinheit, sozusagen die Dauer zwischen zwei Ticks der biologischen Uhr. Sei t = 1 die Zeit, in der im Durchschnitt 1% der Zeichen ersetzt werden. 3 Durch die Multiplikation würden immer kleinere Zahlen entstehen, was selbst bei Fließkommazahlen schnell zu einem Unterlauf führen würde. 4 Die sogenannten log odds ratios. 5
6 Mit Hilfe manuell alignierter Sequenzen dieses evolutionären Abstandes kann man nun einfach die Wahrscheinlichkeit p ab als relative Häufigkeit bestimmen. Die hieraus gemäß s(a, b) = log p ab p a p b (1) entwickelte Distanzmatrix ist die sogenannte PAM1-Matrix. Wir werden nun sehen, wie weitere PAM-Matrizen für längere Zeitabschnitte entwickelt werden. Aus p ab erhält man auch die Wahrscheinlichkeit P (b a, t = 1) = p ab(t = 1) p a dass in einem Zeitschritt ein zu Beginn vorhandenes a durch ein b substituiert wird. Für größere Zeitintervalle ergibt sich P (b a, t = 2) = c P (b c, t = 1) P (c a, t = 1) Trägt man die Werte P (b a, t = 1) in eine Matrix ein, so erhält man P (b a, t = t ) durch Potenzierung der Matrix mit t. Für t = 250 erhält man nach Einsetzen in (1) die sehr häufig verwendete PAM250 Matrix. 2.2 Exakte Lösung mit dynamischer Programmierung Globales Alignment (Needleman-Wunsch) Betrachten wir einige Eigenschaften optimaler Alignments A opt (i, j) von Präfixen x 1...i, y 1...j von Sequenzen x und y. Lemma 2.1 Ist in A opt (i, j) x i mit y j aligniert, so ist auch das darin enthaltene Teilalignment von x 1...i 1 mit y 1...j 1 optimal. Beweis Angenommen, das Teilalignment wäre nicht optimal, also A opt (i 1, j 1). Das Teilalignment hat die Bewertung S(A opt (i, j)) s(x i, y j ). Wähle nun das optimale Alignment A opt (i 1, j 1) und füge am rechten Rand die Spalte x i, y j an. Wegen S(A opt (i 1, j 1)) > S(A opt (i, j)) s(x i, y j ) ist der Wert dieses neuen Alignments von x 1...i mit y 1...j S(A opt (i 1, j 1)) + s(x i, y j ) > S(A opt (i, j)) Widerspruch zur Annahme, dass A opt (i, j) das optimale Alignment von x 1...i mit y 1...j ist. Genau gleich laufen die Beweise zu folgenden Lemmata: Lemma 2.2 Ist in A opt (i, j) x i mit ( gap) aligniert, so ist auch das darin enthaltene Teilalignment von x 1...i 1 mit y 1...j optimal. Lemma 2.3 Ist in A opt (i, j) y j mit ( gap) aligniert, so ist auch das darin enthaltene Teilalignment von x 1...i mit y 1...j 1 optimal. Daraus ergibt sich sofort folgender Satz 1 Entfernt man aus einem optimalen Alignment von Präfixen die letzte Spalte, so entsteht wieder ein optimales Alignment von Präfixen. Jedes optimale paarweise Alignment ist also auf eine von drei Möglichkeiten aus einem optimalen Teilalignment abgeleitet. Der Wert des Gesamtalignments ist der Wert des Teilalignments plus dem Wert der neuen Spalte. 6
7 Sei F (i, j) := S(A opt (i, j)) der optimale Wert eines Alignments der Präfixe x 1...i, y 1...j von Sequenzen x und y. Mit Hilfe von Satz 1 ergibt sich folgende Rekursionsformel für den Wert von F (i, j): F (i 1, j 1) + s(x i, y j ) (x i, y j ) alignieren F (i, j) = max F (i 1, j) d (x i, ) alignieren (2) F (i, j 1) d (, y j ) alignieren wobei d die Lückenkostens sind 5. Mit F (0, 0) = 0 und F (i, 0) = F (0, i) = i ergibt sich ein dynamischer Algorithmus zum Finden des optimalen Alignments. Der Algorithmus berechnet alle F (i, j) und speichert sie in eine Tabelle. Dazu initialisiert er zunächst mit den richtigen Werten für F (i, 0) und F (0, j) und füllt dann die ganze Tabelle der Reihe nach gemäß (2). Abbildung 1 verdeutlicht dies. Abbildung 1: Needleman-Wunsch-Algorithmus zur Bestimmung des optimalen paarweisen globalen Alignments. Im Beispiel aus Abbildung 1 wurden Mismatch- und Gap-Bewertungen von -1 und Matchbewertungen von +1 verwendet. Die roten und grünen Pfeile geben an, von welchem F (i, j ) aus ein bestimmter Wert F (i, j) konstruiert wurde (durch die Maximumsbildung sind ja außer in Randpositionen immer 3 Ausgangspositionen möglich). Blaue Pfeile geben potentielle Pfade an, die einen niedrigeren Wert ergeben hätten und darum bei der Maximumsbildung verworfen wurden. Die linke Bildhälfte zeigt eine Situation nah zu Beginn der Lösungsfindung, die rechte Bildhälfte die Situation am Ende. Der Wert des besten Alignments steht schließlich in der rechten unteren Ecke der Tabelle, an Position (i, j) Verfolgt man von dieser Position aus die Pfeile bis zum Ursprung, so kann man das Alignment rekonstruieren. Je nachdem, in welche Richtung der Pfeil geht, trat einer der drei Fälle aus (2) ein, und man fügt am Anfang des Alignments zwei alignierte Zeichen oder ein Zeichen und eine Lücke ein. Im Beispiel ergeben sich im Pfad Verzweigungsmöglichkeiten, da auf mehreren Wegen der gleiche Wert erzielt wurde. Dies entspricht gleichwertigen Alignments, im Beispiel drei Stück: AATGC AATGC AATGC AG-GC A-GGC -AGGC Lokales Alignment (Smith-Waterman) Oft ist nicht das beste Alignment der Gesamtsequenzen gesucht, sondern das beste lokale Alignment (Abb. 2), d.h. das Alignment von Teilsequenzen mit der höchsten Bewertung. 5 Sollen Lücken je nach Zeichen, mit dem sie gepaart sind, unterschiedlich bewertet werden, so muss man s(.,.) entsprechend erweitern und F (i 1, j) + s(x i, ) anstelle von F (i 1, j) d schreiben, entsprechend auch F (i, j 1) + s(, y j ) statt F (i, j 1) d. 7
8 Statt wie bisher ein Alignment nur auf drei verschiedene Weisen aus Teilalignments fortsetzen Abbildung 2: Ein lokales Alignment zu können, kann an jeder Stelle (i, j) in den Sequenzen auch ein neues Alignment begonnen werden. Darum muss die rekursive Definition der Bewertungsfunktion angepasst werden zu: 0 neu beginnen F (i 1, j 1) + s(x i, y j ) (x i, y j ) alignieren F (i, j) = max (3) F (i 1, j) d (x i, ) alignieren F (i, j 1) d (, y j ) alignieren Die Tabelle für die dynamische Programmierung enthält nun an Position (i, j) die Bewertung des besten Alignments, das irgendwo in x und y beginnt und mit x i und y j aufhört. Insbesondere kann das Alignment leer sein, so dass die Bewertung nie unter 0 fällt. Im Unterschied zum globalen Alignment kann aber auch das Ende des Alignments an einer anderen Position als (i, j) liegen. Darum muss zur Rekonstrukton des Alignments nun die höchste Bewertung in der gesamten Tabelle gesucht werden. Von dieser aus folgt man den Pfeilen bis zur Weitere Varianten Der Ansatz der dynamischen Programmierung kann durch Veränderung der Iterationsformel (2) und der Initialisierung nicht nur an die Bestimmung lokaler Alignments, sondern noch eine Vielzahl von anderen Varianten angepasst werden. Initialisiert man z.b. die erste Zeile und Spalte der Tabelle mit 0, verwendet (2) unverändert und sucht am Ende die beste Lösung nicht nur in der rechten unteren Ecke, sondern in der ganzen rechten Spalte und unteren Zeile der Tabelle, so erhält man das beste globale Alignment unter Vernachlässigung von,,überhängenden Enden. Dies ist z.b. sinnvoll, wenn die eine Sequenz (z.b. ein Genom) viel länger ist als die andere und die zweite Sequenz (z.b. ein Gen) komplett darin gesucht werden soll. Ein anderes Szenario ist das von affinen Lückenkosten. Hier soll das Eröffnen einer Lücke mehr kosten als die Fortführung, zwei kurze Lücken kosten also mehr als eine größere mit der gleichen Gesamtlänge. Dies entspricht durchaus dem tatsächlichen biologischen Modell und kann einfach in unseren Algorithmus integriert werden. Dazu merken wir uns in jeder Tabellenzelle nicht nur das Maximum der drei Werte aus der rechten Seite von (2), sondern alle drei: F (i, j a, b), F (i, j a, ) und F (i, j, b). (i, j a, ) soll dabei beispielsweise ein Alignment charakterisieren, das x 1...i und y 1...j umfasst und mit der Spalte (x i, ) endet. Dann kostet das Erreichen der Position (i, j a, ) aus der Position (i 1, j a, ) nur die Lückenerweiterungskosten, das Erreichen aus der Position (i 1, j a, b) dagegen die Lückeneröffnungskosten denn in letzterem Fall war die Lücke in der zweiten Sequenz ja schon offen. Durch eine andere Variation des Algorithmus kann man alle lokalen Alignments der Sequenz y mit einer Teilsequenz von x finden. Eine genauere Ausführung der Variationen würde den Rahmen dieser Ausarbeitung sprengen, kann aber in [1] nachgelesen werden. 8
9 2.2.4 Laufzeit und Speicherbedarf Laufzeit und Speicherbedarf des vorgestellten Algorithmus sind O( x y ). Der Speicherbedarf kann allerdings auf O( x + y ) gedrückt werden (siehe z.b. [2] Gusfield (1997), S ). 3 Multiples Alignment Ein multiples Alignment ist ein Alignment von mehr als zwei Sequenzen hier zum Beispiel Aminosäuersequenzen für Trypsin und Trypsin-Inhibitoren: EETI-II ----GCPRILMRCKQDSDCLAGCVCGPNG-FCGSP 30 Ii_Mutant ----GCPRLLMRCKQDSDCLAGCVCGPNG-FCG-- 28 BDTI-II ---RGCPRILMRCKRDSDCLAGCVCQKNG-YCG-- 29 CMeTI-B ---VGCPRILMKCKTDRDCLTGCTCKRNG-YCG-- 29 CMTI-IV HEERVCPRILMKCKKDSDCLAECVCLEHG-YCG-- 32 CSTI-IIB ---MVCPKILMKCKHDSDCLLDCVCLEDIGYCGVS 32 MRTI-I ---GICPRILMECKRDSDCLAQCVCKRQG-YCG-- 29 Trypsin ---RICPRIWMECTRDSDCMAKCICVAG--HCG-- 28 ITRA_MOMCH ---RSCPRIWMECTRDSDCMAKCICVAG--HCG-- 28 MCTI-A ---RICPRIWMECKRDSDCMAQCICVDG--HCG-- 28 LCTI-III ---RICPRILMECSSDSDCLAECICLENG-FCG-- 29 **:: *.*. * **: * *.** Die Zahlen am rechten Rand geben die Längen der Sequenzen an, in der letzten Zeile stehen Symbole für vollständige (*), sehr starke (:), starke (.) und weniger starke ( ) Konservierung der einzelnen Positionen. 3.1 Progressiver versus globaler Ansatz Die Verfahren für multiple Alignments lassen sich grob in zwei Kategorien einteilen. Progressive Verfahren bauen ein multiples Alignment sukzessive durch eine Folge von paarweisen Alignments auf. Dazu verschmelzen sie Teilalignments, die entweder einzelne Sequenzen oder ein multiples Alignment von einer Teilmenge der Sequenzen sind. Ein progressiver Ansatz könnte z.b. alle Einzelsequenzen nacheinander zu einem anfangs leeren Alignment hinzufügen oder aber in einer beliebigen anderen Reihenfolge erst einzelne Sequenzen alignieren und dann diese Alignments zu größeren Alignments zusammenfassen. Der Vorteil progressiver Ansätze ist, dass sie schneller und,,einfacher zu greifen sind und die Algorithmen und Bewertungsfunktionen für paarweises Alignment mehr oder weniger unverändert übernommen werden können. Der Nachteil progressiver Verfahren ist, dass sie gierig (greedy) den im begrenzten Blickwinkel paarweiser Alignments optimal erscheinenden Schritt wählen und darum häufig in einem frühen Stadium wegen der fehlenden globalen Sicht Fehler machen (also z.b. fälschlicherweise Lücken einfügen), die dann nicht mehr korrigiert werden können und sich in das Gesamtalignment fortpflanzen. Die andere Klasse von Algorithmen aligniert alle Sequenzen parallel. Die Schwierigkeit dabei ist das Finden einer geeigneten Bewertungsfunktion sowie der viel größere Suchraum, da die Zahl der möglichen Alignments mit steigender Anzahl von Sequenzen sehr schnell wächst. Die Wahl des Ansatzes (progressiv oder nicht) spielt auch eine Rolle für die Wahl von Bewertungsfunktionen. Im folgenden sollen nun zunächst Ansätze für Alignmentbewertungen und dann verschiedene Alignmentverfahren vorgestellt werden. 9
10 3.2 Bewertungsfunktion Wie auch schon bei paarweisen Alignments ist die Wahl einer geeigneten Bewertungsfunktion die halbe Miete, denn ein noch so guter Optimierungsalgorithmus bringt nichts, wenn die gefundenen,,optimalen Alignments tatsächlich nichts taugen. Die folgenden Abschnitte beschreiben verschiedene Ansätze, eine Bewertungsfunktion für multiple Alignments aufzustellen. Tatsächlich können, sobald man eine Bewertungsfunktion hat, ganz allgemeine Ansätze wie z.b. simulated annealing 6 zur Suche guter Lösungen eingesetzt werden. In [5] verwenden Notredame et al. z.b. einen genetischen Algorithmus, um bezüglich ihrer Bewertungsfunktion Coffee, die in Abschnitt vorgestellt wird, gute Alignments zu finden Bewertungsfunktion für k-tupel von Zeichen Der naheliegendste Gedanke wäre, in Analogie zum paarweisen Fall eine Bewertungstabelle aufzustellen, die jeder Spalte eines Aligments, also jedem k-tupel (c 1, c 2,..., c k ) von Zeichen aus ˆΣ eine Bewertung s(c 1, c 2,..., c k ) Z zuordnet. Die Bewertung des Alignments könnte dann wieder die Summe der Bewertungen für die Spalten sein. Dann bräuchte man aber für jedes k eine eigene Tabelle, deren ˆΣ k Parameter man mittels einer hinreichend großen Menge von manuell erstellten k-alignments statistisch abgesichert mit konkreten Werten belegen müsste. Für einigermaßen große k ist das nicht machbar, so dass man nach anderen Wegen sucht SP-Score Eine andere Möglichkeit der Verallgemeinerung ist der direkte Einsatz der paarweisen Bewertungsfunktion s(a, b). Der Wert einer Spalte ist dann die Summe der Werte aller Paare von Zeichen in dieser Spalte (sum-of-pairs Score, SP-Score). Diese Wahl der Bewertungsfunktion hat allerdings zwei Nachteile: Das Problem der Bestimmung des Alignments mit maximalem SP-Score ist NP-hart. Diese Definition der Bewertung ist keine konsequente Fortsetzung des Wahrscheinlichkeitsmodells, auf dem die s(a, b) Tabelle für paarweise Alignments basierte. Wollte man wieder die odd ratio, also das Verhältnis der Wahrscheinlichen, dieses Tupel in homologen bzw. zufälligen Sequenzen zu beobachten, bestimmen, so müsste die Bewertungstabelle z.b. für k = 3 Sequenzen Einträge der Form p abc log p a p b p c enthalten. Der SP-Score entspricht aber Einträgen der Form ( pab log + p ac + p ) ( ) bc pab p c + p ac p b + p bc p a = log p a p b p a p c p b p c p a p b p c Dieser Unterschied wird um so deutlicher, je größer k wird. Der SP-Score taugt also allenfalls für eine,,π mal Daumen Abschätzung der Qualität eines Alignments und ist nicht statistisch abgesichert. 6 Simulated annealing ist ein allgemeines Verfahren zum Lösen von Optimierungsproblemen, basierend auf einem Mutationsoperator und einer Bewertungsfunktion. Man startet mit einer zufällig oder mit einer Heuristik erzeugten Lösung. In jedem Zeitschritt verändert man die Lösung nun durch den Mutationsoperator und bewertet die so erzeugte neue Lösung. Ist die neue Lösung besser als die alte, so übernimmt man sie in jedem Fall; ist sie schlechter, so übernimmt man sie mit einer gewissen Wahrscheinlichkeit. Diese Wahrscheinlichkeit lässt man im Laufe der Zeit mit einer e-funktion langsam abfallen, und die Simulation wird abgebrochen, sobald über einen gewissen Zeitraum keine Verbesserung mehr erzielt wurde. 10
11 3.2.3 Coffee Coffee 7 wurde 1998 von Cédric Notredame et al. in [5] vorgestellt und verfolgt einen neuen, vielversprechenden Ansatz. Coffee bewertet ein Alignment nicht anhand einer Scoretabelle für Zeichentupel, sondern anhand einer Bibliothek von paarweisen Alignments der Einzelsequenzen 8. Die Bewertung eines multiplen Alignments ist um so höher, je konsistenter es mit den Paaralignments ist 9. Konservierte Regionen werden automatisch wesentlich stärker gewichtet, da sie in einem größeren Anteil der Paaralignments aligniert sind. Beachtenswert ist, dass die Bewertung hier jeweils spezifisch für Positionen in den Sequenzen ist, nicht für die dort stehenden Zeichen. Zunächst definiert man einen Unterstützungswert support(i, j) für jedes Paar von alignierten Sequenzen. Dieser Wert ist die Anzahl der Positionen von S i und S j, die sowohl im multiplen als auch im paarweisen Alignment aus der Bibliothek aligniert sind. Der Wert ist eine Ganzzahl zwischen 0 und len(i, j), der Länge des Alignments von S i mit S j aus dem multiplen Alignment Ŝ 10. Ferner führt man für jedes paarweise Aligment von (Ŝi, Ŝj) ein Gewicht w(i, j) ein. Dies ist ein Maß für die Güte des paarweisen Alignments von S i mit S j, und zwar einfach der Anteil der gap-freien Spalten im paarweisen Alignment, die zwei identische Zeichen enthalten. Ausgerüstet mit diesen Definitionen kann man nun den Coffee-Wert eines multiplen Alignments festlegen. Grob gesagt ist er der Anteil der Paare von im multiplen Alignment alignierten Positionen, der auch von dem zugehörigen paarweisen Alignment unterstützt (also in diesem vorhanden) ist gewichtet mit der Vertrauenswürdigkeit w(i, j) der paarweisen Alignments. Formal: CoffeeScore(Ŝ) = 1 i<j k w(i, j) support(i, j) 1 i<j k w(i, j) len(i, j) (4) Man kann die Bibliothek als Menge von Positionspaarungen auffassen. Jede Paarung einer Position aus Sequenz i mit einer Position aus Sequenz j ist mit dem Gewicht w(i, j) beschriftet. Die Bewertung eines Alignments ist dann die Summe der Gewichte aller vom Alignment realisierten Paarungen, die die Bibliothek enthält. Diese wird allerdings noch mit der Summe der gewichteten Sequenzlängen 11 normiert, so dass der Coffee-Wert hinterher stets im Intervall [0, 1] liegt. Man beachte den Unterschied von (4) zur vielleicht intuitiveren Formel 1 i<j k w(i, j) support(i, j) len(i, j) Dadurch, dass die Normierung bezüglich der Sequenzlänge erst nach der Gewichtung und Aufsummierung durchgeführt wird, erhält eine längere Sequenz auch entsprechend mehr Gewicht. 3.3 Maximum Weight Trace Wie gegen Ende von erwähnt, kann die dort verwendete Bibliothek von paarweisen Alignments als Liste von Positionspaarungen betrachtet werden, die jeweils mit einem bestimmten Gewicht markiert sind. Dieses Gewicht entspricht einer Punktzahl, die für die Realisierung speziell dieser Alignierung vergeben wird. 7 Consistency based Objective Function For alignment Evaluation 8 Der Coffee-Wert eines multiplen Alignments hängt also insbesondere von dem Algorithmus ab, mit dem die paarweisen Alignments erzeugt wurden. 9 Anders formuliert: je mehr die Bibliothek von Paaralignments das multiple Aligment unterstützt. 10 Eigentlich sind in Ŝ alle Sequenzen gleich lang, haben also eine Länge von Ŝ := Ŝi. Die Extrafunktion len(i, j) wird von [5] vermutlich eingeführt, um Folgen von Lücken am Anfang und Ende dieses Alignments zu ignorieren. 11 Das ist gerade die höchste erzielbare Punktzahl. 11
12 Vor diesem Hintergrund kann man die Positionen der k Sequenzen als Knoten in einem k- partiten Graphen G = (V, E) 12 auffassen. Knoten zu Positionen verschiedener Sequenzen sind über einer Kante verbunden 13, die mit der Bewertung dieser Zeichenalignierung beschriftet ist. Jedes Alignment entspricht einer Auswahl einer Teilmenge der Kanten. Die Menge der durch ein Alignment realisierten Kanten nennen wir seine Spur (Trace). Nicht jede Teilmenge der Kanten definiert umgekehrt eine gültige Spur (also ein gültiges Alignemnt) z.b. kann eine Position nicht mit mehr als einer Position aus einer bestimmten anderen Sequenz aligniert sein. Ziel ist, die Spur mit maximalem Kantengesamtgewicht (die Maximum Weight Trace) zu bestimmen. Diese Sichtweise und die eingesetzten Algorithmen sind sehr ähnlich zum Bestimmen von Proteinstrukturalignments mittels Maximum Contact Map Overlaps ([4] Lancia et al., 2001). In [5] verwenden die Entwickler der Coffee-Bewertungsfunktion einen genetischen Algorithmus, um bezüglich Coffee optimale bzw. gute Alignments zu finden. Natürlich ist dieser Ansatz auch auf jede andere Beschriftung der Kanten im oben beschriebenen k-partiten Graphen anwendbar, kann also allgemein zum Lösen von Maximum Weight Trace Problemen verwendet werden. Im Abschnitt wird noch ein Branch&Bound-Ansatz für Maximum Weight Trace vorgestellt, in ein progressives Verfahren, das auf einer modifizierten Coffee-Funktion basiert. 3.4 Progressive Verfahren Abbildung 3: Beispiel einer Sequenzmenge, bei der ein zu gieriger, aber kurzsichtiger progressiver Algorithmus ein schlechtes Alignment produziert, wenn er Lücken an den Sequenzenden beim paarweisen Alignment nicht bestraft CLUSTALW Als Beispiel für ein progressives Alignmentverfahren soll hier der CLUSTALW-Algorithmus kurz vorgestellt werden 14. Er geht folgenermaßen vor: 12 V ist die Menge der Knoten (vertices) und E die Menge der Kanten (edges) des Graphen. 13 Die k Sequenzen entsprechen also gerade den k Partitionen des Graphen. 14 Ein weiteres verbreitetes progressives Alignmentverfahren ist der Algorithmus von Feng-Doolittle. 12
13 1. Berechne alle ( ) n 2 = n (n 1) 2 paarweisen Alignmentscores und konvertiere sie in die geschätzten tatsächlichen evolutionären Distanzen Konstruiere einen Baum mittels Neighbour-Joining. Zu Beginn entspricht jede Sequenz einem einzelnen Baum mit nur einem Knoten. Dann werden jeweils die beiden Bäume (Sequenzen oder Alignments) i, j mit der kleinsten Distanz d ij aligniert, und die Distanz des neuen Baums k zu anderen Bäumen m wird auf d km := 1 2 (d im + d jm d ij ) gesetzt. Beim Zusammenfassen zweier Teilbäume zu einem größeren Baum werden die Alignments, die den Teilbäumen entsprechen, ebenfalls zusammengefasst (also aligniert). Es treten Sequenz-Sequenz, Sequenz-Alignment und Alignment-Alignment Paarungen auf. Die Sequenz-Sequenz Paarungen werden wie beim bisherigen paarweisen Alignment aligniert. Zum Alignieren einer Sequenz an ein Alignment wird die Sequenz nacheinander mit allen Sequenzen aus dem Alignment aligniert. Das paarweise Alignment mit der höchsten Punktzahl bestimmt die Art der Alignierung. Für Alignment-Alignment- Verschmelzungen wird von allen paarweisen Alignments das mit der höchsten Wertung herangezogen T-Coffee T-Coffee (tree-based Coffee) ist eine Variante von Coffee, die für die progressive Alignmenterzeugung geeignet ist. Wie beim ursprünglichen Coffee erhält der Algorithmus als Eingabe außer den zu alignierenden Sequenzen S 1,..., S k noch eine Bibliothek von fertigen paarweisen Alignments der Sequenzen. Diese kann sowohl globale als auch lokale Alignments enthalten. Die Bibliothek kann als Liste von Alignierungspaaren aufgefasst werden. Jedes solche Paar kennzeichnet zwei Positionen in unterschiedlichen Sequenzen und ist mit einer Gewichtung versehen. Die Gewichtung ist der Anteil übereinstimmender Zeichen in dem paarweisen A- lignment, also ein Maß für die Güte des Alignments und somit für die Relevanz dieses Alignierungspaars. Bis hierhin ist das Verfahren noch sehr ähnlich zu CLUSTALW bei den in [6] dokumentierten Untersuchungen wurde die Hälfte der paarweisen Alignments sogar mit CLUSTALW erstellt. Bevor die Sequenzen aber nun genau wie bei CLUSTALW zu einem Baum verbunden und aligniert werden, werden die Bewertungen der potentiell alignierten Positionspaare noch einem entscheidenden Zwischenschritt unterworfen. Der Wert einer bestimmten Positionspaarung soll nicht nur die Qualität des zugrundeliegenden Alignments allgemein repräsentieren, sondern Informationen über die Konservierung genau dieser Zuordnung in der Gesamtmenge der Ursprungssequenzen widerspiegeln. Ziel war ja, in den paarweisen Alignments besonders stark unterstützte Alignierungen von hochkonservierten Positionen auch im multiplen Alignment besonders hoch zu bewerten. Dazu werden,,indirekte Alignments über eine Zwischensequenz betrachtet. Seien a, b, c drei Positionen in verschiedenen Sequenzen. Unterstützt ein Alignment mit Gewicht w(a, b) die Alignierung der Positionen a und b, und unterstützt ferner ein Alignment mit Gewicht w(b, c) die Alignierung von b und c, dann wird die Bewertung w(a, c) der Alignierung (a, c) um min(w(a, b), w(b, c)) erhöht. Abbildung 4 verdeutlicht diesen Schritt, mit dem Informationen über die Gesamtbibliothek in jede einzelne Positionspaargewichtung einfließen. 15 Diese Schätzung erfolgt anhand eines statistischen Modells von Kimura, auf das hier nicht näher eingegangen werden soll. Das Modell berücksichtigt, dass aufgrund von Mehrfachmutationen am gleichen Sequenzort besonders für große Zeiträume ein immer kleinerer Anteil der Mutationen,,sichtbar ist. Für t geht ja z.b. die Zahl der tatsächlichen Mutationen, beobachtet wird aber nie mehr als der Unterschied zu einer zufälligen Sequenz. Aus der beobachteten Zahl der Mutationen wird nun die Zahl der unsichtbaren Mutationen (Mehrfachmutationen) und somit die Gesamtzahl an Mutationen, also die evolutionäre Distanz, abgeschätzt. 13
14 Abbildung 4: Erweiterung der Bibliothek bei T-Coffee: Punktzahlen für Alignierungen werden um die Bewertung von Pfaden indirekter Alignierungen über eine Zwischensequenz erhöht. Dies erhöht die Bewertungen von besonders stark konservierten Bereichen und ermöglicht es einem progressiven Alignment, trotz des greedy-ansatzes vorausschauend solche Aspekte der Gesamtmenge von Sequenzen zu berücksichtigen. 3.5 Andere Verfahren Dynamische Programmierung Der vom paarweisen Alignment bekannte Ansatz mit dynamischer Programmierung kann prinzipiell auch auf multiple Alignments verallgemeinert werden. Während im Fall von k = 2 die Matrix aber nur die Größe S 1 S 2 hatte und man zum Bestimmen eines Wertes in der Matrix drei andere Felder anschauen musste, hat die Matrix im allgemeinen Fall die Größe k i=1 S i, und man muss für jeden neu berechneten Wert jeweils 2 k 1 alte Felder anschauen 16. Der Laufzeitaufwand ist im allgemeinen Fall also O(2 k k i=1 S i ), der Speicherbedarf ist O(k 2 + k i=1 S i ) (k 2 für die Bewertungsfunktion) Branch&Bound für Maximum Weight Trace Wie bereits in Abschnitt 3.3 erläutert, kann das Problem des optimalen Sequenzalignments als Graphenproblem, genauer gesagt als die Bestimmung der Spur maximalen Gewichts (Maximum Weight Trace) angesehen werden. Coffee war nur ein Spezialfall der Zuweisung von Gewichten an die Kanten des Graphen, und T-Coffee war eine Heuristik zur Bestimmung guter Alignments bezüglich einer Variante der Coffee-Bewertungsfunktion. In diesem Abschnitt werden wir eine andere Heuristik zur Lösung des Sequenzalignments in Form eines Maximum Weight Trace Problems kennenlernen, die 1997 in [7] vorgeschlagen wurde. In Abschnitt 3.3 wurde erläutert, wie das Sequenzalignierungsproblem als Graphenproblem formuliert wird. Es mag ratsam sein, diesen Abschnitt vor dem Weiterlesen zur Erinnerung nochmals kurz durchzulesen. Ein Alignment entspricht einer Auswahl T E der Kanten des Graphen. Jede Kante entspricht der Alignierung zweier Positionen, und in der Auswahl sind all die Kanten enthalten, deren beide Endknoten (bzw. die zugehörigen Positionen) im 16 Dies liegt daran, dass man für jede Sequenz separat entscheiden kann, ob man in dieser ein Zeichen weiterrückt oder nicht. Jedes Tupel aus {0, 1} k, gibt also einen möglichen letzten Alignierungsschritt an, mit dem man eine bestimmte Position in der Tabelle erreichen kann. Von den 2 k Tupeln fällt lediglich das Nulltupel (0, 0,..., 0) weg, da man in mindestens einer Sequenz weiterrücken muss. 14
15 Alignment in der gleichen Spalte stehen. Eine Menge T E nennen wir eine Spur (Trace) genau dann, wenn sie ein gültiges Alignment codiert. Das Optimierungsproblem der Bestimmung einer maximalgewichteten Spur kann als ganzzahliges lineares Programm 17 beschrieben werden. Ein lineares Programm besteht aus einer Menge von linearen Ungleichungen über einer Menge von Variablen x 1,..., x n sowie einem Vektor b Z n mit Koordinaten b 1,..., b n. Ziel ist es, eine Belegung der Variablen zu finden, die n i=1 b i x i maximiert und alle Ungleichungen berücksichtigt. Bei einem ganzzahligen linearen Programm dürfen die Variablen x i nur ganzzahlige Werte annehmen. In unserem speziellen Fall ist b = (1,..., 1), wir wollen also i x i maximieren. Für jede Kante v des Graphen führen wir eine Variable x v ein, die angibt, ob diese Kante in der Spur enthalten sein soll. Durch Ungleichungen legen wir fest, dass die Variable nur den Wert 0 (Kante ist nicht ausgewählt) oder 1 (Kante ist ausgewählt) annehmen darf. Ferner müssen noch Ungleichungen eingeführt werden, die hinreichende und notwendige Voraussetzungen dafür sind, dass eine Lösung unseres Ungleichungssystems auch wirklich ein gültiges Alignment codiert. Nun könnte man für jede Menge v p1,... von Kanten, von denen nicht mehr als q ausgewählt sein dürfen, eine Ungleichung v p q einführen. Dadurch würde das Ungleichungssystem aber sehr groß werden, was die Lösung des Problems erschwert. Die Menge von Lösungen eines linearen Programms über n Variablen kann anschaulich als konvexer Körper im n-dimensionalen Raum aufgefasst werden 18. Optimale Lösungen liegen auf der (konvexen) Hülle des Körpers, genauer sogar stets in einer Ecke. Da die Variablen bei uns nur die Werte 0 und 1 annehmen können, liegen alle Lösungen auf Eckpunkten des n-dimensionalen Einheitskubus. Der konvexe Körper wird abgegrenzt durch Ebenen. Jede Ebene entspricht einer Ungleichung, die nur eine Hälfte des k-dimensionalen Raums als Lösung zulässt. Nicht jede solche Ebene begrenzt aber tatsächlich eine Seitenfläche des konvexen Lösungsraums z.b., wenn die Einhaltung der zugehörigen Ungleichung schon aus anderen Ungleichungen folgt. Ziel ist es also, eine möglichst kleine Menge von Ungleichungen zu finden, die den Lösungsraum beschreibt. In [7] werden drei Klassen von Ungleichungen entwickelt. Es wird gezeigt, dass für das Alignment von nur zwei Sequenzen diese Ungleichungen exakt den den Lösungsraum begrenzenden Ebenen entsprechen. Ferner werden Separationsalgorithmen angegeben, mit denen für eine teilweise konstruierte Lösung effizient getestet werden kann, ob sie alle Ungleichungen erfüllt andernfalls wird eine nicht erfüllte Ungleichung zurückgeliefert. Hiermit kann nun ein Verfahren zur Bestimmung von Maximum Weight Traces angegeben werden. Der Ansatz ist ein Branch&Bound Verfahren. Das bedeutet, dass die Lösung rekursiv konstruiert wird. Im ersten Schritt wird z.b. die erste Kante des Graphen ausgewählt und dann unter dieser Voraussetzung rekursiv weiter nach einer Lösung gesucht. Anschließend wird die Kante nicht ausgewählt und für die übrigen Kanten nach einer Lösung gesucht. Dieses rekursive Ausprobieren entspricht einem Tiefensuchbaum im Lösungsraum, der aber stark ausgedünnt wird: an jedem Knoten also bei jedem rekursiven Aufruf der Lösungsprozedur wird zunächst getestet, ob eine Ungleichung verletzt wurde. In diesem Fall wird die Lösungssuche abgebrochen. Ferner wird eine obere Schranke für die maximale aus der bisherigen Teillösung noch erzielbaren Punktzahl bestimmt. Liegt diese unter der Punktzahl für die bisher beste gefundene Lösung 19, so kann die rekursive Lösungssuche dort ebenfalls abgebrochen werden. Aus Platzgründen muss ich leider für die genaue Beschreibung des Verfahrens und vor allem der Ungleichungsklassen, die den Lösungsraum begrenzen, auf das Originalpaper [7] verweisen. 17 Integer linear program 18 In [7] wird dieser Körper als trace polytope bezeichnet. 19 Dies ist eine untere Schranke für die Qualität der besten Lösung. 15
16 Literatur [1] Durbin, R. et al. (1998). Biological Sequence Analysis: Probabalistic Models of Proteins and Nucleic Acids. Cambridge University Press. [2] Gusfield, D. (1997). Algorithms on strings, trees and sequences. Cambridge University Press. [3] Hickson, R.E. et al. (2000). The Performance of Several Multiple-Sequence Alignment Programs in Relation to Secondary-Structure Features for an rrna Sequence. Molecular Biology of Evolution 17(4), S [4] Lancia, G. et al. (2001). 101 Optimal PDB Structure Alignments: a Branch-and-Cut Algorithm For The Maximum Contact Map Overlap Problem. In: Proceedings of the Fifth Annual International Conferences on Compututational Molecular Biology (RECOMB01). [5] Notredame, C. et al. (1998). COFFEE: an objective function for multiple sequence alignments. Bioinformatics Vol. 14 no. 5, S [6] Notredame, C. et al. (2000). T-Coffee: A Novel Method for Fast and Accurate Multiple Sequence Alignment. Journal of Molecular Biolgy 302, S [7] Reinert, K. et al. (1997). A branch-and-cut algorithm for multiple sequence alignment. In: Proceedings of the First International Conference on Computational Molecular Biology (RECOMB97), S
Bioinformatik: Neue Paradigmen in der Forschung
 Bioinformatik: Neue Paradigmen in der Forschung Sommerakademie der Studienstiftung St. Johann, 1.-14.9.2002 Leitung: Proff. Ernst W. Mayr & Rolf Backofen Thema 4: Paarweises und multiples (heuristisches)
Bioinformatik: Neue Paradigmen in der Forschung Sommerakademie der Studienstiftung St. Johann, 1.-14.9.2002 Leitung: Proff. Ernst W. Mayr & Rolf Backofen Thema 4: Paarweises und multiples (heuristisches)
Algorithmen für paarweise Sequenz-Alignments. Katharina Hembach
 Proseminar Bioinformatik WS 2010/11 Algorithmen für paarweise Sequenz-Alignments Katharina Hembach 06.12.2010 1 Einleitung Paarweise Sequenz-Alignments spielen in der Bioinformatik eine wichtige Rolle.
Proseminar Bioinformatik WS 2010/11 Algorithmen für paarweise Sequenz-Alignments Katharina Hembach 06.12.2010 1 Einleitung Paarweise Sequenz-Alignments spielen in der Bioinformatik eine wichtige Rolle.
Algorithmen und Datenstrukturen in der Bioinformatik Zweites Übungsblatt WS 05/06 Musterlösung
 Johanna Ploog, Konstantin Clemens Freie Universität Berlin Institut für Mathematik II Arbeitsgruppe für Mathematik in den Lebenswissenschaften Algorithmen und Datenstrukturen in der Bioinformatik Zweites
Johanna Ploog, Konstantin Clemens Freie Universität Berlin Institut für Mathematik II Arbeitsgruppe für Mathematik in den Lebenswissenschaften Algorithmen und Datenstrukturen in der Bioinformatik Zweites
Multiple Alignments. Vorlesung Einführung in die Angewandte Bioinformatik Prof. Dr. Sven Rahmann. Webseite zur Vorlesung
 Multiple Alignments Vorlesung Einführung in die Angewandte Bioinformatik Prof. Dr. Sven Rahmann Webseite zur Vorlesung http://bioinfo.wikidot.com/ Sprechstunde Mo 16-17 in OH14, R214 Sven.Rahmann -at-
Multiple Alignments Vorlesung Einführung in die Angewandte Bioinformatik Prof. Dr. Sven Rahmann Webseite zur Vorlesung http://bioinfo.wikidot.com/ Sprechstunde Mo 16-17 in OH14, R214 Sven.Rahmann -at-
Pairwise Alignment. Steffen Forkmann. Proseminar: BioInformatik
 Pairwise Alignment Steffen Forkmann Proseminar: BioInformatik Wintersemester 2004/2005 Inhaltsverzeichnis 1 Problemstellungen 3 1.1 Rechtschreibkorrektur............................... 3 1.2 DNA- und Aminosäure-Sequenzen........................
Pairwise Alignment Steffen Forkmann Proseminar: BioInformatik Wintersemester 2004/2005 Inhaltsverzeichnis 1 Problemstellungen 3 1.1 Rechtschreibkorrektur............................... 3 1.2 DNA- und Aminosäure-Sequenzen........................
Algorithmische Anwendungen WS 2005/2006
 Algorithmische Anwendungen WS 2005/2006 Sequenzalignment Gruppe F_lila_Ala0506 Allal Kharaz Yassine ELassad Inhaltsverzeichnis 1 Problemstellungen...................................... 3 1.1 Rechtschreibkorrektur...............................
Algorithmische Anwendungen WS 2005/2006 Sequenzalignment Gruppe F_lila_Ala0506 Allal Kharaz Yassine ELassad Inhaltsverzeichnis 1 Problemstellungen...................................... 3 1.1 Rechtschreibkorrektur...............................
Praktikum Algorithmischen Anwendungen WS 2006/07 Bioinformatik Sequence-Aligment Team C rot Ala0607
 Praktikum Algorithmischen Anwendungen WS 2006/07 Bioinformatik Sequence-Aligment Team C rot Ala0607 Blöink, Frank 11038619 ai600@gm.fh-koeln.de Wolters, Benjamin 11037092 ai651@gm.fh-koeln.de 26. Januar
Praktikum Algorithmischen Anwendungen WS 2006/07 Bioinformatik Sequence-Aligment Team C rot Ala0607 Blöink, Frank 11038619 ai600@gm.fh-koeln.de Wolters, Benjamin 11037092 ai651@gm.fh-koeln.de 26. Januar
Bioinformatik. Lokale Alignierung Gapkosten. Silke Trißl / Ulf Leser Wissensmanagement in der. Bioinformatik
 Bioinformatik Lokale Alignierung Gapkosten Silke Trißl / Ulf Leser Wissensmanagement in der Bioinformatik Inhalt dieser Vorlesung Ähnlichkeit Lokales und globales Alignment Gapped Alignment Silke Trißl:
Bioinformatik Lokale Alignierung Gapkosten Silke Trißl / Ulf Leser Wissensmanagement in der Bioinformatik Inhalt dieser Vorlesung Ähnlichkeit Lokales und globales Alignment Gapped Alignment Silke Trißl:
Zentrum für Bioinformatik. Übung 4: Revision. Beispielfragen zur Klausur im Modul Angewandte Bioinformatik (erste Semesterhälfte)
 Andrew Torda Björn Hansen Iryna Bondarenko Zentrum für Bioinformatik Übung zur Vorlesung Angewandte Bioinformatik Sommersemester 2014 20./23.06.2014 Übung 4: Revision Beispielfragen zur Klausur im Modul
Andrew Torda Björn Hansen Iryna Bondarenko Zentrum für Bioinformatik Übung zur Vorlesung Angewandte Bioinformatik Sommersemester 2014 20./23.06.2014 Übung 4: Revision Beispielfragen zur Klausur im Modul
Algorithmische Bioinformatik 1
 Algorithmische Bioinformatik 1 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Paarweises
Algorithmische Bioinformatik 1 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Paarweises
Algorithmen auf Sequenzen
 Algorithmen auf Sequenzen Vorlesung von Prof. Dr. Sven Rahmann im Sommersemester 2008 Kapitel 6 Alignments Webseite zur Vorlesung http://ls11-www.cs.tu-dortmund.de/people/rahmann/teaching/ss2008/algorithmenaufsequenzen
Algorithmen auf Sequenzen Vorlesung von Prof. Dr. Sven Rahmann im Sommersemester 2008 Kapitel 6 Alignments Webseite zur Vorlesung http://ls11-www.cs.tu-dortmund.de/people/rahmann/teaching/ss2008/algorithmenaufsequenzen
Algorithmen auf Sequenzen
 Algorithmen auf Sequenzen Fehlertolerante Mustersuche: Distanz- und Ähnlichkeitsmaße Sven Rahmann Genominformatik Universitätsklinikum Essen Universität Duisburg-Essen Universitätsallianz Ruhr Einführung
Algorithmen auf Sequenzen Fehlertolerante Mustersuche: Distanz- und Ähnlichkeitsmaße Sven Rahmann Genominformatik Universitätsklinikum Essen Universität Duisburg-Essen Universitätsallianz Ruhr Einführung
Algorithmen auf Sequenzen Paarweiser Sequenzvergleich: Alignments
 Algorithmen auf Sequenzen Paarweiser Sequenzvergleich: Alignments Sven Rahmann Genominformatik Universitätsklinikum Essen Universität Duisburg-Essen Universitätsallianz Ruhr Einführung Bisher: Berechnung
Algorithmen auf Sequenzen Paarweiser Sequenzvergleich: Alignments Sven Rahmann Genominformatik Universitätsklinikum Essen Universität Duisburg-Essen Universitätsallianz Ruhr Einführung Bisher: Berechnung
Das Problem des Handlungsreisenden
 Seite 1 Das Problem des Handlungsreisenden Abbildung 1: Alle möglichen Rundreisen für 4 Städte Das TSP-Problem tritt in der Praxis in vielen Anwendungen als Teilproblem auf. Hierzu gehören z.b. Optimierungsprobleme
Seite 1 Das Problem des Handlungsreisenden Abbildung 1: Alle möglichen Rundreisen für 4 Städte Das TSP-Problem tritt in der Praxis in vielen Anwendungen als Teilproblem auf. Hierzu gehören z.b. Optimierungsprobleme
Algorithmen und Datenstrukturen 2
 Algorithmen und Datenstrukturen 2 Sommersemester 2007 11. Vorlesung Peter F. Stadler Universität Leipzig Institut für Informatik studla@bioinf.uni-leipzig.de Das Rucksack-Problem Ein Dieb, der einen Safe
Algorithmen und Datenstrukturen 2 Sommersemester 2007 11. Vorlesung Peter F. Stadler Universität Leipzig Institut für Informatik studla@bioinf.uni-leipzig.de Das Rucksack-Problem Ein Dieb, der einen Safe
Algorithmen und Datenstrukturen 2
 Algorithmen und Datenstrukturen 2 Sommersemester 2009 11. Vorlesung Uwe Quasthoff Universität Leipzig Institut für Informatik quasthoff@informatik.uni-leipzig.de Das Rucksack-Problem Ein Dieb, der einen
Algorithmen und Datenstrukturen 2 Sommersemester 2009 11. Vorlesung Uwe Quasthoff Universität Leipzig Institut für Informatik quasthoff@informatik.uni-leipzig.de Das Rucksack-Problem Ein Dieb, der einen
Algorithmen und Datenstrukturen 2
 Algorithmen und Datenstrukturen 2 Lerneinheit 3: Greedy Algorithmen Prof. Dr. Christoph Karg Studiengang Informatik Hochschule Aalen Sommersemester 2016 10.5.2016 Einleitung Einleitung Diese Lerneinheit
Algorithmen und Datenstrukturen 2 Lerneinheit 3: Greedy Algorithmen Prof. Dr. Christoph Karg Studiengang Informatik Hochschule Aalen Sommersemester 2016 10.5.2016 Einleitung Einleitung Diese Lerneinheit
3.3 Optimale binäre Suchbäume
 3.3 Optimale binäre Suchbäume Problem 3.3.1. Sei S eine Menge von Schlüsseln aus einem endlichen, linear geordneten Universum U, S = {a 1,,...,a n } U und S = n N. Wir wollen S in einem binären Suchbaum
3.3 Optimale binäre Suchbäume Problem 3.3.1. Sei S eine Menge von Schlüsseln aus einem endlichen, linear geordneten Universum U, S = {a 1,,...,a n } U und S = n N. Wir wollen S in einem binären Suchbaum
Primärstruktur. Wintersemester 2011/12. Peter Güntert
 Primärstruktur Wintersemester 2011/12 Peter Güntert Primärstruktur Beziehung Sequenz Struktur Proteinsequenzen, Sequenzdatenbanken Sequenzvergleich (sequence alignment) Sequenzidentität, Sequenzhomologie
Primärstruktur Wintersemester 2011/12 Peter Güntert Primärstruktur Beziehung Sequenz Struktur Proteinsequenzen, Sequenzdatenbanken Sequenzvergleich (sequence alignment) Sequenzidentität, Sequenzhomologie
Aufgabenblatt 5. Silke Trißl Wissensmanagement in der Bioinformatik
 Aufgabenblatt 5 Silke Trißl Wissensmanagement in der Bioinformatik Zuerst! FRAGEN? Silke Trißl: Bioinformatik für Biophysiker 2 Exercise 1 + 2 Modify program to compare protein sequence read substitution
Aufgabenblatt 5 Silke Trißl Wissensmanagement in der Bioinformatik Zuerst! FRAGEN? Silke Trißl: Bioinformatik für Biophysiker 2 Exercise 1 + 2 Modify program to compare protein sequence read substitution
Homologie und Sequenzähnlichkeit. Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de
 Homologie und Sequenzähnlichkeit Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de Homologie Verwandtschaft aufgrund gleicher Abstammung basiert auf Speziation (Artbildung): aus einer
Homologie und Sequenzähnlichkeit Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de Homologie Verwandtschaft aufgrund gleicher Abstammung basiert auf Speziation (Artbildung): aus einer
Theoretische Informatik. Exkurs: Komplexität von Optimierungsproblemen. Optimierungsprobleme. Optimierungsprobleme. Exkurs Optimierungsprobleme
 Theoretische Informatik Exkurs Rainer Schrader Exkurs: Komplexität von n Institut für Informatik 13. Mai 2009 1 / 34 2 / 34 Gliederung Entscheidungs- und Approximationen und Gütegarantien zwei Greedy-Strategien
Theoretische Informatik Exkurs Rainer Schrader Exkurs: Komplexität von n Institut für Informatik 13. Mai 2009 1 / 34 2 / 34 Gliederung Entscheidungs- und Approximationen und Gütegarantien zwei Greedy-Strategien
Algorithmische Bioinformatik
 Algorithmische Bioinformatik Effiziente Berechnung des Editabstands Dynamische Programmierung Ulf Leser Wissensmanagement in der Bioinformatik Inhalt dieser Vorlesung Rekursive Definition des Editabstands
Algorithmische Bioinformatik Effiziente Berechnung des Editabstands Dynamische Programmierung Ulf Leser Wissensmanagement in der Bioinformatik Inhalt dieser Vorlesung Rekursive Definition des Editabstands
Algorithmen und Komplexität Lösungsvorschlag zu Übungsblatt 8
 ETH Zürich Institut für Theoretische Informatik Prof. Dr. Angelika Steger Florian Meier, Ralph Keusch HS 2017 Algorithmen und Komplexität Lösungsvorschlag zu Übungsblatt 8 Lösungsvorschlag zu Aufgabe 1
ETH Zürich Institut für Theoretische Informatik Prof. Dr. Angelika Steger Florian Meier, Ralph Keusch HS 2017 Algorithmen und Komplexität Lösungsvorschlag zu Übungsblatt 8 Lösungsvorschlag zu Aufgabe 1
ADS: Algorithmen und Datenstrukturen 2
 ADS: Algorithmen und Datenstrukturen 2 Teil 6 Prof. Dr. Gerhard Heyer Institut für Informatik Abteilung Automatische Sprachverarbeitung Universität Leipzig 16. Mai 2018 [Letzte Aktualisierung: 18/05/2018,
ADS: Algorithmen und Datenstrukturen 2 Teil 6 Prof. Dr. Gerhard Heyer Institut für Informatik Abteilung Automatische Sprachverarbeitung Universität Leipzig 16. Mai 2018 [Letzte Aktualisierung: 18/05/2018,
Große Lösungsräume. Leon Schmidtchen Hallo Welt Seminar - LS Leon Schmidtchen Große Lösungsräume Hallo Welt Seminar - LS2
 Große Lösungsräume Leon Schmidtchen 1.06.201 Hallo Welt Seminar - LS 2!1 Gliederung Motivation Brute Force Backtracking Pruning Leon Schmidtchen Branch & Bound 1.06.201 Hallo Welt Seminar - LS 2 Wann soll
Große Lösungsräume Leon Schmidtchen 1.06.201 Hallo Welt Seminar - LS 2!1 Gliederung Motivation Brute Force Backtracking Pruning Leon Schmidtchen Branch & Bound 1.06.201 Hallo Welt Seminar - LS 2 Wann soll
Inhalt. 8.1 Motivation. 8.2 Optimierung ohne Nebenbedingungen. 8.3 Optimierung unter Nebenbedingungen. 8.4 Lineare Programmierung
 8. Optimierung Inhalt 8.1 Motivation 8.2 Optimierung ohne Nebenbedingungen 8.3 Optimierung unter Nebenbedingungen 8.4 Lineare Programmierung 8.5 Kombinatorische Optimierung 2 8.1 Motivation Viele Anwendungen
8. Optimierung Inhalt 8.1 Motivation 8.2 Optimierung ohne Nebenbedingungen 8.3 Optimierung unter Nebenbedingungen 8.4 Lineare Programmierung 8.5 Kombinatorische Optimierung 2 8.1 Motivation Viele Anwendungen
ADS: Algorithmen und Datenstrukturen 2
 ADS: Algorithmen und Datenstrukturen 2 Teil 4 Prof. Dr. Gerhard Heyer Institut für Informatik Abteilung Automatische Sprachverarbeitung Universität Leipzig 24. April 2019 [Letzte Aktualisierung: 24/04/2019,
ADS: Algorithmen und Datenstrukturen 2 Teil 4 Prof. Dr. Gerhard Heyer Institut für Informatik Abteilung Automatische Sprachverarbeitung Universität Leipzig 24. April 2019 [Letzte Aktualisierung: 24/04/2019,
Studiengang Informatik der FH Gießen-Friedberg. Sequenz-Alignment. Jan Schäfer. WS 2006/07 Betreuer: Prof. Dr. Klaus Quibeldey-Cirkel
 Studiengang Informatik der FH Gießen-Friedberg Sequenz-Alignment Jan Schäfer WS 2006/07 Betreuer: Prof. Dr. Klaus Quibeldey-Cirkel Überblick Einführung Grundlagen Wann ist das Merkmal der Ähnlichkeit erfüllt?
Studiengang Informatik der FH Gießen-Friedberg Sequenz-Alignment Jan Schäfer WS 2006/07 Betreuer: Prof. Dr. Klaus Quibeldey-Cirkel Überblick Einführung Grundlagen Wann ist das Merkmal der Ähnlichkeit erfüllt?
6. Algorithmen auf Zeichenketten
 6. Algorithmen auf Zeichenketten Motivation Wir lernen nun Algorithmen zur Lösung verschiedener elementarer Probleme auf Zeichenketten zu lösen Zeichenketten spielen eine wichtige Rolle in diversen Gebieten
6. Algorithmen auf Zeichenketten Motivation Wir lernen nun Algorithmen zur Lösung verschiedener elementarer Probleme auf Zeichenketten zu lösen Zeichenketten spielen eine wichtige Rolle in diversen Gebieten
Einführung in die Bioinformatik
 Einführung in die Bioinformatik Ringvorlesung Biologie Sommer 07 Burkhard Morgenstern Institut für Mikrobiologie und Genetik Abteilung für Bioinformatik Goldschmidtstr. 1 Online Materialien zur Ringvorlesung:
Einführung in die Bioinformatik Ringvorlesung Biologie Sommer 07 Burkhard Morgenstern Institut für Mikrobiologie und Genetik Abteilung für Bioinformatik Goldschmidtstr. 1 Online Materialien zur Ringvorlesung:
Algorithmen II Vorlesung am
 Algorithmen II Vorlesung am 17.01.013 Parametrisierte Algorithmen INSTITUT FÜR THEORETISCHE INFORMATIK PROF. DR. DOROTHEA WAGNER KIT Universität des Landes Baden-Württemberg und Algorithmen nationales
Algorithmen II Vorlesung am 17.01.013 Parametrisierte Algorithmen INSTITUT FÜR THEORETISCHE INFORMATIK PROF. DR. DOROTHEA WAGNER KIT Universität des Landes Baden-Württemberg und Algorithmen nationales
Übungsaufgaben zur Einführung in die Bioinformatik - Lösungen
 18.01.2013 Prof. P. Güntert 1 Vorlesung BPC I: Aspekte der Thermodynamik in der Strukturbiologie Übungsaufgaben zur Einführung in die Bioinformatik - Lösungen 1. Hamming und Levenshtein Distanzen a) Was
18.01.2013 Prof. P. Güntert 1 Vorlesung BPC I: Aspekte der Thermodynamik in der Strukturbiologie Übungsaufgaben zur Einführung in die Bioinformatik - Lösungen 1. Hamming und Levenshtein Distanzen a) Was
Übersicht. Aktivitäten-Auswahl-Problem. Greedy Algorithmen. Aktivitäten-Auswahl-Problem. Aktivitäten-Auswahl-Problem. Datenstrukturen & Algorithmen
 Datenstrukturen & Algorithmen Übersicht Greedy Algorithmen Einführung Aktivitäten-Auswahl-Problem Huffman Codierung Matthias Zwicker Universität Bern Frühling 2009 2 Greedy Algorithmen Entwurfsstrategie
Datenstrukturen & Algorithmen Übersicht Greedy Algorithmen Einführung Aktivitäten-Auswahl-Problem Huffman Codierung Matthias Zwicker Universität Bern Frühling 2009 2 Greedy Algorithmen Entwurfsstrategie
Informatik II, SS 2018
 Informatik II - SS 2018 (Algorithmen & Datenstrukturen) Vorlesung 21 (11.7.2018) String Matching (Textsuche) II Greedy Algorithmen I Algorithmen und Komplexität Textsuche / String Matching Gegeben: Zwei
Informatik II - SS 2018 (Algorithmen & Datenstrukturen) Vorlesung 21 (11.7.2018) String Matching (Textsuche) II Greedy Algorithmen I Algorithmen und Komplexität Textsuche / String Matching Gegeben: Zwei
Anwendungen dynamischer Programmierung in der Biologie
 Anwendungen dynamischer Programmierung in der Biologie Überblick Algorithmus zum Finden der wahrscheinlichsten Sekundärstruktur eines RNS Moleküls Sequence Alignment Verbesserung von Sequence Alignment
Anwendungen dynamischer Programmierung in der Biologie Überblick Algorithmus zum Finden der wahrscheinlichsten Sekundärstruktur eines RNS Moleküls Sequence Alignment Verbesserung von Sequence Alignment
Perlen der Informatik I Wintersemester 2012 Aufgabenblatt 7
 Technische Universität München WS 2012 Institut für Informatik Prof. Dr. H.-J. Bungartz Prof. Dr. T. Huckle Prof. Dr. M. Bader Kristof Unterweger Perlen der Informatik I Wintersemester 2012 Aufgabenblatt
Technische Universität München WS 2012 Institut für Informatik Prof. Dr. H.-J. Bungartz Prof. Dr. T. Huckle Prof. Dr. M. Bader Kristof Unterweger Perlen der Informatik I Wintersemester 2012 Aufgabenblatt
Nash-Gleichgewichte in 2-Spieler Systemen. Katharina Klost Freie Universität Berlin
 Nash-Gleichgewichte in 2-Spieler Systemen Katharina Klost Freie Universität Berlin Seminar über Algorithmen, 29.10.2013 Grundlegende Definitionen A Gewinnmatrix für Spieler 1, B Gewinnmatrix für Spieler
Nash-Gleichgewichte in 2-Spieler Systemen Katharina Klost Freie Universität Berlin Seminar über Algorithmen, 29.10.2013 Grundlegende Definitionen A Gewinnmatrix für Spieler 1, B Gewinnmatrix für Spieler
Mischungsverhältnisse: Nehmen wir an, es stehen zwei Substanzen (zum Beispiel Flüssigkeiten) mit spezifischen Gewicht a = 2 kg/l bzw.
 Kapitel 5 Lineare Algebra 51 Lineare Gleichungssysteme und Matrizen Man begegnet Systemen von linearen Gleichungen in sehr vielen verschiedenen Zusammenhängen, etwa bei Mischungsverhältnissen von Substanzen
Kapitel 5 Lineare Algebra 51 Lineare Gleichungssysteme und Matrizen Man begegnet Systemen von linearen Gleichungen in sehr vielen verschiedenen Zusammenhängen, etwa bei Mischungsverhältnissen von Substanzen
TU München. Hauptseminar: WS 2002 / Einführung in Suffix - Bäume
 TU München Hauptseminar: WS 2002 / 2003 Einführung in Suffix - Bäume Bearbeiterin: Shasha Meng Betreuerin: Barbara König Inhalt 1. Einleitung 1.1 Motivation 1.2 Eine kurze Geschichte 2. Tries 2.1 Basisdefinition
TU München Hauptseminar: WS 2002 / 2003 Einführung in Suffix - Bäume Bearbeiterin: Shasha Meng Betreuerin: Barbara König Inhalt 1. Einleitung 1.1 Motivation 1.2 Eine kurze Geschichte 2. Tries 2.1 Basisdefinition
9 Minimum Spanning Trees
 Im Folgenden wollen wir uns genauer mit dem Minimum Spanning Tree -Problem auseinandersetzen. 9.1 MST-Problem Gegeben ein ungerichteter Graph G = (V,E) und eine Gewichtsfunktion w w : E R Man berechne
Im Folgenden wollen wir uns genauer mit dem Minimum Spanning Tree -Problem auseinandersetzen. 9.1 MST-Problem Gegeben ein ungerichteter Graph G = (V,E) und eine Gewichtsfunktion w w : E R Man berechne
Sequenzen-Alignierung in der Bioinformatik. VO Algorithm Engineering Professor Dr. Petra Mutzel Lehrstuhl für Algorithm Engineering, LS11
 Sequenzen-Alignierung in der Bioinformatik VO Algorithm Engineering Professor Dr. Petra Mutzel Lehrstuhl für Algorithm Engineering, LS11 23. VO 24.01.2006 1 1 Literatur für diese VO Volker Heun: Skriptum
Sequenzen-Alignierung in der Bioinformatik VO Algorithm Engineering Professor Dr. Petra Mutzel Lehrstuhl für Algorithm Engineering, LS11 23. VO 24.01.2006 1 1 Literatur für diese VO Volker Heun: Skriptum
Algorithmik WS 07/ Vorlesung, Andreas Jakoby Universität zu Lübeck. 10 Matching-Probleme
 10 Matching-Probleme 10.1 Definition von Matching-Probleme Definition 21 [2-dimensionales Matching] Sei G = (V, E) ein ungerichteter Graph und E E. E ist ein Matching, wenn für alle Kantenpaare e 1, e
10 Matching-Probleme 10.1 Definition von Matching-Probleme Definition 21 [2-dimensionales Matching] Sei G = (V, E) ein ungerichteter Graph und E E. E ist ein Matching, wenn für alle Kantenpaare e 1, e
Algorithmische Geometrie, SoSe 2005 Skriptmitschrift vom 29. April 2005
 Algorithmische Geometrie, SoSe 2005 Skriptmitschrift vom 29. April 2005 Antonia Wittmers Igor Savchenko Konvexe Hüllen Inkrementeller Algorithmus für die konvexe Hülle Dabei heißt inkrementeller Algorithmus,
Algorithmische Geometrie, SoSe 2005 Skriptmitschrift vom 29. April 2005 Antonia Wittmers Igor Savchenko Konvexe Hüllen Inkrementeller Algorithmus für die konvexe Hülle Dabei heißt inkrementeller Algorithmus,
Algorithmen und Datenstrukturen 2 VU 3.0 Nachtragstest SS Oktober 2016
 Technische Universität Wien Institut für Computergraphik und Algorithmen Algorithms and Complexity Group 186.815 Algorithmen und Datenstrukturen 2 VU 3.0 Nachtragstest SS 2016 5. Oktober 2016 Machen Sie
Technische Universität Wien Institut für Computergraphik und Algorithmen Algorithms and Complexity Group 186.815 Algorithmen und Datenstrukturen 2 VU 3.0 Nachtragstest SS 2016 5. Oktober 2016 Machen Sie
4.9.7 Konstruktion der Suffixbäume
 .9.7 Konstruktion der Suffixbäume Beipiel: xabxa (siehe Abbildung.27) Man beginnt mit der Konstruktion eines Suffixbaumes für gesamten String und schreibt eine 1 am Blatt, weil der Suffix xabxa an der
.9.7 Konstruktion der Suffixbäume Beipiel: xabxa (siehe Abbildung.27) Man beginnt mit der Konstruktion eines Suffixbaumes für gesamten String und schreibt eine 1 am Blatt, weil der Suffix xabxa an der
Entscheidungsbäume. Definition Entscheidungsbaum. Frage: Gibt es einen Sortieralgorithmus mit o(n log n) Vergleichen?
 Entscheidungsbäume Frage: Gibt es einen Sortieralgorithmus mit o(n log n) Vergleichen? Definition Entscheidungsbaum Sei T ein Binärbaum und A = {a 1,..., a n } eine zu sortierenden Menge. T ist ein Entscheidungsbaum
Entscheidungsbäume Frage: Gibt es einen Sortieralgorithmus mit o(n log n) Vergleichen? Definition Entscheidungsbaum Sei T ein Binärbaum und A = {a 1,..., a n } eine zu sortierenden Menge. T ist ein Entscheidungsbaum
Bioinformatik Für Biophysiker
 Bioinformatik Für Biophysiker Sommersemester 2009 Silke Trißl / Ulf Leser Wissensmanagement in der Bioinformatik Wissensmanagement in der Bioinformatik Schwerpunkte Algorithmen der Bioinformatik Management
Bioinformatik Für Biophysiker Sommersemester 2009 Silke Trißl / Ulf Leser Wissensmanagement in der Bioinformatik Wissensmanagement in der Bioinformatik Schwerpunkte Algorithmen der Bioinformatik Management
Threading - Algorithmen
 Threading - Algorithmen Florian Lindemann 22.11.2007 Florian Lindemann () Threading - Algorithmen 22.11.2007 1 / 25 Gliederung 1 Prospect Scoring Function Algorithmus Weitere Eigenschaften Komplexität
Threading - Algorithmen Florian Lindemann 22.11.2007 Florian Lindemann () Threading - Algorithmen 22.11.2007 1 / 25 Gliederung 1 Prospect Scoring Function Algorithmus Weitere Eigenschaften Komplexität
Kapitel 7: Sequenzen- Alignierung in der Bioinformatik
 Kapitel 7: Sequenzen- Alignierung in der Bioinformatik 7.3: Paarweise Sequenzen-Alignierung 7.4: Multiple Sequenzen Alignierung VO Algorithm Engineering Professor Dr. Petra Mutzel Lehrstuhl für Algorithm
Kapitel 7: Sequenzen- Alignierung in der Bioinformatik 7.3: Paarweise Sequenzen-Alignierung 7.4: Multiple Sequenzen Alignierung VO Algorithm Engineering Professor Dr. Petra Mutzel Lehrstuhl für Algorithm
2.2 Der Algorithmus von Knuth, Morris und Pratt
 Suchen in Texten 2.1 Grundlagen Ein Alphabet ist eine endliche Menge von Symbolen. Bsp.: Σ a, b, c,..., z, Σ 0, 1, Σ A, C, G, T. Wörter über Σ sind endliche Folgen von Symbolen aus Σ. Wörter werden manchmal
Suchen in Texten 2.1 Grundlagen Ein Alphabet ist eine endliche Menge von Symbolen. Bsp.: Σ a, b, c,..., z, Σ 0, 1, Σ A, C, G, T. Wörter über Σ sind endliche Folgen von Symbolen aus Σ. Wörter werden manchmal
Klausur Algorithmen und Datenstrukturen II 01. Agust 2016
 Technische Universität Braunschweig Sommersemester 2016 Institut für Betriebssysteme und Rechnerverbund Abteilung Algorithmik Prof. Dr. Sándor P. Fekete Dr. Christian Scheffer Klausur Algorithmen und Datenstrukturen
Technische Universität Braunschweig Sommersemester 2016 Institut für Betriebssysteme und Rechnerverbund Abteilung Algorithmik Prof. Dr. Sándor P. Fekete Dr. Christian Scheffer Klausur Algorithmen und Datenstrukturen
MBI: Sequenz-Vergleich mit Alignment
 MBI: Sequenz-Vergleich mit Alignment Bernhard Haubold 28. Oktober 2014 Wiederholung: Was ist Bioinformatik? Historische Übersicht; CABIOS Bioinformatics Gemeinsames Thema: Information in vivo DNA Epigenetik
MBI: Sequenz-Vergleich mit Alignment Bernhard Haubold 28. Oktober 2014 Wiederholung: Was ist Bioinformatik? Historische Übersicht; CABIOS Bioinformatics Gemeinsames Thema: Information in vivo DNA Epigenetik
Algorithmische Bioinformatik 1
 Algorithmische Bioinformatik 1 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Algorithmen
Algorithmische Bioinformatik 1 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Algorithmen
. Die obige Beschreibung der Laufzeit für ein bestimmtes k können wir also erweitern und erhalten die folgende Gleichung für den mittleren Fall:
 Laufzeit von Quicksort im Mittel. Wir wollen die erwartete Effizienz von Quicksort ermitteln. Wir nehmen an, die Wahrscheinlichkeit, dass das gewählte Pivot-Element a j das k-t kleinste Element der Folge
Laufzeit von Quicksort im Mittel. Wir wollen die erwartete Effizienz von Quicksort ermitteln. Wir nehmen an, die Wahrscheinlichkeit, dass das gewählte Pivot-Element a j das k-t kleinste Element der Folge
Satz 324 Sei M wie oben. Dann gibt es für ein geeignetes k Konstanten c i > 0 und Permutationsmatrizen P i, i = 1,...
 Satz 324 Sei M wie oben. Dann gibt es für ein geeignetes k Konstanten c i > 0 und Permutationsmatrizen P i, i = 1,..., k, so dass gilt M = k c i P i i=1 k c i = r. i=1 Diskrete Strukturen 7.1 Matchings
Satz 324 Sei M wie oben. Dann gibt es für ein geeignetes k Konstanten c i > 0 und Permutationsmatrizen P i, i = 1,..., k, so dass gilt M = k c i P i i=1 k c i = r. i=1 Diskrete Strukturen 7.1 Matchings
Informatik II, SS 2016
 Informatik II - SS 2016 (Algorithmen & Datenstrukturen) Vorlesung 22 (20.7.2016) Greedy Algorithmen - Datenkompression Algorithmen und Komplexität Greedy Algorithmen Greedy Algorithmen sind eine Algorithmenmethode,
Informatik II - SS 2016 (Algorithmen & Datenstrukturen) Vorlesung 22 (20.7.2016) Greedy Algorithmen - Datenkompression Algorithmen und Komplexität Greedy Algorithmen Greedy Algorithmen sind eine Algorithmenmethode,
Übersicht. 1 Einführung. 2 Suchen und Sortieren. 3 Graphalgorithmen. 4 Algorithmische Geometrie. 5 Textalgorithmen. 6 Paradigmen
 Übersicht 1 Einführung 2 Suchen und Sortieren 3 Graphalgorithmen 4 Algorithmische Geometrie 5 6 Paradigmen Übersicht 5 Editdistanz (Folie 446, Seite 83 im Skript) Eingabe: Zwei Strings v und w Frage: Kommt
Übersicht 1 Einführung 2 Suchen und Sortieren 3 Graphalgorithmen 4 Algorithmische Geometrie 5 6 Paradigmen Übersicht 5 Editdistanz (Folie 446, Seite 83 im Skript) Eingabe: Zwei Strings v und w Frage: Kommt
Künstliche Intelligenz - Optimierungsprobleme - Suche in Spielbäumen
 Künstliche Intelligenz - Optimierungsprobleme - Suche in Spielbäumen Stephan Schwiebert WS 2009/2010 Sprachliche Informationsverarbeitung Institut für Linguistik Universität zu Köln Optimierungsprobleme
Künstliche Intelligenz - Optimierungsprobleme - Suche in Spielbäumen Stephan Schwiebert WS 2009/2010 Sprachliche Informationsverarbeitung Institut für Linguistik Universität zu Köln Optimierungsprobleme
Algorithmen und Datenstrukturen in der Bioinformatik Drittes Übungsblatt WS 05/06 Musterlösung
 Konstantin Clemens Johanna Ploog Freie Universität Berlin Institut für Mathematik II Arbeitsgruppe für Mathematik in den Lebenswissenschaften Algorithmen und Datenstrukturen in der Bioinformatik Drittes
Konstantin Clemens Johanna Ploog Freie Universität Berlin Institut für Mathematik II Arbeitsgruppe für Mathematik in den Lebenswissenschaften Algorithmen und Datenstrukturen in der Bioinformatik Drittes
Ein sortiertes Feld kann in O(log n) durchsucht werden, z.b. mit Binärsuche. Der Algorithmus 1 gibt den Pseudocode der binären Suche an.
 2.5 Suchen Eine Menge S will nach einem Element durchsucht werden. Die Menge S ist statisch und S = n. S ist Teilmenge eines Universums auf dem eine lineare Ordnung definiert ist und soll so gespeichert
2.5 Suchen Eine Menge S will nach einem Element durchsucht werden. Die Menge S ist statisch und S = n. S ist Teilmenge eines Universums auf dem eine lineare Ordnung definiert ist und soll so gespeichert
Aufgabenblatt 4. Silke Trißl Wissensmanagement in der Bioinformatik
 Aufgabenblatt 4 Silke Trißl Wissensmanagement in der Bioinformatik Zuerst! FRAGEN? Silke Trißl: Bioinformatik für Biophysiker 2 Exercise 1 Global alignment using dynamic programming Write a program to
Aufgabenblatt 4 Silke Trißl Wissensmanagement in der Bioinformatik Zuerst! FRAGEN? Silke Trißl: Bioinformatik für Biophysiker 2 Exercise 1 Global alignment using dynamic programming Write a program to
Bioinformatik. Substitutionsmatrizen BLAST. Ulf Leser Wissensmanagement in der. Bioinformatik
 Bioinformatik Substitutionsmatrizen BLAST Ulf Leser Wissensmanagement in der Bioinformatik Inhalt dieser Vorlesung Substitutionsmatrizen: PAM und BLOSSUM Suche in Datenbanken: Basic Local Alignment Search
Bioinformatik Substitutionsmatrizen BLAST Ulf Leser Wissensmanagement in der Bioinformatik Inhalt dieser Vorlesung Substitutionsmatrizen: PAM und BLOSSUM Suche in Datenbanken: Basic Local Alignment Search
Graphdurchmusterung, Breiten- und Tiefensuche
 Prof. Thomas Richter 18. Mai 2017 Institut für Analysis und Numerik Otto-von-Guericke-Universität Magdeburg thomas.richter@ovgu.de Material zur Vorlesung Algorithmische Mathematik II am 18.05.2017 Graphdurchmusterung,
Prof. Thomas Richter 18. Mai 2017 Institut für Analysis und Numerik Otto-von-Guericke-Universität Magdeburg thomas.richter@ovgu.de Material zur Vorlesung Algorithmische Mathematik II am 18.05.2017 Graphdurchmusterung,
Bioinformatik für Biochemiker
 Bioinformatik für Biochemiker Oliver Kohlbacher WS 2009/2010 9. Multiples Alignment II Abt. Simulation biologischer Systeme WSI/ZBIT, Eberhard Karls Universität Tübingen Übersicht T-COFFEE Probleme bei
Bioinformatik für Biochemiker Oliver Kohlbacher WS 2009/2010 9. Multiples Alignment II Abt. Simulation biologischer Systeme WSI/ZBIT, Eberhard Karls Universität Tübingen Übersicht T-COFFEE Probleme bei
c) {abcde, abcfg, bcade, bcafg} d) {ade, afg, bcde, bcfg} c) {abcabc} d) {abcbc, abc, a} c) {aa, ab, ba, bb} d) {{aa}, {ab}, {ba}, {bb}}
 2 Endliche Automaten Fragen 1. Was ergibt sich bei {a, bc} {de, fg}? a) {abc, defg} b) {abcde, abcfg} c) {abcde, abcfg, bcade, bcafg} d) {ade, afg, bcde, bcfg} 2. Was ergibt sich bei {abc, a} {bc, λ}?
2 Endliche Automaten Fragen 1. Was ergibt sich bei {a, bc} {de, fg}? a) {abc, defg} b) {abcde, abcfg} c) {abcde, abcfg, bcade, bcafg} d) {ade, afg, bcde, bcfg} 2. Was ergibt sich bei {abc, a} {bc, λ}?
Bayesianische Netzwerke - Lernen und Inferenz
 Bayesianische Netzwerke - Lernen und Inferenz Manuela Hummel 9. Mai 2003 Gliederung 1. Allgemeines 2. Bayesianische Netzwerke zur Auswertung von Genexpressionsdaten 3. Automatische Modellselektion 4. Beispiel
Bayesianische Netzwerke - Lernen und Inferenz Manuela Hummel 9. Mai 2003 Gliederung 1. Allgemeines 2. Bayesianische Netzwerke zur Auswertung von Genexpressionsdaten 3. Automatische Modellselektion 4. Beispiel
Routing A lgorithmen Algorithmen Begriffe, Definitionen Wegewahl Verkehrslenkung
 Begriffe, Definitionen Routing (aus der Informatik) Wegewahl oder Verkehrslenkung bezeichnet in der Telekommunikation das Festlegen von Wegen für Nachrichtenströme bei der Nachrichtenübermittlung über
Begriffe, Definitionen Routing (aus der Informatik) Wegewahl oder Verkehrslenkung bezeichnet in der Telekommunikation das Festlegen von Wegen für Nachrichtenströme bei der Nachrichtenübermittlung über
Datenstrukturen und Algorithmen (SS 2013)
 Datenstrukturen und Algorithmen (SS 2013) Übungsblatt 10 Abgabe: Montag, 08.07.2013, 14:00 Uhr Die Übungen sollen in Gruppen von zwei bis drei Personen bearbeitet werden. Schreiben Sie die Namen jedes
Datenstrukturen und Algorithmen (SS 2013) Übungsblatt 10 Abgabe: Montag, 08.07.2013, 14:00 Uhr Die Übungen sollen in Gruppen von zwei bis drei Personen bearbeitet werden. Schreiben Sie die Namen jedes
Greedy Algorithms - Gierige Algorithmen
 Greedy Algorithms - Gierige Algorithmen Marius Burfey 23. Juni 2009 Inhaltsverzeichnis 1 Greedy Algorithms 1 2 Interval Scheduling - Ablaufplanung 2 2.1 Problembeschreibung....................... 2 2.2
Greedy Algorithms - Gierige Algorithmen Marius Burfey 23. Juni 2009 Inhaltsverzeichnis 1 Greedy Algorithms 1 2 Interval Scheduling - Ablaufplanung 2 2.1 Problembeschreibung....................... 2 2.2
2. Woche Eindeutige Entschlüsselbarleit, Sätze von Kraft und McMillan, Huffmancodierung
 2 Woche Eindeutige Entschlüsselbarleit, Sätze von Kraft und McMillan, Huffmancodierung 2 Woche: Eindeutige Entschlüsselbarleit, Sätze von Kraft und McMillan, Huffmancodierung 24/ 44 Zwei Beispiele a 0
2 Woche Eindeutige Entschlüsselbarleit, Sätze von Kraft und McMillan, Huffmancodierung 2 Woche: Eindeutige Entschlüsselbarleit, Sätze von Kraft und McMillan, Huffmancodierung 24/ 44 Zwei Beispiele a 0
a) Fügen Sie die Zahlen 39, 38, 37 und 36 in folgenden (2, 3)-Baum ein:
 1 Aufgabe 8.1 (P) (2, 3)-Baum a) Fügen Sie die Zahlen 39, 38, 37 und 36 in folgenden (2, 3)-Baum ein: Zeichnen Sie, was in jedem Schritt passiert. b) Löschen Sie die Zahlen 65, 70 und 100 aus folgendem
1 Aufgabe 8.1 (P) (2, 3)-Baum a) Fügen Sie die Zahlen 39, 38, 37 und 36 in folgenden (2, 3)-Baum ein: Zeichnen Sie, was in jedem Schritt passiert. b) Löschen Sie die Zahlen 65, 70 und 100 aus folgendem
Grundlagen: Algorithmen und Datenstrukturen
 Technische Universität München Fakultät für Informatik Lehrstuhl für Effiziente Algorithmen Dr. Hanjo Täubig Tobias Lieber Sommersemester 2011 Übungsblatt 1 16. September 2011 Grundlagen: Algorithmen und
Technische Universität München Fakultät für Informatik Lehrstuhl für Effiziente Algorithmen Dr. Hanjo Täubig Tobias Lieber Sommersemester 2011 Übungsblatt 1 16. September 2011 Grundlagen: Algorithmen und
Uninformierte Suche in Java Informierte Suchverfahren
 Uninformierte Suche in Java Informierte Suchverfahren Stephan Schwiebert WS 2008/2009 Sprachliche Informationsverarbeitung Institut für Linguistik Universität zu Köln 8-Damen-Problem Gegeben: Schachbrett
Uninformierte Suche in Java Informierte Suchverfahren Stephan Schwiebert WS 2008/2009 Sprachliche Informationsverarbeitung Institut für Linguistik Universität zu Köln 8-Damen-Problem Gegeben: Schachbrett
NP-vollständige Probleme
 Effiziente Algorithmen Lösen NP-vollständiger Probleme 256 NP-vollständige Probleme Keine polynomiellen Algorithmen, falls P NP. Viele wichtige Probleme sind NP-vollständig. Irgendwie müssen sie gelöst
Effiziente Algorithmen Lösen NP-vollständiger Probleme 256 NP-vollständige Probleme Keine polynomiellen Algorithmen, falls P NP. Viele wichtige Probleme sind NP-vollständig. Irgendwie müssen sie gelöst
Lernmodul 7 Algorithmus von Dijkstra
 Folie 1 von 30 Lernmodul 7 Algorithmus von Dijkstra Quelle: http://www.map24.de Folie 2 von 30 Algorithmus von Dijkstra Übersicht Kürzester Weg von A nach B in einem Graphen Problemstellung: Suche einer
Folie 1 von 30 Lernmodul 7 Algorithmus von Dijkstra Quelle: http://www.map24.de Folie 2 von 30 Algorithmus von Dijkstra Übersicht Kürzester Weg von A nach B in einem Graphen Problemstellung: Suche einer
Algorithmen I - Tutorium 28 Nr. 12
 Algorithmen I - Tutorium 28 Nr. 12 20.07.2017: Spaß mit Dynamischer und Linearer Programmierung Marc Leinweber marc.leinweber@student.kit.edu INSTITUT FÜR THEORETISCHE INFORMATIK (ITI), PROF. DR. JÖRN
Algorithmen I - Tutorium 28 Nr. 12 20.07.2017: Spaß mit Dynamischer und Linearer Programmierung Marc Leinweber marc.leinweber@student.kit.edu INSTITUT FÜR THEORETISCHE INFORMATIK (ITI), PROF. DR. JÖRN
Effiziente Algorithmen 2
 Effiziente Algorithmen 2 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Algorithmen
Effiziente Algorithmen 2 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Algorithmen
Theoretische Grundlagen der Informatik
 Theoretische Grundlagen der Informatik Vorlesung am 20. November 2014 INSTITUT FÜR THEORETISCHE 0 KIT 20.11.2014 Universität des Dorothea Landes Baden-Württemberg Wagner - Theoretische und Grundlagen der
Theoretische Grundlagen der Informatik Vorlesung am 20. November 2014 INSTITUT FÜR THEORETISCHE 0 KIT 20.11.2014 Universität des Dorothea Landes Baden-Württemberg Wagner - Theoretische und Grundlagen der
Approximationsalgorithmen
 Effiziente Algorithmen Lösen NP-vollständiger Probleme 320 Approximationsalgorithmen In polynomieller Zeit lässen sich nicht exakte Lösungen von NP-harten Problemen berechnen. Approximationsalgorithmen
Effiziente Algorithmen Lösen NP-vollständiger Probleme 320 Approximationsalgorithmen In polynomieller Zeit lässen sich nicht exakte Lösungen von NP-harten Problemen berechnen. Approximationsalgorithmen
3 Codierung diskreter Quellen. Quelle Quellcodierer Kanalcodierer reduziert die benötigte Datenmenge. fügt Daten zur Fehlerkorrektur ein.
 3 Codierung diskreter Quellen 3 Einführung 32 Ungleichmäßige Codierung 33 Präfix-Codes 34 Grenzen der Code-Effizienz 35 Optimal-Codierung 3 Zusammenfassung < 24 / 228 > 3 Codierung diskreter Quellen Quelle
3 Codierung diskreter Quellen 3 Einführung 32 Ungleichmäßige Codierung 33 Präfix-Codes 34 Grenzen der Code-Effizienz 35 Optimal-Codierung 3 Zusammenfassung < 24 / 228 > 3 Codierung diskreter Quellen Quelle
Minimal spannende Bäume
 http://www.uni-magdeburg.de/harbich/ Minimal spannende Fakultät für Informatik Otto-von-Guericke-Universität 2 Inhalt Definition Wege Untergraphen Kantengewichtete Graphen Minimal spannende Algorithmen
http://www.uni-magdeburg.de/harbich/ Minimal spannende Fakultät für Informatik Otto-von-Guericke-Universität 2 Inhalt Definition Wege Untergraphen Kantengewichtete Graphen Minimal spannende Algorithmen
Abbildung 1: Reduktion: CLIQUE zu VERTEX-COVER. links: Clique V = {u, v, x, y}. rechts:der Graph Ḡ mit VC V \ V = {w, z}
 u v u v z w z w y x y x Abbildung 1: Reduktion: CLIQUE zu VERTEX-COVER. links: Clique V = {u, v, x, y}. rechts:der Graph Ḡ mit VC V \ V = {w, z} Definition 0.0.1 (Vertex Cover (VC)). Gegeben: Ein ungerichteter
u v u v z w z w y x y x Abbildung 1: Reduktion: CLIQUE zu VERTEX-COVER. links: Clique V = {u, v, x, y}. rechts:der Graph Ḡ mit VC V \ V = {w, z} Definition 0.0.1 (Vertex Cover (VC)). Gegeben: Ein ungerichteter
Operations Research. Ganzzahlige lineare Programme. ganzzahlige lineare Programme. Ganzzahlige lineare Programme. Rainer Schrader. 25.
 Operations Research Rainer Schrader Ganzzahlige lineare Programme Zentrum für Angewandte Informatik Köln 25. Juni 2007 1 / 49 2 / 49 Ganzzahlige lineare Programme Gliederung ganzzahlige lineare Programme
Operations Research Rainer Schrader Ganzzahlige lineare Programme Zentrum für Angewandte Informatik Köln 25. Juni 2007 1 / 49 2 / 49 Ganzzahlige lineare Programme Gliederung ganzzahlige lineare Programme
Unterlagen zur Vorlesung Algebra und Geometrie in der Schule: Grundwissen über Affine Geometrie. Sommersemester Franz Pauer
 Unterlagen zur Vorlesung Algebra und Geometrie in der Schule: Grundwissen über Affine Geometrie Sommersemester 2009 Franz Pauer INSTITUT FÜR MATHEMATIK, UNIVERSITÄT INNSBRUCK, TECHNIKERSTRASSE 13, 6020
Unterlagen zur Vorlesung Algebra und Geometrie in der Schule: Grundwissen über Affine Geometrie Sommersemester 2009 Franz Pauer INSTITUT FÜR MATHEMATIK, UNIVERSITÄT INNSBRUCK, TECHNIKERSTRASSE 13, 6020
Wir wollen nun die Behauptung beweisen, dass die Laufzeit von SELECT linear ist, also dass T (n) = O(n) gilt.
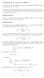 Abschätzung für die Rekursion von SELECT Wir wollen nun die Behauptung beweisen, dass die Laufzeit von SELECT linear ist, also dass T (n) = O(n) gilt. Wir nehmen erst einmal an, dass eine Konstante d existiert,
Abschätzung für die Rekursion von SELECT Wir wollen nun die Behauptung beweisen, dass die Laufzeit von SELECT linear ist, also dass T (n) = O(n) gilt. Wir nehmen erst einmal an, dass eine Konstante d existiert,
Routing Algorithmen. Begriffe, Definitionen
 Begriffe, Definitionen Routing (aus der Informatik) Wegewahl oder Verkehrslenkung bezeichnet in der Telekommunikation das Festlegen von Wegen für Nachrichtenströme bei der Nachrichtenübermittlung über
Begriffe, Definitionen Routing (aus der Informatik) Wegewahl oder Verkehrslenkung bezeichnet in der Telekommunikation das Festlegen von Wegen für Nachrichtenströme bei der Nachrichtenübermittlung über
Datenstrukturen & Algorithmen
 Datenstrukturen & Algorithmen Matthias Zwicker Universität Bern Frühling 2010 Übersicht Dynamische Programmierung Einführung Ablaufkoordination von Montagebändern Längste gemeinsame Teilsequenz Optimale
Datenstrukturen & Algorithmen Matthias Zwicker Universität Bern Frühling 2010 Übersicht Dynamische Programmierung Einführung Ablaufkoordination von Montagebändern Längste gemeinsame Teilsequenz Optimale
Parallele Algorithmen in der Bildverarbeitung
 Seminar über Algorithmen - SoSe 2009 Parallele Algorithmen in der Bildverarbeitung von Christopher Keiner 1 Allgemeines 1.1 Einleitung Parallele Algorithmen gewinnen immer stärker an Bedeutung. Es existieren
Seminar über Algorithmen - SoSe 2009 Parallele Algorithmen in der Bildverarbeitung von Christopher Keiner 1 Allgemeines 1.1 Einleitung Parallele Algorithmen gewinnen immer stärker an Bedeutung. Es existieren
9. Polynom- und Potenzreihenringe
 64 Andreas Gathmann 9. Polynom- und Potenzreihenringe Bevor wir mit der allgemeinen Untersuchung von Ringen fortfahren, wollen wir in diesem Kapitel kurz zwei sehr wichtige weitere Beispiele von Ringen
64 Andreas Gathmann 9. Polynom- und Potenzreihenringe Bevor wir mit der allgemeinen Untersuchung von Ringen fortfahren, wollen wir in diesem Kapitel kurz zwei sehr wichtige weitere Beispiele von Ringen
Mathematik II für Studierende der Informatik. Wirtschaftsinformatik (Analysis und lineare Algebra) im Sommersemester 2016
 und Wirtschaftsinformatik (Analysis und lineare Algebra) im Sommersemester 2016 25. April 2016 Die Dimensionsformel Definition 3.9 Sei f : V W eine lineare Abbildung zwischen zwei K-Vektorräumen. Der Kern
und Wirtschaftsinformatik (Analysis und lineare Algebra) im Sommersemester 2016 25. April 2016 Die Dimensionsformel Definition 3.9 Sei f : V W eine lineare Abbildung zwischen zwei K-Vektorräumen. Der Kern
Effiziente Algorithmen (SS2015)
 Effiziente Algorithmen (SS205) Kapitel 5 Approximation II Walter Unger Lehrstuhl für Informatik 2.06.205 07:59 5 Inhaltsverzeichnis < > Walter Unger 5.7.205 :3 SS205 Z Inhalt I Set Cover Einleitung Approximation
Effiziente Algorithmen (SS205) Kapitel 5 Approximation II Walter Unger Lehrstuhl für Informatik 2.06.205 07:59 5 Inhaltsverzeichnis < > Walter Unger 5.7.205 :3 SS205 Z Inhalt I Set Cover Einleitung Approximation
1 Zahlentheorie. 1.1 Kongruenzen
 3 Zahlentheorie. Kongruenzen Der letzte Abschnitt zeigte, daß es sinnvoll ist, mit großen Zahlen möglichst einfach rechnen zu können. Oft kommt es nicht darauf, an eine Zahl im Detail zu kennen, sondern
3 Zahlentheorie. Kongruenzen Der letzte Abschnitt zeigte, daß es sinnvoll ist, mit großen Zahlen möglichst einfach rechnen zu können. Oft kommt es nicht darauf, an eine Zahl im Detail zu kennen, sondern
MBI: Sequenzvergleich ohne Alignment
 MBI: Sequenzvergleich ohne Alignment Bernhard Haubold 12. November 2013 Wiederholung Exaktes & inexaktes Matching Das exakte Matching Problem Naive Lösung Präprozessierung Muster(Pattern): Z-Algorithmus,
MBI: Sequenzvergleich ohne Alignment Bernhard Haubold 12. November 2013 Wiederholung Exaktes & inexaktes Matching Das exakte Matching Problem Naive Lösung Präprozessierung Muster(Pattern): Z-Algorithmus,
Theoretische Informatik 1
 Theoretische Informatik 1 Approximierbarkeit David Kappel Institut für Grundlagen der Informationsverarbeitung Technische Universität Graz 02.07.2015 Übersicht Das Problem des Handelsreisenden TSP EUCLIDEAN-TSP
Theoretische Informatik 1 Approximierbarkeit David Kappel Institut für Grundlagen der Informationsverarbeitung Technische Universität Graz 02.07.2015 Übersicht Das Problem des Handelsreisenden TSP EUCLIDEAN-TSP
Algorithmische Bioinformatik 1
 Algorithmische Bioinformatik 1 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Algorithmen
Algorithmische Bioinformatik 1 Dr. Hanjo Täubig Lehrstuhl für Effiziente Algorithmen (Prof. Dr. Ernst W. Mayr) Institut für Informatik Technische Universität München Sommersemester 2009 Übersicht Algorithmen
21. Greedy Algorithmen. Aktivitätenauswahl, Fractional Knapsack Problem, Huffman Coding Cormen et al, Kap. 16.1, 16.3
 581 21. Greedy Algorithmen Aktivitätenauswahl, Fractional Knapsack Problem, Huffman Coding Cormen et al, Kap. 16.1, 16.3 Aktivitäten Auswahl 582 Koordination von Aktivitäten, die gemeinsame Resource exklusiv
581 21. Greedy Algorithmen Aktivitätenauswahl, Fractional Knapsack Problem, Huffman Coding Cormen et al, Kap. 16.1, 16.3 Aktivitäten Auswahl 582 Koordination von Aktivitäten, die gemeinsame Resource exklusiv
