Computergestützte Modellierung biologischer Prozesse Computational modelling of biological processes
|
|
|
- Innozenz Acker
- vor 7 Jahren
- Abrufe
Transkript
1 Computergestützte Modellierung biologischer Computational modelling of biological processes Wierling, Christoph; Herwig, Ralf Max-Planck-Institut für molekulare Genetik, Berlin Korrespondierender Autor Zusammenfassung Die Nachbildung biologischer im Computer bietet die Möglichkeit, durch gezielte virtuelle Störungen neue Effekte vorherzusagen, die dann im Experiment überprüft werden können. Solche Vorhersagen sind für viele praktische Anwendungen sehr nützlich, z.b. für die Entwicklung neuer Medikamente. In der Arbeitsgruppe Bioinformatik wurden in den letzten Jahren verschiedene Entwicklungen vorgenommen, die eine Modellierung von biologischen n unterstützen, wie z.b. eine Datenbank zur Integration humaner funktioneller Interaktionen (ConsensusPathDB) und das Modellierungssystem PyBioS. Summary The development of computational models for biological processes opens the possibility to predict new effects of virtual targeted perturbations that can in turn be validated by experimental observations. Such predictions are highly relevant for many practical applications, for example the development of new drugs. Through the last years, the Bioinformatics group has been developing different tools, methods, and databases that support the computational modeling of biological processes, such as the pathway integration database ConsensusPathDB and the modeling system PyBioS. Einführung Die Erstellung mathematischer Modelle ist ein dreistufiges Verfahren. Im ersten Schritt müssen die Randbedingungen des Modells definiert werden. Dazu gehören die Bestimmung der Modellkomponenten; bei biologischen Modellen sind das in der Regel Proteine, Metabolite, Gene und deren Initialwerte. Im zweiten Schritt werden die Interaktionen zwischen den Modellkomponenten festgelegt, also das Reaktionsnetzwerk des Modells. Im dritten Schritt erfolgt die Belegung der Modellreaktionen mit kinetischer Information, das heißt mit den mathematischen Gesetzen und kinetischen Parametern, die die Dynamik des Modells bestimmen [1]. Die Arbeitsgruppe Bioinformatik der Abteilung Analyse des Vertebratengenoms am Max-Planck-Institut für molekulare Genetik beschäftigt sich bereits seit einigen Jahren mit der Entwicklung von Software und Methodologie zur Erstellung derartiger mathematischer Modelle. Diese dienen einer Vielzahl von Anwendungen, zum Beispiel der Entwicklung von Krankheitsmodellen für verschiedene Tumortypen, für Typ Max-Planck-Gesellschaft 1/6
2 Diabetes mellitus, für Modelle zur Beschreibung der Zellantwort auf Chemikalien sowie für Modelle zur Entwicklungsbiologie im Allgemeinen. Markeridentifizierung Krankheiten können durch so genannte molekulare Marker (Gene, Proteine, Metabolite) im Labor diagnostiziert werden. Die Identifizierung spezifischer molekularer Marker, zum Beispiel zur frühzeitigen Diagnose des Krankheitsausbruchs, ist ein wichtiger Bestandteil der Genomforschung. Zur Markeridentifizierung werden zumeist statistische Verfahren der Bioinformatik eingesetzt. Ein typischer Ansatz besteht im statistischen Vergleich (Hypothesentest) von verschiedenen Zuständen, beispielsweise erkrankt gegen gesund oder behandelt gegen unbehandelt [2]. Wenn wenig Vorwissen vorhanden ist, werden dazu im Allgemeinen genomweite experimentelle Techniken wie microarrays oder Hochdurchsatz-Sequenzierungen bei der Genexpressionsanalyse genutzt. Bioinformatische Verfahren zur Markeridentifizierung sind dabei eng mit der Prozessierung der Daten (Qualitätskontrolle, Normalisierung) verbunden. Statistische Testverfahren werden benutzt, um die Signifikanz der Änderungen der Markerexpression beim Vergleich der verschiedenen Zustände zu bewerten. Neben diesen Ansätzen für Einzelvergleiche kommen immer mehr auch integrative Ansätze zum Einsatz. Diese Ansätze beziehen die zahlreichen öffentlich verfügbaren Datensätze in die Analyse mit ein (Abb. 1). Die in Einzelvergleichen gewonnene Information kann so auf ihre Allgemeingültigkeit hin überprüft werden, um möglichst robuste Marker zu identifizieren [3]. Meta-Analyse von Typ-2 Diabetes mellitus-daten. Über einen Webserver ( -genem iner.m olgen.m pg.de) sind die Ergebnisse zugänglich. Ergebnisse für einzelne Gene, hier PDK4, werden über die verschiedenen Einzelstudien hinweg gezeigt (linkes Bild). Das Gen zeigt in diesem Beispiel eine Krankheitsrelevanz bezüglich vieler Einzelstudien verschiedener Gewebe in Mensch und Maus und kann daher als robuster Marker eingestuft werden. Rechtes Bild: Genregulationsnetzwerk aus Markern zum Typ-2 Diabetes m ellitus. Transkriptionsfaktoren und deren Zielgene sind durch Kanten verbunden. Dicke Enden der Kanten zeigen auf die Transkriptionsfaktoren, also die regulierenden Gene, dünne Enden auf die Zielgene, also die regulierten Genen. Max-Planck-Institut für m olekulare Genetik/Herwig Integration humaner funktioneller Netzwerke Es existiert bereits eine Vielzahl (> 200) öffentlich zugänglicher Datenbanken, in denen biologische 2009 Max-Planck-Gesellschaft 2/6
3 annotiert und visualisiert werden. Diese Datenbanken (beispielsweise KEGG [4] oder Reactome [5]) bilden die Basis zur Bestimmung der Netzwerke, die die Modellkomponenten miteinander verbinden. Ein fundamentaler Nachteil in der Bestimmung der Reaktionsnetzwerke besteht in der Diversität der Datenbankannotationen, also der Tatsache, dass Datenbanken oft nur einen speziellen Datentyp abdecken, zum Beispiel Daten für Protein-Protein- Interaktionen, für metabolische Reaktionen oder für Signalwege. Biologische umfassen jedoch üblicherweise mehrere dieser Datentypen. Dies führt in der Praxis dazu, dass ein Anwender viele verschiedene Datenbanken abrufen muss, um die nötige Information über das Reaktionsnetzwerk zu bekommen. In der Arbeitsgruppe Bioinformatik wurde deshalb die integrative Datenbank ConsensusPathDB entwickelt, die die verschiedenen Interaktionstypen integriert und eine umfassende Annotation ermöglicht [6]. ConsensusPathDB bündelt zurzeit den Inhalt von 12 primären Datenbanken für humane funktionelle Interaktionen. Sie enthält Information über verschiedene Komponenten (Proteine, Gene, Metabolite etc.) und verschiedene funktionelle Interaktionen und deckt dabei 1689 humane biologische ab (zum Beispiel den Insulin-Signalweg, Glykolyse etc.). Diese Integration wurde ermöglicht durch die Entwicklung eines einheitlichen Datenbankschemas, das in der Lage ist, die verschiedenen Typen von Interaktionen und Modellkomponenten zu integrieren. ConsensusPathDB ist im Internet über einen Webserver frei zugänglich. Verschiedene Funktionalitäten erlauben beispielsweise die Suche nach Reaktionen von ausgewählten Proteinen, nach kürzesten Wegen zwischen zwei Proteinen, nach Methoden zur Identifizierung angereicherter und nach Visualisierungskomponenten. Netzwerke können in den gängigen Formaten PSI-MI, BioPAX und SBML importiert und exportiert werden, was eine weitgehende Kompatibilität mit anderen Systemen ermöglicht (Abb. 2). ConsensusPathDB. Die Abbildung zeigt die wesentlichen Funktionalitäten der Nutzerschnittstelle, beispielsweise die Suche nach spezifischen Kom ponenten und Netzwerken, Modellim port und export sowie die Visualisierung der verschiedenen Interaktionstypen (Knoten) und deren Herkunft aus den Prim ärdatenbanken (Kanten). Max-Planck-Institut für m olekulare Genetik/Kam burov Modellierung biologischer mit PyBioS Aufbauend auf der Struktur des Reaktions- und Interaktionsnetzwerks kann ein mathematisches Modell erstellt werden, welches beispielsweise durch ein gewöhnliches Differenzialgleichungssystem gegeben ist. Ein derartiges Modell beschreibt die zeitlichen Konzentrationsänderungen der Modellkomponenten. Für die 2009 Max-Planck-Gesellschaft 3/6
4 derartiges Modell beschreibt die zeitlichen Konzentrationsänderungen der Modellkomponenten. Für die Erstellung eines solchen Modells benötigt man zudem kinetische Gesetze, die die Geschwindigkeiten der einzelnen Reaktionen beschreiben. Beispiele für eine derartige Kinetik sind unter anderem die von Michaelis und Menten abgeleitete Kinetik für enzymkatalysierte Reaktionen oder die Massenwirkungskinetik. Die genaue Geschwindigkeit einer einzelnen Reaktion wird dabei durch kinetische Parameter und die Konzentrationen der beteiligten Reaktionspartner festgelegt. Kinetische Parameter müssen experimentell ermittelt oder an gemessene Zustandsänderungen angepasst werden. Vielfach ist es aber auch möglich, Näherungen für die kinetischen Modellparameter zu verwenden, um qualitative Aussagen über das Modellverhalten zu treffen. Für die Erstellung mathematischer Modelle ist in der Arbeitsgruppe Bioinformatik das Computerprogramm PyBioS entwickelt worden [1,7,8]. PyBioS hat eine web-basierte Benutzerschnittstelle (Abb. 3). Das Programm automatisiert die Generierung und Simulation mathematischer Modelle und ermöglicht dadurch auch die Erstellung und Untersuchung großer Modelle. PyBioS automatisiert auch die Integration von Reaktionsnetzwerken aus Datenbanken wie KEGG, Reactome oder ConsensusPathDB und bietet eine Vielzahl vordefinierter kinetischer Gesetze an. Dies ermöglicht eine schnelle Modellerstellung und vermeidet Fehler in der Modellentwicklung. Ferner hat PyBioS auch vielfältige Funktionalitäten für die Visualisierung der Simulationsergebnisse, wie zum Beispiel die Darstellung von zeitlich verlaufenden Konzentrationsänderungen. Neben der reinen Simulation ist auch die Modellanalyse von großer Bedeutung. Hierfür bietet PyBioS beispielsweise die Möglichkeit, den Einfluss eines Modellparameters auf das Modellverhalten im stationären Zustand zu untersuchen. Eine derartige Sensitivitätsanalyse dient der Identifizierung kritischer Modellparameter. Die Web-basierte Benutzerschnittstelle des Modellierungssystems PyBioS beinhaltet eine Sam m lung verschiedener Modelle (A ). Die hierarchische Struktur eines einzelnen Modells orientiert sich dabei an der zellulären Kom partim entierung (B). Über verschiedene Reiter kann der Benutzer auf die unterschiedlichen Funktionalitäten des Program m s zugreifen. Dies um fasst zum Beispiel die Visualisierung des Reaktionsnetzwerks (C), die detaillierte Auflistung der Reaktionen (D) sowie die Darstellung der Sim ulationsergebnisse (E). Max-Planck-Institut für m olekulare Genetik/Wierling Zusammenfassung und Ausblick 2009 Max-Planck-Gesellschaft 4/6
5 In diesem Bericht wurden Werkzeuge für die verschiedenen Schritte bei der Erstellung und Analyse von computergestützten Modellen biologischer n vorgestellt. Diese Modelle ermöglichen eine Vorhersage des Verhaltens eines biologischen Systems,wenn zum Beispiel bestimmte Änderungen auftreten (Medikamentengabe, Umwelteinflüsse, Entwicklungsvorgänge). Die Modellierung von biologischen n, insbesondere von Krankheitsprozessen, besitzt ein hohes wissenschaftliches und wirtschaftliches Potenzial, ist jedoch zur Zeit noch zu generisch, um ein individuelles biologisches System, etwa das eines Patienten, exakt beschreiben zu können. Es wird in Zukunft darauf ankommen, gezielt molekulare Informationen in die mathematischen Modelle einzubauen, um beispielsweise Effekte von Mutationen oder individuelle Gen- und Proteinexpressionsdaten bei der Modellierung berücksichtigen zu können. Originalveröffentlichungen Nach Erweiterungen suchenbilderweiterungchanneltickerdateilistehtml- ErweiterungJobtickerKalendererweiterungLinkerweiterungMPG.PuRe-ReferenzMitarbeiter (Employee Editor)PersonenerweiterungPublikationserweiterungTeaser mit BildTextblockerweiterungVeranstaltungstickererweiterungVideoerweiterungVideolistenerweiterungYouTube- Erweiterung [1] E. Klipp, R. Herwig, A. Kowald, C. Wierling, H. Lehrach: Systems biology in practice. Wiley-VCH, Weinheim (2005). [2] R. Herwig, P. Aanstad, M. Clark, H. Lehrach: Statistical evaluation of differential expression on cdna nylon arrays with replicated experiments. Nucleic Acids Research 29, e117 (2001). [3] A. Rasche, H. Al-Hasani, R. Herwig: Meta-analysis approach identifies candidate genes and associated molecular networks for type-2 diabetes mellitus. BMC Genomics 9, e310 (2008). [4] M. Kanehisa, S. Goto: KEGG: Kyoto encyclopedia of genes and genomes. Nucleic Acids Research 28, (2000). [5] I. Vastrik, P. D Eustachio, E. Schmidt, G. Joshi-Tope, G. Gopinath, D. Croft, B. de Bono, M. Gillespie, B. Jassal, S. Lewis, L. Matthews, G. Wu, E. Birney, L. Stein: Reactome: a knowledge base of biologic pathways and processes. Genome Biology 8, R39 (2007). [6] A. Kamburov, C. Wierling, H. Lehrach, R. Herwig: ConsensusPathDB - a database for integrating human functional interaction networks. Nucleic Acids Research 37, D623-D628 (2008) Max-Planck-Gesellschaft 5/6
6 [7] C. Wierling: PyBioS - ein Modellierungs- und Simulationssystem für komplexe biologische. In: Forschung und wissenschaftliches Rechnen. Beiträge zum Heinz-Billing Preis 2005 (Hrsg.: K. Kremer, V. Macho), Nr. 69: Gesellschaft für wissenschaftliche Datenverarbeitung Göttingen (GWDG) (2006). [8] C. Wierling, R. Herwig, H. Lehrach: Resources, standards and tools for systems biology. Briefings in Functional Genomics and Proteomics 6, (2007) Max-Planck-Gesellschaft 6/6
Computergestützte Modellierung biologischer Prozesse
 Tätigkeitsbericht 2008 Entwicklungs- und Evolutionsbiologie/GenetikInformatik/Mathematik/Komplexe Systeme Computergestützte Modellierung biologischer Prozesse Wierling, Christoph; Herwig, Ralf; Max-Planck-Institut
Tätigkeitsbericht 2008 Entwicklungs- und Evolutionsbiologie/GenetikInformatik/Mathematik/Komplexe Systeme Computergestützte Modellierung biologischer Prozesse Wierling, Christoph; Herwig, Ralf; Max-Planck-Institut
Exercises to Introduction to Bioinformatics Assignment 5: Protein interaction networks. Samira Jaeger
 Exercises to Introduction to Bioinformatics Assignment 5: Protein interaction networks Samira Jaeger Aufgabe 1 Netzwerkzentralität (6P) In der Vorlesung haben Degree Centrality besprochen. Finde drei weitere
Exercises to Introduction to Bioinformatics Assignment 5: Protein interaction networks Samira Jaeger Aufgabe 1 Netzwerkzentralität (6P) In der Vorlesung haben Degree Centrality besprochen. Finde drei weitere
Einführung in die Angewandte Bioinformatik: Netzwerke
 Einführung in die Angewandte Bioinformatik: Netzwerke 15.07.2010 Prof. Dr. Sven Rahmann 1 Modellierung von Interaktionen Ein lebendes System besteht aus Molekülen (verschiedener Art) und funktioniert aufgrund
Einführung in die Angewandte Bioinformatik: Netzwerke 15.07.2010 Prof. Dr. Sven Rahmann 1 Modellierung von Interaktionen Ein lebendes System besteht aus Molekülen (verschiedener Art) und funktioniert aufgrund
Einführung in die Bioinformatik
 Einführung in die Bioinformatik Kay Nieselt Integrative Transkriptomik Zentrum für Bioinformatik Tübingen Kay.Nieselt@uni-tuebingen.de SS 2011 8. Biologische Netzwerke - Gut vernetzt hält besser Überblick
Einführung in die Bioinformatik Kay Nieselt Integrative Transkriptomik Zentrum für Bioinformatik Tübingen Kay.Nieselt@uni-tuebingen.de SS 2011 8. Biologische Netzwerke - Gut vernetzt hält besser Überblick
Untersuchung von Bindungsstellen zur Aktivierung von Genen Analysis of binding sites for the activation of genes
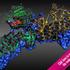 Untersuchung von Bindungsstellen zur Aktivierung von Genen Analysis of binding sites for the activation of genes Vingron, Martin Max-Planck-Institut für molekulare Genetik, Berlin Korrespondierender Autor
Untersuchung von Bindungsstellen zur Aktivierung von Genen Analysis of binding sites for the activation of genes Vingron, Martin Max-Planck-Institut für molekulare Genetik, Berlin Korrespondierender Autor
Einführung in die Bioinformatik
 Einführung in die Bioinformatik Kay Nieselt Integrative Transkriptomik Zentrum für Bioinformatik Tübingen Kay.Nieselt@uni-tuebingen.de SS 2012 5. Biologische Netzwerke - Gut vernetzt hält besser Überblick
Einführung in die Bioinformatik Kay Nieselt Integrative Transkriptomik Zentrum für Bioinformatik Tübingen Kay.Nieselt@uni-tuebingen.de SS 2012 5. Biologische Netzwerke - Gut vernetzt hält besser Überblick
Einführung in die Bioinformatik
 Einführung in die Bioinformatik Kay Nieselt Integrative Transkriptomik Zentrum für Bioinformatik Tübingen Kay.Nieselt@uni-tuebingen.de SS 2014 8. Biologische Netzwerke - Gut vernetzt hält besser Überblick
Einführung in die Bioinformatik Kay Nieselt Integrative Transkriptomik Zentrum für Bioinformatik Tübingen Kay.Nieselt@uni-tuebingen.de SS 2014 8. Biologische Netzwerke - Gut vernetzt hält besser Überblick
Vernetzung zur Integration biologischer Daten Making connections: Data integration in biology
 Vernetzung zur Integration biologischer Daten Making connections: Data integration in biology Schoof, Heiko Max-Planck-Institut für Pflanzenzüchtungsforschung, Köln Korrespondierender Autor E-Mail: schoof@mpiz-koeln.mpg.de
Vernetzung zur Integration biologischer Daten Making connections: Data integration in biology Schoof, Heiko Max-Planck-Institut für Pflanzenzüchtungsforschung, Köln Korrespondierender Autor E-Mail: schoof@mpiz-koeln.mpg.de
Bioinformatik: The Next Generation
 Bioinformatik: The Next Generation Prof. Dr. Caroline Friedel Lehr- und Forschungseinheit Bioinformatik Was ist Bioinformatik? Prof. Dr. Caroline Friedel, Tag der offenen Tür der LMU 2 Was ist Bioinformatik?
Bioinformatik: The Next Generation Prof. Dr. Caroline Friedel Lehr- und Forschungseinheit Bioinformatik Was ist Bioinformatik? Prof. Dr. Caroline Friedel, Tag der offenen Tür der LMU 2 Was ist Bioinformatik?
Bioinformatik: The Next Generation
 Bioinformatik: The Next Generation Prof. Dr. Caroline Friedel Lehr- und Forschungseinheit Bioinformatik Was ist Bioinformatik? Theoretische und Praktische Informatik Statistik, Mathematik Molekularbiologie,
Bioinformatik: The Next Generation Prof. Dr. Caroline Friedel Lehr- und Forschungseinheit Bioinformatik Was ist Bioinformatik? Theoretische und Praktische Informatik Statistik, Mathematik Molekularbiologie,
Institut für Künstliche Intelligenz
 Institut für Künstliche Intelligenz Prof. Sebstaian Rudolph --- Computational Logic Prof. Steffen Hölldobler --- Wissensverarbeitung Prof. Ivo F. Sbalzarini --- Wissenschaftliches Rechnen für Systembiologie
Institut für Künstliche Intelligenz Prof. Sebstaian Rudolph --- Computational Logic Prof. Steffen Hölldobler --- Wissensverarbeitung Prof. Ivo F. Sbalzarini --- Wissenschaftliches Rechnen für Systembiologie
Zurich Open Repository and Archive. Anatomie von Kommunikationsrollen. Methoden zur Identifizierung von Akteursrollen in gerichteten Netzwerken
 University of Zurich Zurich Open Repository and Archive Winterthurerstr. 190 CH-8057 Zurich http://www.zora.uzh.ch Year: 2008 Anatomie von Kommunikationsrollen. Methoden zur Identifizierung von Akteursrollen
University of Zurich Zurich Open Repository and Archive Winterthurerstr. 190 CH-8057 Zurich http://www.zora.uzh.ch Year: 2008 Anatomie von Kommunikationsrollen. Methoden zur Identifizierung von Akteursrollen
Statistische Methoden in der Bioinformatik
 Statistische Methoden in der Bioinformatik Prof. Dr. Jörg Rahnenführer Raum 720 Email: rahnenfuehrer@statistik. tu-.de Voraussetzungen: Vordiplom in Statistik, Mathematik, Datenanalyse, Informatik Zeiten
Statistische Methoden in der Bioinformatik Prof. Dr. Jörg Rahnenführer Raum 720 Email: rahnenfuehrer@statistik. tu-.de Voraussetzungen: Vordiplom in Statistik, Mathematik, Datenanalyse, Informatik Zeiten
VL Bioinformatik für Nebenfächler SS2018 Woche 1. Tim Conrad AG Medical Bioinformatics Institut für Mathematik & Informatik, Freie Universität Berlin
 VL Bioinformatik für Nebenfächler SS2018 Woche 1 Tim Conrad AG Medical Bioinformatics Institut für Mathematik & Informatik, Freie Universität Berlin Die Themen heute 1. Team 2. Tutorien 3. Prüfungsleistungen
VL Bioinformatik für Nebenfächler SS2018 Woche 1 Tim Conrad AG Medical Bioinformatics Institut für Mathematik & Informatik, Freie Universität Berlin Die Themen heute 1. Team 2. Tutorien 3. Prüfungsleistungen
Ontologien und Ontologiesprachen
 Ontologien und Ontologiesprachen Semantische Datenintegration SoSe2005 Uni Bremen Yu Zhao Gliederung 1. Was ist Ontologie 2. Anwendungsgebiete 3. Ontologiesprachen 4. Entwicklung von Ontologien 5. Zusammenfassung
Ontologien und Ontologiesprachen Semantische Datenintegration SoSe2005 Uni Bremen Yu Zhao Gliederung 1. Was ist Ontologie 2. Anwendungsgebiete 3. Ontologiesprachen 4. Entwicklung von Ontologien 5. Zusammenfassung
Hochdurchsatz Generierung und Analyse von Arabidopsis thaliana-proteinen
 MAX-PLANCK-INSTITUT FÜR MOLEKULARE GENETIK Hochdurchsatz Generierung und Analyse von Arabidopsis thaliana-proteinen Dissertation zur Erlangung der Doktorwürde des Fachbereichs Biologie, Chemie, Pharmazie
MAX-PLANCK-INSTITUT FÜR MOLEKULARE GENETIK Hochdurchsatz Generierung und Analyse von Arabidopsis thaliana-proteinen Dissertation zur Erlangung der Doktorwürde des Fachbereichs Biologie, Chemie, Pharmazie
Vorgänger für Pathway Studio und IPA. Herbsttagung der Bibliotheken der BM-Sektion / Elisabeth Schlagberger
 Vorgänger für Pathway Studio und IPA Herbsttagung der Bibliotheken der BM-Sektion 07.11.2016/08.11.2016 Elisabeth Schlagberger STRING und STITCH als Art Vorgänger- Systeme Unter http://string-db.org (Proteine/Gene)
Vorgänger für Pathway Studio und IPA Herbsttagung der Bibliotheken der BM-Sektion 07.11.2016/08.11.2016 Elisabeth Schlagberger STRING und STITCH als Art Vorgänger- Systeme Unter http://string-db.org (Proteine/Gene)
Das Unmessbare messen Softsensoren zur Prozessanalyse und Qualitätskontrolle. Christoph Kugler, Thomas Hochrein 26. Mai 2011
 Das Unmessbare messen Softsensoren zur Prozessanalyse und Qualitätskontrolle Christoph Kugler, Thomas Hochrein 26. Mai 2011 Das in Echtzeit und inline Unmessbare messen Softsensoren zur Prozessanalyse
Das Unmessbare messen Softsensoren zur Prozessanalyse und Qualitätskontrolle Christoph Kugler, Thomas Hochrein 26. Mai 2011 Das in Echtzeit und inline Unmessbare messen Softsensoren zur Prozessanalyse
Einführung Molekulare Bioinformatik
 Einführung Molekulare Bioinformatik Bernhard Haubold 22. Oktober 2013 Übersicht Was ist Bioinformatik? Kursstruktur Was ist Bioinformatik? Geschichtliche Entwicklung Information: Speicherung & Übertragung
Einführung Molekulare Bioinformatik Bernhard Haubold 22. Oktober 2013 Übersicht Was ist Bioinformatik? Kursstruktur Was ist Bioinformatik? Geschichtliche Entwicklung Information: Speicherung & Übertragung
Übungsblatt: Protein interaction networks. Ulf Leser and Samira Jaeger
 Übungsblatt: Protein interaction networks Ulf Leser and Samira Jaeger Aufgabe 1 Netzwerkzentralität (6P) In der Vorlesung haben Degree Centrality besprochen. Finde drei weitere etablierte Zentralitätsmaße
Übungsblatt: Protein interaction networks Ulf Leser and Samira Jaeger Aufgabe 1 Netzwerkzentralität (6P) In der Vorlesung haben Degree Centrality besprochen. Finde drei weitere etablierte Zentralitätsmaße
Metabolismus Modelle. Überblick über Stoffwechselwege aus KEGG Datenbank (Kyoto Encyclopedia of Genes and Genomes)
 Metabolismus Modelle Überblick über Stoffwechselwege aus KEGG Datenbank (Kyoto Encyclopedia of Genes and Genomes) www.genome.p/kegg Edda Klipp, Humboldt- zu Berlin Reproduktion experimenteller Befunde
Metabolismus Modelle Überblick über Stoffwechselwege aus KEGG Datenbank (Kyoto Encyclopedia of Genes and Genomes) www.genome.p/kegg Edda Klipp, Humboldt- zu Berlin Reproduktion experimenteller Befunde
Modell zur Einflussanalyse Ein Modell zur Einflussanalyse von Methodenänderungen in Entwicklungsprozessen
 Modell zur Einflussanalyse Ein Modell zur Einflussanalyse von Methodenänderungen in Entwicklungsprozessen Roland Koppe, Stefan Häusler, Axel Hahn 2 Übersicht Einleitung und Motivation Ansatz und Methodik
Modell zur Einflussanalyse Ein Modell zur Einflussanalyse von Methodenänderungen in Entwicklungsprozessen Roland Koppe, Stefan Häusler, Axel Hahn 2 Übersicht Einleitung und Motivation Ansatz und Methodik
Ausprägungsfach Bioinformatik im Rahmen des Bachelor-Studiengangs Informatik. CIBIV Center for Integrative Bioinformatics Vienna
 Ausprägungsfach Bioinformatik im Rahmen des Bachelor-Studiengangs Informatik Center for Integrative Bioinformatics Vienna (CIBIV) Max F. Perutz Laboratories (MFPL) Vienna, Austria http://www.cibiv.at CIBIV
Ausprägungsfach Bioinformatik im Rahmen des Bachelor-Studiengangs Informatik Center for Integrative Bioinformatics Vienna (CIBIV) Max F. Perutz Laboratories (MFPL) Vienna, Austria http://www.cibiv.at CIBIV
Intelligente Algorithmen zur Analyse zellulärer Spleißmechanismen Intelligent Algorithms for the Analysis of Cellular Splicing Mechanisms
 Intelligente Algorithmen zur Analyse zellulärer Spleißmechanismen Intelligent Algorithms for the Analysis of Cellular Splicing Mechanisms Rätsch, Gunnar Friedrich-Miescher-Laboratorium für biologische
Intelligente Algorithmen zur Analyse zellulärer Spleißmechanismen Intelligent Algorithms for the Analysis of Cellular Splicing Mechanisms Rätsch, Gunnar Friedrich-Miescher-Laboratorium für biologische
Wissenschaftlich-technische Entwicklungen im Bereich der Multiplex- und High-Throughput-Diagnostik. Karl J. Lackner
 Wissenschaftlich-technische Entwicklungen im Bereich der Multiplex- und High-Throughput-Diagnostik Karl J. Lackner Hochdurchsatzverfahren in der molekularen bzw. genetischen Diagnostik Next Generation-Sequenzierung
Wissenschaftlich-technische Entwicklungen im Bereich der Multiplex- und High-Throughput-Diagnostik Karl J. Lackner Hochdurchsatzverfahren in der molekularen bzw. genetischen Diagnostik Next Generation-Sequenzierung
IBE. Ulrich Mansmann 1,2 Jian Li 2,3
 IBE Computerbasierte individuelle Vorhersage des Ansprechens unter zielgerichteter Therapie bei Krebspatienten: Bioinformatische Grundlage und klinische Validierung Ulrich Mansmann 1,2 Jian Li 2,3 ulrich.mansmann@lmu.de
IBE Computerbasierte individuelle Vorhersage des Ansprechens unter zielgerichteter Therapie bei Krebspatienten: Bioinformatische Grundlage und klinische Validierung Ulrich Mansmann 1,2 Jian Li 2,3 ulrich.mansmann@lmu.de
Bioinformatik I (Einführung)
 Kay Diederichs, Sommersemester 2018 Bioinformatik I (Einführung) Algorithmen Sequenzen Strukturen PDFs unter http://strucbio.biologie.unikonstanz.de/~dikay/bioinformatik/ Klausur: Mo 30.7. 14:30-15:30
Kay Diederichs, Sommersemester 2018 Bioinformatik I (Einführung) Algorithmen Sequenzen Strukturen PDFs unter http://strucbio.biologie.unikonstanz.de/~dikay/bioinformatik/ Klausur: Mo 30.7. 14:30-15:30
Bayesianische Netzwerke - Lernen und Inferenz
 Bayesianische Netzwerke - Lernen und Inferenz Manuela Hummel 9. Mai 2003 Gliederung 1. Allgemeines 2. Bayesianische Netzwerke zur Auswertung von Genexpressionsdaten 3. Automatische Modellselektion 4. Beispiel
Bayesianische Netzwerke - Lernen und Inferenz Manuela Hummel 9. Mai 2003 Gliederung 1. Allgemeines 2. Bayesianische Netzwerke zur Auswertung von Genexpressionsdaten 3. Automatische Modellselektion 4. Beispiel
AlgoBio WS 16/17 Protein-DNA Interaktionen ChiP-Seq Datenanalyse. Annalisa Marsico
 AlgoBio WS 16/17 Protein-DNA Interaktionen ChiP-Seq Datenanalyse Annalisa Marsico 6.02.2017 Protein-DNA Interaktionen Häufig binden sich Proteine an DNA, um ihre biologische Funktion zu regulieren. Transkriptionsfaktoren
AlgoBio WS 16/17 Protein-DNA Interaktionen ChiP-Seq Datenanalyse Annalisa Marsico 6.02.2017 Protein-DNA Interaktionen Häufig binden sich Proteine an DNA, um ihre biologische Funktion zu regulieren. Transkriptionsfaktoren
Web Services Die Definition von Web Services in der Theorie und FNT-Command als Web Service in der Praxis
 Web Services Die Definition von Web Services in der Theorie und FNT-Command als Web Service in der Praxis Philipp Tendyra Web Service in kurzen Worten dient der Kommunikation zwischen verschiedenen Systemen
Web Services Die Definition von Web Services in der Theorie und FNT-Command als Web Service in der Praxis Philipp Tendyra Web Service in kurzen Worten dient der Kommunikation zwischen verschiedenen Systemen
Fernwartung mit IPX/S Geräten Konfiguration mit Fritz!Box 7270
 Fernwartung mit IPX/S 3.1.1 Geräten Konfiguration mit Fritz!Box 7270 GPG BUILDING AUTOMATION Dok.-Typ: Schritt-für-Schritt Anleitung Dok.-Nr. 9AKK106713A8893 Dok.-Version: 1.2 Abteilung: Global Support
Fernwartung mit IPX/S 3.1.1 Geräten Konfiguration mit Fritz!Box 7270 GPG BUILDING AUTOMATION Dok.-Typ: Schritt-für-Schritt Anleitung Dok.-Nr. 9AKK106713A8893 Dok.-Version: 1.2 Abteilung: Global Support
Verbindung von Test und Medikament soll Behandlungsstandard für Patienten mit nicht-kleinzelligem Lungenkarzinom (NSCLC) verbessern
 QIAGEN und Pfizer entwickeln Begleitdiagnostikum für neues Lungenkrebs-Präparat Verbindung von Test und Medikament soll Behandlungsstandard für Patienten mit nicht-kleinzelligem Lungenkarzinom (NSCLC)
QIAGEN und Pfizer entwickeln Begleitdiagnostikum für neues Lungenkrebs-Präparat Verbindung von Test und Medikament soll Behandlungsstandard für Patienten mit nicht-kleinzelligem Lungenkarzinom (NSCLC)
Zusammenfassung. Summary
 Medizinische Bioinformatik: Molekulare Modellierung von Krankheiten und Analyse relevanter Proteininteraktionen Medical Bioinformatics: Molecular Modeling of Diseases and Analysis of Relevant Protein Interactions
Medizinische Bioinformatik: Molekulare Modellierung von Krankheiten und Analyse relevanter Proteininteraktionen Medical Bioinformatics: Molecular Modeling of Diseases and Analysis of Relevant Protein Interactions
M0 BIO - Reaktionszeit
 M0 BIO - Reaktionszeit 1 Ziel des Versuches In diesem Versuch haben Sie die Möglichkeit, sich mit Messunsicherheiten vertraut zu machen. Die Analyse von Messunsicherheiten erfolgt hierbei an zwei Beispielen.
M0 BIO - Reaktionszeit 1 Ziel des Versuches In diesem Versuch haben Sie die Möglichkeit, sich mit Messunsicherheiten vertraut zu machen. Die Analyse von Messunsicherheiten erfolgt hierbei an zwei Beispielen.
SKOPOS Webinar 22. Mai 2018
 SKOPOS Webinar 22. Mai 2018 Marktforschung 2020: Künstliche Intelligenz und automatische Text Analysen? Christopher Harms, Consultant Research & Development 2 So? Terminator Exhibition: T-800 by Dick Thomas
SKOPOS Webinar 22. Mai 2018 Marktforschung 2020: Künstliche Intelligenz und automatische Text Analysen? Christopher Harms, Consultant Research & Development 2 So? Terminator Exhibition: T-800 by Dick Thomas
Algorithmen und Anwendungen zur Kartierung von Genomen
 Algorithmen und Anwendungen zur Kartierung von Genomen Dr. Dominik Grimm Probelehrveranstaltung Wissenschaftszentrum Straubing Hochschule Weihenstephan-Triesdorf Straubing, 14. Juli 2017 14. Juli 2017
Algorithmen und Anwendungen zur Kartierung von Genomen Dr. Dominik Grimm Probelehrveranstaltung Wissenschaftszentrum Straubing Hochschule Weihenstephan-Triesdorf Straubing, 14. Juli 2017 14. Juli 2017
Einführung in die Angewandte Bioinformatik: Proteinsequenz-Datenbanken
 Einführung in die Angewandte Bioinformatik: Proteinsequenz-Datenbanken 14.05.2009 Prof. Dr. Sven Rahmann 1 3 Proteinsequenz-Datenbanksysteme NCBI Entrez Proteins EBI SRS Proteins UniProt (empfohlen) 2
Einführung in die Angewandte Bioinformatik: Proteinsequenz-Datenbanken 14.05.2009 Prof. Dr. Sven Rahmann 1 3 Proteinsequenz-Datenbanksysteme NCBI Entrez Proteins EBI SRS Proteins UniProt (empfohlen) 2
Auditive Wahrnehmung in Computerspielen. Funktionelle Aspekte zum Einsatz von Musik und Klang
 Medien Marcus Jobs Auditive Wahrnehmung in Computerspielen. Funktionelle Aspekte zum Einsatz von Musik und Klang Bachelorarbeit Bibliografische Information der Deutschen Nationalbibliothek: Die Deutsche
Medien Marcus Jobs Auditive Wahrnehmung in Computerspielen. Funktionelle Aspekte zum Einsatz von Musik und Klang Bachelorarbeit Bibliografische Information der Deutschen Nationalbibliothek: Die Deutsche
Das Planen von Werkzeughandlungen Action planning in tool use
 Das Planen von Action planning in tool use Massen, Cristina; Lepper, Miriam; Prinz, Wolfgang Max-Planck-Institut für Kognitions- und, Leipzig Korrespondierender Autor E-Mail: massen@cbs.mpg.de Zusammenfassung
Das Planen von Action planning in tool use Massen, Cristina; Lepper, Miriam; Prinz, Wolfgang Max-Planck-Institut für Kognitions- und, Leipzig Korrespondierender Autor E-Mail: massen@cbs.mpg.de Zusammenfassung
Geo 241 Modellierung von Systemen Modulverantwortlicher: Dr. Krause. Sommersemester 2009
 Geo 241 Modellierung von Systemen Modulverantwortlicher: Dr. Krause Sensitivität Tino Wunderlich Matrikel 100028 Gliederung 1. Sensitivität in der Statistik ( Berechnung / Beispiel) 2. Sensitivität in
Geo 241 Modellierung von Systemen Modulverantwortlicher: Dr. Krause Sensitivität Tino Wunderlich Matrikel 100028 Gliederung 1. Sensitivität in der Statistik ( Berechnung / Beispiel) 2. Sensitivität in
Genexpressionsdatenbanken
 Genexpressionsdatenbanken ArrayExpress Gliederung Mikroarrays Struktur von Genexpressionsdatenbanken Arrayexpress Aufbau und Statistik Standardisierung Abfragen und Einstellen von Daten Mikroarrays Glasplatte
Genexpressionsdatenbanken ArrayExpress Gliederung Mikroarrays Struktur von Genexpressionsdatenbanken Arrayexpress Aufbau und Statistik Standardisierung Abfragen und Einstellen von Daten Mikroarrays Glasplatte
Das diesem Dokument zugrundeliegende Vorhaben wurde mit Mitteln des Bundesministeriums für Bildung und Forschung unter dem Förderkennzeichen
 Das diesem Dokument zugrundeliegende Vorhaben wurde mit Mitteln des Bundesministeriums für Bildung und Forschung unter dem Förderkennzeichen 16OH21005 gefördert. Die Verantwortung für den Inhalt dieser
Das diesem Dokument zugrundeliegende Vorhaben wurde mit Mitteln des Bundesministeriums für Bildung und Forschung unter dem Förderkennzeichen 16OH21005 gefördert. Die Verantwortung für den Inhalt dieser
Bioinformatik: The Next Generation
 Bioinformatik: The Next Generation Prof. Dr. Caroline Friedel Lehr- und Forschungseinheit Bioinformatik Was ist Bioinformatik? Theoretische und Praktische Informatik Statistik, Mathematik Molekularbiologie,
Bioinformatik: The Next Generation Prof. Dr. Caroline Friedel Lehr- und Forschungseinheit Bioinformatik Was ist Bioinformatik? Theoretische und Praktische Informatik Statistik, Mathematik Molekularbiologie,
Einführung in die Biophysik
 Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Predictive Analytics für Dummies. Click here if your download doesn"t start automatically
 Predictive Analytics für Dummies Click here if your download doesn"t start automatically Predictive Analytics für Dummies Anasse Bari, Mohamed Chaouchi, Tommy Jung Predictive Analytics für Dummies Anasse
Predictive Analytics für Dummies Click here if your download doesn"t start automatically Predictive Analytics für Dummies Anasse Bari, Mohamed Chaouchi, Tommy Jung Predictive Analytics für Dummies Anasse
Einführung in die Bioinformatik: Lernen mit Kernen
 Einführung in die Bioinformatik: Lernen mit Kernen Dr. Karsten Borgwardt Forschungsgruppe für Maschinelles Lernen und Bioinformatik Max-Planck-Institut für Intelligente Systeme & Max-Planck-Institut für
Einführung in die Bioinformatik: Lernen mit Kernen Dr. Karsten Borgwardt Forschungsgruppe für Maschinelles Lernen und Bioinformatik Max-Planck-Institut für Intelligente Systeme & Max-Planck-Institut für
Vergleich von Methoden zur Rekonstruktion von genregulatorischen Netzwerken (GRN)
 Exposé zur Bachelorarbeit: Vergleich von Methoden zur Rekonstruktion von genregulatorischen Netzwerken (GRN) Fakultät: Informatik, Humboldt-Universität zu Berlin Lijuan Shi 09.05.2013 Betreuer: Prof. Dr.
Exposé zur Bachelorarbeit: Vergleich von Methoden zur Rekonstruktion von genregulatorischen Netzwerken (GRN) Fakultät: Informatik, Humboldt-Universität zu Berlin Lijuan Shi 09.05.2013 Betreuer: Prof. Dr.
Gutartig oder bösartig? Die Diagnose aus dem Rechner
 Gutartig oder bösartig? Die Diagnose aus dem Rechner Eine Krankheit Drei Therapien Klinische Studie Im Mittel 75% 55% 35% Erfolg Drei unterschiedliche Formen dieser Krankheit A B C A B C 100% 60% 65% 40%
Gutartig oder bösartig? Die Diagnose aus dem Rechner Eine Krankheit Drei Therapien Klinische Studie Im Mittel 75% 55% 35% Erfolg Drei unterschiedliche Formen dieser Krankheit A B C A B C 100% 60% 65% 40%
Bioinformatik. Prof. H.-W. Mewes Lehrstuhl für Genomorientierte Bioinformatik Wissenschaftszentrum Weihenstephan Technische Universität München
 Bioinformatik Prof. H.-W. Mewes Lehrstuhl für Genomorientierte Bioinformatik Wissenschaftszentrum Weihenstephan Technische Universität München Prof. D. Frishman Fachgebiet Bioinformatik Wissenschaftszentrum
Bioinformatik Prof. H.-W. Mewes Lehrstuhl für Genomorientierte Bioinformatik Wissenschaftszentrum Weihenstephan Technische Universität München Prof. D. Frishman Fachgebiet Bioinformatik Wissenschaftszentrum
Neues zu advangeo I Umstellung auf ArcGIS 10
 Neues zu advangeo I Umstellung auf ArcGIS 10 Sven Etzold, Silke Noack (Beak) 04.05.2012 Anwendungsgebiete / Methodik advangeo Prediction Software Anwendungsgebiete Vorhersage von Ereignissen Abgrenzung
Neues zu advangeo I Umstellung auf ArcGIS 10 Sven Etzold, Silke Noack (Beak) 04.05.2012 Anwendungsgebiete / Methodik advangeo Prediction Software Anwendungsgebiete Vorhersage von Ereignissen Abgrenzung
Neue Biomarker Kann man die ALS im Blut erkennen?
 Neue Biomarker Kann man die ALS im Blut erkennen? Dagmar Kettemann Ambulanz für ALS und andere Motoneuronenerkrankungen UNIVERSITÄTSMEDIZIN BERLIN 1 Neue Biomarker Kann man die ALS im Blut erkennen? in
Neue Biomarker Kann man die ALS im Blut erkennen? Dagmar Kettemann Ambulanz für ALS und andere Motoneuronenerkrankungen UNIVERSITÄTSMEDIZIN BERLIN 1 Neue Biomarker Kann man die ALS im Blut erkennen? in
Begriffe und Modellbildung. Explizite Finite Elemente Methode
 Explizite Finite Elemente Methode LV01: Masterkurs für MK-M, ME-M und PE-M Begriffe und Modellbildung zur numerischen Simulation Prof. Dr.-Ing. Hans-Dieter Kleinschrodt FB VIII: Maschinenbau, Veranstaltungstechnik,
Explizite Finite Elemente Methode LV01: Masterkurs für MK-M, ME-M und PE-M Begriffe und Modellbildung zur numerischen Simulation Prof. Dr.-Ing. Hans-Dieter Kleinschrodt FB VIII: Maschinenbau, Veranstaltungstechnik,
dacore Datenbanksysteme AG Neue Konzepte zur Optimierung der Auslastungsplanung und Big Data im Informationsmanagement
 dacore Datenbanksysteme AG Neue Konzepte zur Optimierung der Auslastungsplanung und Big Data im Informationsmanagement dacore Datenbanksysteme AG Neue Konzepte zur Optimierung der Auslastungsplanung Die
dacore Datenbanksysteme AG Neue Konzepte zur Optimierung der Auslastungsplanung und Big Data im Informationsmanagement dacore Datenbanksysteme AG Neue Konzepte zur Optimierung der Auslastungsplanung Die
Einführung Molekulare Bioinformatik
 Einführung Molekulare Bioinformatik Bernhard Haubold 21. Oktober 2014 Übersicht Was ist Bioinformatik? Kursstruktur Was ist Bioinformatik? Geschichtliche Entwicklung Information: Speicherung & Übertragung
Einführung Molekulare Bioinformatik Bernhard Haubold 21. Oktober 2014 Übersicht Was ist Bioinformatik? Kursstruktur Was ist Bioinformatik? Geschichtliche Entwicklung Information: Speicherung & Übertragung
Präsentationsorientierte Komposition von Service Frontends durch den Endanwender
 Präsentationsorientierte Komposition von Service Frontends durch den Endanwender Dissertation zur Erlangung des akademischen Grades Doktoringenieur (Dr.-Ing.) vorgelegt an der Technischen Universität Dresden
Präsentationsorientierte Komposition von Service Frontends durch den Endanwender Dissertation zur Erlangung des akademischen Grades Doktoringenieur (Dr.-Ing.) vorgelegt an der Technischen Universität Dresden
Extended Petri Nets for. Systems Biology. LFE Practical Informatics and Bioinformatics. Department Institut für Informatik.
 Extended Petri Nets for Systems Biology Selina Sommer LFE Practical Informatics and Bioinformatics Department Institut für Informatik Outline Extended Petri nets for the simulation of network dynamics
Extended Petri Nets for Systems Biology Selina Sommer LFE Practical Informatics and Bioinformatics Department Institut für Informatik Outline Extended Petri nets for the simulation of network dynamics
Use Cases vs. Funktionale Spezifikation
 Use Cases vs. Funktionale Spezifikation Ein experimenteller Vergleich zweier Methoden zur Anforderungsspezifikation Fraunhofer IESE: Anne Groß (Anne.Gross@iese.fraunhofer.de) & Jörg Dörr (Joerg.Doerr@iese.fraunhofer.de)
Use Cases vs. Funktionale Spezifikation Ein experimenteller Vergleich zweier Methoden zur Anforderungsspezifikation Fraunhofer IESE: Anne Groß (Anne.Gross@iese.fraunhofer.de) & Jörg Dörr (Joerg.Doerr@iese.fraunhofer.de)
STORYBOARDING ZUR ABLEITUNG VON KONTEXTBASIERTEN INTERACTION-CASES FÜR UBIQUITÄRE SYSTEME
 Fakultät Informatik Institut für Software- und Multimediatechnik Juniorprofessur Software Engineering Ubiquitärer STORYBOARDING ZUR ABLEITUNG VON KONTEXTBASIERTEN INTERACTION-CASES FÜR UBIQUITÄRE SYSTEME
Fakultät Informatik Institut für Software- und Multimediatechnik Juniorprofessur Software Engineering Ubiquitärer STORYBOARDING ZUR ABLEITUNG VON KONTEXTBASIERTEN INTERACTION-CASES FÜR UBIQUITÄRE SYSTEME
Ensembl. Steffen Möller
 Ensembl Steffen Möller Zusammenfassung Web Funktionalität Präsentation Genome Aller sequenzierten Säugetiere, Wirbeltiere und Hefe, Fliege, Wurm Identifikation und Annotation von Genen Sequenzvariationen
Ensembl Steffen Möller Zusammenfassung Web Funktionalität Präsentation Genome Aller sequenzierten Säugetiere, Wirbeltiere und Hefe, Fliege, Wurm Identifikation und Annotation von Genen Sequenzvariationen
KISSsys Anwendung: Verwaltung zweistufiger, koaxialer Getriebe
 KISSsoft AG Frauwis 1 CH - 8634 Hombrechtikon Telefon: +41 55 264 20 30 Fax: +41 55 264 20 33 Email: info@kisssoft.ch KISSsys Anwendung: Verwaltung zweistufiger, koaxialer Getriebe Berechnungssoftware
KISSsoft AG Frauwis 1 CH - 8634 Hombrechtikon Telefon: +41 55 264 20 30 Fax: +41 55 264 20 33 Email: info@kisssoft.ch KISSsys Anwendung: Verwaltung zweistufiger, koaxialer Getriebe Berechnungssoftware
You. Das Unsichtbare sichtbar machen
 You & IaaS Das Unsichtbare sichtbar machen EINE REVOLUTION IN FORSCHUNG UND LEHRE Die Softwarefirma Interactive Scientific in Bristol hat sich nicht weniger als die Neugestaltung der naturwissenschaftlichen
You & IaaS Das Unsichtbare sichtbar machen EINE REVOLUTION IN FORSCHUNG UND LEHRE Die Softwarefirma Interactive Scientific in Bristol hat sich nicht weniger als die Neugestaltung der naturwissenschaftlichen
VL Algorithmische BioInformatik (19710) WS2013/2014 Woche 16 - Mittwoch. Annkatrin Bressin Freie Universität Berlin
 VL Algorithmische BioInformatik (19710) WS2013/2014 Woche 16 - Mittwoch Annkatrin Bressin Freie Universität Berlin Vorlesungsthemen Part 1: Background Basics (4) 1. The Nucleic Acid World 2. Protein Structure
VL Algorithmische BioInformatik (19710) WS2013/2014 Woche 16 - Mittwoch Annkatrin Bressin Freie Universität Berlin Vorlesungsthemen Part 1: Background Basics (4) 1. The Nucleic Acid World 2. Protein Structure
1. Absolventenfeier Bioinformatik. 8. Juli 2005
 1. Absolventenfeier Bioinformatik Bioinformatik München (BIM) Ein gemeinsames Studienprogramm der Ludwig-Maximilians-Universität München (LMU) Technischen Universität München (TUM) mit Unterstützung durch
1. Absolventenfeier Bioinformatik Bioinformatik München (BIM) Ein gemeinsames Studienprogramm der Ludwig-Maximilians-Universität München (LMU) Technischen Universität München (TUM) mit Unterstützung durch
Einführung in die Biophysik
 Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Modulliste. für den Masterstudiengang. Data & Knowledge Engineering (alt) an der Otto von Guericke Universität Magdeburg Fakultät für Informatik
 Modulliste für den Masterstudiengang Data & Knowledge Engineering (alt) an der Otto von Guericke Universität Magdeburg Fakultät für Informatik vom Sommersemester 2019 Der Masterstudiengang Data & Knowledge
Modulliste für den Masterstudiengang Data & Knowledge Engineering (alt) an der Otto von Guericke Universität Magdeburg Fakultät für Informatik vom Sommersemester 2019 Der Masterstudiengang Data & Knowledge
Genomics. Ernst W. Mayr Fakultät für Informatik TU München
 Genomics Ernst W. Mayr Fakultät für Informatik TU München http://wwwmayr.in.tum.de/ A. Biologische Hintergründe nde 1. Gene und Phänotypisches 1.1. Beobachtungen nach Mendel 1.2. Eukaryotische Zelle 1.3.
Genomics Ernst W. Mayr Fakultät für Informatik TU München http://wwwmayr.in.tum.de/ A. Biologische Hintergründe nde 1. Gene und Phänotypisches 1.1. Beobachtungen nach Mendel 1.2. Eukaryotische Zelle 1.3.
Neue Diagnostik für akute myeloische Leukämie
 Neue Diagnostik für akute myeloische Leukämie Neuherberg (9. März 2011) - Wissenschaftler des Helmholtz Zentrums München und der Ludwig-Maximilians-Universität München haben eine Methode entwickelt, mit
Neue Diagnostik für akute myeloische Leukämie Neuherberg (9. März 2011) - Wissenschaftler des Helmholtz Zentrums München und der Ludwig-Maximilians-Universität München haben eine Methode entwickelt, mit
Awareness-Verbesserung mit Hilfe von Requirements-Centred Social Networks
 Awareness-Verbesserung mit Hilfe von Requirements-Centred Social Networks Seminar: Informationsfluss Dokumentation und Kommunikation im Unternehmen Vortrag von Sebastian Jaschke 10. Dezember 2008 Inhalt
Awareness-Verbesserung mit Hilfe von Requirements-Centred Social Networks Seminar: Informationsfluss Dokumentation und Kommunikation im Unternehmen Vortrag von Sebastian Jaschke 10. Dezember 2008 Inhalt
Wie liest man Konfidenzintervalle? Teil II. Premiu m
 Wie liest man Konfidenzintervalle? Teil II Premiu m - Hintergrund Anderer Wahrscheinlichkeitsbegriff subjektiver Wahrscheinlichkeitsbegriff Beispiel: Was ist die Wahrscheinlichkeit dafür, dass ein Patient
Wie liest man Konfidenzintervalle? Teil II Premiu m - Hintergrund Anderer Wahrscheinlichkeitsbegriff subjektiver Wahrscheinlichkeitsbegriff Beispiel: Was ist die Wahrscheinlichkeit dafür, dass ein Patient
Principles of heredity Mutations, substitutions and polymorphisms
 Bioinformatics 1 Principles of heredity Mutations, substitutions and polymorphisms Claudia Acquisti Evolutionary Functional Genomics Institute for Evolution and Biodiversity, WWU Münster claudia.acquisti@uni-muenster.de
Bioinformatics 1 Principles of heredity Mutations, substitutions and polymorphisms Claudia Acquisti Evolutionary Functional Genomics Institute for Evolution and Biodiversity, WWU Münster claudia.acquisti@uni-muenster.de
V9 funktionelle Annotation
 V9 funktionelle Annotation Analyse von Gen-Expression Funktionelle Annotation: Gene Ontology (GO) Signifikanz der Annotation: Hypergeometrischer Test Annotationsanalysen z.b. mit NIH-Tool DAVID Ähnlichkeit
V9 funktionelle Annotation Analyse von Gen-Expression Funktionelle Annotation: Gene Ontology (GO) Signifikanz der Annotation: Hypergeometrischer Test Annotationsanalysen z.b. mit NIH-Tool DAVID Ähnlichkeit
20 von 100. an Krebs erkrankten Kindern sterben immer noch. ehemals KIND UND KREBS
 20 von 100 an Krebs erkrankten Kindern sterben immer noch. ehemals KIND UND KREBS Ärzte können es! Die Zahl der an Krebs neu erkrankten Kinder etwa 220 pro Jahr in der Schweiz ist gering, trotzdem sterben
20 von 100 an Krebs erkrankten Kindern sterben immer noch. ehemals KIND UND KREBS Ärzte können es! Die Zahl der an Krebs neu erkrankten Kinder etwa 220 pro Jahr in der Schweiz ist gering, trotzdem sterben
Bioinformatik. Margarethe Kyral
 Bioinformatik Margarethe Kyral Beispiel 1 neuer Krankheitserreger DNA-Sequenzierung Beispiel 1 neuer Krankheitserreger DNA-Sequenzierungsgeräte und -verfahren Beispiel 1 neuer Krankheitserreger Borreliose-Bakterium
Bioinformatik Margarethe Kyral Beispiel 1 neuer Krankheitserreger DNA-Sequenzierung Beispiel 1 neuer Krankheitserreger DNA-Sequenzierungsgeräte und -verfahren Beispiel 1 neuer Krankheitserreger Borreliose-Bakterium
Vorhersage von Protein-Funktionen. Patrick Pfeffer
 Vorhersage von Protein-Funktionen Patrick Pfeffer Überblick Motivation Einleitung Methode Markov Random Fields Der Gibbs Sampler Parameter-Schätzung Bayes sche Analyse Resultate Pfeffer 2 Motivation Es
Vorhersage von Protein-Funktionen Patrick Pfeffer Überblick Motivation Einleitung Methode Markov Random Fields Der Gibbs Sampler Parameter-Schätzung Bayes sche Analyse Resultate Pfeffer 2 Motivation Es
Inhalte der Spezialthemen
 SSM3- Block 24 Praktikum Medizinische Informatik Inhalte der Spezialthemen 1. Bildbearbeitungsmethoden im Rahmen der wissenschaftlichen Publikationserstellung 2. Datenbanken für die molekulare, personalisierte
SSM3- Block 24 Praktikum Medizinische Informatik Inhalte der Spezialthemen 1. Bildbearbeitungsmethoden im Rahmen der wissenschaftlichen Publikationserstellung 2. Datenbanken für die molekulare, personalisierte
Kathrin Thedieck Die Kunst, Signalgeschehen zu entschlüsseln
 Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/kathrin-thedieck-die-kunstsignalgeschehen-zu-entschluesseln/ Kathrin Thedieck Die Kunst, Signalgeschehen zu entschlüsseln
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/kathrin-thedieck-die-kunstsignalgeschehen-zu-entschluesseln/ Kathrin Thedieck Die Kunst, Signalgeschehen zu entschlüsseln
Anforderungen für die Softwareweiterentwicklung
 Anforderungen für die Softwareweiterentwicklung durch Benutzer Feedback ermitteln Melanie Stade, Norbert Seyff, Farnaz Fotrousi, Emitza Guzman, Samuel A. Fricker, Martin Glinz, Ronnie Schaniel melanie.stade@fhnw.ch
Anforderungen für die Softwareweiterentwicklung durch Benutzer Feedback ermitteln Melanie Stade, Norbert Seyff, Farnaz Fotrousi, Emitza Guzman, Samuel A. Fricker, Martin Glinz, Ronnie Schaniel melanie.stade@fhnw.ch
Bachelorstudium Medizinische Informatik. Rudolf Freund
 Bachelorstudium Medizinische Informatik Rudolf Freund Was ist Medizinische Informatik eigentlich? Die Medizinische Informatik beschäftigt sich mit dem Einsatz von Konzepten und Techniken der Informatik
Bachelorstudium Medizinische Informatik Rudolf Freund Was ist Medizinische Informatik eigentlich? Die Medizinische Informatik beschäftigt sich mit dem Einsatz von Konzepten und Techniken der Informatik
Threading - Algorithmen
 Threading - Algorithmen Florian Lindemann 22.11.2007 Florian Lindemann () Threading - Algorithmen 22.11.2007 1 / 25 Gliederung 1 Prospect Scoring Function Algorithmus Weitere Eigenschaften Komplexität
Threading - Algorithmen Florian Lindemann 22.11.2007 Florian Lindemann () Threading - Algorithmen 22.11.2007 1 / 25 Gliederung 1 Prospect Scoring Function Algorithmus Weitere Eigenschaften Komplexität
HANA CLOUD CONNECTOR
 Systemnahe Anwendungsprogrammierung und Beratung GmbH A-1030 Wien, Kölblgasse 8-10/2.OG (Hoftrakt) www.snapconsult.com Tel. +43 1 617 5784 0 Fax +43 1 617 57 84 99 HANA CLOUD CONNECTOR Seite 2 von 9 INHALTSVERZEICHNIS
Systemnahe Anwendungsprogrammierung und Beratung GmbH A-1030 Wien, Kölblgasse 8-10/2.OG (Hoftrakt) www.snapconsult.com Tel. +43 1 617 5784 0 Fax +43 1 617 57 84 99 HANA CLOUD CONNECTOR Seite 2 von 9 INHALTSVERZEICHNIS
Notationen zur Prozessmodellierung
 Notationen zur Prozessmodellierung August 2014 Inhalt (erweiterte) ereignisgesteuerte Prozesskette (eepk) 3 Wertschöpfungskettendiagramm (WKD) 5 Business Process Model and Notation (BPMN) 7 Unified Modeling
Notationen zur Prozessmodellierung August 2014 Inhalt (erweiterte) ereignisgesteuerte Prozesskette (eepk) 3 Wertschöpfungskettendiagramm (WKD) 5 Business Process Model and Notation (BPMN) 7 Unified Modeling
Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen
 Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. des Fachbereichs Biologie, Chemie, Pharmazie der Freien Universität
Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. des Fachbereichs Biologie, Chemie, Pharmazie der Freien Universität
Neuere Ansätze zur Auswahl von Prädiktionsmodellen. Von Veronika Huber
 Neuere Ansätze zur Auswahl von Prädiktionsmodellen Von Veronika Huber Gliederung Anwendungsbereiche von Prädiktionsmodellen Traditionelle Methoden zur Prüfung der Wirksamkeit Neuere Ansätze zur Prüfung
Neuere Ansätze zur Auswahl von Prädiktionsmodellen Von Veronika Huber Gliederung Anwendungsbereiche von Prädiktionsmodellen Traditionelle Methoden zur Prüfung der Wirksamkeit Neuere Ansätze zur Prüfung
Support Technologies based on Bi-Modal Network Analysis. H. Ulrich Hoppe. Virtuelles Arbeiten und Lernen in projektartigen Netzwerken
 Support Technologies based on Bi-Modal Network Analysis H. Agenda 1. Network analysis short introduction 2. Supporting the development of virtual organizations 3. Supporting the development of compentences
Support Technologies based on Bi-Modal Network Analysis H. Agenda 1. Network analysis short introduction 2. Supporting the development of virtual organizations 3. Supporting the development of compentences
Zentrum für Bioinformatik Universität des Saarlandes sowie der Informatik der Johannes-Gutenberg Universität Mainz
 Zentrum für Bioinformatik Universität des Saarlandes sowie der Informatik der Johannes-Gutenberg Universität Mainz Die Rolle der Bioinformatik im Wirkstoffentwurf A. Hildebrandt 1. Einleitung Die erfolgreiche
Zentrum für Bioinformatik Universität des Saarlandes sowie der Informatik der Johannes-Gutenberg Universität Mainz Die Rolle der Bioinformatik im Wirkstoffentwurf A. Hildebrandt 1. Einleitung Die erfolgreiche
Bachelorstudium Medizinische Informatik. Rudolf Freund
 Bachelorstudium Medizinische Informatik Rudolf Freund Was ist Medizinische Informatik eigentlich? Die Medizinische Informatik beschäftigt sich mit dem Einsatz von Konzepten und Techniken der Informatik
Bachelorstudium Medizinische Informatik Rudolf Freund Was ist Medizinische Informatik eigentlich? Die Medizinische Informatik beschäftigt sich mit dem Einsatz von Konzepten und Techniken der Informatik
systems landscape engineering - übung -
 systems landscape engineering - übung - Wintersemester 2010 /2011 Arbeitsgruppe Wirtschaftsinformatik - Managementinformationssysteme - Dipl. Wirt.-Inform. Sven Gerber Arbeitsgruppe Wirtschaftsinformatik
systems landscape engineering - übung - Wintersemester 2010 /2011 Arbeitsgruppe Wirtschaftsinformatik - Managementinformationssysteme - Dipl. Wirt.-Inform. Sven Gerber Arbeitsgruppe Wirtschaftsinformatik
Themen für Bachelorprojekte und Bachelorarbeiten Lehrstuhl Prof. Dr. Pohl, Software Systems Engineering
 Themen für Bachelorprojekte und Bachelorarbeiten Lehrstuhl Prof. Dr. Pohl, Software Systems Engineering 11.07.2018 Vergleich von Metriken zur Bewertung der Genauigkeit binärer Prozessprognosen anhand eines
Themen für Bachelorprojekte und Bachelorarbeiten Lehrstuhl Prof. Dr. Pohl, Software Systems Engineering 11.07.2018 Vergleich von Metriken zur Bewertung der Genauigkeit binärer Prozessprognosen anhand eines
Data Science als Treiber für Innovation Neue Anforderungen und Chancen für Ausbildung und Wirtschaft
 Data Science als Treiber für Innovation Neue Anforderungen und Chancen für Ausbildung und Wirtschaft b Univ-Prof. Dr. Stefanie Lindstaedt b www.know-center.at Know-Center GmbH Know-Center: Austria s Research
Data Science als Treiber für Innovation Neue Anforderungen und Chancen für Ausbildung und Wirtschaft b Univ-Prof. Dr. Stefanie Lindstaedt b www.know-center.at Know-Center GmbH Know-Center: Austria s Research
EAM-Vorlesung (SS2017)
 EAM-Vorlesung (SS2017) V4: TOGAF und das Metamodel Vassilios Goumas Frankfurt University of Applied Science 1 V4: Verlauf Was ist ein Metamodell Aufbau des Metamodells in TOGAF Aufteilung nach Core Metamodel
EAM-Vorlesung (SS2017) V4: TOGAF und das Metamodel Vassilios Goumas Frankfurt University of Applied Science 1 V4: Verlauf Was ist ein Metamodell Aufbau des Metamodells in TOGAF Aufteilung nach Core Metamodel
22. Januar Gruppe 2: TOPCASED
 22. Januar 2008 Aufgabenstellung Modellgetriebene Softwareentwicklung auf Basis von am Beispiel eines Seminarverwaltungssystems Ziel Entwicklungsprozess Anforderungen & Codegenerierung Modellierung & Templates
22. Januar 2008 Aufgabenstellung Modellgetriebene Softwareentwicklung auf Basis von am Beispiel eines Seminarverwaltungssystems Ziel Entwicklungsprozess Anforderungen & Codegenerierung Modellierung & Templates
Corinna Schmitt Dipl. Inf. (Bioinformatik)
 Corinna Schmitt Dipl. Inf. (Bioinformatik) Meluner Str. 42 70569 Stuttgart, Germany Tel.: (+49) 711 94593207 Email: corinna.schmitt@web.de Geboren am 7. Februar 1979 in München Curriculum Vitae Persönliche
Corinna Schmitt Dipl. Inf. (Bioinformatik) Meluner Str. 42 70569 Stuttgart, Germany Tel.: (+49) 711 94593207 Email: corinna.schmitt@web.de Geboren am 7. Februar 1979 in München Curriculum Vitae Persönliche
Optimale Enzymaktivitäten und metabolische Kontrolltheorie
 Seminar Wie Zellen denken : Heute: Optimale Enzymaktivitäten und metabolische Kontrolltheorie Jannis Uhlendorf, Tim Landgraf, WS05/06 Index 1. Crash-Kurs : Metabolische Kontrolltheorie 2. Motivation Optimalität
Seminar Wie Zellen denken : Heute: Optimale Enzymaktivitäten und metabolische Kontrolltheorie Jannis Uhlendorf, Tim Landgraf, WS05/06 Index 1. Crash-Kurs : Metabolische Kontrolltheorie 2. Motivation Optimalität
Janine Czichy, Hamburg SCHUTZ VON VERSUCHSTIEREN. 38 BfR 2 GO
 38 BfR 2 GO GENOMDATENVERGLEICHE VON MENSCH UND TIER Spiegelbild gesucht Neuer Ansatz für Genomdatenvergleiche von Mensch und Tier Das Deutsche Zentrum zum Schutz von Versuchstieren (Bf3R) am BfR zeigt
38 BfR 2 GO GENOMDATENVERGLEICHE VON MENSCH UND TIER Spiegelbild gesucht Neuer Ansatz für Genomdatenvergleiche von Mensch und Tier Das Deutsche Zentrum zum Schutz von Versuchstieren (Bf3R) am BfR zeigt
Realität Virtualität Wirklichkeit
 Realität Virtualität Wirklichkeit 4. Vorlesung (15.5.12): Virtuelle Realitäten, Simulation Christoph Hubig 1 Veränderung der Weltbezüge nichtmenschliche Lebewesen: Individuum Wirkwelt Merkwelt Umwelt (von
Realität Virtualität Wirklichkeit 4. Vorlesung (15.5.12): Virtuelle Realitäten, Simulation Christoph Hubig 1 Veränderung der Weltbezüge nichtmenschliche Lebewesen: Individuum Wirkwelt Merkwelt Umwelt (von
