Virale Ionenkanäle: Bildung, Modelling, Drug Targeting
|
|
|
- Karsten Kramer
- vor 7 Jahren
- Abrufe
Transkript
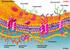
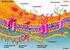
1 1216 Theoretische Chemie Virale Ionenkanäle: Bildung, Modelling, Drug Targeting Viren manipulieren den Stoffwechsel einer infizierten Zelle zu ihren Gunsten. Dafür nutzen sie kleine Proteine, die Ionenkanäle bilden. Doch wie lagern sich diese Proteine zu funktionellen, membrandurchspannenden Einheiten zusammen? Leitfähigkeitsmessungen und Computersimulation sind zwei der vielen Methoden, die dazu beitragen, die Vorgänge zu verstehen und letztlich neue antivirale Wirkstoffe zu entwerfen. Abb. 1. Lebenszyklus eines Virus am Beispiel von HIV-1, schematische Darstellung. Das ionenkanalbildende Protein Vpu ist im endoplasmatischen Retikulum (ER) rot eingezeichnet, farbige Rechtecke stellen virale Makromoleküle dar; siehe auch Infokasten auf S.1217). Erkennung Anbindung Eindringen Infizierte Zelle Lipidmembranen bilden für Ionen und polare Substanzen eine nahezu unüberwindliche Barriere. Für den Metabolismus der Zelle ist es jedoch essentiell, die Barriere für Substrate in geordneten Bahnen permeabel zu machen. Während der Evolution wurden daher hochspezifische Membranproteine, die Ionenkanäle, entwickelt, die zum Beispiel elektrochemische Gradienten über eine Membran hinweg modulieren. Ionenkanäle von Zellen sind Heterooligomere aus vier oder fünf assemblierten Proteinen, welche typischerweise aus jeweils ungefähr 500 Aminosäuren aufgebaut sind. Viren Einbringen des Genoms Modellieren der Zelle Reproduktion ER Virus Budding Assemblierung Reifung nutzen für ihren Lebenszyklus ebenfalls solche Proteine (Abbildung 1). 1) Allerdings bestehen die Proteine, welche die viralen Ionenkanäle konstituieren, lediglich aus ungefähr 50 bis 100 Aminosäuren, sind also miniaturisierte Ausgaben der Kanäle höherer Organismen und bilden Homooligomere (Tabelle). Untersuchungen an viralen Ionenkanälen werden in der Medizin immer wichtiger man denke nur an hochinfektiöse, sich den Menschen anpassende Viren aus tierischen Organismen, für die keine Impfstoffe bereit stehen und zudem liegt die Frage nahe, inwieweit virale Ionenkanäle ihrer geringen Größe wegen in der Bionanotechnik für Sensorarrays, zur Medikamentenentwicklung, als Filter oder zur Konstruktion künstlicher Kanäle genutzt werden können. Auf die Anwendungen wird hier jedoch nicht eingegangen. Virale Ionenkanäle sind für einige Viren, z. B. Poliovirus, überlebenswichtig, für andere Viren wie HIV-1 dienen sie lediglich dazu, die Vermehrung zu steigern. Die bisher bekannten kanalbildenden Proteine bestehen aus ein oder zwei Bereichen, die aus überwiegend hydrophoben Aminosäuren aufgebaut sind und sich daher in Lipidmembranen einlagern und diese durchspannen können. Das Strukturmotiv für diesen Transmembranbereich (TM-Bereich) ist für alle heute bekannten viralen Proteine eine Helix. Influenza, HIV-1, Hepatitis-C-Virus (HCV), Poliovirus oder SARS-Coronavirus sind nur einige der Viren, deren Genom für ionenkanalbildende Proteine kodiert. Die Datenlage Einer der am besten untersuchten Kanäle ist M2 von Influenza A. Um in die Wirtszelle eindringen zu können, bindet das Virus über Erkennungs-
2 Virale Ionenkanäle Magazin 1217 proteine an die Zellmembran der Wirtszelle, die sich daraufhin einstülpt und einen Vesikelkörper (Endosom) um das Virus bildet. Das Innere des Endosom wird dann von der Zelle angesäuert. Dies geschieht mit membranständigen V-Typ-ATPasen (Protonenpumpen). Die Protonierung von Histidinresten im TM-Bereich von M2 führt zur Öffnung der durch M2 gebildeten Kanäle und dem Einfließen von Protonen in das Innere des Viruskörpers. Als Folge tritt eine Reihe struktureller Änderungen an Proteinen innerhalb des Virus auf. Eines dieser Proteine, das Hämagglutinin, leitet daraufhin die Fusion der viralen Membran mit der des Endosoms ein und führt damit zur Infektion der Zelle. Über die Struktur des ionenkanalbildenden Proteins Vpu von HIV-1 ist dank umfangreicher NMR-spektroskopischer Studien und dessen einfacher Zusammensetzung sehr viel bekannt. 2) Vpu besteht aus einem helicalen TM-Bereich und einem längeren cytoplasmatischen Abschnitt. In der infizierten Zelle tritt der cytoplasmatische Abschnitt mit CD4, dem Rezeptorprotein der T-Helferzellen, in Wechselwirkung und induziert dadurch dessen Abbau durch das zelleigene Verdauungssystem. Der TM-Bereich ist indirekt am Austritt neuer HI-Viren aus der infizierten Zelle (Budding) beteiligt. Aller Wahrscheinlichkeit nach assemblieren fünf Vpu-Proteine 1) zu schwach kationenselektiven Kanälen, modulieren dadurch den elektrochemischen Gradienten in der Nähe der Plasmamembran und begünstigen das Budding. Für andere, erst kürzlich entdeckte kanalbildende Proteine stehen spärlichere Strukturinformationen zur Verfügung. Im Gegensatz zu M2 und Vpu werden für p7 von HCV und 2B des Poliovirus zwei TM-Bereiche vermutet. Das Protein Kcv des Pflanzenvirus PBCV-1 soll, das legen Sequenzhomologien nahe, K + -Kanälen ähneln. Andere Proteine werden als ionenkanalbildend eingestuft, da sie, in künstliche Lipidmembranen integriert (Rekonstitution), Kanalaktivität zeigen. Protein Virus Familie Länge Selektivität 3A Poliovirus Picornaviridae 87 2B Polio/Coxsackie B3 Picornaviridae 99 6K Alphavirus Togaviridae Kation E SARS-Coronavirus Coronaviridae 76 Kation Kcv PBCV-1 Phycodnaviridae 94 Kation M2 Influenza A Orthomyxoviridae 97 Proton NB Influenza B Orthomyxoviridae 100 Kation/Proton p7 HCV Flaviviridae 63 Kation Vpr HIV-1 Retroviridae 96 Kation Vpu HIV-1 Retroviridae 81 Kation Die viralen kanalbildenden Proteine werden im rauen endoplasmatischen Retikulum produziert und in die intrazelluläre Membran integriert. Die Zahl der Proteine, die jeweils einen Kanal bilden, ist mit Ausnahme von M2 allerdings für sämtliche Kanäle unbekannt. Wenn die Kanäle gebildet sind, sollten sie schaltbar sein (Gating), d. h. geöffnet und geschlossen werden können und letztlich auch durch Liganden blockierbar oder modulierbar sein wichtig für die Entwicklung von Medikamenten gegen virale Infektionen. In Bezug auf das Gating ist ebenfalls M2 am besten verstanden. Der Lebenszyklus von Viren Der virale Lebenszyklus umfasst in etwa folgende Phasen: Der Virus erkennt und bindet über Erkennungsproteine an die spezifischen Rezeptormoleküle der Wirtszelle und dringt dann in die Zelle ein. Das virale Genom wird in die Zelle eingebracht und verändert das Zellmilieu so, dass die Zelle virale Makromoleküle reproduziert. Die Virushülle sowie das Erbgut des Virus assemblieren an intrazellulären Membranen oder an der Plasmamembran und treten dann schließlich aus der Zelle aus. Eventuell reift das Virus außerhalb der Zelle weiter, d. h. die Makromoleküle innerhalb des Virus ändern ihre Struktur. Experimentelle Strukturund Funktionsanalyse Neben physiologischen Untersuchungen sind Leitfähigkeitsmessungen mit künstlichen Membranen eine wichtige Methode zur Bestimmung der Kanalaktivität (Abbildung 2). Dabei wird das Protein oder auch nur sein TM-Bereich in einer Lipidmembran rekonstituiert. Die Proteine werden über Expression erhalten, während der TM-Bereich üblicherweise über Festphasen-Peptidsynthese hergestellt wird. Die künstlichen Membranen werden über Aperturen von ungefähr 150 µm ge- In all diesen Phasen spielen elektrochemische und Protonengradienten über Membranen hinweg bei der molekularen Erkennung und Faltung der viralen Makromoleküle eine entscheidende Rolle Virale Ionenkanäle dienen dabei dazu, die elektrochemischen Gradienten zugunsten des viralen Lebenszyklus zu verändern. Abbildung 1 zeigt vereinfacht den Lebenszyklus des Human Immunodeficiency Virus Type-1 (HIV-1). Die viralen ionenkanalbildenden Proteine (rot) werden im endoplasmatischen Retikulum (ER), der Proteinproduktionseinheit der Zelle, hergestellt. In die Membran des ER eingesetzt, diffundieren sie als monomere Einheiten, um dann weiter zu homooligomeren Bündeln zu assemblieren. Tabelle. Bekannte virale ionenkanalbildende Membranproteine. Die Kanalaktivität ist durch Rekonstitution der Proteine in künstlichen Lipidmembranen nachgewiesen. Die Länge ist durch die Zahl der Aminosäuren ausgedrückt.
3 1218 Magazin Virale Ionenkanäle A B Für M2 und Vpu ergibt sich aus den Programmen zur Vorhersage der Sekundärstruktur von TM-Bereichen in Membranproteinen ein einziger solcher Bereich. Die Existenz einzelner Aminosäurereste innerhalb dieses Bereiches gibt Hinweise darauf, wie die Proteine im homooligomeren Bündel angeordnet sind. So sind beispielsweise zwei M2-Proteine über Disulfidbrücken (Cysteinreste) kovalent miteinander verbunden, und zwei dieser dimeren Einheiten bilden dann einen tetrameren Kanal. In dieser Anordnung weisen Histidinreste in die Pore; sie sind die Schlüsselaminosäuren zur Öffnung des Kanals und zur Protonenleitung. Vpu hat im TM-Bereich eine hydrophile Aminosäure, Serin (Abbildung 3A). Eine Orientierung des Serinrests ins Bündelinnere erscheint naheliegend und deckt sich mit dem Wissen um dessen Ausrichtung in anderen Kanälen, beispielsweise den nikotinischen Acetylcholinrezeptor. In diesem Fall befindet sich der Tryptophanrest eines Vpu-Proteins am äußeren Rand des Bündels. Dabei handelt es sich um eine Position, die auch bei anderen Kanälen zu fin- Spannungsversorgung 1pA 5 s offen I/V Konverter geschlossen Lipidmembran Ionenkanal Messkammer Pufferlösung Gemessener Strom +90 mv +20 mv 0 mv -20 mv -90 mv Abb. 2. A) Apparatur für Leitfähigkeitsmessungen an Lipidmembranen, schematische Darstellung. Die Lipidmembran (grün) überspannt eine Apertur von ungefähr 150 μm. In dieser Membran wird der Kanal (rot) rekonstituiert. Über Elektroden (schwarz) wird die Spannung angelegt. B) Leitfähigkeitsmessungen mit einem Peptid, das den ersten 32 Aminosäuren von Vpu (HIV-1) entspricht und dessen TM-Bereich umfasst. Aufgetragen ist der gemessene Strom als Funktion der über eine Membran abfallenden Spannung. Die Kanalöffnungen werden durch einen sprunghaften Anstieg des Stromes angezeigt. Diese erhöhten Leitfähigkeiten können einige Millisekunden bis hin zu Sekunden andauern. spannt. Die durch Rekonstitution und nachfolgender Selbstassemblierung gebildeten Kanäle weisen Leitfähigkeiten zwischen 10 und 40 ps auf 3 6) (Abbildung 2B) und liegen damit in der gleichen Größenordnung wie die der großen Kanäle in der Wirtszelle. Auf Grund der geringen Größe der viralen Ionenkanäle gestalten sich Röntgenstrukturanalysen äußerst schwierig. Daher sind Methoden wie NMR-, CD- oder FTIR-Spektroskopie die wichtigsten Quellen für Strukturinformationen (Übersicht in Lit. 1) ). Oftmals mangelt es aber an experimentellen Daten. In diesem Fall bieten insbesondere computergestützte Simulationen eine weitere wesentliche Möglichkeit, um zu Aussagen über Struktur und Funktionsweise viraler Ionenkanäle zu gelangen und ermöglichen ein Screening nach potenziellen Liganden. Modellierung der Kanäle
4 Virale Ionenkanäle Magazin 1219 den ist. Tryptophan verankert Proteine innerhalb der Lipidmembran. Die Analyse der Primärsequenz von p7 ergibt zwei hydrophobe TM- Bereiche, die durch eine kurze Aminosäuresequenz miteinander verbunden sind. Diese müssen sich zunächst in der richtigen Weise zusammenlagern, bevor ein homooligomeres Bündel und damit ein Kanal gebildet werden kann. Diese vergleichsweise komplizierte Kanalkonstruktion erfordert für Computersimulationen die Anwendung differenzierter Analysestrategien. Die möglichen Orientierungen der beiden transmembranen Helices können unter Berücksichtigung intermolekularer Wechselwirkungen bestimmt werden. Für diese Orientierungsexperimente eignet sich das Programm CNS (Crystallography & NMR System) mit entsprechenden Anwendungsroutinen, beispielsweise CHI (CNS Helix Interaction), Damit erhält man ein Monomer, das daraufhin repliziert und in einem Bündel angeordnet werden kann. Aus elektronenmikroskopischen Aufnahmen von p7 geht ein Porendurchmesser von etwa 2,3 nm und ein Außendurchmesser des Bündels von 3 bis 5 nm hervor. Diese Daten können dann mit denen verglichen werden, die sich an Hand der computergestützten Modelle errechnen lassen. Die beste Übereinstimmung ergibt sich, wenn ein hexameres Bündel erstellt wird. Computergestützte Analyse Ist ein Kanalmodell erstellt, müssen strukturelle (z. B. die Neigung der Helices des Proteins) oder funktionelle Daten (z. B. Leitfähigkeit) errechnet werden (Übersicht in Lit. 7) ), die durch Experimente verifiziert werden können. Die Fragen, welche es dann weiterhin zu beantworten gilt, lassen sich wie folgt zusammenfassen: Wie weit dringen Blocker in die Pore, und, wie öffnet und schließt der Kanal? Für die Vorhersage der Leitfähigkeit gibt es eine Reihe von Berechnungsmöglichkeiten. Sie berücksichtigen Strukturdetails in unterschiedlichem Maß und können für alle Ionenkanäle genutzt werden. Ein Kanal kann als einfache Röhre betrachtet und der Ionenfluss auf Basis der kontinuierlichen Elektrodiffusionstheorie (Nernst-Planck-Beziehung) berechnet werden. Der Einfluss des durch die Ionen im Kanal erzeugten Berghof 1/3 hoch S.1219 Strukturvorhersagen und Berechnung der Leitfähigkeit Programme zur Vorhersage der Sekundärstruktur ermöglichen, den membrandurchspannenden Bereich eines Proteins zu ermitteln. Die Vorhersage basiert darauf, dass in den membrandurchspannenden Bereichen vermehrt hydrophobe Aminosäuren wie Isoleucin oder Valin auftreten. Dieses Phänomen zusammen mit anderen Hinweisen gestattet dann, die Länge des Bereichs zu prognostizieren. Ebenso ist aus Experimenten bekannt, dass eine a-helix das überwiegende Strukturmotiv für den membrandurchspannenden Teil ist. Eine b-faltblattmotiv nehmen nur wenige Proteine an. Mit Brownschen-Dynamik-Simulationen wird die Ionenbewegung in einem engen Kanal auf Basis der Langevin-Gleichung in diskreten Zeitschritten unter einem angelegten Potential berechnet. Daraus erhält man die Zahl der Ionen, die den Kanal pro Zeiteinheit passieren (Strom). Erweiterte Rechnungen berücksichtigen das Profil der freien Energie eines Ions im Kanal, um die Vorhersagegenauigkeit zu erhöhen. Das Profil erhält man durch Nichtgleichgewichts-MD-Simulationen, wie Steered MD-Simulationen oder Umbrella Sampling. Im ersten Fall wird das Ion durch die Pore gezogen, im zweiten Fall wird durch ein Potential das Ion an diskreten Positionen entlang des Kanals festgehalten.
5 1220 Magazin Virale Ionenkanäle A B C Wasser C-Terminus N-Terminus C-Terminus Na + Proteinbündel Lipidmembran Abb. 3. Computermodelle der ersten 32 Aminosäuren von Vpu (Vpu 1 32 ). A) Drei Helices, die zu einem Ionenkanal assembliert sind. Um den Blick auf das Poren - innere freizumachen, wurden zwei von insgesamt fünf Helices entfernt. Am oberen Ende (C-Terminus) weisen Serinreste (rot) in das Innere der Pore. Die Tryptophan reste (grün) zeigen nach außen. Zum N-Terminus hin kleiden hydropohobe Aminosäuren (Isoleucin, Leucin, Valin; nicht gezeigt) das Poreninnere aus. B) Seitenansicht des hydratisierten pentameren Bündels von Vpu 1 32 (blaue Helices; Wassermoleküle als rote Winkel), eingebaut in eine Lipidmembran (hellblau). Ein Na + -Ion (grüne Kugel) kann in Steered-MD-Simulationen durch die Pore gezogen werden. Die Aminosäure Arginin in jeder Helix ist in grau gezeigt. C) Blick vom C-Terminus in die Pore eines Vpu Kanals. Die Lipid- und Wassermoleküle sind der Übersichtlichkeit halber weggelassen. Die Strukturen ergaben sich nach einer MD Simulationszeit von 12 ns. Die blockenden Moleküle (gelb) wurden vor Beginn der Simulation in die Bündel eingesetzt; die Koordinaten stammen aus Dockingsimulationen Im linken Bündel ist die Positionierung eines Moleküls des nicht blockenden Wirkstoffs Amilorid (3,5-Diamino- 6-chlor-N-(diaminomethylen) pyrazincarboxamid) gezeigt, im rechten Bündel diejenige des blockierenden Derivats 5-(N,N-Hexamethylen)amilorid). Potenzials auf die Flussdichte wird hierbei mit der Poisson-Gleichung beschrieben und in die Rechnungen implementiert (Poisson-Nernst- Planck-Berechnungen). Die daraus resultierenden gekoppelten partiellen Differentialgleichungen werden dann selbstkonsistent gelöst. Mittlerweile werden jedoch zunehmend auch Strukturinformationen wie die Verteilung von Ladungen entlang der Kanalwand in solche Rechnungen einbezogen. Andere Methoden zur Leitfähigkeitsbestimmung, zum Beispiel Brownsche-Dynamik-Simulationen, berechnen die Bewegung einzelner Ionen innerhalb einer Pore, basierend auf dem in der Pore zur Verfügung stehenden Platz. Die Struktur der Pore wird dabei als starr angenommen und die Eigenschaften der wässrigen Lösung über die Dielektrizitätskonstante berücksichtigt. Realistischere Bedingungen können erzielt werden, wenn eine große Zahl von Ionen in die Rechnungen einbezogen wird. Dazu werden die Brownschen-Dynamik-Simulationen mit Monte-Carlo-Algorithmen gekoppelt, die den Zu- und Abfluss von Ionen zum bzw. vom Kanal simulieren. Letztendlich ist es natürlich wünschenswert, alle Atome und Moleküle, die am Ionenfluss (oder Molekülfluss) beteiligt sind, explizit in die Rechnungen einzubeziehen. Hier haben sich molekulardynamische (MD) Simulationen etabliert. 8) Dabei werden die Koordinatenände- Wolfgang Fischer, Jahrgang 1962, studierte Chemie in Heidelberg und promovierte dort Nach einem Postdoc-Aufenthalt 1992 bis 1993 an der Boston University, USA, ging er an die TU Dresden und habilitierte sich dort Seit 1998 lehrt er an der Universität Oxford biologische Physik und Bionanotechnologie. Am Department für Physik forscht er über die Modellierung viraler Membranproteine, über die Ionendiffussion und arbeitet an der Verbindung von Computersimulation und Experiment.
6 Virale Ionenkanäle Magazin 1221 rungen aller Moleküle oder Atome über einen gewissen Zeitraum hinweg mit den Newtonschen Bewegungsgleichungen verfolgt. Aus den Berechnungen lassen sich dann Gleichgewichtsstrukturen der Proteine und mehrere physikalische Größen ermitteln. Ionenfluss heißt allerdings immer, dass sich das System im Nichtgleichgewicht befindet. Dieser Zustand kann durch die Ausübung einer Kraft auf bestimmte Atome in diesem Fall das Ion in der Pore während einer Simulation dargestellt werden. Zum Beispiel ist es möglich, ein einzelnes Ion durch die Pore zu ziehen (Steered MD- Simulationen), um Informationen über die Wechselwirkungen des Ions mit den Kanalwänden oder den Seitenketten der Aminosäuren zu erhalten (Abbildung 3B). Aus den Simulationen kann die freie Energie des Ions entlang der Pore berechnet und, gekoppelt mit Brownschen-Dynamik-Simulationen, ebenfalls die Leitfähigkeit bestimmt werden. Steered MD-Simulationen erlauben auch Vorhersagen hinsichtlich des Gating. Solche Untersuchungen liefern die Basis für weiterführende Analysen von Wechselwirkungen zwischen Liganden oder Blockern und den kanalbildenden Proteinen 9) (Abbildung 3C). Passagen für die Diffusion von Liganden in Bindungstaschen konnten bereits mit MD-Simulationen berechnet werden. 10) Ionenkanalbildner als Drug Targets Die zunehmende Resistenz von Viren gegen Medikamente, welche die größeren viralen Proteine blockieren, verlangt eine genaue Untersuchung der ionenkanalbildenden Proteine als potentielle Drug Targets. Einige dieser Proteine werden bereits durch Medikamente attackiert: Amantadin, eines der ersten antiviralen Medikamente, blockiert genau den M2-Kanal. Neuere Untersuchungen weisen darauf hin, dass Derivate des Na + -Kanal-Blockers Amilorid auch Vpu inhibieren. Gegen p7 scheinen Aminozucker geeignete Wirkstoffkandidaten zu sein, wie erste Versuche bereits gezeigt haben. Ausblick Zukünftige Arbeiten werden sich weiter mit der Entwicklung potenzieller Blocker beschäftigen. Im Zusammenhang damit ist klar, dass lediglich der TM-Bereich für Wechselwirkungen mit Blockern relevant ist. Da die kurzen TM-Bereiche als Peptide über Festphasensynthese zugänglich sind, könne damit Sensor- arrays für das Drug Screening entwickelt werden. Wolfgang B. Fischer Bionanotechnology Interdisciplinary Research Collaboration Clarendon Laboratory, Department of Physics, Oxford University, UK, and Biomembrane Structure Unit, Department of Biochemistry, Oxford University, wolfgang.fischer@bioch.ox.ac.uk Glossar Blocker: Moleküle, die Durchgänge oder Aktivitäten von Proteinen behindern oder blockieren. Budding: Freisetzung oder Knospung der Viren von der Plasmamembran. Die Assemblierung von Membranproteinen löst Budding in einem begrenzten Bereich der Plasmamembran aus. Endosom: Membranumschlossenes Vesikel oder Bläschen, das sich aus der Zellmembran herausbildet. Dadurch können Makromoleküle in das Zellinnere geschleust werden. Das Endosom wird innerhalb der Zelle durch in der Membran eingebundene Protonenpumpen angesäuert. Die in dem Endosom enthaltenen Makromoleküle werden dadurch hydrolytisch gespalten. Ligand: Meist ein kleines Molekül (z. B. Hormon oder Neurotransmitter), das an große Proteine (Rezeptoren) bindet. Die Bindung ist nicht-kovalent und befindet sich in spezifischen Bereichen des Proteins. 1) Viral ion channels: structure and function : W. B. Fischer, M. S. P. Sansom, Biochim. Biophys. Acta 2002, 1561, ) Vpu from HIV-1 on an atomic scale: experiments and computer simulations : W. B. Fischer, FEBS Lett. 2003, 552, ) Identification of an ion channel activity of the Vpu transmembrane domain and its involment in the regulation of virus release from HIV-1-infected cells : U. Schubert et al., FEBS Lett. 1996, 398, ) Transmembrane peptide NB of influenza B: a simulation, structure, and conductance study : W. B. Fischer et al., Biochemistry 2000, 39, ) Bundles consisting of extended transmembrane segments of Vpu from HIV-1: computer simulations and conductance measurements : F. S. Cordes, A. Tustian, M. S. P. Sansom, A. Watts, W. B. Fischer, Biochemistry 2002, 41, ) The hepatitis C virus p7 protein forms an ion channel that is inhibited by longalkyl-chain iminosugar derivatives : D. Pavlovic et al., Proc. Natl. Acad. Sci. USA 2003, 100, ) Theoretical and computational models of biological ion channels : B. Roux, T. Allen, S. Berneche, W. Im, Quart. Rev. Biophys. 2004, 37, ) Water permeation across biological membranes: mechanism and dynamics of aquaporin-1 and GlpF : B. L. de Groot, H. Grubmüller, Science 2001, 294, ) Interaction of amiloride and one of its deriveative with Vpu from HIV-1: a molecular dynamics simulation : V. Lemaitre, R. Ali, C. G. Kim, A. Watts, W. B. Fischer, FEBS Lett. 2004, 563, ) How do substrates enter and products exit the buried active site of cytochrome P450? 1. Random expulsion molecular dynamics investigation of ligand access channels and mechanisms : S. K. Lüdemann, V. Lounnas, R. C. Wade, J. Mol. Biol. 2000, 303,
Stofftransport durch die Biomembran Variante 1
 Stofftransport durch die Biomembran Variante 1 Aquaporine - die Wasserfilter der Zelle und ihre Entdeckung Im folgenden Text beschreiben Bert L. de Groot und Helmut Grubmüller die Funktionsweise der Aquaporine
Stofftransport durch die Biomembran Variante 1 Aquaporine - die Wasserfilter der Zelle und ihre Entdeckung Im folgenden Text beschreiben Bert L. de Groot und Helmut Grubmüller die Funktionsweise der Aquaporine
5 Zusammenfassung und Ausblick
 5 Zusammenfassung und Ausblick Die Kopplung von sekundären- und primären Aminen an die freie α-carboxylgruppe von L-Glutaminsäure bildet den Zugang zu Monomeren, die zum Aufbau von γ- Peptiden genutzt
5 Zusammenfassung und Ausblick Die Kopplung von sekundären- und primären Aminen an die freie α-carboxylgruppe von L-Glutaminsäure bildet den Zugang zu Monomeren, die zum Aufbau von γ- Peptiden genutzt
Einführung in die Biophysik
 Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Einführung in die Biophysik
 Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Einführung in die Biophysik Quellen Schünemann: Biophysik Cotterill: Biophysik www.biophysics.org www.biophysj.org Sackmann: Lehrbuch der Biophysik Versuch einer Annäherung Biophysics is that branch of
Lipide und Zellmembranen. Stryer ed. 6, Kapitel 12
 Lipide und Zellmembranen Stryer ed. 6, Kapitel 12 Biologische Membranen Die Grenzen, die eine Zelle kennzeichnen, werden von biologischen i Membranen gebildet: sie definieren eine innere un eine äussere
Lipide und Zellmembranen Stryer ed. 6, Kapitel 12 Biologische Membranen Die Grenzen, die eine Zelle kennzeichnen, werden von biologischen i Membranen gebildet: sie definieren eine innere un eine äussere
Aufgabe 5 (Supersekundärstruktur)
 Aufgabe 5 (Supersekundärstruktur) Fragestellung Bei der Untereinheit des Arthropodenhämocyanins aus Limulus polyphemus werden folgende Fragestellungen untersucht: - Welche Supersekundärstrukturen gibt
Aufgabe 5 (Supersekundärstruktur) Fragestellung Bei der Untereinheit des Arthropodenhämocyanins aus Limulus polyphemus werden folgende Fragestellungen untersucht: - Welche Supersekundärstrukturen gibt
Tobler Ackermann Fraefel Allgemeine Virologie. basics
 Tobler Ackermann Fraefel Allgemeine Virologie basics 20 Einführung in die Virologie GFP-Fluoreszenz auch in lebenden Zellen nachgewiesen werden. GFP kann mit den meisten anderen Proteinen (auch Virusproteinen),
Tobler Ackermann Fraefel Allgemeine Virologie basics 20 Einführung in die Virologie GFP-Fluoreszenz auch in lebenden Zellen nachgewiesen werden. GFP kann mit den meisten anderen Proteinen (auch Virusproteinen),
12. Biopolymere. daher Namensunterschied nur durch die Zusätze D (rechtsdrehend) und L (linksdrehend)
 12. Biopolymere 12.1 Die wichtigsten Bausteine a) Zucker Zucker C(H 2 O) n dienen zb als Energiespeicher (Stärke), in der Zellerkennung (Blutgruppen) und als Bausteine der Nukleinsäuren es gibt viele Isomere,
12. Biopolymere 12.1 Die wichtigsten Bausteine a) Zucker Zucker C(H 2 O) n dienen zb als Energiespeicher (Stärke), in der Zellerkennung (Blutgruppen) und als Bausteine der Nukleinsäuren es gibt viele Isomere,
Zellbiologie! Privatdozent Dr. T. Kähne! Institut für Experimentelle Innere Medizin! Medizinische Fakultät
 Zellbiologie! Privatdozent Dr. T. Kähne! Institut für Experimentelle Innere Medizin! Medizinische Fakultät Grundlagen Lipid-Doppelschicht als Barriere für polare Moleküle! Abgrenzung für biochemische
Zellbiologie! Privatdozent Dr. T. Kähne! Institut für Experimentelle Innere Medizin! Medizinische Fakultät Grundlagen Lipid-Doppelschicht als Barriere für polare Moleküle! Abgrenzung für biochemische
Membranen. U. Albrecht
 Membranen Struktur einer Plasmamembran Moleküle gegeneinander beweglich -> flüssiger Charakter Fluidität abhängig von 1) Lipidzusammensetzung (gesättigt/ungesättigt) 2) Umgebungstemperatur Biologische
Membranen Struktur einer Plasmamembran Moleküle gegeneinander beweglich -> flüssiger Charakter Fluidität abhängig von 1) Lipidzusammensetzung (gesättigt/ungesättigt) 2) Umgebungstemperatur Biologische
In der Membran sind Ionenkanäle eingebaut leiten Ionen sehr schnell (10 9 Ionen / s)
 Mechanismen in der Zellmembran Abb 7.1 Kandel Neurowissenschaften Die Ionenkanäle gestatten den Durchtritt von Ionen in die Zelle. Die Membran (Doppelschicht von Phosholipiden) ist hydrophob und die Ionen
Mechanismen in der Zellmembran Abb 7.1 Kandel Neurowissenschaften Die Ionenkanäle gestatten den Durchtritt von Ionen in die Zelle. Die Membran (Doppelschicht von Phosholipiden) ist hydrophob und die Ionen
12. Biopolymere. Anwendungen: Sensoren, Detektoren, Displays, Komponenten in elektrischen Schlatkreisen Modellsysteme
 12. Biopolymere 12.1 Organische dünne Filme Langmuir Filme = organische Polymere auf flüssigen Oberflächen Langmuir-Blodgett Filme = organische Polymere auf festen Oberflächen Anwendungen: Sensoren, Detektoren,
12. Biopolymere 12.1 Organische dünne Filme Langmuir Filme = organische Polymere auf flüssigen Oberflächen Langmuir-Blodgett Filme = organische Polymere auf festen Oberflächen Anwendungen: Sensoren, Detektoren,
Die Zelle. Membranen: Struktur und Funktion
 Die Zelle Membranen: Struktur und Funktion 8.4 Die Fluidität von Membranen. 8.6 Die Feinstruktur der Plasmamembran einer Tierzelle (Querschnitt). (Zum Aufbau der extrazellulären Matrix siehe auch Abbildung
Die Zelle Membranen: Struktur und Funktion 8.4 Die Fluidität von Membranen. 8.6 Die Feinstruktur der Plasmamembran einer Tierzelle (Querschnitt). (Zum Aufbau der extrazellulären Matrix siehe auch Abbildung
Membranen und Potentiale
 Membranen und Potentiale 1. Einleitung 2. Zellmembran 3. Ionenkanäle 4. Ruhepotential 5. Aktionspotential 6. Methode: Patch-Clamp-Technik Quelle: Thompson Kap. 3, (Pinel Kap. 3) 2. ZELLMEMBRAN Abbildung
Membranen und Potentiale 1. Einleitung 2. Zellmembran 3. Ionenkanäle 4. Ruhepotential 5. Aktionspotential 6. Methode: Patch-Clamp-Technik Quelle: Thompson Kap. 3, (Pinel Kap. 3) 2. ZELLMEMBRAN Abbildung
Cholesterolmoleküle. Membranproteine können Zellen. miteinander verknüpfen. tragen Kohlenhydratketten. Manche Lipide (Glykolipide)
 Zellinnenraum Manche Lipide (Glykolipide) tragen Kohlenhydratketten. Membranproteine können Zellen miteinander verknüpfen. Manche Proteine (Glykoproteine) tragen Kohlenhydratketten. Cholesterolmoleküle
Zellinnenraum Manche Lipide (Glykolipide) tragen Kohlenhydratketten. Membranproteine können Zellen miteinander verknüpfen. Manche Proteine (Glykoproteine) tragen Kohlenhydratketten. Cholesterolmoleküle
Übertragung zwischen einzelnen Nervenzellen: Synapsen
 Übertragung zwischen einzelnen Nervenzellen: Synapsen Kontaktpunkt zwischen zwei Nervenzellen oder zwischen Nervenzelle und Zielzelle (z.b. Muskelfaser) Synapse besteht aus präsynaptischen Anteil (sendendes
Übertragung zwischen einzelnen Nervenzellen: Synapsen Kontaktpunkt zwischen zwei Nervenzellen oder zwischen Nervenzelle und Zielzelle (z.b. Muskelfaser) Synapse besteht aus präsynaptischen Anteil (sendendes
Einführung in das Molecular Modelling
 Einführung in das Molecular Modelling Darstellung und Bearbeitung dreidimensionaler Molekülstrukturen Berechnung der physikochemischen Eigenschaften Ziel: Einsicht in die molekularen Mechanismen der Arzneistoffwirkung
Einführung in das Molecular Modelling Darstellung und Bearbeitung dreidimensionaler Molekülstrukturen Berechnung der physikochemischen Eigenschaften Ziel: Einsicht in die molekularen Mechanismen der Arzneistoffwirkung
EINFÜHRUNG IN DIE BIOPHYSIK
 EINFÜHRUNG IN DIE BIOPHYSIK Helmut Grubmüller, Detlev Schild Max Planck-Institut für biophysikalische Chemie Theoretische und Computergestützte Biophysik Am Fassberg 11 37077 Göttingen Tel.: 201-2300 /
EINFÜHRUNG IN DIE BIOPHYSIK Helmut Grubmüller, Detlev Schild Max Planck-Institut für biophysikalische Chemie Theoretische und Computergestützte Biophysik Am Fassberg 11 37077 Göttingen Tel.: 201-2300 /
Vorlesung Biophysik I - Molekulare Biophysik Kalbitzer/Kremer/Ziegler
 Vorlesung Biophysik I - Molekulare Biophysik Kalbitzer/Kremer/Ziegler 23.10. Zelle 30.10. Biologische Makromoleküle I 06.11. Biologische Makromoleküle II 13.11. Nukleinsäuren-Origami (DNA, RNA) 20.11.
Vorlesung Biophysik I - Molekulare Biophysik Kalbitzer/Kremer/Ziegler 23.10. Zelle 30.10. Biologische Makromoleküle I 06.11. Biologische Makromoleküle II 13.11. Nukleinsäuren-Origami (DNA, RNA) 20.11.
Nanostrukturphysik II Michael Penth
 16.07.13 Nanostrukturphysik II Michael Penth Ladungstransport essentiell für Funktionalität jeder Zelle [b] [a] [j] de.academic.ru esys.org giantshoulders.wordpress.com [f] 2 Mechanismen des Ionentransports
16.07.13 Nanostrukturphysik II Michael Penth Ladungstransport essentiell für Funktionalität jeder Zelle [b] [a] [j] de.academic.ru esys.org giantshoulders.wordpress.com [f] 2 Mechanismen des Ionentransports
Computational Chemistry
 The fundamental laws necessary for the mathematical treatment of a large part of physics and the whole of chemistry are thus completely known, and the difficulty lies only in the fact that application
The fundamental laws necessary for the mathematical treatment of a large part of physics and the whole of chemistry are thus completely known, and the difficulty lies only in the fact that application
Simulation von Brownscher Dynamik und Assoziationsraten von PP-Komplexen. Alexander Baldauf Montag
 Simulation von Brownscher Dynamik und Assoziationsraten von PP-Komplexen Alexander Baldauf Montag 25.04.2005 Motivation Typen von Interaktionen Verschiedene Methoden zur Vorhersage Wie kommen die beiden
Simulation von Brownscher Dynamik und Assoziationsraten von PP-Komplexen Alexander Baldauf Montag 25.04.2005 Motivation Typen von Interaktionen Verschiedene Methoden zur Vorhersage Wie kommen die beiden
Passive Transportvorgänge
 Passive Transportvorgänge Diffusion und Osmose sind passive Transportprozesse. Denn die Zelle muss keine Energie aufwenden, um den Transport der Stoffe zu ermöglichen. Diffusion Einzelsubstanzen sind bestrebt,
Passive Transportvorgänge Diffusion und Osmose sind passive Transportprozesse. Denn die Zelle muss keine Energie aufwenden, um den Transport der Stoffe zu ermöglichen. Diffusion Einzelsubstanzen sind bestrebt,
Zellmembran. Zellmembran (TEM) ( x)
 Zellmembran Zellmembran (TEM) (100.000 x) Die Zellmembran ist der äußere Abschluß des Zellinhalts jeder Zelle. Bei Pflanzen- und Bakterienzellen liegt ihr außen noch eine Zellwand auf. Die Zellmembran
Zellmembran Zellmembran (TEM) (100.000 x) Die Zellmembran ist der äußere Abschluß des Zellinhalts jeder Zelle. Bei Pflanzen- und Bakterienzellen liegt ihr außen noch eine Zellwand auf. Die Zellmembran
Proteinogene Aminosäuren. Unpolare, aliphatische Seitenketten Monoaminomonocarbonsäuren
 Proteinogene Aminosäuren Unpolare, aliphatische Seitenketten Monoaminomonocarbonsäuren Proteinogene Aminosäuren Unpolare, heterozyklische Seitenkette Monoaminomonocarbonsäuren Proteinogene Aminosäuren
Proteinogene Aminosäuren Unpolare, aliphatische Seitenketten Monoaminomonocarbonsäuren Proteinogene Aminosäuren Unpolare, heterozyklische Seitenkette Monoaminomonocarbonsäuren Proteinogene Aminosäuren
Protein-Datenbanken. Protein- Datenbanken. Christian Fink
 Protein- Datenbanken Christian Fink 1 » Übersicht «PDB PDB_TM OPM HIC-UP Klotho 2 » PDB «PDB = Protein Data Bank 3D-Strukturen von Proteinen & Nukleinsäuren Gegründet 1971 als freies Archiv für biologische
Protein- Datenbanken Christian Fink 1 » Übersicht «PDB PDB_TM OPM HIC-UP Klotho 2 » PDB «PDB = Protein Data Bank 3D-Strukturen von Proteinen & Nukleinsäuren Gegründet 1971 als freies Archiv für biologische
Kompetenzen und Aufgabenbeispiele Natur und Technik Check S2 / Check S3
 Institut für Bildungsevaluation Assoziiertes Institut der Universität Zürich Kompetenzen und Aufgabenbeispiele Natur und Technik Check S2 / Check S3 Informationen für Lehrpersonen und Eltern 30. Mai 2017
Institut für Bildungsevaluation Assoziiertes Institut der Universität Zürich Kompetenzen und Aufgabenbeispiele Natur und Technik Check S2 / Check S3 Informationen für Lehrpersonen und Eltern 30. Mai 2017
Bioorganische Chemie Enzymatische Katalyse 2011
 Ringvorlesung Chemie B - Studiengang Molekulare Biotechnologie Bioorganische Chemie Enzymatische Katalyse 2011 Prof. Dr. A. Jäschke INF 364, Zi. 308, Tel. 54 48 51 jaeschke@uni-hd.de Lehrziele I Kenntnis
Ringvorlesung Chemie B - Studiengang Molekulare Biotechnologie Bioorganische Chemie Enzymatische Katalyse 2011 Prof. Dr. A. Jäschke INF 364, Zi. 308, Tel. 54 48 51 jaeschke@uni-hd.de Lehrziele I Kenntnis
Konformation von Polypeptiden
 1 Konformation von Polypeptiden Peter Güntert, Wintersemester 2011/12 Primärstruktur Polypeptide sind lineare Ketten, die aus 20 verschiedenen Typen von Bausteinen (Aminosäureresten, AS) aufgebaut sind.
1 Konformation von Polypeptiden Peter Güntert, Wintersemester 2011/12 Primärstruktur Polypeptide sind lineare Ketten, die aus 20 verschiedenen Typen von Bausteinen (Aminosäureresten, AS) aufgebaut sind.
UAufgabe 12: (evolutiv konservierte Aminosäuren)
 UAufgabe 12: (evolutiv konservierte Aminosäuren) Aufgabenstellung Wählen Sie zur Darstellung evolutiv konservierter Aminosäure-Positionen in "1lla" eine ihnen sinnvoll erscheinende Anfärbung. Exportieren
UAufgabe 12: (evolutiv konservierte Aminosäuren) Aufgabenstellung Wählen Sie zur Darstellung evolutiv konservierter Aminosäure-Positionen in "1lla" eine ihnen sinnvoll erscheinende Anfärbung. Exportieren
O H C N S
 Virus-Replikation C 332.662 H 492.338 N 98.245 O 131.196 P 7.500 S 2.340 Capsid (Protein) Nukleokapsid Virion Genom (Nukleinsäure) Nukleokapsid Virion Envelope (Hülle) Virus-Replikation Replikation =
Virus-Replikation C 332.662 H 492.338 N 98.245 O 131.196 P 7.500 S 2.340 Capsid (Protein) Nukleokapsid Virion Genom (Nukleinsäure) Nukleokapsid Virion Envelope (Hülle) Virus-Replikation Replikation =
Fragenkatalog zu BIOPHYSIK I: Teil 2
 Fragenkatalog zu BIOPHYSIK I: Teil 2 1. Was besagt das Lösungsmodell des Membrantransports? Leiten Sie einen mathematischen Ausdruck für die Membranpermeabilität her, der dieses Modell widerspiegelt! 2.
Fragenkatalog zu BIOPHYSIK I: Teil 2 1. Was besagt das Lösungsmodell des Membrantransports? Leiten Sie einen mathematischen Ausdruck für die Membranpermeabilität her, der dieses Modell widerspiegelt! 2.
Fragenkatalog Biophysik II (Stand )
 Fragenkatalog Biophysik II (Stand 18.5.2013) 1. Die Membranproteinfaltung erfolgt in zwei grundlegenden Strukturen. Benennen Sie diese, und gehen Sie auf Gemeinsamkeiten und Unterschiede im Aufbau und
Fragenkatalog Biophysik II (Stand 18.5.2013) 1. Die Membranproteinfaltung erfolgt in zwei grundlegenden Strukturen. Benennen Sie diese, und gehen Sie auf Gemeinsamkeiten und Unterschiede im Aufbau und
Übungsaufgaben. Aufbau und Konformation von Polypeptiden. Einführung in die räumliche Struktur von Proteinen
 Computergestützte Strukturbiologie (Strukturelle Bioinformatik) SS09 P. Güntert Übungsaufgaben Aufbau und Konformation von Polypeptiden 1. Warum haben Proteine im Unterschied zu DNA komplizierte und vielfältige
Computergestützte Strukturbiologie (Strukturelle Bioinformatik) SS09 P. Güntert Übungsaufgaben Aufbau und Konformation von Polypeptiden 1. Warum haben Proteine im Unterschied zu DNA komplizierte und vielfältige
Kapitel 9: KÜNSTLICHE UND NATÜRLICHE MEMBRANEN. Einführung. Inhalt:
 Kapitel 9: KÜNSTLICE UND NATÜRLICE MEMBRANEN Inhalt: EINFÜRUNG... 121 KÜNSTLICE MEMBRANEN... 122 BILGISCE MEMBRANEN... 127 LITERATUR... 129 LINKS... 129 Einführung Jede lebende Zelle ist von einer Membran
Kapitel 9: KÜNSTLICE UND NATÜRLICE MEMBRANEN Inhalt: EINFÜRUNG... 121 KÜNSTLICE MEMBRANEN... 122 BILGISCE MEMBRANEN... 127 LITERATUR... 129 LINKS... 129 Einführung Jede lebende Zelle ist von einer Membran
Tyrosinkinase- Rezeptoren
 Tyrosinkinase- Rezeptoren für bestimmte Hormone gibt es integrale Membranproteine als Rezeptoren Aufbau und Signaltransduktionsweg unterscheiden sich von denen der G- Protein- gekoppelten Rezeptoren Polypeptide
Tyrosinkinase- Rezeptoren für bestimmte Hormone gibt es integrale Membranproteine als Rezeptoren Aufbau und Signaltransduktionsweg unterscheiden sich von denen der G- Protein- gekoppelten Rezeptoren Polypeptide
Ruhemebranpotenzial. den 17 November Dr. Emőke Bódis
 Ruhemebranpotenzial den 17 November 2016 Dr. Emőke Bódis Prüfungsfrage Die Struktur und die Eigenschaften der Zellmembran. Das Ruhemembranpotenzial. Bernstein Kalium Hypothese, Nernst- Gleichung, Donnan-
Ruhemebranpotenzial den 17 November 2016 Dr. Emőke Bódis Prüfungsfrage Die Struktur und die Eigenschaften der Zellmembran. Das Ruhemembranpotenzial. Bernstein Kalium Hypothese, Nernst- Gleichung, Donnan-
Grundlagen der Molekularen Biophysik WS 2011/12 (Bachelor) Dozent: Prof Dr. Ulrike Alexiev (R , Tel /Sekretariat Frau Endrias Tel.
 Grundlagen der Molekularen Biophysik WS 2011/12 (Bachelor) Dozent: Prof Dr. Ulrike Alexiev (R.1.2.34, Tel. 55157/Sekretariat Frau Endrias Tel. 53337) Tutoren: Dr. Kristina Kirchberg, Alex Boreham 6-stündig
Grundlagen der Molekularen Biophysik WS 2011/12 (Bachelor) Dozent: Prof Dr. Ulrike Alexiev (R.1.2.34, Tel. 55157/Sekretariat Frau Endrias Tel. 53337) Tutoren: Dr. Kristina Kirchberg, Alex Boreham 6-stündig
Biologie für Mediziner
 Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Morphologische Grundlagen der Zelle Bakterienzelle
 Morphologische Grundlagen der Zelle Bakterienzelle Entstehung der Eukaryontenzelle Endosymbiontentheorie Tier-Zelle Pflanzen-Zelle Entstehung der Eukaryontenzelle Endosymbiontentheorie (aus Weiler/Nover:
Morphologische Grundlagen der Zelle Bakterienzelle Entstehung der Eukaryontenzelle Endosymbiontentheorie Tier-Zelle Pflanzen-Zelle Entstehung der Eukaryontenzelle Endosymbiontentheorie (aus Weiler/Nover:
2 Coulomb-Kraft Elektrisches Potential... 9
 Inhaltsverzeichnis I Strukturprinzipien 7 1 Konservative Kraft / Potential 7 2 Coulomb-Kraft 8 2.1 Elektrisches Potential....................... 9 3 Abstoßendes Potential 10 4 Bindungsarten 10 4.1 Ionische
Inhaltsverzeichnis I Strukturprinzipien 7 1 Konservative Kraft / Potential 7 2 Coulomb-Kraft 8 2.1 Elektrisches Potential....................... 9 3 Abstoßendes Potential 10 4 Bindungsarten 10 4.1 Ionische
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne
 Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Signale und Signalwege in Zellen
 Signale und Signalwege in Zellen Zellen müssen Signale empfangen, auf sie reagieren und Signale zu anderen Zellen senden können Signalübertragungsprozesse sind biochemische (und z.t. elektrische) Prozesse
Signale und Signalwege in Zellen Zellen müssen Signale empfangen, auf sie reagieren und Signale zu anderen Zellen senden können Signalübertragungsprozesse sind biochemische (und z.t. elektrische) Prozesse
Experimentalphysik 2
 Repetitorium zu Experimentalphysik 2 Ferienkurs am Physik-Department der Technischen Universität München Gerd Meisl 5. August 2008 Inhaltsverzeichnis 1 Übungsaufgaben 2 1.1 Übungsaufgaben....................................
Repetitorium zu Experimentalphysik 2 Ferienkurs am Physik-Department der Technischen Universität München Gerd Meisl 5. August 2008 Inhaltsverzeichnis 1 Übungsaufgaben 2 1.1 Übungsaufgaben....................................
Membranen (3a) Membrane
 Membranen (3a) Biochemie Membrane Funktionen Abgrenzung der Zelle nach außen hin schaffen geeignete Voraussetzungen für einzelne enzymatische Reaktionen Signalübertragung tragen Signalstrukturen für die
Membranen (3a) Biochemie Membrane Funktionen Abgrenzung der Zelle nach außen hin schaffen geeignete Voraussetzungen für einzelne enzymatische Reaktionen Signalübertragung tragen Signalstrukturen für die
2006 Block 3 Phy 1 Aufbau und Funktion der Zellmembran
 2006 Block 3 Phy 1 Aufbau und Funktion der Zellmembran Objectives I Beschreibe die Membran-Eigenschaften in Relation zu den Komponenten der Membran Beschreibe den Aufbau einer Zellmembran Benenne unterschiedliche
2006 Block 3 Phy 1 Aufbau und Funktion der Zellmembran Objectives I Beschreibe die Membran-Eigenschaften in Relation zu den Komponenten der Membran Beschreibe den Aufbau einer Zellmembran Benenne unterschiedliche
Membran-Aufbau. Michael Hertrich Bielefeld University Version 2.1.3
 Membran-Aufbau Michael Hertrich Bielefeld University 27.11.2006 Version 2.1.3 Inhaltsüberblick Membranlipide - Arten und Vorkommen - Anordung - Phospholipid-Beweglichkeit - Selektive Permeabilität - Wechselwirkungen
Membran-Aufbau Michael Hertrich Bielefeld University 27.11.2006 Version 2.1.3 Inhaltsüberblick Membranlipide - Arten und Vorkommen - Anordung - Phospholipid-Beweglichkeit - Selektive Permeabilität - Wechselwirkungen
Zellulärer Abbau von Proteinen in Aminosäuren:! Proteine werden in Zellen durch Proteasom-Komplexe in! einzelne Aminosäuren abgebaut.!
 Zellulärer Abbau von Proteinen in Aminosäuren: Proteine werden in Zellen durch Proteasom-Komplexe in einzelne Aminosäuren abgebaut. Abbau von Aminosäuren: Uebersicht über den Aminosäureabbau Als erster
Zellulärer Abbau von Proteinen in Aminosäuren: Proteine werden in Zellen durch Proteasom-Komplexe in einzelne Aminosäuren abgebaut. Abbau von Aminosäuren: Uebersicht über den Aminosäureabbau Als erster
Übung 6 Vorlesung Bio-Engineering Sommersemester Nervenzellen: Kapitel 4. 1
 Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 21. 04. 08-16:30
Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 21. 04. 08-16:30
Proteinstrukturklassen α-helikale Proteine
 Proteinstrukturklassen α-helikale Proteine Wintersemester 2011/12 Peter Güntert Domäne Modul Faltung Domäne (domain): Polypeptidkette (oder Teil davon), die unabhängig in eine stabile, kompakte Tertiärstruktur
Proteinstrukturklassen α-helikale Proteine Wintersemester 2011/12 Peter Güntert Domäne Modul Faltung Domäne (domain): Polypeptidkette (oder Teil davon), die unabhängig in eine stabile, kompakte Tertiärstruktur
Prof. Dr. Stefan Schuster Lehrstuhl für Tierphysiologie
 Prof. Dr. Stefan Schuster Lehrstuhl für Tierphysiologie Tierphysiologie = Wie Tiere funktionieren Welche Anpassungen. Leistungen, Moleküle etc sie einsetzen um zu leben und möglichst am Leben zu beiben
Prof. Dr. Stefan Schuster Lehrstuhl für Tierphysiologie Tierphysiologie = Wie Tiere funktionieren Welche Anpassungen. Leistungen, Moleküle etc sie einsetzen um zu leben und möglichst am Leben zu beiben
Ionenkanäle der Zellmembran. Seminar Differenzialgleichungen in der Biomedizin SoSe09 Karoline Jäger
 Ionenkanäle der Zellmembran Seminar Differenzialgleichungen in der Biomedizin SoSe09 Karoline Jäger Inhaltsverzeichnis 1. Strom-Spannung Beziehung 2. Unabhängigkeit, Sättigung, Ussing Fluss Rate 3. Elektrodiffusions
Ionenkanäle der Zellmembran Seminar Differenzialgleichungen in der Biomedizin SoSe09 Karoline Jäger Inhaltsverzeichnis 1. Strom-Spannung Beziehung 2. Unabhängigkeit, Sättigung, Ussing Fluss Rate 3. Elektrodiffusions
Conotoxine. Von Dominic Hündgen und Evgeni Drobjasko
 Conotoxine Von Dominic Hündgen und Evgeni Drobjasko Inhalt Vorkommen Aufbau/Arten Wirkungsweise Therapie und Nutzen 2 Vorkommen Gruppe von Toxinen aus der Kegelschnecken-Gattung Conus 600 anerkannte Arten
Conotoxine Von Dominic Hündgen und Evgeni Drobjasko Inhalt Vorkommen Aufbau/Arten Wirkungsweise Therapie und Nutzen 2 Vorkommen Gruppe von Toxinen aus der Kegelschnecken-Gattung Conus 600 anerkannte Arten
Aufbau der Zellmembran Zusammensetzung
 Membranphysiologie Die Zelle Aufbau der Zellmembran Zusammensetzung Der Anteil an Proteinen, Lipiden und Kohlehydraten variiert in biologischen Membranen sehr stark: Membran Anteil an der Trockenmasse
Membranphysiologie Die Zelle Aufbau der Zellmembran Zusammensetzung Der Anteil an Proteinen, Lipiden und Kohlehydraten variiert in biologischen Membranen sehr stark: Membran Anteil an der Trockenmasse
2.2. Peptide. Peptide entstehen durch Kondensation der a-carboxylgruppe einer Aminosäure mit der a-aminogruppe einer anderen Aminosäure
 2.2. Peptide Peptide entstehen durch Kondensation der a-carboxylgruppe einer Aminosäure mit der a-aminogruppe einer anderen Aminosäure Peptid: bis zu ~30 linear über Peptidbindung verknüpfte Aminosäuren
2.2. Peptide Peptide entstehen durch Kondensation der a-carboxylgruppe einer Aminosäure mit der a-aminogruppe einer anderen Aminosäure Peptid: bis zu ~30 linear über Peptidbindung verknüpfte Aminosäuren
Vertiefendes Seminar zur Vorlesung Biochemie
 Vertiefendes Seminar zur Vorlesung Biochemie 31.10.2014 Proteine: Struktur Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/abteilung-van-echten-deckert/abt-van-echten-deckert-startseite/
Vertiefendes Seminar zur Vorlesung Biochemie 31.10.2014 Proteine: Struktur Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/abteilung-van-echten-deckert/abt-van-echten-deckert-startseite/
Entstehung der Eukaryontenzelle Endosymbiontentheorie
 Entstehung der Eukaryontenzelle Endosymbiontentheorie Tier-Zelle Pflanzen-Zelle Entstehung der Eukaryontenzelle Endosymbiontentheorie (aus Weiler/Nover: Allgemeine und molekulare Botanik) Tierzelle Morphologische
Entstehung der Eukaryontenzelle Endosymbiontentheorie Tier-Zelle Pflanzen-Zelle Entstehung der Eukaryontenzelle Endosymbiontentheorie (aus Weiler/Nover: Allgemeine und molekulare Botanik) Tierzelle Morphologische
Molecular Dynamics of Proteorhodopsin in Lipid Bilayers by Solid-State NMR
 Molecular Dynamics of Proteorhodopsin in Lipid Bilayers by Solid-State NMR Yang, Aslimovsk, Glaubitz Moderne Anwendungen der Magnetischen Resonanz Lena Kalinowsky Motivation/Ziel Temperatur Hydrathülle
Molecular Dynamics of Proteorhodopsin in Lipid Bilayers by Solid-State NMR Yang, Aslimovsk, Glaubitz Moderne Anwendungen der Magnetischen Resonanz Lena Kalinowsky Motivation/Ziel Temperatur Hydrathülle
1. Klausur Allgemeine und Anorganische Chemie B.Sc. Chemie
 1. Klausur Allgemeine und Anorganische Chemie B.Sc. Chemie Name: Vorname: Matrikel Nr.: 15.12.2010 Die Durchführung und Auswertung der 12 Aufgaben im zweiten Teil dieser Klausur mit je vier Aussagen (a-d)
1. Klausur Allgemeine und Anorganische Chemie B.Sc. Chemie Name: Vorname: Matrikel Nr.: 15.12.2010 Die Durchführung und Auswertung der 12 Aufgaben im zweiten Teil dieser Klausur mit je vier Aussagen (a-d)
Aufbau und Konformation von Polypeptiden
 1 Aufbau und Konformation von Polypeptiden Peter Güntert, Sommersemester 2009 Hierarchie von Proteinstrukturen Primärstruktur: Aminosäuresequenz Sekundärstruktur: Helices, Faltblätter, Turns, Loops Tertiärstruktur:
1 Aufbau und Konformation von Polypeptiden Peter Güntert, Sommersemester 2009 Hierarchie von Proteinstrukturen Primärstruktur: Aminosäuresequenz Sekundärstruktur: Helices, Faltblätter, Turns, Loops Tertiärstruktur:
3D-Einblicke in die molekulare Teamarbeit in Biomembranen
 Powered by Seiten-Adresse: Rund dreißig Prozent aller Proteine in einer Zelle stehen mit einer Biomembran in Verbindung einige durchqueren die Lipiddoppelschicht, manchmal sogar mehrfach, andere sind an
Powered by Seiten-Adresse: Rund dreißig Prozent aller Proteine in einer Zelle stehen mit einer Biomembran in Verbindung einige durchqueren die Lipiddoppelschicht, manchmal sogar mehrfach, andere sind an
Das Gehirn: Eine Einführung in die Molekulare Neurobiologie. A. Baumann
 Das Gehirn: Eine Einführung in die Molekulare Neurobiologie A. Baumann Das Gehirn Ligandengesteuerte Ionenkanäle Das Gehirn Ligandengesteuerte Kanäle Ligand-gated ion channels LGIC Ionotrope Rezeptoren
Das Gehirn: Eine Einführung in die Molekulare Neurobiologie A. Baumann Das Gehirn Ligandengesteuerte Ionenkanäle Das Gehirn Ligandengesteuerte Kanäle Ligand-gated ion channels LGIC Ionotrope Rezeptoren
TU Bergakademie Freiberg Institut für Werkstofftechnik Schülerlabor science meets school Werkstoffe und Technologien in Freiberg
 TU Bergakademie Freiberg Institut für Werkstofftechnik Schülerlabor science meets school Werkstoffe und Technologien in Freiberg GRUNDLAGEN Modul: Versuch: Elektrochemie 1 Abbildung 1: I. VERSUCHSZIEL
TU Bergakademie Freiberg Institut für Werkstofftechnik Schülerlabor science meets school Werkstoffe und Technologien in Freiberg GRUNDLAGEN Modul: Versuch: Elektrochemie 1 Abbildung 1: I. VERSUCHSZIEL
Diffusion. Prüfungsfrage
 Prüfungsfrage Diffusion Die Diffusion. Erstes Fick sches Gesetz. Der Diffusionskoeffizient. Die Stokes-Einstein Beziehung. Diffusion durch die Zellmembrane: passive, aktive und erleichterte Diffusion Lehrbuch
Prüfungsfrage Diffusion Die Diffusion. Erstes Fick sches Gesetz. Der Diffusionskoeffizient. Die Stokes-Einstein Beziehung. Diffusion durch die Zellmembrane: passive, aktive und erleichterte Diffusion Lehrbuch
Vertiefendes Seminar zur Vorlesung Biochemie I Bearbeitung Übungsblatt 4
 Vertiefendes Seminar zur Vorlesung Biochemie I 20.11.2015 Bearbeitung Übungsblatt 4 Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/
Vertiefendes Seminar zur Vorlesung Biochemie I 20.11.2015 Bearbeitung Übungsblatt 4 Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/
Knospung und Reifung des HI-Virus an der Membran
 Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/knospung-und-reifung-deshi-virus-an-der-membran/ Knospung und Reifung des HI-Virus an der Membran Der Heidelberger
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/knospung-und-reifung-deshi-virus-an-der-membran/ Knospung und Reifung des HI-Virus an der Membran Der Heidelberger
dm A A = D --- (c 1 -c 2 ) = D --- δ c dt d d D: Diffusionskonstante, A: Betrachtete Fläche, d: Strecke c: Konzentration
 Diffusion ist die Bewegung von Teilchen aufgrund der brownschen Molekularbewegung in einem Lösungsmittel. Die Teilchen bewegen sich netto in Richtung der niedrigeren Konzentration. Ficksches Diffusionsgesetz:
Diffusion ist die Bewegung von Teilchen aufgrund der brownschen Molekularbewegung in einem Lösungsmittel. Die Teilchen bewegen sich netto in Richtung der niedrigeren Konzentration. Ficksches Diffusionsgesetz:
Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen. Abb. aus Stryer (5th Ed.)
 Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen Die verschiedenen Ribosomen-Komplexe können im Elektronenmikroskop beobachtet werden Durch Röntgenkristallographie wurden
Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen Die verschiedenen Ribosomen-Komplexe können im Elektronenmikroskop beobachtet werden Durch Röntgenkristallographie wurden
Experimentelle Ermittlung der molaren Lösungswärme von Kaliumchlorid
 Experimentelle Ermittlung der molaren Lösungswärme von Kaliumchlorid Versuchsaufbau : Um den Versuch durchzuführen, benötigen wir 180 g Wasser, welches in ein Becherglas gefüllt wird. Die Temperatur ermitteln
Experimentelle Ermittlung der molaren Lösungswärme von Kaliumchlorid Versuchsaufbau : Um den Versuch durchzuführen, benötigen wir 180 g Wasser, welches in ein Becherglas gefüllt wird. Die Temperatur ermitteln
Universität Stuttgart Institut für Technische Biochemie Lehrstuhl für Biochemie
 Universität Stuttgart Institut für Technische Biochemie Lehrstuhl für Biochemie Address Pfaffenwaldring 55 70569 Stuttgart Deutschland Telephone +49 711 685-64391 Fax +49 711 685-64392 E-Mail elisabeth.tosta@ibc.uni-stuttgart.de
Universität Stuttgart Institut für Technische Biochemie Lehrstuhl für Biochemie Address Pfaffenwaldring 55 70569 Stuttgart Deutschland Telephone +49 711 685-64391 Fax +49 711 685-64392 E-Mail elisabeth.tosta@ibc.uni-stuttgart.de
Die Substratspezifität der Enzyme Trypsin und Chymotrypsin
 Die Substratspezifität der Enzyme Trypsin und Chymotrypsin Einleitung Die Proteine Trypsin und Chymotrypsin sind Enzyme, die im Magen von Säugetieren vorkommen und die Hydrolyse von Peptidbindungen katalysieren.
Die Substratspezifität der Enzyme Trypsin und Chymotrypsin Einleitung Die Proteine Trypsin und Chymotrypsin sind Enzyme, die im Magen von Säugetieren vorkommen und die Hydrolyse von Peptidbindungen katalysieren.
Die Zellmembran und das endoplasmatische Retikulum. Dr. Alpár Alán
 Die Zellmembran und das endoplasmatische Retikulum Dr. Alpár Alán Schema einer eukariotischer Zelle Sekretgranula Golgi-Apparat Selbständig lebensfähig Selbstreduplikation Produktion: Interzellularsubstanz
Die Zellmembran und das endoplasmatische Retikulum Dr. Alpár Alán Schema einer eukariotischer Zelle Sekretgranula Golgi-Apparat Selbständig lebensfähig Selbstreduplikation Produktion: Interzellularsubstanz
Thomas Zöller (Autor) Verbesserung des Auflösungsverhaltens von schwer löslichen schwachen Säuren durch feste Lösungen und Cyclodextrin- Komplexe
 Thomas Zöller (Autor) Verbesserung des Auflösungsverhaltens von schwer löslichen schwachen Säuren durch feste Lösungen und Cyclodextrin- Komplexe https://cuvillier.de/de/shop/publications/334 Copyright:
Thomas Zöller (Autor) Verbesserung des Auflösungsverhaltens von schwer löslichen schwachen Säuren durch feste Lösungen und Cyclodextrin- Komplexe https://cuvillier.de/de/shop/publications/334 Copyright:
Nanotechnologie der Biomoleküle. Aminosäuren und Proteine: Bausteine der Biologie und der Bionanotechnologie. Aufbau Struktur Funktion
 anotechnologie der Biomoleküle Aminosäuren und Proteine: Bausteine der Biologie und der Bionanotechnologie Aufbau Struktur Funktion Zentrum für Mikro- und anotechnologien Das Miller-Urey-Experiment (auch
anotechnologie der Biomoleküle Aminosäuren und Proteine: Bausteine der Biologie und der Bionanotechnologie Aufbau Struktur Funktion Zentrum für Mikro- und anotechnologien Das Miller-Urey-Experiment (auch
Physikalische Chemie Praktikum. Elektrolyte: Dissoziationskonstante von Essigsäure λ von NaCl ist zu ermitteln
 Hochschule Emden/Leer Physikalische Chemie Praktikum Vers. Nr. 16 April 2017 Elektrolyte: Dissoziationskonstante von Essigsäure λ von NaCl ist zu ermitteln In diesem Versuch soll die Dissoziationskonstante
Hochschule Emden/Leer Physikalische Chemie Praktikum Vers. Nr. 16 April 2017 Elektrolyte: Dissoziationskonstante von Essigsäure λ von NaCl ist zu ermitteln In diesem Versuch soll die Dissoziationskonstante
Human Papilloma Virus
 Human Papilloma Virus Gliederung Allgemeine Informationen Virusstruktur Infektion Verschiedene Arten des Infektionsverlaufs nach Infizierung mit HPV Lebenszyklus des HPV Tumorinduktion Virusstruktur Papillomaviren
Human Papilloma Virus Gliederung Allgemeine Informationen Virusstruktur Infektion Verschiedene Arten des Infektionsverlaufs nach Infizierung mit HPV Lebenszyklus des HPV Tumorinduktion Virusstruktur Papillomaviren
Folding Funnel Rädler / Mathias 45 SS2011
 Folding Funnel 45 Proteinfaltung am Computer Snow/Pande: Folding at home Molekulardynamik Simulationen Integration Newtonscher Bewegungsgleichungen Kraftfeld für kovalente Bindungen Elektrostatische Wechselwirkungen,
Folding Funnel 45 Proteinfaltung am Computer Snow/Pande: Folding at home Molekulardynamik Simulationen Integration Newtonscher Bewegungsgleichungen Kraftfeld für kovalente Bindungen Elektrostatische Wechselwirkungen,
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016
 Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 4 (20.06. 24.06.) Regulation der Transkription II, Translation
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 4 (20.06. 24.06.) Regulation der Transkription II, Translation
Kapitel 8, Membranstruktur und Funktion
 Kapitel 8, Membranstruktur und Funktion Verschiedene Modelle von Membranen, science as a process Die Hauptbestandteile der Membrane sind Lipide, Proteine und Kohlenhydrate. Im Laufe der Zeit haben verschiedene
Kapitel 8, Membranstruktur und Funktion Verschiedene Modelle von Membranen, science as a process Die Hauptbestandteile der Membrane sind Lipide, Proteine und Kohlenhydrate. Im Laufe der Zeit haben verschiedene
Grundlagen: Galvanische Zellen:
 E1 : Ionenprodukt des Wassers Grundlagen: Galvanische Zellen: Die Galvanische Zelle ist eine elektrochemische Zelle. In ihr laufen spontan elektrochemische Reaktionen unter Erzeugung von elektrischer Energie
E1 : Ionenprodukt des Wassers Grundlagen: Galvanische Zellen: Die Galvanische Zelle ist eine elektrochemische Zelle. In ihr laufen spontan elektrochemische Reaktionen unter Erzeugung von elektrischer Energie
Erratum zur Diplomarbeit von Andreas Bender. Korrekturverzeichnis: Kapitel 3.2.2, Seiten 39-46
 Erratum zur Diplomarbeit von Andreas Bender Korrekturverzeichnis: Kapitel 3.2.2, Seiten 39-46 3.2.2. Einzelne Aminosäuren in N-terminalen Abschnitten In den Abbildungen 16-22 sind die relativen Aminosäurehäufigkeiten
Erratum zur Diplomarbeit von Andreas Bender Korrekturverzeichnis: Kapitel 3.2.2, Seiten 39-46 3.2.2. Einzelne Aminosäuren in N-terminalen Abschnitten In den Abbildungen 16-22 sind die relativen Aminosäurehäufigkeiten
Weitere Übungsfragen
 1 Strategie bei multiple choice Fragen Wie unterscheidet sich Glucose von Fructose? (2 Punkte) Glucose hat 6 C Atome, Fructose hat nur 5 C Atome. In der Ringform gibt es bei Glucose α und β Anomere, bei
1 Strategie bei multiple choice Fragen Wie unterscheidet sich Glucose von Fructose? (2 Punkte) Glucose hat 6 C Atome, Fructose hat nur 5 C Atome. In der Ringform gibt es bei Glucose α und β Anomere, bei
1 Bau von Nervenzellen
 Neurophysiologie 1 Bau von Nervenzellen Die funktionelle Einheit des Nervensystems bezeichnet man als Nervenzelle. Dendrit Zellkörper = Soma Zelllkern Axon Ranvier scher Schnürring Schwann sche Hüllzelle
Neurophysiologie 1 Bau von Nervenzellen Die funktionelle Einheit des Nervensystems bezeichnet man als Nervenzelle. Dendrit Zellkörper = Soma Zelllkern Axon Ranvier scher Schnürring Schwann sche Hüllzelle
Prof. Dr. Stephan Ludwig. Institut für Molekulare Virologie (IMV)
 Cystus052, ein polyphenolreicher Pflanzenextrakt wirkt gegen Grippeviren durch Blockierung des Viruseintritts in die Wirtszelle Prof. Dr. Stephan Ludwig Institut für Molekulare Virologie (IMV) Die Virusgrippe
Cystus052, ein polyphenolreicher Pflanzenextrakt wirkt gegen Grippeviren durch Blockierung des Viruseintritts in die Wirtszelle Prof. Dr. Stephan Ludwig Institut für Molekulare Virologie (IMV) Die Virusgrippe
Erregungsübertragung an Synapsen. 1. Einleitung. 2. Schnelle synaptische Erregung. Biopsychologie WiSe Erregungsübertragung an Synapsen
 Erregungsübertragung an Synapsen 1. Einleitung 2. Schnelle synaptische Übertragung 3. Schnelle synaptische Hemmung chemische 4. Desaktivierung der synaptischen Übertragung Synapsen 5. Rezeptoren 6. Langsame
Erregungsübertragung an Synapsen 1. Einleitung 2. Schnelle synaptische Übertragung 3. Schnelle synaptische Hemmung chemische 4. Desaktivierung der synaptischen Übertragung Synapsen 5. Rezeptoren 6. Langsame
Studying Invisible Excited Protein States in Slow Exchange with a Major State Conformation
 Studying Invisible Excited Protein States in Slow Exchange with a Major State Conformation JACS 2012 Pramodh Vallurupalli, Guillaume Bouvignies, and Lewis E. Kay MR-Seminar Nina Kubatova 1 13.10.2014 Inhaltverzeichnis
Studying Invisible Excited Protein States in Slow Exchange with a Major State Conformation JACS 2012 Pramodh Vallurupalli, Guillaume Bouvignies, and Lewis E. Kay MR-Seminar Nina Kubatova 1 13.10.2014 Inhaltverzeichnis
Gruppenpuzzle: Stofftransport durch die Biomembran
 1 Bild anfertigen Text 1+2 2 Bild anfertigen Text 3+4 3 Text schreiben Bild 1+2 4 Text schreiben Bild 3+4 Arbeitsaufträge: 1 Versuchen Sie, die zwei beschriebenen Transportvorgänge in geeigneter Weise
1 Bild anfertigen Text 1+2 2 Bild anfertigen Text 3+4 3 Text schreiben Bild 1+2 4 Text schreiben Bild 3+4 Arbeitsaufträge: 1 Versuchen Sie, die zwei beschriebenen Transportvorgänge in geeigneter Weise
Biologie für Mediziner
 Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Inflammasom bei der Wundheilung
 MolCare Consulting Dr. Bernd Becker Schulsiedlung 6 D-93109 Wiesent Mobil: +49 173 66 79 505 - Tel: +49 9482 9099 036 - Fax: +49 9482 9099 037 - E-Mail: becker@molcare-consulting.com Inflammasom bei der
MolCare Consulting Dr. Bernd Becker Schulsiedlung 6 D-93109 Wiesent Mobil: +49 173 66 79 505 - Tel: +49 9482 9099 036 - Fax: +49 9482 9099 037 - E-Mail: becker@molcare-consulting.com Inflammasom bei der
Dieser Anteil ist oft experimentell zumindest näherungsweise zugänglich, zum Beispiel durch optische Messungen
 Dieser Anteil ist oft experimentell zumindest näherungsweise zugänglich, zum Beispiel durch optische Messungen Die Kooperativität zeigt sich in einem sigmoidalen Verlauf des Ordnungsparameters bei Änderung
Dieser Anteil ist oft experimentell zumindest näherungsweise zugänglich, zum Beispiel durch optische Messungen Die Kooperativität zeigt sich in einem sigmoidalen Verlauf des Ordnungsparameters bei Änderung
Einige Grundbegriffe der Elektrostatik. Elementarladung: e = C
 Einige Grundbegriffe der Elektrostatik Es gibt + und - Ladungen ziehen sich an Einheit der Ladung 1C Elementarladung: e = 1.6.10-19 C 1 Abb 14.7 Biologische Physik 2 Parallel- und Serienschaltung von Kondensatoren/Widerständen
Einige Grundbegriffe der Elektrostatik Es gibt + und - Ladungen ziehen sich an Einheit der Ladung 1C Elementarladung: e = 1.6.10-19 C 1 Abb 14.7 Biologische Physik 2 Parallel- und Serienschaltung von Kondensatoren/Widerständen
Membranpotential bei Neuronen
 Membranpotential bei Neuronen J. Almer 1 Ludwig-Thoma-Gymnasium 9. Juli 2012 J. Almer (Ludwig-Thoma-Gymnasium ) 9. Juli 2012 1 / 17 Gliederung 1 Aufbau der Neuronmembran 2 Ruhepotential bei Neuronen Diffusion
Membranpotential bei Neuronen J. Almer 1 Ludwig-Thoma-Gymnasium 9. Juli 2012 J. Almer (Ludwig-Thoma-Gymnasium ) 9. Juli 2012 1 / 17 Gliederung 1 Aufbau der Neuronmembran 2 Ruhepotential bei Neuronen Diffusion
3.1 Zur Photochemie von HNO 3 11 Abbildung 3.1: Das UV-Absorptionsspektrum von HNO 3 nach [71] Abbildung 3.2: Das UV-Absorptionsspektrum von HNO 3 nac
![3.1 Zur Photochemie von HNO 3 11 Abbildung 3.1: Das UV-Absorptionsspektrum von HNO 3 nach [71] Abbildung 3.2: Das UV-Absorptionsspektrum von HNO 3 nac 3.1 Zur Photochemie von HNO 3 11 Abbildung 3.1: Das UV-Absorptionsspektrum von HNO 3 nach [71] Abbildung 3.2: Das UV-Absorptionsspektrum von HNO 3 nac](/thumbs/53/31354364.jpg) Kapitel 3 Grundlagen der Photochemie von HNO 3 und Modellentwicklung 3.1 Zur Photochemie von HNO 3 Salpetersaure ist ein wichtiges Nebenprodukt des photochemischen Smogs [67], und es ist ein relevanter
Kapitel 3 Grundlagen der Photochemie von HNO 3 und Modellentwicklung 3.1 Zur Photochemie von HNO 3 Salpetersaure ist ein wichtiges Nebenprodukt des photochemischen Smogs [67], und es ist ein relevanter
Kapitel I -> Moleküle des Lebens. Biophysikalische Methoden Kapitel I Moleküle des Lebens
 Kapitel I -> Moleküle des Lebens 1 Themen DNA, Polysaccharide, Lipide, Proteine Literatur V. B. Alberts, D. Bray, J. Lewis, M. Raff, K. Roberts, J. D. Watson. 1997. Molecular biology of the cell. New York:
Kapitel I -> Moleküle des Lebens 1 Themen DNA, Polysaccharide, Lipide, Proteine Literatur V. B. Alberts, D. Bray, J. Lewis, M. Raff, K. Roberts, J. D. Watson. 1997. Molecular biology of the cell. New York:
1. Seminar Biotechnologie Natalia Putkaradze, Master Biotechnologie (3. Semester) Matrikel-Nr.:
 1. Seminar Biotechnologie Natalia Putkaradze, Master Biotechnologie (3. Semester) Matrikel-Nr.: 2529735 Faktoren, die von Bedeutung für eine asymmetrische Phospholipid- Verteilung an Membranen roter Blutzellen
1. Seminar Biotechnologie Natalia Putkaradze, Master Biotechnologie (3. Semester) Matrikel-Nr.: 2529735 Faktoren, die von Bedeutung für eine asymmetrische Phospholipid- Verteilung an Membranen roter Blutzellen
-Übersicht. 2. G-Protein-gekoppelte Rezeptoren. 5. Na + -K + -Pumpe REZEPTOREN. 1. Allgemeine Definition: Rezeptoren. 3. Tyrosin-Kinase Rezeptoren
 REZEPTOREN -Übersicht 1. Allgemeine Definition: Rezeptoren 2. G-Protein-gekoppelte Rezeptoren 3. Tyrosin-Kinase Rezeptoren Beispiel: Insulin 4. Steroidhormone 5. Na + -K + -Pumpe EINFÜHRUNG Definition
REZEPTOREN -Übersicht 1. Allgemeine Definition: Rezeptoren 2. G-Protein-gekoppelte Rezeptoren 3. Tyrosin-Kinase Rezeptoren Beispiel: Insulin 4. Steroidhormone 5. Na + -K + -Pumpe EINFÜHRUNG Definition
Funktion der Sinnesrezeptoren, Aktionspotenzial.
 Funktion der Sinnesrezeptoren, Aktionspotenzial. den 17 November 2016 Dr. Emőke Bódis Prüfungsfrage Ionenkanäle. Die Funktion und Klassifizierung der Sinnesrezeptoren. Die Phasen des Aktionspotenzials.
Funktion der Sinnesrezeptoren, Aktionspotenzial. den 17 November 2016 Dr. Emőke Bódis Prüfungsfrage Ionenkanäle. Die Funktion und Klassifizierung der Sinnesrezeptoren. Die Phasen des Aktionspotenzials.
In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit
 In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit in der Nucleotidsequenz der DNA verschlüsselt (codiert)
In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit in der Nucleotidsequenz der DNA verschlüsselt (codiert)
40. Vorlesung. Hybridisierung Biologische Moleküle Photosynthese Sehvorgang Selbstorganisation Molekulare Motoren
 Prof. C. von Borczyskowski Physik für CS + SK 40. Vorlesung Hybridisierung 40.1 Biologische Moleküle 40.1.1 Photosynthese 40.1.2 Sehvorgang 40.2 Selbstorganisation 40.3 Molekulare Motoren Verwendete Literatur:
Prof. C. von Borczyskowski Physik für CS + SK 40. Vorlesung Hybridisierung 40.1 Biologische Moleküle 40.1.1 Photosynthese 40.1.2 Sehvorgang 40.2 Selbstorganisation 40.3 Molekulare Motoren Verwendete Literatur:
