Anhang 6.2.2: Gesamtliste der Microarray-Ergebnisse
|
|
|
- Frieder Martin
- vor 6 Jahren
- Abrufe
Transkript
1 Anhang 6.2.2: Gesamtliste der Microarray-Ergebnisse Experimente mit n Wiederholungen und Standardabweichung () sind oberhalb der Tabelle angegeben. Experimente mit den flia- und flgm -Mutanten wurden mit Oligonukleotid- [Oligo] und PCR-Produkt-Microarrays durchgeführt, alle anderen mit PCR-Produkt-Microarrays. Die Bezeichnung für die Lokalisation der einzelnen Gene in H. pylori (TIGR Nr.) und die englische Genbeschreibung (Gene Description) sind für die einzelnen Quotienten der Signalintensitäten des H. pylori Wildtyps (wt) zur entsprechenden Mutante angegeben Die Hoch- oder Herunterregulation von einzelnen Genen in der Mutante im Vergleich zum Wildtyp ist durch rote bzw. grüne Unterlegung der Felder hervorgenoben. N.D. - kein Wert bestimmt; HpN6 - H. pylori N6; Hp88 - H. pylori TIGR Nr. Gene Description HpN6 wt / flia (n=1) HpN6 wt / flia (n=4) [Oligo] HpN6 wt / flgm (n=1) HpN6 wt / flgm (n=4) [Oligo] HpN6 wt / rpon (n=4) Hp88 wt / rpon (n=3) HpN6 wt / flgs (n=4) HpN6 wt / flgr (n=2) HpN6 wt / flha (n=5) Hp88 wt / flha (n=3) HpN6 wt / flhf (n=4) Hp88 wt / flhf (n=2) HpN6 wt / flha/flgm (n=5) HpN6 wt / flhf /flgm (n=3) HP0001 hypothetical protein 1,2 0,0 0,9 0,1 1,0 0,0 0,8 0,1 1,0 0,3 1,3 0,3 1,1 0,2 1,0 0,1 1,1 0,1 0,8 0,3 1,3 0,3 0,9 0,2 0,8 0,2 1,4 0,8 HP0002 riboflavin synthase beta chain 0,8 N.D. 0,9 0,1 0,7 0,0 0,9 0,0 0,9 0,1 1,5 0,2 1,1 0,1 0,9 0,1 1,2 0,3 0,9 0,2 1,2 0,3 0,9 0,1 0,8 0,6 0,8 0,2 HP deoxy-d-manno-octulosonic acid 8-phosphate synthetase 1,3 0,1 0,8 0,1 1,0 0,0 0,8 0,0 0,9 0,1 1,4 0,3 1,2 0,2 0,9 0,1 1,1 0,1 1,2 0,4 1,2 0,1 1,1 0,1 0,6 0,3 0,5 0,1 HP0004 carbonic anhydrase 1,1 0,1 1,4 0,6 0,9 0,1 0,9 0,3 1,0 0,4 1,5 0,5 1,0 0,1 1,2 0,2 1,3 0,2 1,4 0,6 2,6 0,6 1,2 0,2 1,4 0,3 0,9 0,2 HP0005 orotidine 5'-phosphate decarboxylase 1,1 0,0 0,8 0,1 0,9 0,0 0,9 0,1 0,9 0,2 1,5 0,4 1,1 0,1 0,9 0,0 1,5 0,1 0,9 0,3 1,6 0,2 1,0 0,0 1,1 0,4 1,1 0,1 HP0006 pantoate-beta-alanine ligase 1,3 0,1 1,1 0,1 1,1 0,0 1,0 0,1 0,9 0,3 1,1 0,1 1,1 0,1 0,9 0,1 1,1 0,1 0,7 0,2 1,4 0,3 1,1 0,0 1,1 0,3 1,0 0,1 HP0009 outer membrane protein 1,4 0,0 N.D. N.D. 1,3 0,0 N.D. N.D. 1,0 0,2 1,0 0,1 0,9 0,3 0,8 0,0 0,7 0,1 1,0 0,2 0,8 0,1 0,9 0,1 1,0 0,3 1,0 0,0 HP0010 chaperone and heat shock protein 0,8 0,1 1,0 0,1 1,4 0,1 1,8 0,2 1,2 0,9 0,7 0,1 1,0 0,4 0,7 0,2 0,9 0,1 0,8 0,2 0,8 0,2 0,6 0,1 1,8 0,6 2,2 0,6 HP0011 co-chaperone 0,6 0,1 0,6 0,3 1,0 0,0 1,4 0,7 1,5 1,2 0,6 0,0 1,1 0,4 0,7 0,1 0,9 0,1 1,1 0,3 0,5 0,2 0,9 0,2 2,2 0,9 3,1 1,3 HP0012 DNA primase 0,6 N.D. 1,1 0,1 0,8 0,4 1,2 0,1 1,0 0,2 1,0 0,0 1,1 0,7 1,3 0,0 1,5 1,2 0,3 0,1 1,3 0,5 0,7 0,4 1,3 0,1 1,2 0,7 HP0013 hypothetical protein 0,7 0,1 0,7 0,2 0,8 0,0 0,8 0,2 0,9 0,2 1,3 0,2 1,0 0,3 1,1 0,1 1,0 0,2 0,8 0,1 1,6 0,3 1,4 0,3 1,4 0,2 1,6 0,3 HP0014 hypothetical protein 1,7 0,5 0,9 0,1 1,3 0,2 0,9 0,2 1,2 0,3 1,6 0,6 1,1 0,2 1,1 0,1 0,9 0,2 0,9 0,1 0,8 0,3 1,3 0,1 1,2 0,4 1,7 0,2 HP0015 hypothetical protein 1,3 0,2 1,0 0,2 1,3 0,0 0,9 0,1 1,4 0,4 0,9 0,2 0,9 0,1 0,9 0,1 0,9 0,2 1,6 0,2 0,7 0,1 1,5 0,1 1,3 0,1 1,9 0,3 HP0016 hypothetical protein 2,4 1,4 0,9 0,2 N.D. N.D. 0,8 0,2 1,3 0,7 0,9 0,2 0,9 0,1 1,3 0,1 0,8 0,1 1,1 0,1 1,1 0,2 1,1 0,1 0,9 0,1 1,0 0,2 HP0017 no description available N.D. N.D. 0,6 0,3 N.D. N.D. 1,0 0,4 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0018 hypothetical protein 0,8 N.D. 1,4 0,2 0,8 0,1 1,2 0,1 1,0 0,3 1,1 0,2 0,9 0,2 1,0 0,1 1,2 0,1 1,1 0,1 1,3 0,2 1,2 0,0 1,2 0,1 1,0 0,2 HP0019 CheW/CheY hybrid chemotaxis protein (chev1 ) 2,4 0,2 1,0 0,1 1,3 0,0 0,9 0,0 1,2 0,4 1,6 0,4 1,2 0,0 0,9 0,1 0,9 0,1 1,2 0,4 0,6 0,2 1,2 0,2 0,9 0,4 0,7 N.D. HP0020 carboxynorspermidine decarboxylase 1,9 0,3 0,9 0,2 1,5 0,1 0,9 0,2 1,1 0,2 1,3 0,2 1,1 0,1 1,1 0,1 1,0 0,2 0,8 0,2 1,3 0,5 1,0 0,1 1,0 0,2 0,9 0,1 HP0021 hypothetical protein 1,4 0,1 0,9 0,0 1,4 0,0 1,2 0,1 1,0 0,3 1,1 0,3 1,0 0,2 1,1 0,1 1,0 0,1 0,9 0,3 1,6 0,3 1,1 0,0 0,9 0,2 0,6 0,1 HP0022 conserved hypothetical integral membrane protein 1,4 0,0 1,0 0,2 1,2 0,0 0,9 0,2 1,1 0,2 1,0 0,2 1,0 0,1 1,4 0,2 1,0 0,1 0,7 0,1 1,9 0,2 1,0 0,0 1,3 0,3 1,2 0,3 HP0025 outer membrane protein 1,5 0,0 1,0 0,1 1,2 0,1 0,8 0,1 1,1 0,2 1,1 0,2 1,0 0,1 0,9 0,1 1,0 0,1 1,4 0,3 1,0 0,1 1,1 0,1 1,0 0,4 1,0 0,3 HP0026 citrate synthase 1,1 0,1 0,8 0,1 1,1 0,1 0,8 0,1 1,1 0,4 1,1 0,3 0,9 0,1 0,7 0,1 0,8 0,2 1,1 0,2 0,7 0,1 0,9 0,1 1,0 0,4 2,0 0,2 HP0027 isocitrate dehydrogenase 1,5 0,0 0,7 0,1 1,7 0,0 0,9 0,1 1,0 0,2 1,4 0,6 1,1 0,1 0,6 0,1 1,1 0,1 0,9 0,2 0,8 0,1 0,9 0,2 3,3 0,8 5,8 0,8 HP0028 conserved hypothetical secreted protein 1,5 0,1 0,9 0,1 2,0 0,1 1,1 0,2 1,2 0,4 1,1 0,3 0,9 0,2 0,7 0,1 1,1 0,1 0,9 0,2 0,7 0,1 1,0 0,1 2,6 0,2 4,1 0,4 HP0029 dethiobiotin synthetase 1,3 0,2 1,0 0,2 1,6 0,1 1,4 0,1 1,0 0,2 0,8 0,1 1,0 0,2 0,8 0,1 1,1 0,2 0,9 0,1 0,7 0,1 0,9 0,0 2,1 0,7 1,9 0,4 HP0030 hypothetical protein 0,6 N.D. 1,0 0,1 0,8 0,1 1,1 0,2 1,0 0,2 1,1 0,3 1,3 0,5 1,0 0,1 1,2 0,3 1,0 0,3 1,0 0,1 0,9 0,2 0,9 0,4 1,2 0,8 HP0031 hypothetical protein 1,6 0,2 0,9 0,1 1,2 0,1 0,8 0,1 1,0 0,3 1,3 0,1 1,2 0,2 0,8 0,1 0,9 0,1 1,1 0,2 0,5 0,1 1,2 0,3 0,8 0,3 1,2 0,4 HP0032 conserved hypothetical protein 1,5 0,3 0,9 0,1 1,5 N.D. 0,9 0,1 1,2 0,4 1,1 0,1 0,9 0,2 0,9 0,1 0,9 0,0 0,9 0,1 0,7 0,1 1,0 0,1 0,8 0,1 1,1 0,1 HP0033 ATP-dependent C1p protease 1,2 0,3 1,2 0,1 1,2 0,1 1,0 0,0 1,1 0,3 0,9 0,1 0,9 0,2 0,9 0,0 1,1 0,1 1,1 0,1 0,9 0,1 1,2 0,1 1,0 0,2 1,3 0,1 HP0034 aspartate 1-decarboxylase 0,8 0,2 1,0 0,1 0,9 0,0 1,1 0,3 0,9 0,1 1,0 0,1 0,8 0,2 1,1 0,1 1,0 0,1 0,9 0,1 1,3 0,2 1,2 0,2 1,1 0,1 0,8 0,1 HP0035 conserved hypothetical protein 1,4 0,2 1,0 0,1 1,5 0,0 1,0 0,0 1,0 0,3 0,9 0,2 0,7 0,3 0,9 0,0 1,0 0,2 0,9 0,1 1,3 0,4 1,3 0,1 1,3 0,4 1,2 0,3 HP0036 hypothetical protein 1,2 0,2 0,8 0,2 1,2 N.D. 1,1 0,2 1,0 0,2 0,8 0,1 0,7 0,3 1,1 0,1 0,8 0,1 0,8 0,0 1,3 0,3 1,2 0,1 1,1 0,3 0,9 0,1 HP0037 NADH-ubiquinone oxidoreductase subunit 0,9 0,3 0,9 0,1 1,3 0,1 0,8 0,1 1,1 0,4 1,2 0,1 1,0 0,2 1,2 0,1 0,9 0,1 1,0 0,2 1,3 0,2 1,2 0,0 1,1 0,4 1,2 0,2 HP0038 hypothetical protein 1,7 0,2 0,9 0,2 1,7 0,0 0,8 0,1 1,0 0,2 1,0 0,2 1,0 0,2 1,5 0,1 0,8 0,1 0,6 0,1 1,1 0,0 0,9 0,3 1,1 0,0 1,2 0,2 HP0039 hypothetical protein N.D. N.D. 0,8 0,2 N.D. N.D. 0,8 0,2 1,0 0,3 0,9 0,1 1,1 0,1 1,1 0,1 1,0 0,1 0,7 0,1 1,3 0,3 1,0 0,1 1,0 0,3 0,8 0,1 HP0040 hypothetical protein N.D. N.D. 0,8 0,2 N.D. N.D. 1,2 0,2 1,0 0,3 1,0 0,2 1,2 0,4 1,5 0,1 1,0 0,1 N.D. N.D. 1,7 0,2 9,8 N.D. 0,8 0,5 0,9 0,4 HP0041 hypothetical protein 1,2 0,2 1,2 0,3 N.D. N.D. 1,0 0,1 1,0 0,2 1,1 0,2 1,1 0,3 1,2 0,2 N.D. N.D. 0,8 0,1 1,4 0,1 1,0 N.D. 1,1 0,3 1,4 0,3 HP0042 trbi protein 1,1 N.D. 0,9 0,0 1,4 0,1 1,0 0,0 1,1 0,3 1,3 0,2 1,1 0,2 1,0 0,1 0,7 0,2 1,0 0,2 0,9 0,1 1,0 0,1 1,3 0,3 1,5 0,6 HP0043 mannose-6-phosphate isomerase 1,6 0,1 1,0 0,1 1,3 0,0 1,1 0,2 1,1 0,3 1,2 0,3 1,0 0,0 0,9 0,1 1,1 0,1 1,1 0,4 1,2 0,1 1,0 0,1 1,6 0,3 1,3 0,2 HP0044 GDP-D-mannose dehydratase 0,9 0,0 1,0 0,1 0,9 0,0 1,0 0,1 0,9 0,1 1,8 0,8 1,0 0,2 1,0 0,2 1,0 0,2 1,2 0,3 1,3 0,2 1,1 0,3 1,1 0,3 1,0 N.D. HP0045 nodulation protein 1,5 0,4 1,0 0,1 1,2 0,0 0,9 0,0 1,1 0,2 1,0 0,1 1,0 0,2 1,0 0,1 0,8 0,2 0,6 0,0 1,1 0,5 1,1 0,3 1,2 0,1 1,9 0,4 HP0046 hypothetical protein 1,1 0,1 1,1 0,1 1,3 0,1 1,1 0,1 1,1 0,2 1,1 0,1 1,0 0,1 1,0 0,1 1,0 0,2 1,4 0,2 1,1 0,1 0,9 N.D. 0,9 0,3 0,8 N.D. HP0047 hydrogenase expression/formation protein 2,1 N.D. 0,9 0,1 0,7 0,1 0,8 0,2 1,2 0,2 0,9 0,0 1,0 0,1 1,2 0,1 1,3 0,1 1,7 0,2 1,0 0,2 1,3 0,4 0,9 0,1 1,2 0,2 HP0048 transcriptional regulator 1,2 0,1 1,1 0,2 1,0 0,1 1,0 0,1 1,1 0,2 1,0 0,1 1,1 0,1 0,9 0,1 1,0 0,1 1,2 0,2 0,7 0,1 1,1 0,1 1,1 0,2 1,5 0,2 HP0049 hypothetical protein 1,1 0,3 1,0 0,1 0,9 0,1 1,0 0,2 1,1 0,3 1,2 0,1 1,0 0,1 1,0 0,1 0,8 0,1 0,8 0,1 1,0 0,2 1,1 0,1 1,2 0,3 1,7 0,3 HP0050 adenine specific DNA methyltransferase N.D. N.D. 0,9 0,1 N.D. N.D. 1,0 0,1 1,1 0,2 1,2 0,1 1,1 0,0 1,1 0,2 0,9 N.D. 1,2 N.D. 0,9 0,1 1,1 N.D. 1,1 0,1 N.D. N.D. HP0051 cytosine specific DNA methyltransferase 0,9 0,2 1,0 0,2 1,0 0,0 1,1 0,2 1,1 0,4 1,2 0,2 0,8 0,1 1,0 0,0 1,2 0,2 0,9 0,1 1,3 0,1 1,1 N.D. 1,3 0,3 1,0 0,3 HP0052 hypothetical protein N.D. N.D. 1,1 0,1 N.D. N.D. 1,1 0,2 1,3 0,4 1,0 0,2 1,2 0,2 1,3 0,2 1,3 0,7 N.D. N.D. 1,9 0,6 N.D. N.D. 1,1 0,3 N.D. N.D. HP0053 hypothetical protein N.D. N.D. 1,2 0,2 N.D. N.D. 1,1 0,1 1,1 0,3 1,2 0,1 2,5 N.D. 0,9 0,2 0,8 N.D. 1,6 0,2 0,9 0,4 1,3 N.D. 1,0 0,3 1,1 N.D. HP0054 adenine/cytosine DNA methyltransferase 1,4 N.D. 1,4 0,2 N.D. N.D. 1,1 0,1 1,1 0,2 1,4 0,3 1,0 0,3 0,9 0,1 0,7 0,1 1,3 0,1 1,0 0,1 1,0 N.D. 0,9 0,0 1,2 0,3 HP0055 proline permease 1,1 0,3 1,1 0,1 1,0 0,1 0,9 0,1 1,0 0,1 1,0 0,1 0,9 0,1 0,8 0,1 0,8 0,1 0,9 0,1 1,0 0,1 1,1 0,1 0,8 0,2 1,2 0,1 HP0056 delta-1-pyrroline-5-carboxylate dehydrogenase 1,0 0,2 1,0 0,1 0,9 0,1 0,9 0,1 0,8 0,1 1,0 0,1 1,0 0,2 0,9 0,1 1,0 0,1 1,0 0,1 1,0 0,1 1,1 0,1 1,1 0,1 1,7 0,5 HP0057 hypothetical protein 2,3 0,5 1,3 0,3 1,5 0,2 0,9 0,3 1,2 0,2 1,6 0,0 1,6 0,7 1,0 0,1 1,3 0,1 0,8 0,0 0,9 0,4 1,5 0,2 1,2 0,3 2,7 0,4 HP0058 hypothetical protein N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. 1,3 0,3 1,2 0,2 1,0 0,1 1,0 0,2 1,1 0,5 1,0 0,0 0,9 0,1 0,9 0,2 1,2 0,3 HP0059 hypothetical protein N.D. N.D. 1,1 0,3 N.D. N.D. 1,0 0,1 1,1 0,2 0,8 0,2 1,5 0,8 1,0 0,3 1,2 N.D. 1,0 N.D. 1,4 0,6 0,5 N.D. 1,3 0,3 1,7 1,0 HP0060 hypothetical protein N.D. N.D. 0,7 0,2 N.D. N.D. 1,0 0,2 1,1 0,1 N.D. N.D. 1,2 0,1 1,0 0,1 1,1 0,3 0,8 0,0 N.D. N.D. N.D. N.D. 1,1 0,1 1,0 N.D. HP0061 hypothetical protein 1,2 N.D. 1,0 0,1 1,4 0,1 1,1 0,1 1,0 0,2 1,0 0,2 1,1 0,2 1,2 0,0 0,8 0,2 1,4 0,5 1,4 0,5 0,8 0,1 1,0 0,2 1,1 0,5 HP0062 hypothetical protein 1,3 0,1 0,9 0,1 1,6 0,0 1,0 0,1 1,0 0,3 0,9 0,2 1,0 0,1 0,9 0,1 1,0 0,2 1,4 0,4 1,4 0,3 0,8 0,1 1,2 0,3 1,0 0,3 HP0063 hypothetical protein 1,1 N.D. 0,8 0,1 1,0 0,1 0,8 0,1 0,9 0,2 1,1 0,4 1,1 0,1 1,0 0,1 1,1 0,2 1,6 0,5 1,3 0,4 0,9 0,1 1,4 0,1 1,4 0,4 HP0064 hypothetical protein 1,7 0,2 0,9 0,2 1,4 0,0 1,0 0,1 0,9 0,2 1,1 0,3 1,0 0,1 0,7 0,1 1,2 0,1 1,0 0,3 0,9 0,1 1,0 0,1 2,3 0,2 2,2 0,6 HP0065 hypothetical protein 1,1 0,1 0,8 0,3 1,2 0,0 0,9 0,2 1,0 0,2 1,2 0,2 1,1 0,1 0,9 0,1 1,1 0,1 1,0 0,1 1,0 0,3 0,9 0,1 1,4 0,1 1,3 0,2 HP0066 no description available 0,9 0,1 0,9 0,1 0,8 0,0 1,2 0,3 1,0 0,1 0,9 0,2 1,0 0,1 0,8 0,0 1,1 0,1 0,8 0,1 0,8 0,0 0,8 0,0 1,6 0,2 1,7 0,1 HP0067 urease accessory protein 0,7 0,2 1,4 0,2 0,7 0,1 1,2 0,2 1,0 0,2 1,1 0,1 0,8 0,1 0,8 0,0 1,1 0,2 0,9 0,3 1,1 0,4 1,2 0,1 1,1 0,2 1,6 0,2 HP0068 urease accessory protein 1,0 0,2 1,2 0,2 0,8 0,0 1,2 0,1 1,0 0,1 1,2 0,2 0,9 0,1 0,8 0,1 1,5 0,3 1,3 0,0 1,2 0,3 1,2 0,2 1,1 0,2 1,4 0,4 HP0069 urease accessory protein 0,9 0,2 1,1 0,5 0,9 0,1 1,3 0,2 1,0 0,2 0,9 0,1 0,8 0,1 1,1 0,1 1,7 0,3 1,0 0,1 N.D. N.D. 1,1 0,1 0,8 0,1 0,7 0,1 HP0070 urease accessory protein 1,3 0,2 1,0 0,2 1,2 0,1 1,0 0,2 1,3 0,4 1,1 0,1 0,9 0,1 0,7 0,1 1,4 0,2 0,9 0,2 1,0 0,2 1,0 0,0 1,8 0,3 3,2 0,4 HP0071 urease accessory protein 0,8 0,2 1,1 0,3 0,9 0,1 1,1 0,4 0,9 0,2 1,1 0,0 0,9 0,1 0,6 0,1 1,6 0,1 1,4 0,2 0,9 0,2 1,3 0,0 1,5 0,2 2,6 0,3 HP0072 urease beta subunit (urea amidohydrolase) 0,6 0,1 0,9 0,3 0,7 0,0 1,3 0,2 1,1 0,4 1,4 0,0 1,2 0,2 0,5 0,1 1,0 0,3 1,2 0,2 1,0 0,3 1,2 0,2 1,8 0,6 3,3 0,8 HP0073 urease, alpha subunit 0,4 0,0 0,8 0,4 0,5 0,1 1,5 0,6 1,0 0,3 1,4 0,0 1,2 0,2 0,6 0,1 1,2 0,2 1,3 0,3 1,0 0,2 1,2 0,3 1,9 0,5 3,2 1,0 HP0074 lipoprotein signal peptidase 0,9 0,1 1,0 0,1 1,0 0,0 1,0 0,1 0,9 0,3 1,2 0,2 1,0 0,2 1,0 0,0 1,0 0,1 0,9 0,2 1,3 0,2 1,0 0,0 1,0 0,2 1,0 0,2 HP0075 urease protein 0,9 0,1 0,9 0,1 1,2 N.D. 1,0 0,0 1,0 0,2 1,2 0,2 1,0 0,2 0,9 0,0 0,9 0,1 0,8 0,2 1,6 0,5 0,9 0,0 0,8 0,5 0,8 0,1 HP0076 ribosomal protein S20 N.D. N.D. 0,9 0,2 1,4 0,0 0,9 0,1 1,2 0,3 0,7 0,2 1,1 0,3 1,3 0,1 1,7 0,4 1,2 0,3 1,5 0,1 1,1 0,1 0,7 0,4 0,4 0,0 HP0077 peptide chain release factor RF-1 1,1 0,2 1,1 0,1 1,2 0,1 1,0 0,1 1,2 0,2 0,7 0,2 0,7 0,1 1,6 0,2 1,2 0,2 1,4 0,3 1,6 0,6 0,6 0,1 0,5 0,3 0,2 0,0 HP0078 hypothetical protein 0,8 0,0 0,8 0,1 0,6 0,0 1,0 0,1 0,7 0,1 1,5 0,2 1,4 0,1 1,1 0,0 1,2 0,2 1,2 0,0 8,3 9,8 0,9 N.D. 1,4 0,4 1,6 1,0 HP0079 outer membrane protein 0,9 0,2 1,1 0,1 0,7 0,1 1,1 0,1 0,8 0,1 1,5 0,3 1,3 0,1 1,0 0,1 1,1 0,1 1,2 0,3 N.D. N.D. 0,9 0,0 1,5 0,1 1,5 0,7 HP0080 hypothetical protein 1,3 0,1 0,9 0,2 1,2 0,1 1,1 0,2 1,0 0,2 1,6 0,5 1,2 0,1 1,0 0,1 1,2 0,2 1,0 0,1 1,4 0,1 1,1 0,2 1,2 0,1 1,8 0,3 HP0081 no description available N.D. N.D. 0,7 0,1 N.D. N.D. 0,9 0,1 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0082 receptor protein of chemotaxis signalling system (tlpc ) 0,9 0,1 0,8 0,1 1,0 0,0 0,9 0,1 1,0 0,1 1,0 0,0 1,0 0,1 0,9 0,0 1,1 0,1 1,0 0,1 0,9 0,1 0,9 0,0 1,2 0,2 1,0 0,2 HP0083 ribosomal protein S9 0,7 0,2 0,9 0,2 1,0 0,0 1,1 0,2 1,1 0,2 1,1 0,2 0,9 0,1 0,8 0,1 0,8 0,3 1,0 0,1 0,9 0,4 1,1 0,1 0,4 0,1 0,9 0,1 HP0084 ribosomal protein L13 0,5 0,2 1,0 0,2 0,8 0,1 1,1 0,2 1,0 0,3 1,1 0,0 0,8 0,1 0,8 0,1 1,0 0,2 1,0 0,1 1,2 0,7 1,1 0,0 0,4 0,1 0,3 N.D. HP0085 hypothetical protein 0,9 0,2 1,0 0,1 0,8 0,0 0,9 0,3 1,1 0,3 1,1 0,3 1,0 0,1 0,9 0,1 1,3 0,3 1,1 0,1 1,1 0,3 1,2 0,1 1,1 0,1 1,5 0,1 HP0086 conserved hypothetical protein 0,7 0,2 1,1 0,1 0,7 0,1 1,1 0,1 0,9 0,2 1,0 0,1 1,0 0,1 1,1 0,1 1,1 0,2 0,9 0,1 1,4 0,3 1,0 0,0 0,9 0,1 1,0 0,1 HP0087 hypothetical protein 1,0 0,2 1,0 0,1 1,0 0,1 1,0 0,1 1,1 0,3 0,8 0,1 1,0 0,3 1,1 0,1 1,0 0,1 0,9 0,2 1,1 0,1 0,9 0,0 0,9 0,2 1,1 0,3 HP0088 RNA polymerase sigma-70 factor 0,8 0,1 0,7 0,1 0,9 0,1 0,9 0,1 1,0 0,4 0,8 0,2 1,0 0,2 0,8 0,1 1,0 0,1 0,8 0,1 0,8 0,1 1,0 0,1 1,2 0,3 1,5 0,4 HP0089 pfs protein 0,6 0,1 1,0 0,2 0,8 0,0 0,9 0,2 0,9 0,2 1,1 0,1 1,0 0,2 0,7 0,1 0,8 0,1 N.D. N.D. 0,9 0,1 1,4 0,2 0,8 0,2 0,6 0,2 HP0090 malonyl coenzyme A-acyl carrier protein transacylase 0,6 0,1 0,9 0,1 0,9 0,1 0,9 0,2 1,1 0,2 1,0 0,1 0,9 0,2 0,8 0,1 0,7 0,1 0,8 0,0 1,0 0,1 1,3 0,0 1,5 0,3 1,8 0,3 HP0091 type II restriction enzyme R protein N.D. N.D. 0,9 0,1 N.D. N.D. 0,9 0,1 1,0 0,2 0,6 0,1 1,2 0,1 0,8 0,0 1,1 0,2 1,5 0,6 N.D. N.D. 1,1 0,2 1,3 0,3 1,0 0,1 HP0092 type II restriction enzyme M protein 0,7 0,0 0,9 0,1 0,9 0,0 1,0 0,1 0,8 0,3 0,5 0,1 1,1 0,1 1,1 0,1 0,7 0,1 1,1 0,5 1,7 0,0 0,7 0,1 1,2 0,7 0,7 0,1 HP0093 hypothetical protein 1,7 N.D. 0,9 0,1 1,3 N.D. 1,0 0,1 1,2 0,3 0,8 0,3 1,2 0,3 1,3 0,0 1,2 0,1 0,8 0,1 1,5 0,1 1,1 N.D. 1,0 0,3 1,0 0,4 HP0094 conserved hypothetical protein 0,8 0,0 1,3 0,2 0,8 0,1 1,2 0,1 1,0 0,2 1,3 0,2 1,0 0,2 1,2 0,1 1,5 0,3 1,0 0,2 1,9 0,6 0,8 0,1 1,2 0,4 0,8 0,2 HP0095 hypothetical protein 1,3 0,0 0,9 0,1 1,0 0,0 1,0 0,2 1,1 0,3 1,4 0,5 1,0 0,2 1,1 0,0 1,3 0,2 1,5 0,1 1,4 0,3 1,3 0,2 1,1 0,1 0,9 0,2 HP0096 phosphoglycerate dehydrogenase 1,0 0,1 0,9 0,0 1,0 0,1 1,0 0,1 1,0 0,1 1,6 0,6 1,1 0,1 0,9 0,1 0,9 0,2 1,2 0,2 1,0 0,2 1,1 0,3 0,8 0,2 0,8 0,1 HP0097 hypothetical protein 1,2 0,0 0,8 0,2 1,4 0,0 0,9 0,1 1,1 0,2 1,2 0,3 0,9 0,1 0,7 0,0 0,6 0,2 0,8 0,2 0,7 0,1 1,1 0,2 0,7 0,2 0,7 0,1 HP0098 threonine synthase 1,4 0,1 1,0 0,1 1,8 0,1 1,1 0,1 1,0 0,2 1,4 0,3 1,1 0,1 1,0 0,1 1,0 0,1 1,2 0,2 1,2 0,2 0,8 0,2 0,9 0,4 1,2 0,1 HP0099 Methyl accepting chemotaxis protein (tlpa) 1,0 0,1 0,9 0,0 1,0 0,0 1,0 0,1 1,1 0,2 0,8 0,2 1,0 0,1 0,9 0,0 1,1 0,1 1,7 0,1 0,9 0,3 0,9 0,0 0,7 0,3 1,1 0,1 HP0100 conserved hypothetical protein 0,8 0,1 0,9 0,1 1,1 0,2 1,1 0,2 1,2 0,2 0,8 0,1 1,0 0,2 1,3 0,1 1,1 0,1 2,1 1,5 1,4 0,2 1,0 0,1 0,7 0,3 0,5 0,1 HP0101 hypothetical protein 0,8 0,2 1,0 0,0 0,8 0,0 1,0 0,1 1,2 0,3 0,9 0,2 1,0 0,1 1,0 0,1 1,1 0,1 1,0 0,1 1,0 0,3 1,2 0,2 1,1 0,0 1,9 N.D. HP0102 conserved hypothetical protein 0,7 0,2 1,3 0,2 0,7 0,1 1,4 0,3 1,1 0,4 0,9 0,1 1,0 0,1 1,6 0,2 0,7 0,2 0,9 0,1 1,4 0,6 1,1 0,1 0,8 0,3 0,7 0,2 HP0103 chemotaxis receptor (tlpb ) 0,6 0,1 1,0 0,2 0,9 0,0 1,1 0,2 1,2 0,3 0,9 0,1 0,8 0,2 1,0 0,2 0,8 0,1 1,1 0,1 1,0 0,2 1,1 0,2 0,8 0,2 0,4 0,1 HP0104 2',3'-cyclic-nucleotide 2'-phosphodiesterase 0,8 0,1 0,9 0,1 0,6 0,1 1,1 0,1 0,9 0,2 1,0 0,2 0,9 0,1 0,9 0,1 1,4 0,2 1,0 0,1 1,2 0,3 1,2 0,2 1,4 0,2 1,5 0,2 HP0105 conserved hypothetical protein 1,1 0,2 1,2 0,2 0,6 0,1 1,1 0,1 1,0 0,2 1,2 0,1 1,0 0,1 0,8 0,1 1,6 0,2 N.D. N.D. 1,3 0,3 1,2 0,1 1,3 0,3 1,5 0,1 HP0106 cystathionine gamma-synthase 0,8 0,1 1,5 0,5 0,4 0,1 1,3 0,1 0,8 0,2 1,3 0,0 0,9 0,2 0,8 0,1 1,6 0,1 1,1 0,1 1,9 0,3 1,3 0,0 1,9 0,4 2,9 0,2 HP0107 cysteine synthetase 1,0 0,0 0,9 0,1 0,5 0,0 0,9 0,2 0,9 0,3 2,0 0,7 1,0 0,2 0,8 0,1 1,8 0,8 0,9 0,1 N.D. N.D. 1,1 0,2 1,2 0,3 2,0 0,3 HP0108 hypothetical protein 1,6 0,2 1,0 0,3 1,5 0,1 1,1 0,1 0,9 0,3 1,2 0,3 1,1 0,1 0,9 0,1 1,1 0,3 1,3 0,3 1,0 0,3 1,0 0,2 1,3 0,1 1,5 0,0 HP0109 chaperone and heat shock protein 70 1,1 0,1 0,8 0,3 2,3 0,1 1,2 0,2 1,6 1,3 0,6 0,2 1,0 0,5 0,9 0,0 1,0 0,0 0,8 0,2 0,6 0,1 0,8 0,1 1,1 0,4 1,2 0,3 HP0110 co-chaperone and heat shock protein 0,7 0,1 0,6 0,3 1,5 0,1 1,1 0,4 2,4 2,2 1,1 0,3 0,8 0,2 1,0 0,1 0,7 0,2 1,1 0,1 0,4 0,2 1,2 0,4 1,3 0,6 1,5 0,4 HP0111 hypothetical protein 1,1 0,0 0,5 0,2 1,8 0,0 0,8 0,3 3,6 3,5 1,7 0,6 1,2 0,4 0,9 0,0 0,5 0,1 1,1 0,1 0,3 0,1 1,6 0,3 1,3 0,7 2,4 0,4 HP0112 hypothetical protein 1,5 0,2 0,8 0,2 1,4 0,1 1,0 0,0 1,0 0,2 1,4 0,4 0,9 0,1 1,3 0,1 1,0 0,1 1,1 0,2 1,5 0,3 1,0 0,2 1,0 0,0 0,6 0,1 HP0113 hypothetical protein 1,0 0,1 0,9 0,2 0,9 0,1 0,9 0,2 1,0 0,1 1,2 0,1 0,9 0,1 1,1 0,0 1,3 0,1 0,9 0,2 0,8 0,1 0,7 N.D. 1,4 0,2 2,0 0,2 HP0114 Hypothetical protein (operon with flab?) 1,2 0,0 0,9 0,1 0,8 0,1 0,9 0,0 2,9 1,4 2,4 0,4 2,2 0,4 1,1 0,0 1,5 0,4 1,5 0,1 1,4 0,3 1,2 0,4 0,6 0,3 2,9 0,2 HP0115 Flagellin B (flab) 0,9 0,2 0,9 0,1 0,7 0,1 0,9 0,1 3,4 0,5 5,5 2,7 5,6 2,3 2,6 0,4 3,4 1,6 2,5 0,3 2,8 0,8 2,4 0,2 0,2 0,1 9,4 3,7 HP0116 DNA topoisomerase I 0,8 0,2 0,9 0,1 0,8 0,1 0,8 0,1 1,1 0,2 1,0 0,1 1,1 0,1 0,8 0,1 1,6 0,8 0,9 0,1 1,1 0,1 1,3 0,0 1,4 0,2 1,1 0,0 HP0117 conserved hypothetical protein 1,7 0,4 0,9 0,3 0,6 0,0 1,2 0,3 1,0 0,2 0,9 0,0 0,7 0,2 1,1 0,1 1,4 0,2 1,7 0,1 1,7 0,2 1,5 0,0 0,8 0,2 0,7 0,2 HP0118 hypothetical protein 0,7 0,2 0,9 0,1 0,7 0,1 1,0 0,3 1,0 0,4 1,1 0,3 1,0 0,2 1,0 0,2 0,9 0,3 0,8 0,2 0,9 0,2 1,1 0,2 1,2 0,4 1,3 0,7 HP0119 no description available N.D. N.D. 1,0 0,1 N.D. N.D. 1,0 0,0 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0120 hypothetical protein 1,4 N.D. 0,9 0,2 1,2 0,1 0,9 0,2 0,9 0,2 1,1 0,0 1,1 0,2 0,8 0,2 0,9 0,2 0,9 0,1 0,7 0,0 1,0 0,3 1,5 0,6 2,1 0,4 HP0121 phosphoenolpyruvate synthase 0,9 0,2 1,0 0,1 0,9 0,1 0,9 0,1 0,9 0,2 1,0 0,1 0,9 0,1 0,8 0,1 0,9 0,1 1,0 0,1 1,0 0,1 1,0 0,1 1,0 0,1 1,1 0,3 HP0122 hypothetical protein 1,2 0,2 1,1 0,1 1,1 0,1 1,2 0,1 0,9 0,3 0,9 0,0 1,0 0,0 1,1 0,1 1,2 0,1 0,9 0,2 1,1 0,1 0,9 N.D. 1,2 0,3 1,4 0,5 HP0123 threonyl-trna synthetase 0,6 0,1 1,4 0,2 1,0 0,0 1,6 0,4 1,0 0,2 0,9 0,1 0,8 0,1 0,9 0,1 1,1 0,2 1,2 0,2 1,9 1,0 1,0 0,2 1,1 0,1 0,6 0,1
2 HP0124 translation initiation factor IF-3 0,4 N.D. 1,2 0,1 0,7 0,1 1,1 0,1 1,0 0,4 1,1 0,1 0,8 0,2 1,0 0,2 1,0 0,2 0,9 0,3 1,7 0,8 1,5 0,2 0,8 0,1 0,4 0,1 HP0125 ribosomal protein L35 1,3 0,1 1,1 0,3 1,9 0,0 1,1 0,1 1,0 0,3 1,4 0,3 1,0 0,1 0,8 0,0 1,2 0,4 1,2 0,2 0,9 0,1 1,3 0,2 0,8 0,4 1,1 0,7 HP0126 ribosomal protein L20 1,4 0,2 1,3 0,4 1,4 0,1 1,2 0,2 0,9 0,3 1,3 0,2 0,9 0,1 0,8 0,1 1,2 0,1 1,4 0,1 1,0 0,1 1,4 0,2 0,6 0,3 0,5 0,0 HP0127 outer membrane protein 1,4 0,1 1,0 0,2 1,6 0,0 0,9 0,2 1,1 0,2 1,1 0,3 0,9 0,2 0,8 0,0 1,1 0,2 1,2 0,2 0,9 0,1 1,2 0,0 0,2 0,1 0,2 0,0 HP0128 hypothetical protein N.D. N.D. 1,1 0,3 N.D. N.D. 1,0 0,1 N.D. N.D. 0,7 0,1 0,9 N.D. 0,9 0,1 1,0 0,2 1,2 0,0 0,9 0,0 1,0 N.D. 1,0 0,0 0,7 N.D. HP0129 hypothetical protein 1,1 0,2 0,9 0,1 1,4 0,2 1,0 0,1 0,8 0,1 1,2 0,2 1,2 0,3 1,0 0,1 1,1 0,4 0,8 0,3 0,8 0,3 1,0 0,2 1,3 0,2 1,3 0,3 HP0130 hypothetical protein 1,7 0,1 0,9 0,1 1,3 0,0 0,9 0,1 1,0 0,1 1,4 0,6 0,9 0,3 1,1 0,0 1,1 0,3 1,1 0,1 0,9 0,3 1,1 0,2 0,8 0,1 0,7 0,2 HP0132 L-serine deaminase 1,5 0,1 0,7 0,3 1,5 0,2 1,0 0,2 1,1 0,2 1,6 0,6 1,1 0,2 1,1 0,1 0,7 0,2 1,4 0,3 1,1 0,1 1,3 0,1 1,0 0,4 0,8 0,1 HP0133 serine transporter 1,5 0,1 0,8 0,1 1,6 0,0 0,9 0,1 1,3 0,3 1,3 0,4 1,1 0,1 1,0 0,0 0,6 0,2 1,0 0,2 0,8 0,1 1,3 0,2 0,8 0,2 1,0 0,1 HP deoxy-D-arabino-heptulosonate 7-phosphate synthase 1,0 0,1 0,7 0,3 0,9 0,0 0,8 0,3 1,0 0,2 1,3 0,1 1,1 0,1 0,9 0,1 1,1 0,1 1,1 0,2 1,2 0,1 0,9 0,1 1,1 0,0 1,9 1,0 HP0135 hypothetical protein 0,8 0,2 0,9 0,1 0,6 0,0 0,9 0,0 1,1 0,1 1,0 0,2 1,5 0,4 1,3 0,1 1,8 0,1 1,2 0,2 1,9 0,4 1,1 0,1 0,9 0,1 1,1 0,2 HP0136 bacterioferritin comigratory protein 0,9 0,2 1,0 0,2 0,8 0,1 1,1 0,2 1,1 0,2 1,2 0,2 0,9 0,2 0,9 0,1 1,1 0,1 1,1 0,1 0,9 0,1 1,5 0,0 1,3 0,1 1,4 0,2 HP0137 hypothetical protein 1,1 0,2 0,9 0,1 1,0 0,1 0,9 0,1 1,0 0,2 1,0 0,1 1,0 0,2 0,9 0,1 1,0 0,1 1,1 0,1 1,0 0,2 1,2 0,0 1,0 0,1 1,2 0,4 HP0138 conserved hypothetical iron-sulfur protein 0,7 0,1 1,0 0,2 0,8 0,1 1,1 0,0 0,9 0,2 1,1 0,3 1,1 0,1 0,8 0,2 0,9 0,1 0,9 0,0 1,2 0,2 1,1 0,0 0,9 0,2 1,3 0,3 HP0139 conserved hypothetical secreted protein 0,6 0,1 0,9 0,1 0,7 0,0 1,0 0,1 0,9 0,2 1,0 0,2 1,1 0,2 0,8 0,1 1,3 0,2 1,2 0,2 1,5 0,5 0,8 0,1 1,2 0,1 1,5 0,5 HP0140 L-lactate permease 0,6 0,1 0,9 0,1 1,0 0,1 1,1 0,2 1,3 0,2 0,9 0,2 0,8 0,1 0,8 0,1 0,8 0,4 1,1 0,2 1,1 0,6 1,5 0,0 1,1 0,3 2,2 0,2 HP0141 L-lactate permease 0,6 0,1 0,9 0,2 1,0 0,1 0,9 0,1 1,2 0,3 1,0 0,1 0,7 0,1 0,8 0,1 0,8 0,4 0,8 0,0 1,6 0,9 1,2 0,0 0,9 0,1 1,0 0,2 HP0142 A/G-specific adenine glycosylase 0,6 N.D. 1,1 0,3 0,9 0,1 0,9 0,2 1,1 0,2 1,0 0,2 1,0 0,1 1,2 0,0 0,7 0,1 1,2 0,2 1,0 0,0 1,5 N.D. 1,0 0,2 0,9 0,3 HP oxoglutarate/malate translocator, authentic frameshift 1,7 0,1 0,9 0,2 1,5 0,1 0,8 0,1 1,1 0,3 1,2 0,2 1,0 0,1 0,9 0,0 0,8 0,2 1,1 0,0 1,0 0,1 1,3 0,2 0,9 0,2 1,5 0,9 HP0144 cytochrome c oxidase, heme b and copper-binding subunit, membrane-bound 1,3 0,2 0,8 0,1 1,6 0,2 1,0 0,1 0,9 0,2 1,1 0,1 1,1 0,1 0,8 0,0 0,9 0,1 1,0 0,1 0,8 0,1 1,2 0,0 1,0 0,9 1,4 0,1 HP0145 cytochrome c oxidase, monoheme subunit, membrane-bound 1,4 0,1 0,8 0,2 1,6 0,0 1,1 0,1 1,2 0,5 1,3 0,2 1,0 0,1 0,8 0,0 0,9 0,1 1,1 0,2 0,6 0,0 1,3 0,2 0,7 0,2 1,0 0,1 HP0146 cbb3-type cytochrome c oxidase subunit Q 1,4 0,2 0,8 0,2 1,8 0,2 1,1 0,1 1,2 0,6 1,2 0,0 1,0 0,1 1,0 0,0 1,0 0,2 1,0 0,1 0,7 0,2 1,2 0,3 1,1 0,3 1,2 0,2 HP0147 cytochrome c oxidase, diheme subunit, membrane-bound 1,2 0,1 0,8 0,1 1,5 0,0 1,1 0,1 1,2 0,5 1,6 0,5 1,1 0,2 0,8 0,1 0,9 0,1 1,3 0,1 0,5 0,1 1,5 0,3 0,9 0,2 1,1 0,1 HP0148 hypothetical protein 1,5 0,2 0,8 0,1 1,5 0,1 1,3 0,2 1,1 0,4 1,5 0,1 1,1 0,2 0,9 0,0 1,0 0,3 1,2 0,2 0,6 0,2 1,4 0,5 1,1 0,2 1,1 0,3 HP0149 hypothetical protein N.D. N.D. 1,1 0,2 1,6 0,0 1,0 0,1 1,4 0,5 1,4 0,4 1,1 0,1 0,9 0,0 0,8 0,1 1,4 0,4 0,6 0,1 1,3 0,1 0,9 0,2 0,9 0,2 HP0150 hypothetical protein 1,3 0,1 1,1 0,1 1,4 0,1 1,0 0,1 1,2 0,3 1,2 0,2 1,1 0,2 1,1 0,0 0,9 0,1 1,3 0,1 0,8 0,1 1,1 0,1 0,9 0,2 1,0 0,3 HP0151 conserved hypothetical membrane protein 1,1 0,2 1,1 0,2 1,4 0,1 1,2 0,2 1,0 0,2 0,9 0,0 0,8 0,2 1,0 0,1 0,8 0,1 0,8 0,1 1,4 0,3 1,1 0,0 1,5 0,3 1,1 0,1 HP0152 hypothetical protein 0,8 0,1 1,2 0,2 0,8 0,1 1,0 0,1 1,1 0,2 1,1 0,1 0,9 0,2 1,0 0,1 0,9 0,2 0,9 0,2 1,1 0,2 1,3 0,1 1,4 0,1 1,3 0,1 HP0153 recombinase 0,7 0,1 0,9 0,1 0,7 0,0 1,0 0,1 0,9 0,2 1,1 0,1 1,1 0,2 0,9 0,1 1,1 0,1 1,0 0,1 1,2 0,2 1,1 0,2 1,0 0,1 0,9 0,3 HP0154 enolase 0,7 0,1 0,9 0,1 0,7 0,1 0,8 0,1 0,9 0,3 1,1 0,1 1,0 0,2 0,8 0,2 0,8 0,1 0,8 0,1 1,0 0,2 1,2 0,1 1,0 0,1 1,0 0,3 HP0155 hypothetical protein 0,7 0,1 1,0 0,2 0,7 0,0 1,0 0,0 1,0 0,2 1,1 0,1 1,0 0,2 0,8 0,1 0,9 0,0 1,1 0,1 1,0 0,1 1,0 0,1 1,3 0,1 1,2 0,3 HP0156 hypothetical protein 0,5 N.D. 1,3 0,1 0,6 0,1 1,0 0,2 1,0 0,4 1,1 0,1 0,9 0,2 0,9 0,1 0,8 0,1 0,8 0,1 1,1 0,2 1,2 0,0 0,9 0,2 0,9 0,2 HP0157 shikimic acid kinase I 0,6 0,1 1,2 0,1 0,7 0,0 1,2 0,2 1,1 0,4 0,9 0,1 0,9 0,1 1,0 0,1 0,8 0,1 1,0 0,0 1,4 0,6 1,2 0,1 1,0 0,3 1,3 0,9 HP0158 hypothetical protein 0,5 0,0 1,1 0,2 0,7 0,1 1,1 0,1 1,1 0,3 1,0 0,1 1,1 0,1 1,1 0,1 0,8 0,2 1,0 0,1 1,8 1,0 1,2 0,1 0,9 0,3 0,8 0,0 HP0159 lipopolysaccharide 1,2-glucosyltransferase 1,5 0,1 0,7 0,2 1,2 0,1 1,1 0,1 1,0 0,1 0,9 0,1 1,0 0,1 1,0 0,0 1,0 0,1 0,9 0,1 1,0 0,1 1,2 0,1 1,2 0,2 1,1 0,1 HP0160 conserved hypothetical secreted protein 0,9 0,1 1,0 0,2 1,0 0,1 1,0 0,1 0,8 0,1 1,5 0,5 0,9 0,3 0,9 0,1 1,1 0,3 1,1 0,1 1,3 0,2 0,9 0,1 1,1 0,1 1,0 0,2 HP0162 conserved hypothetical protein 1,1 0,1 1,5 0,3 0,8 0,0 1,0 0,0 0,9 0,2 1,4 0,4 1,0 0,4 1,3 0,1 1,7 0,3 1,1 0,3 1,5 0,2 1,5 0,3 0,9 0,4 0,9 0,1 HP0163 delta-aminolevulinic acid dehydratase 1,0 0,1 1,4 0,5 0,7 0,1 1,2 0,1 0,9 0,2 1,3 0,2 1,1 0,2 1,3 0,0 1,6 0,2 0,7 0,3 1,8 0,4 1,2 0,4 1,1 0,1 0,9 0,2 HP0164 signal-transducing protein, histidine kinase 1,1 0,2 1,4 0,2 1,1 0,1 1,3 0,2 0,9 0,2 1,3 0,3 1,0 0,1 1,2 0,1 1,5 0,4 1,7 0,3 1,3 0,4 1,2 0,1 1,4 0,2 1,2 0,2 HP0165 Histidine kinase 1,0 0,1 1,4 0,3 0,9 0,1 1,4 0,2 0,8 0,2 1,3 0,3 1,0 0,2 1,1 0,0 2,6 0,6 2,1 0,3 1,3 0,1 1,2 0,2 1,6 0,3 1,8 0,6 HP0166 Response regulator 0,8 0,0 1,3 0,3 0,9 0,0 1,3 0,1 0,9 0,2 1,0 0,2 1,1 0,3 1,0 0,0 2,9 0,3 1,5 0,2 1,6 0,2 1,1 0,1 1,4 0,5 1,9 0,9 HP0167 hypothetical protein 0,9 N.D. 1,0 0,1 0,9 0,1 1,1 0,1 1,1 0,2 1,1 0,2 1,0 0,1 1,3 0,1 1,2 0,4 0,8 0,2 1,3 0,0 0,9 0,1 1,2 0,1 1,4 0,3 HP0168 hypothetical protein 0,8 0,1 0,8 0,2 1,0 0,0 1,1 0,2 1,0 0,3 0,9 0,1 1,0 0,1 1,1 0,1 0,9 0,1 0,7 0,0 1,5 0,4 1,0 0,1 1,3 0,3 1,1 0,1 HP0169 collagenase 0,5 0,1 1,2 0,1 0,9 0,1 1,1 0,2 1,0 0,2 0,9 0,0 0,9 0,2 0,9 0,1 0,8 0,1 0,9 0,1 1,2 0,2 1,2 0,0 1,7 0,2 1,9 0,4 HP0170 hypothetical protein 0,7 0,1 0,9 0,1 0,9 0,0 1,0 0,0 1,0 0,3 0,8 0,1 1,0 0,2 0,9 0,1 0,8 0,2 0,7 0,1 0,9 0,2 0,8 0,1 2,1 0,3 2,5 0,9 HP0171 peptide chain release factor RF-2 0,6 N.D. 0,9 0,1 0,8 0,1 0,8 0,0 0,9 0,3 0,9 0,0 0,9 0,2 0,9 0,1 0,8 0,1 0,9 0,1 1,3 0,2 1,1 0,0 1,1 0,2 1,1 0,2 HP0172 molybdopterin biosynthesis protein 1,0 0,2 1,3 0,6 0,9 0,1 1,3 0,1 1,0 0,2 1,0 0,2 1,1 0,1 1,1 0,1 0,9 0,1 0,8 0,1 1,1 0,1 1,1 0,0 1,0 0,2 1,6 0,4 HP0173 flagellar biogenesis regulatory protein (flir ) 0,6 0,1 1,1 0,2 0,9 0,1 1,0 0,1 1,0 0,2 0,9 0,0 1,0 0,1 0,9 0,1 0,9 0,1 0,8 0,1 0,9 0,1 1,4 0,2 1,1 0,0 1,1 0,1 HP0174 hypothetical protein 0,7 0,2 1,0 0,1 0,8 0,0 1,4 0,1 1,1 0,2 0,8 0,1 1,1 0,1 1,2 0,1 1,2 0,1 1,1 0,1 1,2 0,1 1,0 0,1 1,3 0,5 N.D. N.D. HP0175 cell binding factor 2 0,7 0,1 0,9 0,1 1,0 0,1 1,0 0,1 1,0 0,2 0,9 0,0 1,0 0,2 0,8 0,1 1,2 0,1 1,1 0,1 0,9 0,1 1,3 0,1 0,8 0,2 1,2 0,3 HP0176 fructose-bisphosphate aldolase 1,2 0,1 0,9 0,1 1,1 0,0 1,1 0,1 1,0 0,1 1,2 0,2 1,0 0,2 0,7 0,1 1,2 0,2 0,9 0,1 0,9 0,1 1,3 0,1 0,8 0,1 3,7 3,3 HP0177 translation elongation factor EF-P 1,3 0,2 0,9 0,1 1,1 0,1 0,9 0,1 1,2 0,3 1,8 0,6 1,0 0,2 0,9 0,1 1,2 0,1 1,3 0,1 0,8 0,2 1,6 0,5 1,0 0,2 1,2 0,2 HP0178 spore coat polysaccharide biosynthesis protein E 1,0 0,1 1,1 0,2 1,1 0,0 1,0 0,1 1,0 0,2 1,1 0,2 0,9 0,2 1,0 0,0 1,0 0,1 1,0 0,1 1,1 0,1 1,3 0,0 0,7 0,3 0,9 0,2 HP0179 ABC transporter, ATP-binding protein 1,0 0,0 1,2 0,1 1,1 0,1 0,9 0,2 1,0 0,2 1,3 0,4 1,1 0,3 1,3 0,1 0,9 0,1 0,7 0,1 1,5 0,4 1,3 0,2 0,5 0,1 0,6 0,1 HP0180 apolipoprotein N-acyltransferase 1,4 0,1 1,1 0,2 1,3 0,1 1,0 0,1 1,2 0,2 0,9 0,1 1,0 0,1 1,3 0,1 1,0 0,2 0,9 0,1 1,3 0,0 1,2 0,1 1,2 0,2 1,5 0,5 HP0181 hypothetical protein 1,2 0,1 1,2 0,1 1,0 0,0 1,1 0,2 1,0 0,1 1,3 0,5 0,8 0,3 1,0 0,1 0,9 0,1 0,7 0,0 1,7 0,3 0,9 0,2 1,0 0,2 0,8 0,3 HP0182 lysyl-trna synthetase 1,4 0,1 1,0 0,2 0,8 0,0 1,1 0,1 1,0 0,1 1,0 0,1 0,9 0,2 1,1 0,1 1,0 0,1 0,9 0,1 1,4 0,1 1,1 0,1 0,8 0,2 0,8 0,1 HP0183 serine hydroxymethyltransferase 1,2 0,2 1,1 0,1 1,3 0,1 1,0 0,1 1,0 0,2 1,2 0,1 1,0 0,1 1,0 0,1 0,8 0,0 1,0 0,0 1,0 0,0 1,1 0,2 0,9 0,2 0,8 0,1 HP0184 hypothetical protein 0,8 0,1 1,0 0,1 0,7 0,1 1,1 0,1 1,0 0,2 1,0 0,1 1,1 0,3 1,1 0,1 0,7 0,1 0,9 0,1 1,1 0,2 1,2 0,2 0,9 0,1 0,9 0,3 HP0185 hypothetical protein 0,7 0,1 1,0 0,2 0,7 0,1 1,1 0,2 1,1 0,3 1,0 0,1 1,1 0,3 0,9 0,1 0,7 0,1 1,1 0,1 0,8 0,1 1,5 0,2 0,9 0,1 1,2 0,6 HP0186 hypothetical protein 0,9 0,1 0,8 0,2 1,0 0,1 0,9 0,2 1,0 0,1 0,7 0,1 1,1 0,2 0,9 0,1 0,8 0,1 1,3 0,2 0,9 0,1 0,9 0,1 1,2 0,1 1,5 0,3 HP0187 hypothetical protein 0,8 0,2 0,9 0,1 0,9 0,1 1,0 0,1 0,9 0,1 0,7 0,1 1,1 0,2 0,8 0,1 0,8 0,1 0,8 0,2 1,0 0,2 0,9 0,0 1,2 0,1 0,9 N.D. HP0188 hypothetical protein 0,8 N.D. 0,9 0,2 0,7 N.D. 0,8 0,2 1,1 0,2 1,0 0,3 1,1 0,1 0,9 0,1 0,9 0,0 1,0 0,1 0,9 0,1 1,0 0,0 1,1 0,1 1,3 0,1 HP0189 conserved hypothetical integral membrane protein 0,6 0,1 1,0 0,1 0,8 0,1 1,1 0,1 1,1 0,2 1,0 0,2 0,8 0,2 0,9 0,1 1,2 0,2 1,0 0,2 1,1 0,3 1,2 0,0 1,1 0,2 1,2 0,2 HP0190 conserved hypothetical secreted protein 0,7 0,1 1,0 0,2 0,9 0,1 1,0 0,1 1,0 0,2 1,0 0,1 1,0 0,1 1,0 0,1 0,8 0,2 1,6 0,5 1,2 0,3 1,1 0,1 1,0 0,0 1,1 0,2 HP0191 fumarate reductase, iron-sulfur subunit 0,8 0,1 0,8 0,1 1,1 0,1 1,0 0,1 1,0 0,3 1,1 0,3 0,9 0,2 0,8 0,1 0,6 0,1 1,1 0,1 0,7 0,1 1,2 0,1 1,0 0,2 1,0 0,1 HP0192 no description available 1,1 0,0 0,8 0,1 1,7 0,0 1,0 0,1 1,0 0,2 1,0 0,2 1,0 N.D. 0,8 0,1 0,8 0,1 0,9 0,1 0,7 0,1 N.D. N.D. 0,9 0,1 1,0 0,2 HP0193 fumarate reductase, cytochrome b subunit 1,0 0,1 0,8 0,1 1,5 0,1 1,0 0,1 1,1 0,2 1,3 0,4 1,0 0,3 0,8 0,0 0,9 0,1 1,0 0,1 0,8 0,2 1,4 0,4 0,9 0,2 0,9 0,2 HP0194 triosephosphate isomerase 2,4 0,0 1,1 0,1 1,5 0,0 1,0 0,1 1,0 0,1 1,4 0,3 1,1 0,2 1,0 0,0 1,2 0,2 1,1 0,1 0,8 0,2 1,4 0,2 0,9 0,1 1,1 0,1 HP0195 enoyl-(acyl-carrier-protein) reductase (NADH) 1,8 0,2 1,0 0,1 1,4 0,1 0,9 0,1 1,1 0,3 1,5 0,3 1,0 0,1 0,8 0,1 0,9 0,1 0,9 0,1 0,8 0,2 1,2 0,3 1,0 0,1 1,0 0,1 HP0196 UDP-3-0-(3-hydroxymyristoyl) glucosamine N-acyltransferase 1,5 0,1 0,8 0,3 1,2 0,0 1,0 0,1 1,0 0,3 1,2 0,2 1,0 0,1 0,9 0,0 1,0 0,0 0,9 0,1 0,9 0,3 1,2 0,1 0,8 0,1 0,8 0,1 HP0197 S-adenosylmethionine synthetase 2 1,2 0,1 0,9 0,4 1,0 0,1 0,9 0,1 1,0 0,2 1,3 0,4 0,9 0,1 0,8 0,1 1,0 0,1 0,7 0,1 1,3 0,2 1,0 0,2 0,8 0,1 0,8 0,1 HP0198 nucleoside diphosphate kinase 1,3 0,1 1,0 0,1 0,9 0,0 1,1 0,2 1,2 0,2 1,0 0,1 0,9 0,2 1,0 0,0 1,3 0,1 1,0 0,1 1,3 0,1 1,0 0,1 1,1 0,2 1,0 0,1 HP0199 hypothetical protein 1,1 0,1 0,9 0,1 0,9 0,0 0,9 0,0 1,2 0,2 1,3 0,2 1,0 0,1 1,0 0,1 1,3 0,1 1,0 0,1 1,2 0,2 0,8 0,0 1,2 0,2 0,9 0,2 HP0200 ribosomal protein L32 0,7 0,1 1,4 0,2 0,7 0,0 1,4 0,5 0,9 0,3 0,9 0,1 1,0 0,2 1,0 0,2 1,1 0,1 1,4 0,1 1,3 0,3 1,1 0,1 1,1 0,2 0,7 0,1 HP0201 fatty acid/phospholipid synthesis protein 0,4 0,1 1,5 0,1 0,5 0,1 1,3 0,4 0,9 0,5 1,1 0,2 0,9 0,3 0,8 0,2 0,9 0,1 0,8 0,2 1,4 0,5 1,1 0,2 0,7 0,1 0,4 0,2 HP0202 beta-ketoacyl-acyl carrier protein synthase III 0,5 0,1 1,4 0,1 0,6 0,0 1,2 0,2 0,9 0,5 0,8 0,0 0,8 0,3 1,0 0,1 0,9 0,1 1,0 0,1 1,4 0,4 1,2 0,2 0,5 0,0 0,3 0,1 HP0203 hypothetical protein 0,9 0,1 1,0 0,2 1,1 0,2 1,2 0,2 1,0 0,2 0,7 0,0 1,0 0,2 0,9 0,1 0,9 0,1 0,9 0,1 1,1 0,3 1,0 0,0 0,8 0,1 0,5 0,1 HP0204 hypothetical protein 0,8 0,1 0,9 0,1 0,9 0,0 1,4 0,2 0,9 0,2 1,3 0,1 1,4 0,4 1,1 0,1 1,3 0,4 1,2 0,1 1,0 0,2 1,0 0,2 1,3 0,2 1,9 0,4 HP0205 hypothetical protein 0,7 0,1 0,9 0,1 0,8 0,2 1,1 0,2 1,0 0,2 1,0 N.D. 1,0 0,1 0,9 0,1 1,0 0,1 0,9 0,1 0,9 0,1 1,0 N.D. 1,1 0,3 1,0 0,1 HP0206 hypothetical protein 0,7 0,1 1,1 0,2 0,8 0,0 1,2 0,1 1,0 0,2 1,4 0,1 1,1 0,1 1,1 0,1 1,0 0,2 0,8 0,1 1,0 0,1 1,0 N.D. 1,3 0,4 1,3 N.D. HP0207 ATP-binding protein 0,7 N.D. 1,0 0,1 0,5 0,1 1,0 0,1 0,9 0,3 1,3 0,2 0,9 0,2 1,0 0,2 1,7 0,2 0,9 0,2 1,6 0,2 1,2 0,1 1,1 0,4 1,2 0,3 HP0208 lipopolysaccharide 1,2-glucosyltransferase, authentic frameshift 1,1 0,2 N.D. N.D. 0,9 0,0 N.D. N.D. 0,7 0,2 1,1 0,1 1,0 0,1 1,0 0,1 1,5 0,2 0,9 0,1 1,2 0,2 1,1 0,0 1,1 0,2 1,0 0,3 HP0209 hypothetical protein 1,4 0,3 0,9 0,3 1,3 0,2 1,1 0,2 1,2 0,3 0,9 0,0 1,0 0,1 1,0 0,1 1,2 0,1 0,7 0,1 1,1 0,1 0,9 0,2 1,2 0,1 1,0 0,2 HP0210 no description available 1,4 0,0 0,9 0,2 1,3 0,0 0,9 0,1 0,9 0,1 1,2 0,3 0,9 0,1 1,0 0,1 1,2 0,2 1,5 0,5 1,0 0,1 1,2 0,1 1,3 0,1 1,0 0,2 HP0211 conserved hypothetical secreted protein N.D. N.D. 1,1 0,2 1,8 0,0 1,0 0,1 1,3 0,4 1,3 0,2 1,0 0,2 1,0 0,1 0,9 0,2 1,0 0,3 0,7 0,2 1,2 0,3 1,2 0,4 1,4 0,1 HP0212 succinyl-diaminopimelate desuccinylase 1,2 0,1 0,9 0,1 1,2 0,0 0,9 0,1 1,0 0,3 1,1 0,1 0,9 0,1 0,9 0,0 1,1 0,2 1,1 0,1 0,9 0,1 1,2 0,1 1,1 0,1 1,4 0,2 HP0213 glucose inhibited division protein 0,9 0,1 0,9 0,2 0,8 0,0 0,9 0,1 0,9 0,2 1,0 0,1 0,9 0,1 1,0 0,0 1,0 0,0 1,4 0,1 1,1 0,2 1,1 0,3 0,8 0,3 0,6 0,0 HP0214 sodium-dependent transporter 1,8 0,0 1,0 0,1 1,4 0,0 1,2 0,2 1,1 0,2 1,1 0,2 1,1 0,1 0,9 0,0 1,0 0,1 1,4 0,4 0,9 0,1 1,2 0,0 1,1 0,2 1,4 0,2 HP0215 CDP-diglyceride synthetase 1,2 0,2 0,9 0,1 0,9 0,0 0,9 0,1 1,0 0,1 1,6 0,5 0,8 0,2 0,8 0,0 0,9 0,0 1,1 0,2 1,0 0,2 1,1 0,1 1,1 0,3 0,8 0,1 HP0216 no description available 0,6 0,1 0,8 0,1 0,9 0,0 1,0 0,1 1,0 0,2 0,9 0,1 1,1 0,1 1,0 0,2 0,8 0,0 1,0 0,1 1,1 0,2 1,0 0,0 1,1 0,1 1,1 0,1 HP0217 no description available 0,7 0,1 0,9 0,2 0,9 0,2 1,0 0,2 1,0 0,2 0,9 0,2 1,0 0,1 0,8 0,1 0,9 0,1 0,9 0,1 0,9 0,1 1,1 0,0 1,1 0,0 0,9 0,1 HP0218 hypothetical protein 0,8 0,1 0,9 0,3 0,8 0,1 0,9 0,1 1,3 0,5 1,0 0,2 1,0 0,3 0,8 0,1 0,7 0,1 1,6 0,0 0,6 0,1 1,5 0,2 1,0 0,1 1,0 0,2 HP0219 hypothetical protein 0,8 N.D. 0,9 0,1 0,9 0,2 0,9 0,1 1,1 0,2 0,9 0,1 0,9 0,1 1,0 0,1 1,0 0,2 1,1 0,1 1,1 0,3 1,0 0,0 1,3 0,2 0,9 0,0 HP0220 synthesis of [Fe-S] cluster 0,7 0,1 1,2 0,1 0,6 0,0 1,1 0,1 0,7 0,4 1,2 0,1 1,2 0,3 1,2 0,2 1,3 0,3 0,9 0,1 N.D. N.D. 0,9 0,1 1,1 0,4 1,0 0,3 HP0221 nifu-like protein 0,8 0,0 1,2 0,1 0,6 0,1 1,1 0,1 0,7 0,4 N.D. N.D. 1,1 0,4 N.D. N.D. 1,3 0,1 1,1 0,2 1,9 0,1 1,5 N.D. 0,9 0,3 0,7 0,2 HP0222 hypothetical protein 0,8 0,1 0,9 0,1 0,6 0,0 0,9 0,1 0,7 0,3 1,4 0,2 1,1 0,1 1,4 0,2 1,4 0,2 1,2 0,2 1,6 0,2 0,9 0,1 0,6 0,2 0,3 0,0 HP0223 ATP-dependent protease N.D. N.D. 0,9 0,1 N.D. N.D. 0,8 0,1 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0224 peptide methionine sulfoxide reductase 1,9 0,0 0,9 0,2 0,9 0,0 0,9 0,1 1,0 0,3 1,3 0,2 1,2 0,2 0,8 0,1 1,1 0,2 1,3 0,2 0,9 0,4 1,2 0,3 1,0 0,2 0,8 N.D. HP0226 conserved hypothetical integral membrane protein 1,5 0,2 1,0 0,1 1,1 0,0 1,0 0,1 1,1 0,2 1,3 0,0 1,0 0,1 1,0 0,1 1,1 0,1 0,9 0,1 1,0 0,1 0,8 0,1 0,9 0,0 3,1 2,7 HP0227 no description available N.D. N.D. 0,8 0,1 N.D. N.D. 0,9 0,3 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0228 conserved hypothetical integral membrane protein 1,4 0,0 0,9 0,1 1,5 0,2 0,9 0,2 1,1 0,4 1,3 0,0 1,1 0,1 1,1 0,1 1,0 0,0 0,8 0,2 1,0 0,1 1,1 0,1 1,2 0,4 1,2 0,2 HP0229 outer membrane protein 1,3 0,0 0,9 0,2 1,3 0,1 0,8 0,0 1,1 0,2 1,3 0,3 1,0 0,1 0,7 0,0 1,3 0,2 1,5 0,2 0,5 0,2 1,6 0,2 0,9 0,1 1,2 0,3 HP0230 CTP:CMP-3-deoxy-D-manno-octulosonate-cytidylyl-transferase 1,6 0,1 1,3 0,1 1,1 0,1 1,1 0,1 1,2 0,2 1,0 0,1 1,1 0,1 1,1 0,1 1,1 0,1 0,7 0,1 1,0 0,1 0,9 N.D. 1,0 0,1 0,8 0,3 HP0231 hypothetical protein 1,1 0,0 1,0 0,1 1,1 0,0 1,2 0,1 0,9 0,2 1,0 0,2 1,0 0,2 0,7 0,1 1,4 0,2 0,8 0,1 1,3 0,3 1,2 0,1 1,0 0,2 1,6 0,6 HP0232 secreted protein involved in flagellar motility 1,0 0,1 1,0 0,2 1,0 0,1 1,2 0,2 0,9 0,2 1,1 0,3 1,0 0,2 0,8 0,0 1,1 0,1 0,7 0,1 1,0 0,1 0,9 0,1 0,9 0,2 0,9 0,1 HP0233 conserved hypothetical protein 0,6 0,0 0,9 0,1 0,7 0,0 1,0 0,1 0,9 0,2 0,8 0,0 1,1 0,2 0,9 0,1 1,1 0,2 0,8 0,1 1,0 0,2 0,9 0,1 1,0 0,3 0,9 0,2 HP0234 conserved hypothetical integral membrane protein 0,8 0,0 1,2 0,3 0,9 0,1 1,1 0,1 1,2 0,2 0,9 0,1 1,0 0,1 0,9 0,1 1,1 0,2 0,9 0,2 1,0 0,1 1,1 0,1 1,0 0,1 1,2 0,3 HP0235 no description available 0,8 0,1 0,9 0,1 0,9 0,0 0,9 0,1 1,0 0,2 0,8 0,1 1,1 0,0 0,8 0,1 1,0 0,1 1,3 0,2 0,9 0,2 1,2 0,2 0,8 0,1 0,9 0,2 HP0236 hypothetical protein N.D. N.D. 1,0 0,1 0,7 N.D. 1,0 0,3 1,2 0,3 1,0 0,1 1,2 0,1 1,1 0,1 1,1 0,2 1,0 0,1 0,9 0,0 1,0 N.D. 1,1 0,3 0,9 0,0 HP0237 porphobilinogen deaminase 0,7 0,1 0,9 0,1 0,6 0,0 0,9 0,0 1,0 0,2 1,0 0,1 0,9 0,3 1,2 0,2 1,0 0,1 1,3 0,0 1,5 0,3 1,1 0,2 1,0 0,3 0,9 0,2 HP0238 no description available 0,9 0,1 0,9 0,2 0,9 0,2 0,9 0,2 1,0 0,2 1,0 0,0 1,1 0,1 1,1 0,2 1,0 0,1 1,3 0,2 1,2 0,2 1,0 0,1 0,9 0,3 0,8 0,2 HP0239 no description available 0,8 0,0 0,9 0,1 0,8 0,1 0,9 0,1 1,1 0,2 1,0 0,0 1,3 0,6 1,1 0,1 1,1 0,1 1,0 0,1 1,1 0,1 0,9 N.D. 0,9 0,1 0,9 0,2 HP0240 octaprenyl-diphosphate synthase 0,9 0,0 1,2 0,2 1,2 0,2 1,1 0,2 1,2 0,2 0,8 0,0 1,0 0,1 1,3 0,1 1,2 0,0 1,4 0,1 1,2 0,1 1,2 0,1 1,2 0,2 0,8 N.D. HP0241 hypothetical protein 2,4 0,1 1,7 0,3 1,3 0,0 1,2 0,2 1,4 0,4 0,9 0,1 1,0 0,2 1,0 0,0 1,3 0,0 1,2 0,2 1,2 0,1 1,2 0,1 1,3 0,3 0,7 0,3 HP0242 hypothetical protein 2,2 0,4 1,0 0,1 1,7 0,1 0,9 0,1 1,6 0,5 1,3 0,1 1,1 0,4 0,9 0,1 1,5 0,4 1,2 0,4 1,0 0,1 1,5 0,2 1,4 0,2 1,2 0,4 HP0243 neutrophil activating protein (napa) 2,6 0,0 0,8 0,2 1,5 0,1 0,9 0,4 1,5 0,6 1,7 0,4 1,4 0,4 0,8 0,0 2,1 0,8 1,6 0,5 0,7 0,4 2,4 0,3 1,9 0,4 1,1 0,3 HP0244 Histidine kinase specific for flgr 1,1 N.D. 1,2 0,1 1,1 N.D. 1,1 0,2 1,0 0,1 0,9 0,0 1,1 0,0 1,1 0,0 1,2 0,1 1,0 0,0 1,1 0,1 1,0 0,2 0,9 0,1 0,5 0,1 HP0245 hypothetical protein 1,3 0,1 1,0 0,1 1,1 0,1 0,8 0,3 1,0 0,2 1,0 0,2 1,2 0,3 1,1 0,0 1,0 0,1 0,7 0,1 1,1 0,0 1,5 N.D. 0,9 0,1 2,0 0,4 HP0246 Basal body P-ring protein (flgi) 1,7 0,2 1,0 0,1 1,3 0,0 0,9 0,1 1,1 0,2 1,2 0,1 1,0 0,1 1,1 0,0 1,0 0,1 1,0 0,3 1,0 0,1 1,2 0,2 1,0 0,0 1,1 0,3 HP0247 no description available 1,1 0,0 1,1 0,1 0,9 0,0 1,0 0,0 1,0 0,2 0,9 0,0 0,9 0,1 1,0 0,1 1,4 0,2 1,1 0,1 1,2 0,3 1,2 0,1 1,0 0,0 1,0 0,2 HP0248 conserved hypothetical protein 1,1 0,1 0,9 0,1 0,9 0,1 0,9 0,1 0,9 0,1 1,0 0,0 0,9 0,1 0,9 0,0 1,1 0,2 0,8 0,1 1,0 0,1 1,0 0,1 0,9 0,3 0,7 0,1 HP0249 hypothetical protein 0,6 0,0 1,0 0,2 0,7 0,0 1,1 0,1 1,0 0,2 0,9 0,0 1,0 0,2 1,1 0,2 1,0 0,1 1,3 0,2 1,0 0,1 1,2 0,1 1,1 0,1 0,7 0,1 HP0250 oligopeptide ABC transporter, ATP-binding protein 0,7 0,1 0,9 0,2 0,8 0,1 0,7 0,3 1,1 0,3 0,8 N.D. 1,2 0,3 0,9 0,1 0,8 0,0 0,8 0,0 0,8 0,0 1,1 0,1 1,0 0,1 1,2 0,1 HP0251 oligopeptide ABC transporter, permease protein 0,7 0,1 0,9 0,1 1,0 0,1 0,9 0,1 0,9 0,1 0,8 0,1 1,0 0,2 1,1 0,2 0,7 0,1 1,3 0,4 1,2 0,3 1,0 0,1 0,8 0,1 1,1 0,2 HP0252 outer membrane protein 0,8 0,0 1,1 0,2 0,8 0,1 1,2 0,1 1,1 0,2 0,9 0,1 0,9 0,1 0,9 0,1 0,9 0,1 1,3 0,3 1,0 0,2 1,0 0,0 0,9 0,1 0,8 0,1 HP0253 hypothetical protein 0,6 0,0 1,1 0,1 0,7 0,0 1,1 0,1 0,9 0,2 0,7 0,0 1,0 0,1 1,0 0,1 0,8 0,1 1,1 0,2 1,1 0,2 0,8 N.D. 0,9 0,1 0,8 0,1 HP0254 outer membrane protein 0,7 N.D. 1,3 0,2 1,0 N.D. 1,1 0,1 0,9 0,3 0,8 0,1 1,0 0,1 0,9 0,1 0,8 0,1 0,8 0,0 1,1 0,2 1,0 0,1 0,9 0,2 0,6 0,2 HP0255 adenylosuccinate synthetase 0,4 0,1 0,8 0,2 0,5 0,0 0,8 0,2 0,9 0,2 0,7 0,0 0,8 0,0 1,1 0,2 0,6 0,1 1,4 0,3 1,6 0,5 1,1 0,1 0,7 0,2 0,8 0,2 HP0256 hypothetical protein 0,5 0,1 1,6 0,6 0,6 0,1 1,2 0,3 1,0 0,5 0,9 0,1 1,0 0,2 1,1 0,1 0,7 0,2 1,0 0,1 1,4 0,4 0,7 0,1 0,9 0,1 0,5 0,0 HP0257 conserved hypothetical secreted protein 1,0 0,0 1,1 0,1 0,9 0,0 1,1 0,1 1,1 0,4 0,9 0,1 1,1 0,1 1,2 0,0 0,8 0,3 1,1 0,2 N.D. N.D. 1,1 0,1 0,9 0,1 0,7 0,1 HP0258 conserved hypothetical integral membrane protein 1,0 0,1 1,1 0,0 0,9 0,1 1,0 0,1 1,2 0,3 1,1 0,0 0,9 0,2 1,1 0,0 1,1 0,0 1,1 0,1 1,2 0,1 1,4 0,1 1,1 0,1 0,6 0,3 HP0259 no description available 1,6 0,0 1,0 0,2 1,3 0,0 0,9 0,1 1,1 0,4 1,0 0,1 1,1 0,2 1,1 0,1 0,9 0,1 1,0 0,1 1,1 0,1 1,8 0,0 1,3 0,1 0,6 0,1 HP0260 adenine specific DNA methyltransferase 1,4 0,1 0,7 0,1 1,3 0,1 0,9 0,2 1,1 0,2 1,0 0,1 1,0 0,0 1,0 0,0 1,0 0,1 1,2 0,4 1,0 0,1 0,9 0,0 1,0 0,0 1,0 0,1 HP0261 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 1,0 0,1 1,0 0,2 1,1 0,0 1,2 0,2 1,1 0,0 1,2 N.D. 1,1 N.D. 1,0 0,1 2,2 N.D. 1,2 0,1 1,3 0,3 HP0262 hypothetical protein N.D. N.D. 1,0 0,1 1,2 0,1 1,0 0,1 1,0 0,1 1,2 0,2 1,1 0,2 1,4 0,1 1,2 0,1 0,6 0,1 1,6 0,2 2,3 N.D. 1,1 0,1 1,9 0,7 HP0263 adenine specific DNA methyltransferase 1,2 0,2 1,2 0,1 1,0 N.D. 1,0 0,1 1,0 0,2 0,9 0,1 1,0 0,1 1,2 0,1 1,2 0,1 0,8 0,2 1,3 0,1 3,9 N.D. 1,1 0,1 1,2 0,5 HP0264 ATP-dependent protease binding subunit 1,2 0,1 0,8 0,1 1,2 0,1 0,8 0,2 0,9 0,2 0,9 0,1 1,1 0,1 0,8 0,1 0,9 0,1 0,9 0,1 0,9 0,1 0,9 0,1 1,0 0,1 1,3 0,5 HP0265 cytochrome c biogenesis protein 0,8 0,1 0,9 0,1 0,9 0,1 1,0 0,0 1,0 0,3 0,9 0,1 1,1 0,2 0,9 0,1 0,7 0,1 1,2 0,4 0,9 0,1 1,0 0,0 0,8 0,1 1,2 0,1 HP0266 dihydroorotase 0,7 0,0 1,1 0,0 0,8 0,1 1,0 0,1 0,9 0,2 1,0 0,0 0,9 0,2 0,8 0,1 0,8 0,1 1,0 0,1 1,0 0,2 1,1 0,0 0,9 0,1 1,1 0,2 HP0267 chlorohydrolase 0,8 0,1 1,0 0,1 0,8 0,1 0,9 0,1 1,0 0,2 1,0 0,0 1,1 0,2 0,9 0,2 0,8 0,0 1,2 0,2 0,9 0,0 1,2 0,1 0,9 0,2 0,8 0,1 HP0268 hypothetical protein 1,0 0,0 1,0 0,1 1,1 0,2 1,1 0,1 1,1 0,3 1,2 0,2 1,1 0,1 0,8 0,1 0,8 0,1 1,1 0,2 0,7 0,1 1,3 0,1 0,8 0,3 1,1 0,2
3 HP0269 conserved hypothetical ATP-binding protein 0,7 0,1 0,8 0,1 1,1 0,2 1,0 0,2 1,0 0,3 0,9 0,0 0,9 0,2 0,9 0,1 0,7 0,1 1,2 0,3 1,1 0,1 1,0 0,1 0,8 0,1 1,0 0,3 HP0270 hypothetical protein 0,6 0,0 1,0 0,1 0,9 0,1 1,0 0,2 0,9 0,3 0,8 0,0 0,8 0,2 0,9 0,1 0,6 0,1 0,8 0,0 1,3 0,2 1,0 0,0 0,8 0,1 0,8 0,3 HP0271 hypothetical protein 0,7 0,1 1,0 0,2 1,0 0,1 0,9 0,1 1,1 0,2 0,9 0,0 0,9 0,2 1,2 0,2 0,7 0,2 1,0 0,2 1,4 0,4 1,0 0,1 0,9 0,2 0,5 0,0 HP0272 hypothetical protein 1,0 0,0 1,0 0,2 1,2 0,2 0,9 0,1 1,1 0,4 1,0 0,0 1,0 0,2 1,3 0,2 0,9 0,1 0,9 0,2 1,2 0,1 1,3 N.D. 0,9 0,5 0,7 0,0 HP0273 hypothetical protein 1,2 0,0 1,0 0,2 1,2 0,0 1,1 0,2 1,1 0,3 1,2 0,1 1,0 0,2 1,3 0,0 1,0 0,1 0,9 0,2 1,5 0,4 1,4 0,1 1,1 0,2 0,6 0,2 HP0274 conserved hypothetical protein 1,3 0,1 0,9 0,1 1,0 0,1 0,9 0,1 1,1 0,3 1,4 0,1 1,1 0,1 1,3 0,0 1,2 0,1 0,7 0,1 1,1 0,2 1,1 0,1 1,2 0,2 0,8 0,0 HP0275 no description available N.D. N.D. 1,1 0,1 N.D. N.D. 1,2 0,1 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0276 hypothetical protein 1,1 0,2 0,9 0,2 1,1 0,1 1,0 0,2 1,0 0,3 1,4 0,2 0,9 0,1 1,2 0,1 1,3 0,3 1,4 0,5 1,2 0,1 1,7 0,6 1,2 0,2 0,8 0,1 HP0277 ferredoxin 0,7 0,0 1,0 0,1 0,6 0,0 1,1 0,1 1,0 0,4 1,1 0,3 1,2 0,2 1,0 0,1 1,6 0,4 0,8 0,2 N.D. N.D. 1,0 0,1 1,1 0,3 1,3 0,2 HP0278 guanosine pentaphosphate phosphohydrolase 0,4 0,0 1,3 0,2 0,6 0,1 1,4 0,4 1,0 0,4 1,0 0,1 1,0 0,3 1,2 0,1 1,6 0,3 0,8 0,1 N.D. N.D. 0,9 0,0 1,0 0,3 0,9 0,2 HP0279 lipopolysaccharide heptosyltransferase-1 0,8 0,1 1,5 0,4 0,9 0,0 1,1 0,1 1,1 0,4 1,1 0,1 1,0 0,2 1,4 0,1 1,4 0,4 1,1 0,2 2,3 1,1 1,4 0,0 1,2 0,5 0,7 0,3 HP0280 heat shock protein B 1,0 0,1 0,9 0,2 0,8 0,1 1,4 0,4 0,9 0,2 1,4 0,2 1,2 0,2 1,2 0,1 1,3 0,2 1,2 0,2 1,4 0,3 1,3 0,2 1,3 0,3 0,9 0,1 HP0281 no description available 0,8 0,1 1,2 0,3 0,8 0,0 1,0 0,3 1,1 0,2 0,8 0,1 1,3 0,4 1,0 0,1 0,9 0,1 0,9 0,1 0,9 0,1 1,0 0,0 0,9 0,1 1,4 0,3 HP0282 hypothetical protein 0,7 0,0 1,4 0,1 0,8 0,1 1,1 0,1 0,9 0,3 0,9 0,1 1,0 0,0 0,8 0,1 0,8 0,1 1,1 0,2 1,0 0,1 1,1 0,1 1,0 0,0 1,1 0,1 HP dehydroquinate synthase 1,0 0,0 0,9 0,1 0,9 0,1 0,9 0,1 1,0 0,2 0,9 0,1 1,1 0,2 1,2 0,2 1,0 0,1 0,8 0,3 1,3 0,3 0,9 0,0 0,9 0,3 0,7 0,1 HP0284 conserved hypothetical integral membrane protein 1,0 0,0 1,1 0,1 0,6 0,3 1,0 0,2 1,1 0,3 1,0 0,1 1,0 0,1 1,1 0,1 1,1 0,1 0,9 0,1 1,0 0,1 0,9 N.D. 1,0 0,1 0,8 0,1 HP0285 conserved hypothetical protein 0,8 0,0 0,9 0,1 1,0 0,0 0,9 0,2 1,0 0,2 1,0 0,1 1,1 0,0 1,0 0,1 0,9 0,1 1,1 0,2 1,0 0,0 1,1 0,2 0,9 0,1 1,1 N.D. HP0286 cell division protein 0,8 N.D. 1,1 0,1 0,7 0,1 1,1 0,2 1,1 0,3 0,8 0,1 0,9 0,2 0,9 0,1 0,6 0,1 0,9 0,0 1,3 0,2 1,1 0,1 0,9 0,2 1,1 N.D. HP0287 hypothetical protein 0,7 0,0 1,2 0,2 0,9 0,0 1,2 0,1 1,0 0,3 1,0 0,0 0,9 0,2 1,3 0,2 0,9 0,2 1,5 0,4 1,3 0,1 1,0 0,0 1,0 0,3 0,7 0,1 HP0288 hypothetical protein 0,8 0,0 1,1 0,3 0,8 0,1 1,1 0,3 1,0 0,4 1,0 0,0 1,0 0,1 1,2 0,1 1,0 0,2 1,3 0,1 1,1 0,1 1,0 0,1 1,0 0,1 0,9 0,1 HP0289 toxin-like outer membrane protein 1,4 0,1 1,3 0,1 1,3 0,0 1,1 0,1 1,1 0,2 1,0 0,2 1,1 0,0 1,1 0,0 1,4 0,4 1,0 0,1 1,2 0,1 1,3 0,1 1,1 0,1 0,8 0,2 HP0290 diaminopimelate decarboxylase (dap decarboxylase) 0,7 0,1 1,1 0,2 0,7 0,1 1,2 0,1 0,9 0,3 1,2 0,2 0,8 0,2 1,2 0,0 1,0 0,1 1,0 0,1 N.D. N.D. 1,4 0,0 0,9 0,1 0,8 0,0 HP0291 hypothetical protein 0,8 0,0 0,9 0,1 0,5 0,2 1,1 0,2 0,9 0,4 1,1 0,1 1,0 0,3 1,1 0,1 1,0 0,2 0,9 0,1 1,8 0,8 1,9 0,6 0,9 0,1 0,5 0,1 HP0292 hypothetical protein 0,7 0,1 1,0 0,1 0,6 0,1 0,8 0,2 0,9 0,3 1,3 0,2 1,1 0,2 1,5 0,0 1,0 0,1 1,0 0,2 N.D. N.D. 1,3 0,1 0,7 0,1 0,5 0,1 HP0293 para-aminobenzoate synthetase 1,1 0,0 0,8 0,2 1,0 0,1 0,9 0,1 1,0 0,3 1,1 0,2 1,1 0,2 1,0 0,0 1,2 0,2 1,0 0,1 1,3 0,1 1,1 N.D. 1,0 0,2 0,4 0,1 HP0294 aliphatic amidase 0,7 0,1 1,3 0,8 0,1 0,0 1,1 0,5 1,8 1,4 0,7 0,1 0,8 0,3 0,6 0,1 11,0 3,3 0,8 0,4 6,2 0,8 0,3 0,0 16,9 3,4 1,2 0,3 HP0295 Flagellar hook associated protein 3/HAP3 (flgl ) 1,2 0,0 1,0 0,1 1,1 0,1 0,9 0,1 2,2 0,8 3,2 0, ,9 5,8 0,7 8,4 1,4 5,0 4,9 5,6 1,3 6,3 1,6 0,4 0,1 10,4 6,9 HP0296 ribosomal protein L21 0,6 0,0 1,0 0,1 0,7 0,1 1,0 0,1 0,9 0,3 1,1 0,2 1,1 0,2 0,7 0,1 2,2 0,6 1,0 0,2 1,6 0,6 1,1 0,3 0,9 0,1 1,9 0,2 HP0297 ribosomal protein L27 0,5 0,0 1,0 0,1 0,7 0,0 1,1 0,2 0,9 0,2 0,8 0,1 1,1 0,3 0,8 0,1 1,5 0,2 1,4 0,2 1,0 0,3 1,2 0,3 0,5 0,1 0,6 0,2 HP0298 no description available 0,6 0,1 0,8 0,1 1,0 0,1 0,9 0,1 1,0 0,3 0,6 0,0 1,0 0,1 0,8 0,0 0,9 0,2 1,4 0,2 0,8 0,1 0,9 0,0 0,9 0,1 0,5 0,2 HP0299 dipeptide ABC transporter, permease protein 0,8 0,0 0,8 0,1 0,9 0,0 0,9 0,1 0,9 0,2 0,6 0,1 0,8 0,2 0,9 0,1 0,6 0,2 0,9 0,0 1,0 0,4 0,9 0,0 0,6 0,2 0,9 N.D. HP0300 dipeptide ABC transporter, permease protein 0,9 0,0 0,9 0,1 1,0 0,1 0,9 0,1 1,0 0,2 0,7 0,0 0,9 0,1 0,7 0,1 0,6 0,2 0,9 0,1 1,1 0,4 0,9 0,0 0,6 0,1 0,7 0,1 HP0301 no description available 0,7 0,0 0,8 0,4 1,0 N.D. 0,9 0,1 0,9 0,1 0,7 0,1 0,9 0,2 0,9 0,1 0,6 0,1 1,2 0,0 0,9 0,1 0,7 N.D. 0,8 0,1 0,6 0,1 HP0302 dipeptide ABC transporter, ATP-binding protein 0,7 0,0 0,9 0,1 0,9 0,1 0,9 0,1 0,9 0,3 0,7 0,0 0,9 0,1 0,7 0,1 0,6 0,1 0,8 0,1 1,1 0,4 0,9 0,0 0,6 0,2 0,9 N.D. HP0303 GTP-binding protein 0,7 0,0 0,9 0,1 0,9 0,1 0,9 0,2 0,8 0,2 0,7 0,1 1,0 0,1 1,0 0,1 0,6 0,2 1,0 0,2 1,2 0,4 0,9 0,1 0,8 0,0 0,6 0,2 HP0304 hypothetical protein 0,8 0,0 0,9 0,1 0,7 0,1 0,9 0,1 1,0 0,2 0,8 0,0 0,9 0,1 0,9 0,1 0,6 0,2 1,0 0,1 1,3 0,4 1,0 0,0 0,8 0,4 0,6 0,0 HP0305 hypothetical protein 1,3 0,1 1,0 0,4 0,9 0,0 1,1 0,2 0,8 0,1 1,3 0,1 1,2 0,2 0,9 0,1 1,8 0,6 1,7 0,3 1,0 0,2 1,6 0,4 1,2 0,3 0,7 0,1 HP0306 glutamate-1-semialdehyde 2,1-aminomutase 1,0 0,1 1,3 0,1 0,9 0,0 1,5 0,4 1,0 0,2 1,2 0,0 0,9 0,2 0,8 0,0 1,8 0,3 1,2 0,2 1,5 0,6 1,0 0,3 1,4 0,1 2,0 0,4 HP0307 hypothetical protein 1,1 0,0 0,9 0,4 1,1 N.D. 0,8 0,5 1,2 0,3 1,3 0,3 1,0 0,1 1,1 0,0 1,8 0,2 0,9 0,1 N.D. N.D. 1,1 N.D. 1,3 0,6 1,1 0,0 HP0308 hypothetical protein 1,1 0,1 1,1 0,1 0,9 0,1 1,2 0,3 1,2 0,4 1,1 0,1 1,0 0,1 1,2 0,1 1,6 0,2 1,0 0,2 1,3 0,1 1,2 N.D. 1,4 0,3 0,9 0,2 HP0309 conserved hypothetical protein 1,2 0,1 1,1 0,1 1,0 0,0 1,1 0,1 1,0 0,2 0,8 0,1 1,0 0,1 1,1 0,0 1,2 0,2 0,9 0,1 1,8 0,4 1,1 0,1 0,9 0,1 1,3 0,5 HP0310 conserved hypothetical protein 1,2 0,1 0,9 0,0 0,8 0,1 0,9 0,1 1,1 0,3 0,6 0,1 1,0 0,1 0,9 0,1 1,0 0,1 0,7 0,2 1,1 0,1 0,7 0,1 1,5 0,7 0,6 0,1 HP0311 hypothetical protein 1,6 0,1 0,9 0,1 1,1 0,1 0,9 0,1 1,1 0,3 0,6 0,1 0,9 0,1 0,9 0,0 1,0 0,2 0,7 0,1 1,3 0,4 0,7 0,1 1,4 0,4 1,0 0,3 HP0312 conserved hypothetical ATP-binding protein 1,1 0,1 0,9 0,2 0,9 0,1 0,9 0,1 1,0 0,2 0,7 0,1 0,8 0,1 0,9 0,0 1,0 0,2 0,8 0,1 1,4 0,4 0,8 0,1 1,5 0,4 0,9 0,0 HP0313 nitrite extrusion protein 0,8 0,0 1,0 0,0 1,0 0,1 1,0 0,2 1,1 0,2 0,8 0,1 1,0 0,1 1,1 0,1 1,1 0,2 1,1 0,0 1,1 0,2 0,9 0,0 1,1 0,3 1,0 0,2 HP0315 virulence associated protein D N.D. N.D. 0,9 0,1 N.D. N.D. 0,9 0,1 N.D. N.D. 1,0 0,2 1,1 N.D. 1,1 0,0 1,2 0,2 0,5 0,0 1,2 0,1 1,1 N.D. 1,3 N.D. 1,3 0,0 HP0316 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 1,0 0,1 N.D. N.D. 1,3 0,3 1,1 N.D. 1,1 0,2 1,3 N.D. 0,7 0,1 1,3 0,2 1,3 N.D. 1,1 N.D. 1,3 N.D. HP0317 outer membrane protein 0,7 0,1 0,7 0,2 0,8 0,1 0,9 0,1 0,9 0,3 1,1 N.D. 1,4 0,7 1,0 0,1 0,8 0,1 0,8 0,0 1,0 0,0 0,9 0,1 0,9 0,0 0,9 N.D. HP0318 conserved hypothetical protein 1,1 0,0 0,7 0,1 1,5 0,1 0,8 0,1 1,3 0,6 1,3 0,2 1,5 0,3 0,8 0,1 0,6 0,2 1,4 0,1 0,3 0,0 1,0 0,1 0,5 0,1 0,7 N.D. HP0319 arginyl-trna synthetase 0,7 0,0 1,1 0,1 0,8 0,0 1,1 0,1 1,0 0,2 0,9 0,0 0,8 0,0 0,8 0,1 0,9 0,2 0,7 0,1 0,9 0,1 1,0 N.D. 1,0 0,1 0,7 0,2 HP0320 conserved hypothetical secreted protein 1,0 0,0 1,0 0,2 1,1 0,2 0,9 0,1 1,0 0,2 0,7 0,1 1,0 0,1 1,1 0,1 0,8 0,1 1,2 0,1 0,9 0,1 1,0 0,1 1,0 0,2 0,9 0,2 HP0321 5'-guanylate kinase 0,8 0,0 0,9 0,2 0,8 0,1 0,9 0,1 0,9 0,3 0,8 0,2 0,9 0,1 0,9 0,1 1,0 0,2 1,3 0,1 0,9 0,1 1,3 0,0 0,9 0,1 1,0 0,1 HP0322 no description available N.D. N.D. 0,9 0,1 N.D. N.D. 0,9 0,1 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0323 membrane bound endonuclease 1,3 0,0 0,8 0,2 1,0 0,1 0,9 0,1 0,8 0,2 1,1 0,2 1,2 0,3 0,8 0,1 1,2 0,3 1,0 0,4 0,8 0,3 1,3 0,1 1,3 0,2 1,6 0,5 HP0324 outer membrane protein 1,3 0,2 1,0 0,1 1,2 0,0 1,0 0,2 1,0 0,1 1,1 0,2 1,0 0,1 0,9 0,1 1,0 0,2 0,9 0,1 1,0 0,2 1,3 0,1 1,2 0,1 1,5 0,2 HP0325 Basal body L-ring protein (flgh ) 1,1 0,1 0,8 0,1 0,9 0,1 0,8 0,1 1,0 0,1 1,1 0,2 1,1 0,2 1,1 0,1 1,3 0,1 0,9 0,1 1,3 0,2 1,0 0,1 0,6 0,2 0,4 0,0 HP0326 (neua) 1,4 0,1 1,1 0,1 1,3 0,1 1,2 0,1 1,1 0,3 1,0 0,1 0,9 0,0 1,1 0,1 1,6 0,1 0,8 0,1 1,3 0,0 1,2 N.D. 1,0 0,2 1,3 0,4 HP0327 (flag1/flmh ) N.D. N.D. 1,7 0,7 N.D. N.D. 1,4 0,3 1,2 0,4 1,1 0,2 1,1 0,1 1,5 0,1 1,6 0,4 0,9 0,0 1,5 0,1 1,1 N.D. 1,3 0,4 1,1 N.D. HP0328 conserved hypothetical protein 1,3 0,1 1,1 0,2 1,4 0,0 1,0 0,1 1,1 0,3 1,0 0,1 0,9 0,0 1,2 0,1 1,0 0,2 1,0 0,2 1,3 0,1 1,0 0,0 0,9 0,2 1,2 0,2 HP0329 NH(3)-dependent NAD+ synthetase 1,4 0,3 1,0 0,1 1,1 0,2 1,1 0,1 1,2 0,2 0,8 0,2 1,0 0,1 0,9 0,0 0,8 0,0 0,8 0,1 1,1 0,1 0,7 0,0 1,4 0,5 0,8 0,2 HP0330 ketol-acid reductoisomerase 1,4 0,2 0,9 0,2 1,5 0,1 0,9 0,2 0,9 0,2 0,7 0,1 0,9 0,0 0,9 0,0 0,8 0,1 1,2 0,1 0,9 0,1 1,1 0,4 0,9 0,1 0,8 0,1 HP0331 cell division inhibitor 1,6 0,2 0,9 0,1 1,0 0,1 0,9 0,1 1,1 0,3 0,7 0,1 1,0 0,2 1,1 0,1 0,9 0,1 1,3 0,3 0,8 0,2 1,5 0,2 1,2 0,2 0,8 0,1 Hp0332 no description available 1,2 0,2 1,0 0,1 1,0 0,1 0,9 0,1 1,0 0,3 0,5 0,0 1,0 0,1 1,1 0,1 0,8 0,1 1,5 0,2 1,0 0,2 0,9 0,0 1,0 0,1 1,2 0,1 HP0333 DNA processing chain A 0,7 0,0 1,0 0,2 0,8 0,1 1,0 0,1 1,0 0,3 0,6 0,1 0,9 0,3 1,0 0,1 0,8 0,1 1,4 0,2 1,0 0,2 1,2 0,4 0,9 0,3 1,0 0,1 HP0334 conserved hypothetical protein 1,0 0,1 0,9 0,3 1,4 0,2 1,1 0,1 1,0 0,2 0,8 0,0 1,2 0,1 1,0 0,1 0,7 0,1 1,2 0,1 0,5 0,1 0,9 0,1 0,9 0,1 0,6 0,1 HP0335 hypothetical protein 1,3 0,1 0,9 0,1 1,2 0,1 1,0 0,2 1,0 0,4 0,8 0,0 1,0 0,0 1,0 0,1 1,9 0,7 1,3 0,1 0,9 0,0 0,9 N.D. 1,2 0,0 0,8 0,1 HP0336 no description available N.D. N.D. 0,9 0,2 N.D. N.D. 0,9 0,2 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0337 hypothetical protein 0,8 0,1 1,1 0,2 0,8 0,1 1,1 0,1 0,9 0,4 0,7 0,1 0,9 0,1 1,2 0,1 1,3 0,3 1,0 0,2 0,6 0,0 0,7 N.D. 1,1 0,3 0,7 N.D. HP0338 hypothetical protein 0,8 0,0 0,9 0,1 0,8 0,1 1,3 0,2 0,9 0,2 0,7 0,1 1,0 0,2 1,3 0,2 0,9 0,0 1,2 0,3 0,7 0,1 0,7 0,1 1,0 0,2 0,6 0,1 HP0339 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 0,9 0,0 N.D. N.D. 0,9 0,0 1,1 0,3 1,0 0,3 1,0 0,0 N.D. N.D. 1,2 0,1 1,0 N.D. 0,6 N.D. 0,7 0,2 HP0340 hypothetical protein N.D. N.D. 0,9 0,3 N.D. N.D. 0,9 0,2 N.D. N.D. 0,9 0,0 1,0 N.D. 0,9 0,1 N.D. N.D. N.D. N.D. 1,0 0,1 1,0 N.D. 1,1 0,1 0,9 N.D. HP0342 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 1,1 0,2 N.D. N.D. N.D. N.D. N.D. N.D. 1,2 0,2 1,2 0,1 N.D. N.D. 1,2 0,1 0,8 N.D. N.D. N.D. 1,3 N.D. HP0343 hypothetical protein N.D. N.D. 1,1 0,1 N.D. N.D. 1,0 0,1 N.D. N.D. N.D. N.D. 1,5 N.D. 0,9 0,1 N.D. N.D. 0,7 N.D. N.D. N.D. 1,0 N.D. 1,0 0,2 1,5 N.D. HP0344 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 0,9 0,1 1,1 0,1 N.D. N.D. 0,8 N.D. 0,9 0,0 1,3 0,2 N.D. N.D. 1,2 0,0 1,1 N.D. 1,3 0,3 0,7 N.D. HP0345 hypothetical protein N.D. N.D. 0,7 0,3 N.D. N.D. 0,9 0,2 N.D. N.D. N.D. N.D. 0,9 0,1 1,3 0,4 1,0 N.D. 0,9 0,1 1,5 0,3 1,0 N.D. 1,0 0,1 1,3 N.D. HP0346 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 1,0 0,1 N.D. N.D. 0,8 N.D. 1,0 N.D. 0,9 0,0 1,1 0,1 1,2 0,2 1,0 0,0 1,2 N.D. 1,0 0,1 N.D. N.D. HP0347 conserved hypothetical protein 0,7 0,1 1,0 0,1 0,8 0,2 1,2 0,1 1,0 0,3 0,9 0,1 1,0 0,1 0,9 0,1 1,3 0,3 1,2 0,1 1,4 0,2 1,1 0,0 1,0 0,2 1,0 N.D. HP0348 single-stranded-dna-specific exonuclease 0,7 0,1 1,0 0,2 0,9 0,1 1,0 0,1 1,1 0,2 0,8 0,1 1,0 0,1 1,0 0,1 0,9 0,0 4,8 0,3 0,8 0,1 0,9 N.D. 1,1 0,2 0,7 0,1 HP0349 CTP synthetase 0,8 0,0 1,0 0,2 0,7 0,1 1,0 0,2 1,1 0,4 1,0 0,1 1,0 0,3 0,8 0,1 0,9 0,1 1,2 0,1 0,8 0,1 1,3 0,2 1,0 0,2 0,8 0,2 HP0350 hypothetical protein 1,3 0,1 1,4 0,2 1,6 0,0 1,2 0,2 0,9 0,2 0,9 0,0 0,9 0,2 0,9 0,1 1,1 0,2 1,2 0,3 0,8 0,1 1,6 0,3 1,0 0,1 0,9 0,3 HP0351 Basal body M-ring protein (flif ) 1,0 0,1 1,2 0,1 1,2 0,1 1,1 0,2 1,0 0,1 1,3 0,1 1,0 0,1 0,8 0,0 1,4 0,3 0,8 0,1 1,0 0,2 0,8 N.D. 0,9 0,1 0,7 0,3 HP0352 Flagellar motor switch protein (flih ) 1,1 0,1 1,2 0,1 1,0 0,0 1,2 0,1 1,0 0,2 1,0 0,2 0,9 0,2 1,2 0,1 1,0 0,1 1,0 0,2 1,2 0,2 1,1 0,1 0,9 0,0 0,9 0,1 HP0353 flagellar export protein 1,1 0,0 1,0 0,1 0,9 N.D. 1,0 0,2 1,1 0,3 0,9 0,1 0,9 0,2 1,4 0,1 1,1 0,1 1,0 0,0 1,2 0,1 1,0 N.D. 1,0 0,1 1,0 0,3 HP0354 deoxyxylulose-5-phosphate synthase, putative 1,2 0,1 1,1 0,0 1,3 0,1 1,1 0,1 1,0 0,3 0,9 0,2 1,1 0,2 1,1 0,1 1,1 0,3 0,8 0,0 1,3 0,0 1,0 N.D. 0,9 0,1 0,6 0,1 HP0355 GTP-binding membrane protein 1,1 0,1 1,0 0,0 1,2 0,2 1,1 0,0 1,0 0,2 1,2 0,2 0,9 0,1 1,0 0,0 0,8 0,1 0,8 0,1 1,1 0,1 1,0 0,1 1,0 0,5 0,8 0,2 HP0356 hypothetical protein 0,8 0,1 1,0 0,3 N.D. N.D. 1,0 0,1 1,1 0,5 1,1 0,2 0,9 0,1 1,6 0,0 0,9 0,2 0,8 0,3 1,0 0,0 1,1 0,4 0,9 0,2 0,8 0,1 HP0357 short chain alcohol dehydrogenase 1,3 0,2 0,9 0,1 0,9 0,1 1,0 0,1 1,1 0,2 0,9 0,0 0,9 0,1 0,9 0,0 1,1 0,1 1,0 0,1 0,9 0,1 1,2 0,1 1,2 0,1 0,7 0,1 HP0358 hypothetical protein 0,7 0,1 0,9 0,2 0,6 0,1 1,0 0,1 0,9 0,2 0,9 0,2 1,3 0,4 1,1 0,2 1,1 0,1 1,3 0,2 1,0 0,1 1,0 0,1 1,2 0,0 1,1 0,2 HP0359 hypothetical protein 1,0 N.D. 1,0 0,1 1,0 0,1 1,1 0,1 1,0 0,2 0,7 0,1 1,1 0,0 1,0 0,1 0,9 0,0 0,8 N.D. 1,0 0,1 0,8 N.D. 1,1 0,0 1,1 N.D. HP0360 UDP-glucose 4-epimerase 0,8 0,0 0,7 0,3 1,0 0,2 0,9 0,1 0,9 0,3 1,0 0,1 1,2 0,1 0,9 0,1 0,9 0,1 1,0 0,1 1,0 0,2 1,3 0,3 1,1 0,1 1,0 0,1 HP0361 pseudouridylate synthase I 0,7 0,0 1,0 0,1 0,8 0,0 1,0 0,1 0,7 0,3 0,8 0,1 1,0 0,1 1,2 0,1 1,0 0,1 1,0 0,0 0,8 0,0 1,5 N.D. 1,0 0,3 1,7 0,4 HP0362 conserved hypothetical integral membrane protein 0,7 0,0 0,9 0,1 0,8 0,1 0,9 0,2 1,0 0,2 0,8 0,0 1,1 0,1 1,1 0,1 0,8 0,0 1,2 0,1 0,7 0,1 0,9 N.D. 0,9 0,2 0,7 0,1 HP0363 L-isoaspartyl-protein carboxyl methyltransferase 0,8 0,0 1,1 0,2 0,7 0,1 1,0 0,1 1,1 0,2 0,5 0,1 0,9 0,1 1,1 0,1 0,8 0,1 1,0 0,1 1,3 0,2 1,2 0,2 0,9 0,2 0,7 0,3 HP0364 ribonucleoside diphosphate reductase, beta subunit 0,7 0,0 0,9 0,1 0,9 0,1 0,9 0,1 1,0 0,2 1,0 0,1 1,0 0,3 1,3 0,1 1,1 0,1 1,2 0,2 1,3 0,1 1,0 0,2 1,2 0,2 0,7 0,0 HP0366 spore coat polysaccharide biosynthesis protein C 0,6 0,0 0,8 0,1 0,7 0,1 0,9 0,2 1,5 0,6 0,8 0,0 3,8 2,1 1,1 0,1 1,5 0,1 1,2 0,5 1,4 0,5 1,4 N.D. 0,5 0,2 4,6 2,5 HP0367 hypothetical protein 1,0 0,1 0,8 0,2 1,2 0,0 1,0 0,2 1,2 0,3 1,4 0,3 3,5 1,4 2,7 0,3 1,5 0,2 2,1 1,0 2,0 0,4 3,3 0,7 0,1 0,1 4,0 0,9 HP0368 hypothetical protein N.D. N.D. 0,9 0,1 N.D. N.D. 1,0 0,2 1,4 0,4 0,9 0,0 1,2 0,2 1,2 0,1 1,4 0,2 0,8 0,1 1,2 0,0 1,0 N.D. 1,0 0,2 1,1 0,2 HP0369 hypothetical protein N.D. N.D. 1,0 0,1 N.D. N.D. 1,0 0,1 1,2 0,3 0,9 0,2 1,6 0,6 1,1 0,1 1,4 0,2 0,8 0,1 1,1 0,1 1,2 N.D. 0,9 0,3 N.D. N.D. HP0370 biotin carboxylase 0,9 0,1 1,1 0,2 1,1 0,1 1,1 0,1 1,0 0,3 1,0 0,3 0,8 0,1 1,0 0,0 0,9 0,1 0,7 0,1 1,4 0,3 1,0 0,0 1,0 0,4 1,4 N.D. HP0371 biotin carboxyl carrier protein 1,3 0,2 1,1 0,1 1,4 0,1 1,2 0,3 1,1 0,2 1,0 0,3 0,8 0,1 1,0 0,1 1,0 0,3 0,9 0,1 1,5 0,6 0,8 0,1 1,1 0,2 0,6 0,1 HP0372 deoxycytidine triphosphate deaminase 1,0 0,1 0,9 0,1 1,1 0,1 0,9 0,2 1,0 0,2 1,2 0,1 0,9 0,1 0,9 0,1 1,0 0,1 0,7 0,1 1,0 0,1 0,9 0,1 1,2 0,2 0,6 0,0 HP0373 conserved hypothetical protein 1,5 0,0 0,9 0,2 2,1 0,0 0,9 0,2 1,2 0,3 0,8 0,1 1,1 0,1 1,1 0,1 0,6 0,1 0,7 0,1 0,8 0,0 0,8 N.D. 1,0 0,2 1,4 0,2 HP0374 conserved hypothetical protein 1,3 0,0 1,0 0,1 1,3 0,2 1,0 0,1 1,1 0,2 1,0 0,2 0,9 0,1 1,0 0,0 0,8 0,2 1,1 0,2 1,0 0,1 1,2 0,3 1,0 0,3 1,1 0,6 HP0375 hypothetical protein 1,0 0,0 0,9 0,1 1,0 0,2 0,9 0,2 1,1 0,3 0,9 0,2 1,1 0,1 1,2 0,1 0,6 0,1 1,6 0,3 0,7 0,2 1,2 0,3 1,0 0,2 0,9 0,1 HP0376 ferrochelatase 0,9 0,0 1,4 0,5 0,8 0,1 0,7 0,3 1,1 0,3 0,9 0,0 1,0 0,2 1,3 0,2 1,2 0,4 1,1 0,1 1,9 1,0 0,8 N.D. 1,4 0,6 1,3 0,1 HP0377 thiol:disulfide interchange protein (dsbc), putative 0,8 0,0 0,8 0,2 1,0 0,1 0,8 0,1 0,9 0,4 1,0 0,1 1,0 0,3 0,9 0,1 1,1 0,2 1,7 0,2 0,7 0,0 1,4 0,4 1,0 0,2 1,5 0,3 HP0378 cytochrome c biogenesis protein 0,7 0,0 1,2 0,1 0,7 0,1 1,0 0,2 0,9 0,2 1,1 0,2 0,9 0,1 1,1 0,1 0,9 0,1 2,1 0,6 1,0 0,2 1,3 0,0 1,2 0,1 1,8 0,5 HP0379 fucosyltransferase 0,8 0,0 1,1 0,1 0,8 0,1 1,0 0,0 0,9 0,3 1,2 0,0 0,9 0,1 1,4 0,1 0,8 0,1 1,8 0,5 1,0 0,2 1,8 0,7 1,2 0,2 1,7 0,5 HP0380 glutamate dehydrogenase 0,9 0,1 0,9 0,1 0,6 0,1 0,9 0,1 1,1 0,3 1,0 0,1 1,0 0,2 1,0 0,1 1,3 0,1 1,1 0,1 1,3 0,1 1,1 0,1 1,2 0,2 1,0 0,0 HP0381 HemK family methylase, putative 0,9 0,0 0,9 0,2 0,9 0,1 1,0 0,3 1,1 0,3 0,9 0,0 1,1 0,1 1,0 0,1 0,9 0,1 1,6 0,4 1,2 0,4 0,9 0,1 1,0 0,1 1,5 0,1 HP0382 zinc-metallo protease 0,8 0,1 0,9 0,1 0,9 0,0 0,9 0,1 1,0 0,4 1,0 0,1 1,2 0,1 0,9 0,1 0,8 0,1 0,9 0,1 0,8 0,1 0,8 0,1 0,9 0,1 0,9 0,1 HP0383 hypothetical protein 1,2 0,1 1,4 0,3 0,9 0,0 1,1 0,1 1,0 0,3 1,0 0,1 1,1 0,2 1,4 0,2 1,5 0,2 1,4 0,3 1,5 0,2 1,3 0,2 1,2 0,8 1,2 0,3 HP0384 hypothetical protein 1,0 0,2 0,7 0,4 1,1 0,1 0,9 0,4 1,1 0,1 1,0 0,0 1,2 0,1 1,0 0,1 1,3 0,1 0,9 0,0 1,1 0,0 0,9 0,1 0,9 0,1 1,5 0,1 HP0385 hypothetical protein 0,9 0,0 1,1 0,1 1,0 0,0 1,0 0,1 0,9 0,1 1,0 0,2 1,1 0,2 1,0 0,1 1,4 0,3 N.D. N.D. 1,5 0,3 1,0 N.D. 1,3 0,3 0,8 N.D. HP0386 hypothetical protein 0,9 0,1 1,0 0,2 1,3 N.D. 1,3 0,4 0,9 0,3 0,9 0,2 1,0 0,1 1,1 0,0 1,3 0,3 0,7 0,0 1,3 0,1 1,1 N.D. 1,3 0,2 1,5 0,2 HP0387 no description available N.D. N.D. 1,0 0,2 N.D. N.D. 0,9 0,2 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0388 conserved hypothetical protein 1,2 0,2 0,8 0,2 1,4 0,3 0,9 0,1 1,0 0,2 1,0 0,2 1,3 0,3 1,1 0,0 0,6 0,1 1,6 0,1 0,5 0,1 1,6 0,0 1,0 0,3 0,8 0,1 HP0389 superoxide dismutase 1,8 0,2 0,5 0,2 2,1 0,1 0,6 0,1 1,0 0,3 1,1 0,3 1,8 0,8 0,9 0,0 0,7 0,1 1,1 0,2 0,4 0,1 1,7 0,2 0,6 0,1 1,5 0,4 HP0390 adhesin-thiol peroxidase 1,4 0,2 0,9 0,4 1,0 0,2 0,9 0,2 1,1 0,3 1,4 0,2 1,2 0,1 0,7 0,1 2,0 0,5 1,5 1,0 0,9 0,2 1,9 0,2 1,9 0,4 0,9 0,2 HP0391 chemotaxis adaptor protein (chew) 1,0 0,0 0,9 0,1 1,2 0,2 0,9 0,1 1,3 0,5 0,8 0,0 1,2 0,1 1,0 0,1 1,1 0,2 1,4 0,2 0,7 0,2 1,1 0,0 0,9 0,0 1,2 0,2 HP0392 receptor coupled histidine kinase (chea ) 0,7 0,0 0,9 0,0 0,7 0,1 1,0 0,2 1,1 0,4 1,1 0,0 1,0 0,2 0,9 0,1 1,0 0,1 1,2 0,1 0,9 0,1 1,1 0,2 1,0 0,3 1,1 0,3 HP0393 hybrid CheW-CheY chemotaxis protein (chev3 ) 0,9 0,0 0,9 0,2 0,8 0,1 0,9 0,2 1,0 0,3 1,0 0,0 1,1 0,1 1,1 0,1 1,0 0,0 1,2 0,2 0,8 0,1 1,2 0,3 1,0 0,3 1,6 0,4 HP0394 hypothetical protein 0,7 0,0 1,4 0,1 0,7 0,1 1,2 0,2 1,0 0,4 1,0 0,1 0,9 0,3 1,3 0,1 1,1 0,1 1,2 0,3 1,2 0,0 N.D. N.D. 1,5 0,4 3,2 0,4 HP0395 conserved hypothetical protein 0,7 0,0 0,9 0,1 0,7 0,1 0,9 0,1 1,0 0,3 1,0 0,0 1,0 0,3 1,3 0,1 1,0 0,1 1,7 0,6 1,5 0,5 1,2 0,4 1,2 0,6 1,0 0,2 HP0396 no description available N.D. N.D. 1,1 0,1 N.D. N.D. 1,0 0,2 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0397 phosphoglycerate dehydrogenase 0,7 0,0 0,7 0,2 0,7 0,1 1,0 0,1 0,9 0,2 0,9 0,1 1,0 0,1 1,0 0,1 1,0 0,1 1,2 0,2 1,4 0,4 0,9 0,1 0,9 0,1 0,9 0,0 HP0398 hypothetical protein 0,4 0,0 1,0 0,2 0,5 0,0 1,1 0,3 0,9 0,3 0,8 0,0 0,9 0,2 0,8 0,1 0,9 0,2 1,6 0,1 1,2 0,3 0,9 0,2 0,9 0,2 0,9 0,2 HP0399 no description available N.D. N.D. 1,0 0,1 N.D. N.D. 1,0 0,1 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0400 penicillin tolerance protein 1,1 0,3 1,1 0,2 1,0 0,1 1,0 0,2 1,0 0,2 1,0 0,0 1,1 0,2 1,0 0,0 1,2 0,1 1,2 0,1 1,2 0,1 1,2 0,2 1,2 0,1 0,9 N.D. HP phosphoshikimate 1-carboxyvinyltransferase 0,8 0,2 0,8 0,2 0,8 0,0 1,0 0,1 1,0 0,2 1,2 0,2 1,0 0,1 1,0 0,1 1,2 0,2 1,1 0,2 1,5 0,3 1,3 0,1 0,9 0,1 1,3 0,0 HP0402 no description available 0,7 0,1 1,2 0,1 1,0 N.D. 1,1 0,1 1,1 0,2 1,1 0,3 1,1 0,3 0,9 0,0 1,1 0,0 1,1 0,1 1,0 0,1 0,9 N.D. 1,0 0,1 0,8 0,2 HP0403 phenylalanyl-trna synthetase, alpha subunit 0,8 0,1 0,9 0,1 0,8 0,1 0,9 0,1 1,0 0,1 0,9 0,1 0,9 0,2 0,9 0,1 1,6 0,3 0,7 0,1 1,7 0,5 0,9 0,1 1,2 0,3 1,1 0,5 HP0404 protein kinase C inhibitor 1,0 0,1 0,8 0,1 1,1 0,1 0,9 0,0 0,9 0,2 1,0 0,2 1,1 0,1 0,9 0,1 1,1 0,2 1,0 0,2 0,9 0,1 1,1 0,1 1,3 0,3 1,2 0,0 HP0405 nifs-like protein 1,2 0,3 1,0 0,1 1,6 0,0 1,0 0,1 0,9 0,2 0,8 0,1 1,2 0,3 0,9 0,1 0,9 0,3 1,1 0,1 0,9 0,4 1,2 0,1 0,7 0,1 1,6 0,3 HP0406 hypothetical protein 1,2 0,2 1,2 0,4 1,0 0,1 1,0 0,0 1,0 0,2 1,0 0,1 1,0 0,1 0,9 0,1 1,3 0,1 1,0 0,3 1,5 0,4 1,3 0,1 1,5 0,4 0,9 0,1 HP0407 no description available N.D. N.D. 0,8 0,2 N.D. N.D. 1,0 0,1 N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. N.D. HP0408 hypothetical protein 0,7 0,1 0,8 0,2 0,8 0,1 0,9 0,2 0,9 0,3 0,8 0,1 1,0 0,1 1,0 0,1 1,0 0,1 1,0 0,1 0,9 0,1 1,1 0,0 0,8 0,2 1,0 N.D. HP0409 GMP synthase 0,8 0,1 1,0 0,1 1,0 0,1 1,0 0,1 0,9 0,2 1,0 0,0 1,0 0,1 1,0 0,1 0,8 0,1 1,2 0,2 0,8 0,2 1,0 0,0 0,9 0,1 0,7 0,2 HP0410 Flagellar sheath associated protein (hpa2 ) 0,7 0,0 0,9 0,1 0,7 0,1 0,9 0,1 0,7 0,3 0,9 0,2 1,0 0,2 1,0 0,1 2,1 0,9 1,0 0,0 1,6 0,3 1,1 0,2 0,8 0,2 0,9 0,3 HP0411 hypothetical protein 2,4 0,1 1,4 0,2 1,9 0,0 0,6 0,0 1,4 0,6 0,8 0,1 1,1 0,1 1,0 0,2 1,1 0,1 0,9 0,1 1,2 0,5 1,0 0,1 1,0 0,4 0,5 0,1 HP0412 hypothetical protein N.D. N.D. 1,5 0,4 1,0 N.D. 0,7 0,3 0,9 0,4 0,6 0,0 1,1 0,3 1,0 0,1 0,8 0,3 2,4 0,6 0,9 0,2 0,8 N.D. 1,3 0,5 1,2 0,5 HP0413 transposase-like protein, PS3IS N.D. N.D. 0,9 0,3 N.D. N.D. 1,0 0,1 N.D. N.D. 0,8 0,1 0,9 N.D. 1,0 0,1 1,0 0,0 0,9 0,2 0,9 0,1 0,9 N.D. 1,1 0,1 1,0 0,1
Regulation der Glykolyse: Phosphofructokinase
 Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
11 ANHANG Haushaltsgene
 11 ANHANG 11.1 Haushaltsgene Tabelle 11.1: Auflistung der Gene, die durch ESTs repräsentiert werden (Bezeichnung und UniGene Accession Nummer). Diese ESTs wurden zur Normalisierung der Mikroarray-Experimente
11 ANHANG 11.1 Haushaltsgene Tabelle 11.1: Auflistung der Gene, die durch ESTs repräsentiert werden (Bezeichnung und UniGene Accession Nummer). Diese ESTs wurden zur Normalisierung der Mikroarray-Experimente
Biologische Katalysatoren
 Biologische Katalysatoren Die Entdeckung der biologischen Katalysatoren (Enzyme) ist eng mit der Entstehungsgeschichte der Biochemie verknüpft: Ende 19. Jhdt: Speichel - Fleischverdauung durch Magensäfte
Biologische Katalysatoren Die Entdeckung der biologischen Katalysatoren (Enzyme) ist eng mit der Entstehungsgeschichte der Biochemie verknüpft: Ende 19. Jhdt: Speichel - Fleischverdauung durch Magensäfte
52: Welche der Coenzyme sind an der oxidativen Decarboxylierung von Pyruvat beteiligt? 1) Thiaminpyrophosphat
 2. Testat Biochemie 93/94 - Teil B (Leider ist dies nur ein Teil der Fragen!) 52: Welche der Coenzyme sind an der oxidativen Decarboxylierung von Pyruvat beteiligt? 1) Thiaminpyrophosphat 3) Biotin 2)
2. Testat Biochemie 93/94 - Teil B (Leider ist dies nur ein Teil der Fragen!) 52: Welche der Coenzyme sind an der oxidativen Decarboxylierung von Pyruvat beteiligt? 1) Thiaminpyrophosphat 3) Biotin 2)
Gluconeognese Neusynthese von Glucose aus Pyruvat
 Gluconeognese Neusynthese von Glucose aus Pyruvat Warum notwendig? Das Gehirn ist auf eine konstante Versorgung mit Glucose angewiesen. Eine Unterzuckerung (< 3 4 mmol/l) führt unweigerlich zur Bewußtlosigkeit
Gluconeognese Neusynthese von Glucose aus Pyruvat Warum notwendig? Das Gehirn ist auf eine konstante Versorgung mit Glucose angewiesen. Eine Unterzuckerung (< 3 4 mmol/l) führt unweigerlich zur Bewußtlosigkeit
Molekulargenetik der Eukaryoten WS 2014/15, VL 12. Erwin R. Schmidt Institut für Molekulargenetik
 Molekulargenetik der Eukaryoten WS 2014/15, V 12 Erwin R. Schmidt Institut für Molekulargenetik Der basale Transkriptionskomplex plus genspezifische TFs Zusammenfassung: Präinitiationskomplexe der verschiedenen
Molekulargenetik der Eukaryoten WS 2014/15, V 12 Erwin R. Schmidt Institut für Molekulargenetik Der basale Transkriptionskomplex plus genspezifische TFs Zusammenfassung: Präinitiationskomplexe der verschiedenen
Biochemie II - Tutorium
 Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 04.01.2016 Ablauf des Tutoriums Einführung und Wiederholung Vorlesungszusammenfassung Übungsaufgaben Selbststudium
Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 04.01.2016 Ablauf des Tutoriums Einführung und Wiederholung Vorlesungszusammenfassung Übungsaufgaben Selbststudium
DNA-Replikation. Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination
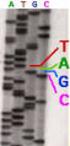 DNA-Replikation Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination Die Initiation der DNA-Replikation bei Eukaryoten am ori erfolgt erst nach der Lizensierung durch ORC und weitere Proteine
DNA-Replikation Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination Die Initiation der DNA-Replikation bei Eukaryoten am ori erfolgt erst nach der Lizensierung durch ORC und weitere Proteine
Biochemie (für Bioinformatiker) WS 2010/2011, 2. Klausur (50 Punkte)
 Datum: 07.02.2011 Name: Matrikel-Nr.: Vorname: Studiengang: Bioinformatik Biochemie (für Bioinformatiker) WS 2010/2011, 2. Klausur (50 Punkte) Modulnr.: FMI-BI0027 Hiermit bestätige ich meine Prüfungstauglichkeit.
Datum: 07.02.2011 Name: Matrikel-Nr.: Vorname: Studiengang: Bioinformatik Biochemie (für Bioinformatiker) WS 2010/2011, 2. Klausur (50 Punkte) Modulnr.: FMI-BI0027 Hiermit bestätige ich meine Prüfungstauglichkeit.
Atmung Übersicht. Atmung der Mitochondrien
 Atmung der Mitochondrien Atmung Übersicht e - Transportkette REAKTION: C 6 H 12 O 6 + 6O 2 6CO 2 + 6H 2 O + Energie (Glucose) (Sauerstoff) (Kohlendioxid) (Wasser) Nur ca. 40% der Energie wird zu ATP Der
Atmung der Mitochondrien Atmung Übersicht e - Transportkette REAKTION: C 6 H 12 O 6 + 6O 2 6CO 2 + 6H 2 O + Energie (Glucose) (Sauerstoff) (Kohlendioxid) (Wasser) Nur ca. 40% der Energie wird zu ATP Der
Translation benötigt trnas und Ribosomen. Genetischer Code. Initiation Elongation Termination
 8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
Klausurbogen im Studiengang B.Sc. Biologie Modul BSc-Bio-7, Teilklausur Biochemie vom
 Johann Wolfgang Goethe-Universität Frankfurt am Main Deckblatt Fachbereich Biowissenschaften Klausurbogen im Studiengang B.Sc. Biologie Modul BSc-Bio-7, Teilklausur Biochemie vom 08.12.2008 Name: Vorname:
Johann Wolfgang Goethe-Universität Frankfurt am Main Deckblatt Fachbereich Biowissenschaften Klausurbogen im Studiengang B.Sc. Biologie Modul BSc-Bio-7, Teilklausur Biochemie vom 08.12.2008 Name: Vorname:
1 Einleitung 1. 2 Materialien und Methoden 17
 Inhaltsverzeichnis 1 Einleitung 1 1.1 Kurze Darstellung der Hitzestreßantwort pflanzlicher Systeme 1 1.2 Die Hitzeschockproteine 4 1.3 Signaltransduktion in Streßsituationen 14 1.4 Zielstellung 16 2 Materialien
Inhaltsverzeichnis 1 Einleitung 1 1.1 Kurze Darstellung der Hitzestreßantwort pflanzlicher Systeme 1 1.2 Die Hitzeschockproteine 4 1.3 Signaltransduktion in Streßsituationen 14 1.4 Zielstellung 16 2 Materialien
Pharmazeutische Biologie Grundlagen der Biochemie
 harmazeutische Biologie Grundlagen der Biochemie A Enzyme E1 E2 E3 E4 Biosynthese A B D E B E7 E2 E6 E1 E3 E5 E4 E1 E2 E5 E4 rof. Dr. Theo Dingermann Institut für harmazeutische Biologie Goethe-Universität
harmazeutische Biologie Grundlagen der Biochemie A Enzyme E1 E2 E3 E4 Biosynthese A B D E B E7 E2 E6 E1 E3 E5 E4 E1 E2 E5 E4 rof. Dr. Theo Dingermann Institut für harmazeutische Biologie Goethe-Universität
Translation benötigt trnas und Ribosomen. Genetischer Code. Initiation Elongation Termination
 8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
Vertiefendes Seminar zur Vorlesung Biochemie I Bearbeitung Übungsblatt 5
 Vertiefendes Seminar zur Vorlesung Biochemie I 27.11.2015 Bearbeitung Übungsblatt 5 Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/
Vertiefendes Seminar zur Vorlesung Biochemie I 27.11.2015 Bearbeitung Übungsblatt 5 Gerhild van Echten-Deckert Fon. +49-228-732703 Homepage: http://www.limes-institut-bonn.de/forschung/arbeitsgruppen/unit-3/
BIOCHEMIE DER ERNÄHRUNG II Oxidation von Lipiden. Gastrointestinaltrakt. Fettsäureoxidation zu Acetyl-CoA
 xidation von Lipiden ß-xidation: Fettsäureoxidation zu Acetyl-oA BIEMIE DER ERNÄRUNG II 07.05.2012 wichtigste Fettsäurequelle in Nahrung: Triacylglyceride Import freier Fettsäuren ins ytosol oder Erzeugung
xidation von Lipiden ß-xidation: Fettsäureoxidation zu Acetyl-oA BIEMIE DER ERNÄRUNG II 07.05.2012 wichtigste Fettsäurequelle in Nahrung: Triacylglyceride Import freier Fettsäuren ins ytosol oder Erzeugung
Kataboler und Anaboler Stoffwechsel
 Vorlesung Vom Molekül zur Zelle Ao.Univ.Prof. Dr. Georg Weitzer Fortsetzung von Block 3 nach Prof. Müllner, ab 8.1. Kataboler und Anaboler Stoffwechsel Aktuelle Folien unter http://homepage.univie.ac.at/georg.weitzer/lectures.html
Vorlesung Vom Molekül zur Zelle Ao.Univ.Prof. Dr. Georg Weitzer Fortsetzung von Block 3 nach Prof. Müllner, ab 8.1. Kataboler und Anaboler Stoffwechsel Aktuelle Folien unter http://homepage.univie.ac.at/georg.weitzer/lectures.html
3.4 Identifizierung von genetisch kartierten Proteinspots durch Massenspektrometrie
 3.4 von genetisch kartierten Proteinspots durch Massenspektrometrie Zur der kartierten Proteinspots mit MALDI Massenspektrometrie wurden Proteinvarianten aus vier silbergefärbten 2D-Gelen (Shevchenko et
3.4 von genetisch kartierten Proteinspots durch Massenspektrometrie Zur der kartierten Proteinspots mit MALDI Massenspektrometrie wurden Proteinvarianten aus vier silbergefärbten 2D-Gelen (Shevchenko et
Genexpressionsanalyse von DNA-Reparaturgenen in primären. Fibroblasten von Tumorpatienten mittels Real Time-PCR
 Aus dem Institut für Humangenetik der Universitätsmedizin der Johannes Gutenberg-Universität Mainz Genexpressionsanalyse von DNA-Reparaturgenen in primären Fibroblasten von Tumorpatienten mittels Real
Aus dem Institut für Humangenetik der Universitätsmedizin der Johannes Gutenberg-Universität Mainz Genexpressionsanalyse von DNA-Reparaturgenen in primären Fibroblasten von Tumorpatienten mittels Real
Es ist die Zeit gekommen, zu verstehen, wie es zur Proteinbiosynthese kommt?! Wobei jeweils eine AS von 3 Basen codiert wird..
 Proteinbiosynthese Es ist die Zeit gekommen, zu verstehen, wie es zur Proteinbiosynthese kommt?! Alle Proteine, sind über die DNA codiert Wobei jeweils eine AS von 3 Basen codiert wird.. GENETISCHER CODE
Proteinbiosynthese Es ist die Zeit gekommen, zu verstehen, wie es zur Proteinbiosynthese kommt?! Alle Proteine, sind über die DNA codiert Wobei jeweils eine AS von 3 Basen codiert wird.. GENETISCHER CODE
Citratzyklus. Citratzyklus
 Der hat in der Zelle verschiedene Aufgaben. Teilschritte werden z.b. bei manchen Gärungen eingesetzt (Methyl-Malonyl-CoA-Weg). Er ist wichtig zur Bereitstellung verschiedener Vorstufen für Biosynthesen,
Der hat in der Zelle verschiedene Aufgaben. Teilschritte werden z.b. bei manchen Gärungen eingesetzt (Methyl-Malonyl-CoA-Weg). Er ist wichtig zur Bereitstellung verschiedener Vorstufen für Biosynthesen,
Die innere Mitochondrienmebran ist durchlässig für: 1. Pyruvat 2. Malat 3. Aspartat 4. Citrat
 Der Malat-Shuttle Die innere Mitochondrienmebran ist durchlässig für: 1. Pyruvat 2. Malat 3. Aspartat 4. Citrat Die innere Mitochondrienmembran ist undurchlässig für: 1. Wasserstoffatomen > 2. Acetyl-
Der Malat-Shuttle Die innere Mitochondrienmebran ist durchlässig für: 1. Pyruvat 2. Malat 3. Aspartat 4. Citrat Die innere Mitochondrienmembran ist undurchlässig für: 1. Wasserstoffatomen > 2. Acetyl-
KOHLENHYDRATE. Die Glykolyse
 Die Glykolyse Hexokinase Glucose Kostet 1ATP Mg 2+ Glucose-6-P Die Glucokinase kann durch Insulin induziert werden in : 1) Den Fettzellen 2) Den ß-Zellen des Pankreas 3) Der Nierenrinde 4) Der Leber Die
Die Glykolyse Hexokinase Glucose Kostet 1ATP Mg 2+ Glucose-6-P Die Glucokinase kann durch Insulin induziert werden in : 1) Den Fettzellen 2) Den ß-Zellen des Pankreas 3) Der Nierenrinde 4) Der Leber Die
mrna S/D UTR: untranslated region orf: open reading frame S/D: Shine-Dalgarno Sequenz
 1. Nennen Sie die verschiedenen RNA-Typen, die bei der Translation wichtig sind. Erklären Sie die Funktion der verschiedenen RNA-Typen. Skizzieren Sie die Struktur der verschiedenen RNA-Typen und bezeichnen
1. Nennen Sie die verschiedenen RNA-Typen, die bei der Translation wichtig sind. Erklären Sie die Funktion der verschiedenen RNA-Typen. Skizzieren Sie die Struktur der verschiedenen RNA-Typen und bezeichnen
1) Erklären sie die Begriffe Primär Sekundär und Tertiärstruktur von Proteinen. Nennen Sie drei typische Sekundärstrukturelemente (6P)
 1. Klausur zum Modul 5.3 Biochemie WS 09/10 12.2.2010 1) Erklären sie die Begriffe Primär Sekundär und Tertiärstruktur von Proteinen. Nennen Sie drei typische Sekundärstrukturelemente (6P) 2) Welche Funktion
1. Klausur zum Modul 5.3 Biochemie WS 09/10 12.2.2010 1) Erklären sie die Begriffe Primär Sekundär und Tertiärstruktur von Proteinen. Nennen Sie drei typische Sekundärstrukturelemente (6P) 2) Welche Funktion
- der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen
 Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Pentosephosphat-Weg: alternativer Abbau von Glucose. Wird auch als Hexosemonophosphatweg (HMW) oder Phosphogluconat-Cyclus bezeichnet.
 Pentosephosphat-Weg: alternativer Abbau von Glucose Wird auch als Hexosemonophosphatweg (HMW) oder Phosphogluconat-Cyclus bezeichnet. Er liefert NADPH und Ribose-5-phosphat. NADPH ist die 2. Währung der
Pentosephosphat-Weg: alternativer Abbau von Glucose Wird auch als Hexosemonophosphatweg (HMW) oder Phosphogluconat-Cyclus bezeichnet. Er liefert NADPH und Ribose-5-phosphat. NADPH ist die 2. Währung der
1. Biochemie-Klausur Zahnmediziner, WS 03/04
 1. Biochemie-Klausur Zahnmediziner, WS 03/04 1. Welche Aussage zur ß-Oxidation von Fettsäuren in Peroxisomen ist falsch? A) Die Aufnahme langkettiger Fettsäuren in die Peroxisomen erfolgt Carnitin-unabhängig!
1. Biochemie-Klausur Zahnmediziner, WS 03/04 1. Welche Aussage zur ß-Oxidation von Fettsäuren in Peroxisomen ist falsch? A) Die Aufnahme langkettiger Fettsäuren in die Peroxisomen erfolgt Carnitin-unabhängig!
Verteilen von Proteinen innerhalb der Zelle
 Verteilen von Proteinen innerhalb der Zelle cytosolische Proteine Proteine, die direkt in Organellen transportiert werden Proteine, die über das ER transportiert werden Regulation der eukaryontischen Genexpression
Verteilen von Proteinen innerhalb der Zelle cytosolische Proteine Proteine, die direkt in Organellen transportiert werden Proteine, die über das ER transportiert werden Regulation der eukaryontischen Genexpression
Teilbereiche der Biophysik. Literatur:
 Teilbereiche der Biophysik Molekulare Biophysik Membranbiophysik Neurobiophysik Zellbiophysik Biomechanik Kybernetik Evolution Strahlenbiophysik Leistungskriterien Übungszettel (bewertet) Klausur (Zulassung
Teilbereiche der Biophysik Molekulare Biophysik Membranbiophysik Neurobiophysik Zellbiophysik Biomechanik Kybernetik Evolution Strahlenbiophysik Leistungskriterien Übungszettel (bewertet) Klausur (Zulassung
1.Frankfurter Consilium in 2015
 Regulationsmechanismen der Glykolyse und Tumorstoffwechsel Glykolyse leicht gemacht 1.Frankfurter Consilium in 2015 Alfons Meyer Facharzt f. Allgemeinmedizin Notfallmedizin, Naturheilverfahren, Akupunktur,
Regulationsmechanismen der Glykolyse und Tumorstoffwechsel Glykolyse leicht gemacht 1.Frankfurter Consilium in 2015 Alfons Meyer Facharzt f. Allgemeinmedizin Notfallmedizin, Naturheilverfahren, Akupunktur,
Organische Chemie der biologischen Stoffwechselwege
 Organische Chemie der biologischen Stoffwechselwege John McMurry / Tadhg Begley aus dem Amerikanischen übersetzt von Karin Beifuss ELSEVIER SPEKTRUM AKADEMISCHER VERLAG Spektrum k_/l AKADEMISCHER VERLAG
Organische Chemie der biologischen Stoffwechselwege John McMurry / Tadhg Begley aus dem Amerikanischen übersetzt von Karin Beifuss ELSEVIER SPEKTRUM AKADEMISCHER VERLAG Spektrum k_/l AKADEMISCHER VERLAG
Um welches Molekül handelt es sich? Was ist dessen Funktion? Benennen Sie die funktionellen Gruppen. (2)
 Prüfungsfragen Biochemie; Teil Schroeder Ad Einheit 1 Vergleichen Sie die Stärke von molekularen Wechselwirkungen in kj/mol. In welcher Größenordnung ist der Abstand dieser Wechselwirkungen? Im Vergleich
Prüfungsfragen Biochemie; Teil Schroeder Ad Einheit 1 Vergleichen Sie die Stärke von molekularen Wechselwirkungen in kj/mol. In welcher Größenordnung ist der Abstand dieser Wechselwirkungen? Im Vergleich
Entropie ist ein Maß für den Ordnungszustand eines thermodynamischen Systems (chem. Reaktion in einem geschlossenen System); kann auch als Maß für
 Entropie ist ein Maß für den Ordnungszustand eines thermodynamischen Systems (chem. Reaktion in einem geschlossenen System); kann auch als Maß für die Nichtumkehrbarkeit (Irreversibilität) eines Vorganges
Entropie ist ein Maß für den Ordnungszustand eines thermodynamischen Systems (chem. Reaktion in einem geschlossenen System); kann auch als Maß für die Nichtumkehrbarkeit (Irreversibilität) eines Vorganges
Was ist denn NAD / NADP??
 Exkurs Oxidative Desaminierung: Was ist denn NAD / NADP?? Exkurs Oxidative Desaminierung: NAD / NADP gehört zum Vitamin B2- Komplex Nikotinsäureamid = Nikotinamid= Vit B3 Exkurs Oxidative Desaminierung:
Exkurs Oxidative Desaminierung: Was ist denn NAD / NADP?? Exkurs Oxidative Desaminierung: NAD / NADP gehört zum Vitamin B2- Komplex Nikotinsäureamid = Nikotinamid= Vit B3 Exkurs Oxidative Desaminierung:
Gunnar Schröder Computational Structural Biology Group Institute of Complex Systems (ICS-6) Forschungszentrum Jülich
 Gunnar Schröder Computational Structural Biology Group Institute of Complex Systems (ICS-6) Forschungszentrum Jülich! gu.schroeder@fz-juelich.de!! http://www.schroderlab.org/vorlesung.html Teilbereiche
Gunnar Schröder Computational Structural Biology Group Institute of Complex Systems (ICS-6) Forschungszentrum Jülich! gu.schroeder@fz-juelich.de!! http://www.schroderlab.org/vorlesung.html Teilbereiche
BIOCHEMIE. Teil 2: Aminosäuren, Proteine, Enzyme. Urheberrechtlich geschütztes Material. Lernkarteikarten Veterinärmedizin - BIOCHEMIE
 Lernkarteikarten Veterinärmedizin - BIOCHEMIE BIOCHEMIE 2014 Teil 2: Aminosäuren, Proteine, Enzyme Vetbrainfood - Tiermedizinische Lernkarteikarten - Diplombiologin und Tierärztin G. Glück Liebe/r Biochemie-Interessierte,
Lernkarteikarten Veterinärmedizin - BIOCHEMIE BIOCHEMIE 2014 Teil 2: Aminosäuren, Proteine, Enzyme Vetbrainfood - Tiermedizinische Lernkarteikarten - Diplombiologin und Tierärztin G. Glück Liebe/r Biochemie-Interessierte,
Das ist der Ort, wo die Proteine Synthetisiert werden. Zusammen mit mrna und trna bilden sie eine Einheit, an der die Proteine synthetisiert werden.
 DAS RIBOSOM Das ist der Ort, wo die Proteine Synthetisiert werden. Zusammen mit mrna und trna bilden sie eine Einheit, an der die Proteine synthetisiert werden. Das Ribosom besteht aus 2 zusammengelagerten
DAS RIBOSOM Das ist der Ort, wo die Proteine Synthetisiert werden. Zusammen mit mrna und trna bilden sie eine Einheit, an der die Proteine synthetisiert werden. Das Ribosom besteht aus 2 zusammengelagerten
KOHLENHYDRATE PYRUVAT-DEHYDROGENASE
 PYRUVAT-DEHYDROGENASE PYRUVAT-DEHYDROGENASE: Um ein Optimum Beute garantieren zu können, Wird das entstandene Pyruvat (bei der aeroben) Glykolyse, durch die PDH in Acetyl-CoA umgewandelt, um dann, Teil
PYRUVAT-DEHYDROGENASE PYRUVAT-DEHYDROGENASE: Um ein Optimum Beute garantieren zu können, Wird das entstandene Pyruvat (bei der aeroben) Glykolyse, durch die PDH in Acetyl-CoA umgewandelt, um dann, Teil
KV: Translation Michael Altmann
 Institut für Biochemie und Molekulare Medizin KV: Translation Michael Altmann Herbstsemester 2008/2009 Übersicht VL Translation 1.) Genexpression 2.) Der genetische Code ist universell 3.) Punktmutationen
Institut für Biochemie und Molekulare Medizin KV: Translation Michael Altmann Herbstsemester 2008/2009 Übersicht VL Translation 1.) Genexpression 2.) Der genetische Code ist universell 3.) Punktmutationen
Basen Nucleoside - Nucleotide Synthese und Abbau
 Basen ucleoside - ucleotide Synthese und Abbau Stickstoff-Basen Die heterocyclischen und aromatischen Stickstoffbasen sind planar Sie leiten sich ab von den -eterocyclen: Purin und Pyrimidin Die ucleinsäure-basen
Basen ucleoside - ucleotide Synthese und Abbau Stickstoff-Basen Die heterocyclischen und aromatischen Stickstoffbasen sind planar Sie leiten sich ab von den -eterocyclen: Purin und Pyrimidin Die ucleinsäure-basen
Lecture 3 Regulation of Initiation: Met-tRNA-binding
 Institut für Biochemie und Molekulare Medizin Lecture 3 Regulation of Initiation: Met-tRNA-binding Michael Altmann FS 2014 Model of initiation Category of initiation factors eif1 Members eif1 eif1a Function
Institut für Biochemie und Molekulare Medizin Lecture 3 Regulation of Initiation: Met-tRNA-binding Michael Altmann FS 2014 Model of initiation Category of initiation factors eif1 Members eif1 eif1a Function
Präsentation STOFFWECHSEL STOFFWECHSEL. Fettstoffwechsel im Sport. Biologische Oxidation Zitratzyklus und Atmungskette
 STOFFWESEL GRUNDLAGEN STÖRUNGEN:Diagnose, Therapie, Prävention 6 Bedeutung der körperlichen Aktivität Präsentation Fettstoffwechsel im Sport Glukose exokinase 1ATP -> 1ADP Glukose-6-Phosphat Phosphohexoisomerase
STOFFWESEL GRUNDLAGEN STÖRUNGEN:Diagnose, Therapie, Prävention 6 Bedeutung der körperlichen Aktivität Präsentation Fettstoffwechsel im Sport Glukose exokinase 1ATP -> 1ADP Glukose-6-Phosphat Phosphohexoisomerase
PROTEINBIOSYNTHESE "Das zentrale Dogma der Molekularbiologie"
 PROTEINBIOSYNTHESE "Das zentrale Dogma der Molekularbiologie" Die für die Synthese von Eiweißstoffen notwendigen Schritte sind: (1) Replikation der DNA: Vor jeder Zellteilung wird die gesamte zelluläre
PROTEINBIOSYNTHESE "Das zentrale Dogma der Molekularbiologie" Die für die Synthese von Eiweißstoffen notwendigen Schritte sind: (1) Replikation der DNA: Vor jeder Zellteilung wird die gesamte zelluläre
Grundlagen der Biochemie (BCh 5.3) Prof. Dr. Christoph Thiele WS 2014/15 1. Klausur ( )
 Grundlagen der Biochemie (BCh 5.3) Prof. Dr. Christoph Thiele WS 2014/15 1. Klausur (6.2.2015) Aufgabe 1: Erläutern Sie die Primär-, Sekundär- und die Tertiärstruktur von Proteinen. [3 P] Aufgabe 2: Erklären
Grundlagen der Biochemie (BCh 5.3) Prof. Dr. Christoph Thiele WS 2014/15 1. Klausur (6.2.2015) Aufgabe 1: Erläutern Sie die Primär-, Sekundär- und die Tertiärstruktur von Proteinen. [3 P] Aufgabe 2: Erklären
Biosynthese und Enzymfamilien
 Biosynthese und Enzymfamilien The vast array of plant natural products can be viewed as the current status of nature s activities in combinatorial chemistry. R.A. Dixon Kombinatorische Chemie Technologie
Biosynthese und Enzymfamilien The vast array of plant natural products can be viewed as the current status of nature s activities in combinatorial chemistry. R.A. Dixon Kombinatorische Chemie Technologie
Institut für Biochemie und Molekulare Medizin. Lecture 1 Translational components. Michael Altmann FS 2011
 Institut für Biochemie und Molekulare Medizin Lecture 1 Translational components Michael Altmann FS 2011 Gene Expression Fliessdiagramm der eukaryotischen Genexpression Die Expression eines Gens kann auf
Institut für Biochemie und Molekulare Medizin Lecture 1 Translational components Michael Altmann FS 2011 Gene Expression Fliessdiagramm der eukaryotischen Genexpression Die Expression eines Gens kann auf
Datenspeicherung und Datenfluß in der Zelle - Grundlagen der Biochemie
 Datenspeicherung und Datenfluß in der Zelle - Grundlagen der Biochemie Datenspeicherung und Datenfluß der Zelle Transkription DNA RNA Translation Protein Aufbau I. Grundlagen der organischen Chemie und
Datenspeicherung und Datenfluß in der Zelle - Grundlagen der Biochemie Datenspeicherung und Datenfluß der Zelle Transkription DNA RNA Translation Protein Aufbau I. Grundlagen der organischen Chemie und
Schrittweise Oxidation und Decarboxylierung von Glucose-6-phosphat zu Ribulose-5- phosphat
 1. Der plastidäre oxidative Pentosephosphatweg Abbau von Hexose unter NADPH+H + -Synthese Schlüsselenzym=Glucose-6-phosphat-Dehydrogenase Glucose-6-phosphat-Dehydrogenase wird durch Thioredoxin im reduzierten
1. Der plastidäre oxidative Pentosephosphatweg Abbau von Hexose unter NADPH+H + -Synthese Schlüsselenzym=Glucose-6-phosphat-Dehydrogenase Glucose-6-phosphat-Dehydrogenase wird durch Thioredoxin im reduzierten
Was bisher geschah 1
 Was bisher geschah 1 Zellatmung (Übersicht) Der Citratcyclus ist die erste Stufe der Zellatmung 2 Citratzyklus Synonyme: Tricarbonsäurezyklus (TCA-Zyklus) Krebszyklus, Zitronensäurezyklus Der Zyklus ist
Was bisher geschah 1 Zellatmung (Übersicht) Der Citratcyclus ist die erste Stufe der Zellatmung 2 Citratzyklus Synonyme: Tricarbonsäurezyklus (TCA-Zyklus) Krebszyklus, Zitronensäurezyklus Der Zyklus ist
Die regulatorische Rolle der TFIIH Proteinkinasen in Pflanzen Regulatory roles of plant TFIIH protein kinases
 Die regulatorische Rolle der TFIIH Proteinkinasen in Pflanzen Regulatory roles of plant TFIIH protein kinases Koncz, Csaba Max-Planck-Institut für Pflanzenzüchtungsforschung, Köln Korrespondierender Autor
Die regulatorische Rolle der TFIIH Proteinkinasen in Pflanzen Regulatory roles of plant TFIIH protein kinases Koncz, Csaba Max-Planck-Institut für Pflanzenzüchtungsforschung, Köln Korrespondierender Autor
Citratzyklus. Biochemie Maria Otto,Bo Mi Ok Kwon Park
 Citratzyklus Biochemie 13.12.2004 Maria Otto,Bo Mi Ok Kwon Park O CH 3 C Acetyl-CoA + H 2 O HO C COO C NADH O C H Citrat Cis-Aconitat H C Malat Citratzyklus HO C H Isocitrat CH H 2 O Fumarat C = O FADH
Citratzyklus Biochemie 13.12.2004 Maria Otto,Bo Mi Ok Kwon Park O CH 3 C Acetyl-CoA + H 2 O HO C COO C NADH O C H Citrat Cis-Aconitat H C Malat Citratzyklus HO C H Isocitrat CH H 2 O Fumarat C = O FADH
Bachelor Studiengang Ernährungswissenschaften Seminar Biochemie der Ernährung (Modul BE2.3) Übersicht Seminarthemen
 Biochemie der Ernährung (Modul BE2.3) Prof. Dr. Stefan Lorkowski 1 Bachelor Studiengang Ernährungswissenschaften Seminar Biochemie der Ernährung (Modul BE2.3) Übersicht Seminarthemen Biosynthese von Aminosäuren
Biochemie der Ernährung (Modul BE2.3) Prof. Dr. Stefan Lorkowski 1 Bachelor Studiengang Ernährungswissenschaften Seminar Biochemie der Ernährung (Modul BE2.3) Übersicht Seminarthemen Biosynthese von Aminosäuren
Biochemie und Stoffwechsel Biochemie Aufklärung der Stoffwechselwege und -teilschritte
 Biochemie und Stoffwechsel Biochemie Aufklärung der Stoffwechselwege und -teilschritte Identifikation der Ausgangs-, Zwischen- und Endprodukte (Stoffwechselprodukte) Enzyme sind Proteine mit Katalysatorwirkung.
Biochemie und Stoffwechsel Biochemie Aufklärung der Stoffwechselwege und -teilschritte Identifikation der Ausgangs-, Zwischen- und Endprodukte (Stoffwechselprodukte) Enzyme sind Proteine mit Katalysatorwirkung.
Stoffwechsel - Energiebereitstellung - Biomoleküle
 Biochemie und Stoffwechsel Biochemie Aufklärung der Stoffwechselwege und -teilschritte Identifikation der Ausgangs-, Zwischen- und Endprodukte (Stoffwechselprodukte) Enzyme sind Proteine mit Katalysatorwirkung.
Biochemie und Stoffwechsel Biochemie Aufklärung der Stoffwechselwege und -teilschritte Identifikation der Ausgangs-, Zwischen- und Endprodukte (Stoffwechselprodukte) Enzyme sind Proteine mit Katalysatorwirkung.
Translation. Auflesung- Proteinsynthese
 Translation Auflesung- Proteinsynthese Proteinsynthese DNA mrna Transkription elágazási hely Translation Polypeptid Vor dem Anfang Beladen der trnas spezifische Aminosäure + spezifische trna + ATP Aminoacyl-tRNA
Translation Auflesung- Proteinsynthese Proteinsynthese DNA mrna Transkription elágazási hely Translation Polypeptid Vor dem Anfang Beladen der trnas spezifische Aminosäure + spezifische trna + ATP Aminoacyl-tRNA
1 Bausteine der Nucleinsäuren - Dezember 2008
 Page 1 of 5 GRUNDLAGEN DER MOLEKULARBIOLOGIE Prof. Dr. Anne Müller 1 Bausteine der Nucleinsäuren 1.1 Stickstoffhaltige Basen: Pyrimidine und Purine 1.2 Pentosen und Phosphate 1.3 Nucleoside 1.4 Nucleotide
Page 1 of 5 GRUNDLAGEN DER MOLEKULARBIOLOGIE Prof. Dr. Anne Müller 1 Bausteine der Nucleinsäuren 1.1 Stickstoffhaltige Basen: Pyrimidine und Purine 1.2 Pentosen und Phosphate 1.3 Nucleoside 1.4 Nucleotide
Unterrichtsmaterialien in digitaler und in gedruckter Form. Auszug aus:
 Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Die gentechnische Produktion von Insulin - Selbstlerneinheit zur kontextorientierten Wiederholung der molekularen Genetik Das komplette
Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Die gentechnische Produktion von Insulin - Selbstlerneinheit zur kontextorientierten Wiederholung der molekularen Genetik Das komplette
Biochemie Übungsblatt Nr Fettsäuresynthese:
 Fettsäuresynthese: 1) Mit Hilfe des mitochondriale ACP? AcetylCoA Carrboxylase (Biothin als prosth. Gruppe) AcetylCoA + Kohlendioxid + ATP MalonylCoA + ADP + P 2)? Reguliert durch Insulin (Insulin hemmt
Fettsäuresynthese: 1) Mit Hilfe des mitochondriale ACP? AcetylCoA Carrboxylase (Biothin als prosth. Gruppe) AcetylCoA + Kohlendioxid + ATP MalonylCoA + ADP + P 2)? Reguliert durch Insulin (Insulin hemmt
Struktur und Funktion der DNA
 Struktur und Funktion der DNA Wiederholung Nucleotide Nucleotide Nucleotide sind die Untereinheiten der Nucleinsäuren. Sie bestehen aus einer N-haltigen Base, einer Pentose und Phosphat. Die Base hängt
Struktur und Funktion der DNA Wiederholung Nucleotide Nucleotide Nucleotide sind die Untereinheiten der Nucleinsäuren. Sie bestehen aus einer N-haltigen Base, einer Pentose und Phosphat. Die Base hängt
Expressionskontrolle in Eukaryonten
 Expressionskontrolle in Eukaryonten Warum muss Genexpression kontrolliert werden? 1. Gewebsspezifische Kontrolle - nicht jedes Genprodukt ist in allen Zellen erforderlich - manche Genprodukte werden ausschliesslich
Expressionskontrolle in Eukaryonten Warum muss Genexpression kontrolliert werden? 1. Gewebsspezifische Kontrolle - nicht jedes Genprodukt ist in allen Zellen erforderlich - manche Genprodukte werden ausschliesslich
Biochemie II - Tutorium
 Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 16.11.2016 Ablauf des Tutoriums Einführung und Wiederholung Vorlesungszusammenfassung Übungsaufgaben Selbststudium
Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 16.11.2016 Ablauf des Tutoriums Einführung und Wiederholung Vorlesungszusammenfassung Übungsaufgaben Selbststudium
Wirkungsmechanismen regulatorischer Enzyme
 Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
8. Translation. Konzepte: Translation benötigt trnas und Ribosomen. Genetischer Code. Initiation - Elongation - Termination
 8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation - Elongation - Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse human bacteria
8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation - Elongation - Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse human bacteria
Leibniz-Institut für Naturstoff-Forschung und Infektionsbiologie - Hans-Knöll-Institut! Chemie Mikrobieller Kommunikation!
 Leibniz-Institut für aturstoff-forschung und Infektionsbiologie - ans-knöll-institut! Chemie Mikrobieller Kommunikation! Pierre Stallforth - pierre.stallforth@gmail.com! Ringvorlesung! Vertiefungsfach
Leibniz-Institut für aturstoff-forschung und Infektionsbiologie - ans-knöll-institut! Chemie Mikrobieller Kommunikation! Pierre Stallforth - pierre.stallforth@gmail.com! Ringvorlesung! Vertiefungsfach
RNA und Expression RNA
 RNA und Expression Biochemie RNA 1) Die Transkription. 2) RNA-Typen 3) RNA Funktionen 4) RNA Prozessierung 5) RNA und Proteinexpression/Regelung 1 RNA-Typen in E. coli Vergleich RNA-DNA Sequenz 2 Die Transkriptions-Blase
RNA und Expression Biochemie RNA 1) Die Transkription. 2) RNA-Typen 3) RNA Funktionen 4) RNA Prozessierung 5) RNA und Proteinexpression/Regelung 1 RNA-Typen in E. coli Vergleich RNA-DNA Sequenz 2 Die Transkriptions-Blase
Um die Gestalt des Nukleotid langsam zu vervollständigen, kommt es nun zum Anhängen eines aktivierten Ribosephosphats. Enzym:
 Um die Gestalt des Nukleotid langsam zu vervollständigen, kommt es nun zum Anhängen eines aktivierten Ribosephosphats. Enzym: Orotat-Phosphoribosyltransferase Durch Abspaltung des P-P, wird die hier frei
Um die Gestalt des Nukleotid langsam zu vervollständigen, kommt es nun zum Anhängen eines aktivierten Ribosephosphats. Enzym: Orotat-Phosphoribosyltransferase Durch Abspaltung des P-P, wird die hier frei
Einführung in die Biochemie Antworten zu den Übungsaufgaben
 Einführung in die Biochemie Antworten zu den Übungsaufgaben Dank Die vorliegenden Antworten zu den Übungsaufgaben für das Seminar zum Modul Einführung in die Biochemie wurden im Wintersemester 2014/2015
Einführung in die Biochemie Antworten zu den Übungsaufgaben Dank Die vorliegenden Antworten zu den Übungsaufgaben für das Seminar zum Modul Einführung in die Biochemie wurden im Wintersemester 2014/2015
Biochemisches Grundpraktikum
 Biochemisches Grundpraktikum Dr. Ellen Hornung; Email: ehornun@gwdg.de; Tel: 39-5748 Einteilung der Praktikumsplätze: Eintragen in Listen am - Dienstag, 10.11.2009, von 12:00 13:00 - Freitag, 13.11.2009,
Biochemisches Grundpraktikum Dr. Ellen Hornung; Email: ehornun@gwdg.de; Tel: 39-5748 Einteilung der Praktikumsplätze: Eintragen in Listen am - Dienstag, 10.11.2009, von 12:00 13:00 - Freitag, 13.11.2009,
Biochemie II - Tutorium
 Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 23.11.2016 Ablauf des Tutoriums Einführung und Wiederholung Vorlesungszusammenfassung Übungsaufgaben Selbststudium
Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 23.11.2016 Ablauf des Tutoriums Einführung und Wiederholung Vorlesungszusammenfassung Übungsaufgaben Selbststudium
COPYRIGHTED MATERIAL. Index
 253 Index ACE siehe Angiotensin-konvertierendes Enzym Acetaldehyd 174 Acetyl-CoA/Acetyl-Coenzym A 164, 175 Acetylcholinesterase 151 Acetylsalicylsäure 137, 141 Aconitase 191 Aconitat 190 Actin 37 Acyl-Enzym-Intermediat
253 Index ACE siehe Angiotensin-konvertierendes Enzym Acetaldehyd 174 Acetyl-CoA/Acetyl-Coenzym A 164, 175 Acetylcholinesterase 151 Acetylsalicylsäure 137, 141 Aconitase 191 Aconitat 190 Actin 37 Acyl-Enzym-Intermediat
Hauptwege des Stoffwechsels
 Hauptwege des Stoffwechsels Auf- und Abbau der Stoffgruppen Kohlenhydrate 1 Enzyme Hauptklassen 1 2 3 4 5 6 Biochemie 08/2 Enzyme 1. Erhöhung der Reaktionsgeschwindigkeit ΔΔG kat = ΔG unkat - ΔG kat Erniedrigung
Hauptwege des Stoffwechsels Auf- und Abbau der Stoffgruppen Kohlenhydrate 1 Enzyme Hauptklassen 1 2 3 4 5 6 Biochemie 08/2 Enzyme 1. Erhöhung der Reaktionsgeschwindigkeit ΔΔG kat = ΔG unkat - ΔG kat Erniedrigung
Aufbau, Struktur, Funktion von DNA, RNA und Proteinen
 Aufbau, Struktur, Funktion von DNA, RNA und Proteinen Mitarbeiterseminar der Medizinischen Fakultät Ruhr-Universität Bochum Andreas Friebe Abteilung für Pharmakologie und Toxikologie Aufbau, Struktur,
Aufbau, Struktur, Funktion von DNA, RNA und Proteinen Mitarbeiterseminar der Medizinischen Fakultät Ruhr-Universität Bochum Andreas Friebe Abteilung für Pharmakologie und Toxikologie Aufbau, Struktur,
Biochemie II - Tutorium
 Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 04.01.2016 Zellkern Lipidtröpfchen Nucleotidmetabolismus Glykogen- Stoffwechsel Pentosephosephatweg Glucose Glucose
Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 04.01.2016 Zellkern Lipidtröpfchen Nucleotidmetabolismus Glykogen- Stoffwechsel Pentosephosephatweg Glucose Glucose
Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt. Abb. aus Stryer (5th Ed.)
 Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt Die Initiation der Translation bei Eukaryoten Der eukaryotische Initiationskomplex erkennt zuerst das 5 -cap der mrna und
Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt Die Initiation der Translation bei Eukaryoten Der eukaryotische Initiationskomplex erkennt zuerst das 5 -cap der mrna und
Anabole Prozesse in der Zelle
 Anabole Prozesse in der Zelle DNA Vermehrung RNA Synthese Protein Synthese Protein Verteilung in der Zelle Ziel: Zellteilung (Wachstum) und Differenzierung (Aufgabenteilung im Organismus). 2016 Struktur
Anabole Prozesse in der Zelle DNA Vermehrung RNA Synthese Protein Synthese Protein Verteilung in der Zelle Ziel: Zellteilung (Wachstum) und Differenzierung (Aufgabenteilung im Organismus). 2016 Struktur
Vertiefendes Seminar zur Vorlesung Biochemie I
 Vertiefendes Seminar zur Vorlesung Biochemie I 30.01.2015 Klausurvorbereitung: Gerhild van Echten-Deckert Rekombinante DNA Fon. +49-228-732703 Homepage: http://www.limes.uni-bonn.de Klärung einiger Begriffe:
Vertiefendes Seminar zur Vorlesung Biochemie I 30.01.2015 Klausurvorbereitung: Gerhild van Echten-Deckert Rekombinante DNA Fon. +49-228-732703 Homepage: http://www.limes.uni-bonn.de Klärung einiger Begriffe:
Charakterisierung der PII-P Phosphatase in Cyanobakterien
 Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Der Fettsäurestoffwechsel. Basierend auf Stryer Kapitel 22
 Der Fettsäurestoffwechsel Basierend auf Stryer Kapitel 22 1 CoA 2 3 Überblick 4 Ein paar Grundlagen... Carbonsäure Alkohol Carbonsäureester Eine Acyl-Gruppe 5 Eine Acyl-Gruppe H O Formyl H 3 C O Acetyl
Der Fettsäurestoffwechsel Basierend auf Stryer Kapitel 22 1 CoA 2 3 Überblick 4 Ein paar Grundlagen... Carbonsäure Alkohol Carbonsäureester Eine Acyl-Gruppe 5 Eine Acyl-Gruppe H O Formyl H 3 C O Acetyl
Energiestoffwechsel --- Aminosäuren. Prof. Dr. Martin van der Laan
 Energiestoffwechsel --- Aminosäuren Prof. Dr. Martin van der Laan Stoffwechsel von Aminosäuren (Überblick) Aminosäure N 3 + R C α Desaminierung Transaminierung Decarboxylierung CO 2 N 3 + R C (Biogenes)
Energiestoffwechsel --- Aminosäuren Prof. Dr. Martin van der Laan Stoffwechsel von Aminosäuren (Überblick) Aminosäure N 3 + R C α Desaminierung Transaminierung Decarboxylierung CO 2 N 3 + R C (Biogenes)
Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!!
 Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!! Indirekte ATP synthese! Protonen können in wässriger Lösung sehr! schnell transportiert werden.! Ionen können biologische Membranen nicht spontan
Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!! Indirekte ATP synthese! Protonen können in wässriger Lösung sehr! schnell transportiert werden.! Ionen können biologische Membranen nicht spontan
Seminar Biochemie. Lipide und Lipidstoffwechsel. Dr. Christian Hübbers
 Seminar Biochemie Lipide und Lipidstoffwechsel Dr. Christian Hübbers Lernziele Lipidklassen: Fettsäuren, Triglyceride, Phospholipide, Cholesterin, Sphingolipide, Glykolipide. ß-Oxidation, FS-Biosynthese,
Seminar Biochemie Lipide und Lipidstoffwechsel Dr. Christian Hübbers Lernziele Lipidklassen: Fettsäuren, Triglyceride, Phospholipide, Cholesterin, Sphingolipide, Glykolipide. ß-Oxidation, FS-Biosynthese,
Seminar Biochemie. Lipide und Lipidstoffwechsel. Dr. Christian Hübbers
 Seminar Biochemie Lipide und Lipidstoffwechsel Dr. Christian Hübbers Lernziele Lipidklassen: Fettsäuren, TG, Phospholipide, Cholesterin, Sphingolipide, Glykolipide. ß-Oxidation, FS-Biosynthese, MalonylCoA,
Seminar Biochemie Lipide und Lipidstoffwechsel Dr. Christian Hübbers Lernziele Lipidklassen: Fettsäuren, TG, Phospholipide, Cholesterin, Sphingolipide, Glykolipide. ß-Oxidation, FS-Biosynthese, MalonylCoA,
Vom Gen zum Protein. Zusammenfassung Kapitel 17. Die Verbindung zwischen Gen und Protein. Gene spezifizieren Proteine
 Zusammenfassung Kapitel 17 Vom Gen zum Protein Die Verbindung zwischen Gen und Protein Gene spezifizieren Proteine Zellen bauen organische Moleküle über Stoffwechselprozesse auf und ab. Diese Prozesse
Zusammenfassung Kapitel 17 Vom Gen zum Protein Die Verbindung zwischen Gen und Protein Gene spezifizieren Proteine Zellen bauen organische Moleküle über Stoffwechselprozesse auf und ab. Diese Prozesse
4. Hydrolytisch aktive Enzyme und Gruppentransfer-Reaktionen
 4. Hydrolytisch aktive Enzyme und Gruppentransfer-Reaktionen In der Natur gibt es prinzipiell drei Arten von Hydrolasen (hydrolytisch aktiven Enzymen), die die unterschiedlichen in der Natur vorhandenen
4. Hydrolytisch aktive Enzyme und Gruppentransfer-Reaktionen In der Natur gibt es prinzipiell drei Arten von Hydrolasen (hydrolytisch aktiven Enzymen), die die unterschiedlichen in der Natur vorhandenen
Kohlenstoffdioxid wird in lichtabhängigen Reaktion freigesetzt Sauerstoff wird wie in der Atmung verbraucht. Lichtatmung
 1. Kompartimentierung bei der Photorespiration Kohlenstoffdioxid wird in lichtabhängigen Reaktion freigesetzt Sauerstoff wird wie in der Atmung verbraucht. Lichtatmung Kosten: o Energie, da ATP verbraucht
1. Kompartimentierung bei der Photorespiration Kohlenstoffdioxid wird in lichtabhängigen Reaktion freigesetzt Sauerstoff wird wie in der Atmung verbraucht. Lichtatmung Kosten: o Energie, da ATP verbraucht
Bioinformatik I: Grundlagen der Gentechnik
 Bioinformatik I: Grundlagen der Gentechnik Dr. Maik Böhmer Institut für Biologie und Biotechnologie der Pflanzen Schlossplatz 7 Schwerpunkte: Vorlesung 1: Einführung & Enzyme der Gentechnik Vorlesung 2:
Bioinformatik I: Grundlagen der Gentechnik Dr. Maik Böhmer Institut für Biologie und Biotechnologie der Pflanzen Schlossplatz 7 Schwerpunkte: Vorlesung 1: Einführung & Enzyme der Gentechnik Vorlesung 2:
Die Pyrimidinsynthese
 Die Pyrimidinsynthese Bei der Pyrimidinsynthese wird zunächst der 6-Ring, also die Base zusammengefügt. Erst anschließend kommt es zum Anknüpfen der Ribose. Lokalisation: Zytoplasma Der Ausgang, der
Die Pyrimidinsynthese Bei der Pyrimidinsynthese wird zunächst der 6-Ring, also die Base zusammengefügt. Erst anschließend kommt es zum Anknüpfen der Ribose. Lokalisation: Zytoplasma Der Ausgang, der
ORGANISCHE CHEMIE. Aminosäuren und Proteine
 RGAISE EMIE Aminosäuren und Proteine Aminosäuren (AS) R 2 arbonsäuren mit einer Aminogruppe Die (etwa) 20 proteinogenen Aminosäuren sind alle α-aminosäuren (Aminogruppe an -2), unterscheiden sich durch
RGAISE EMIE Aminosäuren und Proteine Aminosäuren (AS) R 2 arbonsäuren mit einer Aminogruppe Die (etwa) 20 proteinogenen Aminosäuren sind alle α-aminosäuren (Aminogruppe an -2), unterscheiden sich durch
Atmung Respiration 1
 Atmung Respiration 1 Atmung Die oxidative Phosphorylierung ist der letzte Schritt einer Serie von Energieumwandlungen, die insgesamt als Zellatmung oder einfach Atmung (Respiration) bezeichnet werden 2
Atmung Respiration 1 Atmung Die oxidative Phosphorylierung ist der letzte Schritt einer Serie von Energieumwandlungen, die insgesamt als Zellatmung oder einfach Atmung (Respiration) bezeichnet werden 2
Was ist der Promotor? Antwort: Eine spezielle Nucleotidsequenz auf der DNA, an der die RNA-Polymerase bindet um die Transkription zu starten.
 Was ist der Promotor? Antwort: Eine spezielle Nucleotidsequenz auf der DNA, an der die RNA-Polymerase bindet um die Transkription zu starten. Wie bezeichnet man den Strang der DNA- Doppelhelix, der die
Was ist der Promotor? Antwort: Eine spezielle Nucleotidsequenz auf der DNA, an der die RNA-Polymerase bindet um die Transkription zu starten. Wie bezeichnet man den Strang der DNA- Doppelhelix, der die
Pathology Department - North Devon District Hospital Biochemistry Reference Ranges - Amendments (Shown in Pink) As from 14/03/2018
 ALT Alanine Aminotransferase No Change u/l Blood F 0-36 - Local/Historical ALT Alanine Aminotransferase No Change u/l Blood M 0-40 - Local/Historical ALB Albumin Amended g/l Blood B
ALT Alanine Aminotransferase No Change u/l Blood F 0-36 - Local/Historical ALT Alanine Aminotransferase No Change u/l Blood M 0-40 - Local/Historical ALB Albumin Amended g/l Blood B
Molekulare Wirkmechanismen von Flavonoiden
 Molekulare Wirkmechanismen von Flavonoiden R 2 R 3 R 4 R 7 R 8 A R 6 R 5 O C O B R 3 R 6 R 5 Krebs Arteriosklerose Altern Prof. Dr. Uwe Wenzel, Leipzig, Juni 21 Flavonoide und Kolonkrebs Flavonoide hemmen
Molekulare Wirkmechanismen von Flavonoiden R 2 R 3 R 4 R 7 R 8 A R 6 R 5 O C O B R 3 R 6 R 5 Krebs Arteriosklerose Altern Prof. Dr. Uwe Wenzel, Leipzig, Juni 21 Flavonoide und Kolonkrebs Flavonoide hemmen
LIPIDE. Periphere Gruppe mit Cysteinylrest. FS-Synthase. Zentrale Gruppe Phosphopanthetinarm kovalente Bindung
 Periphere Gruppe mit Cysteinylrest FS-Synthase Zentrale Gruppe Phosphopanthetinarm kovalente Bindung Die FS-Biosynthse aus Acetyl-CoA und Malonyl-CoA, wird komplett an dem dimeren Multienzymkomplex FETTSÄURESYNTHETASE
Periphere Gruppe mit Cysteinylrest FS-Synthase Zentrale Gruppe Phosphopanthetinarm kovalente Bindung Die FS-Biosynthse aus Acetyl-CoA und Malonyl-CoA, wird komplett an dem dimeren Multienzymkomplex FETTSÄURESYNTHETASE
Expression der genetischen Information Skript: Kapitel 5
 Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Warum brauchen Pflanzen Mikronährstoffe?
 Warum brauchen Pflanzen Mikronährstoffe? Mikronährstoffe besitzen bereits in kleinen Mengen (ppm) eine hohe physiologische Wirksamkeit. Bei höheren Gehalten (ppm!) wirken sie toxisch. Wirkung in Enzymen
Warum brauchen Pflanzen Mikronährstoffe? Mikronährstoffe besitzen bereits in kleinen Mengen (ppm) eine hohe physiologische Wirksamkeit. Bei höheren Gehalten (ppm!) wirken sie toxisch. Wirkung in Enzymen
Petr Necas wies auf die essentielle Bedeutung von Bienenpollen als regelmäßiger Bestandteil des Chamäleon Futters hin.
 Folgendes basiert auf Angaben von Petr Necas welche er bei seinem Vortrag Anfang Januar in Frankfurt am Main hielt sowie Post s welche er in einer Englischsprachigen Gruppe auf Englisch schrieb. Bienenpollen,
Folgendes basiert auf Angaben von Petr Necas welche er bei seinem Vortrag Anfang Januar in Frankfurt am Main hielt sowie Post s welche er in einer Englischsprachigen Gruppe auf Englisch schrieb. Bienenpollen,
Der Citratzyklus (= Trikarbonsäurezyklus, Krebszyklus)
 Der Citratzyklus (= Trikarbonsäurezyklus, Krebszyklus) Biochemischer Kreisprozeß Ablauf in der mitochondrialen Matrix Glykolyse β-oxidation Atmungskette AS-Abbau Der Citratzyklus Der Citratzyklus: Übersicht
Der Citratzyklus (= Trikarbonsäurezyklus, Krebszyklus) Biochemischer Kreisprozeß Ablauf in der mitochondrialen Matrix Glykolyse β-oxidation Atmungskette AS-Abbau Der Citratzyklus Der Citratzyklus: Übersicht
