3 DNA-Synthese (Replikation) - Dezember 2008
|
|
|
- Gregor Holst
- vor 8 Jahren
- Abrufe
Transkript
1 Page 1 of 16 GRUNDLAGEN DER MOLEKULARBIOLOGIE Prof. Dr. Anne Müller 3 DNA-Synthese (Replikation) 3.1 Ursprung der Replikation 3.2 Primase 3.3 DNA-Polymerasen 3.4 Leitstrang-Synthese 3.5 Folgestrang-Synthese 3.6 DNA-Polymerase I von E. coli 3.7 DNA-Ligase 3.8 Superspiralisierung 3.9 Topoisomerasen 3.10 Telomerasen 3.11 Organisation des Eukaryotischen Genoms 3.12 Hemmstoffe der Replikation 3.13 Der Zellzyklus Die DNA-Replikation erfolgt semikonservativ. Der DNA-Doppelstrang wird geöffnet und jeder Einzelstrang determiniert jeweils einen komplementären Tochterstrang. Durch fortlaufende Synthese entstehen zwei neue DNA-Doppelstränge, welche je zur Hälfte aus einem alten und einem neu synthetisierten Strang bestehen. Bilder: Nachweis der semikonservativen Replikation in E.coli durch Dichtegradientenzentrifugation. Die Lage einer DNA-Bande hängt von ihrem Gehalt an 14 N und 15 N ab. Nach 1,0 Generationen sind alle DNA-Moleküle Hybride mit gleichen Mengen an 14 N und 15 N, und es ist keine Eltern-DNA ( 15 N) mehr übrig.
2 Page 2 of Ursprung der Replikation Die DNA Replikation fängt generell an einem bestimmten Ort an, dessen DNA-Sequenz sehr AT-reich ist. In E. coli wird der Replikationsstartpunkt "Ori C" genannt. Der Replikationsstartpunkt in der Hefe Saccharomyces cerevisiae heisst ARS (autonomously-replicating sequence). Replikationsursprünge in menschlicher DNA wurden bis anhin nicht identifiziert. Bild: Replikationsinitiation in E. coli. Durch sequenzspezifische Bindung von DnaA, B und C Proteinen wird die Doppelhelix an dieser Stelle geöffnet, so dass die Primer- Synthese stattfinden kann. Das DnaA-Protein bindet spezifische ATreiche Sequenzen, während DnaB und DnaC für die Trennung der DNA- Stränge notwendig sind. DnaB ist eine ATP-abhängige DNA-Helikase, die als Homohexamer wirkt. Bild: Replikationsinitiation in Eukaryoten. Der Replikationsursprung wird in G2/M-Phasen des Zellzyklus vom (inaktiven) Origin Recognition Complex (ORC) gebunden. Während der G1-Phase wird ORC von der (inaktiven) MCM2-7- Helikase und anderen zellzyklus-abhängigen Faktoren gebunden (origin licensing). Am Anfang der S-phase werden MCM2-7 und ORC phosphoryliert, was zur Dissoziation von ORC und Aktivierung von MCM2-7 führt. Der Ursprung wird geöffnet, was die Bindung des Primase/Polymerasekomplexes ermöglicht. Sobald die Replikation angefangen hat, werden die Ursprungssequenzen von ORG wieder gebunden, um weitere Replikationsrunden zu vermeiden.
3 Page 3 of Primase Neue DNA kann nicht ohne das Vorhandensein eines Primers synthetisiert werden. Ein RNA-Primer (aus ungefähr 5 Nucleotiden) wird von der Primase synthetisiert und ermöglicht den Start der DNA-Synthese. Die Primase ist eine RNA- Polymerase, die an der DNA-Replikation beteiligt ist. In E. coli ist die Primase vom DnaG-Gen codiert, und ist Bestandteil eines Primosoms. In Eukaryoten assoziiert die Primase mit DNA- Polymerase-α. 3.3 DNA-Polymerasen Die DNA-Polymerase ist das Enzym, welches die schrittweise Addition von Desoxyribonucleotid-Einheiten an den wachsenden DNA-Strang katalysiert. Das Prinzip dieser Addition besteht im nucleophilen Angriff der 3'-OH- Gruppe des wachsenden Strangs auf das innerste (α) Phosphoratom des neuen Desoxyribonucleosidtriphosphats. Demnach ist die Verlängerung der DNA-Kette nur in 5' >3'-Richtung möglich. Die DNA-Polymerase braucht folgende Komponenten, um eine DNA-Kette zu synthetisieren: Sämtliche vier Desoxyribonucleosid-triphosphate (datp, dgtp, dttp, dctp; abgekürzt mit dntp). Die Hydrolyse des Pyrophosphats (PP i ) ist die treibende Kraft der Synthese. Mg 2+. Primer (Startermolekül), d.h. eine freie 3'-OH-Gruppe eines bereits vorhandenen DNA- (oder RNA-) Strangs. DNA-Matrize.
4 Page 4 of 16 In unserem Prototyp-Organismus, Escherichia coli, sind 5 DNA-Polymerasen identifiziert worden: Pol I - V. Pol I ist vorwiegend an der Folgestrang-Reparatur und bei der DNA-Reparatur beteiligt. Die Rolle der Pol II liegt höchstwahrscheinlich auch bei der DNA-Reparatur. Pol III, die als ein Holoenzym vorkommt, das aus mehreren Untereinheiten zusammengesetzt ist, ist für die Replikation des bakteriellen Genoms verantwortlich. Die Pol IV und Pol V Enzyme sind bei der SOS-Antwort, bzw. beim Überqueren von DNA-Schäden ("By-pass"), notwendig (siehe Kapitel 4). Ausser dem Pol-III Holoenzym erfordert die DNA-Replikation in E. coli auch das Einzelstrangbindungsprotein Ssb (Single Strand Binding protein), das die einzelsträngige DNA stabilisiert, und die DnaB-Helikase, die die DNA entwindet. Nach heutigem Wissen sind mindestens 15 Proteine direkt an der bakteriellen DNA-Replikation beteiligt. Man nimmt an, dass die Komplexität des Replikationsapparates notwendig ist, um u.a. die sehr hohe Wiedergabetreue zu gewährleisten. Die Fehlerquote beträgt etwa 1 pro 10 9 bis kopierter Basenpaare. In Eukaryoten sind bis anhin zwölf DNA-Polymerasen entdeckt worden: Polα, Pol δ und Polε sind an der DNA- Replikation beteiligt, Polβ sorgt für die Basenexzisionsreparatur (siehe 4.2), Polγ repliziert das mitochondriale Genom, und die Polymerasen ζ, η, θ, ι, κ und λ überqueren DNA-Schäden als sogenannte By-pass- Polymerasen (siehe 4.6). Polµ spielt höchstwahrscheinlich bei der VDJ-Rekombination und der Class-Switch- Rekombination von Immunoglobulin-Genen eine Hauptrolle (siehe 6.3c). 3.4 Leitstrang-Synthese In E. coli wird der Leitstrang kontinuierlich von Pol III synthetisiert. Die Prozessivität der Pol III wird von der ringförmigen β-untereinheit garantiert. Die β-untereinheit, auch gleitende Klammer genannt, ist ein Homodimer (gelb und rot), das die DNA-Matrize (blau) umschliesst. Das Aufladen der gleitenden Klammer auf die DNA wird von der γ-untereinheit ( Clamp Loader ) ausgeführt; diese Reaktion geschieht unter ATP-Verbrauch. Bild: Pol III Holoenzym
5 Page 5 of 16 Bild: β-untereinheit der Polymerase III Bild: γ-untereinheit ( Clamp Loader ) In Eukaryoten wird die Leitstrang-Synthese von Polymerase-δ katalysiert. Die gleitende Klammer -Funktion wird von PCNA (Proliferating Cell Nuclear Antigen), einem Homotrimer, erfüllt. RFC (Replication Factor C) ist der Klammer-Lader. Die Rolle des Ssb wird in Eukaryoten von RPA (Replication Protein A) erfüllt. In menschlichen Zellen wird die Rolle der replikations-assoziierten DNA-Helikase höchstwahrscheinlich vom MCM2-7-Komplex erfüllt. Bei der SV40-Replikation wird diese Aufgabe vom Large T antigen übernommen (siehe Bild unten). Bild: Replikation der SV40-DNA
6 Page 6 of Folgestrang-Synthese Der Folgestrang, welcher scheinbar von der Replikationsgabel wegwächst, wird stückweise aufgebaut. Diese sogenannten Okazaki-Stücke besitzen jeweils einen RNA- Primer, der von der Primase (resp. Primase/Pol-α Komplex in Eukaryoten) synthetisiert wird und dann von Pol III (resp. von Pol-δ/ε) verlängert wird. Die Bildung einer Schleife ermöglicht das Wachstum des Folgestrangs in 3' ---> 5'- Richtung, obwohl das Anhängen der einzelnen Nucleotide in 5' ---> 3'-Richtung erfolgt. 3.6 DNA-Polymerase I Die DNA-Polymerase I-Aktivität füllt die Lücken zwischen benachbarten Okazaki-Stücken auf.
7 Page 7 of 16 Die 5-3 -Exonuclease wird beim Abbau der RNA- Primer der Okazaki-Stücke gebraucht. Die 3-5 -Exonuclease wird beim Korrekturlesen ( proofreading ) gebraucht. In Eukaryoten ist die 5 -->3 -Exonuclease Funktion von Pol I durch die "Flap Endonuclease 1" (FEN1) ersetzt. Das Korrekturlesen ist von der 3 -->5 -Exonuclease Untereinheit der Pol-δ/ε katalysiert.
8 Page 8 of DNA-Ligase Die DNA-Ligase verknüpft DNA-Enden in Doppelhelixregionen. Das Enzym katalysiert die Bildung einer Phosphodiesterbindung zwischen der 3 - OH-Gruppe am Ende des einen DNA- Fragments und der 5 -Phosphatgruppe am Ende des anderen Fragments. 3.8 Superspiralisierung Die meisten natürlich vorkommenden DNA-Moleküle sind negativ superspiralisiert (untergewunden). Die negative Superspiralisierung bereitet die DNA auf Prozesse vor, die eine Trennung der beiden Stränge erfordern. Dazu gehören die Replikation, die Rekombination und die Transkription. Die Verwindungszahl der DNA ist aber ein dynamisches Phänomen. Prozesse, die Proteinbewegungen auf der DNA-Matrize erfordern, ändern kontinuierlich den Superspiralisierungsgrad vor und hinter sich.
9 Page 9 of 16 Wenn Lk die Verwindungszahl (linking number) der DNA ist, Tw (twisting number) der Zahl der Watson-Crick- Windungen entspricht und Wr die Anzahl der Superhelixwindungen bezeichnet, besteht die folgende Beziehung: Lk = Tw + Wr Das Ausmass der Superspiralisierung einer DNA kann durch die Superhelixdichte σ ausgedrückt werden: σ = (Lk - Lk o )/Lk o (Lk o entspricht der Verwindungszahl des entspannten zirkulären DNA-Moleküls.) Beispiel: (23-25)/25 = -2/25 = Die Superhelixdichte σ der in vivo superspiralisierter DNA ist in der Regel Topoisomerasen Die Überführung verschiedener Topoisomere (DNA-Moleküle mit unterschiedlicher Verwindungszahl) einer DNA ineinander wird durch Enzyme katalysiert, die man als Topoisomerasen bezeichnet. Diese Enzyme verändern die Verwindungszahl der DNA, indem sie einen dreistufigen Prozess katalysieren: 1) Spaltung eines oder beider DNA-Stränge. 2) Durchtritt eines DNA-Abschnitts durch die entstandene Lücke. 3) Verknüpfung der DNA-Bruchstelle.
10 Page 10 of 16 Topoisomerase I Die Typ-I-Topoisomerasen spalten nur einen DNA-Strang. Die Reaktion der Entspannung einer negativsuperspiralisierten DNA ist thermodynamisch gesehen mit Energiegewinn verbunden. Die Verwindungszahl der superspiralisierten DNA ändert sich bei jedem Katalysedurchgang um 1. Die Topoisomerase I von E. coli relaxiert nur negativ-superhelikale DNA. Die menschliche Topoisomerase I (und Topoisomerase III) relaxieren sowohl negativ- als auch positiv-superspiralisierte DNA. Bild: Das eine Ende der DNA-Doppelhelix kann gegenüber dem anderen Ende nicht rotieren Typ I DNA-Topoisomerase mit Tyrosin in der aktiven Stelle DNA-Topoisomerase bindet kovalent an ein DNA-Phosphat und öffnet dabei eine Phosphodiesterbindung in diesem Strang Die beiden Enden der DNA-Doppelhelix können nun relativ zueinander rotieren Die ursprüngliche Bindungs-Energie des Phosphodiesters wird in der Phospho- Tyrosinbindung gespeichert - Reversibilität Spontane Wiederbildung der Phosphodiesterbindung regeneriert sowohl die DNA-Helix als auch die Topoisomerase in unveränderter Form
11 Page 11 of 16 Topoisomerase II Enzyme des Typs II spalten beide Stränge. Sie relaxieren sowohl negativ- als auch positiv-superspiralisierte DNA. Die Topoisomerase II von E. coli (DNA-Gyrase) führt dazu negative Superhelices in die DNA ein. In Eukaryoten ist dies nicht notwendig, da die DNA durch die Verpackung ins Chromatin automatisch negativsuperspiralisiert wird (siehe Kapitel 3.11). Die DNA-Topoisomerase II wandelt die freie Energie des ATP in die Torsionsenergie von Superhelices um. Die Verwindungszahl der DNA wird bei jeder Reaktion um 2 geändert. Hemmstoffe der Topoisomerasen Die DNA-Gyrase (bakterielle Topo-II) ist Angriffspunkt verschiedener Antibiotika, die das prokaryotische Enzym viel stärker hemmen als das eukaryotische. Novobiocin blockiert die Bindung des ATP an die Gyrase. Nalidixinsäure und Ciprofloxacin, die häufig zur Behandlung von Harnweg- und anderen Infektionen eingesetzt werden, beeinträchtigen dagegen die Spaltung und Wiederverknüpfung von DNA-Ketten. Eukaryotische Topoisomerasen sind Angriffspunkte der modernen Krebstherapie; Camptothecin (Topo-I Inhibitor) und Etoposid (Topo-II Inhibitor) hemmen die Proliferation der schnell-replizierenden Krebszellen Telomerasen Telomere, die Enden der Chromosomen, werden von einer Reversen Transkriptase (RNA-abhängige DNA- Polymerase) repliziert, die eine RNA-Matrize enthält. Die Telomerase synthetisiert Tandemwiederholungen von G-reichen Hexanucleotiden (AGGGTT in menschlicher DNA), um die Synthese des Folgestrangs zu vollenden. Weil somatische Zellen keine oder nur wenig Telomeraseaktivität haben, wird die Telomerlänge mit jeder Zellteilung kürzer. Daraus ist die Hypothese entstanden, dass die Telomeraseaktivität die Geschwindigkeit des Alterungsprozesses festlegt. Krebszellen zeigen eine erhöhte Telomeraseaktivität und haben die Tendenz "immortal" zu werden. Die Telomerase könnte deswegen ein Ziel der Tumortherapie werden.
12 Page 12 of 16 Bild: Synthese des G-reichen Stranges der Telomer-DNA. Die RNA-Matrize der Telomerase ist rot, die an den G-reichen Strang des Primers angehängten Nucleotide sind blau dargestellt.
13 Page 13 of Organisation des Eukaryotischen-Genoms Die DNA bildet mit den basischen Histonen geordnete Strukturen, die Nucleosomen. Ein Nucleosom Core besteht aus etwa 146 Basenpaaren, die um je zwei H2A, H2B, H3 und H4 Histonmoleküle gewunden sind, so dass 1 3 /4 Windungen einer linksgängigen Superhelix entstehen. Einzelne Nucleosomen sind über kurze DNA- Abschnitte perlschnurartig miteinander verbunden. Die einzelnen Chromatinfäden liegen in den 46 Chromosomen in stark kondensierter Form vor: Totale Länge der DNA = 2 m, totale Länge aller 46 Chromosomen (in Metaphase) = 200 µm. Chromatinkondensierung ist auch abhängig vom DNA- Methylierungszustand und der Histonmodifikation (Acetylierung, Methylierung, ADP-Ribosylierung, Phosphorylierung). Nucleosomen, die H1 enthalten, besitzen ungefähr 166 Basenpaare DNA. H1 wird unmittelbar vor der Mitose phosphoryliert und nach der Mitose wieder dephosphoryliert, was vermuten lässt, dass diese kovalente Modifikation dessen Fähigkeit steuert, die DNA zu verdichten. Acetylierung an Lysinresten von Histonen (z. B. H4) kann deren Affinität für DNA beeinflussen. Das Zusammenspiel von Histon-Acetylasen, -Deacetylasen und Histon-Kinasen ist insgesamt für die Dichte der Chromatinverpackung verantwortlich. Die am stärksten komprimierte DNA findet man in Spermienköpfen, wo die Histone durch Protamine ersetzt sind. Die Protamine sind argininreiche Proteine, welche durch Bindung an die DNA hochgradig α-helikal werden. Menschliche DNA enthält pro diploide Zelle ca. 6.6 x 10 9 Basenpaare. Dies würde für ca. 3 x 10 6 Gene reichen. Tatsächlich rechnet man aber nur mit ca Genen, was theoretisch eine viel kürzere DNA zur Folge hätte. Das Vorhandensein von stark repetitiver DNA und Introns (nicht-kodierende DNA-Abschnitte zwischen den Exons) gleicht jedoch diese Diskrepanz aus und ist für die wahre Länge der menschlichen DNA verantwortlich.
14 Page 14 of Hemmstoffe der Replikation Hemmstoffe der Replikation können als Cytostatika zur Krebsbekämpfung eingesetzt werden (Cytosin-Arabinosid). Die Wirksamkeit von Acyclovir bei Herpes simplex Infektionen ist dadurch gegeben, dass diese Substanz erst durch die virale Thymidin- Kinase phosphoryliert wird. Acyclovirtriphosphat ist nur Substrat für die virale, nicht aber für die menschliche DNA-Polymerase. Deswegen wird es ausschliesslich in die virale DNA eingebaut, wo es zur Kettenverlängerungstermination der DNA-Synthese führt, da ACV keine freie 3 -OH-Gruppe besitzt. ddc, ddi und AZT sind Hemmer der Reversen Transkriptase des HIV- Virus. Diese Substanzen werden von Enzymen der Wirtszelle in Triphosphate umgewandelt, werden aber von Polymerase α, δ und ε nicht als Substrate erkannt. Ihre Nebenwirkungen sind mit dem Einbau in die DNA durch Polymerase β, die in der Basenexzisionsreparatur tätig ist, verbunden. ACYCLOVIR 3.13 Der Zellzyklus Voraussetzung für jede Zellteilung (Mitose) ist die Replikation der DNA, um identische Kopien für jede Tochterzelle bereitzustellen. Die Replikation erfolgt in der synthetischen oder S-Phase während der Interphase (zwischen zwei Mitosen). Ein ganzer Zellzyklus (G 1 - S - G 2 - M) dauert bei sich kontinuierlich teilenden Zellen (z.b. Darmmucosa, Hautepithel, Erythrozyten-Vorstufe) ca Std. Andere Zellarten teilen sich nur auf ein Signal hin (z.b. Lymphozyten), selten (Leber- und Nierenzellen) oder sozusagen nie (Nerven- und Muskelzellen). In diesen Fällen ist die G 1 -Phase von ca. 5 Std. auf Tage oder Wochen verlängert. Eine derart lange G 1 -Phase wird auch als G 0 bezeichnet. S-, G 2 - und M-Phase dauern ca. 7, 3 und 2 Std.
15 Page 15 of 16 Bild: Zellzyklus der Eukaryoten. M (Mitose);S (DNA-Synthese); G 1 (gap1; Lücke) und G 2 (gap2) Der Eintritt in die Mitose wird von einer Protein-Kinase (CDK2-Kinase; cdc2 in Hefe) kontrolliert. Sie wird durch Cyclin B aktiviert, ein im Zellzyklus synthetisiertes und abgebautes Protein. Die katalytische Aktivität der cdc2- Kinase hängt auch von ihrem Phosphorylierungszustand ab, der durch Signale von Wachstumsfaktoren kontrolliert wird. Bild: Cyclin B (gelb), das während der Interphase gebildet wird, formt mit der cdc2-kinase (blau) einen inaktiven reifungsfördernden Faktor (MPF). Aufgrund der Phosphorylierung an Tyr 15 (hemmende Stelle) und danach Thr 161 (stimulierende Stelle) bleibt MPF inaktiv. Die Kinaseaktivität wird angeschaltet, sobald Cdc25, eine Phosphatase, die Phosphorylgruppe vom Tyr 15 entfernt. Der aktivierte MPF phosphoryliert dann seinerseits viele Zielproteine und initiiert damit die Mitose. Der ebenfalls durch die Kinase ausgelöste Abbau von Cyclin beendet die enzymatische Aktivität des Komplexes wieder.
16 Page 16 of 16 Lernziele Konzept der semikonservativen DNA-Synthese grundsätzlich verstehen Notwendigkeit des Primers kennen Kriterien der DNA-Synthese kennen (Matrize, Primer, dntps, Mg++, Richtung der Synthese) Rolle von PolI und PolIII in der DNA-Replikation von E. coli kennen Rolle der 5`--> 3`- und 3`--> 5`-Exonucleasen verstehen (Beispiel: Pol I von E. coli) Rolle von Pol α und Pol δ(ε) in der eukaryotischen DNA-Synthese kennen Unterschied zwischen Leitstrang- und Folgestrang-Synthese kennen Konzept der Prozessivität verstehen (E. coli: β-untereinheit der Pol III, Eukaryoten: PCNA) Funktion der DNA-Ligase kennen Konzept der Superspiralisierung verstehen Topoisomerasen: Klassen kennen und Mechanismen verstehen Telomerase: Funktion, enzymatische Aktivitäten und Matrize kennen Chromatin-Bestandteile kennen Unterschied zwischen Euchromatin und Heterochromatin kennen (Rolle der posttranskriptionellen Modifikation von DNA und Histonen) Hemmstoffe der DNA-Synthese Zellzyklus: alle Phasen kennen, Rolle der verschiedenen Zykline und deren Phosphorylierung während des Zyklus verstehen
Weitergabe genetischer Information: DNA-Replikation Beispiel: Escherichia coli.
 Weitergabe genetischer Information: DNA-Replikation Beispiel: Escherichia coli. zirkuläres bakterielles Chromosom Replikation (Erstellung einer identischen Kopie des genetischen Materials) MPM 1 DNA-Polymerasen
Weitergabe genetischer Information: DNA-Replikation Beispiel: Escherichia coli. zirkuläres bakterielles Chromosom Replikation (Erstellung einer identischen Kopie des genetischen Materials) MPM 1 DNA-Polymerasen
DNA Replikation ist semikonservativ. Abb. aus Stryer (5th Ed.)
 DNA Replikation ist semikonservativ Entwindung der DNA-Doppelhelix durch eine Helikase Replikationsgabel Eltern-DNA Beide DNA-Stränge werden in 5 3 Richtung synthetisiert DNA-Polymerasen katalysieren die
DNA Replikation ist semikonservativ Entwindung der DNA-Doppelhelix durch eine Helikase Replikationsgabel Eltern-DNA Beide DNA-Stränge werden in 5 3 Richtung synthetisiert DNA-Polymerasen katalysieren die
Die DNA Replikation. Exakte Verdopplung des genetischen Materials. Musterstrang. Neuer Strang. Neuer Strang. Eltern-DNA-Doppelstrang.
 Die DNA Replikation Musterstrang Neuer Strang Eltern-DNA-Doppelstrang Neuer Strang Musterstrang Exakte Verdopplung des genetischen Materials Die Reaktion der DNA Polymerase 5`-Triphosphat Nächstes Desoxyribonucleosidtriphosphat
Die DNA Replikation Musterstrang Neuer Strang Eltern-DNA-Doppelstrang Neuer Strang Musterstrang Exakte Verdopplung des genetischen Materials Die Reaktion der DNA Polymerase 5`-Triphosphat Nächstes Desoxyribonucleosidtriphosphat
Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr
 Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr Name: Matrikel-Nr.: Code Nummer: Bitte geben Sie Ihre Matrikel-Nr. und Ihren Namen an. Die Code-Nummer erhalten Sie zu Beginn
Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr Name: Matrikel-Nr.: Code Nummer: Bitte geben Sie Ihre Matrikel-Nr. und Ihren Namen an. Die Code-Nummer erhalten Sie zu Beginn
DNA-Replikation. Konrad Beyreuther. Stefan Kins
 DNA-Replikation Konrad Beyreuther Stefan Kins DNA-Replikation Originalgetreue Verdopplung des genetischen Materials als Voraussetzung für die kontinuierliche Weitergabe der in der DNA verschlüsselten Information
DNA-Replikation Konrad Beyreuther Stefan Kins DNA-Replikation Originalgetreue Verdopplung des genetischen Materials als Voraussetzung für die kontinuierliche Weitergabe der in der DNA verschlüsselten Information
6. DNA -Bakteriengenetik
 6. DNA -Bakteriengenetik Konzepte: Francis Crick DNA Struktur DNA Replikation Gentransfer in Bakterien Bakteriophagen 2. Welcher der folgenden Sätze entspricht der Chargaff-Regel? A) Die Menge von Purinen
6. DNA -Bakteriengenetik Konzepte: Francis Crick DNA Struktur DNA Replikation Gentransfer in Bakterien Bakteriophagen 2. Welcher der folgenden Sätze entspricht der Chargaff-Regel? A) Die Menge von Purinen
DNA-Sequenzierung. Martina Krause
 DNA-Sequenzierung Martina Krause Inhalt Methoden der DNA-Sequenzierung Methode nach Maxam - Gilbert Methode nach Sanger Einleitung Seit 1977 stehen zwei schnell arbeitende Möglichkeiten der Sequenzanalyse
DNA-Sequenzierung Martina Krause Inhalt Methoden der DNA-Sequenzierung Methode nach Maxam - Gilbert Methode nach Sanger Einleitung Seit 1977 stehen zwei schnell arbeitende Möglichkeiten der Sequenzanalyse
Anabole Prozesse in der Zelle
 Anabole Prozesse in der Zelle DNA Vermehrung RNA Synthese Protein Synthese Protein Verteilung in der Zelle Ziel: Zellteilung (Wachstum) und Differenzierung (Aufgabenteilung im Organismus). 2016 Struktur
Anabole Prozesse in der Zelle DNA Vermehrung RNA Synthese Protein Synthese Protein Verteilung in der Zelle Ziel: Zellteilung (Wachstum) und Differenzierung (Aufgabenteilung im Organismus). 2016 Struktur
Dr. Jens Kurreck. Otto-Hahn-Bau, Thielallee 63, Raum 029 Tel.: 83 85 69 69 Email: jkurreck@chemie.fu-berlin.de
 Dr. Jens Kurreck Otto-Hahn-Bau, Thielallee 63, Raum 029 Tel.: 83 85 69 69 Email: jkurreck@chemie.fu-berlin.de Prinzipien genetischer Informationsübertragung Berg, Tymoczko, Stryer: Biochemie 5. Auflage,
Dr. Jens Kurreck Otto-Hahn-Bau, Thielallee 63, Raum 029 Tel.: 83 85 69 69 Email: jkurreck@chemie.fu-berlin.de Prinzipien genetischer Informationsübertragung Berg, Tymoczko, Stryer: Biochemie 5. Auflage,
KV: DNA-Replikation Michael Altmann
 Institut für Biochemie und Molekulare Medizin KV: DNA-Replikation Michael Altmann Herbstsemester 2008/2009 Übersicht VL DNA-Replikation 1.) Das Zentraldogma der Molekularbiologie 1.) Semikonservative Replikation
Institut für Biochemie und Molekulare Medizin KV: DNA-Replikation Michael Altmann Herbstsemester 2008/2009 Übersicht VL DNA-Replikation 1.) Das Zentraldogma der Molekularbiologie 1.) Semikonservative Replikation
Überblick von DNA zu Protein. Biochemie-Seminar WS 04/05
 Überblick von DNA zu Protein Biochemie-Seminar WS 04/05 Replikationsapparat der Zelle Der gesamte Replikationsapparat umfasst über 20 Proteine z.b. DNA Polymerase: katalysiert Zusammenfügen einzelner Bausteine
Überblick von DNA zu Protein Biochemie-Seminar WS 04/05 Replikationsapparat der Zelle Der gesamte Replikationsapparat umfasst über 20 Proteine z.b. DNA Polymerase: katalysiert Zusammenfügen einzelner Bausteine
Das Prinzip der DNA-Sequenzierung Best.- Nr. 201.3055
 Das Prinzip der DNA-Sequenzierung Best.- Nr. 201.3055 Allgemeine Informationen Prinzip der DNA-Sequenzierung Ziel dieses Versuchs ist einen genauen Überblick über die Verfahrensweise der DNA- Sequenzierung
Das Prinzip der DNA-Sequenzierung Best.- Nr. 201.3055 Allgemeine Informationen Prinzip der DNA-Sequenzierung Ziel dieses Versuchs ist einen genauen Überblick über die Verfahrensweise der DNA- Sequenzierung
DNA-Replikation. Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination
 DNA-Replikation Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination Die Initiation der DNA-Replikation bei Eukaryoten am ori erfolgt erst nach der Lizensierung durch ORC und weitere Proteine
DNA-Replikation Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination Die Initiation der DNA-Replikation bei Eukaryoten am ori erfolgt erst nach der Lizensierung durch ORC und weitere Proteine
Klonierung von S2P Rolle der M19-Zellen. POL-Seminar der Biochemie II 13.02.2007 Sebastian Gabriel
 Klonierung von S2P Rolle der M19-Zellen POL-Seminar der Biochemie II 13.02.2007 Sebastian Gabriel Inhalt 1. Was ist eine humane genomische DNA-Bank? 2. Unterschied zwischen cdna-bank und genomischer DNA-Bank?
Klonierung von S2P Rolle der M19-Zellen POL-Seminar der Biochemie II 13.02.2007 Sebastian Gabriel Inhalt 1. Was ist eine humane genomische DNA-Bank? 2. Unterschied zwischen cdna-bank und genomischer DNA-Bank?
Zeichen bei Zahlen entschlüsseln
 Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Expression der genetischen Information Skript: Kapitel 5
 Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Biochemisches Grundpraktikum
 Biochemisches Grundpraktikum Dr. Ellen Hornung; Email: ehornun@gwdg.de; Tel: 39-5748 Einteilung der Praktikumsplätze: Eintragen in Listen am - Dienstag, 10.11.2009, von 12:00 13:00 - Freitag, 13.11.2009,
Biochemisches Grundpraktikum Dr. Ellen Hornung; Email: ehornun@gwdg.de; Tel: 39-5748 Einteilung der Praktikumsplätze: Eintragen in Listen am - Dienstag, 10.11.2009, von 12:00 13:00 - Freitag, 13.11.2009,
Zellzyklus, Replikation und Chromosomen
 Zellzyklus, Replikation und Chromosomen Wiederholung: Größenverhältnisse im DNA-Molekül 3 5 Das größte menschliche Chromosom enthält 247 Millionen Basenpaare Moleküllänge: 8.4 cm Die Länge des gesamten
Zellzyklus, Replikation und Chromosomen Wiederholung: Größenverhältnisse im DNA-Molekül 3 5 Das größte menschliche Chromosom enthält 247 Millionen Basenpaare Moleküllänge: 8.4 cm Die Länge des gesamten
Biochemie Seminar. Struktur und Organisation von Nukleinsäuren Genomorganisation DNA-Replikation
 Biochemie Seminar Struktur und Organisation von Nukleinsäuren Genomorganisation DNA-Replikation Dr. Jessica Tröger jessica.troeger@med.uni-jena.de Tel.: 938637 Adenosin Cytidin Guanosin Thymidin Nukleotide:
Biochemie Seminar Struktur und Organisation von Nukleinsäuren Genomorganisation DNA-Replikation Dr. Jessica Tröger jessica.troeger@med.uni-jena.de Tel.: 938637 Adenosin Cytidin Guanosin Thymidin Nukleotide:
Von der DNA zum Eiweißmolekül Die Proteinbiosynthese. Ribosom
 Von der DNA zum Eiweißmolekül Die Proteinbiosynthese Ribosom Wiederholung: DNA-Replikation und Chromosomenkondensation / Mitose Jede Zelle macht von Teilung zu Teilung einen Zellzyklus durch, der aus einer
Von der DNA zum Eiweißmolekül Die Proteinbiosynthese Ribosom Wiederholung: DNA-Replikation und Chromosomenkondensation / Mitose Jede Zelle macht von Teilung zu Teilung einen Zellzyklus durch, der aus einer
Übung 8. 1. Zellkommunikation. Vorlesung Bio-Engineering Sommersemester 2008. Kapitel 4. 4
 Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 05. 05. 08
Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 05. 05. 08
Klausur zur Vorlesung Biochemie III im WS 2000/01
 Klausur zur Vorlesung Biochemie III im WS 2000/01 am 15.02.2001 von 15.30 17.00 Uhr (insgesamt 100 Punkte, mindestens 40 erforderlich) Bitte Name, Matrikelnummer und Studienfach unbedingt angeben (3 1.
Klausur zur Vorlesung Biochemie III im WS 2000/01 am 15.02.2001 von 15.30 17.00 Uhr (insgesamt 100 Punkte, mindestens 40 erforderlich) Bitte Name, Matrikelnummer und Studienfach unbedingt angeben (3 1.
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren
 Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
Unterrichtsmaterialien in digitaler und in gedruckter Form. Auszug aus: Antibiotikaresistenz bei Pseudomonaden
 Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Antibiotikaresistenz bei Pseudomonaden Das komplette Material finden Sie hier: School-Scout.de S 1 M 6 M 7 Erarbeitung II, Präsentation
Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Antibiotikaresistenz bei Pseudomonaden Das komplette Material finden Sie hier: School-Scout.de S 1 M 6 M 7 Erarbeitung II, Präsentation
Outlook. sysplus.ch outlook - mail-grundlagen Seite 1/8. Mail-Grundlagen. Posteingang
 sysplus.ch outlook - mail-grundlagen Seite 1/8 Outlook Mail-Grundlagen Posteingang Es gibt verschiedene Möglichkeiten, um zum Posteingang zu gelangen. Man kann links im Outlook-Fenster auf die Schaltfläche
sysplus.ch outlook - mail-grundlagen Seite 1/8 Outlook Mail-Grundlagen Posteingang Es gibt verschiedene Möglichkeiten, um zum Posteingang zu gelangen. Man kann links im Outlook-Fenster auf die Schaltfläche
Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig)
 Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Jetzt liegen diese Stränge einzeln
Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Jetzt liegen diese Stränge einzeln
Grundlagen der Theoretischen Informatik, SoSe 2008
 1. Aufgabenblatt zur Vorlesung Grundlagen der Theoretischen Informatik, SoSe 2008 (Dr. Frank Hoffmann) Lösung von Manuel Jain und Benjamin Bortfeldt Aufgabe 2 Zustandsdiagramme (6 Punkte, wird korrigiert)
1. Aufgabenblatt zur Vorlesung Grundlagen der Theoretischen Informatik, SoSe 2008 (Dr. Frank Hoffmann) Lösung von Manuel Jain und Benjamin Bortfeldt Aufgabe 2 Zustandsdiagramme (6 Punkte, wird korrigiert)
4. Genetische Mechanismen bei Bakterien
 4. Genetische Mechanismen bei Bakterien 4.1 Makromoleküle und genetische Information Aufbau der DNA Phasen des Informationsflusses Vergleich der Informationsübertragung bei Pro- und Eukaryoten 4.2 Struktur
4. Genetische Mechanismen bei Bakterien 4.1 Makromoleküle und genetische Information Aufbau der DNA Phasen des Informationsflusses Vergleich der Informationsübertragung bei Pro- und Eukaryoten 4.2 Struktur
Professionelle Seminare im Bereich MS-Office
 Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2014
 Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2014 Fragen für die Übungsstunde 8 (14.07-18.07.) 1) Von der DNA-Sequenz zum Protein Sie können
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2014 Fragen für die Übungsstunde 8 (14.07-18.07.) 1) Von der DNA-Sequenz zum Protein Sie können
Übung 11 Genregulation bei Prokaryoten
 Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Regulation des Zellcylus
 Regulation des Zellcylus Dr. F. Neuschäfer-Rube Cyclin-abhängige Kinasen (CDKs) Cyclin-abhängige Kinasen: Motoren und Schalter des Zellzyclus Dr. F. Neuschäfer-Rube Der Zellzyklus M S Der Zellzyklus M
Regulation des Zellcylus Dr. F. Neuschäfer-Rube Cyclin-abhängige Kinasen (CDKs) Cyclin-abhängige Kinasen: Motoren und Schalter des Zellzyclus Dr. F. Neuschäfer-Rube Der Zellzyklus M S Der Zellzyklus M
Q1 B1 KW 49. Genregulation
 Q1 B1 KW 49 Genregulation Transkription Posttranskription elle Modifikation Genregulation bei Eukaryoten Transkriptionsfaktoren (an TATA- Box) oder Silencer (verringert Transkription) und Enhancer (erhöht
Q1 B1 KW 49 Genregulation Transkription Posttranskription elle Modifikation Genregulation bei Eukaryoten Transkriptionsfaktoren (an TATA- Box) oder Silencer (verringert Transkription) und Enhancer (erhöht
Das große ElterngeldPlus 1x1. Alles über das ElterngeldPlus. Wer kann ElterngeldPlus beantragen? ElterngeldPlus verstehen ein paar einleitende Fakten
 Das große x -4 Alles über das Wer kann beantragen? Generell kann jeder beantragen! Eltern (Mütter UND Väter), die schon während ihrer Elternzeit wieder in Teilzeit arbeiten möchten. Eltern, die während
Das große x -4 Alles über das Wer kann beantragen? Generell kann jeder beantragen! Eltern (Mütter UND Väter), die schon während ihrer Elternzeit wieder in Teilzeit arbeiten möchten. Eltern, die während
Würfelt man dabei je genau 10 - mal eine 1, 2, 3, 4, 5 und 6, so beträgt die Anzahl. der verschiedenen Reihenfolgen, in denen man dies tun kann, 60!.
 040304 Übung 9a Analysis, Abschnitt 4, Folie 8 Die Wahrscheinlichkeit, dass bei n - maliger Durchführung eines Zufallexperiments ein Ereignis A ( mit Wahrscheinlichkeit p p ( A ) ) für eine beliebige Anzahl
040304 Übung 9a Analysis, Abschnitt 4, Folie 8 Die Wahrscheinlichkeit, dass bei n - maliger Durchführung eines Zufallexperiments ein Ereignis A ( mit Wahrscheinlichkeit p p ( A ) ) für eine beliebige Anzahl
Versuch 8. Plasmid - Isolierung
 Versuch 8 Plasmid - Isolierung Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@quantentunnel.de X X X C. Weindel & M. Schwarz Wird benotet?: Einleitung Ein Plasmid
Versuch 8 Plasmid - Isolierung Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@quantentunnel.de X X X C. Weindel & M. Schwarz Wird benotet?: Einleitung Ein Plasmid
Dieser Ablauf soll eine Hilfe für die tägliche Arbeit mit der SMS Bestätigung im Millennium darstellen.
 Millennium SMS Service Schnellübersicht Seite 1 von 6 1. Tägliche Arbeiten mit der SMS Bestätigung Dieser Ablauf soll eine Hilfe für die tägliche Arbeit mit der SMS Bestätigung im Millennium darstellen.
Millennium SMS Service Schnellübersicht Seite 1 von 6 1. Tägliche Arbeiten mit der SMS Bestätigung Dieser Ablauf soll eine Hilfe für die tägliche Arbeit mit der SMS Bestätigung im Millennium darstellen.
Während der Synthese synthetisiert die Polymerase den neuen Strang in 5 3 Richtung und bewegt sich in 3 5 -Richtung am Matrizenstrang entlang:
 4.4 Replikation und PCR Ablauf der Replikation in vivo: Die Replikation wird von einer DNA-abhängigen DNA- Polymerase katalysiert. Jede DNA-Polymerase synthetisiert den neuen Strang in 5 3 Richtung, hierzu
4.4 Replikation und PCR Ablauf der Replikation in vivo: Die Replikation wird von einer DNA-abhängigen DNA- Polymerase katalysiert. Jede DNA-Polymerase synthetisiert den neuen Strang in 5 3 Richtung, hierzu
Anleitung über den Umgang mit Schildern
 Anleitung über den Umgang mit Schildern -Vorwort -Wo bekommt man Schilder? -Wo und wie speichert man die Schilder? -Wie füge ich die Schilder in meinen Track ein? -Welche Bauteile kann man noch für Schilder
Anleitung über den Umgang mit Schildern -Vorwort -Wo bekommt man Schilder? -Wo und wie speichert man die Schilder? -Wie füge ich die Schilder in meinen Track ein? -Welche Bauteile kann man noch für Schilder
Artikel Schnittstelle über CSV
 Artikel Schnittstelle über CSV Sie können Artikeldaten aus Ihrem EDV System in das NCFOX importieren, dies geschieht durch eine CSV Schnittstelle. Dies hat mehrere Vorteile: Zeitersparnis, die Karteikarte
Artikel Schnittstelle über CSV Sie können Artikeldaten aus Ihrem EDV System in das NCFOX importieren, dies geschieht durch eine CSV Schnittstelle. Dies hat mehrere Vorteile: Zeitersparnis, die Karteikarte
Stellen Sie bitte den Cursor in die Spalte B2 und rufen die Funktion Sverweis auf. Es öffnet sich folgendes Dialogfenster
 Es gibt in Excel unter anderem die so genannten Suchfunktionen / Matrixfunktionen Damit können Sie Werte innerhalb eines bestimmten Bereichs suchen. Als Beispiel möchte ich die Funktion Sverweis zeigen.
Es gibt in Excel unter anderem die so genannten Suchfunktionen / Matrixfunktionen Damit können Sie Werte innerhalb eines bestimmten Bereichs suchen. Als Beispiel möchte ich die Funktion Sverweis zeigen.
Online Schulung Anmerkungen zur Durchführung
 Online Schulung Anmerkungen zur Durchführung 1.0 Einleitung Vielen Dank, dass Sie sich für die Online Schulung von SoloProtect entschieden haben. Nachfolgend finden Sie Informationen für Identicomnutzer
Online Schulung Anmerkungen zur Durchführung 1.0 Einleitung Vielen Dank, dass Sie sich für die Online Schulung von SoloProtect entschieden haben. Nachfolgend finden Sie Informationen für Identicomnutzer
Bei der Focus Methode handelt es sich um eine Analyse-Methode die der Erkennung und Abstellung von Fehlerzuständen dient.
 Beschreibung der Focus Methode Bei der Focus Methode handelt es sich um eine Analyse-Methode die der Erkennung und Abstellung von Fehlerzuständen dient. 1. F = Failure / Finding An dieser Stelle wird der
Beschreibung der Focus Methode Bei der Focus Methode handelt es sich um eine Analyse-Methode die der Erkennung und Abstellung von Fehlerzuständen dient. 1. F = Failure / Finding An dieser Stelle wird der
Erfahrungen mit Hartz IV- Empfängern
 Erfahrungen mit Hartz IV- Empfängern Ausgewählte Ergebnisse einer Befragung von Unternehmen aus den Branchen Gastronomie, Pflege und Handwerk Pressegespräch der Bundesagentur für Arbeit am 12. November
Erfahrungen mit Hartz IV- Empfängern Ausgewählte Ergebnisse einer Befragung von Unternehmen aus den Branchen Gastronomie, Pflege und Handwerk Pressegespräch der Bundesagentur für Arbeit am 12. November
Stellvertretenden Genehmiger verwalten. Tipps & Tricks
 Tipps & Tricks INHALT SEITE 1. Grundlegende Informationen 3 2.1 Aktivieren eines Stellvertretenden Genehmigers 4 2.2 Deaktivieren eines Stellvertretenden Genehmigers 11 2 1. Grundlegende Informationen
Tipps & Tricks INHALT SEITE 1. Grundlegende Informationen 3 2.1 Aktivieren eines Stellvertretenden Genehmigers 4 2.2 Deaktivieren eines Stellvertretenden Genehmigers 11 2 1. Grundlegende Informationen
Simulation LIF5000. Abbildung 1
 Simulation LIF5000 Abbildung 1 Zur Simulation von analogen Schaltungen verwende ich Ltspice/SwitcherCAD III. Dieses Programm ist sehr leistungsfähig und wenn man weis wie, dann kann man damit fast alles
Simulation LIF5000 Abbildung 1 Zur Simulation von analogen Schaltungen verwende ich Ltspice/SwitcherCAD III. Dieses Programm ist sehr leistungsfähig und wenn man weis wie, dann kann man damit fast alles
Anleitung zur Daten zur Datensicherung und Datenrücksicherung. Datensicherung
 Anleitung zur Daten zur Datensicherung und Datenrücksicherung Datensicherung Es gibt drei Möglichkeiten der Datensicherung. Zwei davon sind in Ges eingebaut, die dritte ist eine manuelle Möglichkeit. In
Anleitung zur Daten zur Datensicherung und Datenrücksicherung Datensicherung Es gibt drei Möglichkeiten der Datensicherung. Zwei davon sind in Ges eingebaut, die dritte ist eine manuelle Möglichkeit. In
1 Mathematische Grundlagen
 Mathematische Grundlagen - 1-1 Mathematische Grundlagen Der Begriff der Menge ist einer der grundlegenden Begriffe in der Mathematik. Mengen dienen dazu, Dinge oder Objekte zu einer Einheit zusammenzufassen.
Mathematische Grundlagen - 1-1 Mathematische Grundlagen Der Begriff der Menge ist einer der grundlegenden Begriffe in der Mathematik. Mengen dienen dazu, Dinge oder Objekte zu einer Einheit zusammenzufassen.
Anleitung für den Zugriff auf Mitgliederdateien der AG-KiM
 Anleitung für den Zugriff auf Mitgliederdateien der AG-KiM Hinweise: - Dies ist eine schrittweise Anleitung um auf den Server der Ag-Kim zuzugreifen. Hierbei können Dateien ähnlich wie bei Dropbox hoch-
Anleitung für den Zugriff auf Mitgliederdateien der AG-KiM Hinweise: - Dies ist eine schrittweise Anleitung um auf den Server der Ag-Kim zuzugreifen. Hierbei können Dateien ähnlich wie bei Dropbox hoch-
AGROPLUS Buchhaltung. Daten-Server und Sicherheitskopie. Version vom 21.10.2013b
 AGROPLUS Buchhaltung Daten-Server und Sicherheitskopie Version vom 21.10.2013b 3a) Der Daten-Server Modus und der Tresor Der Daten-Server ist eine Betriebsart welche dem Nutzer eine grosse Flexibilität
AGROPLUS Buchhaltung Daten-Server und Sicherheitskopie Version vom 21.10.2013b 3a) Der Daten-Server Modus und der Tresor Der Daten-Server ist eine Betriebsart welche dem Nutzer eine grosse Flexibilität
DNA-Synthese in vivo und in vitro
 DNA-Synthese in vivo und in vitro 4 Einführung Replikation der DNA Entwindung der DNA Priming der DNA-Synthese Struktur und Funktion der DNA-Polymerase Synthese des Folgestranges Fehlpaarungsreparatur
DNA-Synthese in vivo und in vitro 4 Einführung Replikation der DNA Entwindung der DNA Priming der DNA-Synthese Struktur und Funktion der DNA-Polymerase Synthese des Folgestranges Fehlpaarungsreparatur
IBIS Professional. z Dokumentation zur Dublettenprüfung
 z Dokumentation zur Dublettenprüfung Die Dublettenprüfung ist ein Zusatzpaket zur IBIS-Shopverwaltung für die Classic Line 3.4 und höher. Dubletten entstehen dadurch, dass viele Kunden beim Bestellvorgang
z Dokumentation zur Dublettenprüfung Die Dublettenprüfung ist ein Zusatzpaket zur IBIS-Shopverwaltung für die Classic Line 3.4 und höher. Dubletten entstehen dadurch, dass viele Kunden beim Bestellvorgang
Labortechniken (2) Amplifikationskurve (linear linear) Realtime-PCR
 Realtime PCR Quantitative PCR Prinzip: Nachweis der PCR-Produkte in Echtzeit und Quantifizierung anhand eines fluoreszenten Reporters R Q Taq-Polymerase Synthese- und Nuklease-Einheit R=Reporter, Q=Quencher
Realtime PCR Quantitative PCR Prinzip: Nachweis der PCR-Produkte in Echtzeit und Quantifizierung anhand eines fluoreszenten Reporters R Q Taq-Polymerase Synthese- und Nuklease-Einheit R=Reporter, Q=Quencher
AZK 1- Freistil. Der Dialog "Arbeitszeitkonten" Grundsätzliches zum Dialog "Arbeitszeitkonten"
 AZK 1- Freistil Nur bei Bedarf werden dafür gekennzeichnete Lohnbestandteile (Stundenzahl und Stundensatz) zwischen dem aktuellen Bruttolohnjournal und dem AZK ausgetauscht. Das Ansparen und das Auszahlen
AZK 1- Freistil Nur bei Bedarf werden dafür gekennzeichnete Lohnbestandteile (Stundenzahl und Stundensatz) zwischen dem aktuellen Bruttolohnjournal und dem AZK ausgetauscht. Das Ansparen und das Auszahlen
Tutorial: Entlohnungsberechnung erstellen mit LibreOffice Calc 3.5
 Tutorial: Entlohnungsberechnung erstellen mit LibreOffice Calc 3.5 In diesem Tutorial will ich Ihnen zeigen, wie man mit LibreOffice Calc 3.5 eine einfache Entlohnungsberechnung erstellt, wobei eine automatische
Tutorial: Entlohnungsberechnung erstellen mit LibreOffice Calc 3.5 In diesem Tutorial will ich Ihnen zeigen, wie man mit LibreOffice Calc 3.5 eine einfache Entlohnungsberechnung erstellt, wobei eine automatische
Lineare Gleichungssysteme
 Lineare Gleichungssysteme 1 Zwei Gleichungen mit zwei Unbekannten Es kommt häufig vor, dass man nicht mit einer Variablen alleine auskommt, um ein Problem zu lösen. Das folgende Beispiel soll dies verdeutlichen
Lineare Gleichungssysteme 1 Zwei Gleichungen mit zwei Unbekannten Es kommt häufig vor, dass man nicht mit einer Variablen alleine auskommt, um ein Problem zu lösen. Das folgende Beispiel soll dies verdeutlichen
Datensicherung. Beschreibung der Datensicherung
 Datensicherung Mit dem Datensicherungsprogramm können Sie Ihre persönlichen Daten problemlos Sichern. Es ist möglich eine komplette Datensicherung durchzuführen, aber auch nur die neuen und geänderten
Datensicherung Mit dem Datensicherungsprogramm können Sie Ihre persönlichen Daten problemlos Sichern. Es ist möglich eine komplette Datensicherung durchzuführen, aber auch nur die neuen und geänderten
FlowFact Alle Versionen
 Training FlowFact Alle Versionen Stand: 29.09.2005 Rechnung schreiben Einführung Wie Sie inzwischen wissen, können die unterschiedlichsten Daten über verknüpfte Fenster miteinander verbunden werden. Für
Training FlowFact Alle Versionen Stand: 29.09.2005 Rechnung schreiben Einführung Wie Sie inzwischen wissen, können die unterschiedlichsten Daten über verknüpfte Fenster miteinander verbunden werden. Für
Adami CRM - Outlook Replikation User Dokumentation
 Adami CRM - Outlook Replikation User Dokumentation Die neue Eigenschaft der Adami CRM Applikation macht den Information Austausch mit Microsoft Outlook auf vier Ebenen möglich: Kontakte, Aufgaben, Termine
Adami CRM - Outlook Replikation User Dokumentation Die neue Eigenschaft der Adami CRM Applikation macht den Information Austausch mit Microsoft Outlook auf vier Ebenen möglich: Kontakte, Aufgaben, Termine
Dokumentation für die software für zahnärzte der procedia GmbH Onlinedokumentation
 Dokumentation für die software für zahnärzte der procedia GmbH Onlinedokumentation (Bei Abweichungen, die bspw. durch technischen Fortschritt entstehen können, ziehen Sie bitte immer das aktuelle Handbuch
Dokumentation für die software für zahnärzte der procedia GmbH Onlinedokumentation (Bei Abweichungen, die bspw. durch technischen Fortschritt entstehen können, ziehen Sie bitte immer das aktuelle Handbuch
Unterrichtsmaterialien in digitaler und in gedruckter Form. Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis
 Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis Das komplette Material finden Sie hier: Download bei School-Scout.de
Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis Das komplette Material finden Sie hier: Download bei School-Scout.de
Kurzanleitung MAN E-Learning (WBT)
 Kurzanleitung MAN E-Learning (WBT) Um Ihr gebuchtes E-Learning zu bearbeiten, starten Sie bitte das MAN Online- Buchungssystem (ICPM / Seminaris) unter dem Link www.man-academy.eu Klicken Sie dann auf
Kurzanleitung MAN E-Learning (WBT) Um Ihr gebuchtes E-Learning zu bearbeiten, starten Sie bitte das MAN Online- Buchungssystem (ICPM / Seminaris) unter dem Link www.man-academy.eu Klicken Sie dann auf
Nucleophiler Angriff Ein Nucleophil greift ein positiv polarisiertes Kohlenstoff in einer Verbindung an.
 Weiter im Text: Der RNA-Primer, kann die DNA nucleophil angreifen. Nucleophil: ein stark "kernliebendes" Teilchen, das negativ polarisiert ist (z.b. OH-) und ein positiv polarisiertes (elektronenarmes)
Weiter im Text: Der RNA-Primer, kann die DNA nucleophil angreifen. Nucleophil: ein stark "kernliebendes" Teilchen, das negativ polarisiert ist (z.b. OH-) und ein positiv polarisiertes (elektronenarmes)
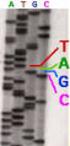
1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet und
 10. Methoden 1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet und welche Funktion haben diese bei der Sequenzierungsreaktion?
10. Methoden 1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet und welche Funktion haben diese bei der Sequenzierungsreaktion?
Fax einrichten auf Windows XP-PC
 Um ein PC Fax fähig zu machen braucht man einen sogenannten Telefon Anschluss A/B das heißt, Fax funktioniert im Normalfall nur mit Modem nicht mit DSL. Die meisten neueren PCs haben ein Modem integriert.
Um ein PC Fax fähig zu machen braucht man einen sogenannten Telefon Anschluss A/B das heißt, Fax funktioniert im Normalfall nur mit Modem nicht mit DSL. Die meisten neueren PCs haben ein Modem integriert.
Hinweise zum Ausfüllen der Zeiterfassung
 Hinweise zum Ausfüllen der Zeiterfassung Generelle Hinweise zu Excel Ab Version VSA 4.50 wird die dezimale Schreibweise für Zeiteingaben verwendet. Die Zeiterfassung, die Sie erhalten haben wurde für Excel
Hinweise zum Ausfüllen der Zeiterfassung Generelle Hinweise zu Excel Ab Version VSA 4.50 wird die dezimale Schreibweise für Zeiteingaben verwendet. Die Zeiterfassung, die Sie erhalten haben wurde für Excel
1. Einschränkung für Mac-User ohne Office 365. 2. Dokumente hochladen, teilen und bearbeiten
 1. Einschränkung für Mac-User ohne Office 365 Mac-User ohne Office 365 müssen die Dateien herunterladen; sie können die Dateien nicht direkt öffnen und bearbeiten. Wenn die Datei heruntergeladen wurde,
1. Einschränkung für Mac-User ohne Office 365 Mac-User ohne Office 365 müssen die Dateien herunterladen; sie können die Dateien nicht direkt öffnen und bearbeiten. Wenn die Datei heruntergeladen wurde,
ACDSee Pro 2. ACDSee Pro 2 Tutorials: Übertragung von Fotos (+ Datenbank) auf einen anderen Computer. Über Metadaten und die Datenbank
 Tutorials: Übertragung von Fotos (+ ) auf einen anderen Computer Export der In dieser Lektion erfahren Sie, wie Sie am effektivsten Fotos von einem Computer auf einen anderen übertragen. Wenn Sie Ihre
Tutorials: Übertragung von Fotos (+ ) auf einen anderen Computer Export der In dieser Lektion erfahren Sie, wie Sie am effektivsten Fotos von einem Computer auf einen anderen übertragen. Wenn Sie Ihre
How to do? Projekte - Zeiterfassung
 How to do? Projekte - Zeiterfassung Stand: Version 4.0.1, 18.03.2009 1. EINLEITUNG...3 2. PROJEKTE UND STAMMDATEN...4 2.1 Projekte... 4 2.2 Projektmitarbeiter... 5 2.3 Tätigkeiten... 6 2.4 Unterprojekte...
How to do? Projekte - Zeiterfassung Stand: Version 4.0.1, 18.03.2009 1. EINLEITUNG...3 2. PROJEKTE UND STAMMDATEN...4 2.1 Projekte... 4 2.2 Projektmitarbeiter... 5 2.3 Tätigkeiten... 6 2.4 Unterprojekte...
Finanzierung: Übungsserie III Innenfinanzierung
 Thema Dokumentart Finanzierung: Übungsserie III Innenfinanzierung Lösungen Theorie im Buch "Integrale Betriebswirtschaftslehre" Teil: Kapitel: D1 Finanzmanagement 2.3 Innenfinanzierung Finanzierung: Übungsserie
Thema Dokumentart Finanzierung: Übungsserie III Innenfinanzierung Lösungen Theorie im Buch "Integrale Betriebswirtschaftslehre" Teil: Kapitel: D1 Finanzmanagement 2.3 Innenfinanzierung Finanzierung: Übungsserie
Studieren- Erklärungen und Tipps
 Studieren- Erklärungen und Tipps Es gibt Berufe, die man nicht lernen kann, sondern für die man ein Studium machen muss. Das ist zum Beispiel so wenn man Arzt oder Lehrer werden möchte. Hat ihr Kind das
Studieren- Erklärungen und Tipps Es gibt Berufe, die man nicht lernen kann, sondern für die man ein Studium machen muss. Das ist zum Beispiel so wenn man Arzt oder Lehrer werden möchte. Hat ihr Kind das
Biologie für Mediziner WS 2007/08
 Biologie für Mediziner WS 2007/08 Teil Allgemeine Genetik, Prof. Dr. Uwe Homberg 1. Endozytose 2. Lysosomen 3. Zellkern, Chromosomen 4. Struktur und Funktion der DNA, Replikation 5. Zellzyklus und Zellteilung
Biologie für Mediziner WS 2007/08 Teil Allgemeine Genetik, Prof. Dr. Uwe Homberg 1. Endozytose 2. Lysosomen 3. Zellkern, Chromosomen 4. Struktur und Funktion der DNA, Replikation 5. Zellzyklus und Zellteilung
IV. Übungsaufgaben für die Jahrgangstufe 9 & 10
 IV. Übungsaufgaben für die Jahrgangstufe 9 & 10 Von der Erbanlage zum Erbmerkmal: 34) Welche Aufgaben haben Chromosomen? 35) Zeichne und benenne die Teile eines Chromosoms, wie sie im Lichtmikroskop während
IV. Übungsaufgaben für die Jahrgangstufe 9 & 10 Von der Erbanlage zum Erbmerkmal: 34) Welche Aufgaben haben Chromosomen? 35) Zeichne und benenne die Teile eines Chromosoms, wie sie im Lichtmikroskop während
Was meinen die Leute eigentlich mit: Grexit?
 Was meinen die Leute eigentlich mit: Grexit? Grexit sind eigentlich 2 Wörter. 1. Griechenland 2. Exit Exit ist ein englisches Wort. Es bedeutet: Ausgang. Aber was haben diese 2 Sachen mit-einander zu tun?
Was meinen die Leute eigentlich mit: Grexit? Grexit sind eigentlich 2 Wörter. 1. Griechenland 2. Exit Exit ist ein englisches Wort. Es bedeutet: Ausgang. Aber was haben diese 2 Sachen mit-einander zu tun?
icloud nicht neu, aber doch irgendwie anders
 Kapitel 6 In diesem Kapitel zeigen wir Ihnen, welche Dienste die icloud beim Abgleich von Dateien und Informationen anbietet. Sie lernen icloud Drive kennen, den Fotostream, den icloud-schlüsselbund und
Kapitel 6 In diesem Kapitel zeigen wir Ihnen, welche Dienste die icloud beim Abgleich von Dateien und Informationen anbietet. Sie lernen icloud Drive kennen, den Fotostream, den icloud-schlüsselbund und
Download von Hörbüchern
 Manual PocketBook LUX und ebookit Download von Hörbüchern Wie kommen Hörbücher auf den PocketBook LUX? Rolf 11.10.2013 Inhalt Download von Hörbüchern... 2 Einleitung/Ausgangslage... 2 Download auf Notebook/PC...
Manual PocketBook LUX und ebookit Download von Hörbüchern Wie kommen Hörbücher auf den PocketBook LUX? Rolf 11.10.2013 Inhalt Download von Hörbüchern... 2 Einleitung/Ausgangslage... 2 Download auf Notebook/PC...
Kreativ visualisieren
 Kreativ visualisieren Haben Sie schon einmal etwas von sogenannten»sich selbst erfüllenden Prophezeiungen«gehört? Damit ist gemeint, dass ein Ereignis mit hoher Wahrscheinlichkeit eintritt, wenn wir uns
Kreativ visualisieren Haben Sie schon einmal etwas von sogenannten»sich selbst erfüllenden Prophezeiungen«gehört? Damit ist gemeint, dass ein Ereignis mit hoher Wahrscheinlichkeit eintritt, wenn wir uns
Eigenen Farbverlauf erstellen
 Diese Serie ist an totale Neulinge gerichtet. Neu bei PhotoLine, evtl. sogar komplett neu, was Bildbearbeitung betrifft. So versuche ich, hier alles einfach zu halten. Ich habe sogar PhotoLine ein zweites
Diese Serie ist an totale Neulinge gerichtet. Neu bei PhotoLine, evtl. sogar komplett neu, was Bildbearbeitung betrifft. So versuche ich, hier alles einfach zu halten. Ich habe sogar PhotoLine ein zweites
Anleitung RÄUME BUCHEN MIT OUTLOOK FÜR VERWALTUNGSANGESTELLTE
 Anleitung RÄUME BUCHEN MIT OUTLOOK FÜR VERWALTUNGSANGESTELLTE Dezernat 6 Abteilung 4 Stand: 14.Oktober 2014 Inhalt 1. Einleitung 3 2. Räume & gemeinsame Termine finden 3 3. Rüstzeit 8 4. FAQ: Oft gestellte
Anleitung RÄUME BUCHEN MIT OUTLOOK FÜR VERWALTUNGSANGESTELLTE Dezernat 6 Abteilung 4 Stand: 14.Oktober 2014 Inhalt 1. Einleitung 3 2. Räume & gemeinsame Termine finden 3 3. Rüstzeit 8 4. FAQ: Oft gestellte
Powermanager Server- Client- Installation
 Client A Server Client B Die Server- Client- Funktion ermöglicht es ein zentrales Powermanager Projekt von verschiedenen Client Rechnern aus zu bedienen. 1.0 Benötigte Voraussetzungen 1.1 Sowohl am Server
Client A Server Client B Die Server- Client- Funktion ermöglicht es ein zentrales Powermanager Projekt von verschiedenen Client Rechnern aus zu bedienen. 1.0 Benötigte Voraussetzungen 1.1 Sowohl am Server
Handbuch ECDL 2003 Basic Modul 5: Datenbank Grundlagen von relationalen Datenbanken
 Handbuch ECDL 2003 Basic Modul 5: Datenbank Grundlagen von relationalen Datenbanken Dateiname: ecdl5_01_00_documentation_standard.doc Speicherdatum: 14.02.2005 ECDL 2003 Basic Modul 5 Datenbank - Grundlagen
Handbuch ECDL 2003 Basic Modul 5: Datenbank Grundlagen von relationalen Datenbanken Dateiname: ecdl5_01_00_documentation_standard.doc Speicherdatum: 14.02.2005 ECDL 2003 Basic Modul 5 Datenbank - Grundlagen
SICHERN DER FAVORITEN
 Seite 1 von 7 SICHERN DER FAVORITEN Eine Anleitung zum Sichern der eigenen Favoriten zur Verfügung gestellt durch: ZID Dezentrale Systeme März 2010 Seite 2 von 7 Für die Datensicherheit ist bekanntlich
Seite 1 von 7 SICHERN DER FAVORITEN Eine Anleitung zum Sichern der eigenen Favoriten zur Verfügung gestellt durch: ZID Dezentrale Systeme März 2010 Seite 2 von 7 Für die Datensicherheit ist bekanntlich
Molekulargenetik der Eukaryoten WS 2014/15, VL 10. Erwin R. Schmidt Institut für Molekulargenetik
 Molekulargenetik der Eukaryoten WS 2014/15, VL 10 Erwin R. Schmidt Institut für Molekulargenetik Replikationsgabel bei Prokaryoten Replikationsgabel bei Eukaryoten Pol e Pol d GINS (Go, Ichi, Nii, and
Molekulargenetik der Eukaryoten WS 2014/15, VL 10 Erwin R. Schmidt Institut für Molekulargenetik Replikationsgabel bei Prokaryoten Replikationsgabel bei Eukaryoten Pol e Pol d GINS (Go, Ichi, Nii, and
1 Einleitung. Lernziele. Symbolleiste für den Schnellzugriff anpassen. Notizenseiten drucken. eine Präsentation abwärtskompatibel speichern
 1 Einleitung Lernziele Symbolleiste für den Schnellzugriff anpassen Notizenseiten drucken eine Präsentation abwärtskompatibel speichern eine Präsentation auf CD oder USB-Stick speichern Lerndauer 4 Minuten
1 Einleitung Lernziele Symbolleiste für den Schnellzugriff anpassen Notizenseiten drucken eine Präsentation abwärtskompatibel speichern eine Präsentation auf CD oder USB-Stick speichern Lerndauer 4 Minuten
APP-GFP/Fluoreszenzmikroskop. Aufnahmen neuronaler Zellen, mit freund. Genehmigung von Prof. Stefan Kins, TU Kaiserslautern
 Über die Herkunft von Aβ42 und Amyloid-Plaques Heute ist sicher belegt, dass sich die amyloiden Plaques aus einer Vielzahl an Abbaufragmenten des Amyloid-Vorläufer-Proteins (amyloid-precursor-protein,
Über die Herkunft von Aβ42 und Amyloid-Plaques Heute ist sicher belegt, dass sich die amyloiden Plaques aus einer Vielzahl an Abbaufragmenten des Amyloid-Vorläufer-Proteins (amyloid-precursor-protein,
Binäre Bäume. 1. Allgemeines. 2. Funktionsweise. 2.1 Eintragen
 Binäre Bäume 1. Allgemeines Binäre Bäume werden grundsätzlich verwendet, um Zahlen der Größe nach, oder Wörter dem Alphabet nach zu sortieren. Dem einfacheren Verständnis zu Liebe werde ich mich hier besonders
Binäre Bäume 1. Allgemeines Binäre Bäume werden grundsätzlich verwendet, um Zahlen der Größe nach, oder Wörter dem Alphabet nach zu sortieren. Dem einfacheren Verständnis zu Liebe werde ich mich hier besonders
Wollen Sie einen mühelosen Direkteinstieg zum Online Shop der ÖAG? Sie sind nur einen Klick davon entfernt!
 Wollen Sie einen mühelosen Direkteinstieg zum Online Shop der ÖAG? Sie sind nur einen Klick davon entfernt! Sehr geehrte(r) Geschäftspartner(in), Um Ihre Transaktionen schneller durchzuführen, bieten wir
Wollen Sie einen mühelosen Direkteinstieg zum Online Shop der ÖAG? Sie sind nur einen Klick davon entfernt! Sehr geehrte(r) Geschäftspartner(in), Um Ihre Transaktionen schneller durchzuführen, bieten wir
DNA versus RNA. RNA Instabilität
 DNA versus RNA DNA stellt den eigentlichen Speicher genetischer Information dar, während RNA als Informationsüberträger und katalytisch in der Proteinbiosynthese agiert. Warum dient DNA und nicht RNA als
DNA versus RNA DNA stellt den eigentlichen Speicher genetischer Information dar, während RNA als Informationsüberträger und katalytisch in der Proteinbiosynthese agiert. Warum dient DNA und nicht RNA als
Bereich METIS (Texte im Internet) Zählmarkenrecherche
 Bereich METIS (Texte im Internet) Zählmarkenrecherche Über die Zählmarkenrecherche kann man nach der Eingabe des Privaten Identifikationscodes einer bestimmten Zählmarke, 1. Informationen zu dieser Zählmarke
Bereich METIS (Texte im Internet) Zählmarkenrecherche Über die Zählmarkenrecherche kann man nach der Eingabe des Privaten Identifikationscodes einer bestimmten Zählmarke, 1. Informationen zu dieser Zählmarke
4. BEZIEHUNGEN ZWISCHEN TABELLEN
 4. BEZIEHUNGEN ZWISCHEN TABELLEN Zwischen Tabellen können in MS Access Beziehungen bestehen. Durch das Verwenden von Tabellen, die zueinander in Beziehung stehen, können Sie Folgendes erreichen: Die Größe
4. BEZIEHUNGEN ZWISCHEN TABELLEN Zwischen Tabellen können in MS Access Beziehungen bestehen. Durch das Verwenden von Tabellen, die zueinander in Beziehung stehen, können Sie Folgendes erreichen: Die Größe
Welche Unterschiede gibt es zwischen einem CAPAund einem Audiometrie- Test?
 Welche Unterschiede gibt es zwischen einem CAPAund einem Audiometrie- Test? Auch wenn die Messungsmethoden ähnlich sind, ist das Ziel beider Systeme jedoch ein anderes. Gwenolé NEXER g.nexer@hearin gp
Welche Unterschiede gibt es zwischen einem CAPAund einem Audiometrie- Test? Auch wenn die Messungsmethoden ähnlich sind, ist das Ziel beider Systeme jedoch ein anderes. Gwenolé NEXER g.nexer@hearin gp
= i (V) = d 2. v = d! p! n da v 1 = v 2 gilt auch d 1 ÿ p ÿ n 1 = d 2 ÿ p ÿ n 2 (III) p kürzen (Division durch p) d 1 ÿ n 1 = d 2 ÿ n 2 (IV) oder
 v = d! p! n da v 1 = v 2 (I) (II) gilt auch d 1 ÿ p ÿ n 1 = d 2 ÿ p ÿ n 2 (III) p kürzen (Division durch p) d 1 ÿ n 1 = d 2 ÿ n 2 (IV) oder i = Übersetzungsverhältnis n 1 n 2 = d 2 d 1 = i (V) Beispiel
v = d! p! n da v 1 = v 2 (I) (II) gilt auch d 1 ÿ p ÿ n 1 = d 2 ÿ p ÿ n 2 (III) p kürzen (Division durch p) d 1 ÿ n 1 = d 2 ÿ n 2 (IV) oder i = Übersetzungsverhältnis n 1 n 2 = d 2 d 1 = i (V) Beispiel
DNA: Aufbau, Struktur und Replikation
 DNA: Aufbau, Struktur und Replikation Biochemie Die DNA als Träger der Erbinformation Im Genom sind sämtliche Informationen in Form von DNA gespeichert. Die Information des Genoms ist statisch, d. h. in
DNA: Aufbau, Struktur und Replikation Biochemie Die DNA als Träger der Erbinformation Im Genom sind sämtliche Informationen in Form von DNA gespeichert. Die Information des Genoms ist statisch, d. h. in
trivum Multiroom System Konfigurations- Anleitung Erstellen eines RS232 Protokolls am Bespiel eines Marantz SR7005
 trivum Multiroom System Konfigurations- Anleitung Erstellen eines RS232 Protokolls am Bespiel eines Marantz SR7005 2 Inhalt 1. Anleitung zum Einbinden eines über RS232 zu steuernden Devices...3 1.2 Konfiguration
trivum Multiroom System Konfigurations- Anleitung Erstellen eines RS232 Protokolls am Bespiel eines Marantz SR7005 2 Inhalt 1. Anleitung zum Einbinden eines über RS232 zu steuernden Devices...3 1.2 Konfiguration
GeoPilot (Android) die App
 GeoPilot (Android) die App Mit der neuen Rademacher GeoPilot App machen Sie Ihr Android Smartphone zum Sensor und steuern beliebige Szenen über den HomePilot. Die App beinhaltet zwei Funktionen, zum einen
GeoPilot (Android) die App Mit der neuen Rademacher GeoPilot App machen Sie Ihr Android Smartphone zum Sensor und steuern beliebige Szenen über den HomePilot. Die App beinhaltet zwei Funktionen, zum einen
Schrittweise Anleitung zur Erstellung einer Angebotseite 1. In Ihrem Dashboard klicken Sie auf Neu anlegen, um eine neue Seite zu erstellen.
 Schrittweise Anleitung zur Erstellung einer Angebotseite 1. In Ihrem Dashboard klicken Sie auf Neu anlegen, um eine neue Seite zu erstellen. Klicken Sie auf Neu anlegen, um Ihre neue Angebotseite zu erstellen..
Schrittweise Anleitung zur Erstellung einer Angebotseite 1. In Ihrem Dashboard klicken Sie auf Neu anlegen, um eine neue Seite zu erstellen. Klicken Sie auf Neu anlegen, um Ihre neue Angebotseite zu erstellen..
Die Forschung mit embryonalen Stammzellen ist ethisch nicht akzeptabel
 Die Forschung mit embryonalen Stammzellen ist ethisch nicht akzeptabel Das Schweizer Volk soll sich selber äussern bezüglich der Gesetzgebung zur embryonalen Stammzellenforschung. Die ethische Bedeutung
Die Forschung mit embryonalen Stammzellen ist ethisch nicht akzeptabel Das Schweizer Volk soll sich selber äussern bezüglich der Gesetzgebung zur embryonalen Stammzellenforschung. Die ethische Bedeutung
Die Liebe und der Verlust
 Die Liebe und der Verlust Jeder Mensch hat in seinem Leben Zuneigung, Affinität oder Liebe zu einem anderen Menschen gehabt in einer Partnerschaft oder sogar einer Ehe. Gemeint ist eine Zeit, in der man
Die Liebe und der Verlust Jeder Mensch hat in seinem Leben Zuneigung, Affinität oder Liebe zu einem anderen Menschen gehabt in einer Partnerschaft oder sogar einer Ehe. Gemeint ist eine Zeit, in der man
Musterlösung - Übung 5 Vorlesung Bio-Engineering Sommersemester 2008
 Aufgabe 1: Prinzipieller Ablauf der Proteinbiosynthese a) Erklären Sie folgende Begriffe möglichst in Ihren eigenen Worten (1 kurzer Satz): Gen Nukleotid RNA-Polymerase Promotor Codon Anti-Codon Stop-Codon
Aufgabe 1: Prinzipieller Ablauf der Proteinbiosynthese a) Erklären Sie folgende Begriffe möglichst in Ihren eigenen Worten (1 kurzer Satz): Gen Nukleotid RNA-Polymerase Promotor Codon Anti-Codon Stop-Codon
