Biochemische Untersuchungen zur Interaktion der aeroben sn-glycerin-3-phosphat-dehydrogenase aus Escherichia coli mit der cytoplasmatischen Membran
|
|
|
- Hede Frei
- vor 5 Jahren
- Abrufe
Transkript
1 Biochemische Untersuchungen zur Interaktion der aeroben sn-glycerin-3-phosphat-dehydrogenase aus Escherichia coli mit der cytoplasmatischen Membran Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften Universität Konstanz Fachbereich Biologie Antje-Christine Walz Konstanz, 2002
2 Dissertation der Universität Konstanz Referenten: Prof. Dr. Rupert Mutzel Prof. Dr. Winfried Boos Datum der mündlichen Prüfung: 12. Februar 2002 Prüfer: Prof. Dr. Hans-Jürgen Apell HD Dr. Michael Dahl Prof. Dr. Gottfried Seebaß
3 Eine Tasse faßt nur eine Tasse Wasser, und wenn ich Liter in sie gösse. Ludwig Wittgenstein
4 Teile dieser Arbeit wurden veröffentlicht: Walz, A.-C., R.A. Demel, B. de Kruijff, und R. Mutzel, Aerobic sn-glycerol-3-phosphate dehydrogenase from Escherichia coli binds to the cytoplasmic membrane through an amphipathic α-helix. Biochem. J. (im Druck)
5 Zusammenfassung Diese Arbeit beschreibt den molekularen Mechanismus der Bindung der löslichen sn- Glycerin-3-Phosphat-Dehydrogenase (GlpD) aus Escherichia coli an die cytoplasmatische Membran. Zunächst wurde der spezifische Bindungsmechanismus zwischen GlpD und eukaryontischem Ca 2+ /Calmodulin (Ca 2+ /CaM) durch Kompetitionsstudien analysiert. Von den eingesetzten chemisch unterschiedlichen Calmodulin-Antagonisten wurde die GlpD-Calmodulin-Bindung lediglich durch anionische Liposomen und durch Melittin gehemmt, einem kleinen Peptid, dessen Sekundärstruktur durch eine basisch amphiphile α-helix charakterisiert ist. In einem weiteren Schritt wurde die Ca 2+ /CaM-Bindedomäne von GlpD auf den Bereich der Aminosäuren eingegrenzt. Sowohl das GlpD-Fragment CBRIII, das C-terminal um 190 Aminosäuren verkürzt ist, als auch die GlpD-Mutante GlpD (351-72), die eine Deletion im Bereich der Aminosäuren aufweist, konnten nicht mehr an Ca 2+ /CaM binden. Sekundärstrukturvorhersagen ermittelten eine basisch amphiphile α-helix für diesen Bereich. Durch gerichtete Mutagenese wurden Mutanten (R359E, K360E, AH362/4DE) erzeugt, die im α-helicalen Bereich negativ geladen waren, während Wildtyp-GlpD in diesem Bereich positiv geladen war. Die beiden Mutanten R359E und K360E präsentieren einen Überschuss an negativer Ladung im hydrophilen Teil der Helix und konnten nicht mehr an Ca 2+ /CaM binden. Dagegen konnte die Mutante AH362/4DE, deren hydrophile Seite der Helix neutral war, noch an Ca 2+ /CaM binden. Die Vermutung, dass dieselben molekulare Mechanismen bei der GlpD-Calmodulin- und der GlpD-Lipid-Bindung vorherrschen, konnte durch Experimente mit Lipid-Monolayern bestätigt werden. GlpD induzierte eine Druckänderung in Monolayern, die aus Gesamt- Phospholipidextrakt von E. coli (TLE) bestanden, indem es in den hydrophoben Bereich der Lipidschicht eindrang. Die Membraninsertion von GlpD war abhängig von der Lipidzusammensetzung. In anionischen Monolayern war die penetrative Kraft von GlpD deutlich höher als bei zwitterionischen Monolayern. In Experimenten mit gemischten Phospholipiden wurde eine positive Korrelation zwischen dem Gehalt an negativ geladenen Phospholipiden und der induzierten Druckänderung beobachtet. Die Punktmutanten, die nicht mehr an Ca 2+ /CaM banden, konnten bei physiologisch relevanten Drücken nicht mehr in I
6 TLE-Monolayer inserieren. Die AH362/4DE-Mutante war dagegen im Vergleich zum Wildtyp in ihrer Insertionskraft nur geringfügig eingeschränkt. Versuche mit anionischen und zwitterionischen Phospholipiden zeigten, dass die penetrative Kraft der Punktmutanten nur in anionischen Monolayern nicht jedoch in zwitterionischen Lipidschichten reduziert war. Experimente mit Lipid-Bilayern bestätigten, dass eine direkte GlpD-Lipid-Bindung besteht. In Bindungsstudien mit GlpD und TLE-Vesikeln wurde gezeigt, dass GlpD an TLE-Liposomen bindet. Die Lipidspezifität dieser Bindung unterschied sich deutlich von der des Monolayer- Systems. Die Affinität von GlpD war gegenüber zwitterionischen Phospholipiden höher als gegenüber anionischen Phospholipiden. Die Tatsache, dass dieser Effekt auch mit den Punktmutanten erzielt wurde, zeigt, dass im Lipid-Bilayer-System ein weiterer Mechanismus der GlpD-Lipid-Interaktion beobachtet wurde. Die Enzymaktivität von GlpD war abhängig von der Phospholipid-Zusammensetzung. Es konnte ausgeschlossen werden, dass die geringere Stimulation durch PG- gegenüber PE- Vesikeln ausschließlich auf Ladungs- bzw. auf ph-effekten beruht. Die mittels Liposomenbindungstests beobachtete spezifische Bindung von GlpD an PE wirkt sich offensichtlich begünstigend auf die PE-vermittelte Aktivierung aus, möglicherweise indem dies GlpD in seiner enzymatisch aktiven Konformation stabilisiert. II
7 Abkürzungsverzeichnis ATP Adenosin-3 Monophosphat nuclear magnetic resonance NMR spectroscopy CaM Calmodulin NTA Nitriloessigsäure CDP Cytosindiphosphat OD optische Dichte CL Cardiolipin PAGE Polyacrylamid Gelelektrophorese DMSO Dimethylsulfoxid PC Phosphatidylcholin dntp Desoxyribonukleotide PE Phosphatidylethanolamin DHAP Dihydroxyaceton-Phosphat PG Phosphatidylglycerin DOPC Dioleoylphosphatidylcholin PGP Phosphatidylglycerinphosphat DOPE Dioleoylphosphatidylethanolamin PS Phosphatidylserin DOPG Dioleoylphosphatidylglycerin R24571 Calmidazolium DPG Dipalmitoylglycerin PCR Polymerase Kettenreaktion DTT Dithiothreitol PMS Phenazin Methosulfat ECL enhanced chemiluminescence RGS regulator of G protein signaling EGTA Ethylenglycol-O-O -bis(2- RT Raumtemperatur aminoethyl)-n,n,n,n -tetraacetat FAD Flavin-Adenin-Dinukleotid SDS Natriumdodecylsulfat G3P Glycerin-3-Phosphat SRP Signalerkennungspartikel GlpD Glycerin-3-Phosphat-Dehydrogenase SUVs small unilamellar vesicles GUT glycerol utilization TBS Tris buffered saline IPTG Isopropyl-β-D-thiogalactopyranosid TCA Trichloressigsäure kb Kilobasen TFP Trifluoperazin kda Kilodalton T m Schmelztemperatur LUVETs large unilamellar vesicles prepared TLE Gesamtphospholipid Extrakt by extrusion technique aus Escherichia coli MARCKS myristoylierte Alanin-reiche Tris Tris-(hydroxymethyl)- Proteinkinase C-Substrate aminomethan MTT 3-(4,5-dimethylthiazolyl-2)-2,5- diphenyltetrazolium Bromid W7 N-(6-Aminohexyl)-5-chlor-1- naphthalinsulfonamidhydrochlorid NADH reduziertes Nicotinamid-Adenin- Dinucleotidphosphat III
8 Inhaltsverzeichnis Inhaltsverzeichnis Einleitung... 1 Zentrale Rolle von G3P... 1 Membranaufbau von E. coli... 1 Phospholipidsynthese... 3 Charakteristika der Phospholipide... 5 Nicht-Bilayer Lipids... 5 Anionische Phospholipide... 6 Glycerintransport und -metabolismus... 7 Das glp-regulon... 9 Regulation des Glycerinmetabolismus Die sn-glycerin-3-phosphat-dehydrogenasen Die anaerobe Glycerin-3-Phosphat-Dehydrogenase Die aerobe Glycerin-3-Phosphat-Dehydrogenase Klassifizierung peripherer Membranproteine Eukaryontisches Calmodulin und bakterielle Calmodulin-Bindeproteine Ziel dieser Arbeit Material und Methoden Verwendete Materialien und deren Bezugsquellen Chemikalien und sonstige Materialien Oligonukleotidprimer Verwendete Bakterienstämme IV
9 Inhaltsverzeichnis Verwendete Plasmide Verwendete Kulturmedien Mikrobiologische Methoden Aufbewahrung von Bakterienstämmen Wachstumsbedingungen Bestimmung der Zelldichte in Flüssigkulturen Molekularbiologische Methoden Standardmethoden Polymerase-Kettenreaktion Gerichtete Mutagenese mittels Fusions-PCR Konstruktion der Vektoren TSS-Transformation Biochemische Methoden SDS-Polyacrylamid-Gelelektrophorese Coomassie Blue-Färbung von SDS-PAA-Gelen Western Blot und Immunfärbung von Proteinen Protein-Präzipitation mit 10% Trichloressigsäure Proteinbestimmung Enzymaktivitätstest Reinigung von rekombinantem GlpD über Chitinaffinitätschromatographie Reinigung der Wildtyp-, CBRIII-, GlpDD( ), R349E-, K360E, AH362/4DE-Fusionsproteine über Ni2+/NTA-Affinitätschromatographie Bindungsstudien von Wildtyp- und mutagenisiertem GlpD an Calmodulin-Sepharose 4B Präparation von SUVs (Small Unilamellar Vesicles) V
10 Inhaltsverzeichnis Präparation LUVETs (Large Unilamellar Vesicles prepared by Extrusion Technique) Lipid Extraktion nach Bligh and Dyer Phosphor Bestimmung nach Fiske Subbarow Isolierung und Reinigung des gesamten Phospholid- Extrakts aus E. coli (TLE) Monolayer-Experimente Vesikel-Bindungsstudien Ergebnisse Spezifität der Bindung von GlpD an CaM Klonierung, Überexpression und Reinigung von GlpD Calcium-abhängige CaM-Bindung Inhibitionsstudien zur Bindung von GlpD an CaM Bindung von GlpD an saure und hydrophobe Oberflächen Lokalisation der CaM-Bindedomänen Basisch amphiphile Helix: CaM-Bindemotiv von GlpD? Charakterisierung der GlpD-Lipid-Bindung Direkte Protein-Lipid-Interaktion Lipidspezifität der Insertion von GlpD Insertion von GlpD in gemischte Phospholipid-Monolayer Insertion von GlpD und Punktmutanten in TLE-Monolayer Insertion von wt-glpd und Punktmutanten in PG- bzw. PE-Monolayer GlpD-Bindung an TLE-Bilayer Liposomen-Bindungsspezifität von GlpD Einfluss von Liposomen auf die GlpD-Aktivität VI
11 Inhaltsverzeichnis Stärkste Stimulation mit neutralen SUVs Dosis-Abhängigkeit der Liposomen-stimulierten GlpD Aktivität Enzymaktivität nach Vorinkubation mit LUVETs Liposomen-stimulierte GlpD-Aktivität bei hohen Salzkonzentrationen ph-abhängigkeit der Liposomen-stimulierten Enzymaktivität Diskussion Direkte GlpD-Lipid-Interaktion Membraninsertion von GlpD Insertion erfolgt durch basisch amphiphile α-helix Spezifische Bindung an PE-enthaltende Membranen Lipid-stimulierte Enzymaktivität Lokalisierung an Mikrodomänen? α-helix: generelles Membranbindemotiv bei Glycerin-3-Phosphat-Dehydrogenasen? Identität von CaM- und Membranbindedomäne Ausblick: Identifikation von Membranbindemotiven durch CaM Bindetests Literaturverzeichnis VII
12 Einleitung Einleitung Escherichia coli gehört der Familie der Enterobacteriaceae an, einer Gruppe Gram-negativer Bakterien, deren natürliches Habitat der Darm warmblütiger Organismen ist (Sanderson, 1976). In seiner natürlichen Umgebung sind Fettsäuren, Triacylglyceride und Phospholipide häufig vorkommende Substanzen. Zentraler Bestandteil von Triacylglyceriden und Phospholipiden ist Glycerin-3-Phosphat (G3P), das von E. coli entweder für den Membranaufbau oder zur Energiegewinnung genutzt werden kann. Zentrale Rolle von G3P Im Metabolismus von E. coli spielt das wasserlösliche G3P eine zentrale Rolle. Einerseits wird es für die Lipidbiosynthese als Grundbaustein von Phospholipiden und als Vorläufermolekül der polaren Kopfgruppe des Phosphatidylglycerins benötigt (Raetz, 1986). Andererseits kann es als Energiequelle in Form von Glycerin, G3P und Glycerophosphatdiester genutzt werden (Lin, 1976). Ist kein externes G3P vorhanden, so wird es von E. coli durch eine NADH-abhängige Reduktion von Glycerinaldehyd-3-Phosphat synthetisiert (Yun et al., 2000). Die Tatsache, dass G3P als Substrat zweier unterschiedlicher Reaktionswege genutzt werden kann, ermöglicht den Bakterien, flexibel und schnell auf die jeweilige Umweltsituation zu reagieren und stellt sie gleichzeitig vor regulatorische Herausforderungen. Wird die Expression der Strukturgene des Glycerin-Metabolismus nicht mehr reguliert, z.b. durch Mutationen im Glp-Repressor, der die Expression dieser Strukturgene negativ kontrolliert, so führt dies zu einem verstärktem G3P-Abbau, so dass nicht mehr genügend G3P für den Aufbau von Phospholipiden vorhanden ist, was letztendlich die Lebensfähigkeit der Bakterien einschränkt (Holtman et al., 2001b). In E. coli müssen Glycerintransport und metabolismus folglich auf verschiedenen Ebenen reguliert werden. Membranaufbau von E. coli Bakterielle Membranen stellen eine Permeationsbarriere für viele Substrate dar. Die Zellhülle Gram-negativer Bakterien besteht aus drei Kompartimenten: der cytoplasmatischen Membran, dem Periplasma und der Außenmembran (Abb. 1). Alle von Gram-negativen Bakterien aufgenommenen Substanzen müssen daher zwei Membranen passieren. Die äußere 1
13 Einleitung Membran ist durch einen asymetrischen Aufbau gekennzeichnet, der ungewöhnlich für biologische Membranen ist. Während die äußere Schicht aus Lipopolysacchariden (LPS) besteht, sind in der zum Periplasma gerichteten Schicht Glycerophospholipide enthalten (Raetz und Dowhan, 1990). Die äußere Membran stellt eine Permeationsbarriere dar und bietet der Zelle Schutz vor antimikrobiellen Agenzien. Sie enthält nur wenige Porine, die die äußere Membran gegenüber hydrophilen Substanzen, die kleiner als 600 Da sind, durchlässig machen (Nikaido und Vaara, 1985). Eine Peptidoglykanschicht trennt die innere von der äußeren Membran und bietet der Zelle mechanischen Schutz (Cooper, 1991). Die Aufnahme von Substraten durch die äußere Membran erfolgt durch passive Diffusion, weswegen die Nährstoffkonzentration im Periplasma niedriger als im Extrazellulärraum gehalten werden muss. Da Mikroorganismen meist unter limitiertem Substratangebot leben, kann dies nur erfüllt werden, wenn die Substanzen durch aktiven und spezifischen Transport über die cytoplasmatische Membran schnell entfernt werden. Die cytoplasmatische Membran, bestehend aus einem Glycerophospholipidbilayer mit darin eingelagerten Proteinen, entspricht dem typischen Aufbau von Plasmamembranen. Abbildung 1: Zellhülle von E. coli (Raetz und Dowhan, 1990) 2
14 Einleitung Die integralen Membranproteine übernehmen verschiedene Funktionen, wie Energieerzeugung, Transport und Austausch von Nährstoffen und Metaboliten und sind die Vebindungsglieder zwischen Umweltbedingungen und physiologischer Antwort. Die wichtigsten Glycerophospholipide von E. coli sind Phosphatidylethanolamin (PE), Phosphatidylglycerin (PG) und Cardiolipin (CL) (Abb2B). Circa 75% des gesamten cytoplasmatischen Phospholipidgehalts besteht aus PE (Raetz, 1978). Die relativen Anteile von CL und PG hängen vom Wachstumsstadium der Zellen ab. In der logarithmischen Wachstumsphase wird vermehrt PG gebildet, in der stationären Phase wird ein erhöhter Anteil von CL beobachtet (Ames, 1968; Cronan, 1968; Raetz, 1986). Phospholipidsynthese Die Biosynthese von PE, PG und CL läuft in mehreren Stufen ab (Abbildung 2A). Vorläufer von Fettsäuren und polaren Kopfgruppen werden im Cytoplasma synthetisiert (Raetz, 1986). Die Endprodukte der Fettsäuresynthese werden zu den an der cytoplasmatischen Membran lokalisierten G3P-Molekülen transferiert. Dort werden sie zu den verschiedenen Phospholipidkopfgruppen synthetisiert. Die Synthese der Phospholipide beginnt mit der Acetylierung zu 1-Acyl-Glycerin-3-Phosphat, katalysiert von der G3P-Acyltransferase PlsB (Larson et al., 1984; Raetz, 1986). 1-Acyl-G3P wird durch eine zweite Acyltransferase (Rajasekharan et al., 1988) zu Diacylglycerin-3-Phosphat, auch Phosphatidat genannt, umgewandelt. Aus Phosphatidat wird Cytosidindiphosphat (CDP)-Diacylglycerin, das gemeinsame Vorläufermolekül von PE, PG und CL. Die von pssa kodierte Phosphatidylserin-Synthase und die von psd kodierte Phosphatidylserin (PS)-Decarboxylase wandeln CDP-Diacylglycerin zu PE um. Diese Reaktion läuft so schnell ab, dass in E. coli kaum PS nachgewiesen werden kann. Die Synthese von PG erfolgt aus CDP-Diacylglycerin über das Zwischenprodukt Phosphatidylglycerinphosphat (PGP). Die zweistufige Reaktion wird von der pgs-kodierten PG- Phosphatsynthase und der von pgp kodierten PGP-Phosphatase katalysiert. Aus der Kondensation von zwei PG-Molekülen entsteht durch die von dem cls-gen kodierte CL-Synthase ein CL-Molekül. 3
15 Einleitung A sn -glycero-3-phosphate fatty acyl-acp ACP phosphatic acid CTP PP i CDP-diacylglycerol L-serine CMP phosphatidylserine (PS) psd CO 2 phosphatidylethanolamine (PE) sn-glycerol-3-p CMP phosphatidylglycerol-p pgp P i phosphatidylglycerol (PG) PG cls glycerol B O cardiolipin (CL) R C O CH 2 Phosphatidylethanolamine R' C O CH O O H 2 C O P O CH 2 CH 2 NH 3 + O _ O R C O CH 2 Phosphatidylglycerol R' C O CH O H O H 2 C O P O CH 2 C CH 2 OH O _ OH O O R C O CH 2 Cardiolipin H 2 C O C R R' C O CH O H O HC O C R' O H 2 C O P O CH 2 C CH 2 O P O CH 2 O O _ OH O _ Abbildung 2: A: Syntheseweg der Glycerophospholipide nach Raetz (1986) und B: chemische Struktur von PE, PG und Cl. R und R bezeichnen Fettsäurereste. 4
16 Einleitung Charakteristika der Phospholipide Glycerophospholipide bestehen aus dem Rückgrat-bildenden Glycerin, zwei Fettsäureketten, die den hydrophoben Teil darstellen und einem phosphorylierten Alkohol als Kopfgruppe. Nicht-Bilayer Lipids Aufgrund seiner kleinen Kopfgruppe ist PE (Abb. 2B) im Gegensatz zu PG und CL nicht in der Lage, sich spontan zu einem Lipidbilayer zusammenzuschließen. Obwohl biologische Membranen Bilayer sind, enthält die Membran von E. coli einen hohen Anteil an Nicht- Bilayer-Lipiden. Diese Lipide bilden eine invertierte hexagonale Phase (Abb. 3B), die jedoch in Biomembranen nicht vorkommt. In Biomembranen induzieren Nicht-Bilayer-Lipide eine kubische Phase (Abb. 3C). Nicht-Bilayer-Lipide sind in E. coli essentiell, wie die Arbeiten mit dem Stamm AD93 zeigen, der aufgrund einer Mutation im pssa-gen kein PS synthetisiert und folglich auch kein PE mehr bilden kann (Dechavigny et al., 1991a und 1991b). Unter normalen Wachstumsbedingungen ist diese Mutation lethal. AD93-Zellen können nur durch Zusatz divalenter Kationen wie Calcium, Magnesium und Strontium wachsen (Dechavigny et al., 1991a). Durch NMR-Analyse wurde gezeigt, dass unter diesen Bedingungen der Übergang von Bilayer- zu Nicht-Bilayer-Strukturen bei Lipiden des AD93-Stammes in einem ähnlichen Temperaturbereich erfolgt wie bei Lipiden des parentalen Stammes W3899 (Rietveld et al., 1993). Rietveld und Mitarbeiter folgern, dass CL zusammen mit Kationen die für die Membranfunktion essentielle Struktur von PE einnehmen kann. Elektronenmikroskopische Untersuchungen zeigen, dass bei AD93-Zellen Calcium-Ionen auf der periplasmatischen Seite wirken und die Bilayer-Struktur der Membran durch die cytosolische Membranhälfte stabilisiert werden (Rietveld et al., 1997). Die funktionelle Bedeutung von PE besteht folglich darin, dass sich Biomembranen in einem Zustand befinden, der nahe des Bilayer-Nichtbilayer Übergangs ist. Dieser Zustand ist in E. coli von zentraler Bedeutung, da dies eine Neuanordnung der Lipide gemäß ihrer jeweiligen Aufgabe ermöglicht (De Kruijff, 1997b) und ist folglich in E. coli strikt reguliert (Morein et al., 1996; Rietveld et al., 1993). In AD93-Zellen sind der Einbau und die Funktion wichtiger Transportproteine wie beispielsweise die Lac-Permease deutlich geschwächt (Bogdanov et al., 1996) und die Protein-Translokation deutlich reduziert (Rietveld et al., 1995). Abbildung 3E und 3G verdeutlichen, wie PE die Packungsdichte von Lipiden in der Membran erhöht und damit die 5
17 Einleitung Faltung integraler Proteine beeinflusst (Cantor, 1999; Curran et al., 1999; De Kruijff, 1997a). Dies erklärt die Beobachtung, dass eine Membran mit steigender Anzahl an funktionellen Aufgaben einen steigenden Anteil an Nicht-Bilayer-Lipiden aufweist. Zudem stimulieren die kleinen Kopfgruppen von Nicht-Bilayer-Lipiden die partielle Insertion von Proteinen in die Kopfgruppenregion der Membran (Van Den Brink-Van Der Laan et al., 2001; Van Klompenburg et al., 1998). Abbildung 3: Organisation von Nicht-Bilayerlipiden. A: Die Assemblierung des Nicht-Bilayer Lipids PE zu einer konkaver Struktur führt zur Bildung von B: einer hexagonalen Phase oder C: einer kubischen Phase. D: Nicht-Bilayer-Lipide, die in einem planaren Lipid-Bilayer zusamengefasst sind, erzeugen einen höheren lateralen Druck im Inneren der Membran und einen niedrigeren Druck im Bereich der Kopfgruppen. E: Dieses Druckprofil stabilisiert transmembrane Proteine. F: Bilayer-Lipide, wie Phosphatidylcholin haben eine zylindrische Form und führen deshalb zu einer gleichmäßigen Packungsdichte in Bilayern. G: Der laterale Druck in PC-Bilayern ist geringer und führt zu einer relaxierten Orientierung von transmembranen Proteinen (De Kruijff, 1997b). Anionische Phospholipide Bei physiologischem ph (7.4) ist PE neutral, PG und CL sind dagegen negativ geladen. Während die Mutanten noch überlebensfähig sind, die aufgrund eines Defekts im cls-gen zwar noch PG jedoch kein CL mehr produzieren können, sind Mutationen im pgsa-gen lethal (Heacock und Dowhan, 1987). Durch die Konstruktion einer Mutante, deren pgsa-expression durch ein lac-operon gesteuert ist, wurde der minimale Gehalt an PG für die Überlebensfähigkeit mit 3% bestimmt. Kikuchi und Mitarbeiter zeigten, dass die pgsa-mutante 6
18 Einleitung überlebensfähig ist, wenn gleichzeitig die Synthese von Lipoprotein gehemmt ist. Die Autoren folgern, dass PG und CL für elementare Prozesse nicht essentiell sind, da ihre Rolle von Phosphatidsäure übernommen werden kann (Kikuchi et al., 2000). Anionische Lipide spielen bei der Proteintranslokation (De Kruijff, 1994; De Vrije et al., 1988; Kusters et al., 1991; Kusters et al., 1994) und DNA Replikation (Garner et al., 1998) eine zentrale Rolle. So wurde gezeigt, dass das periphere Membranprotein DnaA, das die Initiation der DNA-Replikation katalysiert, anionische Phospholipide mit einer ein kda großen Domäne penetriert (Garner und Crooke, 1996; Garner et al., 1998). Die Helical- Wheel-Projektion dieser 18 Aminosäuren zeigt, dass das Fragment eine amphiphile Helix formieren kann. Die Aktivität von SecA, einer cytoplasmatischen Komponente des Protein- Translokationskomplexes, wird durch saure Phospholipide stimuliert (Lill et al., 1990). Es wurde gezeigt, dass SecA in Phospholipid-Monolayer inseriert (Breukink et al., 1992). FtsY, eine GTPase mit Homologie zur α-untereinheit des eukaryontischen Signalerkennungspartikel(SRP)-Rezeptors, ist ein cytosolisches Protein, das bei der Proteintranslokation das Preprotein an die Membran transloziert (Luirink et al., 1994). FtsY ist ein cytoplasmatisches Protein, das durch anionische Phospholipide stimuliert wird und durch Insertion in anionische Lipide an die Membran bindet (De Leeuw et al., 2000). Eine weitere wichtige Funktion anionischer Phospholipide besteht darin, die korrekte Orientierung und Topologie transmembraner Proteine zu kontrollieren (Van Klompenburg et al., 1997; Van Klompenburg und De Kruijff, 1998). Glycerintransport und -metabolismus E. coli nutzt unterschiedliche G3P-Quellen. Zum einen das durch Phosphorylierung von Glycerin gewonnene und zum anderen das aus dem Phospholipidabbau stammende G3P. Transport und Katabolismus von Glycerin, G3P und Glycerophosphatdiester durch die Genprodukte des glp-regulons sind in Abbildung 4 dargestellt. Das metabolische System dient im wesentlichen dazu, das aus dem Abbau von Phospholipiden und Triacylgylcerin stammenden Glycerin zu verstoffwechseln. Glycerophosphatdiester, die deacetylierten Abbauprodukte der Phospholipide (Lin, 1987), werden im Periplasma von der dort lokalisierten Phosphodiesterase GlpQ zu Alkohol und G3P hydrolysiert (Larson et al., 1983). G3P wird durch die Permease GlpT (Eiglmeier et al., 1987; Larson et al., 1982) im Austausch gegen anorganisches Phosphat ins Zellinnere transportiert (Auer et al., 2001; Elvin et al., 1985). Die für den Transport benötigte Energie wird 7
19 Einleitung durch diesen Antiport bereitgestellt und gleichzeitig wird eine Akkumulation des toxisch wirkenden Phosphats verhindert (Xavier et al., 1995). Wird Glycerin aus dem Nährmedium aufgenommen, so gelangt es durch erleichterte Diffusion ins Zellinnere. Dies wird von dem Glycerinfascilitator GlpF katalysiert (Borgnia und Agre, 2001; Heller et al., 1980), der einen substratspezifischen Kanal mit einer Porengröße von 0.4 nm bildet (Heller et al., 1980). Cytoplasmatisches Glycerin wird sofort von der ATP-abhängigen Glycerinkinase K phosphoryliert, die in ihrer enzymatisch aktiven Form mit dem GlpF-Fascilitator assoziiert vorliegt (Voegele et al., 1993). Extracellular Medium Periplasma Cytoplasma Glycerol Glycerol Facilitator (glpf) Glycerol Feedback Inhibition Enzyme III Gle Kinase +ATP (glpk) Fructose-1,6-P 2 Aerobic dehydrogenase (glpd) G3P ROH P-diesterase (glpq) Permease (glpt) G3P Anaerobic dehydrogenase (glpacb) G3P Oxidoreductase gpsa DHAP Glyceraldehyde-3-P Phosphatidic acid G3P-OR phosphatidylgycerol-3-p PE PG Cardiolipin Abb. 4: Glycerintransport und metabolismus Im Cytosol wird G3P entweder als Ausgangsstoff für die Lipidsynthese oder für die Dissimilation verwendet. Zur Energiegewinnung wird G3P von zwei FAD-abhängigen Dehydrogenasen zu Dihydroxyaceton-Phosphat (DHAP) oxidiert. DHAP kann als Intermediat in die Glykolyse eingehen. Im aeroben Milieu wird diese Oxidation von der aeroben Glycerin-3-Phosphat-Dehydrogenase (GlpD) katalysiert, im anaeroben Milieu dagegen von der anaeroben G3P Dehydrogenase (GlpABC). Die bei dieser Oxidationsreaktion frei werdenden Reduktionsäquivalente werden von den beiden Flavoenzymen auf einen membranassoziierten Cytochromkomplex übertragen, an dessen Ende ein terminaler 8
20 Einleitung Elektronenakzeptor sitzt. Im Fall der aeroben Oxidation ist dies molekularer Sauerstoff bzw. Nitrat, unter anaeroben Bedingungen dagegen Fumarat oder Nitrat (Lin, 1987). Während die Elektronen durch den Cytochromkomplex geschleust werden, wird die frei werdende Energie dazu genutzt, Protonen durch die Membran von der cytoplasmatischen auf die periplasmatische Seite zu pumpen. Mit dem an der Membran erzeugten Protonengradienten ändert sich sowohl das elektrische als auch das chemische Potential, womit die membrangebundene ATPase angetrieben wird und dadurch ATP erzeugen kann. Das glp-regulon Die Expression der Enzyme, die am Glycerintransport und metabolismus beteiligt sind, wird strikt reguliert. Dies wird durch die strukturelle Organisation der glp-gene ermöglicht. Das Glycerophosphatregulon (glp) besteht aus fünf Operons, die an drei verschiedenen Genorten auf dem E. coli-chromosom liegen (Cozzarelli et al., 1968). Alle Gene des glp-systems stehen unter der Kontrolle des GlpR-Repressors (Schweizer und Larson, 1987). Der Genlocus von glpr liegt bei 75 min und wird im Gegenuhrzeigersinn transkribiert (Lin, 1987). Der Repressor bildet im aktiven Zustand ein Tetramer aus identischen Untereinheiten von 30 kda (Larson et al., 1987) und bindet spezifisch an die Kontrollregionen des glp-regulons. Sekundärstrukturanalysen zeigen, dass die spezifische DNA-Bindung des Repressors durch ein Helix-Schleife-Helix-Motiv in der Nähe des N-Terminus möglich ist (Zeng et al., 1996). Diese DNA-Bindung kann durch den Induktor G3P aufgelöst werden (Larson et al., 1987). Durch Versuche mit einer Mutante mit einer inaktiven Glycerinkinase wurde gezeigt, dass für eine Induktion der glp-gene die Phosphorylierung von Glycerin notwendig ist. Folglich ist G3P der alleinige Induktor (Hayashi und Lin, 1965; Lin, 1976). Als Anti-Induktor wurde D- Galaktose-1-Phosphat identifiziert (Sundarajan, 1963). Bei 88 min findet man das Operon glpfkx (Cozzarelli und Lin, 1966; Weissenborn et al., 1992), das für den Glycerinfascilitator GlpF und die Glycerinkinase GlpK und eine Fructose-1,6-Bisphosphatase GlpX (Donahue et al., 2000) kodiert. In dem bei 49 min kartierten Operon sind die Gene glpt (G3P-Transporter) und glpq (periplasmatische Glycerinphosphodiesterase) zusammengefasst, die im Gegenuhrzeigersinn transkribiert werden. Gegenläufig dazu ist das glpabc-operon angeordnet, dessen Gene für die drei Untereinheiten der anaeroben heterotrimeren sn-glycerin-3-phosphat-dehydrogenase kodieren (Cole et al., 1988; Ehrmann et al., 1987; Kistler und Lin, 1971; Miki et al., 1979). Bei 75 min auf dem Chromosom liegt das glpdeg-operon. Während die physiologische 9
21 Einleitung Funktion von glpg noch unbekannt ist (Zeng et al., 1996), wurden GlpE als Schwefeltransferase und GlpD als aerobe Glycerin-3-Phosphat-Dehydrogenase (Cozzarelli et al., 1968) identifiziert. Die vom glpe-gen kodierte saure, cytoplasmatische Rhodanese besteht lediglich aus 108 Aminosäuren mit einem Molekulargewicht von 12 kda und katalysiert als Dimer die Übertragung von Schwefel auf den Schwefelakzeptor Thioredoxin 1. Welche physiologische Rolle GlpE spielt, ist noch unklar (Ray et al., 2000). Das offene Leseraster des glpd-gens besteht aus 501 Codons und die translatierte Sequenz kodiert ein Protein mit einem Molekulargewicht von 57 kda (Austin und Larson, 1991). Das glpg-genprodukt ist ein basisches, cytoplasmatisches oder membranassoziiertes Protein mit einem Molekulargewicht von 28 kda (Zeng et al., 1996). Regulation des Glycerinmetabolismus E. coli verfügt über ein breitgefächertes kataboles Repertoire. Dabei wird immer diejenige Kohlenstoffquelle bevorzugt, die ein schnelleres Wachstum ermöglicht. Produktion und Konzentration von Syntheseprodukten werden sowohl auf metabolischer Ebene mittels Änderungen von Enzymaktivitäten als auch durch eine regulierte Transkription der jeweiligen Enzyme gesteuert. Auf genetischer Ebene wird die Expression der glp-gene negativ durch den Repressor GlpR kontrolliert, der mit unterschiedlichen Affinitäten an die Operatorsequenzen bindet. So zeigt der GlpR-Repressor eine deutlich höhere Affinität zu dem glpd-operator als zu denen der anderen glp-operatoren (Lin, 1987). Bei Glycerinmangel resultiert daraus eine niedrige Basalexpression von GlpD. Mutationen in GlpR, die zu einer konstitutiven Expression von GlpD führen, beeinträchtigen die Phospholipidbiosynthese und bewirken ein Ungleichgewicht in der Membranzusammensetzung (Flower, 2001), das letztendlich die Lebensfähigkeit der Bakterien dramatisch einschränkt. Neben der negativen Regulation durch GlpR unterliegen alle glp-gene der Katabolitrepression durch den camp-cap (camp catabolite gene activator protein)-komplex (Cozzarelli et al., 1968; Fraser und Yamazaki, 1980). Stehen der Zelle neben Glycerin andere, energiereichere Kohlenstoffquellen zur Verfügung wie beispielsweise die dem PTS (Phosphoenolpyruvat-abhängigen Phosphotransferase)-System angehörigen Zucker (Glucose, Mannitol und Mannose), so wird durch Katabolitrepression die Transkription der glp-gene verhindert (Übersicht in Postma et al., 1993). Das Schlüsselenzym im Glycerinmetabolismus ist die Glycerinkinase GlpK, die den 10
22 Einleitung geschwindigkeitsbestimmenden Schritt katalysiert (Zwaig et al., 1970). Sie ist ein wichtiger Regulator zwischen der Genexpression des glp-systems und dem Angebot an Kohlenstoffquellen. Die Inhibition durch das nichtphosphorylierte Enzym EIIA Glc des PTS-Systems, auch Inducer Exclusion bezeichnet, erfolgt durch spezifische Bindung von EIIA Glc an GlpK, womit eine reduzierte Synthese des Induktors G3P bewirkt wird. Dies ist außergewöhnlich, denn in der Regel bindet EIIA Glc an Transportproteine. In diesem Fall bindet es jedoch nicht an den Fascilitator GlpF, sondern an das metabole Enzym GlpK (De Boer et al., 1986; Novotny et al., 1985). Ferner wird die Enzymaktivität von GlpK allosterisch durch Fructose- 1,6-Bisphosphat gehemmt (Holtman et al., 2001a). Während die Kinase bereits bei gering induziertem Zustand durch EIIA Glc reguliert wird, tritt die Feedback Inhibition durch Fructose-1,6-Bisphosphat erst in voll induzierten Zellen ein (Liu et al., 1994). Durch diese strikte Kontrolle der Glycerinkinase wird verhindert, dass G3P akkumuliert, denn eine intrazelluläre Konzentration von 20 mm G3P wirkt sich bakteriostatisch aus (Cozzarelli et al., 1965). Die sn-glycerin-3-phosphat-dehydrogenasen Die anaerobe und die aerobe Glycerin-3-Phosphat-Dehydrogenase befinden sich an zentraler Position zwischen dem Energiestoffwechsel und dem Metabolismus von Membranlipiden (siehe Abb. 4). Beide katalysieren die Oxidation von G3P zu DHAP, übertragen jedoch die bei der Oxidation frei werdenden Reduktionsäquivalente auf unterschiedliche Cytochromoxidasen. Im Fall der aeroben Oxidation überträgt GlpD die Reduktionsäquivalente auf das bei hohem O 2 -Gehalt synthetisierte und vom cyo-operon exprimierte Cytochrom O. Bei der anaeroben Oxidation von G3P wird die Übertragung durch GlpABC auf das bei niedrigem O 2 -Gehalt synthetisierte und vom cyd-operon exprimierte Cytochrom D übermittelt (Kistler und Lin, 1972; Spiro und Guest, 1987). Die Expression der beiden Cytochromoxidasen steht unter der respiratorischen Kontrolle der ArcA und ArcB Produkte, die ein Zweikomponenten- Regulationssystem bilden. Der response-regulator ArcA wird bei geringer Sauerstoffkonzentration von dem Sensor ArcB, einer Histidinkinase, phosphoryliert und reprimiert die GlpD-Expression (Iuchi et al., 1990). Die anaerobe Glycerin-3-Phosphat-Dehydrogenase Die anaerobe Glycerin-3-Phosphat-Dehydrogenase (GlpABC) ist ein heterotrimeres Enzym, das aus drei unterschiedlichen Untereinheiten zusammengesetzt ist (Kuritzkes et al., 11
23 Einleitung 1984; Schryvers und Weiner, 1981; Schryvers und Weiner, 1982). Dieser Komplex besteht aus dem Polypeptid GlpA (62 kda) mit dem nicht-kovalent gebundenen Kofaktor FAD, ferner GlpB (43 kda), das vermutlich FMN-assoziiert vorliegt, und dem Membrananker GlpC (44 kda), dessen Aminosäuresequenz auf zwei Eisen-Schwefel-Zentren schließen lässt (Cole et al., 1988). Die Expression von GlpABC ist das Resultat zweier Einflüsse: einer Fumarat und Nitrat Reduktase (FNR)-abhängigen Regulation (Kuritzkes et al., 1984) und eines weiteren Mechanismus, der über den Redoxstatus der Zelle kontrolliert wird (Iuchi et al., 1990). Die aerobe Glycerin-3-Phosphat-Dehydrogenase Die aerobe Glycerin-3-Phosphat-Dehydrogenase (GlpD) liegt im nativen Zustand als homodimeres Enzym vor (Schryvers et al., 1978). Sie ist aus zwei identischen Untereinheiten von 58 kda zusammengesetzt (Schweizer und Larson, 1987) mit FAD als nichtkovalentem Kofaktor (Robinson und Weiner, 1980). Das periphere Membranprotein GlpD katalysiert die aerobe Oxidation von G3P zu DHAP unter Verwendung von molekularem Sauerstoff bzw. Nitrat als Elektronenakzeptor (Kuritzkes et al., 1984). Die Transkription von GlpD ist strikt reguliert. Es wurden insgesamt vier Repressorbindestellen identifiziert, zwei davon außerhalb und zwei innerhalb des offenen Leseraster von glpd (Yang und Larson, 1996). Die Bindung von GlpR an die internen Repressorbindestellen führt zu einer fünf- bis siebenfach stärkeren Kontrolle des glpd-operons im Vergleich zum glpfkx-operon (Yang und Larson, 1996). Zellen in der stationären Wachstumsphase zeigen eine erhöhte GlpD-Expression (Weichart et al., 1993). Die Autoren diskutieren, dass dies mit der verstärkten Bildung des Induktors G3P als Nebenprodukt der erhöhten Cardiolipinsynthese zusammenhängt. Neben der Regulation auf Transkriptionsebene wird die Aktivität von GlpD über die Lokalisation geregelt. In vivo ist enzymatisch aktives GlpD mit der Membran assoziiert (Weiner, 1974). In vitro ist das lösliche GlpD vollständig aktiv, wenn Phospholipide oder Detergenzien zugesetzt werden (Robinson und Weiner, 1980; Schryvers et al., 1978). Der Bindungsmechanismus von GlpD an die Membran konnte bislang noch nicht aufgeklärt werden. Es kann lediglich ausgeschlossen werden, dass transmembrane Helices das Protein an der Membran verankern. Denn die einzige mögliche transmembrane Helix, die durch Sekundärstrukturvorhersage ermittelt wurde, befindet sich im Bereich der FAD-Bindedomäne (Austin und Larson, 1991). 12
24 Einleitung Möglicherweise ist GlpD in die Lipid A-Biosynthese involviert. Milla und Raetz (Milla und Raetz, 1996) reinigten die Disaccharid-Synthase LpxB, ein Enzym des Lipid A- Syntheseweges, als 8xHis-LpxB-Fusionsprotein, um Interationen mit anderen E. coli Proteinen untersuchen zu können. GlpD konnte selbst bei geringer Expressionsinduktion des 8xHis-LpxB-Fusionsproteins kogereinigt werden. Die Autoren diskutieren, dass eine Interaktion zwischen GlpD und LpxB in vivo möglicherweise ein regulatorisches Bindeglied zwischen dem Katabolismus von Glycerophospholipiden und der Lipid A-Biosynthese darstellt. Außerdem wurde eine spezifische Interaktion zwischen GlpD und eukaryontischem Calmodulin beobachtet (Hellstern, 1997), deren physiologische Bedeutung noch ungeklärt ist. Klassifizierung peripherer Membranproteine Aufgrund der Beobachtung, dass die Reinigung von GlpD aus E. coli den Einsatz von Detergenzien und hohen Salzkonzentrationen erfordert, wurde GlpD als peripheres Membranprotein klassifiziert (Weiner und Heppel, 1972). Die Gemeinsamkeit der so bezeichneten Proteine besteht darin, dass sie sich in ihrem aktiven Zustand an die Membran anheften. Die jeweils zugrunde liegenden molekularen Mechanismen können sehr verschieden sein. Die Bindung an die Membran kann entweder durch Protein-Protein-Wechselwirkungen mit einem integralen Membranprotein dem sogenannten Membrananker, oder durch direkte Protein- Lipid-Interaktion vermittelt werden, die entweder durch Bindung an Phospholipide oder durch Penetration der cytoplasmatischen Schicht des Lipid-Bilayers erfolgt. Die intermolekularen Kräfte, die die Proteine an der Membran verankern, können auf hydrophoben oder elektrostatischen Wechselwirkungen beruhen. Im Fall von hauptsächlich elektrostatischen Kräften wird eine elektropositive Protein-Oberfläche präsentiert, die mit den elektronegativen Lipid-Kopfgruppen interagiert (Lloyd et al., 1999; Stams et al., 1998). Im Fall von hydrophoben und elektrostatischen Wechselwirkungen kann das periphere Membranprotein eine unpolare α-helix ausbilden, die eine Lipidschicht des Bilayers penetriert (Picot et al., 1994). Häufiger wird jedoch die Bildung unpolarer Schleifen beobachtet, die den hydrophoben Bereich der Membran penetrieren (Macedo-Ribeiro et al., 1999). In beiden Fällen interagieren polare Aminosäuren mit den Phospholipid-Kopfgruppen. 13
25 Einleitung Eukaryontisches Calmodulin und bakterielle Calmodulin-Bindeproteine Zwar kommt Calmodulin in Prokaryonten nicht vor, dennoch wurden einige Proteine identifiziert, die in vitro spezifisch mit Calmodulin interagieren. Die Adenylatcyclase aus den drei Spezies Anabaena species (Bianchini et al., 1990), Bordetella pertussis (Gordon et al., 1989; Wolff et al., 1980) und Bacillus anthracis (Gordon et al., 1989; Leppla, 1982) wird von Calcium-gebundenem Calmodulin aktiviert. In E. coli wurden spezifische Interaktionen zwischen Calmodulin und GlpD, GlpC, TraC bzw. SecA beobachtet (Hellstern, 1997). Die physiologische Bedeutung dieser Eigenschaft ist jedoch noch unklar. Calmodulin ist ein eukaryontisches, hochkonserviertes hitzestabiles Protein mit vier Calcium-Bindestellen, das in Eukaryonten an einer Vielzahl Ca 2+ -abhängiger Signaltransduktionswege beteiligt ist (Cohen, 1988; Hinrichsen, 1993). Ob Calciumabhängige Signaltransduktion in Prokaryonten ebenfalls eine Rolle spielen, bleibt spekulativ (Norris et al., 1996). Die vier Calciumbindestellen des 17 kda großen Calmodulin liegen paarweise in Helix-Schleife-Helix-Motiven (auch EF-Hand genannt) an beiden Enden des Moleküls (Babu et al., 1988; Kretsinger et al., 1991). Die Bindung von Calcium-Ionen verursacht eine Konformationsänderung, so dass hydrophobe Bereiche präsentiert werden, die von elektronegativen Aminosäuren flankiert sind (James et al., 1995). Damit kann Calmodulin an die jeweiligen Zielproteine binden. Es wurde gezeigt, dass Calmodulin positiv geladene Peptide bindet, die eine basisch amphipathische α-helix ausbilden. Zudem wird beobachtet, dass die Calmodulin-Bindedomänen der Zielproteine trotz geringer Sequenzhomologie basisch amphipathische α-helices aufweisen (O'Neil und Degrado, 1990). Sequenzvergleiche zeigten, dass einige hydrophobe Reste konserviert sind. Aufgrund dieser Homologien können die Helices in drei Bindungsmotivtypen unterschieden werden (Rhoads und Friedberg, 1997). Ziel dieser Arbeit Während in Eukaryonten physiologische Prozesse in Organellen kompartimentiert ablaufen, ist die prokaryontische Cytoplasma-Membran zentraler Dreh- und Angelpunkt vieler Reaktionen. Neben der Regulation auf Transkriptions- und Translationsebene ist die Translokation von Proteinen zu ihrem Wirkort eine weitere Regulationsebene. 14
26 Einleitung Die aerobe sn-glycerin-3-phosphat-dehydrogenase (GlpD) aus E. coli ist ein lösliches Enzym, das aktiv ist, sobald es an die Membran bindet. Der molekulare Mechanismus dieser Anheftung wurde in der vorliegenden Arbeit untersucht. Zunächst wurde die spezifische Interaktion zwischen GlpD und Calmodulin biochemisch charakterisiert. Da in vivo die identifizierten Calmodulin-Bindeproteine aus E. coli mit der cytoplasmatischen Membran assoziiert sind, können diese Untersuchungen Aufschluss über die Art der GlpD-Membran- Anheftung geben. Die Frage, ob GlpD direkt an die cytoplasmatische Membran bindet, wurde durch Monolayer-Experimente und Vesikelbindungsstudien beantwortet. Mit Hilfe der Monolayer-Technik wurde untersucht, ob GlpD Phospholipid-Monolayer penetrieren kann und welche Phospholipide diese Membraninsertion begünstigen. Mit Vesikelbindungsstudien wurde die Bindung zwischen GlpD und den verschiedenen Lipidtypen quantifiziert. Um den Einfluss von Lipiden auf die biologische Aktivität charakterisieren zu können, wurde die Enzymaktivität von GlpD in Anwesenheit von Liposomen gemessen. 15
27 Material und Methoden Material und Methoden Verwendete Materialien und deren Bezugsquellen Chemikalien und sonstige Materialien Agar Agarose Alkalische Phosphatase Ammoniummolybdat Bactotrypton CaM-Sepharose 4B Calmidazolium (R24571) Cardiolipin aus Rinderherz Chitin Beads Chlorpromazin Compound 48/80 Coomassie Brilliant Blue Coomassie Protein Assay Reagent Dialysemembran (Spectra/Por 6, MWCO 3500 Da) D,L-sn-Glycerin-3-Phosphat Dithiothreitol 1,2-Dioleoyl-sn-Glycerol-3-Phosphatidylcholin 1,2-Dioleoyl-sn-Glycerol-3-Phosphatidylethanolamin 1,2-Dioleoyl-sn-Glycerol-3-Phosphatidylglycerin DMSO EGTA Expand TM -Polymerase FAD Fendilin IPTG Luminol 2-Mercaptoethanol Magermilchpulver (Frema Reform) Melittin (Bienengift) Molekulargewichtsstandard, LMW Molecular-Weight-Marker (SDS-PAGE) MTT Naphthalinsulfonamid (W7) Ni 2+ -NTA Agarose Nitrozellulosemembran (BA 85, 0.45 µm) Nucleoside Nonidet P-40 NZ-Amine A (Caseinhydrolysat) PMS p-coumarsäure Restriktionsenzyme inkl. Puffer Röntgenfilme Fuji-RX Rotiblock SAWADY Pwo-DNA-Polymerase Sepharose 4B Natriumdisulfit Natriumsulfit Trifluoperazin Tween20 GibcoBRL, Eggenstein GibcoBRL Eggenstein Boehringer Mannheim Merck, Darmstadt GibcoBRL Eggenstein Pharmacia, Freiburg Sigma, Deisendorf Avanti Polar Lipids, Alabaster (USA) New England Biolabs Sigma, Deisendorf Sigma, Deisendorf Serva, Heidelberg Pierce, Rockford (USA) Spectrum Medical Industries (L.A., USA) Sigma, Deisendorf Serva, Heidelberg Avanti Polar Lipids, Alabaster (USA) Avanti Polar Lipids, Alabaster (USA) Avanti Polar Lipids, Alabaster (USA) Sigma Chemical Co, St. Louis (USA) Fluka, Neu-Ulm Boehringer Mannheim Sigma, Deisendorf Sigma, Deisendorf BTS (BioTech Trade & Service GmbH) Fluka, Neu-Ulm Fluka, Neu-Ulm Fink GmbH, Herrenberg Merck, Darmstadt Pharmacia, Freiburg Sigma, Deisendorf Sigma, Deisendorf Fluka, Neu-Ulm Qiagen GmbH, Hilden Schleicher & Schuell, Dassel Sigma Chemical Co, St. Louis (USA) ICN, Eschwege Interorgana, Köln Sigma, Deisendorf Fluka, Neu-Ulm Boehringer Mannheim, Gibco, BRL, New England Biolabs, Pharmacia, MBI, USB Allmedt, Essen Roth, Karlsruhe peqlab, Erlangen Pharmacia, Freiburg Merck, Darmstadt Merck, Darmstadt Sigma, Deisendorf Sigma, Deisendorf 16
28 Material und Methoden Oligonukleotidprimer Alle aufgelisteten Oligonukleotide wurden von der Firma MWG-Biotech (Ebersberg) synthetisiert. pqe30-glpd.fwd pqe30-glpd.rev PTYB11-glpD.fwd pqe30-glpd.rev pqe30-cbriii.fwd pqe30-cbriii.rev pqe30-glpd ( ).fwd pqe30-glpd ( ).rev pqe30-glpd-r359e.fwd pqe30-glpd-r359e.rev pqe30-glpd-k360e.fwd pqe30-glpd-k360e.rev pqe30-glpd-ah362/4de.fwd pqe30-glpd-ah362/4de.rev 5 -cgcggatccgaaaccaaagatctgattgtg-3 5 -gcgcctaggctttggtttctagactaacac-3 5 -ggtggttgctctaccatggaaaccaaagatctgattgtg-3 5 -ggtggtctgcagtcacgacgccagcgataacc-3 5 -cgcggatccgaaaccaaagatctgattgtg-3 5 -gggttcgaacccgccttaactgctttttaaagtgc-3 5 -cggggtaccccgtattatcagggtattggcccggcatgg-3 5 -cggggtaccccggccgaataccgacagcagcggtgctttgcc-3 5 -cggtaagctgaccacctacgaaaaactggcggaacatgcg-3 5 -cgcatgttccgccagtttttcgtaggtggtcagcttacc-3 5 -ctgaccacctaccgagaactggcggaacatgcg-3 5 -cgcatgttccgccagttctcggtaggtggtcag-3 5 -cctaccgaaaactggacgaagatgcgctggaaaaactaacgc-3, 5 -gcgtaagtttttccagcgcatcttcctccagttttcggtagg-3 Verwendete Bakterienstämme Stammname relevanter Genotyp Referenz/Quelle MC4100 F - arad139 (argf-lac)u169 rpsl 150 (Str r ) rela1 flb5301 deoc1 (Casadaban, 1976) ptsf25 rbsr XL1-Blue F ::Tn10 proa + B + lacl q (lacz)m15/reca1 enda1 gyra96 (Nal r ) (Bullock et al., 1987) - thi hsdr17(r k m k+ ) supe44 rela1 lac M15[pREP4] F - lac - ara - gal - - mtl - reca + uvr + Nal S Str S rif S (Villarejo und Zabin, 1974) W3899 F + nadb7 supe (Dechavigny et al., 1991a) ER2566 F - λ - fhua2 [lon] ompt lacz::t7 gene1 gal sula11 (mcrcmrr)114::is10 New England Biolabs R (mcr-73::minitn10-tets) R(zgb-210::Tn10) (TetS) enda1 [dcm] 17
29 Material und Methoden Verwendete Plasmide Plasmid Beschreibung/Genotyp/ Referenz/Quelle pqe 30 pds56/rbsii Amp r Stüber et al.,1990 ptyb11 N-terminaler Intein-Fusionsvektor New England Biolabs paw4 pqe30-glpd diese Arbeit paw7 ptyb11-glpd diese Arbeit PAW9 pqe30-glpd ( ) diese Arbeit paw13 pqe30-cbriii diese Arbeit paw16 pqe30-glpd-k359e diese Arbeit paw17 pqe30-glpd-k360e diese Arbeit paw18 pqe30-glpd-ah362/4de diese Arbeit Verwendete Kulturmedien Die Medien wurden nach Miller (Miller, 1972) hergestellt. Sie wurden 20 Minuten bei 120 C und einem Druck von 1 bar autoklaviert, auf C abgekühlt und anschließend je nach Bedarf mit hitzelabilen Medienzusätzen versetzt. LB-Medium NZA-Medium SOC-Medium 2xTSS-Medium 1% Bactotrypton (w/v), 0.5% Hefeextrakt (w/v), 0.5% NaCl(w/v) 1% NZ-Amine-A (w/v), 0.5% Hefeextrakt (w/v), 0.5% NaCl(w/v) 2% Bactotrypton (w/v), 0.5% Hefeextrakt (w/v), 10mM NaCl, 2.5 mmkcl, 10 mm MgCl 2, 10 mm MgSO 4, 20 mm Glucose 1% Bactotrypton (w/v), 0.5% Hefeextrakt (w/v), 0.5% NaCl(w/v), 20% (w/v) PEG-6000, 10% (v/v) DMSO, 100 mm MgSO 4 Für Agarplatten wurden je Liter Medium 20g Select-Agar zugegeben. Nachdem die Medien auf C abgekühlt waren, wurden je nach Bedarf Antibiotika zugesetzt. Mikrobiologische Methoden Aufbewahrung von Bakterienstämmen Die verwendeten Bakterienstämme wurden als DMSO-Kulturen bei 70 C aufbewahrt. Hierzu wurde eine Übernachtkultur mit DMSO [10% Endkonzentration (v/v)] gemischt und als Suspension eingefroren. 18
30 Material und Methoden Wachstumsbedingungen Flüssigkulturen wurden in Gefäßen, die eine ausreichende Belüftung zuließen, in Reagenzglasrollständern oder in thermostatisierten Schütteltruhen bei 37 C bzw. 28 C inkubiert. Bestimmung der Zelldichte in Flüssigkulturen Die Zellzahl von Flüssigkulturen wurde bestimmt, indem die optische Dichte der Kultur bei einer Wellenlänge von λ = 578 nm (OD 578 ) photometrisch gemessen wurde. Nach Miller entspricht eine optische Dichte OD 578 von 1 etwa 107 µg/ml Protein und einer Zellzahl von 10 9 Zellen pro ml (Miller, 1972). Molekularbiologische Methoden Standardmethoden Die routinemäßigen molekularbiologischen Arbeiten wie DNA-Restriktion, Ligation, Ethanolfällung, Dephosphorylierung mit alkalischer Phosphatase, Analyse von Restriktionsfragmenten durch Agarose- bzw. PAA-Gelelektrophorese, Isolation von Plasmid DNA aus E. coli-zellen mittels Cäsiumchlorid-Dichtegradienten-Zentrifugation, Transformation durch Elektroporation und Plasmid-Minipräparation (Concert Rapid Plasmid Miniprep Kit, Gibco Karlsruhe) wurden nach Standardprotokollen (Sambrook et al., 1989) bzw. nach Angaben der Hersteller durchgeführt. Polymerase-Kettenreaktion Die Polymeraskettenreaktion (PCR) wurde durchgeführt, um glpd bzw. glpd-fragmente in die Überexpressionsvektoren pqe30 bzw. ptyb11 zu klonieren. Der Reaktionsansatz setzte sich standardmäßig aus 0.5 µg chromosomaler DNA von E. coli [MC4100] bzw. 10 ng Plasmid-DNA, jeweils 100 pmol Vorwärts- bzw. Rückwärts- Oligonukleotidprimer (Primer), jeweils 4 µm Nukleotide, PCR-Puffer (nach Angaben des Herstellers) und 2.6 Units Polymerase (SAWADY Pwo-DNA-Polymerase). Die Polymerasekettenreaktion wurde in einem Thermocycler (Perkin Elmer GeneAmp PCR System 2400) durchgeführt. Nach einer initialen Denaturierung bei 95 C für 5 min erfolgten 30 Zyklen, zusammengesetzt aus jeweils einem Denaturierungsschritt, bei dem die DNA-Stränge 19
31 Material und Methoden voneinander getrennt werden (30 s bei 95 C), einem Annealingschritt, um die Primer mit dem Template (chromosomale DNA) zu hybridisieren (45 s, T m, s.u.) und einem abschließenden Elongationsschritt, bei dem die Primer komplementär zur Template-DNA von der Polymerase verlängert werden (90 s 72 C). Die Schmelztemperatur T m wurde nach folgender Formel bestimmt: T m = 69.3 C x (GC-Gehalt in %) 650/Oligonukleotidlänge 2 C Die amplifizierten DNA-Fragmente wurden mit dem Concert Rapid PCR Purification System (Gibco, Karlsruhe) gereinigt. Gerichtete Mutagenese mittels Fusions-PCR Punktmutationen sowie Deletionen wurden gerichtet in die kodierenden Sequenzen eingefügt, nach der von Ho und Mitarbeitern etablierten Methode (Ho et al., 1989). Hierfür wurden flankierende Primer, die das 5 -Ende bzw. das 3 -Ende beinhalten, sowie Mutationsprimer eingesetzt, die in der Mitte die gewünschte Mutation tragen und komplementär zueinander sind. Mit je einem mutierten und einem der flankierenden Primer wurde die vordere bzw. die hintere Hälfte der Sequenz durch PCR amplifiziert. Die beiden PCR-Produkte tragen die Punktmutationen und wurden in einer dritten PCR fusioniert. Da die PCR-Produkte im mutagenisierten Bereich überlappen, hybridisieren sie miteinander und wurden durch die PCR verlängert. Die Ausbeute der Fusions-PCR wurde durch Zusatz der flankierenden Primer gesteigert. Deletionen wurden ebenfalls durch Fusions-PCR hergestellt. Hierzu wurden Primer upstream bzw. downstream der Deletionsstelle ausgewählt. Da die Primer zusätzlich eine Schnittstelle trugen und in der Restriktionsschnittstelle komplementär zu einander waren, überlappten die PCR-Fragmente und konnten so wie oben beschrieben in einer dritten PCR verlängert werden. Die PCR-Produkte wurden durch elelektrophoretische Auftrennung, gefolgt von Elution aus einem Agarose-Gel mittels Qiaquick Gelextraction Kit (Qiagen) gereinigt. 20
32 Material und Methoden Konstruktion der Vektoren Zur Herstellung des Polyhistidin-GlpD-Fusionsprotein kodierenden Plasmids paw4 wurde das offene Leseraster glpd aus chromosomaler E. coli DNA [MC4100] ohne das Startcodon ATG amplifiziert. Dabei wurden als Vorwärtsprimer pqe30-glpd.fwd und als Rückwärtsprimer pqe30-glpd.rev eingesetzt. Das PCR-Produkt sowie der Überexpressionsvektor pqe30 wurden mit den Restriktionsenzymen BamHI und HindIII geschnitten. Das geschnittene PCR-Produkt wurde mit dem geschnittenen und dephosphorylierten Vektor ligiert. Das C-terminal um 190 Aminosäuren verkürzte, GlpD kodierende Plasmid paw13 wurde wie oben beschrieben mit den Primern CBRIII-glpD.fwd und CBRIII-glpD.rev erzeugt. Die GlpD ( )- GlpD-R359E, GlpD-K360E bzw. AH362/4DE kodierenden Plasmide paw9, paw16, paw17 bzw. paw18 wurden mittels PCR amplifiziert und gerichtet in pqe30-vektoren kloniert. Durch gerichtete Klonierung von glpd in den Überexpressionsvektor ptyb11 entstand das Plasmid paw7, das für ein Intein-GlpD-Fusionsprotein kodiert. Aus chromosomaler E. coli-dna wurde glpd mit den Primern glpd-ptyb11.fwd und glpd-ptyb11.rev amplifiziert. Vektor und PCR-Produkt wurden mit den Restriktionsenzymen PstI und SapI geschnitten und ligiert. Alle klonierten Konstrukte wurde von der Firma GATC (Konstanz) kontrollsequenziert. TSS-Transformation Die TSS Transformation nach Chung und Mitarbeitern ist ein schnelle und zuverlässige Methode, E. coli-zellen mit Plasmid-DNA zu transformieren (Chung et al., 1989). Einer Übernachtkultur der zu transformierenden Zellen wurde 1:100 mit LB-Medium verdünnt. Die bei 37 C wachsenden Zellen wurden in der logarithmischen Phase aliquotiert (50 µl) und auf Eis mit demselben Volumen von kaltem 2xTSS-Medium versetzt. 10 ng Plasmid-DNA wurde zu den kompetenten Zellen gegeben und 20 min auf Eis inkubiert. Der Transformationsansatz wurde in 1 ml SOC-Medium überführt und bei 37 C im Roller inkubiert. Anschließend wurden die Zellen auf Selektionsplatten ausplattiert. Da diese Methode keine hohe Transformationseffizienz ermöglicht, wurden zur Transformation von Ligationsansätzen eine Transformation durch Elektroschock durchgeführt (Sambrook et al., 1989). 21
33 Material und Methoden Biochemische Methoden SDS-Polyacrylamid-Gelelektrophorese SDS-PAGE wurde nach der Methode von Laemmli durchgeführt (Lämmli, 1970). Zur Auftrennung von rekombinanten GlpD-Proteinen wurden 10% Polyacrylamid (PAA)-Gele verwendet, die 0.1% SDS enthielten. Die Auftrennung des verkürzten CBRII-GlpD- Fragments erfolgte in 15% PAA-Gelen. Die aufzutrennenden Proben wurden 1:1 (v/v) mit SDS-Probenpuffer (10 mm Tris-HCl ph % SDS, 5% β-mercaptoethanol, 0.05% Bromphenolblau) versetzt, 5 min bei 95 C erhitzt und einer SDS-PAGE unterzogen. Zur Bestimmung des apparenten Molekulargewichts wurden die Molekulargewichtsstandards SDS 7B (Sigma) und LMW-Marker (Pharmacia) verwendet. Coomassie Blue-Färbung von SDS-PAA-Gelen Nach der gelelektrophoretischen Auftrennung wurden die Proteinbanden mit 0.05% Coomassie Brilliant Blue R250 in 25% Isopropanol, 10% Essigsäure für mindestens eine Stunde angefärbt. Proteinbanden wurden durch Entfärben in 20% Methanol und 10% Essigsäure sichtbar gemacht. Western Blot und Immunfärbung von Proteinen Für einen spezifischen Nachweis wurden die mittels SDS-PAGE aufgetrennten Proteine nach der Semi-dry-Methode 1 h bei einem konstanten Stromfluss von 0.8 ma/cm 2 auf eine Nitrozellulosemembran transferiert (Kyhse-Andersen, 1984). Die Membran wurde zweimal jeweils 5 min mit TBSI-Puffer (10 mm Tris-HCl ph 7.4, 150 mm NaCl, 0.05% Nonidet P-40) gewaschen. Zur Absättigung der Protein-Bindestellen wurde die Membran mindestens eine Stunde in einer Blocklösung (Rotiblock, Roth) inkubiert. Nach zweimaligem Waschen (5 min) mit TBSI-Puffer erfolgte die Inkubation mit dem primären anti-glpd-antikörper (gelöst in TBSI, 3% BSA, 0.02% NaN 3 ). Dann wurde die Membran 3 x 3 min mit TBSI gewaschen und eine Stunde mit sekundärem Peroxidasegekoppelten anti-kaninchen-antikörper inkubiert. Nach 3 x 3-minütigem Waschen wurde der Blot mit 2 ml ECL-Detektionsreagenz für 1 min inkubiert. Die Chemilumineszenz des sekundären Antikörpers wurde auf einem Röntgenfilm nachgewiesen. 22
34 Material und Methoden Das ECL-Reagenz wurde wie folgt hergestellt: ECL-Lösung I: 100 mm Tris-HCl (ph 8.5, V = 500 ml) wurde unter ständigem Rühren entgast (Zugabe einiger Tropfen flüssigen Stickstoffs zur Verdrängung von O 2 ). Tropfenweise wurden unter ständigem Rühren 2.5 ml einer 0.18 M p-cumarsäure (in DMSO gelöst) und 1.2 ml einer 0.5 M Luminol-Lösung (3-Aminophtalhydrazid, unter N 2 -Begasung in DMSO gelöst) zugegeben. Die Lösung wurde sterilfiltriert und in einer braunen Flasche bei RT aufbewahrt. Luminol-Aliquots wurden bei -20 C gelagert. ECL II besteht aus einer 18%igen H 2 O 2 -Lösung, die dunkel und kühl gelagert wurde. Die gebrauchsfertige Lösung wurde unmittelbar vor der Anwendung gemischt, wobei 2 ml ECL-Lösung I mit 10 µl ECL II gemischt wurden. Protein-Präzipitation mit 10% Trichloressigsäure Durch Trichloressigsäure-Fällung wurden Detergenzien und Lipide aus den Proteinproben entfernt. Hierzu wurde die wässrige Probe mit demselben Volumen an eisgekühlter 10% Trichloressigsäure gemischt (vortexen) und 20 min auf Eis inkubiert. Nach 15 min Zentrifugation (10.000xg, 4 C) wurde der Überstand dekantiert. Trichloressigsäure im Proteinpellet wurde durch mehrmaliges Waschen mit eisgekühltem 80% Aceton und H 2 O entfernt. Für SDS-PAGE wurden denaturierte Proteinproben mit SDS-Probenpuffer versetzt bzw. zur BCA-Proteinbestimmung in 0.1% SDS gelöst. Proteinbestimmung Proteinkonzentrationen wurden mit dem Coomassie Protein Assay Reagent (Pierce) nach der Methode von Bradford (Bradford, 1976) bestimmt. Proben, die Detergenzien oder Lipide enthielten, wurden mit 10% TCA präzipitiert. Die Proteinkonzentration wurde mit dem BCA Detection Kit (Pierce) nach der Methode von Smith (Smith et al., 1985) bestimmt. Die Konzentration homogener Proteinlösungen mit bekannter Aminosäurezusammensetzung wurden ferner durch UV-Absorption bei 280 nm nach Lambert Beer mit folgender Formel bestimmt : c = A 280 M W ( ε d) 23
35 Material und Methoden Wobei c = Proteinkonzentration (g/l), A = Absorption, Schichtdicke (cm), der Extinktionskoeffizient ε wurde wie folgt berechnet: M W = Molekulargewicht, d = ε 1 1 = ( ntryptophan ntyro sin nphenylalanin 10 + ncystein 30) mol cm Enzymaktivitätstest Die Enzymaktivität der aeroben Glycerin-3-Phosphat-Dehydrogenase wurde 10 min lang photometrisch bei Raumtemperatur mittels der PMS-gekoppelten Reduktion von MTT bei einer Wellenlänge von 570 nm gemessen (Kistler und Lin, 1972). Proteinmenge [μg] Der Testansatz (V=250 µl) enthielt 50 mm Tris-HCl, ph 7.4, 75 mm NaCl, 0.5 mm MTT, 0.2 mm PMS, 10 µm FAD, 75 ng gereinigtes GlpD. Die Reaktion wurde mit 20 mm D,L-Glycerin-3-Phosphat gestartet. Die spezifische Aktivität wurde angegeben in [nmol] reduziertes MTT pro [µg] GlpD und Minute. Sie wurde nach folgender Formel berechnet: spez. Aktivität GlpD = A 1 min ε 1 Gesamtvolumen 0.5cm Pr oteinmenge[ µ ] 570 g Der Extinktionskoeffizient ε des reduzierten MTTs beträgt 17 mm -1 cm -1 (Kistler und Lin, 1972), A bezeichnet die Absorbtionsänderung. 24
36 Material und Methoden Reinigung von rekombinantem GlpD über Chitinaffinitätschromatographie Rekombinantes GlpD wurde als Intein-Fusionsprotein überexprimiert und über Chitin- Affinitätschromatographie mit anschließender Abspaltung des Inteinrestes gereinigt. Das GlpD-Intein-Fusionsprotein (MW = 113 kda) kodierende Plasmid paw7 wurde in den Überexpressionsstamm ER2566 transformiert. Für die Reinigung wurde eine Übernachtkultur des GlpD-Expressionsstamms 1:100 mit NZA-Medium (100 µg/ml Ampicillin, V = 2 Liter) verdünnt. Nachdem die bei 37 C wachsenden Zellen die logarithmische Wachstumsphase (OD 600 = ) erreicht hatten, wurde die Proteinexpression mit 0.5 mm IPTG induziert. Vor Erreichen der stationären Phase (OD 600 = 1.0) wurden die Zellen durch Zentrifugation (8000xg, 30 min, 4 C) geerntet. Alle nun folgenden Schritte wurden bei 4 8 C durchgeführt. Die Zellen wurden 20fach konzentriert in Lysepuffer (20 mm Tris-HCl ph 7.4, 500 mm NaCl, 1 mm EDTA, 1% Tween20) resuspendiert und mittels einer zweifache Passage durch eine French-Pressure Cell Press (12000 p.s.i., 4 C, American Instrument Co, Silver Spring, MD, USA) lysiert. Der Überstand des abzentrifugierten (12000xg, 4 C) Lysats wurde auf eine mit Lysepuffer äquilibrierte Chitinsäule (V = 2 ml, Durchmesser 1 cm) bei einer konstanten Flussrate von 0.25 ml/min aufgetragen. Unspezifisch gebundene Proteine wurden mit 100 ml Waschpuffer (20 mm Tris-HCl ph 8.0, 1 M NaCl, 1 mm EDTA, 0.1% Tween20) bei einer Flussrate von 1 ml/min entfernt. Die Chitinmatrix wurde anschließend 20 h bei 23 C in einem Cleavagepuffer (20 mm Tris-HCl ph 8.0, 500 mm NaCl, 100 mm DTT, 0.1% Tween20) inkubiert. In Anwesenheit des Thiols DTT erfolgte die Abspaltung ( Splicing ) des rekombinanten GlpD von seinem N-terminalen Inteinrest (MW = 55 kda), der an die Chitinbeads gebunden hat. Das Protein wurde mit Elutionspuffer (20 mm Tris-HCl ph 7.4, 500 mm NaCl, 1 mm EDTA, 0.1% Tween20) von der Säule eluiert. Eluatfraktionen, die gereinigtes und abgespaltenes Protein enthielten, wurden dreimal gegen Dialysepuffer (1000faches Volumen, 50 mm Tris-HCl ph 7.4, 150 mm NaCl, 0.1% Tween20) dialysiert. Reinigung der Wildtyp-, CBRIII-, GlpD ( ), R349E-, K360E, AH362/4DE-Fusionsproteine über Ni 2+ /NTA-Affinitätschromatographie Rekombinantes GlpD-Wildtyp-Protein sowie das verkürzte CBRIII Fragment, die Deletionsmutante GlpD ( ) und die mutierten Isoformen R349E-, K360E, AH362/4DE wurden als N-terminale 6xHistidin-Fusionsproteine in einer einstufigen Affinitätschromatographie gereinigt. 25
37 Material und Methoden Die klonierten Plasmide, die anstelle des ersten Methionins sechs Histidine kodieren, wurden zur Expression in den Stamm M15 transformiert. Wegen einer beobachteten Plasmid- Instabilität wurde vor jeder Reinigungsprozedur das jeweilige Plasmid frisch transformiert. Die Expression unterlag der Kontrolle des Lactose-Operons und konnte durch IPTG induziert werden. Um zu vermeiden, dass die Zellen vor der logarithmischen Wachstumsphase rekombinantes Protein exprimieren ( ground leakage ), wurde Lactose-freies Medium verwendet. Für die Reinigung wurde eine Übernachtkultur des GlpD-Expressionsstamms 1:100 mit NZA-Medium (V = 4 Liter) verdünnt. Nachdem die bei 28 C wachsenden Zellen die logarithmische Phase erreicht hatten (OD 600 = ), wurden sie mit 1 mm IPTG induziert. Drei Stunden nach der Induktion wurden die Zellen durch Zentrifugation (8000xg, 30 min, 4 C) geerntet. Das Zellpellet wurde in flüssigem Stickstoff eingefroren und 15 min auf Eis aufgetaut. Alle nachfolgenden Schritte wurden bei 4-8 C durchgeführt. Die aufgetauten Zellen wurden in Lysepuffer (50 mm NaPO 4 ph 8.0, 300 mm NaCl, 1% Tween20, 10 mm β-mercaptoethanol, 10 mm Imidazol) 20fach konzentriert resuspendiert und mittels zweimaliger Passage durch eine French-Pressure Cell Press (12000 p.s.i., 4 C) lysiert. Nach Zentrifugation (40.000xg, 30 min, 4 C) wurde der Überstand auf eine mit Lysepuffer äquilibrierte Ni 2+ -NTA-Agarose-Säule (Qiagen, V = 1ml) bei einer konstanten Flussrate von 5 ml/min aufgetragen. Unspezifisch gebundene Proteine wurden durch Waschen mit Waschpuffer I (50 mm NaPO 4 ph 7.0, 10 mm Imidazol, 300 mm NaCl, 1% Tween20, 10 mm β-mercaptoethanol) sowie Waschpuffer II (50 mm NaPO 4 ph 7.0, 30 mm Imidazol, 300 mm NaCl, 1% Tween20, 10 mm β-mercaptoethanol) entfernt. Die Polyhistidin-Fusionsproteine wurden mit Elutionspuffer (50 mm NaPO 4 ph 7.4, 250 mm Imidazol, 300 mm NaCl, 1% Tween20, 10 mm β-mercaptoethanol) bei einer Flussrate von 0.5 ml/min eluiert. Eluatfraktionen, die gereinigtes Protein enthielten, wurden dreimal gegen Dialysepuffer (1000faches Volumen, 50 mm Tris-HCl ph 7.4, 150 mm NaCl, 0.1% Tween20) dialysiert. Für Liposomen-stimulierte Enzymaktivitätsmessungen sowie Monolayer-Experimente und LUVET-Bindungsstudien wurde die Reinigungsprozedur mit folgenden Modifikationen durchgeführt: Die Puffer enthielten weder Tween20 noch andere Detergenzien. Im Lysepuffer waren 500 mm NaCl, im Waschpuffer II 1 M NaCl und im Elutionspuffer 250 mm NaCl und 10% Glycerin enthalten. Gereinigtes Protein wurde dreimal gegen Dialysepuffer (1000faches Volumen, 50 mm Tris-HCl ph 7.4, 75 mm NaCl und 10% Glycerin) dialysiert. 26
38 Material und Methoden Bindungsstudien von Wildtyp- und mutagenisiertem GlpD an Calmodulin- Sepharose 4B Die Bindungsstudien wurden in einem sogenannten Batchverfahren durchgeführt. Alle Schritte wurden bei 8 C durchgeführt, und von jedem Schritt wurde ein Aliquot abgenommen, das anschließend mittels SDS-PAGE und Immunoblotting analysiert wurde. Um die Bindung von GlpD an Calmodulin (CaM) zu charakterisieren, wurde, falls nicht anders angegeben, 1 µg GlpD in einem Testansatz von 250 µl mit 15 µl CaM-Sepharose 4B in Anwesenheit von Ca 2+ /CaM-Bindepuffer (50 mm Tris-HCl ph 7.4, 150 mm NaCl, 2 mm CaCl 2, 0.1% Tween20) auf einem Roller für 1 h bei 8 C inkubiert. In den Kontrollansätzen wurde Ca 2+ /CaM-Bindepuffer, der 10 mm EGTA (anstelle von 2 mm CaCl 2 ) enthielt, verwendet bzw. statt der CaM-gekoppelten Sepharose wurde nur Sepharose 4B eingesetzt. Bei Kompetitionsstudien wurden CaM-Antagonisten (50 µg/ml Compound 48/80, 150 µm TFP, 20 mm R24571, 60 µm Melittin, 150 µm Fendilin, 100 µm Chlorpromazin) in die Reaktionsansätze gegeben. Die Proben wurden bei 8 C 1 h auf dem Roller inkubiert. Der Überstand wurde nach kurzer Zentrifugation des Testansatzes abgenommen und das Sepharose-Sediment wurde in einem Gesamtvolumen von 1 ml CaM-Bindepuffer resuspendiert. Dieser Waschschritt wurde zunächst dreimal mit Ca 2+ /CaM-Bindepuffer und dreimal mit Waschpuffer (50 mm TrisHCl ph 7.4, 500 mm NaCl, 2 mm CaCl 2, 0.1% Tween20) wiederholt, um unspezifisch gebundenes Protein zu entfernen. Um spezifisch gebundenes Protein zu eluieren, wurde das Sediment in 250 µl Elutionspuffer (50 mm Tris-HCl ph 7.4, 150 mm NaCl, 10 mm EGTA, 0.1% Tween20) resuspendiert. Nach einer 30-minütigen Inkubation auf dem Roller bei 8 C wurde nach kurzer Zentrifugation der Überstand abgenommen. Nach zwei weiteren Waschschritten mit Elutionspuffer (V = 1 ml) wurde das Pellet mit SDS-Probenpuffer (in einem Gesamtvolumen von 250 µl) versetzt. Bei Kompetitionsstudien mit kleinen Liposomen (SUVs) wurde der Bindungstest wie oben beschrieben jedoch ohne Tween20 durchgeführt. Präparation von SUVs (Small Unilamellar Vesicles) Zur Herstellung von Lipidlösungen (10-20 mg Lipid/ml) wurden organische Lösungsmittel eingesetzt: 100% Chloroform bei zwitterionischen Phospholipiden bzw. ein Chloroform:Methanol-Gemisch (75/25 (v/v)) bei anionischen Phospholipiden. Es wurden ausschließlich Glasgefäße verwendet. Sollten Liposomen mit unterschiedlicher Lipid- Zusammensetzung hergestellt werden, so wurden die Lipide in organischem Lösungsmittel 27
39 Material und Methoden gemischt. Dann wurde ein Lipidfilm hergestellt, indem das organische Lösungsmittel unter Stickstoff-Begasung verdampft wurde. Der Lipidfilm wurde im Exsikkator getrocknet. Mehrschichtige große Liposomen (multilamellar vesicles, MLV) bilden sich spontan, wenn der Lipidfilm in wässriger Lösung (50 mm Tris-HCl ph 7.4, 75 mm NaCl) vollständig hydratisiert wurde, z.b. durch vortexen unter Zuhilfenahme von Glaskügelchen. Kleine, einschichtige Liposomen (mit einem Durchmesser von nm) wurden durch Ultraschallbehandlung hergestellt. Dazu wurde die MLVs enthaltende Lipidsuspension auf Eis unter Stickstoff-Begasung 10mal 30s bei 50 W sonifiziert, wobei nach jedem Burst eine 30s Pause erfolgte. Bildeten sich SUVs, so wurde die Suspension klarer, die Lösung war leicht neblig transparent. Größere multilamellare Strukturen wurden durch Zentrifugation (30 min, xg) sedimentiert, die SUVs waren im Überstand enthalten. Präparation LUVETs (Large Unilamellar Vesicles prepared by Extrusion Technique) Große, einschichtige Liposomen (Durchmesser nm) wurden aus MLVs hergestellt, die nach 10 Einfrier-Auftau-Zyklen in einem Ethanol/Trockeneisbad (-80 C) 10mal durch einen Polycarbonatfilter (200 nm Porengröße) gepresst wurden (Extrusion). Aufgrund ihrer Form sind LUVETs geeignet, Biomembranen zu modellieren. Lipid Extraktion nach Bligh und Dyer Phospholipide wurden aus wässrigen Lösungen nach der Methode von Bligh und Dyer isoliert (Bligh und Dyer, 1959). Wurden Phospholipide aus kleinen Volumina (1-5 ml) zur quantitativen Analyse gereinigt, so wurden in einem ersten Schritt 0.8 Volumeneinheiten der zu untersuchenden Probe (enthält 0.1 M HCl) mit 2 Volumeneinheiten Methanol und 1 Volumeneinheit Chloroform durch vortexen gemischt. Zu diesem Ein-Phasen-Gemisch wurde eine Volumeneinheit 0.1 M HCl und eine Volumeneinheit Chloroform gegeben und gemischt. Es bildet sich ein zwei Phasen-Gemisch, wobei sich Chloroform mit den gelösten Lipiden in der unteren Phase befindet. Die Chloroformschicht wurde quantitativ in ein Glasröhrchen überführt. Die wässrige Phase wurde zweimal mit 1.2 Volumeneinheiten Chloroform gewaschen. 28
40 Material und Methoden Phosphor Bestimmung nach Fiske Subbarow Die Menge an Phospholipiden wurde durch eine Phosphorbestimmung nach der Methode von Fiske-Subbarow bestimmt (Fiske und Subbarow, 1925). Die zu analysierenden Proben wurden in phosphorfreie Glasröhrchen überführt und im Trockenofen (200 C) eingeengt. Zu den getrockneten Proben wurde jeweils 0.3 ml 70% Perchlorsäure gegeben. Das mit einer Glasmurmel abgedichtete Röhrchen wurde 3 h bei 180 C inkubiert, um die Phospholipide zu zerstören. Zu den abgekühlten Proben wurden jeweils 3 ml Ammoniummolybdatlösung und 120 µl Fiske-Subbarow Reagenz gegeben. Die gevortexten Proben wurden 15 min in einem Wasserbad gekocht und nach Abkühlung photometrisch bei 830 nm gemessen. Eine Eichgerade wurde aus Proben mit nmol anorganischem Phosphat (0.5 mm KH 2 PO 4 ) erstellt. Dazu wurden folgende Reagenzien hergestellt: Ammoniummolybdat-Lösung: 2.2 g NH 4 -Molybdat wurde in 20 ml 98% H 2 SO 4 gelöst (= 90 mm (NH 4 ) 6 MO 7 O 24 x4h 2 O). Nach 15 min Rühren wurde die Lösung mit H 2 O 20fach verdünnt. Fiske-Subbarow Reagenz: In 200 ml H 2 O wurden g Natriumdisulfit (= 720 mm Na 2 S 2 O 5 ) und 1 g Natriumsulfit (= 40 mm Na 2 SO 3 ) gelöst. Dann wurden 0.5 g 1-Amino-2-Naphtalen-4-Sulfonsäure zugegeben und unter ständigem Rühren bei 40 C gelöst und über Nacht stehen gelassen. Das filtrierte Reagenz wurde in einer lichtundurchlässigen Flasche aufbewahrt. Isolierung und Reinigung des gesamten Phospholid-Extrakts aus E. coli (TLE) W3899-Zellen (V= 5 Liter) wurden in der späten logarithmischen Phase (OD 600 = 0.8) geerntet (30 min, 8000xg, 4 C) und mit physiologischer Salzlösung (PSS, 0.9% w/v NaCl) gewaschen. Das Nassgewicht der Zellpellets wurde bestimmt. 12 g Zellpellet wurden in ml PSS resuspendiert. Die Extraktion aller Phospholipide aus E. coli erfolgte nach der Methode von Bligh und Dyer unter Verwendung von Glasgefäßen. Eine Volumeneinheit Zellsuspension (= ml) wurde mit einer Volumeneinheit Chloroform und 2.2 Volumeneinheiten Methanol in einem Scheidetrichter gemischt. Das Gemisch wurde nun im Scheidetrichter mindestens 5 29
41 Material und Methoden min geschwenkt (hin und wieder wurde Chloroform durch einen Hahn entweichen gelassen), bis sich ein Ein-Phasensystem eingestellt hatte. Ein optimales Einphasengemisch erkennt man an vielen rasch entweichenden Luftblasen. Dann wurde jeweils eine Volumeneinheit Chloroform und PSS zugegeben und das Gemisch erneut für mindestens 5 min geschwenkt. Sobald eine Phasentrennung auftrat, wurde die Chloroformphase (unterste Phase: enthält Phospholipide, neutrale Lipide und Fettsäuren) abgelassen und gesammelt. Die wässrige Phase wurde erneut mit einer Volumeneinheit Chloroform unter Schwenken gewaschen und nachdem sich das Gemisch in zwei Phasen aufgetrennt hatte, wurde die Chloroformphase abgelassen und gesammelt. Dies wurde noch ein weiteres Mal wiederholt. Das gesammelte Lipid-Chloroform-Gemisch wurde in einem Rotationsevaporator eingedampft. Da der rohe Lipidextrakt noch viel Protein enthält, wurde die Prozedur erneut wiederholt. Um neutrale Lipide, Fettsäuren und andere Kontaminationen aus dem Lipidextrakt zu entfernen, wurde der rohe Lipidextrakt in einem kleinen Volumen (1-10 ml) auf eine Silicagel-Säule (Baker 60 mm Partikel, Durchmesser 2 cm, Länge 25 cm) geladen. Die Säule wurde mit 500 ml Chloroform gewaschen, dabei eluierten alle neutralen Lipide und andere Kontaminationen. Phospholipide wurden durch 500 ml Chloroform:Methanol (1:1, v/v) eluiert. Die Reinheit des Extraktes wurde durch Dünnschicht-Chromatographie bestimmt (Fine und Sprecher, 1982). Monolayer-Experimente Lipid-Moleküle orientieren sich an einer Pufferoberfläche spontan zu einem Lipid-Monolayer. Die polaren Kopfgruppen sind in Kontakt mit der wässrigen Phase, die hydrophoben Kohlenwasserstoffketten ragen heraus. Mit der Wilhelmy Plate Methode (Demel, 1994) wurde der Druck von Lipidmonolayern gemessen. Lagert sich ein Peptid zwischen die Kohlenwasserstoffketten der Lipide ein, so bewirkt dies eine messbare Änderung der Oberflächenspannung. Da die Oberflächenspannung der Pufferlösung bekannt ist, entspricht die Differenz zwischen der gemessenen Spannung und der Pufferoberflächenspannung dem Druck des Lipidlayers. Informationen über einen aufgesprühten Lipidmonolayer erhält man durch die Messung des Drucks an der Oberfläche. Ein dünnes Platinplättchen (1.96 cm breit, 1 cm lang) mit einer rauen Oberfläche wurde in eine wässrige Lösung getaucht. Das Gewicht des Plättchens wurde durch eine Waage (Cahn 2000 Microbalance) bestimmt und diente als Maß für den Oberflächendruck. Der Druck an der Oberfläche π wird definiert als die Differenz 30
42 Material und Methoden zwischen der Oberflächenspannung der wässrigen Phase (γ Puffer = 72 mn/m) und der Oberflächenspannung des die Oberfläche bedeckenden Lipidfilms: π = γ Puffer γ Monolayer Die Wilhelmy Plate Methode misst den Druck nicht direkt, sondern die jeweilige Oberflächenspannung. Somit sind Messungen kleinster Druckänderung mit einer Genauigkeit von 0.01 mn/m möglich. Mit dem teilweise eingetauchten Plättchen wird die Spannungsänderung gemessen. Auf das Platinplättchen wirken verschiedene Kräfte. Einerseits wirkt das Gewicht P und die Oberflächenspannung nach unten, anderereits die Archimedes Auftriebskraft A nach oben. Die Netto-Kraft, die nach unten wirkt, wird folgendermaßen berechnet: F = P + 2 ( w + t) γ cosθ A Wobei w die Breite, t die Dicke des Plättchens ist (da t <<w, kann t vernachlässigt werden). θ ist der Kontaktwinkel zwischen der flüssigen und festen Phase. Ist das Plättchen vollständig benetzt (begünstigt durch raue Oberfläche), so ist der Kontaktwinkel θ = 0, cos θ = 1, so dass gilt: F = P + 2 w γ A Wird ein Monolayer auf die Oberfläche gesprüht, so gilt: F = ( P + 2 w γ A) ( P + 2 w γ A) Puffer Monolayer da P und A konstant bleiben, kann man folgendermaßen vereinfachen: F = 2 w ( γ Puffer γ Monolayer ) Da der Druck an der Oberfläche der Differenz zwischen der Oberflächenspannung des Puffers und des Lipidfilms entspricht, gilt: 31
43 Material und Methoden F = 2 w π daraus folgt: F π = 2 w Für Monolayer-Experimente wurde der Druck einer Oberfläche bei 28+1 C und kontinuierlichem Rühren gemessen. Filtrierter und entgaster Subphasenpuffer (50 mm Tris-HCl, 75 mm NaCl ph 7.4) wurde in ein Teflongefäß gefüllt (5 ml Volumen, 8.81 cm 2 Oberfläche). Die Lipide wurden aus einer Chloroform- bzw. Chloroform:Methanol-(1:1 v/v) Stammlösung auf die Pufferoberfläche gesprüht. Bei den Messungen wurden Monolayer mit einem Oberflächendruck zwischen 25 und 35 mn/m verwendet. Experimente mit geringeren Anfangsdrücken wurden nicht durchgeführt, da die durch GlpD induzierte Oberflächenspannung bei 24 mn/m lag. In allen Experimenten wurden sättigende Mengen an gereinigten Proteinen (3 µg/ml) durch ein Loch am Rande des Teflongefäßes in die Subphase injiziert. Druckänderungen wurden verfolgt, bis der Anstieg ein Gleichgewicht erreicht hatte (gewöhnlich nach min). Nach jeder Messung wurde das Teflongefäß mechanisch mit nichtionischer Seife (Zeep) gesäubert und unter kontinuierlichem Rühren mit Methanol/Essigsäure (10:1, v/v) für 20 h gereinigt. Die Messungen erfolgten unter N 2 -Atmosphäre. Vesikel-Bindungsstudien Vesikel-Bindungsstudien wurden mit LUVETs aus TLE, DOPE:PC (6:4 molares Verhältnis) bzw. DOPG und gereinigtem wt-glpd bzw. den R359E- K360E- und AH362DE-Mutanten durchgeführt. LUVETs wurden mit gereinigtem Protein in 400 µl 50 mm Tris-HCl ph 7.4, 75 mm NaCl zwei Stunden bei Raumtemperatur inkubiert. Durch einstündige Zentrifugation ( xg, 4 C) sedimentierten die Vesikel vollständig. In einem Kontrollexperiment wurden Vesikel ohne Protein sedimentiert und der Phospholipidgehalt von Überstand und Pellet bestimmt. Es zeigte sich, dass mindestens 95% der Vesikel sedimentierten. Die Proteine wurden dagegen in Abwesenheit von Vesikeln zu 99% im Überstand nachgewiesen. Eine qualitative Analyse von kosedimentiertem GlpD erfolgte durch SDS-PAGE und anschließendem Immunoblotting. Im Überstand und Pellet der LUVET-Protein-Ansätze wurde die Proteinkonzentration nach Fällung von Aliquots des Überstands und des resupendierten Pellets mit 10% Trichloressigsäure mit dem Pierce BCA Detection Kit bestimmt. 32
44 Ergebnisse Ergebnisse Spezifität der Bindung von GlpD an CaM Klonierung, Überexpression und Reinigung von GlpD Die Isolierung von rekombinantem GlpD wurde auf zwei unterschiedlichen Wegen erzielt. Einerseits wurde GlpD als Fusionsprotein mit 6 N-terminalen Histidinen (vom Plasmid paw4) exprimiert und über eine einstufige Affinitätschromatographie an einer Ni 2+ /NTA- Säule gereinigt (Abb. 5A) und andererseits als Intein-Fusionsprotein (vom Plasmid paw7) exprimiert und über eine Chitin-Affinitätschromatographie nach Abspaltung des fusionierten Inteins gereinigt (Abb. 5B). In beiden Fällen lag das rekombinante, überexprimierte Protein in löslicher Form vor, so dass unter nativen Bedingungen gereinigt werden konnte. Abbildung 5: Reinigung von rekombinantem GlpD. GlpD wurde als A: N-terminales poly-histidin-fusionsprotein überexprimiert und über Ni 2+ /NTA-Affinitätschromatographie gereinigt bzw. B: als N-terminales Intein- Fusionsprotein überexprimiert und über eine Chitin-Affinitätschromatographie mit Intein-Abspaltung gereinigt. Die Proben wurden durch 10% SDS-PAGE aufgetrennt und mit Coomassie Blue Färbung sichtbar gemacht. Folgende Fraktionen sind gezeigt: n.i.: Zellextrakt von nicht induzierten Zellen RE: Zellextrakt induzierter Zellen FP-S: Überstand des abzentrifugierten French-Press-Zelllysats FP-P: Pellet des abzentrifugierten French- Press-Lysats E: GlpD-Eluatfraktion. 33
45 Ergebnisse Photometrisch wurde die Aktivität beider GlpD-Proteine durch einen PMS-gekoppelten MTT-Enzymaktivitätstest bestimmt. Die spezifische Enzymaktivität des von paw4 exprimierten GlpD-6xHistidin-Fusionsproteins bzw. des von Plasmid paw7 exprimierten GlpD-Proteins wurde in mindestens drei Experimenten ermittelt. Der mittlere Wert und die Standardabweichung wurde mit [nmol/(µg x min)] bzw [nmol/(µg x min)] bestimmt. Die Aktivität beider Proteine wird durch das nichtionische Detergenz Tween20 ca. 3-fach stimuliert (Daten nicht gezeigt). Beide rekombinanten Proteine sind enzymatisch aktiv und verhalten sich bezüglich ihrer Aktivität wie Wildtyp-GlpD (Weiner und Heppel, 1972). Calcium-abhängige CaM-Bindung Um die Bindungseigenschaften von GlpD an CaM zu untersuchen, wurde gereinigtes GlpD mit immobilisiertem Rinder-CaM inkubiert. In einem sogenannten Batchverfahren wurde gereinigtes 6-Histidin-GlpD Fusionsprotein (95%ige Reinheit) mit CaM-Sepharose 4B in Anwesenheit von 2 mm Calcium bzw. 10 mm EGTA inkubiert. Das Gel wurde mit 500 mm NaCl gewaschen und anschließend mit EGTA inkubiert, um gebundenes GlpD zu eluieren. Der in Abbildung 6 gezeigte Western Blot dokumentiert die spezifische Bindung von GlpD an CaM. Im Kontrollansatz, der nur Sepharose-Matrix enthielt, ist kein gebundenes GlpD vorhanden. GlpD bindet in Anwesenheit von 2 mm Calcium nahezu vollständig an CaM und wird durch 10 mm EGTA eluiert. Im Ansatz mit 10 mm EGTA bindet dagegen sehr wenig GlpD. Nach EGTA-Elution wurden die CaM-Sepharose-Pellets mit SDS-Probenpuffer versetzt und über SDS-PAGE und anschließendem Immunoblot analysiert (Daten nicht gezeigt). Es konnte keine Bindung von GlpD an die Trägermatrix festgestellt werden. Dagegen wurden Spuren von GlpD bei einer Analyse der CaM-Pellets nach Elution sowohl im Ansatz mit Calcium als auch im Ansatz mit EGTA nachgewiesen (Daten nicht gezeigt). GlpD bindet offensichtlich an Bindungsstellen, die sowohl in der Calcium-gebundenen als auch der Calcium-freien Konformation präsent sind. Diese spezifische Wechselwirkung wurde ebenfalls mit GlpD beobachtet, das aus mit paw7 transformierten ER2566-Zellen isoliert wurde (Daten nicht gezeigt). Folglich kann ausgeschlossenen werden, dass der N-terminale Histidinrest die CaM-Bindung beeinflusst. 34
46 Ergebnisse Abbildung 6: Bindungsstudien von GlpD an immobilisiertes Rinder-CaM. Im Batch-Verfahren wurden in einem Testansatz von 250 µl 35 nm GlpD mit 30 µl CaM-Sepharose 4B in Anwesenheit von 2mM Calcium (Calcium-CaM) bzw. von 10 mm EGTA (EGTA-CaM) bei 8 C 1 h inkubiert. Als Kontrolle wurden 35 nm GlpD mit 30 µl Trägermatrix Sepharose 4B inkubiert (Control). Nach dreimaligem Waschen mit 500 mm NaCl wurde nach einer 30 minütigen Inkubation mit 10 mm EGTA gebundenes GlpD eluiert. Von jedem Schritt wurden Aliquots abgenommen, durch 10% SDS-PAGE aufgetrennt, auf Nitrozellulose transferiert und mit 1: verdünntem anti-glpd Antikörper immunmarkiert. D: Überstand mit nicht gebundenem GlpD; W1: erster NaCl-Waschschritt; E1: erste Eluatfraktion; E2: zweite Eluatfraktion. Inhibitionsstudien zur Bindung von GlpD an CaM Die Bindungsspezifität wurde durch Kompetitionsstudien mit CaM-Antagonisten näher charakterisiert. Es wurde geprüft, welche der chemisch unterschiedlichen CaM-Antagonisten die spezifische Bindung zwischen GlpD und CaM hemmen (Abb. 7). Folgende CaM-Antagonisten konnten die GlpD-CaM-Bindung nicht hemmen: die aus einer Familie von homologen hydrophoben Polykationen bestehende Substanz Compound 48/80 (Gietzen, 1983), Trifluoperazin, der Mikonazol-Abkömmling R24571 (Gietzen et al., 1981), Chlorpromazin, Fendilin sowie das Naphthalinsulfonamid W7. Lediglich das Bienengift Melittin konnte die Bindung wirksam inhibieren. Melittin ist ein kleines Peptid, das eine basisch amphiphile Helix ausbilden kann. In Anwesenheit von Ca 2+ /CaM wird die Bildung α-helicaler Strukturen von 5% auf 72% erhöht (Maulet und Cox, 1983). Aufgrund der spezifischen Kompetition zwischen GlpD und Melitittin kann gefolgert werden, dass GlpD in Anwesenheit von Ca 2+ /CaM ebenfalls eine basisch amphiphile α- helicale Domäne exponiert und an CaM bindet. 35
47 Ergebnisse Abbildung 7: Kompetitionsstudien zur Charakterisierung der GlpD-CaM Bindung. Je 35 nm GlpD wurden mit 15 µl Ca 2+ /CaM-Sepharose 4B in Anwesenheit von 50µg/ml Compound 48/80, 150 µm Trifluoperazin (TFP), 20 mm R24571, 60 µm Melittin, 150 µm Fendilin bzw. 100 µm Chlorpromazin in einem Gesamtvolumen von 250 µl 2 h bei 8 C inkubiert. Der Kontrollansatz enthielt nur Ca 2+ /CaM-Sepharose und GlpD. Durch dreimaliges Waschen mit 500 mm NaCl wurde unspezifisch gebundenes GlpD entfernt. Nach einer 30 minütigen Inkubation in 10 mm EGTA wurde gebundenes Protein eluiert. Nach jedem Zentrifugationsschritt wurde jeweils ein Aliquot abgenommen, durch 10 %ige SDS-PAGE aufgetrennt und nach Transfer auf Nitrozellulose mit anti-glpd-antikörper imunmarkiert. D: Überstand mit nicht gebundenem GlpD; W1: erster NaCl- Waschschritt; W3: letzter Waschschritt; E1: erste Eluatfraktion; E2: zweite Eluatfraktion. Bindung von GlpD an saure und hydrophobe Oberflächen Ca 2+ /CaM präsentiert eine saure und hydrophobe Oberfläche (James et al., 1995). Durch Komplexierung von Calcium mit EGTA wird eine Konformationsänderung bewirkt, in deren Folge weniger saure und hydrophobe Aminosäuren auf der Proteinoberfläche präsentiert werden. Die Beobachtung, dass GlpD bevorzugt an Ca 2+ /CaM bindet und dass GlpD mit Melittin um die CaM-Bindestellen kompetiert, deutet darauf hin, dass GlpD in Anwesenheit von Ca 2+ /CaM durch hydrophobe und elektrostatische Wechselwirkungen an Ca 2+ /CaM bindet. Um dies zu testen, wurde GlpD mit CaM in Anwesenheit von unterschiedlichen hydrophoben Substanzen inkubiert. Abbildung 8 dokumentiert, dass die GlpD-CaM-Bindung lediglich durch kleine Liposomen aus negativ geladenen Phospholipiden (DOPG-SUVs) gehemmt wird. Neutrale Liposomen, die aus jeweils 50% DOPE und DOPC gebildet wurden, 36
48 Ergebnisse beeinträchtigen die GlpD-Ca 2+ /CaM-Interaktion nicht. Die Zugabe von Dipalmitoylglycerin (DPG)-Mizellen, einer neutralen Fettsäure, hatte ebenfalls keinen Effekt. Folglich beruht die spezifische Interaktion zwischen GlpD und CaM auf hydrophoben und elektrostatischen Wechselwirkungen. Abbildung 8: Hemmung der GlpD-CaM Bindung. Dargestellt sind Bindungsstudien von GlpD an CaM, die in Anwesenheit von 70 mm DOPG-SUVs, 70 mm DOPE/PC-SUVs bzw. 5 mm DPG-Mizellen durchgeführt wurden. Der Bindungstest enthielt in einem Ansatz (V=250 µl) 35 nm GlpD und 15 µl Ca 2+ /CaM-Sepharose 4B. Im Kontrollansatz wurde nur GlpD mit CaM-Sepharose inkubiert. Nach 30 min Inkubation wurde das CaM- Pellet dreimal mit 500 mm NaCl gewaschen und anschließend für 30 min in 10 mm EGTA inkubiert, um gebundenes GlpD zu eluieren. Es wurden jeweils Aliquots zur 10 % SDS-PAGE abgenommen. Mittels Immunoblotting wurde GlpD in den einzelnen Fraktionen nachgewiesen. D: Überstand mit nicht gebundenem GlpD; W3: letzter NaCl-Waschschritt; E1: erste Eluatfraktion. Lokalisation der CaM-Bindedomänen Um die CaM-Bindedomäne von GlpD zu identifizieren, wurden glpd-fragmente kloniert, die für C-terminal verkürzte Proteine kodieren. Diese Proteine wurden überexprimiert und gereinigt. Anschließend wurden sie bezüglich ihrer Fähigkeit getestet, spezifisch an Ca 2+ /CaM-Sepharose 4B zu binden. Abbildung 9A dokumentiert die Reinigung des um 190 Aminosäuren verkürzten CBRIII-Fragments. Die apparente Größe des gereinigten Fragments betrug 30 kda. Wie Abbildung 9B zeigt, bindet das CBRIII-Fragment nicht an die Ca 2+ /CaM-Sepharose 4B, da 37
49 Ergebnisse es nur im Überstand nachgewiesen werden kann. Bereits eine C-terminale Deletion von 190 Aminosäuren verhindert folglich eine Bindung an Ca 2+ /CaM. Abbildung 9: Reinigung und CaM-Bindungstest des C-terminal verkürzten GlpD-Fragments CBRIII. A: Überexpression und Reinigung des C-terminal um 190 Aminosäuren verkürzten GlpD-Fragments als N- terminales poly-histidin-fragment. Die Proben wurden durch 15% SDS-PAGE aufgetrennt und mit Coomassie Blue Färbung sichtbar gemacht. Die Abbildung zeigt folgende Fraktionen: 1: Zellextrakt uninduzierter Zellen 2: Zellextrakt induzierter Zellen 3: Überstand des abzentrifugierten French-Press-Zelllysats 4: Eluatfraktion des gereinigten CBRIII-Fragments B: Der Bindungstest enthielt in einem Ansatz (V=250 µl) 35 nm CBRIII und 15 µl Ca 2+ /CaM-Sepharose 4B. Nach 30 min Inkubation wurde das CaM-Pellet dreimal mit 500 mm NaCl gewaschen und anschließend für 30 min in 10 mm EGTA inkubiert, um gebundenes CBRIII zu eluieren. Es wurden jeweils Aliquots zur 15 % SDS-PAGE abgenommen. Mittels Immunoblotting wurde CBRIII in den einzelnen Fraktionen nachgewiesen. D: Überstand mit nicht gebundenem CBRIII; W3: letzter NaCl- Waschschritt; E1 und E2: Eluatfraktionen. Basisch amphiphile Helix: CaM-Bindemotiv von GlpD? Sekundärstrukturvorhersageprogramme wie PHD (Rost et al., 1994), SOPM (Geourjon und Deleage, 1994), PREDATOR (Frishman und Argos, 1996) und GOR3 (Gibrat et al., 1987) ermittelten für GlpD im Bereich von Aminosäuren eine basische α-helix mit amphiphiler Ausprägung (Eisenberg et al., 1984). Um zu überprüfen, ob dieser Bereich für die CaM-Bindung notwendig ist, wurde durch FusionsPCR die Deletionsmutante GlpD ( ) kloniert, überexprimiert und gereinigt. CaM-Bindungsstudien zeigen, dass GlpD ohne die basisch amphiphile Helix nicht mehr an CaM binden kann (Abb. 10). 38
50 Ergebnisse Abbildung 10: Bindungsstudien mit gereinigtem GlpD ( ) und immobilisiertem Rinder-CaM. In einem Testansatz von 250 µl wurden 35 nm GlpD ( ) mit 15 µl Ca 2+ /CaM-Sepharose 4B inkubiert (delta351-72). Als Kontrolle wurden 35 nm GlpD mit 15 µl Ca 2+ /CaM-Sepharose 4B inkubiert (wt-glpd). Nach dreimaligem Waschen mit 500 mm NaCl wurde 30 min mit 10 mm EGTA inkubiert, um gebundenes GlpD zu eluieren. Von jedem Schritt wurden Aliquots abgenommen, durch 10% SDS-PAGE aufgetrennt, auf Nitrozellulose transferiert und mit 1:50000 verdünntem anti-glpd Antikörper immunmarkiert. Spur 1 zeigt durch 10% SDS-PAGE aufgetrenntes und Coomassie Blue gefärbtes Wildtyp-GlpD sowie GlpD ( ). D: Überstand mit nicht gebundenem GlpD; W3: letzter NaCl-Waschschritt; E1: erste Eluatfraktion; E2: zweite Eluatfraktion. Welche Rolle spielen die positiv geladenen Reste im α-helicalen Bereich von GlpD für die Bindung an CaM? Werden die Aminosäuren in einer Helix-Projektion dargestellt, wird die basisch amphiphile Struktur deutlich (Abbildung 11). Die eine Hälfte der Helix hat einen hydrophoben, die andere Seite einen hydrophilen Charakter, der zudem positiv geladen ist. Die Nettoladung der α-helix beträgt unter physiologischen Bedingungen +1. Es stellt sich nun die Frage, ob die positive Ladung der Helix für die CaM-Bindung notwendig ist. Mittels gerichteter Mutagenese wurden drei Punktmutanten hergestellt, die durch eine elektronegative anstelle einer elektropositiven Helix charakterisiert sind. Es wurden zwei Mutanten (R359E, K360E) erzeugt, bei denen jeweils eine basische Aminosäure (Arginin359 bzw. Lysin360) gegen die saure Aminosäure Glutamat ausgetauscht wurden. Bei der Doppelmutante wurden zwei Aminosäuren, nämlich Alanin362 gegen Glutamat, sowie Histidin364 gegen Aspartat ausgetauscht. Alle drei Mutanten haben im Bereich der α-helix eine negative Nettoladung von 1. Das jeweils gezielt mutagenisierte offene Leseraster von glpd wurde in einen pqe30-vektor kloniert. Die Punktmutanten wurden überexprimiert und über Ni 2+ /NTA-Affinitätschromatographie gereinigt (Abb. 12A). 39
51 Ergebnisse Wildtyp -L 355 T T Y R 359 K 360 L A 362 E H 364 A L E K L T 370 R359E L 355 T T Y E 359 K L A E H A L E K L T 370 K360E L 355 T T Y R E 360 L A E H A L E K L T 370 AH362/4DE L 355 T T Y R K L D 362 E E 364 A L E K L T 370 M,I,L,V F,Y,W G,A,P D,E K,H,R S,T 355 L L R 370 T 363 E T 356 E A 369 L K Y A 365 L 361 K 368 T H Abbildung 11: Aminosäuresequenz und Helical-Wheel-Projektion vonglpd im Bereich der Aminosäuren Wildtyp-Sequenz der vorhergesagten α-helix im Bereich der Aminosäuren Die Strategien für die Punktmutation sind darunter dargestellt. In der Helical-Wheel Projektion der Wildtyp-Sequenz entsprechen die Graustufen dem Hydrophobizitätsgrad der Aminosäuren, wobei Reste mit größter Hydrophobizität schwarz dargestellt sind. Die mutierten GlpD-Proteine wurden als 6xHistidin-Fusionsproteine überexprimiert und gereinigt (Abb. 12a). CaM-Bindungsstudien mit den gereinigten Proteinen zeigten, dass die Doppelmutante ebenso wie der Wildtyp an CaM bindet (Abb. 12B). Keine Bindung wurde bei den Punktmutanten K360E und R359E beobachtet. 40
52 Ergebnisse Abbildung 12:Reinigung von GlpD-Punktmutanten und Bindungsstudien mit immobilisiertem Rinder- CaM. wt-glpd, R359E-, K360E- und AH362/4DE-Mutanten wurden als N-terminale poly-histidin- Fusionsproteine überexprimiert und über Ni 2+ /NTA Affinitätschromatographie gereinigt. A: Die gereinigten und dialysierten Proteine wurden durch 10% SDS-PAGE aufgetrennt und durch Coomassie Blue Färbung sichtbar gemacht. B: Bindungstest von wt-glpd und Punktmutanten mit immobilisiertem CaM. In einem Testansatz von 250 µl wurden jeweils 35 nm wt-glpd, AH362/4DE, R359E bzw. K360E mit 15 µl Ca 2+ /CaM-Sepharose 4B inkubiert. Als Kontrolle wurden 35 nm GlpD mit 15 µl Ca 2+ /CaM-Sepharose 4B inkubiert (wt-glpd). Nach dreimaligem Waschen mit 500 mm NaCl wurden die Ansätze für 30 min in 10 mm EGTA inkubiert, um gebundenes GlpD zu eluieren. Von jedem Schritt wurden Aliquots abgenommen, durch 10% SDS-PAGE aufgetrennt, auf Nitrozellulose transferiert und mit 1:50000 verdünntem anti-glpd Antikörper immunmarkiert. D Überstand mit nicht gebundenem GlpD; W3: letzter NaCl-Waschschritt; E1: erste Eluatfraktion; E2: zweite Eluatfraktion. 41
53 Ergebnisse Zusammenfassend wird aus den hier präsentierten Ergebnissen gefolgert, dass GlpD eine basische amphiphile α-helix ausbildet, die mit der sauren und hydrophoben Oberfläche von CaM interagiert. Es stellt sich nun die Frage, welche physiologische Bedeutung der in vitro beobachtete Befund hat. Aufgrund dieser biochemischen Charakterisierung wird postuliert, dass die in vitro beobachtete Protein-Protein-Wechselwirkung die Assoziation von GlpD an die innere Plasma-Membran widerspiegelt. Die Hypothese einer direkten Lipid- Protein-Interaktion zwischen GlpD und der Cytoplasma-Membran wurde sowohl in Lipid- Monolayer- als auch in Lipid-Bilayer-Systemen überprüft. Charakterisierung der GlpD-Lipid-Bindung Eine biochemische Charakterisierung von Lipid-Protein-Wechselwirkungen kann nur unter Detergenz-freien Bedingungen erfolgen. Deshalb wurden sowohl Wildtyp-GlpD als auch die mutierten GlpD-Isoformen ohne Verwendung von Detergenz gereinigt. Hohe Salzkonzentrationen im Waschpuffer verhinderten eine Verunreinigung mit Lipiden in der Präparation. Der Lipidgehalt der gereinigten Proteine wurde bestimmt, indem Aliquots einer Lipid- Extraktion nach der Methode von Bligh und Dyer (1959) unterzogen wurden. Anschließend wurde der Phosphorgehalt des Extrakts nach der Methode von Fiske-Subbarrow (1925) bestimmt. Es konnte keine Verunreinigung mit Lipiden nachgewiesen werden (Daten nicht gezeigt). Direkte Protein-Lipid-Interaktion Mit der Wilhelmy Plate Methode wurde die Insertion von GlpD zwischen die Kohlenwasserstoffketten von Lipiden gemessen. Gereinigtes GlpD wurde bei einem Anfangsdruck von 27 mn/m unter einen Lipid-Monolayer injiziert, der aus einem Phospholipid-Extrakt von E. coli (TLE) bestand. Der zeitliche Verlauf der Druckänderung wurde bis zum Erreichen eines konstanten Endwertes protokolliert (Abb. 13). 40 Minuten nach der Injektion von GlpD wurde eine Druckänderung von 5 mn/m gemessen. Eine signifikante Druckänderung wird nur bei Substanzen beobachtet, die zwischen den polaren Kopfgruppen bis in die Kohlenwasserstoffkettenregion der Lipide eindringen (Demel, 1994). Eine bloße Bindung an die polaren Kopfgruppen der Lipide bewirkt dagegen keine signifikante Druckänderung (Mayer et al., 1983). Dieses Ergebnis kann folglich als effiziente Insertion von GlpD in Lipidschichten interpretiert werden. Ferner zeigt dieses Experiment, dass eine direkte Protein- 42
54 Ergebnisse Lipid-Interaktion zwischen GlpD und E. coli Phospholipiden stattfindet. 33 surface pressure (mn/m) tim e (m in) TLE Abbildung 13: Interaktion zwischen GlpD und TLE-Monolayer. Nach Injektion von 3 µg GlpD unter einen TLE-Monolayer mit einem initialen Oberflächendruck von 27 mn/m wurde die Druckänderung über die Zeit verfolgt. Lipidspezifität der Insertion von GlpD Experimente mit reinen Phospholipid-Monolayern geben Aufschluss darüber, ob die Insertion von GlpD von bestimmten Lipidtypen abhängt. Um den Einfluss verschiedener Lipid- Kopfgruppen zu testen, wurden artifizielle Phospholipide mit derselben Fettsäurezusammensetzung und unterschiedlichen Kopfgruppen eingesetzt. 39 surface pressure (mn/m) CL DOPG PE tim e (m in) Abbildung 14: Vergleich von Insertionsprofilen bei unterschiedlichen Monolayern. Es wurden Monolayer aus reinem DOPE, DOPG bzw. Rinderherz-Cardiolipin (CL) auf eine Pufferoberfläche mit einem Oberflächendruck von 27 mn/m aufgesprüht. Die durch GlpD induzierte Druckänderung wurde aufgezeichnet. 43
55 Ergebnisse Abbildung 14 zeigt, dass GlpD eine dreifach höhere Druckänderung in CL-Monolayern und eine 2.2-fach höhere Druckänderung in DOPG-Monolayern als in DOPE-Monolayern bewirkt. Folglich inserierten mehr GlpD-Moleküle in anionische Phospholipide als in zwitterionische. Um die penetrative Kraft von GlpD zu untersuchen, wurde in Abhängigkeit des Anfangsdrucks die Insertion von GlpD in DOPE-, DOPC-, DOPG- und CL-Monolayern gemessen. Ein höherer Anfangsdruck korreliert mit einer dichteren Packung der Lipide und einer reduzierten Insertion von Proteinen (Demel, 1994). surface pressure increase (mn/m) Cardiolipin DOPG DOPE DOPC initial surface pressure (mn/m) Abbildung 15: Penetrative Kraft von GlpD bei unterschiedlichen Monolayern. Die Druckänderung nach Injektion von GlpD wurde als Funktion des Anfangsdruckes gemessen. Monolayer aus DOPC (offene Raute), DOPE (offenes Dreieck), DOPG (gefüllter Kreis) und Cardiolipin aus Rinderherz (gefülltes Quadrat) wurden eingesetzt. Abbildung 15 zeigt, dass bei allen vier Lipiden eine deutliche Druckänderung bewirkt wird. Unabhängig vom Anfangsdruck induzierte GlpD bei negativ geladenen Monolayern aus DOPG bzw. Cardiolipin eine deutlich höhere Druckänderung als bei den ungeladenen Monolayern aus DOPC bzw. DOPE (Abb. 15). Folglich inseriert GlpD in anionische Lipide stärker als in ungeladene. Ein Maß für die penetrative Kraft eines Proteins ist der maximale Initialdruck π max, nämlich der höchstmögliche Anfangsdruck, bei dem das Protein eine Druckänderung bewirkt. Der maximale Anfangsdruck für CL wurde mit 38.5 mn/m, für DOPG mit 37 mn/m, für DOPE mit 33.5 mn/m und für DOPC mit 30 mn/m extrapoliert. Obwohl sowohl DOPE als auch DOPC zwitterionische Lipide sind, ist die penetrative Kraft 44
56 Ergebnisse von GlpD bei DOPE-Monolayern deutlich stärker als bei DOPC-Monolayern. Da PC in Bakterien nicht vorkommt, PE dagegen 70% der Phospholipide ausmacht, könnte die beobachtete Diskrepanz auf der Lipidspezifität von GlpD beruhen. Monolayer-Experimente, die bei hohen Anfangsdrücken durchgeführt werden, sind ein verlässliches Modellsystem, um Proteininsertion zu untersuchen (Übersicht in Demel, 1994). Für biologische Membranen liegt der Druck zwischen mn/m (Demel et al., 1975). Werden in vitro in diesem physiologisch relevanten Bereich Druckänderungen durch ein Protein induziert, so lässt dies darauf schließen, dass es auch in vivo die jeweiligen Lipidschichten penetriert. Insertion von GlpD in gemischte Phospholipid-Monolayer Um zu testen, ob anionische Lipide die Membraninsertion von GlpD begünstigen, wurden Experimente mit gemischten Phospholipiden durchgeführt. Bei einem konstanten Anfangsdruck von 30 mn/m wurde die induzierte Druckänderung von GlpD in DOPE-Monolayern mit unterschiedlichem DOPG-Gehalt gemessen. Abbildung 16 demonstriert eine lineare Abhängigkeit zwischen dem DOPG-Gehalt und der Druckdifferenz. Je größer der Anteil an anionischen DOPG war, desto stärkere Druckänderungen wurden gemessen. Insertion von GlpD findet folglich verstärkt in Anwesenheit von anionischen Lipiden statt. surface pressure increase (mn/m) mole % DOPG Abbildung 16: Insertion von GlpD als Funktion des prozentualen DOPG-Anteils in einem DOPE- Monolayer. GlpD wurde unter gemischte Monolayer aus DOPE und DOPG bei einem Anfangsdruck von 30 mn/m injiziert. 45
57 Ergebnisse Insertion von GlpD und Punktmutanten in TLE-Monolayer Apolipoproteine binden Phospholipide, indem sie eine basisch amphiphile α-helix exponieren (Segrest et al., 1992; Segrest et al., 1998). Die Helical-Wheel-Projektion der Aminosäuren ähnelt den helicalen Strukturen von Apolipoprotein A Klasse I- Molekülen. Bei beiden Helices taucht an den Übergängen zwischen hydrophober und hydrophiler Hälfte jeweils eine negativ geladene Aminosäure auf (Abb. 11). Um zu testen, ob die α-helix, die an die CaM-Oberfläche bindet, an der Membraninsertion beteiligt ist, wurden die Punktmutanten unter TLE-Monolayer injiziert. Die jeweils induzierte Druckänderung wurde mit der des Wildtyps verglichen. Abbildung 17A vergleicht den zeitlichen Verlauf der Druckänderung, nachdem Wildtyp- bzw. mutiertes GlpD injiziert wurde. Sowohl die Kinetik als auch die maximale Druckänderung unterscheiden sich deutlich. Betrachtet man die Druckänderung in Abhängigkeit des Anfangsdrucks (Abb. 17B) wird klar, dass unabhängig vom Anfangsdruck die Insertionskraft der Punktmutanten deutlich geringer ist als die des Wildtyps. Die geringste Penetrationskraft wurde bei den Punktmutanten R359E (π i = 31.3 mn/m) und K360E (π i =30.2) beobachtet. Die Doppelmutante AH362/4DE ist im Vergleich zum Wildtyp bezüglich ihrer Fähigkeit, Lipide zu penetrieren, dagegen nur geringfügig geschwächt (π i =33.0). Diese Experimente bestätigen die These, dass die basisch amphiphile α-helix für die Protein-Lipid-Bindung entscheidend ist. surface pressure (mn/m) A wt -GlpD AH362/4DE K360E R359E surface pressure increase (mn/m) B wt-glpd AH362/4DE K360E R359E 25 27, ,5 35 time (min) initial surface pressure (m N/m) Abbildung 17: Insertion von Wildtyp- und mutiertem GlpD in TLE-Monolayer A: Zeitverlauf der Insertion von Wildtyp- und mutierten GlpD-Proteinen in TLE-Monolayer mit einem Oberflächendruck von 27 mn/m. B: Insertion von Wildtyp und mutierten Proteinen in TLE-Monolayer als Funktion des initialen Oberflächendruck. 46
58 Ergebnisse Insertion von wt-glpd und Punktmutanten in PG- bzw. PE-Monolayer Aufgrund der bisherigen Datenlage wird vermutet, dass die basisch amphiphile α-helix von GlpD anionische Phospholipide penetriert. Diese These wurde mit folgenden Experimenten geprüft. DOPG- bzw. DOPE-Monolayer wurden mit unterschiedlichem Anfangsdruck auf die Pufferoberfläche gesprüht. Nach Injektion von Wildtyp-GlpD bzw. Punktmutanten-GlpD wurde die induzierte Druckänderung gemessen und verglichen. Abbildung 18 stellt die gemessenen Druckdifferenzen als Funktion des Anfangsdrucks dar. Im Vergleich zum Wildtyp ist die penetrative Kraft der Punktmutanten in DOPG-Monolayer deutlich geringer (Abb. 18A). Dagegen ergaben Messungen mit DOPE-Monolayern keine signifikanten Unterschiede zwischen Wildtyp und den Mutanten (Abb. 18B). Dies beweist, dass die α-helix an der Insertion in anionische Phospholipide beteiligt ist. Vergleicht man die maximalen Initialdrucke π max der einzelnen Mutanten, so zeigt die Doppelmutante AH362/4DE (π max =35.5 mn/m) das größte Potential, in DOPG-Monolayer zu inserieren. Die beiden Mutanten R359E und K360E sind bei einem Anfangsdruck von 31.5 mn/m nicht mehr fähig, eine Druckänderung zu bewirken. Folglich ist bei der Insertion in Lipide weniger die Nettoladung der Helix als vielmehr die Ladungsverteilung in diesem Bereich ausschlaggebend. A B surface pressure increase (mn/m) wt-glpd AH362/4DE R359E K360E 25 27, ,5 35 initial surface pressure increase (mn/m) surface pressure increase (mn/m) 6 wt-glpd 5 AH362/4DE R359E 4 K360E , ,5 35 initial surface pressure (m N/m ) Abbildung 18: Lipidspezifische Insertion von wt-glpd und Punktmutanten. Die Änderung des Oberflächendrucks nach Injektion von Wildtyp- (offene Raute), AH362/4DE (gefülltes Quadrat), R359E (gefülltes Dreieck) und K360E (gefüllter Kreis) unter A: DOPG-Monolayer bzw. B: DOPE-Monolayern ist als Funktion des initialen Oberflächendrucks dargestellt. 47
59 Ergebnisse GlpD-Bindung an TLE-Bilayer Nachdem im Monolayer-System eine direkte GlpD-Lipid Interaktion gezeigt und charakterisiert wurde, wurde diese Interaktion im Bilayer-System weiter analysiert. In Vesikelbindungsexperimenten wurde gereinigtes GlpD mit LUVETs ( Large unilamellar vesicles prepared by Extrusion ) aus TLE für zwei Stunden vorinkubiert. Anschließend wurden die Liposomen durch Ultrazentrifugation abzentrifugiert. In Abwesenheit von Liposomen wurde GlpD quantitativ im Überstand nachgewiesen (Abb.19, Spuren 1+2). In einem weiteren Kontrollexperiment wurden TLE-Liposomen ohne GlpD abzentrifugiert. Die Bestimmung des Lipidgehalts von Überstand und Pellet ergab, dass die Liposomen unter diesen Bedingungen quantitativ sedimentierten (Daten nicht gezeigt). Wurde dagegen GlpD nach Vorinkubation mit TLE-Liposomen abzentrifugiert, so sedimentierte ein beträchtlicher Teil von GlpD mit den Liposomen (Abb. 19, Spur 4), was auf eine Bindung von GlpD an die Membranen schließen lässt. Abbildung 19: Liposomenbindungsstudien mit gereinigtem GlpD und TLE-Liposomen. Proben mit gereinigtem GlpD (250 ng) wurden ohne (Spur1+2) und mit 500 nmol LUVETs (Spur 3+4) nach zweistündiger Vorinkubation abzentrifugiert. Die Überstände (Spuren 1+3) und die Pellets (Spuren 2+4) der abzentrifugierten Ansätze wurden durch 10 % SDS-PAGE und Western Blot mit anschließender Immunfärbung mit anti-glpd Antikörper analysiert. Liposomen-Bindungsspezifität von GlpD Um die Lipidspezifität der GlpD-Assoziation an Membranen zu untersuchen, wurde gereinigtes GlpD einerseits mit neutralen LUVETs, die 60% PE und 40% PC enthielten und andererseits mit negativ geladenen LUVETs, die aus 100% PG bestanden, inkubiert. Da PE ein Nicht-Bilayer-Lipid ist, konnten keine reinen PE-Liposomen hergestellt werden. Der Phospholipidgehalt der eingesetzten Liposomen lag bei 200 nmol, die Proteinmenge variierte 48
Entwicklungs /gewebespezifische Genexpression. Coexpression funktional überlappender Gene
 Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Coexpression funktional überlappender Gene Positive Genregulation Negative Genregulation cis /trans Regulation
Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Coexpression funktional überlappender Gene Positive Genregulation Negative Genregulation cis /trans Regulation
Entwicklungs /gewebespezifische Genexpression
 Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Positive Genregulation Negative Genregulation cis /trans Regulation 1. Auf welchen Ebenen kann Genregulation
Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Positive Genregulation Negative Genregulation cis /trans Regulation 1. Auf welchen Ebenen kann Genregulation
Abbildungsverzeichnis. Tabellenverzeichnis. Abkürzungsverzeichnis. 1 Einleitung 1
 Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis VI Tabellenverzeichnis VIII Abkürzungsverzeichnis IX 1 Einleitung 1 1.1 Wasser, ein limitierender Faktor für die Landwirtschaft 1 1.2 Anpassungen
Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis VI Tabellenverzeichnis VIII Abkürzungsverzeichnis IX 1 Einleitung 1 1.1 Wasser, ein limitierender Faktor für die Landwirtschaft 1 1.2 Anpassungen
Übung 11 Genregulation bei Prokaryoten
 Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA
 Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu
Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016
 Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 4 (20.06. 24.06.) Regulation der Transkription II, Translation
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 4 (20.06. 24.06.) Regulation der Transkription II, Translation
Materialien und Methoden...16
 Inhaltsverzeichnis I. Einleitung...1 1. Mitochondrien - Energielieferanten der eukaryontischen Zelle... 1 2. Die mitochondriale DNA-Replikation... 5 2.1. Transkriptionsabhängige mtdna-replikation... 5
Inhaltsverzeichnis I. Einleitung...1 1. Mitochondrien - Energielieferanten der eukaryontischen Zelle... 1 2. Die mitochondriale DNA-Replikation... 5 2.1. Transkriptionsabhängige mtdna-replikation... 5
LIPIDE. -Biosynthese:
 -Biosynthese: LIPIDE -Fettsäuren -Fettsäuresynthese: Acyl Carrier Protein, Desaturation -physiologische Bedeutung ungesättigter Fettsäuren -essentielle ungesättigte Fettsäuren -Glycerolipide: -Phosphatidatsynthese:
-Biosynthese: LIPIDE -Fettsäuren -Fettsäuresynthese: Acyl Carrier Protein, Desaturation -physiologische Bedeutung ungesättigter Fettsäuren -essentielle ungesättigte Fettsäuren -Glycerolipide: -Phosphatidatsynthese:
Untersuchungen zur Wirkung von Pasteurella multocida Toxin
 Institut fiir Experimentelle und Klinische Pharmakologie und Toxikologie der Albert-Ludwigs-Universitat Freiburg Untersuchungen zur Wirkung von Pasteurella multocida Toxin Dissertation zur Erlangung des
Institut fiir Experimentelle und Klinische Pharmakologie und Toxikologie der Albert-Ludwigs-Universitat Freiburg Untersuchungen zur Wirkung von Pasteurella multocida Toxin Dissertation zur Erlangung des
6. Induktion der Galactosidase von Escherichia coli
 Johannes Gutenberg-Universität Mainz Institut für Mikrobiologie und Weinforschung FI-Übung: Identifizierung, Wachstum und Regulation (WS 2004/05) Sebastian Lux Datum: 19.1.2005 6. Induktion der Galactosidase
Johannes Gutenberg-Universität Mainz Institut für Mikrobiologie und Weinforschung FI-Übung: Identifizierung, Wachstum und Regulation (WS 2004/05) Sebastian Lux Datum: 19.1.2005 6. Induktion der Galactosidase
Membranen. U. Albrecht
 Membranen Struktur einer Plasmamembran Moleküle gegeneinander beweglich -> flüssiger Charakter Fluidität abhängig von 1) Lipidzusammensetzung (gesättigt/ungesättigt) 2) Umgebungstemperatur Biologische
Membranen Struktur einer Plasmamembran Moleküle gegeneinander beweglich -> flüssiger Charakter Fluidität abhängig von 1) Lipidzusammensetzung (gesättigt/ungesättigt) 2) Umgebungstemperatur Biologische
Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen. Abb. aus Stryer (5th Ed.)
 Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen Die verschiedenen Ribosomen-Komplexe können im Elektronenmikroskop beobachtet werden Durch Röntgenkristallographie wurden
Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen Die verschiedenen Ribosomen-Komplexe können im Elektronenmikroskop beobachtet werden Durch Röntgenkristallographie wurden
Biochemie und. chemischer Mechanismus
 Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München Biochemie und chemischer Mechanismus der Thiomethylierung von RNA Dorothea Maria
Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München Biochemie und chemischer Mechanismus der Thiomethylierung von RNA Dorothea Maria
1 Einleitung Material und Methoden
 1 Einleitung 1 1.1 P-Typ ATPasen 2 1.1.1 Struktur und Funktion von P-Typ ATPasen 4 1.1.2 CPx-ATPasen 7 1.1.2.1 Nukleotidbindungs-/Phosphorylierungsdomäne 8 1.1.2.2 Aktuatordomäne 13 1.1.2.3 Cytoplasmatische
1 Einleitung 1 1.1 P-Typ ATPasen 2 1.1.1 Struktur und Funktion von P-Typ ATPasen 4 1.1.2 CPx-ATPasen 7 1.1.2.1 Nukleotidbindungs-/Phosphorylierungsdomäne 8 1.1.2.2 Aktuatordomäne 13 1.1.2.3 Cytoplasmatische
Membranen (3a) Membrane
 Membranen (3a) Biochemie Membrane Funktionen Abgrenzung der Zelle nach außen hin schaffen geeignete Voraussetzungen für einzelne enzymatische Reaktionen Signalübertragung tragen Signalstrukturen für die
Membranen (3a) Biochemie Membrane Funktionen Abgrenzung der Zelle nach außen hin schaffen geeignete Voraussetzungen für einzelne enzymatische Reaktionen Signalübertragung tragen Signalstrukturen für die
Charakterisierung der PII-P Phosphatase in Cyanobakterien
 Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Praktikum Biochemie B.Sc. Water Science WS Enzymregulation. Marinja Niggemann, Denise Schäfer
 Praktikum Biochemie B.Sc. Water Science WS 2011 Enzymregulation Marinja Niggemann, Denise Schäfer Regulatorische Strategien 1. Allosterische Wechselwirkung 2. Proteolytische Aktivierung 3. Kovalente Modifikation
Praktikum Biochemie B.Sc. Water Science WS 2011 Enzymregulation Marinja Niggemann, Denise Schäfer Regulatorische Strategien 1. Allosterische Wechselwirkung 2. Proteolytische Aktivierung 3. Kovalente Modifikation
Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt. Abb. aus Stryer (5th Ed.)
 Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt Die Initiation der Translation bei Eukaryoten Der eukaryotische Initiationskomplex erkennt zuerst das 5 -cap der mrna und
Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt Die Initiation der Translation bei Eukaryoten Der eukaryotische Initiationskomplex erkennt zuerst das 5 -cap der mrna und
2.1 Das prä-mrna Spleißen 3
 I 1. Zusammenfassung 1 2. Einleitung 3 2.1 Das prä-mrna Spleißen 3 2.1.1 Die Konsensussequenzen zweier Klassen von prä-mrna Introns 3 2.1.2 Die katalytischen Transesterifizierungsreaktionen 5 2.2 Das U2-abhängige
I 1. Zusammenfassung 1 2. Einleitung 3 2.1 Das prä-mrna Spleißen 3 2.1.1 Die Konsensussequenzen zweier Klassen von prä-mrna Introns 3 2.1.2 Die katalytischen Transesterifizierungsreaktionen 5 2.2 Das U2-abhängige
Induktion der β-galaktosidase von Escherichia coli
 Induktion der β-galaktosidase von Escherichia coli 1. Einleitung Das Bakterium Escherichia coli ist in der Lage verschiedene Substrate für seinen Stoffwechsel zu nutzen. Neben Glucose und Acetat kann es
Induktion der β-galaktosidase von Escherichia coli 1. Einleitung Das Bakterium Escherichia coli ist in der Lage verschiedene Substrate für seinen Stoffwechsel zu nutzen. Neben Glucose und Acetat kann es
1. Welche Auswirkungen auf die Expression des lac-operons haben die folgenden Mutationen:
 Übung 10 1. Welche Auswirkungen auf die Expression des lac-operons haben die folgenden Mutationen: a. Eine Mutation, die zur Expression eines Repressors führt, der nicht mehr an den Operator binden kann.
Übung 10 1. Welche Auswirkungen auf die Expression des lac-operons haben die folgenden Mutationen: a. Eine Mutation, die zur Expression eines Repressors führt, der nicht mehr an den Operator binden kann.
Molekulare Klonierung Praktikum
 Molekulare Klonierung Praktikum 2013 Praktikumsaufgabe Ligation 1. Zusammensetzung des Ligationsreaktions-ansatzes : 1 l Vektor DNA 1 l Insert DNA 2 l 10X Ligase Puffer 15 l destillertes Wasser 1 l Ligase
Molekulare Klonierung Praktikum 2013 Praktikumsaufgabe Ligation 1. Zusammensetzung des Ligationsreaktions-ansatzes : 1 l Vektor DNA 1 l Insert DNA 2 l 10X Ligase Puffer 15 l destillertes Wasser 1 l Ligase
Zusammenfassung. Abkürzungsverzeichnis
 Zusammenfassung Abkürzungsverzeichnis I IX 1. Einleitung 1 1.1. Glutamatrezeptoren.............................. 1 1.1.1. Modularer Aufbau von ionotropen Glutamatrezeptoren....... 3 1.1.2. AMPA-Rezeptoren...........................
Zusammenfassung Abkürzungsverzeichnis I IX 1. Einleitung 1 1.1. Glutamatrezeptoren.............................. 1 1.1.1. Modularer Aufbau von ionotropen Glutamatrezeptoren....... 3 1.1.2. AMPA-Rezeptoren...........................
Abbildungs- und Tabellenverzeichnis Abkürzungsverzeichnis Genbezeichnung. 1. Einleitung 1
 Abbildungs- und Tabellenverzeichnis Abkürzungsverzeichnis Genbezeichnung VI VIII X 1. Einleitung 1 1.1 Das Plastom 1 1.2 Transkription in Chloroplasten 4 1.2.1 Die plastidäre Transkriptionsmaschinerie
Abbildungs- und Tabellenverzeichnis Abkürzungsverzeichnis Genbezeichnung VI VIII X 1. Einleitung 1 1.1 Das Plastom 1 1.2 Transkription in Chloroplasten 4 1.2.1 Die plastidäre Transkriptionsmaschinerie
Gluconeognese Neusynthese von Glucose aus Pyruvat
 Gluconeognese Neusynthese von Glucose aus Pyruvat Warum notwendig? Das Gehirn ist auf eine konstante Versorgung mit Glucose angewiesen. Eine Unterzuckerung (< 3 4 mmol/l) führt unweigerlich zur Bewußtlosigkeit
Gluconeognese Neusynthese von Glucose aus Pyruvat Warum notwendig? Das Gehirn ist auf eine konstante Versorgung mit Glucose angewiesen. Eine Unterzuckerung (< 3 4 mmol/l) führt unweigerlich zur Bewußtlosigkeit
Übung 11 Genregulation bei Prokaryoten
 Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Regulation der Expression, Funktion und Internalisierung von muscarinischen Acetylcholinrezeptoren
 00G Regulation der Expression, Funktion und Internalisierung von muscarinischen Acetylcholinrezeptoren INAUGURAL-DISSERTATION zur Erlangung der Doktorwürde im Fachbereich Pharmazie der Freien Universität
00G Regulation der Expression, Funktion und Internalisierung von muscarinischen Acetylcholinrezeptoren INAUGURAL-DISSERTATION zur Erlangung der Doktorwürde im Fachbereich Pharmazie der Freien Universität
Neue Enzyme für ein altes Organeil: Kryptische peroxisomale Lokalisationssignale in Pilzen. Dissertation
 Neue Enzyme für ein altes Organeil: Kryptische peroxisomale Lokalisationssignale in Pilzen Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften (Dr. rer. nat.) Dem Fachbereich Biologie der
Neue Enzyme für ein altes Organeil: Kryptische peroxisomale Lokalisationssignale in Pilzen Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften (Dr. rer. nat.) Dem Fachbereich Biologie der
Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen
 Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. des Fachbereichs Biologie, Chemie, Pharmazie der Freien Universität
Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. des Fachbereichs Biologie, Chemie, Pharmazie der Freien Universität
Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle. in bioinformatisch identifizierten,
 Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle in bioinformatisch identifizierten, Pathogen-responsiven c/s-elementen aus Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften
Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle in bioinformatisch identifizierten, Pathogen-responsiven c/s-elementen aus Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften
Abbildung 1: Schematische Darstellung der Domänenorganisation von Survivin
 Abbildung 1: Schematische Darstellung der Domänenorganisation von Survivin Survivin weist eine N-terminale BIR-Domäne (grün) und eine C-terminale α-helix (blau) auf. Das intrinsische nuclear export signal
Abbildung 1: Schematische Darstellung der Domänenorganisation von Survivin Survivin weist eine N-terminale BIR-Domäne (grün) und eine C-terminale α-helix (blau) auf. Das intrinsische nuclear export signal
3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung
 3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung Transkriptionsfaktoren erfahren oft eine posttranslationelle Modifikation in Form von Phosphorylierung und werden dadurch in ihrer Aktivität
3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung Transkriptionsfaktoren erfahren oft eine posttranslationelle Modifikation in Form von Phosphorylierung und werden dadurch in ihrer Aktivität
Wirkungsmechanismen regulatorischer Enzyme
 Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
1. Einleitung Biologische Membranen...1
 I Inhaltsverzeichnis 1. Einleitung... 1 1.1 Biologische Membranen...1 1.1.1 Aufbau der Biomembran...1 1.1.2 Funktion der Plasmamembran...3 1.1.3 Membranlipide...4 1.1.4 Cholesterin als Membranbestandteil...5
I Inhaltsverzeichnis 1. Einleitung... 1 1.1 Biologische Membranen...1 1.1.1 Aufbau der Biomembran...1 1.1.2 Funktion der Plasmamembran...3 1.1.3 Membranlipide...4 1.1.4 Cholesterin als Membranbestandteil...5
TEV Protease, rekombinant (Kat.-Nr )
 GEBRAUCHSANLEITUNG TEV Protease, rekombinant (Kat.-Nr. 36403) SERVA Electrophoresis GmbH - Carl-Benz-Str. 7-69115 Heidelberg Phone +49-6221-138400, Fax +49-6221-1384010 e-mail: info@serva.de -http://www.serva.de
GEBRAUCHSANLEITUNG TEV Protease, rekombinant (Kat.-Nr. 36403) SERVA Electrophoresis GmbH - Carl-Benz-Str. 7-69115 Heidelberg Phone +49-6221-138400, Fax +49-6221-1384010 e-mail: info@serva.de -http://www.serva.de
The Arabidopsis F-box protein TIR1 is an auxin receptor. Von Stefan Kepinski & Ottoline Leyser
 The Arabidopsis F-box protein TIR1 is an auxin receptor Von Stefan Kepinski & Ottoline Leyser Bekanntes Modell Was war bekannt? In der Zwischenzeit gefunden: - ABP1 kann große Mengen Auxin binden und ist
The Arabidopsis F-box protein TIR1 is an auxin receptor Von Stefan Kepinski & Ottoline Leyser Bekanntes Modell Was war bekannt? In der Zwischenzeit gefunden: - ABP1 kann große Mengen Auxin binden und ist
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016
 Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 2 (06.06. 10.06.) DNA-Schäden, Mutationen und Reparatur 1.
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 2 (06.06. 10.06.) DNA-Schäden, Mutationen und Reparatur 1.
Untersuchungen zur Funktion von Stärke-Synthasen in der Kartoffel (Solanum tuberosum L.)
 Untersuchungen zur Funktion von Stärke-Synthasen in der Kartoffel (Solanum tuberosum L.) Inaugural-Dissertation Erlangung der Doktorwürde im Fachbereich Biologie der Freien Univeisität Berlin vorgelegt
Untersuchungen zur Funktion von Stärke-Synthasen in der Kartoffel (Solanum tuberosum L.) Inaugural-Dissertation Erlangung der Doktorwürde im Fachbereich Biologie der Freien Univeisität Berlin vorgelegt
Tyrosinkinase- Rezeptoren
 Tyrosinkinase- Rezeptoren für bestimmte Hormone gibt es integrale Membranproteine als Rezeptoren Aufbau und Signaltransduktionsweg unterscheiden sich von denen der G- Protein- gekoppelten Rezeptoren Polypeptide
Tyrosinkinase- Rezeptoren für bestimmte Hormone gibt es integrale Membranproteine als Rezeptoren Aufbau und Signaltransduktionsweg unterscheiden sich von denen der G- Protein- gekoppelten Rezeptoren Polypeptide
Protokoll Versuch A2 Expressionstests von D1D2 in verschiedenen E. coli-stämmen
 Protokoll Versuch A2 Expressionstests von D1D2 in verschiedenen E. coli-stämmen Gruppe 8 Susanne Duncker und Friedrich Hahn Einleitung und Aufgabenstellung In Versuch A1 stellten wir einen das Gen für
Protokoll Versuch A2 Expressionstests von D1D2 in verschiedenen E. coli-stämmen Gruppe 8 Susanne Duncker und Friedrich Hahn Einleitung und Aufgabenstellung In Versuch A1 stellten wir einen das Gen für
Pharmakologische Untersuchungen zur Neuropeptid Y Yi-, Y 2 - und Ys-Rezeptorselektivität peptidischer und nichtpeptidischer Liganden:
 o Pharmakologische Untersuchungen zur Neuropeptid Y Yi-, Y 2 - und Ys-Rezeptorselektivität peptidischer und nichtpeptidischer Liganden: Klonierung und funktioneile Expression des humanen Ys-Rezeptors in
o Pharmakologische Untersuchungen zur Neuropeptid Y Yi-, Y 2 - und Ys-Rezeptorselektivität peptidischer und nichtpeptidischer Liganden: Klonierung und funktioneile Expression des humanen Ys-Rezeptors in
https://cuvillier.de/de/shop/publications/2978
 Andreas Christmann (Autor) Zur bakteriellen Oberflächenpräsentation kombinatorischer Peptidbibliotheken und deren Anwendung auf Fragestellungen der Immunologie, Molekularbiologie und der Molekualren Medizin
Andreas Christmann (Autor) Zur bakteriellen Oberflächenpräsentation kombinatorischer Peptidbibliotheken und deren Anwendung auf Fragestellungen der Immunologie, Molekularbiologie und der Molekualren Medizin
Herstellung und Charakterisierung von zellpermeablem Interferon-α des Waldmurmeltieres (Marmota monax)
 Aus der Abteilung Innere Medizin II der Medizinischen Universitätsklinik der Albert-Ludwigs-Universität Freiburg im Breisgau Herstellung und Charakterisierung von zellpermeablem Interferon-α des Waldmurmeltieres
Aus der Abteilung Innere Medizin II der Medizinischen Universitätsklinik der Albert-Ludwigs-Universität Freiburg im Breisgau Herstellung und Charakterisierung von zellpermeablem Interferon-α des Waldmurmeltieres
Molekularbiologische und naturstoffchemische Untersuchungen ausgewählter Bakterienstämme
 Molekularbiologische und naturstoffchemische Untersuchungen ausgewählter Bakterienstämme - Dissertation" zur Erlangung des Doktorgrades (Dr. rer. nat.) ' der. Mathematisch-Naturwissenschaftlichen Fakultät
Molekularbiologische und naturstoffchemische Untersuchungen ausgewählter Bakterienstämme - Dissertation" zur Erlangung des Doktorgrades (Dr. rer. nat.) ' der. Mathematisch-Naturwissenschaftlichen Fakultät
12. Biopolymere. daher Namensunterschied nur durch die Zusätze D (rechtsdrehend) und L (linksdrehend)
 12. Biopolymere 12.1 Die wichtigsten Bausteine a) Zucker Zucker C(H 2 O) n dienen zb als Energiespeicher (Stärke), in der Zellerkennung (Blutgruppen) und als Bausteine der Nukleinsäuren es gibt viele Isomere,
12. Biopolymere 12.1 Die wichtigsten Bausteine a) Zucker Zucker C(H 2 O) n dienen zb als Energiespeicher (Stärke), in der Zellerkennung (Blutgruppen) und als Bausteine der Nukleinsäuren es gibt viele Isomere,
Hochdurchsatz Generierung und Analyse von Arabidopsis thaliana-proteinen
 MAX-PLANCK-INSTITUT FÜR MOLEKULARE GENETIK Hochdurchsatz Generierung und Analyse von Arabidopsis thaliana-proteinen Dissertation zur Erlangung der Doktorwürde des Fachbereichs Biologie, Chemie, Pharmazie
MAX-PLANCK-INSTITUT FÜR MOLEKULARE GENETIK Hochdurchsatz Generierung und Analyse von Arabidopsis thaliana-proteinen Dissertation zur Erlangung der Doktorwürde des Fachbereichs Biologie, Chemie, Pharmazie
Kulturen von humanen Nabelschnur-Endothelzellen (HUVEC) produzieren konstant Endothelin-1 (ET-1) und geben dieses in das Kulturmedium ab.
 4 ERGEBNISSE 4.1 Endothelin Kulturen von humanen Nabelschnur-Endothelzellen (HUVEC) produzieren konstant Endothelin-1 (ET-1) und geben dieses in das Kulturmedium ab. 4.1.1 Dosisabhängige Herabregulation
4 ERGEBNISSE 4.1 Endothelin Kulturen von humanen Nabelschnur-Endothelzellen (HUVEC) produzieren konstant Endothelin-1 (ET-1) und geben dieses in das Kulturmedium ab. 4.1.1 Dosisabhängige Herabregulation
Sophia Buhs. Dissertation. Diplom-Biochemikerin
 Beeinflussung Phosphotyrosin-abhängiger Signalwege in humanen unter Einsatz von SH2-Domänen und Phosphatasen in Kombination mit dem TAT-Transduktionssystem Dissertation Zur Erlangung der Würde des Doktors
Beeinflussung Phosphotyrosin-abhängiger Signalwege in humanen unter Einsatz von SH2-Domänen und Phosphatasen in Kombination mit dem TAT-Transduktionssystem Dissertation Zur Erlangung der Würde des Doktors
Regulation der Glykolyse: Phosphofructokinase
 Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Jens Tilsner (Autor) Aminosäuretransport in Raps unter besonderer Berücksichtigung des Entwicklungsstadiums der Pflanze und der Stickstoffdüngung
 Jens Tilsner (Autor) Aminosäuretransport in Raps unter besonderer Berücksichtigung des Entwicklungsstadiums der Pflanze und der Stickstoffdüngung https://cuvillier.de/de/shop/publications/3133 Copyright:
Jens Tilsner (Autor) Aminosäuretransport in Raps unter besonderer Berücksichtigung des Entwicklungsstadiums der Pflanze und der Stickstoffdüngung https://cuvillier.de/de/shop/publications/3133 Copyright:
Prostaglandinstoffwechsel in Trypanosoma brucei: Klonierung und Charakterisierung der Prostaglandin F 2a Synthase und der PGD 2 induzierte Zelltod
 Prostaglandinstoffwechsel in Trypanosoma brucei: Klonierung und Charakterisierung der Prostaglandin F 2a Synthase und der PGD 2 induzierte Zelltod DISSERTATION der Fakultät für Chemie und Pharmazie der
Prostaglandinstoffwechsel in Trypanosoma brucei: Klonierung und Charakterisierung der Prostaglandin F 2a Synthase und der PGD 2 induzierte Zelltod DISSERTATION der Fakultät für Chemie und Pharmazie der
Phosphati ylethanolamine
 Phosphatidic acid Ethanolamine Phosphati ylethanolamine Phosphatidylcholine Choline Phosphate Abb. 1.1 Struktur verschiedener Phospholipide. Das Grundgerüst von Phosphoglyceriden besteht aus zwei Fettsäuren,
Phosphatidic acid Ethanolamine Phosphati ylethanolamine Phosphatidylcholine Choline Phosphate Abb. 1.1 Struktur verschiedener Phospholipide. Das Grundgerüst von Phosphoglyceriden besteht aus zwei Fettsäuren,
1. EINLEITUNG Der eukaryontische Translationsinitiationsfaktor 5A (eif5a) Der Polyaminbiosyntheseinhibitor Agmatin...
 1. EINLEITUNG... 14 1.1 Lebenszyklus der Malaria Erreger Plasmodium falciparum und Plasmodium vivax... 16 1.2 Molekulares Targeting... 18 1.2.1 Die alternative Fettesäurebiosynthese II in Plasmodium falciparum...
1. EINLEITUNG... 14 1.1 Lebenszyklus der Malaria Erreger Plasmodium falciparum und Plasmodium vivax... 16 1.2 Molekulares Targeting... 18 1.2.1 Die alternative Fettesäurebiosynthese II in Plasmodium falciparum...
Translation benötigt trnas und Ribosomen. Genetischer Code. Initiation Elongation Termination
 8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
3 Ergebnisse. 3.1 Die Speckling-Domäne von Wt1 umfaßt die Aminosäuren
 3 Ergebnisse 3.1 Die Speckling-Domäne von Wt1 umfaßt die Aminosäuren 76-120 Die Existenz verschiedener Isoformen von WT1 ist unter anderem auf die Verwendung einer für die Aminosäuren KTS kodierenden alternativen
3 Ergebnisse 3.1 Die Speckling-Domäne von Wt1 umfaßt die Aminosäuren 76-120 Die Existenz verschiedener Isoformen von WT1 ist unter anderem auf die Verwendung einer für die Aminosäuren KTS kodierenden alternativen
6.3 Phospholipide und Signaltransduktion. Allgemeines
 6.3 Phospholipide und Signaltransduktion Allgemeines Bei der Signaltransduktion, das heißt der Weiterleitung von Signalen über die Zellmembran in das Innere der Zelle, denkt man zuerst einmal vor allem
6.3 Phospholipide und Signaltransduktion Allgemeines Bei der Signaltransduktion, das heißt der Weiterleitung von Signalen über die Zellmembran in das Innere der Zelle, denkt man zuerst einmal vor allem
Glycerin Produktion am Beispiel von Saccharomyces cerevisiae
 Glycerin Produktion am Beispiel von Saccharomyces cerevisiae Inhalt Sinn und Zweck Biosynthese Glycerinphosphatdehydrogenase Überexpression des GPD1-Gens Nebenprodukte Zusammenfassung Literatur Sinn und
Glycerin Produktion am Beispiel von Saccharomyces cerevisiae Inhalt Sinn und Zweck Biosynthese Glycerinphosphatdehydrogenase Überexpression des GPD1-Gens Nebenprodukte Zusammenfassung Literatur Sinn und
6.2 Membranen Struktur der Membranen
 6.2 Membranen Struktur der Membranen Für die Integrität und Entwicklungsfähigkeit der Zellen ist eine Abgrenzung gegenüber der Umwelt essentiell. Sie wird durch eine Lipiddoppelschicht geleistet, die Plasmamembran.
6.2 Membranen Struktur der Membranen Für die Integrität und Entwicklungsfähigkeit der Zellen ist eine Abgrenzung gegenüber der Umwelt essentiell. Sie wird durch eine Lipiddoppelschicht geleistet, die Plasmamembran.
Fakten und Fragen zur Vorbereitung auf das Seminar Signaltransduktion
 Prof. Dr. KH. Friedrich, Institut für Biochemie II Fakten und Fragen zur Vorbereitung auf das Seminar Signaltransduktion Voraussetzung für einen produktiven und allseits erfreulichen Ablauf des Seminars
Prof. Dr. KH. Friedrich, Institut für Biochemie II Fakten und Fragen zur Vorbereitung auf das Seminar Signaltransduktion Voraussetzung für einen produktiven und allseits erfreulichen Ablauf des Seminars
Promotor kodierende Sequenz Terminator
 5.2 Genexpression Sequenz in eine RNA-Sequenz. Die Enzyme, die diese Reaktion katalysieren, sind die DNA-abhängigen RNA-Polymerasen. Sie bestehen aus mehreren Untereinheiten, die von den Pro- bis zu den
5.2 Genexpression Sequenz in eine RNA-Sequenz. Die Enzyme, die diese Reaktion katalysieren, sind die DNA-abhängigen RNA-Polymerasen. Sie bestehen aus mehreren Untereinheiten, die von den Pro- bis zu den
Der Einfluss von multiple antibiotic resistance (mar) Mutationen auf die Fitness fluorchinolonresistenter Mutanten von Escherichia coli.
 Der Einfluss von multiple antibiotic resistance (mar) Mutationen auf die Fitness fluorchinolonresistenter Mutanten von Escherichia coli Dissertation zur Erlangung des naturwissenschaftlichen Doktorgrades
Der Einfluss von multiple antibiotic resistance (mar) Mutationen auf die Fitness fluorchinolonresistenter Mutanten von Escherichia coli Dissertation zur Erlangung des naturwissenschaftlichen Doktorgrades
Signale und Signalwege in Zellen
 Signale und Signalwege in Zellen Zellen müssen Signale empfangen, auf sie reagieren und Signale zu anderen Zellen senden können Signalübertragungsprozesse sind biochemische (und z.t. elektrische) Prozesse
Signale und Signalwege in Zellen Zellen müssen Signale empfangen, auf sie reagieren und Signale zu anderen Zellen senden können Signalübertragungsprozesse sind biochemische (und z.t. elektrische) Prozesse
Biologie für Mediziner
 Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Fachverlag Köhler Giessen VETERINÄRMEDIZIN
 VETERINÄRMEDIZIN Kälteanpassung \bi\ Listeria monocytogenes: Klonierung, D^Ä-Sequenzanalyse und Funktionsstudien der Gene CS/JL, CS/JLB und flar aus dem Wildtypstamm Listeria monocytogenes EGD Eva-Maria
VETERINÄRMEDIZIN Kälteanpassung \bi\ Listeria monocytogenes: Klonierung, D^Ä-Sequenzanalyse und Funktionsstudien der Gene CS/JL, CS/JLB und flar aus dem Wildtypstamm Listeria monocytogenes EGD Eva-Maria
Abschlussklausur zur Vorlesung Biomoleküle II WS 2004/05
 16.02.2005 Abschlussklausur zur Vorlesung Biomoleküle II WS 2004/05 Name: Studienfach: Matrikelnummer: Fachsemester: Hinweise: 1. Bitte tragen Sie Ihren Namen, Matrikelnummer, Studienfach und Semesterzahl
16.02.2005 Abschlussklausur zur Vorlesung Biomoleküle II WS 2004/05 Name: Studienfach: Matrikelnummer: Fachsemester: Hinweise: 1. Bitte tragen Sie Ihren Namen, Matrikelnummer, Studienfach und Semesterzahl
Translation benötigt trnas und Ribosomen. Genetischer Code. Initiation Elongation Termination
 8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation Elongation Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse huma n bacter
Georg-August-Universität Göttingen
 Georg-August-Universität Göttingen Abteilung für Allgemeine Mikrobiologie Institut für Mikrobiologie und Genetik Prof. Dr. Jörg Stülke Email:jstuelk@gwdg.de http://wwwuser.gwdg.de/~genmibio/stuelke.html
Georg-August-Universität Göttingen Abteilung für Allgemeine Mikrobiologie Institut für Mikrobiologie und Genetik Prof. Dr. Jörg Stülke Email:jstuelk@gwdg.de http://wwwuser.gwdg.de/~genmibio/stuelke.html
Bestimmung des Schlüsselenzyms für den biologischen Abbau mittels der Planreal DNA-Methodik PRA-DNA
 Bestimmung des Schlüsselenzyms für den biologischen Abbau mittels der Planreal DNA-Methodik PRA-DNA Dr. Christoph Henkler, Planreal AG Der biologische Abbau von Schadstoffen erfolgt in den Zellen durch
Bestimmung des Schlüsselenzyms für den biologischen Abbau mittels der Planreal DNA-Methodik PRA-DNA Dr. Christoph Henkler, Planreal AG Der biologische Abbau von Schadstoffen erfolgt in den Zellen durch
2.1 Die Entstehung des Gehirns aus neuralen Stammzellen Transkriptionsfaktoren in der Gehirnentwicklung...16
 Inhaltsverzeichnis Danksagung...3 Inhaltsverzeichnis...5 Abkürzungverzeichnis...1 1 Zielsetzung...4 2 Einleitung...6 2.1 Die Entstehung des Gehirns aus neuralen Stammzellen...6 2.2 Radiale Gliazellen...9
Inhaltsverzeichnis Danksagung...3 Inhaltsverzeichnis...5 Abkürzungverzeichnis...1 1 Zielsetzung...4 2 Einleitung...6 2.1 Die Entstehung des Gehirns aus neuralen Stammzellen...6 2.2 Radiale Gliazellen...9
Grundlagen der Physiologie
 Grundlagen der Physiologie Abbau eines Zuckermoleküls www.icbm.de/pmbio Lebensweise eines heterotrophen Aerobiers 1 Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien Anabolismus
Grundlagen der Physiologie Abbau eines Zuckermoleküls www.icbm.de/pmbio Lebensweise eines heterotrophen Aerobiers 1 Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien Anabolismus
Eukaryontische DNA-Bindedomänen
 1. Viele eukaryotische (und auch prokaryotische) Transkriptionsfaktoren besitzen eine DNA-bindende Domäne, die an eine ganz bestimmte DNA- Sequenz binden kann. Aufgrund von Ähnlichkeiten in der Struktur
1. Viele eukaryotische (und auch prokaryotische) Transkriptionsfaktoren besitzen eine DNA-bindende Domäne, die an eine ganz bestimmte DNA- Sequenz binden kann. Aufgrund von Ähnlichkeiten in der Struktur
VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN
 INHALTSVERZEICHNIS I VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VII VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN XIII 1 EINLEITUNG 1 1.1 Osmoadaption halophiler Mikroorganismen 2 1.1.1 Salt-in-cytoplasm
INHALTSVERZEICHNIS I VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VII VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN XIII 1 EINLEITUNG 1 1.1 Osmoadaption halophiler Mikroorganismen 2 1.1.1 Salt-in-cytoplasm
Regulation der Carotinoidbiosynthese in Nostoc PCC Dissertation Zur Erlangung des Doktorgrades der Naturwissenschaften
 Regulation der Carotinoidbiosynthese in Nostoc PCC 7120 Dissertation Zur Erlangung des Doktorgrades der Naturwissenschaften vorgelegt beim Fachbereich Biowissenschaften der Johann Wolfgang Goethe-Universität
Regulation der Carotinoidbiosynthese in Nostoc PCC 7120 Dissertation Zur Erlangung des Doktorgrades der Naturwissenschaften vorgelegt beim Fachbereich Biowissenschaften der Johann Wolfgang Goethe-Universität
12. Biopolymere. Anwendungen: Sensoren, Detektoren, Displays, Komponenten in elektrischen Schlatkreisen Modellsysteme
 12. Biopolymere 12.1 Organische dünne Filme Langmuir Filme = organische Polymere auf flüssigen Oberflächen Langmuir-Blodgett Filme = organische Polymere auf festen Oberflächen Anwendungen: Sensoren, Detektoren,
12. Biopolymere 12.1 Organische dünne Filme Langmuir Filme = organische Polymere auf flüssigen Oberflächen Langmuir-Blodgett Filme = organische Polymere auf festen Oberflächen Anwendungen: Sensoren, Detektoren,
7. Arachidonsäurestoffwechsel: Prostaglandine
 7. Arachidonsäurestoffwechsel: Prostaglandine Synthese von Prostaglandin (PG) Vorstufen Arachidonsäure wird durch Hydrolyse von Phospholipiden erzeugt Cyclischer und linearer Weg des Arachidonsäurestoffwechsels
7. Arachidonsäurestoffwechsel: Prostaglandine Synthese von Prostaglandin (PG) Vorstufen Arachidonsäure wird durch Hydrolyse von Phospholipiden erzeugt Cyclischer und linearer Weg des Arachidonsäurestoffwechsels
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne
 Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Die Zelle. Membranen: Struktur und Funktion
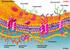 Die Zelle Membranen: Struktur und Funktion 8.4 Die Fluidität von Membranen. 8.6 Die Feinstruktur der Plasmamembran einer Tierzelle (Querschnitt). (Zum Aufbau der extrazellulären Matrix siehe auch Abbildung
Die Zelle Membranen: Struktur und Funktion 8.4 Die Fluidität von Membranen. 8.6 Die Feinstruktur der Plasmamembran einer Tierzelle (Querschnitt). (Zum Aufbau der extrazellulären Matrix siehe auch Abbildung
Genexpression und Genregulation in Prokaryoten
 Genexpression und Genregulation in Prokaryoten Genregulation: Mechanismen bakterieller Genregulation, Grundbegriffe: nicht alle Gene eines Bakteriums sind ständig aktiv deren Induktion der Expression erfolgt
Genexpression und Genregulation in Prokaryoten Genregulation: Mechanismen bakterieller Genregulation, Grundbegriffe: nicht alle Gene eines Bakteriums sind ständig aktiv deren Induktion der Expression erfolgt
Kerstin Brachhold (Autor) Das Basalapparat-Subproteom von Chlamydomonas reinhardtii
 Kerstin Brachhold (Autor) Das Basalapparat-Subproteom von Chlamydomonas reinhardtii https://cuvillier.de/de/shop/publications/1867 Copyright: Cuvillier Verlag, Inhaberin Annette Jentzsch-Cuvillier, Nonnenstieg
Kerstin Brachhold (Autor) Das Basalapparat-Subproteom von Chlamydomonas reinhardtii https://cuvillier.de/de/shop/publications/1867 Copyright: Cuvillier Verlag, Inhaberin Annette Jentzsch-Cuvillier, Nonnenstieg
- der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen
 Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Zellulärer Abbau von Proteinen in Aminosäuren:! Proteine werden in Zellen durch Proteasom-Komplexe in! einzelne Aminosäuren abgebaut.!
 Zellulärer Abbau von Proteinen in Aminosäuren: Proteine werden in Zellen durch Proteasom-Komplexe in einzelne Aminosäuren abgebaut. Abbau von Aminosäuren: Uebersicht über den Aminosäureabbau Als erster
Zellulärer Abbau von Proteinen in Aminosäuren: Proteine werden in Zellen durch Proteasom-Komplexe in einzelne Aminosäuren abgebaut. Abbau von Aminosäuren: Uebersicht über den Aminosäureabbau Als erster
Antibiotika sind oft Inhibitoren der Genexpression
 Antibiotika sind oft Inhibitoren der Genexpression Inhibitoren der Transkription: Rifampicin, Actinomycin α-amanitin Inhibitoren der Translation: Puromycin, Streptomycin, Tetracycline, Chloramphenicol
Antibiotika sind oft Inhibitoren der Genexpression Inhibitoren der Transkription: Rifampicin, Actinomycin α-amanitin Inhibitoren der Translation: Puromycin, Streptomycin, Tetracycline, Chloramphenicol
Abkürzungsverzeichnis... VII. 1. Einleitung Regulation intrazellulärer Signalwege durch camp Optogenetische Methoden...
 I Abkürzungsverzeichnis... VII 1. Einleitung... 13 1.1 Regulation intrazellulärer Signalwege durch camp... 13 1.2 Optogenetische Methoden... 15 1.3 camp-abhängige Mechanismen synaptischer Plastizität...
I Abkürzungsverzeichnis... VII 1. Einleitung... 13 1.1 Regulation intrazellulärer Signalwege durch camp... 13 1.2 Optogenetische Methoden... 15 1.3 camp-abhängige Mechanismen synaptischer Plastizität...
Z 11 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: 2) ÜBERSICHT ÜBER DEN ENERGIESTOFFWECHSEL
 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE Zusammenfassung Zusammenfassung Kapitel 11 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung.
GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE Zusammenfassung Zusammenfassung Kapitel 11 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung.
Stammzüchtung. Selektion von natürlichen Varianten. Ungerichtete genetische Veränderungen zufallsverteilte induzierte Mutagenese
 Stammzüchtung Selektion von natürlichen Varianten Ungerichtete genetische Veränderungen zufallsverteilte induzierte Mutagenese Kreuzungen genetische Rekombination Sexuelle Kreuzungen Induzierte Zellfusion
Stammzüchtung Selektion von natürlichen Varianten Ungerichtete genetische Veränderungen zufallsverteilte induzierte Mutagenese Kreuzungen genetische Rekombination Sexuelle Kreuzungen Induzierte Zellfusion
Z11 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: 2) ÜBERSICHT ÜBER DEN ENERGIESTOFFWECHSEL
 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung. Proteine aus der Nahrung werden
GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung. Proteine aus der Nahrung werden
STOFFTRANSPORT DURCH BIOMEM- BRANEN
 DIE BIOMEMBRAN Vorkommen Plasmalemma Grenzt Cytoplasma nach außen ab Tonoplast Grenzt Vakuole vom Cytoplasma ab Zellkernmembran Mitochondrienmembran Plastidenmembran ER Kompartimente Durch Zellmembran
DIE BIOMEMBRAN Vorkommen Plasmalemma Grenzt Cytoplasma nach außen ab Tonoplast Grenzt Vakuole vom Cytoplasma ab Zellkernmembran Mitochondrienmembran Plastidenmembran ER Kompartimente Durch Zellmembran
Bioinformatik I: Grundlagen der Gentechnik
 Bioinformatik I: Grundlagen der Gentechnik Dr. Maik Böhmer Institut für Biologie und Biotechnologie der Pflanzen Schlossplatz 7 Schwerpunkte: Vorlesung 1: Einführung & Enzyme der Gentechnik Vorlesung 2:
Bioinformatik I: Grundlagen der Gentechnik Dr. Maik Böhmer Institut für Biologie und Biotechnologie der Pflanzen Schlossplatz 7 Schwerpunkte: Vorlesung 1: Einführung & Enzyme der Gentechnik Vorlesung 2:
Frage 1 A: Wieviele Codone des "Universellen genetisches Codes" kodieren:
 Frage 1 A: Wieviele Codone des "Universellen genetisches Codes" kodieren: Aminosäuren Translationsstart Translationsstop? B: Welche biochemische Reaktion wird von Aminoazyl-tRNA-Synthetasen katalysiert?
Frage 1 A: Wieviele Codone des "Universellen genetisches Codes" kodieren: Aminosäuren Translationsstart Translationsstop? B: Welche biochemische Reaktion wird von Aminoazyl-tRNA-Synthetasen katalysiert?
Mikrobiologie 2. Vertretung. Prof. Dr. Nicole Frankenberg-Dinkel. Dr. Susanne Zehner. Follow us on:
 Mikrobiologie 2 Prof. Dr. Nicole Frankenberg-Dinkel Vertretung Dr. Susanne Zehner Follow us on: Transkription Kontrolle der Transkription Translation Wiederholung Transkription Erster Schritt der Expression
Mikrobiologie 2 Prof. Dr. Nicole Frankenberg-Dinkel Vertretung Dr. Susanne Zehner Follow us on: Transkription Kontrolle der Transkription Translation Wiederholung Transkription Erster Schritt der Expression
Musterlösung- Übung 9
 Teil Molekularbiologie 1. Über Mutationen a) In welchem Teil eines Operons befinden sich Mutationen, welche die Menge eines Enzyms in einem Organismus beeinflussen? Solche Mutationen befinden sich im Operator.
Teil Molekularbiologie 1. Über Mutationen a) In welchem Teil eines Operons befinden sich Mutationen, welche die Menge eines Enzyms in einem Organismus beeinflussen? Solche Mutationen befinden sich im Operator.
Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!!
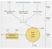 Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!! Indirekte ATP synthese! Protonen können in wässriger Lösung sehr! schnell transportiert werden.! Ionen können biologische Membranen nicht spontan
Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!! Indirekte ATP synthese! Protonen können in wässriger Lösung sehr! schnell transportiert werden.! Ionen können biologische Membranen nicht spontan
Das Rcs-System und die RcsAB-Box: Identifikation eines neuen, essentiellen Operators für die. Regulation der enterobakteriellen Kapselbiosynthese
 Das Rcs-System und die RcsAB-Box: Identifikation eines neuen, essentiellen Operators für die Regulation der enterobakteriellen Kapselbiosynthese Inaugural-Dissertation zur Erlangung der Doktorwürde vorgelegt
Das Rcs-System und die RcsAB-Box: Identifikation eines neuen, essentiellen Operators für die Regulation der enterobakteriellen Kapselbiosynthese Inaugural-Dissertation zur Erlangung der Doktorwürde vorgelegt
Hemmung der Enzym-Aktivität
 Enzym - Inhibitoren Wie wirkt Penicillin? Wie wirkt Aspirin? Welche Rolle spielt Methotrexat in der Chemotherapie? Welche Wirkstoffe werden gegen HIV entwickelt? Hemmung der Enzym-Aktivität Substrat Kompetitiver
Enzym - Inhibitoren Wie wirkt Penicillin? Wie wirkt Aspirin? Welche Rolle spielt Methotrexat in der Chemotherapie? Welche Wirkstoffe werden gegen HIV entwickelt? Hemmung der Enzym-Aktivität Substrat Kompetitiver
Strukturelle und funktionelle Analyse des fibronektinbindenden Serumopazitätsfaktors aus Streptococcus pyogenes (Gruppe A Streptokokken)
 A Strukturelle und funktionelle Analyse des fibronektinbindenden Serumopazitätsfaktors aus Streptococcus pyogenes (Gruppe A Streptokokken) Von der Naturwissenschaftlichen Fakultät der Technischen Universität
A Strukturelle und funktionelle Analyse des fibronektinbindenden Serumopazitätsfaktors aus Streptococcus pyogenes (Gruppe A Streptokokken) Von der Naturwissenschaftlichen Fakultät der Technischen Universität
Rekombinante Antikorper. fur,,lab-on-chip"
 Rekombinante Antikorper fur,,lab-on-chip" Von der Fakultat fur Lebenswissenschaften der Technischen Universitat Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades einer Doktorin der Naturwissenschaften
Rekombinante Antikorper fur,,lab-on-chip" Von der Fakultat fur Lebenswissenschaften der Technischen Universitat Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades einer Doktorin der Naturwissenschaften
Fettsäurebiosynthese
 Fettsäurebiosynthese Inhalt Fettsäuren Triacylglyceride FS-Biosynthese und einzelne Schritte Fettsäuren Lange CH-Ketten mit einer endständigen Carboxylgruppe 3 Gruppen: -> gesättigte FS -> einfach ungesättigte
Fettsäurebiosynthese Inhalt Fettsäuren Triacylglyceride FS-Biosynthese und einzelne Schritte Fettsäuren Lange CH-Ketten mit einer endständigen Carboxylgruppe 3 Gruppen: -> gesättigte FS -> einfach ungesättigte
Rekombinante Antikörper
 Frank Breitling und Stefan Dübel 2008 AGI-Information Management Consultants May be used for personal purporses only or by libraries associated to dandelon.com network. Rekombinante Antikörper Technische
Frank Breitling und Stefan Dübel 2008 AGI-Information Management Consultants May be used for personal purporses only or by libraries associated to dandelon.com network. Rekombinante Antikörper Technische
