Biochemie- Seminar 10
|
|
|
- Ruth Schäfer
- vor 8 Jahren
- Abrufe
Transkript
1 Biochemie- Seminar 10 Verweise mit [DR] bezieht sich auf Duale Reihe - Biochemie von Rassow, Hauser, Netzker und Deutzmann in 2. Auflage; [L] steht für Biochemie und Pathobiochemie von Löffler, Petrides, Heinrich in 8.Auflage. Replikation und Zellzyklus von Leif Ablauf des Zellzyklus - G 1 - Phase (Gap): Zellwachstum, Proteinsynthese, RNA-Synthese anschließend Kontrollpunkt: Zellgröße, DNA-Integrität, Substratverfügbarkeit - S- Phase (Synthese): DNA wird verdoppelt, Synthese von RNA und Proteinen - Kontrollpunkt in früher G2-Phase: Zellgröße, vollständige Replikation, DNA-Schäden G 2 - Phase (Gap): weitere Synthese; Vorbereitung auf Teilung - Mitose (M-Phase) Kontrollpunkt in später M-Phase: korrekte Aufteilung der Chromosomensätze - bei schnell wachsender Zelle dauert Zellzyklus ca. 24 Stunden o Interphase: 23 Std [Interphase = G-Phase und S-Phase] o M-Phase: 1 Std - Verlassen des Zellzyklus bedeutet Übergang von der G 1 -Phase in die G 0 - Phase (Ruhephase): o bei ausdifferenzierten Zellen o bei Entzug von Nährstoffen und Wachstumsfaktoren Regulation des Zellzyklus - inneres Uhrwerk der Zelle hilft bei Entscheidung über Checkpoint- Passage; besteht aus: o Cykline o Cyklin- abhängige Proteinkinasen (cycline-dependend kinases; cdks) o cdk- Inhibitoren (CKI; binden spezifisch an Cyclin/cdk-Komplexe) - Cycline = strukturell verwandte Proteine, deren Gehalt während Zellzyklus schwankt (durch proteolytischen Abbau); aktivieren die cdks - cdks= durch Cycline aktiviert ( Bildung von Cyclin/cdk- Komplexen); Gehalt während des Zellzyklus ist konstant Aktivität durch Phosphorylierung und Dephosphorylierung reguliert z.b.: cdk aktivierende Kinase (CAK; aus Cyclin H + cdk7 + Mat1) cdks werden durch Inhibitoren kontrolliert z.b.: p21- Familie, p16- Familie Aktivität der cdks wird über subzelluläre Lokalisation reguliert z.b.: Cyclin B/cdk1- Komplex: während Zellzyklus im Zytosol und enzymatisch inaktiv, in G 2 - Phase Phosphorylierung und Translokation in den Nucleus; ebenso Cyclin C/cdk4- Komplex Beispiele - CyclinB und CDK1 assoziieren zu MPF (M-phase promoting factor; steuert Eintritt der Zelle in die M-Phase) o CDK1-Spiegel ist konstant o CyclinB-Spiegel schwankt zyklusabhängig - 1 -
2 - RB-Protein (Retinoblastom-Protein; Wachstumsfaktor) blockiert Transkriptionsfaktor E2F o Phosphorylierung von RB-Prot. E2F kann Gene aktivieren deren Produkte den Zellzyklus stimulieren Apoptose - [L] S. 227 Abb gezielte Einzel- Zell- Eliminierung durch biochemisches Programm; induzierbar, energieabhängig - Vorgänge: o Fragmentierung des ER + Golgi- Apparat o Chromosomenkondensation o Abbau der Kernhülle o Cyclinabbau - katalysiert durch proteolytische Enzyme: Caspasen o Cysteinproteasen, spalten Proteine hinter Aspartyl- Resten o liegen intrazellulär als enzymatisch inaktive Procaspasen vor; Aktivierung durch limitierte Proteolyse Effektorcaspasen spalten zelluläre Proteine (wie z.b. Proteinkinase C, Reparaturenzyme) Inaktivierung dieser Proteine aktivieren spezielle DNase: Caspase- aktivierte DNase (CAD) Initiatorcaspasen aktiviert über TNF- Rezeptorweg (extrinsisch, s.u.) oder mitochondrialen Weg (intrinsisch, s.u.) - TNF- Rezeptorweg o Bindung von Liganden an TNF- Rezeptor (z.b. TNFα- Rezeptor, Fas- Rezeptor) o Anlagerung von FADD- Adaptermolekül an Rezeptor o Anlagerung und proteolytische Aktivierung von Procaspase 8 (= Initiatorcaspase) zu Caspase 8 o Aktivierung von Effektorcaspasen Apoptose - mitochondrialer Weg o proapoptotische und antiapoptotische Proteine der Bcl-2-Familie regulieren Cytochrom c- Freisetzung aus Mitochondrien; proapoptotisches Heterodimer Bax/Bak bildet hierfür wahrscheinlich eine Pore Cytochrom c bindet an Apaf-1 (apoptotic protease-activating factor) Apaf-1 aktiviert unter ATP- Verbrauch Initiator- Procaspase 9 zu Caspase 9 Aktivierung von Effektorcaspasen Apoptose o Bim aus Cytoskelett aktiviert bei Störungen Bax/Bak o Bad wird inaktiviert durch Proteinkinase B (aktiviert durch Wachstumsfaktoren) Hemmung der proapoptotischen Wirkung o Bid wird von Caspase 8 zu tbid gespalten aktiviert Bax/Bak [Verbindung zwischen extrinsischem und intrinsischem Weg] - Bcl-2 und einige Proteine aus der Bcl-2- Familie wirken antiapoptotisch - Schicksal der Zelle wird bestimmt durch Balance der Pro- und Antiapoptotischen Proteine Verknüpfung Apoptose und Zellzyklus - Tumorsuppressor p53 bewirkt nach DNA-Schädigung einen Verbleib der Zelle in der G 1 -Phase o Zelle gewinnt Zeit, Schäden zu beheben o p53 bindet an Promotor für cdk- Inhibitor p21 WAF1 p21 WAF1 - Expression - 2 -
3 o p21waf1 hemmt Cyclin D/cdk4, Cyclin E/cdk2, Cyclin A/cdk2 Zellzyklus wird angehalten - ist Schaden zu umfangreich oder irreparabel induziert p53 die Expression von bax Apoptoseweg wird in Gang gesetzt (intrinsisch) DNA- Replikation - Replikation = Verdopplung der DNA vor Zellteilung o semikonservativ: der Doppelstrang wird aufgespalten und jeder Einzelstrang dient als Vorlage für einen neuen Strang Prinzip - Primer leitet Replikation ein [Proc.: Primase; Euc.: DNA-Polymerase α- Teilaktivität] - 3 -OH-Gruppe des neugebildeten Strangs greift nucleophil das α-phosphoratom des nächsten dntp (desoxy-nucleotidphosphat) an Synthese 5-3 ; PP i wird abgespalten DNA-Polymerasen - *Skript 2 Replikation_neu.pdf + Folie 6 - unentbehrlicher Cofaktor ist Mg ++ - einige DNA-Polymerasen haben Fähigkeit zum Korrekturlesen via Exonucleaseaktivität: falsche eingebaute Nucleotide können erkannt und unmittelbar nach Einbau wieder hydrolytisch gespalten werden - wichtiger Faktor von DNA-Polymerasen ist Prozessivität = Zahl der Nucleotide, die von Polymerase an Kette angefügt wird bevor Polymerase wieder abdissoziiert - 3 -
4 Initiation - Protein DnaA bindet an Replikationsursprung (origin of replication); öffnet den Doppelstrang o meist an Orten mit vielen A-T- Basenpaaren, da diese nur 2 Wasserstoffbindungen haben und leichter zu trennen sind [G-C- Paare haben 3 Bindungen] - DnaB und DnaC- Proteine sind Helicasen: entwindet DNA unter ATP- Verbrauch - Topoisomerase I (Einzelstrangbruch) und Topoisomerase II (Doppelstrangbruch) reduzieren physikalische Belastung auf die verwundenen DNA-Stränge - entwundene Stränge werden durch SSBs (single strand binding protein; Tetramere) stabilisiert, diese verhindern eine Reassoziation der Stränge Replikationsgabel wird geformt am Replikon (diejenige DNA- Einheit, in der die einzelnen Replikationsschritte stattfinden) Elongation - DNA- Doppelstränge verlaufen Antiparallel, DNA- Polymerasen können aber nur in Richtung synthetisieren - Führungsstrang ( leading strand ) wird kontinuierlich synthetisiert - verzögerter Strang ( lagging strand ) wird diskontinuierlich synthetisiert o Okazaki- Fragmente aus Basen werden nacheinander abgearbeitet 1. Primersynthese 2. Synthese erstes Okazaki-Fragment und zweiter Primer 3. Synthese zweites Okazaki-Fragment und dritter Primer 4. o bei Prokaryoten: DNA-Polymerase I mit Exonucleaseaktivität entfernt den RNA- Primer und füllt die entstanden Lücken auf; DNA-Ligase verknüpft neu entstandene Stränge (siehe [L] S. 233 Abb. 7.16); Polymerase-Dimer ermöglicht simultane Synthese von lagging strand und leadings strand durch Polymerase-Komplex-Bildung lagging strand bildet eine Schleife zurück zum Komplex o bei Eukaryoten: Polymerase α-primase-komplex macht Initiation der Synthese und Synthese des lagging strand ; Polymerase δ synthetisiert den leadings strand Telomerase - am 3 - Ende des lagging strand kann keine Replikation stattfinden da hier der Primer liegt, der von der Exonuclease entfernt wird DNA-Polymerase kann Lücke nicht auffüllen DNA wird mit jeder Replikation um ein Stück kleiner - Problem wird durch G-reiche Sequenzen an den Telomere (DNA-Enden) gelöst: o Telomerasen (entsprechen reversen Transcriptasen) enthalten RNA mit zur telomeren Sequenz passenden komplementären Basen (A-reich) o Nucleotide des Telomers paaren sich mit Telomerase- RNA - Telomerasen sind aktiv bei Keimbahnzellen und nach maligner Zelltransformation (so kommt es zu keiner normalen Zellalterung sondern z.b. zu neoplastischem Wachstum von Tumoren) - 4 -
5 Inhibitoren der DNA-Replikation Mitomycin - kovalente Quervernetzung der DNA verhindert Trennung der Stränge Biochemie- Seminar 10 Actinomycin - bildet Komplexe mit Guanin-Resten hemmt DNA-abhängige RNA- Biosynthese, in höheren Dosen auch DNA-Replikation Fluorouracil - irreversibler Inhibitor der Thymidylat-Synthase keine drmp-synthese keine Replikation und Reparatur Metothrexat, Aminopterin - kompetetiver Inhibitor der Dihydrofolat-Reductase keine Bildung von THF keine Nucleotid-Synthese Cisplatin - DNA-Schäden Apoptose virale Oncogene - Tumorviren können Gene der Wirtszelle in eigenes Genom integrieren - durch Rekombination und Mutationen entstehen daraus Onkogene - Onkogenprodukte können auf allen Ebenen der Signaltransduktion proliferative Signale auslösen - Prototyp: v-raf, Entstehung aus humanem Protooncogen raf - virales Onkogen v-erbb produziert verkürzten Rezeptor ohne Ligandbindungsdomäne Kontrolle durch EGF (Epidermal Growth Factor) unwirksam konstitutive Aktivität - Zelltransformation durch permanente proliferative Signale retrovirale Vermehrung - durch reverse Transkriptase Einbau des Virus-Genoms ins Wirts-Genoms mittels Integrase 1. Virus-RNA wird mittels reverser Transkriptase in Virus-DNA gewandelt 2. Virus-DNA wird mittels Integrase in Wirts-DNA eingebaut 3. Transkription und Translation dieser Hybrid-DNA schafft neue Virus-Bausteine die dann neue Viruspartikel bilden - 5 -
6 DNA/Protein- Wechselwirkungen und DNA- Reparatur von Leif Wodurch wird DNA geschädigt? Welche Schäden können auftreten? Biochemie- Seminar 10 - Chemikalien und Strahlung führen zu Depurinierung (z.b. durch thermische Spaltung der N- glycosidischen Bindungen); ca Purinbasen pro Zelle in 24 Stunden - Chemikalien und Strahlung führen zu Desaminierung von Cytosin (es entsteht Uracil mutagen wg. Paarung mit Adenin und nicht Guanin) - UV-Licht führt zur Entstehung von Thymidindimeren - Schäden durch Sauerstoffradikale oxidative Schädigung - hydrolytische Angriffe - unkontrollierte Methylierung Schäden, die zum Austausch von Einzelbasen führen - veränderte Sequenz, keine Auswirkungen auf DNA-Gesamtstruktur - Transkription und Replikation unbeeinflußt - Konsequenzen für Tochtergenerationen - Bsp.: o Desaminierung von Basen (Cytosin Uracil) o Fehlpaarungen nach Replikation (Cytosin Adenin) o Fehlpaarung durch Tautomerie (Imino-Adenin paart mit Cytosin statt mit Thymin) o Strangverschiebung durch Polymerase-Slippage Schäden, die zu strukturellen Verzerrungen der DNA führen - physikalische Behinderung bei Transkription und Replikation - entstehende Probleme sind nach Reparatur aufgehoben - keine Folgen für Tochtergeneration - Bsp.: o Einführung kovalenter Bindungen zwischen den Basen (z.b. durch UV-Strahlung Thymin + Thymin wird zu Thymindimer verknüpft) o chemische Modifikation (z.b. durch Alkylierung) o Verlust von Basen (durch Depurinierung) Reparaturmechanismen - allgemeine Strategie: Erkennen des Fehlers Entfernen des Fehlers Schließen der Lücke Ligation direkte Reparatur (nur bei Prokaryoten) - durch photoaktivierte Enzyme - siehe *Skript+ 4 Reparatur.pdf Folie 14 Fehlpaarungsreparatur - verbessert Genauigkeit der Replikation um Mut S gleitet auf dem Strang (hemimethyliert) entlang und tastet auf Fehler ab, findet es einen Fehler, rekrutiert es Mut L und Mut H. - Endonuclease Mut H spaltet nun den Strang an einer GATC-Sequenz - zusammen mit Mut S, Mut L und einer Helicase II baut die Endonuklease I den defekten Strang über die Fehlpaarungsstelle hinaus ab - DNA-Polymerase III füllt wieder auf und DNA-Ligase versiegelt die Bruchstellen - 6 -
7 Basenexzisionsreparatur - Basenveränderung wird erkannt von spezifischen Proteinen, die mit DNA-Glycosylasen aktive Komplexe bilden defekte Base wird entfernt - AP-Endonuclease (apurinic bzw. apyrimidinic; trennt Ribose und Phosphat) und Phosphodiesterase entfernen zur herausgeschnittenen Base gehöriges Desoxyribosephosphat - mittels DNA-Polymerasen und DNA-Ligasen kann die Lücke gefüllt und geschlossen werden - prinzipiell gleiches Verfahren auch bei Depurinierung Nucleotid- Exzisionsreparatur - dient zur Behebung größerer Defekte - nach Erkennung des Schadens durch Multienzymkomplex wird Nucleotide langes Oligonucleotid durch Nucleaseaktivität aus geschädigtem Einzelstrang entfernt - entstandene Lücke wird mittels DNA-Polymerase und DNA-Ligase gefüllt und geschlossen Reparatur bei schweren DNA-Schäden NHEJ (non homologous end-joining) und HEJ (homologous end-joining) - zur Reparatur von Doppelstrangschäden (z.b. durch ionisierende Strahlung oder oxidative Reagenzien) - NHEJ: DNA- Enden werden einfach miteinander verknüpft; trotz Mutation an Bruchstelle anscheinend akzeptable Lösung - HEJ: das doppelt vorhandene intakte Chromosom wird als Matrize zur Reparatur des defekten Chromosoms genutzt Reparatur ohne DNA- Verlust ist so möglich wenn der DNA-Schaden zu groß ist: - p53 induziert Zellzyklus-Inhibitor Cdk-Inhibitor p21 Hemmung des Zellzyklus (Onkogen-induzierte Inaktivierung von p53 führt zu Tumor-Entstehung) Reparaturdefekte Xeroderma pigmentosum - Lichtschrumpfhaut, extreme Empfindlichkeit gegenüber UV-Licht - starke Hautschädigung, hohe Krebsanfälligkeit - Ursache: u.a. Defekt in Excinuclease (Reparatur von Thymindimeren) Fanconi-Anämie - Fehlbildungen,Leukämie - durch fehlerhafte Reparatur von Strangüberkreuzungen BRCA-2 - Brust-u. Eierstockkrebs - Reparatur durch homologe Rekombination fehlerhaft MSH2,3,6 - Dickdarmkrebs - Defekt der Fehlpaarungsreparatur - 7 -
8 Transkription und posttranskriptionelle Modifikation von Leif Grundlagen Biochemie- Seminar 10 Funktionen der RNA - RNA- Biosynthese unterscheidet sich von DNA- Biosynthese: o bei Transkription werden nur einzelne DNA- Abschnitte abgelesen o DNA- Bestand einer Zelle bleibt konstant, RNA wird immer wieder neu synthetisiert - Form der mrna dient als Matrize für Proteinbiosynthese - trna (transfer-rna) bildet den Adapter für Übersetzung des Nucleinsäuren- Codes in eine Aminosäuresequenz - rrna ist Strukturbestandteil der Ribosomen (für Proteinbiosynthese) - RNA dient in Form von Ribozymen als Katalysator - viele kleine RNA- Moleküle haben regulatorische Funktion konstitutive Transkription - Genen für Aufrechterhaltung der basalen Funktionen in Zellen werden konstant exprimiert - Gene = house keeping genes regulierte Gene - Expression variiert je nach Bedarf - durch viele intra- und extrazelluläre Faktoren beeinflusst Prinzip der Transkription - Prinzip ähnelt dem der DNA- Replikation - anders als DNA- Replikation benötigt RNA- Biosynthese keinen Primer - zuerst wird DNA geöffnet und entwunden, beide Stränge haben unterschiedliche Funktion: o o Strang der als Matrize für RNA- Synthese dient = Matrizenstrang ( antisense strand ) anderer Strang entspricht Basensequenz des RNA- Transkripts = codierender Strang ( sense strand ) - DNA-abhängige RNA-Polymerasen sind für Transkription verantwortlich, durch Rotation der DNA bedingte topographische Probleme werden von Topoisomerasen bewältigt Initiation - korrekte Startstelle für Transkription ist durch einen sog. Promotor bzw. eine Promotorregion gekennzeichnet; hierdurch wird die Transkription gesteuert - Promotorregionen auf der DNA werden auch cis- Elemente genannt, die an sie bindenden spezifischen Proteine sind trans- Elemente Elongation - Verknüpfung von Nucleosidtriphosphate (NTPs) durch RNA-Polymerasen zur RNA - chemisch entspricht Reaktionsmechanismus der RNA-Polymerasen dem der DNA- Polymerasen: o 3 -OH-Ende eines Nucleotids greift am α-phosphat des anzufügenden Ribonucleotids an Einbau in wachsende RNA- Kette o anstelle von Thymin enthält die RNA Uracil (bildet aber auch mit Adenin zwei Wasserstoffbrückenbindungen) Termination - spezifische Signale auf der DNA verhindern eine (energieträchtige) überschießende Transkription - 8 -
9 Transkription bei Prokaryoten - RNA-Polymerase (Pentamer mit Untereinheiten 2,,, ) kann RNA selbsttätig synthetisieren, benötigt aber zusätzlichen Sigmafaktor σ zum Auffinden der Promotorregion ( Initiation) - nach Auffinden lagert RNA-Polymerase erstes Nucleosidtriphosphat an (immer ATP oder GTP) Bildung des Initiationskomplexes - nach Knüpfung ca. 10 weiterer Phosphodiesterbindungen dissoziiert Sigmafaktor, da er für Elongation nicht benötigt wird - RNA-Polymerase liest DNA in Richtung ab, synthetisiert RNA in Richtung; dabei findet sog. proofreading statt - Termination findet Rho-abhängig oder Rho-unabhängig statt o ρ abhängig: Proteinfaktor Rho (ρ) bildet hexamere Struktur um entstehende RNA Abtrennung der Polymerase o ρ unabhängig: Ausbildung von Haarnadelstrukturen in der RNA durch Terminationssignale (palindromische repetitive Sequenzen ( inverted repeat )) in den Genen Transkription bei Eukaryoten - RNA-Polymerase I; im Nucleolus; transkribiert für ribosomale RNA (rrna) o α-amanitin-unempfindlich - RNA-Polymerase II; im Zellkern; transkribiert für prä-mrna/ mrna o α-amanitin-empfindlich - RNA-Polymerase III; Zellkern; transkribiert für trna o α-amanitin-wenig empfindlich (große Mengen Gift nötig) - sind ohne allgemeine Transkriptionsfaktoren nicht zur Initiation imstande RNA-Polymerase II - Gene haben bestimmte Erkennungssequenzen für Bindung von Transkriptionsfaktoren; z.b. CAAT-Box, TATA-Box o zur Bildung von Initiationskomplexen notwendig; für optimale Funktion mindestens 2-3 Elemente im Promotor enthalten - eukaryote RNA-Polymerasen können nicht von selbst an DNA binden, benötigen allgemeine Transkriptionsfaktoren (TF) bilden Initiationskomplexe aus RNA-Polymerase II, TFs und Mediator- Proteinen - erhöhte Effektivität durch multiple Initiation mehrere Polymerasen arbeiten gleichzeitig 1. Initiation beginnt mit der Anlagerung des TATA-Bindungsproteins (UE von Transkriptionsfaktor IID) an die TATA-Box [ DNA wird um 80 verbogen, Wechselwirkung zwischen AT-Basenpaaren gelockert] 2. Transkriptionsfaktor IID rekrutiert RNA-Polymerase II; Transkriptionsfaktoren IIB,E,F,H,J lagern sich an C-terminale Domäne (CTD) der RNA- Polymerase II an [C-terminale Domäne (CTD) der RNA-Polymerase II spielt eine so wichtige Rolle, dass die humane RNA-Polymerase II 52 Kopien davon enthält; besteht aus repetitiver Sequenz des Heptapeptids Tyr-Ser-Pro-Thr-Ser-Pro-Ser; wichtig weil: geht sehr spezifische Wechselwirkung mit TATA-Box-Bindeprotein ein] 3. Enhancer oder Silencer regulieren über Konformationsänderungen der DNA oder Interaktion mit den Transkriptionsfaktoren oder RNA-Polymerase II die Transkription - 9 -
10 - für Elongation sind viele Proteine des Initiationskomplexes nicht mehr nötig; nur TFIIH verbleibt wegen weiter benötigter Helicase- Aktivität 4. in jedem Heptapeptid der CTD [also 52 beim Menschen] befinden sich 2 Serinreste Phosphorylierung durch CyclinH/cdk7/Mat1-Proteinkinase 5. Verlust der Affinität von RNA-Polymerase II zu Initiatorkomplex [TFIID; verbleibt an TATA- Box] und den meisten Transkriptionsfaktoren - reife mrna entsteht durch einige Modifikationen an der prä-mrna; auch hierbei spielt CTD eine wichtige Rolle Kappe = methyliertes Guaninnucleotid am 5 -Ende a. durch RNA-Triphosphatase wird am 5 -NTP-Ende der prä-mrna ein Phosphatrest abgespalten Diphosphatende entsteht b. Guanylyl-Transferase heftet ein GTP an 5-5 -Triphosphat- Verknüpfung entsteht ([L] S. 263 Abb. 8.10) c. Methyltransferasen methylieren angeheftetes Guaninnucleotid in Position 7 [gelegentlich auch Ribosereste der beiden folgenden Nucleotide] - Vorgang findet kurz nach Beginn der Elongation statt; Kappe hat wichtige Funktionen: o schützt RNA vor Abbau durch 5 -Exonucleasen o ist Signal für Transport der RNA durch Kernporen o Erkennungsmerkmal für Ribosomen (Bindung an 40S-ribosomale UE) - Termination ist Teil der posttranskriptionalen Modifikation 7. Spaltungs-und PolyA-Signale (s.u.) am 3 -Ende der mrna werden zunächst mit transkribiert; CstF (cleavage stimulation factor) und CPSF (cleavage and polyadenylation specificity factor) sitzen an RNA-Polymerase bis Spaltungssequenz zugänglich ist 8. RNA-Polymerase arbeitet nach Spaltung weiter und verliert dann Affinität zur DNA posttranskriptionelle Modifikation Spleißen die meisten Gene höherer Eukaryoten sind diskontinuierlich angeordnet Introns: intervenierende Sequenzen die nicht codieren, also nicht in der mrna erscheinen Exons: codierende Sequenzen, erscheinen in der mrna - DNA-Polymerase II kann nicht zwischen Introns und Extrons unterscheiden Introns müssen aus prä-mrna entfernt werden durch spleißen - bedeutsam sind die 5 - und 3 - Spleißstelle und die Verzweigungsstelle innerhalb des Introns o 5 -Spleißstelle durch GU[A/G]G gekennzeichnet o 3 -Spleißstelle durch pyrimidinreiche Sequenz + AG gekennzeichnet o an Verzweigungsstelle ist das Adenin von besonderer Bedeutung - [L] S.265 Abb Prinzip: 1. Spaltung der 5 -Spleißstelle durch Bildung eines cyclischen Intermediats (Bruch des RNA- Strangs und Bildung einer Lasso- Struktur) 2. Abspaltung des 3 -Endes des Introns und Verknüpfung der Exons Spleißen bei einfachen Eukaryoten - RNA kann durch ihre Raumstruktur bei einfachen Eukaryoten die Spleißung selbst katalysieren (= proteinfreies Spleißen), RNA- Moleküle mit dieser Fähigkeit nennt man Ribozyme
11 Spleißen bei höheren Eukaryoten - bei höheren Eukaryoten ist komplexer Apparat aus Proteinen und RNA für korrektes Spleißen nötig Spleißosom - kleine RNA- Moleküle (snrnas) liegen in Komplexen mit Proteinen vor (= snrnps ( small nuclear ribonucleoproteins )); Enfernung von Introns erfolgt in folgenden Schritten: 1. snrnp U1 lagert an 5 - Ende des Introns an 2. snrnp U2 bindet an Verzweigungsstelle innerhalb des Introns 3. snrnps U4, U5 und U6 verdrängen U1 Aktivierung der katalytischen Funktion Spleißdefekte Mutationen können die Spleißstellen unkenntlich machen; dies führt zu verlängerten oder verkürzten Proteinen - alternatives Spleißen o Herstellung unterschiedlicher mrna-transkripte/proteine aus einer prä-mrna o Beispiel: Calcitonin/CGRP-Gen; gewebsspezifische Spleißvarianten liefern Hormone mit unterschiedlicher Funktion Anheftung des Poly-A- Endes - laut Skript posttranslational, laut [L] cotranslational - letzter Vorgang der Transkription besteht in Spaltung am 3 - Ende und Anheftung des Poly-A- Endes (Sequenz aus Adenylresten) durch Polyadenylat-Polymerase - Signal ist die Polyadenylierungssequenz AAUAAA, ausgelöst wird Vorgang von Faktoren aus CTD der RNA-Polymerase II mrna-editing - Veränderung in der Sequenz der prä-mrna ermöglicht Herstellung unterschiedlicher Proteine aus einer mrna (versch. Isoformen) - Bsp: Apolipoprotein B o in Leber synthetisierte Apolipoprotein B 100 hat 513 kda o in Darm synthetisierte Apolipoprotein B 48 hat 250 kda RNA-Polymerase I - transkribiert ribosomale RNA - Promotor besteht aus 2 Teilen: o core promotor im Bereich der Startstelle o Kontrollelement etwa Basenpaare oberhalb - beide Promotorregionen reich an GC-Basenpaaren Prozessierung der Transkripte - Bildung von rrna verläuft bei Pro- und Eukaryoten im Prinzip gleich RNA-Polymerase III - einige Promotoren für 5S-RNA- und trna- Gene liegen innerhalb codierender Sequenz, Promotoren für snrna sind oberhalb des Startpunkts lokalisiert - trna- Moleküle entstehen aus langen Transkripten, die von Nucleasen prozessiert werden müssen; Basen werden modifiziert und Teile durch Spleißen entfernt
12 Abbau und Stabilität der mrna - [L] und die Struktur der mrna determiniert ihre Stabilität deadenylierungsabhängiger mrna-abbau - schrittweiser Abbau des 3 -PolyA-Schwanzes durch PolyA-Nuclease o = Signal zur Entfernung der 5 -Kappe Abbau der RNA durch 5-3 -Exonucleasen endonucleolytischer Abbau - sequenzspezifische Spaltung durch Endonucleasen Signal für 5-3 -Exonucleasen Export der mrna aus dem Zellkern - selektiver Transport (nur voll funktionstüchtige RNA-Moleküle) - reife mrna muss als transportkompetent markiert werden o durch Proteinfaktoren, die während Spleißen an mrna gebunden werden - an markierte mrna lagern sich Proteine, die für Wechselwirkung mit Kernporenkomplex notwendig sind binden an Transportrezeptoren o Bsp: NXF1/p15 - Transport erfolgt unidirektional (nur raus), ist energieaufwendig - unmittelbar nach Export binden cytosolische RNA-Bindeproteine an mrna o wichtige Cofaktoren für ribosomale Translation o dienen der cytoplasmatischen Lokalisation der mrna o führen mrna dem Verdau zu Hemmstoffe der Transkription Actinomycin D - Interkalation zwischen CG-Paaren führt zu Verformung und Transkriptionsabbruch - Pro- und Eukaryonten 3 -Desoxadenosin - Transkriptionsabbruch - Pro- und Eukaryonten Rifamipicin - hemmt RNA-Polymerase - Prokaryoten α-amantin - Gift des Knollenblätterpilzes ( s.o.); hemmt RNA-Polymerase II - Eukaryonten
13 Regulation der Genexpression von Leif Übersichtsvergleich Prokaryonten/ Eukaryonten Biochemie- Seminar 10 Prokaryonten - Transkription und Translation simultan - sehr kurze mrna-hwz - Regulation auf Ebene der Transkription gemäß Operonmodell o Repressorprotein (an Operator gebunden) verhindert Transkription des Gens o Repressorprotein kann durch Induktor entfernt werden Eukaryoten - [L] S. 272 Tab Transkription und Translation räumlich und zeitlich getrennt o Transkription und posttransskriptionelle Prozesse finden im Zellkern statt, Proteinbiosynthese im Cytosol Export der mrna durch Kernporen notwendig - z.t. lange mrna-hwz - Transkriptionsfaktoren steuern die Expression einzelner Gene durch Kontakt mit regulatorischen cis-elementen - Regulation durch o Zugänglichkeit des Chromatins o Transkription o posttranskriptionelle Modifikation o Stabilität der mrna Prokaryonten - Prokaryonten regulieren die Proteinmengen fast ausschließlich auf der Ebene der Transkription: o eine Transkriptionseinheit besteht aus mehreren Genen (polycistronische mrna) o metabolisch kooperierende Genprodukte werden von einem genetischen Element gesteuert o regulierbare Repressor-Proteine determinieren die Genaktivität - Beispiele o Lac- Operon; kontrolliert alle Genprodukte für den Abbau von Lactose; bilateral reguliert über Lactose- und Glucose-Angebot: Lactose und Glucose vorhanden schwache Transkription der Lac-Gene nur Lactose vorhanden Hungersignal camp aktiviert CRP CRP bindet an CRP-Region oberhalb Promotor CRP rekrutiert RNA-Polymerase Transkription der Lac-Gene nur Glucose vorhanden Repressor der Lac-Gene aktiv keine Lac-Transkription keine Lactose oder Glucose vorhanden camp-reaktion (s.o.) aber Repressor aktiv keine Lac-Transkription o Tryptophan-Promotor codiert 5 Enzyme der Tryptophansynthese Tryptophan hemmt als Corepressor die Transkription der Enzymgene zusätzlicher Mechanismus passt die Transkriptionsrate dem aktuellen Bedarf genau an: Attenuation
14 Attenuation - Regulation der trp-mrna-synthese durch Komponenten der Translation und Transkription - Leitsequenz enthält mehrere Codons für Tryptophan - je nach Tryptophan-Angebot der Zelle bildet die mrna unterschiedliche Schleifen die entweder die Transkription blockieren oder erlauben Eukaryonten - eucaryotische Gene werden individuell reguliert - für die Transkription muss ein basaler Transkriptionskomplex am Promotor etabliert werden - Gene werden konstitutiv ( Haushalts-Gene ) abgelesen oder gezielt reguliert o konstitutive Genaktivierung: GC-Boxen, keine TATA-Box, häufig keine CCAAT-Box - Möglichkeiten der Regulation o Zugänglichkeit des Chromatins o Transkription o Posttranskriptionelle Modifikation o Stabilität der mrna o Differenzierung von Geweben o induzierbare/konstitutive Expression; zellspezifische Expression o Veränderung der Genexpression durch extrazelluläre Signale Zugänglichkeit des Chromatins - Auflockerung der Nucleosomenstruktur erfolgt durch Modifikation der Histone - Histon-Acetyltransferasen acetylieren Histone an Lysylresten - weitere Histonmodifikationen: Phosphorylierung, Ribosylierung, Methylierung - Euchromatin: transkriptionell aktiv Heterochromatin: transkriptionell inaktiv - Histonacetylierung o Acetylierung von Lysinresten der Histonprotein- Schwanzregion durch Histon-Acetyl- Transferasen trägt zur Auflockerung der Struktur bei o Histon- Deacetylasen machen Strukturänderungen wieder rückgängig - Histonphosphorylierung o unterschiedliche Konsequenzen je nachdem ob in Interphase oder Mitose Interphase: Phosphorylierung von Serinresten Mitose: dient hier der Chromatinkondensation - Histonmethylierung o durch Methyltransferasen o führt zur Anlagerung von Proteinen die eine spezielle Chromodomäne besitzen Chromatinkondensation und Inaktivierung von Genen o anscheinend irreversibel!
15 Transkriptionsregulation durch Aktivierung und Inaktivierung von Genen - durch DNA-Methylierung o jede differenzierte Zelle entwickelt ein Muster aus an- und abgeschalteten Genen o hierfür wird Methylierungsmuster in das Genom eingebracht Inaktivierung von Genen durch Methylierung am C-Atom 5 eines Cytosinrests in einer Kontrollregion; Zielsequenz sind sog. CpG-Inseln (5 -CG-3 ) o Weitergabe des Methylierungsmusters durch Methyltransferase o Methyltransferasen modifizieren ca. 4% der Cytosinreste o o o Bindung von Methyl-CpG-Bindungs-Protein (MeCP) Blockade der transkriptionellen Aktivität der Methylierungsstatus korreliert mit der Genexpression Methylierung auch für genomic imprinting verantwortlich (Allele paternaler und maternaler Gene werden unterschiedlich exprimiert) Epigenetik - Vererbung von Merkmalen, die nicht auf DNA-Sequenz zurückgeführt werden können - zur An/Abschaltung zelltypspezifischer Genprogramme bei Differenzierung; z.b. Zellzyklusregulator p53 + Tumorsuppressorgene in Tumoren hypermethyliert - Histonmodifikation und DNA-Methylierung regulieren die Genexpression durch Veränderungen der DNA/Protein-Interaktion o Histondeacetylasen (HDAC) verdichten Chromatin o methylbindende Proteine (MDB) senken Genaktivität Transkriptionsfaktoren und -aktivatoren - cis-aktivierende Elemente ( Enhancer ; [L] Tab. 8.3) auf der DNA bieten Bindungsstellen für Transkriptionsaktivatoren entstehender Komplex wirkt als zusätzlicher Faktor für RNA- Polymerase II und stimuliert Initiation der Transkription (= Transaktivierung ) - Transkriptionsaktivator benötigt mindestens 2 Domänen: für DNA-Bindung und Transaktivierungsdomäne - die spezifische Wechselwirkung von TF s mit regulatorischen Sequenzen ist die Basis der eucaryotischen Genregulation o Ausbildung von Wasserstoffbrücken o hydrophobe Interaktionen o symmetrische Strukturen im Protein (Dimere) und in der Zielsequenz (Palindrome) - 4 Hauptklassen von Transkriptionsfaktoren o Helix-loop-Helix (HLH)-Proteine o Helix-turn-Helix (HTH)-Proteine o Zinkfinger-Proteine o Leucin-Zipper-Proteine o Zinkfinger siehe [L] S. 276 Abb Cyteinyl- oder Histidylreste in Peptidkette können durch Zink-Atom komplexiert werden schleifenförmige Struktur ( Finger ) entsteht können in der großen Furche der DNA basenspezifische Kontakte knüpfen kommen z.b. in Familie der Steroidhormonrezeptoren vor
16 Strukturmotiv des Leucin-zippers gewährleistet spezifische DNA-Bindung und Dimerisierung des Bindeproteins - in vielen DNA-Bindeproteinen - enthalten als Monomere 2 Domänen; besondere Fähigkeit zur Bildung von Dimeren (homo und hetero) - viele Transkriptionsaktivatoren treten über Leucin-zippers mit anderen Transkriptionsproteinen in Wechselwirkung in vielen DNA-Bindeproteinen kommen Helix-loop-Helix- Strukturen vor - mit Leucin-zipper verwandt - zwei α-helices sind durch flexible Schleife verbunden - Bildung von Homo- und Heterodimeren gut möglich - DNA-Bindeproteine mit Helix-loop-Helix spielen z.b. bei Muskeldifferenzierung eine Rolle negative Regulation - Transkriptionsfaktor NF-κB (z.b.) reguliert Transport von Transkriptionsaktivatoren aus dem Cytosol in den Nucleus
DNA Replikation ist semikonservativ. Abb. aus Stryer (5th Ed.)
 DNA Replikation ist semikonservativ Entwindung der DNA-Doppelhelix durch eine Helikase Replikationsgabel Eltern-DNA Beide DNA-Stränge werden in 5 3 Richtung synthetisiert DNA-Polymerasen katalysieren die
DNA Replikation ist semikonservativ Entwindung der DNA-Doppelhelix durch eine Helikase Replikationsgabel Eltern-DNA Beide DNA-Stränge werden in 5 3 Richtung synthetisiert DNA-Polymerasen katalysieren die
Die DNA Replikation. Exakte Verdopplung des genetischen Materials. Musterstrang. Neuer Strang. Neuer Strang. Eltern-DNA-Doppelstrang.
 Die DNA Replikation Musterstrang Neuer Strang Eltern-DNA-Doppelstrang Neuer Strang Musterstrang Exakte Verdopplung des genetischen Materials Die Reaktion der DNA Polymerase 5`-Triphosphat Nächstes Desoxyribonucleosidtriphosphat
Die DNA Replikation Musterstrang Neuer Strang Eltern-DNA-Doppelstrang Neuer Strang Musterstrang Exakte Verdopplung des genetischen Materials Die Reaktion der DNA Polymerase 5`-Triphosphat Nächstes Desoxyribonucleosidtriphosphat
RNA und Expression RNA
 RNA und Expression Biochemie RNA 1) Die Transkription. 2) RNA-Typen 3) RNA Funktionen 4) RNA Prozessierung 5) RNA und Proteinexpression/Regelung 1 RNA-Typen in E. coli Vergleich RNA-DNA Sequenz 2 Die Transkriptions-Blase
RNA und Expression Biochemie RNA 1) Die Transkription. 2) RNA-Typen 3) RNA Funktionen 4) RNA Prozessierung 5) RNA und Proteinexpression/Regelung 1 RNA-Typen in E. coli Vergleich RNA-DNA Sequenz 2 Die Transkriptions-Blase
Biochemie Tutorium 9. RNA, Transkription
 Biochemie Tutorium 9 RNA, Transkription IMPP-Gegenstandskatalog 3 Genetik 3.1 Nukleinsäuren 3.1.1 Molekulare Struktur, Konformationen und Funktionen der Desoxyribonukleinsäure (DNA); Exon, Intron 3.1.2
Biochemie Tutorium 9 RNA, Transkription IMPP-Gegenstandskatalog 3 Genetik 3.1 Nukleinsäuren 3.1.1 Molekulare Struktur, Konformationen und Funktionen der Desoxyribonukleinsäure (DNA); Exon, Intron 3.1.2
Musterlösung - Übung 5 Vorlesung Bio-Engineering Sommersemester 2008
 Aufgabe 1: Prinzipieller Ablauf der Proteinbiosynthese a) Erklären Sie folgende Begriffe möglichst in Ihren eigenen Worten (1 kurzer Satz): Gen Nukleotid RNA-Polymerase Promotor Codon Anti-Codon Stop-Codon
Aufgabe 1: Prinzipieller Ablauf der Proteinbiosynthese a) Erklären Sie folgende Begriffe möglichst in Ihren eigenen Worten (1 kurzer Satz): Gen Nukleotid RNA-Polymerase Promotor Codon Anti-Codon Stop-Codon
Expression der genetischen Information Skript: Kapitel 5
 Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Prof. A. Sartori Medizin 1. Studienjahr Bachelor Molekulare Zellbiologie FS 2013 12. März 2013 Expression der genetischen Information Skript: Kapitel 5 5.1 Struktur der RNA 5.2 RNA-Synthese (Transkription)
Übung 11 Genregulation bei Prokaryoten
 Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Überblick von DNA zu Protein. Biochemie-Seminar WS 04/05
 Überblick von DNA zu Protein Biochemie-Seminar WS 04/05 Replikationsapparat der Zelle Der gesamte Replikationsapparat umfasst über 20 Proteine z.b. DNA Polymerase: katalysiert Zusammenfügen einzelner Bausteine
Überblick von DNA zu Protein Biochemie-Seminar WS 04/05 Replikationsapparat der Zelle Der gesamte Replikationsapparat umfasst über 20 Proteine z.b. DNA Polymerase: katalysiert Zusammenfügen einzelner Bausteine
Expressionskontrolle in Eukaryonten
 Expressionskontrolle in Eukaryonten Warum muss Genexpression kontrolliert werden? 1. Gewebsspezifische Kontrolle - nicht jedes Genprodukt ist in allen Zellen erforderlich - manche Genprodukte werden ausschliesslich
Expressionskontrolle in Eukaryonten Warum muss Genexpression kontrolliert werden? 1. Gewebsspezifische Kontrolle - nicht jedes Genprodukt ist in allen Zellen erforderlich - manche Genprodukte werden ausschliesslich
Von der DNA zum Eiweißmolekül Die Proteinbiosynthese. Ribosom
 Von der DNA zum Eiweißmolekül Die Proteinbiosynthese Ribosom Wiederholung: DNA-Replikation und Chromosomenkondensation / Mitose Jede Zelle macht von Teilung zu Teilung einen Zellzyklus durch, der aus einer
Von der DNA zum Eiweißmolekül Die Proteinbiosynthese Ribosom Wiederholung: DNA-Replikation und Chromosomenkondensation / Mitose Jede Zelle macht von Teilung zu Teilung einen Zellzyklus durch, der aus einer
Weitergabe genetischer Information: DNA-Replikation Beispiel: Escherichia coli.
 Weitergabe genetischer Information: DNA-Replikation Beispiel: Escherichia coli. zirkuläres bakterielles Chromosom Replikation (Erstellung einer identischen Kopie des genetischen Materials) MPM 1 DNA-Polymerasen
Weitergabe genetischer Information: DNA-Replikation Beispiel: Escherichia coli. zirkuläres bakterielles Chromosom Replikation (Erstellung einer identischen Kopie des genetischen Materials) MPM 1 DNA-Polymerasen
Es ist die Zeit gekommen, zu verstehen, wie es zur Proteinbiosynthese kommt?! Wobei jeweils eine AS von 3 Basen codiert wird..
 Proteinbiosynthese Es ist die Zeit gekommen, zu verstehen, wie es zur Proteinbiosynthese kommt?! Alle Proteine, sind über die DNA codiert Wobei jeweils eine AS von 3 Basen codiert wird.. GENETISCHER CODE
Proteinbiosynthese Es ist die Zeit gekommen, zu verstehen, wie es zur Proteinbiosynthese kommt?! Alle Proteine, sind über die DNA codiert Wobei jeweils eine AS von 3 Basen codiert wird.. GENETISCHER CODE
Regulation des Zellcylus
 Regulation des Zellcylus Dr. F. Neuschäfer-Rube Cyclin-abhängige Kinasen (CDKs) Cyclin-abhängige Kinasen: Motoren und Schalter des Zellzyclus Dr. F. Neuschäfer-Rube Der Zellzyklus M S Der Zellzyklus M
Regulation des Zellcylus Dr. F. Neuschäfer-Rube Cyclin-abhängige Kinasen (CDKs) Cyclin-abhängige Kinasen: Motoren und Schalter des Zellzyclus Dr. F. Neuschäfer-Rube Der Zellzyklus M S Der Zellzyklus M
Dr. Jens Kurreck. Otto-Hahn-Bau, Thielallee 63, Raum 029 Tel.: 83 85 69 69 Email: jkurreck@chemie.fu-berlin.de
 Dr. Jens Kurreck Otto-Hahn-Bau, Thielallee 63, Raum 029 Tel.: 83 85 69 69 Email: jkurreck@chemie.fu-berlin.de Prinzipien genetischer Informationsübertragung Berg, Tymoczko, Stryer: Biochemie 5. Auflage,
Dr. Jens Kurreck Otto-Hahn-Bau, Thielallee 63, Raum 029 Tel.: 83 85 69 69 Email: jkurreck@chemie.fu-berlin.de Prinzipien genetischer Informationsübertragung Berg, Tymoczko, Stryer: Biochemie 5. Auflage,
Eukaryotische messenger-rna
 Eukaryotische messenger-rna Cap-Nukleotid am 5 -Ende Polyadenylierung am 3 -Ende u.u. nicht-codierende Bereiche (Introns) Spleißen von prä-mrna Viele Protein-codierende Gene in Eukaryoten sind durch nicht-codierende
Eukaryotische messenger-rna Cap-Nukleotid am 5 -Ende Polyadenylierung am 3 -Ende u.u. nicht-codierende Bereiche (Introns) Spleißen von prä-mrna Viele Protein-codierende Gene in Eukaryoten sind durch nicht-codierende
Transkription Teil 2. - Transkription bei Eukaryoten -
 Transkription Teil 2 - Transkription bei Eukaryoten - Inhalte: Unterschiede in der Transkription von Pro- und Eukaryoten Die RNA-Polymerasen der Eukaryoten Cis- und trans-aktive Elemente Promotoren Transkriptionsfaktoren
Transkription Teil 2 - Transkription bei Eukaryoten - Inhalte: Unterschiede in der Transkription von Pro- und Eukaryoten Die RNA-Polymerasen der Eukaryoten Cis- und trans-aktive Elemente Promotoren Transkriptionsfaktoren
Q1 B1 KW 49. Genregulation
 Q1 B1 KW 49 Genregulation Transkription Posttranskription elle Modifikation Genregulation bei Eukaryoten Transkriptionsfaktoren (an TATA- Box) oder Silencer (verringert Transkription) und Enhancer (erhöht
Q1 B1 KW 49 Genregulation Transkription Posttranskription elle Modifikation Genregulation bei Eukaryoten Transkriptionsfaktoren (an TATA- Box) oder Silencer (verringert Transkription) und Enhancer (erhöht
Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr
 Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr Name: Matrikel-Nr.: Code Nummer: Bitte geben Sie Ihre Matrikel-Nr. und Ihren Namen an. Die Code-Nummer erhalten Sie zu Beginn
Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr Name: Matrikel-Nr.: Code Nummer: Bitte geben Sie Ihre Matrikel-Nr. und Ihren Namen an. Die Code-Nummer erhalten Sie zu Beginn
Posttranskriptionale RNA-Prozessierung
 Posttranskriptionale RNA-Prozessierung Spaltung + Modifikation G Q Spleissen + Editing U UUU Prozessierung einer prä-trna Eukaryotische messenger-rna Cap-Nukleotid am 5 -Ende Polyadenylierung am 3 -Ende
Posttranskriptionale RNA-Prozessierung Spaltung + Modifikation G Q Spleissen + Editing U UUU Prozessierung einer prä-trna Eukaryotische messenger-rna Cap-Nukleotid am 5 -Ende Polyadenylierung am 3 -Ende
Genaktivierung und Genexpression
 Genaktivierung und Genexpression Unter Genexpression versteht man ganz allgemein die Ausprägung des Genotyps zum Phänotyp einer Zelle oder eines ganzen Organismus. Genotyp: Gesamtheit der Informationen
Genaktivierung und Genexpression Unter Genexpression versteht man ganz allgemein die Ausprägung des Genotyps zum Phänotyp einer Zelle oder eines ganzen Organismus. Genotyp: Gesamtheit der Informationen
Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen. Abb. aus Stryer (5th Ed.)
 Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen Die verschiedenen Ribosomen-Komplexe können im Elektronenmikroskop beobachtet werden Durch Röntgenkristallographie wurden
Elektronenmikroskopie zeigte die Existenz der A-, P- und E- trna-bindungsstellen Die verschiedenen Ribosomen-Komplexe können im Elektronenmikroskop beobachtet werden Durch Röntgenkristallographie wurden
Klausur zur Vorlesung Biochemie III im WS 2000/01
 Klausur zur Vorlesung Biochemie III im WS 2000/01 am 15.02.2001 von 15.30 17.00 Uhr (insgesamt 100 Punkte, mindestens 40 erforderlich) Bitte Name, Matrikelnummer und Studienfach unbedingt angeben (3 1.
Klausur zur Vorlesung Biochemie III im WS 2000/01 am 15.02.2001 von 15.30 17.00 Uhr (insgesamt 100 Punkte, mindestens 40 erforderlich) Bitte Name, Matrikelnummer und Studienfach unbedingt angeben (3 1.
Biochemisches Grundpraktikum
 Biochemisches Grundpraktikum Dr. Ellen Hornung; Email: ehornun@gwdg.de; Tel: 39-5748 Einteilung der Praktikumsplätze: Eintragen in Listen am - Dienstag, 10.11.2009, von 12:00 13:00 - Freitag, 13.11.2009,
Biochemisches Grundpraktikum Dr. Ellen Hornung; Email: ehornun@gwdg.de; Tel: 39-5748 Einteilung der Praktikumsplätze: Eintragen in Listen am - Dienstag, 10.11.2009, von 12:00 13:00 - Freitag, 13.11.2009,
4. Genetische Mechanismen bei Bakterien
 4. Genetische Mechanismen bei Bakterien 4.1 Makromoleküle und genetische Information Aufbau der DNA Phasen des Informationsflusses Vergleich der Informationsübertragung bei Pro- und Eukaryoten 4.2 Struktur
4. Genetische Mechanismen bei Bakterien 4.1 Makromoleküle und genetische Information Aufbau der DNA Phasen des Informationsflusses Vergleich der Informationsübertragung bei Pro- und Eukaryoten 4.2 Struktur
Übung 8. 1. Zellkommunikation. Vorlesung Bio-Engineering Sommersemester 2008. Kapitel 4. 4
 Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 05. 05. 08
Bitte schreiben Sie Ihre Antworten direkt auf das Übungsblatt. Falls Sie mehr Platz brauchen verweisen Sie auf Zusatzblätter. Vergessen Sie Ihren Namen nicht! Abgabe der Übung bis spätestens 05. 05. 08
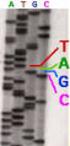
DNA-Sequenzierung. Martina Krause
 DNA-Sequenzierung Martina Krause Inhalt Methoden der DNA-Sequenzierung Methode nach Maxam - Gilbert Methode nach Sanger Einleitung Seit 1977 stehen zwei schnell arbeitende Möglichkeiten der Sequenzanalyse
DNA-Sequenzierung Martina Krause Inhalt Methoden der DNA-Sequenzierung Methode nach Maxam - Gilbert Methode nach Sanger Einleitung Seit 1977 stehen zwei schnell arbeitende Möglichkeiten der Sequenzanalyse
Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt. Abb. aus Stryer (5th Ed.)
 Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt Die Initiation der Translation bei Eukaryoten Der eukaryotische Initiationskomplex erkennt zuerst das 5 -cap der mrna und
Transkription und Translation sind in Eukaryoten räumlich und zeitlich getrennt Die Initiation der Translation bei Eukaryoten Der eukaryotische Initiationskomplex erkennt zuerst das 5 -cap der mrna und
Promotor kodierende Sequenz Terminator
 5.2 Genexpression Sequenz in eine RNA-Sequenz. Die Enzyme, die diese Reaktion katalysieren, sind die DNA-abhängigen RNA-Polymerasen. Sie bestehen aus mehreren Untereinheiten, die von den Pro- bis zu den
5.2 Genexpression Sequenz in eine RNA-Sequenz. Die Enzyme, die diese Reaktion katalysieren, sind die DNA-abhängigen RNA-Polymerasen. Sie bestehen aus mehreren Untereinheiten, die von den Pro- bis zu den
Kapitel 8 Ò Chromosomen und Genregulation
 Kapitel 8 Ò Chromosomen und Genregulation 8.1 Struktur eukaryontischer Chromosomen Ein menschlicher Zellkern ist nur zehn Mikrometer gross und (10-9 ) hat zwei Meter DNA drin. Damit es da kein Durcheinander
Kapitel 8 Ò Chromosomen und Genregulation 8.1 Struktur eukaryontischer Chromosomen Ein menschlicher Zellkern ist nur zehn Mikrometer gross und (10-9 ) hat zwei Meter DNA drin. Damit es da kein Durcheinander
Anabole Prozesse in der Zelle
 Anabole Prozesse in der Zelle DNA Vermehrung RNA Synthese Protein Synthese Protein Verteilung in der Zelle Ziel: Zellteilung (Wachstum) und Differenzierung (Aufgabenteilung im Organismus). 2016 Struktur
Anabole Prozesse in der Zelle DNA Vermehrung RNA Synthese Protein Synthese Protein Verteilung in der Zelle Ziel: Zellteilung (Wachstum) und Differenzierung (Aufgabenteilung im Organismus). 2016 Struktur
Antwort: 2.Uracil. Antwort: 2. durch Wasserstoffverbindungen. Adenin, Cystein und Guanin kommen alle in der RNA und DNA vor.
 Antwort: 2.Uracil Adenin, Cystein und Guanin kommen alle in der RNA und DNA vor. Thymin kommt nur in der DNA vor; Uracil nimmt seinen Platz in den RNA- Molekülen ein. Antwort: 2. durch Wasserstoffverbindungen
Antwort: 2.Uracil Adenin, Cystein und Guanin kommen alle in der RNA und DNA vor. Thymin kommt nur in der DNA vor; Uracil nimmt seinen Platz in den RNA- Molekülen ein. Antwort: 2. durch Wasserstoffverbindungen
Seminar Biochemie. Nukleotide - Nukleinsäuren - Nukleotidstoffwechsel - DNA-Replikation. Dr. Christian Hübbers
 Seminar Biochemie Nukleotide - Nukleinsäuren - Nukleotidstoffwechsel - DNA-Replikation Dr. Christian Hübbers Lernziele Zusammensetzung der Nukleotide (Basen, Zucker) Purin-und Pyrimidinbiosynthese (prinzipieller
Seminar Biochemie Nukleotide - Nukleinsäuren - Nukleotidstoffwechsel - DNA-Replikation Dr. Christian Hübbers Lernziele Zusammensetzung der Nukleotide (Basen, Zucker) Purin-und Pyrimidinbiosynthese (prinzipieller
Aufbau und Funktion des Genoms: Von der Genstruktur zur Funktion
 Assoc. Prof. PD Mag. Dr. Aufbau und Funktion des Genoms: Von der Genstruktur zur Funktion Wien, 2013 Währinger Straße 10, A-1090 Wien helmut.dolznig@meduniwien.ac.at www.meduniwien.ac.at/medizinische-genetik
Assoc. Prof. PD Mag. Dr. Aufbau und Funktion des Genoms: Von der Genstruktur zur Funktion Wien, 2013 Währinger Straße 10, A-1090 Wien helmut.dolznig@meduniwien.ac.at www.meduniwien.ac.at/medizinische-genetik
7. Regulation der Genexpression
 7. Regulation der Genexpression 7.1 Regulation der Enzymaktivität Stoffwechselreaktionen können durch Kontrolle der Aktivität der Enzyme, die diese Reaktionen katalysieren, reguliert werden Feedback-Hemmung
7. Regulation der Genexpression 7.1 Regulation der Enzymaktivität Stoffwechselreaktionen können durch Kontrolle der Aktivität der Enzyme, die diese Reaktionen katalysieren, reguliert werden Feedback-Hemmung
Bei der Translation wird die Aminosäuresequenz eines Polypeptids durch die Sequenz der Nukleotide in einem mrna- Molekül festgelegt
 Bei der Translation wird die Aminosäuresequenz eines Polypeptids durch die Sequenz der Nukleotide in einem mrna- Molekül festgelegt 5 mrna Nukleotid 3 N-Terminus Protein C-Terminus Aminosäure Es besteht
Bei der Translation wird die Aminosäuresequenz eines Polypeptids durch die Sequenz der Nukleotide in einem mrna- Molekül festgelegt 5 mrna Nukleotid 3 N-Terminus Protein C-Terminus Aminosäure Es besteht
KV: DNA-Replikation Michael Altmann
 Institut für Biochemie und Molekulare Medizin KV: DNA-Replikation Michael Altmann Herbstsemester 2008/2009 Übersicht VL DNA-Replikation 1.) Das Zentraldogma der Molekularbiologie 1.) Semikonservative Replikation
Institut für Biochemie und Molekulare Medizin KV: DNA-Replikation Michael Altmann Herbstsemester 2008/2009 Übersicht VL DNA-Replikation 1.) Das Zentraldogma der Molekularbiologie 1.) Semikonservative Replikation
Kontrolle des Zellzyklus: Checkpoints. kann an bestimmten Kontrollpunkten gestoppt werden
 Kontrolle des Zellzyklus: Checkpoints kann an bestimmten Kontrollpunkten gestoppt werden G2 checkpoint komplette DNA Replikation? sind DNA Schäden vorhanden? ist die Zelle groß genug? ist die Umgebung
Kontrolle des Zellzyklus: Checkpoints kann an bestimmten Kontrollpunkten gestoppt werden G2 checkpoint komplette DNA Replikation? sind DNA Schäden vorhanden? ist die Zelle groß genug? ist die Umgebung
Spleißen und Prozessieren von mrna
 Spleißen und Prozessieren von mrna Spleißen, die Aneinanderreihung von Exons: Prä-mRNAs sind 4-10x länger als die eigentlichen mrnas. Funktionelle Sequenzabschnitte in den Introns der Prä-mRNA: 5 -Spleißstelle
Spleißen und Prozessieren von mrna Spleißen, die Aneinanderreihung von Exons: Prä-mRNAs sind 4-10x länger als die eigentlichen mrnas. Funktionelle Sequenzabschnitte in den Introns der Prä-mRNA: 5 -Spleißstelle
PROTEINBIOSYNTHESE "Das zentrale Dogma der Molekularbiologie"
 PROTEINBIOSYNTHESE "Das zentrale Dogma der Molekularbiologie" Die für die Synthese von Eiweißstoffen notwendigen Schritte sind: (1) Replikation der DNA: Vor jeder Zellteilung wird die gesamte zelluläre
PROTEINBIOSYNTHESE "Das zentrale Dogma der Molekularbiologie" Die für die Synthese von Eiweißstoffen notwendigen Schritte sind: (1) Replikation der DNA: Vor jeder Zellteilung wird die gesamte zelluläre
Übung 11 Genregulation bei Prokaryoten
 Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
Übung 11 Genregulation bei Prokaryoten Konzepte: Differentielle Genexpression Positive Genregulation Negative Genregulation cis-/trans-regulation 1. Auf welchen Ebenen kann Genregulation stattfinden? Definition
DNA: Aufbau, Struktur und Replikation
 DNA: Aufbau, Struktur und Replikation Biochemie Die DNA als Träger der Erbinformation Im Genom sind sämtliche Informationen in Form von DNA gespeichert. Die Information des Genoms ist statisch, d. h. in
DNA: Aufbau, Struktur und Replikation Biochemie Die DNA als Träger der Erbinformation Im Genom sind sämtliche Informationen in Form von DNA gespeichert. Die Information des Genoms ist statisch, d. h. in
Datenspeicherung und Datenfluß in der Zelle - Grundlagen der Biochemie
 Datenspeicherung und Datenfluß in der Zelle - Grundlagen der Biochemie Datenspeicherung und Datenfluß der Zelle Transkription DNA RNA Translation Protein Aufbau I. Grundlagen der organischen Chemie und
Datenspeicherung und Datenfluß in der Zelle - Grundlagen der Biochemie Datenspeicherung und Datenfluß der Zelle Transkription DNA RNA Translation Protein Aufbau I. Grundlagen der organischen Chemie und
Verbesserte Basenpaarung bei DNA-Analysen
 Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/verbesserte-basenpaarungbei-dna-analysen/ Verbesserte Basenpaarung bei DNA-Analysen Ein Team aus der Organischen
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/verbesserte-basenpaarungbei-dna-analysen/ Verbesserte Basenpaarung bei DNA-Analysen Ein Team aus der Organischen
Apoptose 69. 1. Einleitung 2. Extrinsischer Weg 3. Intrinsischer Weg
 Teil III. Signalweg des Zelltodes im Herzen Apoptose 69. 1. Einleitung 2. Extrinsischer Weg 3. Intrinsischer Weg >50% der Neuronen Während der Entwicklung 70. Kontraktionsband Nekrose Retina DNA Abbau
Teil III. Signalweg des Zelltodes im Herzen Apoptose 69. 1. Einleitung 2. Extrinsischer Weg 3. Intrinsischer Weg >50% der Neuronen Während der Entwicklung 70. Kontraktionsband Nekrose Retina DNA Abbau
Entwicklungs /gewebespezifische Genexpression
 Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Positive Genregulation Negative Genregulation cis /trans Regulation 1. Auf welchen Ebenen kann Genregulation
Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Positive Genregulation Negative Genregulation cis /trans Regulation 1. Auf welchen Ebenen kann Genregulation
Zentrales Dogma der Biologie
 Zentrales Dogma der Biologie Transkription: von der DNA zur RNA Biochemie 01/1 Transkription Biochemie 01/2 Transkription DNA: RNA: Biochemie 01/3 Transkription DNA: RNA: Biochemie 01/4 Transkription RNA:
Zentrales Dogma der Biologie Transkription: von der DNA zur RNA Biochemie 01/1 Transkription Biochemie 01/2 Transkription DNA: RNA: Biochemie 01/3 Transkription DNA: RNA: Biochemie 01/4 Transkription RNA:
Wiederholunng. Klassische Genetik
 Wiederholunng Klassische Genetik Mendelsche Regeln Uniformitätsregel Spaltungsregel Freie Kombinierbarkeit Koppelung von Genen Polygene: mehre Gene für ein Merkmal Pleiotropie: 1 Gen steuert mehrere Merkmale
Wiederholunng Klassische Genetik Mendelsche Regeln Uniformitätsregel Spaltungsregel Freie Kombinierbarkeit Koppelung von Genen Polygene: mehre Gene für ein Merkmal Pleiotropie: 1 Gen steuert mehrere Merkmale
Biochemie Vorlesung Die ersten 100 Seiten
 Biochemie Vorlesung 11-15 Die ersten 100 Seiten 1. Unterschiede der Zellen Eukaryoten- Prokaryoten Eukaryoten: - Keine Zellwand - Intrazelluläre Membransysteme - Kernhülle mit 2 Membranen und Kernporen
Biochemie Vorlesung 11-15 Die ersten 100 Seiten 1. Unterschiede der Zellen Eukaryoten- Prokaryoten Eukaryoten: - Keine Zellwand - Intrazelluläre Membransysteme - Kernhülle mit 2 Membranen und Kernporen
IV. Übungsaufgaben für die Jahrgangstufe 9 & 10
 IV. Übungsaufgaben für die Jahrgangstufe 9 & 10 Von der Erbanlage zum Erbmerkmal: 34) Welche Aufgaben haben Chromosomen? 35) Zeichne und benenne die Teile eines Chromosoms, wie sie im Lichtmikroskop während
IV. Übungsaufgaben für die Jahrgangstufe 9 & 10 Von der Erbanlage zum Erbmerkmal: 34) Welche Aufgaben haben Chromosomen? 35) Zeichne und benenne die Teile eines Chromosoms, wie sie im Lichtmikroskop während
9.) Wie heißen die kurzen RNA-Moleküle, mit deren Hilfe die Polymerase die Replikation der DNA starten kann? a) Starter b) Primer c) Beginner
 Lernkontrolle M o d u l 1 A w i e... A n k r e u z e n! 1.) Wie viele Chromosomen besitzt eine menschliche Körperzelle? a) 23 b) 46 c) 44 2.) In welchem Zellorganell befindet sich die DNA? a) Zellkern
Lernkontrolle M o d u l 1 A w i e... A n k r e u z e n! 1.) Wie viele Chromosomen besitzt eine menschliche Körperzelle? a) 23 b) 46 c) 44 2.) In welchem Zellorganell befindet sich die DNA? a) Zellkern
DNA mrna Protein. Initiation Elongation Termination. RNA Prozessierung. Unterschiede Pro /Eukaryoten
 7. Transkription Konzepte: DNA mrna Protein Initiation Elongation Termination RNA Prozessierung Unterschiede Pro /Eukaryoten 3. Aus welchen vier Nukleotiden ist RNA aufgebaut? 4. DNA RNA 5. Ein Wissenschaftler
7. Transkription Konzepte: DNA mrna Protein Initiation Elongation Termination RNA Prozessierung Unterschiede Pro /Eukaryoten 3. Aus welchen vier Nukleotiden ist RNA aufgebaut? 4. DNA RNA 5. Ein Wissenschaftler
3.5 Moderne Genetik - Vorgänge
 3.5 Moderne Genetik - Vorgänge Der genetische Code Jedes Gen besteht aus sogenannten Basentriplets. Das ist eine Sequenz von drei aufeinanderfolgenden Nukleinbasen, die für eine bestimmte Aminosäure stehen.
3.5 Moderne Genetik - Vorgänge Der genetische Code Jedes Gen besteht aus sogenannten Basentriplets. Das ist eine Sequenz von drei aufeinanderfolgenden Nukleinbasen, die für eine bestimmte Aminosäure stehen.
Transkription in Prokaryoten (Bakterien)
 Einführung Transkription in Prokaryoten (Bakterien) Transkription in Eukaryoten - Eukaryotische RNA-Polymerasen - Transkription durch RNA-Polymerase II - Transkription durch RNA-Polymerase I & III - Genregulation
Einführung Transkription in Prokaryoten (Bakterien) Transkription in Eukaryoten - Eukaryotische RNA-Polymerasen - Transkription durch RNA-Polymerase II - Transkription durch RNA-Polymerase I & III - Genregulation
Träger der Erbinformation sind die Nukleinsäuren. Es handelt sich hierbei um hochmolekulare lineare Kettenmoleküle, die aus durch
 Achtung Die folgenden Texte sind als Stichworte für die Klausurvorbereitung zu sehen. Keinesfalls sind die Fragen in der Klausur auf den Inhalt dieser Folien beschränkt, sondern werden aus dem Stoff der
Achtung Die folgenden Texte sind als Stichworte für die Klausurvorbereitung zu sehen. Keinesfalls sind die Fragen in der Klausur auf den Inhalt dieser Folien beschränkt, sondern werden aus dem Stoff der
Entwicklungs /gewebespezifische Genexpression. Coexpression funktional überlappender Gene
 Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Coexpression funktional überlappender Gene Positive Genregulation Negative Genregulation cis /trans Regulation
Übung 11 Genregulation bei Prokaryoten Konzepte: Entwicklungs /gewebespezifische Genexpression Coexpression funktional überlappender Gene Positive Genregulation Negative Genregulation cis /trans Regulation
Vom Gen zum Protein. Zusammenfassung Kapitel 17. Die Verbindung zwischen Gen und Protein. Gene spezifizieren Proteine
 Zusammenfassung Kapitel 17 Vom Gen zum Protein Die Verbindung zwischen Gen und Protein Gene spezifizieren Proteine Zellen bauen organische Moleküle über Stoffwechselprozesse auf und ab. Diese Prozesse
Zusammenfassung Kapitel 17 Vom Gen zum Protein Die Verbindung zwischen Gen und Protein Gene spezifizieren Proteine Zellen bauen organische Moleküle über Stoffwechselprozesse auf und ab. Diese Prozesse
Genregulation bei Eukaryoten II
 Genregulation bei Eukaryoten II Aktivierung und Repression der Transkription erfolgen durch Protein-Protein-Wechselwirkungen Protein-Protein-Wechselwirkungen spielen bei der Genregulation der Eukaryoten
Genregulation bei Eukaryoten II Aktivierung und Repression der Transkription erfolgen durch Protein-Protein-Wechselwirkungen Protein-Protein-Wechselwirkungen spielen bei der Genregulation der Eukaryoten
1. Beschriften Sie in der Abbildung die verschiedenen Bereiche auf der DNA und beschreiben Sie ihre Funktion! nicht-codogener Strang.
 ARBEITSBLATT 1 Transkription 1. Beschriften Sie in der Abbildung die verschiedenen Bereiche auf der DNA und beschreiben Sie ihre Funktion! Bindungsstelle für RNA-Polymerase RNA-Polymerase nicht-codogener
ARBEITSBLATT 1 Transkription 1. Beschriften Sie in der Abbildung die verschiedenen Bereiche auf der DNA und beschreiben Sie ihre Funktion! Bindungsstelle für RNA-Polymerase RNA-Polymerase nicht-codogener
APP-GFP/Fluoreszenzmikroskop. Aufnahmen neuronaler Zellen, mit freund. Genehmigung von Prof. Stefan Kins, TU Kaiserslautern
 Über die Herkunft von Aβ42 und Amyloid-Plaques Heute ist sicher belegt, dass sich die amyloiden Plaques aus einer Vielzahl an Abbaufragmenten des Amyloid-Vorläufer-Proteins (amyloid-precursor-protein,
Über die Herkunft von Aβ42 und Amyloid-Plaques Heute ist sicher belegt, dass sich die amyloiden Plaques aus einer Vielzahl an Abbaufragmenten des Amyloid-Vorläufer-Proteins (amyloid-precursor-protein,
Molekularbiologie 6c Proteinbiosynthese. Bei der Proteinbiosynthese geht es darum, wie die Information der DNA konkret in ein Protein umgesetzt wird
 Molekularbiologie 6c Proteinbiosynthese Bei der Proteinbiosynthese geht es darum, wie die Information der DNA konkret in ein Protein umgesetzt wird 1 Übersicht: Vom Gen zum Protein 1. 2. 3. 2 Das Dogma
Molekularbiologie 6c Proteinbiosynthese Bei der Proteinbiosynthese geht es darum, wie die Information der DNA konkret in ein Protein umgesetzt wird 1 Übersicht: Vom Gen zum Protein 1. 2. 3. 2 Das Dogma
DNA mrna Protein. Initiation Elongation Termination. RNA Prozessierung. Unterschiede Pro /Eukaryoten
 7. Transkription Konzepte: DNA mrna Protein Initiation Elongation Termination RNA Prozessierung Unterschiede Pro /Eukaryoten 1. Aus welchen vier Nukleotiden ist RNA aufgebaut? 2. RNA unterscheidet sich
7. Transkription Konzepte: DNA mrna Protein Initiation Elongation Termination RNA Prozessierung Unterschiede Pro /Eukaryoten 1. Aus welchen vier Nukleotiden ist RNA aufgebaut? 2. RNA unterscheidet sich
Proteinbiosynthese. Prof. Dr. Albert Duschl
 Proteinbiosynthese Prof. Dr. Albert Duschl DNA/RNA/Protein Im Bereich von Genen sind die beiden Stränge der DNA nicht funktionell äquivalent, weil nur einer der beiden Stränge transkribiert, d.h. in RNA
Proteinbiosynthese Prof. Dr. Albert Duschl DNA/RNA/Protein Im Bereich von Genen sind die beiden Stränge der DNA nicht funktionell äquivalent, weil nur einer der beiden Stränge transkribiert, d.h. in RNA
Klonierung von S2P Rolle der M19-Zellen. POL-Seminar der Biochemie II 13.02.2007 Sebastian Gabriel
 Klonierung von S2P Rolle der M19-Zellen POL-Seminar der Biochemie II 13.02.2007 Sebastian Gabriel Inhalt 1. Was ist eine humane genomische DNA-Bank? 2. Unterschied zwischen cdna-bank und genomischer DNA-Bank?
Klonierung von S2P Rolle der M19-Zellen POL-Seminar der Biochemie II 13.02.2007 Sebastian Gabriel Inhalt 1. Was ist eine humane genomische DNA-Bank? 2. Unterschied zwischen cdna-bank und genomischer DNA-Bank?
Exkurs 4: Oligonucleotide als Antisense Wirkstoffe
 Exkurs 4: ligonucleotide als Antisense Wirkstoffe Pharmazeutische Biochemie Antisense Wirkstoffe am Markt Fomivirsen (INN) Handelsname Vitravene Einsatz: Lokale Behandlung von Zytomegalie-Virus Infektionen
Exkurs 4: ligonucleotide als Antisense Wirkstoffe Pharmazeutische Biochemie Antisense Wirkstoffe am Markt Fomivirsen (INN) Handelsname Vitravene Einsatz: Lokale Behandlung von Zytomegalie-Virus Infektionen
DNA mrna Protein. Initiation Elongation Termination. RNA Prozessierung. Unterschiede Pro /Eukaryoten
 7. Transkription Konzepte: DNA mrna Protein Initiation Elongation Termination RNA Prozessierung Unterschiede Pro /Eukaryoten 1. Aus welchen vier Nukleotiden ist RNA aufgebaut? 2. RNA unterscheidet sich
7. Transkription Konzepte: DNA mrna Protein Initiation Elongation Termination RNA Prozessierung Unterschiede Pro /Eukaryoten 1. Aus welchen vier Nukleotiden ist RNA aufgebaut? 2. RNA unterscheidet sich
VORANGEGANGENE MODELLE
 VORANGEGANGENE MODELLE UNSER THEMA HEUTE Ziel dieses Papers: - Nähere Charakterisierung der AuxREs - Analyse eines zu den AuxREs gehörenden Transkriptionsfaktors WAS BEREITS BEKANNT WAR: - Auxin moduliert
VORANGEGANGENE MODELLE UNSER THEMA HEUTE Ziel dieses Papers: - Nähere Charakterisierung der AuxREs - Analyse eines zu den AuxREs gehörenden Transkriptionsfaktors WAS BEREITS BEKANNT WAR: - Auxin moduliert
TRANSKRIPTION I. Die Herstellung von RNA bei E-Coli
 TRANSKRIPTION I Die Herstellung von RNA bei E-Coli Inhalt Aufbau der RNA-Polymerase Promotoren Sigma-Untereinheit Entwindung der DNA Elongation Termination der Transkription Modifizierung der RNA Antibiotika
TRANSKRIPTION I Die Herstellung von RNA bei E-Coli Inhalt Aufbau der RNA-Polymerase Promotoren Sigma-Untereinheit Entwindung der DNA Elongation Termination der Transkription Modifizierung der RNA Antibiotika
Grundideen der Gentechnik
 Grundideen der Gentechnik Die Gentechnik kombiniert Biotechnik und Züchtung. Wie in der Züchtung wird die Erbinformation eines Lebewesen verändert. Dabei nutzte man in den Anfängen der Gentechnik vor allem
Grundideen der Gentechnik Die Gentechnik kombiniert Biotechnik und Züchtung. Wie in der Züchtung wird die Erbinformation eines Lebewesen verändert. Dabei nutzte man in den Anfängen der Gentechnik vor allem
8. Translation. Konzepte: Translation benötigt trnas und Ribosomen. Genetischer Code. Initiation - Elongation - Termination
 8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation - Elongation - Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse human bacteria
8. Translation Konzepte: Translation benötigt trnas und Ribosomen Genetischer Code Initiation - Elongation - Termination 1. Welche Typen von RNAs gibt es und welches sind ihre Funktionen? mouse human bacteria
Molekulargenetik der Eukaryoten WS 2014/15, VL 11. Erwin R. Schmidt Institut für Molekulargenetik
 Molekulargenetik der Eukaryoten WS 2014/15, VL 11 Erwin R. Schmidt Institut für Molekulargenetik Abhängig von der Genklasse: Genstruktur der Eukaryoten 1. RNA Pol I Gene: 18S, 5,8S, 28S rrna 2. RNA Pol
Molekulargenetik der Eukaryoten WS 2014/15, VL 11 Erwin R. Schmidt Institut für Molekulargenetik Abhängig von der Genklasse: Genstruktur der Eukaryoten 1. RNA Pol I Gene: 18S, 5,8S, 28S rrna 2. RNA Pol
In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit
 In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit in der Nucleotidsequenz der DNA verschlüsselt (codiert)
In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit in der Nucleotidsequenz der DNA verschlüsselt (codiert)
6. DNA -Bakteriengenetik
 6. DNA -Bakteriengenetik Konzepte: Francis Crick DNA Struktur DNA Replikation Gentransfer in Bakterien Bakteriophagen 2. Welcher der folgenden Sätze entspricht der Chargaff-Regel? A) Die Menge von Purinen
6. DNA -Bakteriengenetik Konzepte: Francis Crick DNA Struktur DNA Replikation Gentransfer in Bakterien Bakteriophagen 2. Welcher der folgenden Sätze entspricht der Chargaff-Regel? A) Die Menge von Purinen
KV: Genexpression und Transkription Michael Altmann
 Institut für Biochemie und Molekulare Medizin KV: Genexpression und Transkription Michael Altmann Herbstsemester 2008/2009 Übersicht VL Genexpression / Transkription 1.) Was ist ein Gen? 2.) Welche Arten
Institut für Biochemie und Molekulare Medizin KV: Genexpression und Transkription Michael Altmann Herbstsemester 2008/2009 Übersicht VL Genexpression / Transkription 1.) Was ist ein Gen? 2.) Welche Arten
Molekulargenetik Biologie am Inhaltsverzeichnis Die Begriffe DNA, Nukleotid, Gen, Chromosom und Epigenom definieren...
 Molekulargenetik Inhaltsverzeichnis Die Begriffe DNA, Nukleotid, Gen, Chromosom und Epigenom definieren... 2 Beschreiben, wie die DNA aufgebaut ist... 3 Den Ablauf der Replikation erklären und dabei die
Molekulargenetik Inhaltsverzeichnis Die Begriffe DNA, Nukleotid, Gen, Chromosom und Epigenom definieren... 2 Beschreiben, wie die DNA aufgebaut ist... 3 Den Ablauf der Replikation erklären und dabei die
GENETIK. für Studierende. Michaela Aubele. für Ahnungslose. Eine Einstiegshilfe. 2. Auflage. Dr. Michaela Aubele, München.
 Michaela Aubele GENETIK für Ahnungslose Eine Einstiegshilfe für Studierende 2. Auflage von Prof. Dr. Michaela Aubele, München Mit 52 Abbildungen und 33 Tabellen S. Hirzel Verlag die VII Vorwort V Kurzer
Michaela Aubele GENETIK für Ahnungslose Eine Einstiegshilfe für Studierende 2. Auflage von Prof. Dr. Michaela Aubele, München Mit 52 Abbildungen und 33 Tabellen S. Hirzel Verlag die VII Vorwort V Kurzer
Inhaltsverzeichnis. 1. Lebensformen: Zellen mit und ohne Kern... 3. 2. DNA: Träger der genetischen Information... 9
 Vorwort IX Teil I Grundlagen 1. Lebensformen: Zellen mit und ohne Kern... 3 Eukaryoten... 4 Prokaryoten... 6 Literatur... 8 2. DNA: Träger der genetischen Information... 9 Bausteine: Nucleotide... 10 Doppelhelix...
Vorwort IX Teil I Grundlagen 1. Lebensformen: Zellen mit und ohne Kern... 3 Eukaryoten... 4 Prokaryoten... 6 Literatur... 8 2. DNA: Träger der genetischen Information... 9 Bausteine: Nucleotide... 10 Doppelhelix...
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2014
 Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2014 Fragen für die Übungsstunde 8 (14.07-18.07.) 1) Von der DNA-Sequenz zum Protein Sie können
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2014 Fragen für die Übungsstunde 8 (14.07-18.07.) 1) Von der DNA-Sequenz zum Protein Sie können
Genetik - The Human Genome Project. Überblick über die Genetik. Die gesamte Erbinformation eines Menschen befindet sich in jedem Zellkern
 Genetik - The Human Genome Project Überblick über die Genetik Die gesamte Erbinformation eines Menschen befindet sich in jedem Zellkern seines Körpers. 1 2 Im Organismus müsssen nun ständig Enzyme u. a.
Genetik - The Human Genome Project Überblick über die Genetik Die gesamte Erbinformation eines Menschen befindet sich in jedem Zellkern seines Körpers. 1 2 Im Organismus müsssen nun ständig Enzyme u. a.
Alternative Methoden der RNA-Analyse
 Alternative Methoden der RNA-Analyse In diesem Versuch wurde die Northern Blot Hybridisierung zur Analyse isolierter mrna eingesetzt. Mit dieser Technik können Größe und Menge einer spezifischen RNA bestimmt
Alternative Methoden der RNA-Analyse In diesem Versuch wurde die Northern Blot Hybridisierung zur Analyse isolierter mrna eingesetzt. Mit dieser Technik können Größe und Menge einer spezifischen RNA bestimmt
Versuch 8. Plasmid - Isolierung
 Versuch 8 Plasmid - Isolierung Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@quantentunnel.de X X X C. Weindel & M. Schwarz Wird benotet?: Einleitung Ein Plasmid
Versuch 8 Plasmid - Isolierung Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@quantentunnel.de X X X C. Weindel & M. Schwarz Wird benotet?: Einleitung Ein Plasmid
Transgene Organismen
 Transgene Organismen Themenübersicht 1) Einführung 2) Komplementäre DNA (cdna) 3) Vektoren 4) Einschleusung von Genen in Eukaryontenzellen 5) Ausmaß der Genexpression 6) Genausschaltung (Gen-Knockout)
Transgene Organismen Themenübersicht 1) Einführung 2) Komplementäre DNA (cdna) 3) Vektoren 4) Einschleusung von Genen in Eukaryontenzellen 5) Ausmaß der Genexpression 6) Genausschaltung (Gen-Knockout)
Die Entwicklung der Keimbahn. wahrend der Embryogenese von Caenorhabditis elegans
 Die Entwicklung der Keimbahn wahrend der Embryogenese von Caenorhabditis elegans Von der Fakultat fur Lebenswissenschaften der Technischen Universitat Carolo-Wilhelmina zu Braunschweig zur Erlangung des
Die Entwicklung der Keimbahn wahrend der Embryogenese von Caenorhabditis elegans Von der Fakultat fur Lebenswissenschaften der Technischen Universitat Carolo-Wilhelmina zu Braunschweig zur Erlangung des
Zelluläre Reproduktion: Zellzyklus. Regulation des Zellzyklus - Proliferation
 Zelluläre Reproduktion: Zellzyklus Regulation des Zellzyklus - Proliferation Alle Zellen entstehen durch Zellteilung Der Zellzyklus kann in vier Haupt-Phasen eingeteilt werden Interphase Zellwachstum;
Zelluläre Reproduktion: Zellzyklus Regulation des Zellzyklus - Proliferation Alle Zellen entstehen durch Zellteilung Der Zellzyklus kann in vier Haupt-Phasen eingeteilt werden Interphase Zellwachstum;
Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig)
 Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Jetzt liegen diese Stränge einzeln
Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Die doppelsträngige Helix wird zunächst aufgetrennt. Enzym: Helicase (ATP-abhängig) Jetzt liegen diese Stränge einzeln
Einführung Nukleinsäuren
 Einführung Nukleinsäuren Dr. Kristian M. Müller Institut für Biologie III Albert-Ludwigs-Universität Freiburg Einführung 1. Semester, WiSe 2007/2008 Historischer Überblick Literatur Bilder aus: Taschenatlas
Einführung Nukleinsäuren Dr. Kristian M. Müller Institut für Biologie III Albert-Ludwigs-Universität Freiburg Einführung 1. Semester, WiSe 2007/2008 Historischer Überblick Literatur Bilder aus: Taschenatlas
Im Folgenden wird Ihnen an einem Beispiel erklärt, wie Sie Excel-Anlagen und Excel-Vorlagen erstellen können.
 Excel-Schnittstelle Im Folgenden wird Ihnen an einem Beispiel erklärt, wie Sie Excel-Anlagen und Excel-Vorlagen erstellen können. Voraussetzung: Microsoft Office Excel ab Version 2000 Zum verwendeten Beispiel:
Excel-Schnittstelle Im Folgenden wird Ihnen an einem Beispiel erklärt, wie Sie Excel-Anlagen und Excel-Vorlagen erstellen können. Voraussetzung: Microsoft Office Excel ab Version 2000 Zum verwendeten Beispiel:
Transkription 3. Teil. Posttranskriptionale Modifikationen
 Transkription 3. Teil Posttranskriptionale Modifikationen Gliederung des Vortrags 1. Reifung der t-rna 2. Modifikationen der Prä-mRNA 5 Capping 3 Schwanzbildung RNA-Editing Spleißen Alternatives Spleißen
Transkription 3. Teil Posttranskriptionale Modifikationen Gliederung des Vortrags 1. Reifung der t-rna 2. Modifikationen der Prä-mRNA 5 Capping 3 Schwanzbildung RNA-Editing Spleißen Alternatives Spleißen
Frage 1 A: Wieviele Codone des "Universellen genetisches Codes" kodieren:
 Frage 1 A: Wieviele Codone des "Universellen genetisches Codes" kodieren: Aminosäuren Translationsstart Translationsstop? B: Welche biochemische Reaktion wird von Aminoazyl-tRNA-Synthetasen katalysiert?
Frage 1 A: Wieviele Codone des "Universellen genetisches Codes" kodieren: Aminosäuren Translationsstart Translationsstop? B: Welche biochemische Reaktion wird von Aminoazyl-tRNA-Synthetasen katalysiert?
Zentrales Dogma der Biochemie Zyklus eines Retrovirus Der Fluss der genetischen Information verläuft von der DNA zur RNA zum Protein. Zumindest bis 19
 Unterschiede DNA < > RNA Posttranskriptionale Veränderungen EML BIORUNDE DNA/RNA II Zentrales Dogma der Biochemie Der Fluss der genetischen Information verläuft von der DNA zur RNA zum Protein. Outline
Unterschiede DNA < > RNA Posttranskriptionale Veränderungen EML BIORUNDE DNA/RNA II Zentrales Dogma der Biochemie Der Fluss der genetischen Information verläuft von der DNA zur RNA zum Protein. Outline
Eva Douma: Die Vorteile und Nachteile der Ökonomisierung in der Sozialen Arbeit
 Eva Douma: Die Vorteile und Nachteile der Ökonomisierung in der Sozialen Arbeit Frau Dr. Eva Douma ist Organisations-Beraterin in Frankfurt am Main Das ist eine Zusammen-Fassung des Vortrages: Busines
Eva Douma: Die Vorteile und Nachteile der Ökonomisierung in der Sozialen Arbeit Frau Dr. Eva Douma ist Organisations-Beraterin in Frankfurt am Main Das ist eine Zusammen-Fassung des Vortrages: Busines
Eine neue RNA-Welt. Uralte RNA-Welt Am Anfang der Entstehung des Lebens. Bekannte RNA-Welt Protein-Synthese. Neue RNA-Welt Regulatorische RNA-Moleküle
 RNAs Eine neue RNA-Welt 1. Uralte RNA-Welt Am Anfang der Entstehung des Lebens Bekannte RNA-Welt Protein-Synthese Neue RNA-Welt Regulatorische RNA-Moleküle 2. Eine neue RNA-Welt die Anzahl der nicht-kodierenden
RNAs Eine neue RNA-Welt 1. Uralte RNA-Welt Am Anfang der Entstehung des Lebens Bekannte RNA-Welt Protein-Synthese Neue RNA-Welt Regulatorische RNA-Moleküle 2. Eine neue RNA-Welt die Anzahl der nicht-kodierenden
DNA-Replikation. Konrad Beyreuther. Stefan Kins
 DNA-Replikation Konrad Beyreuther Stefan Kins DNA-Replikation Originalgetreue Verdopplung des genetischen Materials als Voraussetzung für die kontinuierliche Weitergabe der in der DNA verschlüsselten Information
DNA-Replikation Konrad Beyreuther Stefan Kins DNA-Replikation Originalgetreue Verdopplung des genetischen Materials als Voraussetzung für die kontinuierliche Weitergabe der in der DNA verschlüsselten Information
Professionelle Seminare im Bereich MS-Office
 Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Das Leitbild vom Verein WIR
 Das Leitbild vom Verein WIR Dieses Zeichen ist ein Gütesiegel. Texte mit diesem Gütesiegel sind leicht verständlich. Leicht Lesen gibt es in drei Stufen. B1: leicht verständlich A2: noch leichter verständlich
Das Leitbild vom Verein WIR Dieses Zeichen ist ein Gütesiegel. Texte mit diesem Gütesiegel sind leicht verständlich. Leicht Lesen gibt es in drei Stufen. B1: leicht verständlich A2: noch leichter verständlich
DNA-Replikation. Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination
 DNA-Replikation Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination Die Initiation der DNA-Replikation bei Eukaryoten am ori erfolgt erst nach der Lizensierung durch ORC und weitere Proteine
DNA-Replikation Ein Prozess in drei Stufen 1. Initiation 2. Elongation 3. Termination Die Initiation der DNA-Replikation bei Eukaryoten am ori erfolgt erst nach der Lizensierung durch ORC und weitere Proteine
Zellzyklus, Replikation und Chromosomen
 Zellzyklus, Replikation und Chromosomen Wiederholung: Größenverhältnisse im DNA-Molekül 3 5 Das größte menschliche Chromosom enthält 247 Millionen Basenpaare Moleküllänge: 8.4 cm Die Länge des gesamten
Zellzyklus, Replikation und Chromosomen Wiederholung: Größenverhältnisse im DNA-Molekül 3 5 Das größte menschliche Chromosom enthält 247 Millionen Basenpaare Moleküllänge: 8.4 cm Die Länge des gesamten
Zeichen bei Zahlen entschlüsseln
 Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Thema Transkription und Genregulation Erwin R. Schmidt Institut für Molekulargenetik Gentechnologische Sicherheitsforschung & Beratung
 Thema Transkription und Genregulation 21.12.2012 Erwin R. Schmidt Institut für Molekulargenetik Gentechnologische Sicherheitsforschung & Beratung Thema: Gene und Transkription Was ist ein Gen? Heute: Gendefinition:
Thema Transkription und Genregulation 21.12.2012 Erwin R. Schmidt Institut für Molekulargenetik Gentechnologische Sicherheitsforschung & Beratung Thema: Gene und Transkription Was ist ein Gen? Heute: Gendefinition:
