Kinetische und einzelmolekülspektroskopische Untersuchungen an H + -ATPsynthasen aus Escherichia coli und Saccharomyces cerevisiae
|
|
|
- Julius Gerstle
- vor 6 Jahren
- Abrufe
Transkript
1 Kinetische und einzelmolekülspektroskopische Untersuchungen an H + -ATPsynthasen aus Escherichia coli und Saccharomyces cerevisiae Dissertation zur Erlangung der Doktorwürde der Fakultät für Chemie und Pharmazie der Albert-Ludwigs-Universität Freiburg im Breisgau vorgelegt von Ilka Starke aus Dresden 2014
2
3
4 Vorsitzender des Promotionsausschusses: Referent: Korreferent: Prof. Dr. S. Weber Prof. Dr. P. Gräber Prof. Dr. T. Koslowski Tag der Bekanntgabe des Prüfungsergebnisses:
5 Publikationen I. Starke, S. Osterwald, P. Gräber und J. Petersen (2012). The H + -ATPsynthase from Yeast: Construction and Characterisation of an EGFP-MF 0 F 1 Fusion Protein with Cysteine Mutations for FRET Investigations. BBA 1817, (8-28):23-24 I. Starke, S. Loschonsky, S. Fischer und P. Gräber (2010). Isolation of the H + -ATPsynthase from E. coli and the stability of its subcomplexes. BBA 1797, (26-40):38 Vorträge I. Starke: In vitro Aktivitätsmessungen an H + -ATPsynthasen Seminar der Hermann-Staudinger-Graduiertenschule in St. Märgen (2011) I. Starke: ΔpH induzierte Fluoreszenzlöschung von 9-Amino-6-chloro-2-methoxyacridin Am Institut für Physikalische Chemie der Albert-Ludwigs-Universität Freiburg (2009) Posterpräsentationen I. Starke, S. Osterwald und J. Petersen (2012). The H + -ATPsynthase from Yeast: Construction and Characterisation of an EGFP-MF 0 F 1 Fusion Protein with Cysteine Mutations for FRET Investigations. 3 th International Workshop on Expression, Structure and Function of Membrane Proteins (Florenz, Italien) I. Starke, S. Osterwald und J. Petersen (2012). The H + -ATPsynthase from Yeast: Construction and Characterisation of an EGFP-MF 0 F 1 Fusion Protein with Cysteine Mutations for FRET Investigations. 17 th European Bioenergetics Conference (Freiburg im Breisgau, Deutschland) I. Starke, S. Loschonsky, S. Fischer und P. Gräber (2010). Isolation of the H + -ATPsynthase from E. coli and the stability of its subcomplexes. 16 th European Bioenergetics Conference (Warschau, Polen)
6
7 Inhaltsverzeichnis Abkürzungen...VII Zusammenfassung... XI 1 Einleitung Die H + -ATPsynthase Die H + -ATPsynthase aus Escherichia coli Die H + -ATPsynthase aus Saccharomyces cerevisiae Katalysemechanismus der F-Typ H + -ATPsynthase Fluoreszierende Proteine Fluoreszenzspektroskopie Konfokales Fluoreszenzmikroskop Fluoreszenz-Korrelations-Spektroskopie (FCS) Fluoreszenz-Resonanz-Energie-Transfer (FRET) Ziel der Arbeit Material Geräte Chemikalien Verwendete Zellstämme Biochemische Methoden Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Anzucht von E. coli Isolation von EF 0 F 1 aus E. coli Isolation und Reinigung von EF 0 F 1 durch Chromatographie Isolation und Reinigung von EF 0 F 1 durch Dichtegradientenzentrifugation Isolation der H + -ATPsynthase aus Saccharomyces cerevisiae (YF 0 F 1 ) Anzucht von S. cerevisiae Isolation von YF 0 F 1 aus S. cerevisiae I
8 Inhaltsverzeichnis 3.3 Fluoreszenzmarkierung Markierung von YF 0 F 1 aus YEBTA-c-γ1-b-K174C mit ATTO Konzentrationsbestimmungsmethoden Konzentrationsbestimmung mit UV/VIS-Spektroskopie Konzentrationsbestimmung von Proteinen mit einem Bicinchoninsäure-Test Konzentrationsbestimmung von Proteinen mit Amidoschwarz Vergleich der Proteinkonzentrationen Polyacrylamidgelelektrophorese (SDS-PAGE) Gelelektrophorese Färbung mit Coomassie Blau Färbung mit kolloidalem Silber Darstellung von Dialyseliposomen Rekonstitution von F 0 F 1 in Dialyseliposomen Messung der ATP-Synthese Kalibrierung der ATP-Synthesemessung ATP-Synthesemessung Optimierung der Reaktionsbedingungen bei der Messung der Phosphatabhängigkeit der H + -ATPsynthase Messung der ATP-Hydrolyse Messung der ATP-Hydrolyse nach der One-step-Methode Messung der mit dem Protonentransport gekoppelten ATP-Hydrolyse anhand der Fluoreszenzlöschung von 9-Amino-6-Chloro-2-methoxyacridin (ACMA) Kalibrierung der ACMA-Fluoreszenzlöschung Fluoreszenzspektroskopische Methoden Messung von Fluoreszenzanregungs- und Fluoreszenzemissionsspektren Total-Reflexions-Fluoreszenz-Mikroskopie (TIRF) Fluoreszenz-Resonanz-Energie-Transfer-Messungen an Einzelmolekülen (FRET) Justage des konfokalen Mikroskops Einzelmolekülmessungen an H + -ATPsynthasen Einzelmolekülmessungen in Gegenwart von Nukleotiden Einzelmolekülmessungen an mit NaN 3 und ADP inhibierten H + -ATPsynthasen 69 Auswertung der Einzelmolekülmessungen II
9 Inhaltsverzeichnis 5 Die H + -ATPsynthase aus Escherichia coli Anzucht von E. coli Isolation der H + -ATPsynthase aus E. coli Isolation durch Ionenaustausch-/Größenausschlusschromatographie Isolation durch Dichtegradientenzentrifugation Fazit Fluoreszenzlöschung von ACMA in Dialyseliposomen Quantitative Auswertung durch Kalibrierung der ACMA-Fluoreszenzlöschung Qualitative Auswertung der ACMA-Fluoreszenzlöschung Bestimmung des K M -Wertes von Phosphat Michaelis-Menten-Kinetik Bestimmung der Michaelis-Menten-Konstante von Phosphat Bestimmung der Michaelis-Menten-Konstante von Dihydrogenphosphat (H 2 PO 4 ) Diskussion Die H + -ATPsynthase aus Saccharomyces cerevisiae Der S. cerevisiae-stamm YEBTA-c-γ1-b-K174C Anzucht von S. cerevisiae Isolation der H + -ATPsynthase aus S. cerevisiae Untersuchung unbekannter Proteine mittels HPLC-MS Disskusion Fluoreszenzspektroskopische Charakterisierung Fluoreszenzmarkierung von F 0 b Syntheseaktivität nach der Markierung Bestimmung des Försterradius des Fluorophorenpaars EGFP/ATTO Bestimmung des Korrekturfaktors γ Bestimmung der Fluoreszenzquantenausbeute von ATTO Bestimmung der Detektionseffizienzen Berechung des Korrekturfaktors γ Ensemble Fluoreszenzspektroskopie Test auf Anwesenheit eines aktiven FRET-Systems Test auf konformative Änderung durch Zugabe von AMPPNP Diskussion Einzelmolekül-FRET-Spektroskopie Auswertung der Einzelmolekülmessungen III
10 Inhaltsverzeichnis Korrektur der Fluoreszenzintensitäten Bestimmung der FRET-Effizienz Auswahlkriterien und Klassifizierung der Photonenbursts Messung unter Hydrolysebedingungen FRET-Messungen unter nicht katalytischen Bedingungen (AMPPNP) FRET-Messungen an inhibierten H + -ATPsynthasen (NaN 3 und ADP) Vergleich der FRET-Daten mit einem Homologiemodell Ausblick Literaturverzeichnis 147 A Die H + i Konzentrationsbestimmung mit der Aminmethode 167 B Einheiten der Fluoreszenzspektroskopie 171 B.1 Einheit der Fluoreszenz B.2 Einheit des Überlappungsintegrals B.3 Einheit des Försterradius C Präparation von submitochondrialen Partikeln aus Rinderherz 175 D Justage des konfokalen Fluoreszenzmikroskops 179 Danksagung 186 IV
11
12
13 Abkürzungen Chemikalien und Proteine ADP Adenosin-5 -diphosphat Amp Ampicillin AMPPNP Adenosin-5 -(β, γ-imido)triphosphat APS Ammoniumperoxodisulfat ATP Adenosin-5 -triphosphat BCA Bicinchoninsäure Bicin (N,N-Bis-(2-hydroxyethyl)-glycin) BPB Bromphenolblau Cam Chloramphenicol CBB Coomassie Brillant Blue DDM n-dodecyl-β-maltosid DHB Dihydroxybenzoesäure DMSO Dimetylsulfoxid DTT 1,4-Dithiothreit E. coli Escherichia coli EDTA Ethylendiamin-N,N,N,N -tetraessigsäure, Dinatriumsalz EF 0 F 1 H + -ATPsynthase aus Escherichia coli EGFP verbessertes grün fluoreszierendes Protein (Enhanced Green Fluorescence Protein) EPA Phosphatidsäure EPC Phosphatidylcholin EYFP verbessertes gelb fluoreszierendes Protein (Enhanced Yellow Fluorescence Protein) F 0 -Teil membranintegraler, hydrophober Teil der H + - ATPsynthase F 1 -Teil peripherer, hydrophiler Teil der H + -ATPsynthase G-418 Geneticin GFP grün fluoreszierendes Protein (Green Fluorescence Protein) HEPES N-(2-Hydroxyethl)piperazin-N -(2-ethansulfonsäure) Kan Kanamycin VII
14 LDAO N,N-Dimethyldodecylamin-N-oxid LDH Lactat-Dehydrogenase MES 2-(N-Morpholino)ethansulfonsäure MF 0 F 1 H + -ATPsynthase aus Rinderherz (Mammalian) MOPS 3-(N-Morpholino)propansulfonsäure NAD + Nicotinamid-Adenin-Dinucleotid (oxidiert) NADH Nicotinamid-Adenin-Dinucleotid (reduziert) PABA Paraaminobenzoesäure PEG Polyethylenglycol PEP Phosphoenolpyruvat P i PK PMSF (anorganisches) Phosphat Pyruvat-Kinase Phenylmethylsulfonylfluorid S. cerevisiae Saccharomyces cerevisiae SDS Natriumdodecylsulfonylfluorid TEMED N,N,N,N -Tetrametyletylendiamin Tet Tetracyclin TEV Tabakätzvirus (Tobacco etch virus) Tricin N-[Tris-(hydroxymethyl)-methyl]-glycin Tris Tris(hydroxymethyl)-aminomethan Triton X-100 t-octylphenoxypolyethoxxyethanol YF 0 F 1 H + -ATPsynthase aus Saccharomyces cerevosoae (Yeast) YFP gelb fluoreszierendes Protein (Yellow Fluorescence Protein) zip-helix Leucin-Zipper Helix Geräte-Abkürzungen, technische und physikalische Größen Å Ångström (1 Å = m) APD Avalanche Photo Diode CCD-Kamera Charge-coupled device-kamera ΔΦ transmembrane elektrische Potentialdifferenz ΔpH transmembrane ph-differenz Δ μ H + transmembrane elektrochemische Potentialdifferenz der Protonen VIII
15 FCS Fluoreszenzkorrelationsspektroskopie FRET Fluoreszenz-Resonanz-Energie-Transfer g Erdbeschleunigung (1 g = 9,81 m s 1 ) GPC Gelpermeationschromatographie I A I D kbp MWCO PAGE rpm RT UV Fluoreszenzintensität des Akzeptorfluorophors Fluoreszenzintensität des Donorfluorophors Kilobasen (1000 Basenpaare) Molekularmassen-Trenngrenze (Molecular Weight Cutoff ) Polyacrylamid-Gelelektrophorese Umdrehungen pro Minute (rounds per minute) Raumtemperatur Fluoreszenzlebensdauer Ultraviolettes Licht (λ < 400 nm) VIS sichtbarer Wellenlängenbereich des Lichts (visible, λ nm) v/v Volumenanteil w/v Masse pro Volumen verwendete Ausdrücke BioBeads Photonenburst Rate SMP Turnover Vortexen Polystyrolkügelchen große Anhäufung von Photonen Geschwindigkeit submitochondriale Partikel Wechselzahl (umgesetztes Substrat pro Sekunde und Enzym) mit einem Rüttler für kurze Zeit stark durchrütteln IX
16 X
17 Zusammenfassung H + -ATPsynthasen sind zentrale Enzyme des Energiestoffwechsels in allen Zellen. Es sind membranintegrierte Enzyme, die in den Plasmamembranen der Bakterien, den Thylakoidmembranen der Chloroplasten und den inneren Mitochondrienmembranen vorkommen. Sie koppeln den transmembranen Protonentransport mit der Synthese von ATP aus ADP und anorganischem Phosphat und stellen somit die Energieversorgung der Zellen sicher. Um die Kenntnis der Funktion dieser Enzyme zu erweitern, wurden in dieser Arbeit enzymkinetische Untersuchungen an der H + -ATPsynthase aus Escherichia coli und einzelmolekülspektroskopische Arbeiten am Enzym aus Saccharomyces cerevisiae durchgeführt. H + -ATPsynthasen aus E. coli wurden aus zwei verschiedenen Stämmen (RA1/pRA100 und DK8/pBWU13) isoliert und mit zwei unterschiedlichen Methoden gereinigt (Ionenaustausch- Chromatographie, Dichtegradientenzentrifugation). Zum Vergleich der Methoden wurde die Reinheit der verschiedenen Fraktionen mit Hilfe der SDS-Gel-Chromatographie bestimmt und deren Synthese- und Hydrolyseaktivität gemessen. Zur Untersuchung der Syntheseaktivität wurde das Enzym in Liposomen rekonstituiert. Mit Hilfe eines Säure-Base-Sprungs wurde eine transmembrane elektrochemische Potentialdifferenz der Protonen (ΔpH und Δϕ) erzeugt und die Anfangsgeschwindigkeit der ATP-Synthese luminometrisch mit Luciferin/Luciferase gemessen. Die Geschwindigkeit der ATP-Hydrolyse wurde zum einen mit Pyruvat/NADH gemessen, zum anderen wurde die ACMA (9-Amino-6-Chloro-2-methoxyacridin) Methode etabliert, um den mit der ATP-Hydrolyse gekoppelten Aufbau der transmembranen ph-differenz zu messen. Die höchste ATP-Syntheseaktivität (100 s 1 ) wurde mit dem Enzym aus dem Stamm DK8/pBWU13 beobachtet. Mit beiden Reinigungsmethoden wurde festgestellt, dass die höchsten Syntheseaktivitäten in anderen Fraktionen als die höchsten Hydrolyseaktivitäten auftreten, obwohl die Zusammensetzungen der Untereinheiten dieser Fraktionen im SDS-Gel keine signifikanten Unterschiede aufwiesen. Bei der Messung der ATP-Synthese liegen sowohl die Nukleotide als auch Phosphat in verschiedenen Protonierungszuständen und Mg 2+ -Komplexen vor. Um die Phosphatspezies zu identifizieren, die an das Enzym bindet, wurde die Abhängigkeit der Syntheseaktivität von der Phosphatkonzentration bei verschiedenen ph-werten untersucht. Die Phosphatabhängigkeit lässt sich bei den untersuchten ph-werten zwischen 7,3 und 8,8 jeweils mit einer Michaelis- Menten Kinetik beschreiben, die K M Werte variieren zwischen 0,09 mm und 1,76 mm. Berechnet man aus der Einwaagekonzentration des Phosphats den Anteil der Spezies H 2 PO 4 bei dem jeweiligen ph-wert aus und trägt die Daten gegen die Konzentration dieser Spezies auf, lassen sich alle Daten mit einem K M -Wert (K M = (30 ± 5) μm) beschreiben. Daraus wird geschlossen, dass diese Spezies an das Enzym bindet. XI
18 Die Kopplung des Protonentransports durch das Enzym mit der ATP-Synthese erfolgt nach den derzeitigen Vorstellungen durch Rotation der Rotor-Untereinheiten (γɛc 10 ) relativ zu den Stator-Untereinheiten (α 3 β 3 δab 2 ). Dies konnte mit Hilfe der Einzelmolekülspektroskopie an bakteriellen Enzymen (PS3 und E. coli) gezeigt werden, jedoch ist dies bislang mit mitochondrialen Enzymen nicht gelungen. In unserer Arbeitsgruppe (Doktorarbeit von J. Petersen, Diplomarbeit von S. Osterwald) wurde eine Mutante der mitochondrialen H + -ATPsynthase aus Saccharomyces cerevisiae molekularbiologisch hergestellt, die für fluoreszenzspektroskopische Einzelmolekül- Messungen geeignet ist (YEBTA-c-γ1-b-K174C). In diesem Enzym ist ein enhanced green fluorescent protein (EGFP) über einen Linker an die γ-untereinheit (Rotor) fusioniert und in der b-untereinheit wurde die Mutation b-k174c eingeführt. Dieses Fusionsprotein wurde isoliert und seine Eigenschaften mit dem entsprechenden Wildtyp und der Doppelcysteinmutante YRD15-b-K174C-γ-C84S/T121C verglichen. Das SDS-Gel zeigt die Abwesenheit der γ- Untereinheit und das Auftreten einer neuen Bande bei 66 kda, die der Masse des γ-egfp Fusionsproteins entspricht. Nach Rekonstitution in Liposomen zeigt das Fusionsprotein eine ATP-Syntheseaktivität von 5 s 1 d.h. 15 % der Aktivität des entsprechenden Wildtyps. Um Abstandsänderungen bei der Rotation zu untersuchen, wurde das Fusionsprotein mit ATTO565 am Cystein b-k174c markiert. In diesem System ist nach Anregung des EGFPs ein Fluoreszenz-Resonanz-Energie-Transfer (FRET) möglich, dessen Größe vom Abstand der beiden Fluorophore abhängt. Die Fluoreszenzemissionsspektren zeigen die Anwesenheit von EGFP und ATTO565 im markierten Fusionsprotein und zeigen einen FRET zwischen beiden Fluorophoren an. Die Bestimmung des Überlappungsintegrals und der Fluoreszenzquantenausbeute des gebundenen EGFPs ergab einen Försterradius von 55 Å. Für die Einzelmolekül- FRET-Messungen wurden in Liposomen rekonstituierte H + -ATPsynthasen (YEBTA-c-γ1-b- K174C-ATTO565) verwendet. Das gebundene EGFP (Donor) wurde mit einem Laser bei 476 nm angeregt und die Fluoreszenz von Donor und ATTO565 (Akzeptor) bei der Diffusion einzelner Liposomen durch das konfokale Volumen gemessen ( Burst ). Aus den korrigierten Fluoreszenzzeitspuren von Donor und Akzeptor wurden die FRET-Effizienzen und aus diesen die Donor-Akzeptor-Abstände berechnet. Bursts mit konstanter FRET-Effizienz wurden inaktiven Enzymen, Bursts mit Änderungen in der FRET-Effizinezen aktiven Enzymen zugeordnet. Bei aktiven Enzymen wurden drei verschiedene Abstände zwischen EGFP und ATTO565 bestimmt. Der Vergleich mit einem Molekülmodell des doppeltmarkierten Enzyms ergab, dass diese Abstände anhand der Rotation der γ-untereinheit mit dem EGFP um 120 Schritten beschrieben werden können. Aus den Daten der FRET-Stufen wurde eine Hydrolyseaktivität der aktiven Enzyme von 63 s 1 ermittelt. Dieses System ist geeignet, die Rotation der γ- Untereinheit bei der Katalyse in mitochondrialen H + -ATPsynthasen zu untersuchen. XII
19 Einleitung
20
21 1 Einleitung 1.1 Die H + -ATPsynthase H + -ATPsynthasen des F-Typs (EC ) koppeln die Synthese von ATP aus ADP und anorganischem Phosphat mit einem transmembranen Protonentransport. ADP + P i + H + in ATP + H 2 O + H + out Sie kommen in allen lebenden Organismen vor, da sie eine zentrale Rolle im Energiehaushalt der Zelle spielen. Eine durch die Atmungskette aufgebaute transmembrane ph-differenz [1] liefert die Triebkraft für die Synthese. In Struktur und Funktion zeigen die ATPsynthasen aus allen Organismen große Analogien [2 6]. Erste grundlegende Kenntnisse der Teilstrukturen wurden durch die Arbeitsgruppe von Walker 1994 [7] durch Kristallisation eines Teils des Komplexes aus Rinderherz gewonnen (Nobelpreis 1997). In Abbildung 1.1 ist schematisch das einfachste dieser Enzyme, die H + -ATPsynthase aus Escherichia coli (E. coli), dargestellt. ATP Stator H + b 2 ADP + P i Rotor a c 10 Abbildung 1.1: Schematische Darstellung der F-Typ H + -ATPsynthasen. Hier beispielhaft gezeigt für die H + -ATPsynthase aus E. coli. Die c-untereinheiten (L bei Na + -ATPsynthasen) bilden ein ringförmiges Oligomer, dessen Stöchiometrie je nach Organismus variiert. Die folgenden Stöchiometrien wurden gefunden; H + 3
22 1 Einleitung für die H + -ATPsynthasen aus Rinderherz n = 8 [8], für die H + -ATPsynthase aus E. coli und Mitochondrien aus Saccharomyces cerevisiae n = 10 [9, 10], für Na + -ATPsynthasen aus Ilyobacter tartaricus n = 11 [11], für H + -ATPsynthasen aus Thermus thermophilus n=12[12] und für H + -ATPsynthasen aus Chlorophlasten n=14[13].derc-ring ist in der Membran lokalisiert und ist mit weiteren membranständigen Untereinheiten für die Translokation von Protonen über die Membran verantwortlich. Eine c-untereinheit ist aus zwei hydrophoben α-helices aufgebaut [14 17], sie bilden zusammen eine Haarnadelstruktur, welche durch eine polare loop-region verbunden ist, die aus der Membran herausragt [18]. Die Zahl der Protonen, die pro gebildetem ATP über die Membran transportiert wird, hängt wiederum vom Organismus ab, so wurde für die H + -ATPsynthase aus Chloroplasten ein H + /ATP-Verhältnis von 4, für die aus Hefe von 3 bestimmt [19]. Der membranintegrierte hydrophobe F 0 -Teil ist mit dem hydrophilen F 1 -Teil durch zwei Verbindungsstiele verknüpft. Zum einen durch einen peripheren Stiel (b 2 -Untereinheiten), zum anderen durch einen zentralen Stiel (γ-untereinheit). Mit dem zentralen Stiel wird die Bewegung des c-rings auf die γ-untereinheit übertragen und dies führt zu Konformationsänderungen an den katalytischen Untereinheiten [20 22]. Der periphere Stiel dient der Aufnahme der mechanischen Kräfte, die bei der Rotation auftreten [23]. An dem zentralen Stiel sind je nach Organismus weitere Untereinheiten gebunden, die vor allem regulatorische Aufgaben in der Drehbewegung übernehmen [24]. Der zentrale Stiel bewegt sich im Zentrum der ringförmig und alternierend angeordneten katalytischen und nichtkatalytischen Untereinheiten (α 3 β 3 -Hexamer) [25]. An den Grenzflächen zwischen den α- und β-untereinheiten befinden sich die Nukleotidbindungsplätze. Die Synthese bzw. Hydrolyse von ATP findet an den Bindungsplätzen statt, die hauptsächlich auf den β-untereinheiten lokalisiert sind [26]. Auch an den nicht-katalytischen Bindungsplätzen, die sich auf den α- Untereinheiten befinden, können Nukleotide binden, diese werden aber im Katalysezyklus nicht direkt umgesetzt. Die Funktion der nicht-katalytischen Bindungsplätze ist noch nicht endgültig geklärt [27] Die H + -ATPsynthase aus Escherichia coli Zu den am besten untersuchten Modellorganismen gehört das gramnegative, stabförmige und rund um die Zelle begeißelte Bakterium Escherichia coli (E. coli). Es kommt im menschlichen und tierischen Darm vor, sein Genom ist vollständig sequenziert und zum Großteil charakterisiert. Es kann zusätzlich zu seinem ringförmigen Chromosom (ca kb) noch weitere ringförmige, doppelsträngige DNA-Moleküle (Plasmide) enthalten. Dies ermöglicht die Verwendung von E. coli zur Herstellung von rekombinanten Proteinen (z.b. Insulin) und für molekularbiologische Arbeiten unter Verwendung von Stämmen, die idealerweise das zu 4
23 1.1 Die H + -ATPsynthase untersuchende Protein überexprimieren. Die H + -ATPsynthase aus E. coli besteht aus acht verschiedenen Untereinheiten mit einer Stöchiometrie von ab 2 c 10 α 3 β 3 γδɛ (molekulare Masse 531 kda). Die Untereinheiten können weiter zusammen gefasst werden, entweder strukturell in den hydrophoben, membranständigen F 0 -Teil (ab 2 c 10 ) und den hydrophilen, peripheren F 1 -Teil (α 3 β 3 γδɛ), oder nach ihrer Funktion in den beweglichen Rotor (c 10 γɛ) und den unbeweglichen Stator (ab 2 α 3 β 3 δ). Für kinetische Untersuchungen in Ensemble- und Einzelmolekülmessungen ist die Unterscheidung in Rotor und Stator von Bedeutung. Ein Strukturmodell ist in Abbildung 1.2 gezeigt, die Untereinheiten, welche zum Rotor gehören sind in Blau und Grün, die die zum Stator gehören in Rot- und Gelbtönen dargestellt (schematische Darstellung siehe auch Abbildung 1.1). Für E. coli wurde b F 1 F 0 c 10 * a Abbildung 1.2: Strukturmodell der H + -ATPsynthase aus E. coli. Das Homologiemodell wurde erstellt aus pdb-files von Rombach-Riegraf 2009 [28]. Die mit * gekennzeichneten Untereinheiten sind Teil des Rotors der H + -ATPsynthase. für den c-ring eine Stöchiometrie von n = 10 [9, 29] gefunden, der Protonentransport über die Membran erfolgt durch Bindung des Protons an die Aminosäure Asp 61 [30], welche in der Mitte der Membran lokalisiert ist. Der Zugang zu dieser wird durch die a-untereinheit ermöglicht. Deren räumliche Struktur ist noch nicht bekannt [31], man geht aber davon aus, dass sie aus 5 Transmembranhelices besteht die einen Halbkanal für die Zuführung und einen Halbkanal für den Austritt der Protonen bilden [32]. Die a-untereinheit interagiert über 2 ihrer Helices direkt mit dem c-ring [33, 34], gleichzeitig grenzt diese Untereinheit auch an 5
24 1 Einleitung das membranständige N-terminale Ende der b-untereinheiten, was zu einer Stabilisierung der Struktur führt [35, 36]. Die α-helikale Struktur [37, 38] der beiden b-untereinheiten bildet zusammen eine coiled-coil -Struktur [39] und stellt zusammen mit der δ-untereinheit den peripheren Stiel der H + -ATPsynthase aus E. coli dar. Die δ-untereinheit liegt oberhalb des α 3 β 3 -Hexamers, ihre Wechselwirkung mit dem α 3 β 3 -Hexamer bzw. mit der b 2 -Untereinheit ist noch nicht vollständig geklärt [40, 41]. Bei der H + -ATPsynthase aus E. coli besteht der zentrale Stiel aus der γ-untereinheit und der ɛ-untereinheit [42, 43]. Die ɛ-untereinheit reguliert [24] die Katalyse. Bei der Synthese interagiert sie mit dem α 3 β 3 -Hexamer und lässt nur eine Drehrichtung zu [44]. Bestehend aus drei Domänen, zwei α-helices und einem β- Faltblatt, wurden für die ɛ-untereinheit drei verschiedene Konformationen gefunden, die mit den regulatorischen Aufgaben zusammen hängen [45 48] Die H + -ATPsynthase aus Saccharomyces cerevisiae Saccharomyces cerevisiae (S. cerevisiae), auch Bäcker- oder Bierhefe ist ein eukaryotischer Einzeller, der zur Gattung der Pilze gehört. Aufgrund der einfachen Kultivierbarkeit und der starken Verwandtschaft mit anderen eukaryotischen Zellen in der Pflanzen- und Tierwelt, eignet er sich als Modellorganismus, da die Ergebnisse sich auch auf höhere Organismen übertragen lassen. Die Aminosäuresequenz des Genoms von S. cerevisiae ist vollständig ermittelt, dadurch lassen sich gezielte molekularbiologische Veränderungen vornehmen. Der Stoffwechsel ist fakultativ aerob, wodurch sich S. cerevisiae besonders für Untersuchungen an den Enzymkomplexen der mitochondrialen Atmungskette eignet. Die Mitochondrien kommen im Cytosol von Eukaryoten vor und ihre Anzahl richtet sich nach dem Energiebedarf der Zelle (z.b in Leberzellen [49], in Eizellen [50]). Aufgrund ihrer Größe von 1 μm und ihres Aufbaus wird nach der Endosymbiontentheorie davon ausgegangen, dass sie aus Bakterien hervorgegangen sind [51 53]. Dafür sprechen der Besitz einer eigenen mitochondrialen DNA, eine eigene Proteinsynthese, Fortpflanzung durch Zellteilung (Mitose) und die innere Membran, die sich deutlich von der äußeren Zellmembran unterscheidet. Die innere Membran ist für Proteine und Ionen weitgehend undurchlässig und fasst den mitochondrialen Matrixraum ein. Der Austausch von Proteinen und Molekülen erfolgt über selektive Transportwege [54]. In der inneren Membran sind auch die Enzyme der Atmungskette lokalisiert. Das bei der Glykolyse oder beim Citrat-Zyklus reduzierte NADH überträgt an der inneren Mitochondrienmembran Elektronen über die Enzymkomplexe der Elektronentransportkette auf Sauerstoff, dabei werden Protonen aus der Matrix in den Raum zwischen der inneren und äußeren Membran transportiert. Es kommt zum Aufbau einer Energetisierung der inneren Membran durch die transmembrane elektrochemische Potentialdifferenz der Protonen (Δ μ H +), die von der H + -ATPsynthase zur 6
25 1.1 Die H + -ATPsynthase Synthese von ATP genutzt wird. Die mitochondriale H + -ATPsynthase ist deutlich komplexer aufgebaut als die bakterielle H + - ATPsynthase. Sie hat eine molekulare Masse von ca. 600 kda und besteht aus vermutlich 17 Untereinheiten [55]. Wie auch bei der bakteriellen H + -ATPsynthase können die Un- F 0 Stator H + f d H 5 ATP g J 4 a ADP + P i Rotor Matrixraum F 1 Innere Mitochondrienmembran e Intermembranraum Abbildung 1.3: Schematische Darstellung eines Mitochondriums und der H + - ATPsynthase aus S. cerevisiae. Links: Schematische Darstellung eines Mitochondriums, Bild aus commons/3/3b/animal mitochondrion diagram en.svg Datum: Rechts: Schematische Darstellung der H + -ATPsynthase aus S. cerevisiae. H + tereinheiten des Komplexes aus S. cerevisiae nach ihrer Struktur oder nach ihrer Funktion eingeteilt werden. Zu dem hydrophoben, membranständigen F 0 -Teil zählen die Untereinheiten a, d, 4 (b), 9 10, 8, e, f, g, H, J und K und zu dem hydrophilen, peripheren F 1 -Teil die Untereinheiten α 3, β 3, γ, 5 (OSCP), δ und ɛ. Die Untereinheiten des beweglichen Rotors sind in Abbildung 1.3 in Blau und Grün, die Untereinheiten, die zum Stator gehören in Rot- und Gelbtönen dargestellt. Der F 1 -Teil unterscheidet sich nicht wesentlich von dem F 1 -Teil der H + -ATPsynthase aus E. coli. Lediglich die mitochondriale Untereinheit ɛ tritt dort nicht auf. Auch im F 0 -Teil finden sich analog die für die Syntheseaktivität wichtigen Untereinheiten a, d, 4 (b) und 9 10 (entsprechen den bakteriellen Untereinheiten a, b 2 und c 10 ). Die Aufgaben der zusätzlichen Untereinheiten ( 8, e, f, g, H, J, K) sind noch nicht vollständig geklärt [55, 56]. Es wird vermutet, dass die Untereinheiten e, g und K, sowie J und H an der Dimerisierung der H + -ATPsynthasen beteiligt [57] und somit wichtig für die Ausbildung der Cristae sind [58 62]. 7
26 1 Einleitung Die genaue Funktion der Untereinheit 8 ist nicht geklärt, sie nimmt nicht am Protonentransport teil, scheint aber wichtig für die Integration der Untereinheit a zu sein [63], die Funktion von f ist gänzlich unbekannt. Der periphere Stiel wird von den Untereinheiten 4 (b), d und H gebildet. Ein assoziiertes Protein der H + -ATPsynthase aus S. cerevisiae ist das Inhibitorprotein INH1 (nicht gezeigt in Abbildung 1.3). Bei geringen transmembranen Protonengradienten inhibiert es das Enzym und verhindert damit die Hydrolyse von ATP [64 66] Katalysemechanismus der F-Typ H + -ATPsynthase Der von Boyer beschriebene binding-change Mechanismus (siehe Abbildung 1.4) für die Katalyse von ATP durch die H + -ATPsynthase geht von drei identischen Bindungsplätzen aus, bei dem auf allen Bindungsplätzen die gleiche Abfolge von Reaktionen durchlaufen wird, wobei der Start jeweils um 120 verschoben ist [67]. In Syntheserichtung durchlaufen die Nukleotid- Abbildung 1.4: Schematische Darstellung des binding-change Mechanismus nach Boyer [67]. Bild verändert nach K. Förster [68]. bindungsplätze die Konformationen loose (L) = tight (T) = open (O) = loose (L). In der L-Konformation sind die Substrate ADP und P i locker gebunden, in der T-Konformation findet die Synthese von ATP statt und es bleibt fest an die Bindungsstelle gebunden. Beim Übergang in die O-Konformation weist die Nukleotidbindungsstelle eine geringe Affinität für das gebildete ATP auf, worauf dieses abdissoziiert. Durch den anschließenden Übergang zurück in die L- Konformation beginnt der Synthesezyklus von vorne. Bei der Hydrolyse erfolgen die Schritte in entgegengesetzter Reihenfolge. Die für den Zyklus notwendigen Konformationsänderungen in den β-untereinheiten werden durch die Rotation des zentralen Stiels hervorgerufen [69 74]. Bei Anwesenheit von sehr geringen Konzentrationen an ATP zeigte es sich, dass die Rotation des zentralen Stiels in weitere Teilschritte von 90 und 30 [75] bzw. von 80 und 40 [76 78] unterteilt war. Die Rotation der γ-untereinheit wird durch eine Protonentranslokation 8
27 1.1 Die H + -ATPsynthase über den c- bzw. 9-Ring bewirkt [79, 80]. Durch Protonierung einer Aminosäure, welche eine Carboxylat-Seitenkette enthält (Asp 61 bei E. coli [30], Glu 59 bei S. cerevisiae), verlagert sich eine Salzbrücke zwischen dem Carboxylatrest und einem Arginin in der Untereinheit a zu einem nicht protonierten Carboxylatrest der benachbarten c- bzw. 9-Untereinheit. Dadurch kommt es zu der Drehbewegung des c- bzw. 9-Rings [30, 81]. Die Protonen werden durch den zweiten Halbkanal der a-untereinheit schließlich in den Matrixraum abgegeben. In Abbildung 1.5 ist dieser Vorgang schematisch dargestellt. Abbildung 1.5: Schematische Darstellung der Protonentranslokation nach Junge et al. [82]. Bild modifiziert aus [21]. 9
28 1 Einleitung 1.2 Fluoreszierende Proteine Die Fähigkeit von lebenden Organismen Licht durch eine enzymatische Reaktion zu erzeugen wird als Biolumineszenz bezeichnet. Eine solche chemische Umwandlung findet man zum Beispiel bei der Oxidation von Luciferin in Leuchtkäfern. Neben dieser bei verschiedenen Organismen verbreiteten Biolumineszenz, findet man bei Meeresbewohnern auch Lichtemission, die auf Photolumineszenz, also auf Absorption und Emission von Photonen beruht. Verantwortlich dafür sind fluoreszierende Proteine (FP). Als erstes fluoreszierendes Protein wurde bereits 1962 das grün fluoreszierende Protein (GFP) aus der Qualle Aequorea victoria isoliert [83]. Allerdings erst die Klonierung und erfolgreiche Transformation in andere Organismen (Escherichia coli und Caenorhabditis elegans) [84], sowie die Herstellung des ersten GFP- Fusionsproteins [85] ermöglichte die breite Anwendung in der Biotechnologie. GFP ist ein globuläres Protein bestehend aus 238 Aminosäuren [86]. Kristallographische Untersuchungen haben eine zylinderförmige Struktur gezeigt, bestehend aus 11 β-faltblättern um eine einzelne zentrale α-helix, welche den Chromophor enthält [87, 88]. Der Zylinder (βbarrel) hat einen Durchmesser von ca. 30 Å und eine Länge von ca. 40 Å [89] (siehe Abbildung 1.6). Der Chromophor besteht aus den drei Aminosäuren Serin-Tyrosin-Glycin (SYG) an den Positionen 65 bis 67. Dadurch entsteht ein ausgedehntes π-elektronenresonanzsystem [90], bestehend aus zwei aromatischen Ringen mit einer dazwischen liegenden Brücke, welches Licht im Wellenlängenbereich zwischen 395 bis 475 nm absorbiert und ein Emissionsmaximum bei 508 nm hat. Das aus der Qualle isolierte Wildtyp GFP hat einige für die Fluoreszenzspektroskopie ungeeignete Eigenschaften, wie zum Beispiel eine niedrige Quantenausbeute bei Anregung mit blauem Licht [91 93]. Um die spektralen Eigenschaften zu verbessern, wurde eine verbesserte GFP-Variante (EnhancedGFP) durch Mutation des Chromophors (F64L/S65T) entwickelt (EGFP), welches eine Steigerung der Fluoreszenz um das 35-fache im Vergleich zum Wildtyp erbrachte [94], sowie eine Verschiebung des Absorptionsmaximums auf etwa 488 nm und des Emissionsmaximum auf 511 nm. Gleichzeitig faltet sich das EGFP schneller als der Wildtyp und neigt weniger stark zum Ausbleichen. Bei der Expression faltet sich funktionsfähiges GFP völlig selbstständig, ohne dass Cofaktoren oder Chaperone benötigt werden. Es zeigte sich, dass GFP sowie EGFP sehr stabil sind gegenüber der Behandlung mit Detergenz [95, 96], gegenüber Proteasen [84] und dass sie temperaturstabil [97, 98] sind. Neben dem GFP aus A. victoria hat man eine große Anzahl von weiteren natürlich vorkommenden GFP-ähnlichen Proteinen entdeckt. Obwohl die Aminosäuresequenz von allen fluoreszierenden Proteinen nur bis zu 30 % identisch ist, weisen sie als gut konserviertes Strukturmerkmal das β-barrel auf [87, 99, 100]. Auch die Sequenz des Tripeptids (XYG) des Chromophors ist für die beiden Positionen von Y und G streng konserviert. Die Fluoreszenzeigenschaften 10
29 1.2 Fluoreszierende Proteine Abbildung 1.6: Struktur des grün fluoreszierenden Proteins (GFP) aus Aequorea victoria. Der Chromophor (grün, AS-Sequenz SYG) befindet sich im Zentrum des β-barrel, dieses besteht aus 11 β-faltblättern (grau) und einer zentralen α-helix (rot) [89] (pdb 1GFL). und Emissionswellenlängen der fluoreszierenden Proteine beruhen vor allem auf unterschiedlichen Strukturen und Wechselwirkungen mit der direkten Umgebung des Chromophors. Diese wirken als Erweiterung des π-elektronensystems und ändern die Energien der Orbitale des Chromophors [ ]. Durch entsprechende Auswahl der fluoreszierenden Proteine können so individuelle Fluorophorenpaare für Fluoreszenz-Resonanz-Energietransfer-Messungen (FRET) zusammengestellt werden [104]. Fusionsproteine ermöglichen zudem eine selektive Markierung ausgewählter Proteine. Entsprechende Fusionsproteine von fluoreszierenden Proteinen und dem C-Terminus der γ-untereinheit der H + -ATPsynthase aus Saccharomyces cerevisiae wurden zur Messung des Rotationsmechanismus bei der Katalyse von ATP hergestellt. Es wurde im Rahmen der Doktorarbeit von J. Petersen ein Lego -System für das fluoreszierende Protein (siehe Abbildung 1.7) entwickelt, um unterschiedliche Anforderungen an das FRET- System erfüllen zu können [105]. Die Konstrukte wurden zunächst in E. coli exprimiert. Um die Fluoreszenzeigenschaften zu überprüfen wurde dann die entsprechende DNA-Sequenz in S. cervisiae eingebracht. Im Rahmen dieser Arbeit wurde das Konstrukt aus dem Plasmid pqgbta-c verwendet. 11
30 1 Einleitung Abbildung 1.7: Ausschnitt aus den Variationsmöglichkeiten für Zip-Helix-FP- Konstrukte. Es sind die Namen der zipfp-plasmide angegeben und schematisch die Struktur der Proteine gezeigt. Die Konstrukte bestehen aus den fluoreszierenden Proteinen Y = gelb fluoreszierendes Protein, G = grün fluoreszierendes Protein und einem Linker zur kovalenten Bindung des fluoreszierenden Proteins an den C-Terminus der γ-untereinheit der H + -ATPsynthase. Die Linker bestehen aus einer antiparallelen homologen (weiß unterlegter Abschnitt) oder aus einer heterologen (grau unterlegter Abschnitt) Leucin-Zipper-Doppelhelix A = Acoil, B = Bcoil und aus einem zweiteiligen Linkermodul (T) mit einem Strep-tag II (a), einer TEV-Proteaseschnittstelle (c) sowie zwischen den beiden Modulteilen einer weiteren Restriktionsschnittstelle BcII (b). Dies führt zu sechs verschiedenen Möglichkeiten zur Insertion des fluoreszierenden Proteins (a, b, c, ab, bc, ac). Rot eingerahmt ist das in dieser Arbei verwendete Konstrukt, die Abbildung wurde aus J. Petersen [105] entnommen. 1.3 Fluoreszenzspektroskopie Für die Aufklärung von Struktur und Mechanismus von Proteinen werden Methoden benötigt, die eine hohe Empfindlichkeit aufweisen. Dafür eignen sich insbesondere Methoden, die auf Lumineszenz basieren. Hier wird die Lichtemission einer Substanz gemessen, die aus einem elektronisch angeregten Zustand wieder in den Grundzustand übergeht. 12
31 1.3 Fluoreszenzspektroskopie 3 2 S IC hν A IC 3 2 S IC hν A hν F T 1 0 IC ISC IC hν P IC 3 2 S IC Abbildung 1.8: Jablonski-Termschema. Darstellung der Absorption (hν A ) und der Emission von Photonen (hν F = Fluoreszenz, hν P = Phosphoreszenz). Die Innere Umwandlung IC ist in schwarz dargestellt und das Intersystem Crossing ISC in orange. Am Rand ist der jeweilige Spinzustand gezeigt. Die Phänomene der Absorption und Fluoreszenz können anhand des Jablonski-Termschemas (siehe Abbildung 1.8) erklärt werden. Durch Absorption eines Photons wird ein Molekül aus dem Grundzustand (S 0 ) in einen angeregten Singulett-Zustand (S 1 oder S 2 ) überführt. Zumeist erfolgt diese Anregung in ein höheres Schwingungsniveau dieser Singulett-Zustände. Durch innere Umwandlung (IC) relaxiert das Molekül unter Abgabe von Wärme in den Schwingungsgrundzustand des elektronisch angeregten Zustands S 1. Nach einer gewissen Verweildauer (Fluoreszenzlebensdauer 1-10ns)findet der Übergang zurück in den Grundzustand S 0 statt. Wird dabei ein Photon frei, spricht man von Fluoreszenz. Durch den Vorgang der inneren Umwandlung, und damit dem Verlust von Energie, ist das emittierte Licht im Vergleich zum absorbierten Licht zu größerer Wellenlänge verschoben, dies wird als Stokes-Shift bezeichnet [106]. Andere Wege zurück in den Grundzustand stellen die thermische Relaxation unter Abgabe von Wärme, die Energieübertragung auf andere Moleküle z.b. durch Stöße mit Lösungsmittelmolekülen (Fluoreszenzlöschung) und der deutlich langsamere Prozess des Intersystem Crossing (ISC), und damit verbunden die Phosphoreszenz, dar. Bei der Phosphoreszenz erfolgt zunächst aus dem angeregten Singulett-Zustand ein spinverbotener Übergang in den Triplettzustand (T 1 ). Dieser ist relativ stabil und langlebig, da beim Übergang zurück in den Grundzustand S 0 wiederum eine Spinumkehr erfolgen muss. 13
32 1 Einleitung Konfokales Fluoreszenzmikroskop Durch den Einsatz von hochempfindlichen Avalanche-Photodioden (APD) können einzelne Photonen detektiert werden. Dies ermöglicht die zeitaufgelöste Beobachtung einzelner fluoreszierender Moleküle und damit die Möglichkeit Informationen über individuelle Eigenschaften einzelner Moleküle zu erhalten. Für entsprechende Messungen werden die zu untersuchenden Biomoleküle mit organischen Fluorophoren, mit fluoreszierenden Proteinen (z.b. GFP, siehe Kapitel 1.2) oder auch Nanokristallen (Quantenpunkte) [ ] markiert und mit einem konfokalen Fluoreszenzmikroskop gemessen (siehe Abbildung 1.9). Fokusebene Probe Objektiv dichroitische Spiegel Laser Lochblende Detektor (APD) Abbildung 1.9: Konfokales Fluoreszenzmikroskop. Die Fokusebenen des Lasers und der Lochblende werden in der Probe aufeinander justiert. Dadurch wird Licht außerhalb der Fokusebene (roter und grauer Strahlengang) nicht durch die Blende durchgelassen. Dies führt zu einem kleinen Detektionsvolumen und einem guten Signal-Rausch-Verhältnis bei den Fluoreszenzmessungen. Durch Einbringen einer Lochblende wird nur die Fluoreszenz aus dem konfokalen Volumen detektiert, Licht von außerhalb dieses Bereichs wird geblockt. Das Detektionsvolumen eines solchen Aufbaus liegt im Femtoliterbereich und durch Einsatz von Probenkonzentrationen im 14
33 1.3 Fluoreszenzspektroskopie subnanomolaren Bereich, kann sicher gestellt werden, dass sich durchschnittlich weniger als ein Molekül im Detektionsvolumen befindet. Die fluoreszierenden Biomoleküle diffundieren dabei entweder frei durch den Fokus [111] oder werden an einer Oberfläche immobilisiert [112, 113] Fluoreszenz-Korrelations-Spektroskopie (FCS) Bei der Fluoreszenz-Korrelations-Spektroskopie (FCS) werden zeitliche Änderungen der Fluoreszenzintensität im Beobachtungsvolumen des konfokalen Fluoreszenzmikroskops gemessen. Dabei wird nicht die Fluoreszenz eines einzelnen Moleküls detektiert, sondern deren Konzentration wird so gewählt, dass eine geringe Zahl an frei diffundierenden, fluoreszierenden Molekülen im Beobachtungsvolumen vorhanden ist. Die gemessene Gesamtfluoreszenz hängt damit mit der Zahl der Teilchen zusammen. Die Fluktuation der Fluoreszenzintensität ist zum Beispiel durch die Diffusion der fluoreszierenden Teilchen aus dem Beobachtungsvolumen hinaus bzw. hinein gegeben oder durch photophysikalische Übergänge (Triplett-Zustand). Die aus solchen Messungen erhaltenen Daten können mittels einer Autokorrelationsfunktion (siehe Gleichung 1.1) beschrieben werden. Mit einer Modellfunktion, die die verschiedenen Einflussgrößen enthält (Diffusion, Triplettlebensdauer), können durch Anpassung die Diffusionszeit und die Triplettlebensdauer bestimmt werden. Durch die Verwendung eines Eichfluorophors, dessen Diffusionskoeffizient bekannt ist, wird die Justage des konfokalen Mikroskops überprüft. G(t C )= δf (t + τ) δf (t) F (t) 2 (1.1) G(t C ) δ F t+τ F(t) Autokorrelationsfunktion Änderung der Fluoreszenz Zeitintervall zeitlicher Mittelwert der Fluoreszenz Fluoreszenz-Resonanz-Energie-Transfer (FRET) Bei einem Fluoreszenz-Resonanz-Energie-Transfer wird Energie strahlungslos von einem angeregten (*) Fluorophor (Donor, D) auf einen anderen Fluorophor (Akzeptor, A) übertragen. Dieser wird dadurch in einen elektronisch angeregten Zustand überführt und kann Photonen emittieren (vgl. oberer Reaktionsweg in Abbildung 1.10). 15
34 1 Einleitung D* k T k F D + A* D + h (D) D + A + h (A) Abbildung 1.10: Schematische Darstellung der Relaxationswege für einen angeregten Fluorophor (Donor, D*). Oben: Anregung eines zweiten Fluorophors (Akzeptor, A*) durch Fluoreszenz-Resonanz-Energie-Transfer mit der Reaktionsgeschwindigkeitskonstante k T und Relaxation dieses Fluorophors (A) unter Emission der Fluoreszenz hν(a). Unten: Relaxation des Fluorophors D mit der Reaktionsgeschwindigkeitskonstante k F unter Emission der Fluoreszenz hν(d). Die Effizienz dieser Energieübertragung kann mit Gleichung 1.2 beschrieben werden: E FRET = k T k T + k F = F A F D + F A (1.2) k T k F F D F A Geschwindigkeitskonstante des Energie-Transfers Geschwindigkeitskonstante der Fluoreszenz des Donors Fluoreszenzintensität Donor Fluoreszenzintensität Akzeptor Die Effizienz der Energieübertragung hängt maßgeblich vom Abstand der beiden Fluorophore ab. Dieser Zusammenhang wird durch die Förster-Theorie beschrieben [114]. Damit ein Energietransfer stattfinden kann, müssen Emissionsspektrum des Donors und Absorptionsspektrum des Akzeptors überlappen, d.h. sie müssen gleiche Energieniveaus haben. Das Überlappungsintegral J(λ) gibt die Größe der Überlappung an (Gleichung 1.3). J(λ) = 0 f D (λ) ɛ A (λ) λ 4 dλ (1.3) f D (λ) ɛ A (λ) λ normierte Fluoreszenzintensität Donor Absorptionsspektrum des Akzeptors Wellenlänge Befinden sich die beiden Fluorophore in räumlicher Nähe (für organische Fluorophore Å) kommt es zu Dipol-Dipol-Wechselwirkungen der Übergangsdipolmomente der Fluoro- 16
35 1.3 Fluoreszenzspektroskopie phore und damit zum Energietransfer. Die Abhängigkeit der FRET-Effizienz vom Abstand ist in Abbildung 1.11 dargestellt und entspricht Gleichung 1.4. Beträgt die FRET-Effizienz 50 % Abbildung 1.11: Abstandsabhängigkeit der Transfereffizienz (Förster-Kurve). Der Abstand bei einer Energieübertragung von 50 % (FRET-Effizienz = 0,5) entspricht dem Försterradius R 0. entspricht der zugehörige Abstand dem Förster-Radius R 0. In diesem Bereich bewirken bereits kleine Abstandsänderungen eine große Änderung in der FRET-Effizienz. Damit können Molekülbewegungen und Konformationsänderungen bei enzymatischen Prozessen direkt beobachtet werden. r DA = R 0 ( 1 E FRET 1 ) 1/6 (1.4) Die Transfereffizienz ist zusätzlich von der Orientierung der Übergangsdipolmomente der Fluorophore relativ zueinander abhängig. Dies wird durch den Orientierungsfaktor κ 2 wiedergegeben. Für frei bewegliche Fluorophore, welche z.b. an exponierten Positionen im Enzym gebunden sind, nimmt κ 2 den Wert 2/3 an. Damit kann der Försterradius nach Gleichung 1.5 berechnet werden [115] (siehe auch Anhang B). R 0 =0.211[κ 2 n 4 Φ D J(λ)] 1/6 (1.5) κ 2 n Φ D J(λ) Orientierungsfaktor Brechungsindex der Lösung Quantenausbeute des Donors Überlappungsintegral 17
36 1 Einleitung 1.4 Ziel der Arbeit Im Rahmen dieser Arbeit wurden zwei Fragestellungen zum Mechanismus von H + -ATPsynthasen bearbeitet. Dazu wurde für die enzymkinetischen Untersuchungen die H + -ATPsynthase aus E. coli und für die einzelmolekülspektroskopischen Untersuchungen die H + -ATPsynthase aus S. cerevisiae verwendet. Phosphat, das als Substrat bei der ATP-Synthese dient, liegt in wässriger Lösung in mehreren Protonierungsstufen vor. Es sollte mit Hilfe enzymkinetischer Messungen geklärt werden, ob das Enzym eine dieser Protonierungsstufen bevorzugt. In Anknüpfung an frühere Untersuchungen sollte hierfür die Abhängigkeit der Geschwindigkeit der ATP-Synthese von der Phosphatkonzentration bei verschiedenen ph-werten gemessen und die Ergebnisse mit den Dissoziationskonstanten des Phosphats verglichen werden. Um für diese Untersuchungen Enzyme mit möglichst hoher Aktivität verwenden zu können, sollten zwei Methoden für die Reinigung solubilisierter H + -ATPsynthasen aus E. coli miteinander verglichen werden, die Dichtegradientenzentrifugation mit der Ionenaustauschchromatographie. In diesem Zusammenhang sollte auch die ACMA-Methode erstmalig in der Arbeitsgruppe eingesetzt werden, die es ermöglicht, die bei der ATP-Hydrolyse erzeugte transmembrane ph-differenz zu messen. Die zweite Fragestellung war, ob es möglich ist, die Rotation von Untereinheiten in mitochondrialen H + -ATPsynthasen mit Hilfe der Einzelmolekülspektroskopie zu zeigen. Bisherige Arbeiten zur Rotation von Untereinheiten wurden vor allem mit bakteriellen Enzymen durchgeführt, die einfacher gebaut sind und molekularbiologisch leicht modifiziert werden können. In unserer Arbeitsgruppe lag zu Beginn dieser Arbeit eine modifizierte H + -ATPsynthase aus S. cerevisiae vor, an deren γ-untereinheit ein fluoreszierendes Protein (EGFP) fusioniert war und die zusätzlich ein Cystein in der b-untereinheit enthielt. Es sollte versucht werden, dieses modifizierte Enzym in S. cerevisiae zu exprimieren und zu charakterisieren (Untereinheiten, Aktivität). Anschließend sollte versucht werden, das Cystein in der b-untereinheit mit einem für FRET-Messungen geeigneten Fluorophor zu markieren und die Fluoreszenzemissionsspektren des Enzyms zu messen. Falls in diesem doppelt markierten Enzym ein FRET beobachtet wird, sollte es in Liposomen rekonstituiert und die Untersuchungen an einzelnen Molekülen mit Hilfe eines konfokalen Mikroskopaufbaus durchgeführt werden. Hier sollte vor allem die Frage geklärt werden, ob die FRET-Effizienz in den beobachteten Photonbursts konstant ist oder ob bei der ATP-Hydrolyse Änderungen der FRET-Effizienz auftreten, die auf Bewegungen der mit EGFP fusionierten γ-untereinheit relativ zu der mit einem organischen Fluorophor markierten b-untereinheit schließen lassen. Die aus den Änderungen der FRET-Effizienzen berechneten Abstandsänderungen zwischen Fluorophor und EGFP werden mit den aus einem molekularen Modell der H + -ATPsynthase ermittelten Abständen bei der Rotation der γ-untereinheit verglichen und erlauben eine Aussage über die Relativbewegungen der Untereinheiten. 18
37 Material und Methoden
38
39 2 Material 2.1 Geräte Analysewaage Mettler AE200 Feinwaage Kern EW NM Kern PLS Airfuge Beckman Air-driven Ultrazentrifuge Kühlzentrifuge Heraeus Biofuge 28RS Rotoren: 28.1-RS RS 3746 Heraeus Megafuge 1.0R Beckman Instruments J2-HS Rotoren: JA20 JLA JLA Tischzentrifuge Eppendorf Mini Spin Eppendorf 5412 Fisher Scientific MicroV Ultrazentrifuge Beckman Instruments L7 Ultracentrifuge Rotoren: Ti-60 Ti-70 Schüttler Janke & Kunkel IKA-VIBRAX-VXR Janke & Kunkel KS
40 2 Material Schüttler-Inkubator New Brunswick Scientific G25 Incubator-Shaker Inkubator Eppendorf Thermomixer 5436 Heraeus Incubator Typ L150 Sterilbank Microflow Class 2 Biological Safety Cabinet Dampfsterilisator Zirbus VST 50/70S Fermenter Bioengeneering Laborpilot LP351 French Press SLM Aminco French pressure cell press Chromatographie-Anlage Amersham ÄKTApurifier PerSeptive Biosystems BioCad Schallgerät Branson Sonifier 250 Vortexer Heidolph REAX 2000 ph-meter Schott Geräte CG 802 Luminometer BioOrbit 1250 Elektrophorese-Apperatur BioRad Mini Protean 3 Cell UV/Vis-Spektrometer Perkin Elmer Lambda 2 UV/Vis Amersham Pharmacia Spectrophotometer Biotech Ultraspec 3000 pro Fluoreszenz-Spektrometer Perkin Elmer LS 45 Umwälzthermostat Haake G Modell D8 22
41 2.2 Chemikalien 2.2 Chemikalien Chemikalien Hersteller Acrylamid/Bisacrylamid 40 % Sigma, 99 % Adenosin-5 -diphosphat (ADP) Alexis, 98 % Adenosin-5 -triphosphat (ATP) Sigma, 98 % Adenylyl-imidodiphosphat (AMPPNP) Roche Agar-Agar Fluka Alexa488 Molecular Probes, Invitrogen Amidoschwarz 10 B Merck α-aminohexansäure Sigma Ammoniumperoxodisulfat (APS) Sigma, 98 % Ammoniumsulfat Applichem, p.a. Ampicillin (Amp) Sigma ATP-Monitoring Kit Roche ATTO565 ATTO-tec BCA Protein Assay Kit Uptima Bernsteinsäure (Succinat) Merck, p.a. BioBeads SM-2 BioRad Borsäure Merck Bromphenolblau (Mononatriumsalz) (BPB) Serva, reinst Calciumchlorid-Dihydrat Merck, p.a. Chloramphenicol Sigma Coomassie Brillant Blue R250 (CBB) Merck Cobaltchlorid-Hexahydrat Merck, p.a. Dihydroxybenzoesäure (DHB) Aldrich, 99 % Dimetylsulfoxid (DMSO, wasserfrei) Sigma, 99,5 % 1,4-Dithiothreit (DTT) AppliChem, 99,5 % n-dodecyl-β-maltosid (DDM) Glycon, 99 % Eisen-(II)-chlorid-Tetrahydrat Applichem, p.a. Eisen-(III)-chlorid-Hexahydrat Roth, reinst Ethanol redest. Ethylendiamintetraessigsäure (EDTA) Sigma, 95 % Essigsäure Riedel de Haën, p.a. 99,8 % Formaldehyd Fluka, 36,5 % in H 2 O 23
42 2 Material Geneticin (G-418) Sigma Glucose Applichem Glutaraldehyd Fluka, 2,6 M (25 % in H 2 O) Glycerin Fluka, p.a. Hefeextrakt Applichem HEPES Sigma, >99,5 % Histidin Fluka, 99 % Isoleucin Fluka Kaliumacetat Merck, reinst Kaliumchlorid Riedel de Haën Kaliumdihydrogenphosphat Sigma-Aldrich Kaliumhydroxid Applichem Kanamycin (Kan) Sigma Lactatdehydrogenase Roche Lauryl-Dimethylaminoxid (LDAO) Fluka, 30 % in H 2 O Leucin Fluka, >99,5 % Lithiumacetat Roth, purum Methanol Merck, p.a. 2-(N-Morpholino)ethansulfonsäure (MES) Applichem, mind. 99 % Magnesiumchlorid-Hexahydrat Fluka, p.a. Magnesiumsulfat-Heptahydrat Applichem Manganchlorid-Dihydrat Merck, p.a. (N-Morpholino)-Propansulfonsäure(MOPS) Applichem, 99,5 % Natriumacetat-Trihydrat Riedel de Haën, 99,5 % Natriumchlorid Riedel de Haën, p.a. Natriumcholat Serva Natriumdihydrogenphosphat Merck, reinst Natriumdodecylsulfat (SDS) Sigma, 99 % Natriumhydroxid Merck, p.a. Nicotinamidadenindinukleotid (NADH) Roche Octylglycosid Alexis, 98 % Paraaminobenzoesäure (PABA) Peptone from Casein Fluka Phenylmethansulfonylfluorid (PMSF) Fluka Phosphoenolpyruvat (PEP) Fluka, 99,5 % Phosphatidsäure Sigma 24
43 2.2 Chemikalien Phosphatidylcholin Avanti Polyethylenglycol (PEG) 3350 Sigma Pyruvatkinase Boehringer, in Glycerin Saccharose Fluka, f.d.mikrobiol. Salzsäure, konz Sigma-Aldrich Sephadex G-50 fine GE Healthcare Silbernitrat Fluka, p.a. Tetracyclin (Tet) Sigma N,N,N,N -Tetrametyletylendiamin (TEMED) Sigma N-(Tri(hydroxymethyl)methyl)glycin (Tricin) Applichem, 99 % Tris(hydroxymethyl)-aminomethan (Tris) Applichem, 99,9 % Triton X-100 Roch, 10 % (w/v) in H 2 O Trypton Fluka Uracil Sigma Valin Fluka Valinomycin Sigma, 90 % Yeast Nitrogen Base Becton, Dickinson and Company Wasser wurde mit der Inenaustauschanlage ELIX 10 Millipore deonisiert. Ionenleitfähigkeit < 223μS 25
44 2 Material 2.3 Verwendete Zellstämme Die in dieser Arbeit verwendeten Escherichia coli Zellstämme sind in Tabelle 2.3, die verwendeten Saccharomyces cerevisiae Zellstämme in Tabelle 2.4 aufgeführt. Stamm / Plasmid Tabelle 2.3: Verwendete E. coli Zellstämme. Genotyp Resistenz Verwendung / Besonderheit RA1 / pra100 DK8 / pbwu13 F-, thi1, rpsl, Δ(atpB-C), Δb0, Δcyo::kan ilv::tn10 / uncbefhagdc bgir, thi1, rel1, Hfr801, Δ(atpB-C), ilv::tn10 / uncbefhagdc Tet, Kan / Cam E. coli Referenzstamm (H + -ATPsynthase Wildtyp) [116] - / Amp Ampicillin-Resistenz und Erhöhung des Anteils an H + -ATPsynthasen auf bis zu 25 % durch Multicopy-Plasmid [ ] Tabelle 2.4: Verwendete S. cerevisiae Zellstämme. Stamm Genotyp Auxotrophie / Resistenz Verwendung / Besonderheit YRD15 MATαhis3-11,3-15 leu2-3,2-112, ura3-251,3-373[rho + ] Uracil, Histidin, Leucin S. cerevisiae Referenzstamm (H + -ATPsynthase Wildtyp) [120, 121] YRD15- b-k174c-γ- C84S/T121C MATαhis3-11,3-15 leu2-3,2-112, ura3-251,3-373[rho + ] ATP3 C84S/T 121C ::ADHT- CaUra3MX ATP4 K174C ::KanMX Histidin, Leucin / G418, Ampicilin Tochterstamm von YRD15 Geneticin-Resistenz durch KanMX-Kassette Ampicin-Resistenz durch ADHT-CaUra3MX-Kasette Exprimiert H + -ATPsynthase, bei der in Untereinheit b an Position 174 ein K gegen ein C, in γ an 84 ein C gegen ein S bzw. an 121 ein T gegen ein C ausgetauscht wurde. [68, 122] YEBTA-cγ1-b-K174C MATαhis3-11,3-15 leu2-3,2-112, ura3-251,3-373[rho + ] ATP3-EBTA-c::His5MX + ATP4 K174C ::KanMX Leu, Uracil / G418 Tochterstamm von YEBTA-c-γ1 [105] Geniticin-Resistenz durch KanMX-Kassette [88] Exprimiert Fusionsprotein von EGFP und H + -ATPsynthase, bei dem an Position 174 in Untereinheit b ein K gegen ein C ausgetauscht wurde. [122] 26
45 3 Biochemische Methoden 3.1 Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Anzucht von E. coli Es wurden E. coli-zellen der Stämme RA1 und DK8 nach Vorschrift [123] und [124] in einem 30 l Fermenter kultiviert. Abbildung 3.1 zeigt den schematischen Ablauf. Lösungen Stamm RA1 Medium für Agar-Platten Trypton 1,6 % (w/v) Hefeextrakt 1 % (w/v) NaCl 85 mm Agar 20 mg/ml nach autoklavieren: Chloramphenicol 30 mg/l Lagerung bei 4 C Salzlösung 1000-fach ZnCl 2 15 mm MnCl 2 2H 2 O 1 mm H 3 BO 3 50 mm CaCl 2 2H 2 O 2,5 mm CoCl 2 6H 2 O 1 mm FeCl 2 4H 2 O 3 mm lösen in 1 M NaOH, steril filtrieren, Lagerung lichtgeschützt bei 4 C RA1-Medium II KH 2 PO mm KOH 71 mm Trypton 0,1 % (w/v) Hefeextrakt 0,05 % (w/v) NaCl 17 mm (NH 4 ) 2 SO 4 15 mm Uracil 0,4 mm Salz-Lösung 0,1 % (v/v) autoklavieren, 2 x 1 l, Lagerung bei RT Medium Fermenter RA1 KH 2 PO mm KOH 71 mm Trypton 0,1 % (w/v) Hefeextrakt 0,05 % (w/v) NaCl 17 mm (NH 4 ) 2 SO 4 15 mm Uracil 0,4 mm Antifoam 0,01 % (v/v) Salz-Lösung 0,1 % (v/v) autoklavieren, Lagerung bei RT 27
46 3 Biochemische Methoden RA1-Medium I Trypton 1,6 % (w/v) Hefeextrakt 1 % (w/v) NaCl 85 mm autoklavieren, 10 ml Fraktionen, Lagerung bei RT MgSO 4 -Lösung 1000-fach MgSO 4 1M autoklavieren, Lagerung bei RT Aminosäurelösung Arginin 250 mm Thiamin-HCl 1,5 mm DHB 40 mm Isoleucin 250 mm Valin 350 mm lösen in 1 M NaOH, steril filtrieren, Lagerung lichtgeschützt bei 4 C Glukoselösung 67-fach Glukose 20 % (w/v) autoklavieren, Lagerung bei RT Lösungen Stamm DK8 Luria-Medium Pepton 1 % (w/v) Hefeextrakt 0,5 % (w/v) NaCl 85 mm nach autoklavieren: Ampicillin 100 mg/l 2 x 100 ml, Lagerung bei RT Medium für Agarplatten Pepton 1 % (w/v) Hefeextrakt 0,5 % (w/v) NaCl 85 mm Agar 20 mg/ml nach autoklavieren: Ampicillin 100 mg/l Lagerung bei 4 C Kultermedium NaH 2 PO 4 34 mm K 2 HPO 4 64 mm (NH 4 ) 2 SO 4 20 mm Salz-Lösung 0,1 % (v/v) autoklavieren, 2 x 1 l, Lagerung bei RT Salzlösung 1000-fach ZnCl 2 MgSO 4 2H 2 O CaCl 2 2H 2 O FeSO 4 7H 2 O Lagerung bei 4 C 1mM 0,3 mm 10 mm 1 mm Thiaminlösung Thiamin 60 mm steril filtrieren, Lagerung lichtgeschützt bei 4 C Glycerin Glycerin 3,75 M autoklavieren, Lagerung bei RT 28
47 3.1 Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Aminosäurelösung Asparagin 230 mm Thymin 240 mm Isoleucin 230 mm Valin 260 mm lösen in 1 M NaOH, steril filtrieren, Lagerung lichtgeschützt bei 4 C Medium Fermenter DK8 NaH 2 PO 4 34 mm K 2 HPO 4 64 mm (NH 4 ) 2 SO 4 20 mm Antifoam 0,01 % Salz-Lösung 0,1 % (v/v) autoklavieren, Lagerung bei RT Antibiotika Chloramphenicol Chloramphenicol 30 g/l in Ethanol 50 % (v/v) steril filtrieren, Lagerung bei -20 C Kanamycin Kanamycin 30 g/l in Ethanol 50 % (v/v) steril filtrieren, Lagerung bei -20 C Ampicillin Ampicillin in H 2 O 200 g/l steril filtrieren, Lagerung bei -20 C Festkultur Zellen aus bei -80 C gelagerten Stammkulturen wurden unter sterilen Bedingungen auf für den verwendeten Stamm entsprechenden Agarplatten ausgestrichen. Die Festkulturen wurden über Nacht bei 37 C inkubiert. Vorkultur Zur Kultivierung des RA1-Stamms wurden vier Vorkulturen hergestellt. Ein Reagenzglas mit 10 ml RA1-Medium I, Chloramphenicol (30 mg/l) und Kanamycin (30 mg/l) wurde mit einer Kolonie von der Festkultur unter sterilen Bedingungen überimpft und 7 Stunden (200 rpm, 37 C) inkubiert. Jeweils zwei Vorkulturen wurden in 2lSchikanekolben mit 1 l RA1-Medium II, 2 ml Aminosäurelösung, 1 ml MgCl 2 -Lösung, 15 ml Glukoselösung und Chloramphenicol (30 mg/l) überführt. Das Zellwachstum erfolgte über 9 Stunden bei 37 C und 180 rpm. Für den DK8-Stamm wurden je zwei Kolonien in zwei 100 ml Kolben Luria-Medium gegeben und bei 37 C und 230 rpm 9 Stunden inkubiert. Anschließend wurden je 60 ml eines Kolbens in einen 2 l Schikanekolben mit 1 l Kulturmedium, 1650 μl Aminosäurelösung, 150 μl 29
48 3 Biochemische Methoden Glycerinstammkultur pra100/ra1 pbwu13/dk8 Agarplatten 37 C, ca. 9h Vorkultur I 37 C RA1-Medium I, 7h Luria-Medium, 9h Vorkultur II 37 C, 9h RA1-Medium II Kulturmedium Hauptkultur Fermenter, 37 C Medium RA1, ca. 5h Medium DK8, ca. 12h Escherichia coli-zellen Abbildung 3.1: Zellanzucht von Escherichia coli: Schematischer Verlauf der Kultivierung von Zellen der E. coli-stämme RA1 mit dem Plasmid pra100 und DK8 mit dem Plasmid pbwu13. Thiaminlösung und 20 ml Glycerin unter sterilen Bedingungen überführt und über Nacht bei 37 C und 230 rpm inkubiert. Hauptkultur Die Zellanzucht in der Hauptkultur erfolgte für beide Stämme in einem 30 l Fermenter bei 37 C und 380 rpm bis zu einer OD 578 von ca. 1,5. Für den Stamm RA1 wurden zu 26 l des Fermentermediums 56 ml Aminosäurelösung, 420 ml Glukoselösung, 28 ml Chloramphenicol- Stammlösung und 2 l Vorkultur gegeben. Nach ca. 5 Stunden wurde der Übergang in die stationäre Phase erreicht und mit der Zellernte begonnen. Die Kultivierung des DK8-Stamms wurde in 26 l Fermentermedium für DK8, 46,2 ml Aminosäurelösung, 4,2 ml Thiaminlösung, 560 ml Glycerin, 14 ml Ampicillin und 2 l Vorkultur durchgeführt. Mit der Zellernte konnte nach ca. 12 Stunden begonnen werden. 30
49 3.1 Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Zellernte Die Kultivierungstemperatur wurde unverzüglich auf 15 C abgekühlt und die Zellen durch Zentrifugation (Beckman Instruments J2-HS, Rotor JLA10.500, 9000 rpm, 4 C) vom Medium abgetrennt. Die Lagerung erfolgte nach Schockfrostung mit flüssigem Stickstoff in g Portionen bei -80 C Isolation von EF 0 F 1 aus E. coli Die Isolation der H + -ATPsynthase kann wie unter [124] beschrieben durch eine Dichtegradientenzentrifugation oder wie unter [123] beschrieben durch die Reinigung über eine Anionenaustauschsäule erfolgen. Dabei liefert die Isolation per Dichtegradient ein syntheseaktiveres Enzym, welches für Kinetikmessungen verwendet wurde. Durch Chromatographie gereinigtes Enzym wird in der Einzelmolekülspektroskopie benötigt. Neben der Reinigungsmethode, sind auch die verwendeten E. coli-stämme unterschiedlich geeignet für die weiteren Messmethoden. So zeigt der DK8-Stamm eine höhere Syntheseaktivität. Der Stamm RA1 kann in der Kombination mit Plasmiden auf pra100-basis kein Cytochrom b0 synthetisieren und damit können reine ATPsynthasen für Einzelmolekülmessungen isoliert werden [123]. Bis zur Solubilisierung der Membranproteine ist der schematische Ablauf für beide Methoden identisch (siehe Abbildung 3.2). Im Detail unterscheiden sich bei der Isolation für die beiden in dieser Arbeit isolierten Stämme die verwendeten Lösungen und Detergenzien. Die abweichenden Werte bei der Isolation von DK8 mittels Dichtegradientenzentrifugation sind im Folgenden in Klammern angegeben. Lösungen für Solubilisierung Waschpuffer Tris-HCl, ph 8 Solubilisierungspuffer MES-KOH, ph 7 Tricin-KOH MgCl 2 DTT PMSF (frisch zugeben) (Thioglycerin 50 mm 20 mm 20 mm 5 (2) mm 2 mm (entfällt) 0,001 % (entfällt) 5 mm) French Press Puffer Tris-HCl, ph 8 20 (10) mm KCl 140 mm MgCl 2 5mM DTT 2 (1) mm Glycerin 10 % PABA 5 mm PMSF (frisch zugeben) 0,002 % 31
50 3 Biochemische Methoden E. coli Zellen French Press Zentrifugation Überstand: Membranen & Cytoplasma Sediment: Nicht aufgeschlossene Zellen Ultrazentrifugation Überstand: Cytoplasma Sediment: Membranen Solubilisierung Ultrazentrifugation Sediment: Membranen Überstand: Protein-Detergenz-Komplex Größenausschlusschromatographie S-300 Anionenaustauschchromatographie Größenausschlusschromatographie S-400 Dichtegradientenzentrifugation EF 0 F 1 Abbildung 3.2: Isolation von H + -ATPsynthase aus Escherichia coli: Schematischer Verlauf der Isolation von EF 0 F 1. 32
51 3.1 Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Zellaufschluss Die folgenden Schritte wurden bei 4 C durchgeführt. In 300 ml Waschpuffer wurden über Nacht 50 g Zellen unter Rühren langsam aufgetaut. Die Zellsuspension wurde zentrifugiert (Beckman Instruments J2-HS, Rotor JLA , rpm, 10 min, 4 C), das Pellet in 300 ml Waschpuffer aufgenommen, 45 min gerührt und erneut zentrifugiert (siehe oben). Das Pellet wurde in 200 ml French Press Puffer resuspendiert, durch Nylongaze filtriert und nach Zugabe einer Spatelspitze DNAse 30 min gerührt. Die Lösung wurde in die vorgekühlte French Press gegeben und die Zellen mit einem Druck von 1300 psi und einer Tropfengeschwindigkeit von 1-2 Tropfen pro Sekunde aufgeschlossen. Die Tropfen wurden in flüssigem Stickstoff aufgefangen und bei -80 C gelagert. Isolierung der Membranen Das aufgeschlossene Zellhomogenat wurde aufgetaut und zentrifugiert (Beckman Instruments J2-HS, Rotor JA-20, rpm, 15 min, 4 C). Der Überstand wurde erneut zentrifugiert (Beckman Instruments J2-HS, Rotor JA-20, rpm, 15 min, 4 C). Der Überstand mit den Membranen wurde in der Ultrazentrifuge zentrifugiert (Beckman Instruments L7, Rotor Ti60, rpm, 90 min, 4 C) und die Membranen so sedimentiert. Isolation und Reinigung von EF 0 F 1 durch Chromatographie Verwendete Lösungen (NH 4 ) 2 SO 4 -Lösung gesättigt, ph 7,5 Lagerung bei 4 C MOPS-Puffer MOPS-NaOH, ph 7 MgCl 2 50 mm 100 μm Puffer A MOPS-KOH, ph 7,5 40 mm KCl 80 mm Saccharose 2 % (w/v) DTT 2 mm MgCl 2 4mM Glycerin 10 % (v/v) DDM 0,1 % Puffer filtrieren PMSF (frisch zugeben) 0,001 % Puffer B MOPS-KOH, ph 7,5 80 mm KCl 160 mm Saccharose 4 % (w/v) DTT 4 mm MgCl 2 8mM Glycerin 20 % (v/v) DDM 0,2 % Puffer filtrieren PMSF (frisch zugeben) 0,002 % 33
52 3 Biochemische Methoden Membranenpuffer Tris-HCl, ph 8 KCl MgCl 2 Saccharose DTT Glycerin PABA 50 mm 100 mm 5mM 2 % (w/v) 2 mm 10 % (v/v) 5 mm Puffer C MOPS-KOH, ph 7,5 40 mm KCl 80 mm Saccharose 2 % (w/v) DTT 2 mm MgCl 2 4mM Glycerin 10 % (v/v) DDM 0,1 % Puffer filtrieren Solubilisierung der Membranproteine Das Sediment wurde in Membranenpuffer resuspendiert und erneut zentrifugiert (Beckman Instruments L7, Rotor Ti60, rpm, 90 min, 4 C). Das Pellet wurde gewogen und in Solubilisierungspuffer (10 ml/g Membranen) resuspendiert. Zu der Suspension wurden 1,75 % DDM (w/v) frisch zugegeben, die Lösung 30 min gerührt und zentrifugiert (Beckman Instruments L7, Rotor Ti60, rpm, 90 min, 4 C). Der Überstand wurde mit 0,001 % PMSF (v/v) versetzt, das Pellet verworfen. Gelpermeationschromatographie S-300 Zu den solubilisierten Membranproteinen wurde in zwei Schritten langsam gesättigte Ammoniumsulfat-Lösung zunächst bis zu einer Gesamtsättigung von 45 %, dann bis 65 % zugetropft, die Lösung mit 0,001 % PMSF versetzt und über Nacht gerührt. Die Ammoniumsulfat-Fällung wurde zentrifugiert (Beckman Instruments J2-HS, Rotor JA-20, rpm, 15 min, 4 C), das Pellet in 5 ml Puffer A resuspendiert, die Lösung filtriert (Whatman, Filtereinheit 0,45 μm, Celluloseacetat, steril) und auf die Gelpermeationssäule S-300 aufgetragen. Die Parameter der Chromatographie sind in Tabelle 3.1 aufgeführt. Anionenaustauschchromatographie HQ20 Fraktionen mit deutlicher Absorbanz bei 280 nm (A 280nm > 0,3, Fraktion 19-26) wurden auf eine Anionenaustauschsäule Poros HQ20 aufgetragen. Die Chromatographie erfolgte an der BioCad Workstation nach den in Tabelle 3.1 aufgeführten Parametern. 34
53 3.1 Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Tabelle 3.1: Parameter der Chromatographien für die Isolation von EF0F1. Die Reinigung und Isolation der H + -ATPsynthase erfolgte durch eine Gelpermationschromatographie S-300, eine Anionenaustauschchromatographie Poros HQ20 und eine weitere Gelpermationschromatographie S-400. Parameter S-300 HQ20 S-400 Säulenvolumen (CV) 60 ml 1,7 ml 60 ml Säulenmaterial Sephacryl S-300 HR Poros, 20 HQ Sephacryl HR Äquilibrierungspuffer Puffer A Puffer B Puffer C Elutionspuffer Puffer A 3 M KCl-Lösung Puffer C Äquilibrierungsvolumen - 17 ml (10 CV) - Probenvolumen 5 ml 5-15 ml 500 μl Flussrate 40 ml/h 3,5 ml/min 20 ml/min Größe der Elutionsfraktionen 4 ml 1 ml 5 ml Detektion UV/VIS (278 nm) UV/VIS (280 nm), Leitfähigkeit UV/VIS (278 nm) Gradient % Elutionspuffer - 35
54 3 Biochemische Methoden Gelpermeationschromatographie S-400 Fraktionen mit deutlicher Absorbanz bei 280 nm (> 0,1) wurden vereinigt und gesättigte Ammoniumsulfat-Lösung bis 65 % der Sättigungskonzentration langsam unter Rühren zugetropft, die Lösung mit 0,001 % PMSF versetzt und über Nacht gerührt. Die AS-Fällung wurde zentrifugiert (Beckman Instruments J2-HS, Rotor JA-20, rpm, 15 min, 4 C), das Pellet in 500 μl Puffer C mit 1 % DDM resuspendiert und erneut zentrifugiert (Heraeus Biofuge 28RS, Rotor HFA , rpm, 5 min, 4 C). Der Überstand wurde auf die Gelpermeationssäule S-400 aufgetragen und die Chromatographie mit den Parametern in Tabelle 3.1 durchgeführt. Die Fraktionen wurden in 500 μl Strohhalme fraktioniert, schockgefroren und unter flüssigem Stickstoff gelagert. Konzentrierung 1 ml Eluat wurde mit 2 ml gesättigter Ammoniumsulfat-Lösung gemischt, 90 min auf Eis inkubiert und zentrifugiert (Heraeus Biofuge 28RS, Rotor HFA , rpm, 20 min, 4 C). Das Pellet wurde in 75 μl MOPS-Puffer mit 0,1 % DDM resuspendiert und zur Abtrennung von überschüssigem Ammoniumsulfat über eine Zentrifugensäule (Sephadex G50 fine in MOPS-Puffer mit 0,1 % DDM suspendiert, vorzentrifugiert Heraeus Megafuge 1.0R, 1600 rpm, 2 min) eluiert (Heraeus Megafuge 1.0R, rpm, 3 min). Zur Lagerung wurden die Proben mit 10 % Glycerin (v/v) versetzt, schockgefroren und bei -80 C gelagert. Isolation und Reinigung von EF 0 F 1 durch Dichtegradientenzentrifugation Verwendete Lösungen Gradientenpuffer MES-NaOH, ph 7 Tricin MgCl 2 Thioglycerin Octylglucosid 10 mm 10 mm 0,5 mm 5 mm 10 g/l Solubilisierung der Membranproteine Das Pellet der Ultrazentrifugation (vgl. oben) wurde in 15 ml Solubilisierungspuffer resuspendiert und mit einem Homogenisator homogenisiert. Die Proteinkonzentration wurde mit einem BCA-Test bestimmt und mit Solubilisierungspuffer auf 10 mg/ml eingestellt. Zu der 36
55 3.1 Isolation der H + -ATPsynthase aus Escherichia coli (EF 0 F 1 ) Tabelle 3.2: Pipettierschema des Stufengradienten für die Dichtegradientenzentrifugation zur Isolierung von EF 0 F 1. Der Stufengradient wurde durch Unterschichtung mit je 2 ml Lösung gegossen. Stufe Gradientenpuffer Saccharose % % (w/w) % (w/w) Suspension wurde in Solubilisierungspuffer gelöstes Octylglycosid bis zu einer Endkonzentration von 20 mg/ml langsam zugetropft, die Lösung 30 min bei 4 C gerührt und zentrifugiert (Beckman Instruments L7 Ultracentrifuge, Rotor SW40, rpm, 60 min, 4 C). Dichtegradientenzentrifugation Auf einen Stufengradienten (Pipettierschema siehe Tabelle 3.2) wurden je 2 ml des Überstands aufgetragen und über Nacht zentrifugiert (Beckman Instruments L7 Ultracentrifuge, Rotor SW40, rpm, 16 h, 4 C). Nach der Zentrifugation wurde der Gradient von oben in 250 μl Schritten fraktioniert, die Fraktionen aus der gleichen Schicht vereinigt und auf Hydrolyseaktivität (siehe Kapitel 3.9.1) untersucht. Aktive Fraktionen wurden in flüssigem Stickstoff eingefroren und bei -80 C gelagert. 37
56 3 Biochemische Methoden 3.2 Isolation der H + -ATPsynthase aus Saccharomyces cerevisiae (YF 0 F 1 ) Anzucht von S. cerevisiae Es wurden Zellen der Hefestammes YRD15 (Wildtyp-YF 0 F 1 ) und YEBTA-c-γ1-b-K174C (EGFP-Fusionprotein-Cysteinmutante von YF 0 F 1 ) nach Vorschrift [68] in einem 30 l Fermenter kultiviert. Abbildung 3.3 zeigt den schematischen Ablauf. YEPD-Medium Pepton 1 % (w/v) Hefeextrakt 1 % (w/v) Glukose 2 % (w/v) für Festmedien: Agar 18 mg/ml autoklavieren, Lagerung bei 4 C YEPE-Medium Pepton 1 % (w/v) Hefeextrakt 1 % (w/v) Ethanol 2 % (v/v) für Festmedien: Agar 18 mg/ml autoklavieren, Lagerung bei 4 C YMM-Medium Yeast Nitrogen Base 0,17 % (w/v) (NH 4 ) 2 SO 4 0,5 % (w/v) Glukose 2 % (w/v) Aminosäuren 2 ml/l für Festmedien: Agar 18 mg/ml autoklavieren, Lagerung bei 4 C SACC-Medium Hefeextrakt 1 % (w/v) 10 %ig FeCl 3 50 μl/l CaCl 2 2H 2 O 67 mg/l SACC-Salze 1 % (v/v) Aminosäuren 2 ml/l autoklavieren für SACC-E-Medium: + Ethanol 2 % (v/v) Lagerung bei RT SACC-Salze 100-fach (NH 4 ) 2 SO 4 12 % (w/v) NaCl 5 % (w/v) MgCl 2 6H 2 O 7 % (w/v) KH 2 PO 4 10 % (w/v) Lagerung bei RT Aminosäurelösungen 500-fach Histidin 1 % (w/v) Uracil 1 % (w/v) Leucin 1 % (w/v) steril filtrieren, Lagerung lichtgeschützt bei 4 C 38
57 3.2 Isolation der H + -ATPsynthase aus Saccharomyces cerevisiae (YF 0 F 1 ) STA-Puffer Tris/HCl ph 7,0 10 mm Saccharose 250 mm 6-Aminohexansäure 5 mm steril filtrieren, Lagerung bei 4 C Antibiotika Geneticin (G-418) G-418 in H 2 O 200 g/l steril filtrieren, Lagerung bei -20 C YRD15 Glycerinstammkultur YEBTA-c- l-b-k174c Agarplatten 28 C, ca. 4 Tage Vorkultur I 28 C, YEPD-Medium, ca. 25h Vorkultur II 28 C, SACC-E-Medium 25h 38h Hauptkultur Fermenter, 28 C, SACC-E-Medium ca. 22h ca. 45h Saccharomyces cerevisiae- Zellen Abbildung 3.3: Zellanzucht von Saccharomyces cerevisiae: Schematischer Verlauf der Kultivierung von Zellen der Hefe-Stämme YRD15 und YEBTA-c-γ1-b- K174C. Selektionstest und Festkultur Es wurde auf vorhandene Auxotrophien und Antibiotikaresistenzen geprüft. Zellen aus bei - 80 C gelagerten Stammkulturen wurden unter sterilen Bedingungen auf YMM-Agarplatten mit unterschiedlichen Kombinationen von Aminosäuren, auf YEPD-Agarplatten mit G-418 ausgestrichen und 24 Stunden bei 28 C inkubiert. Nur Zellkulturen, die entsprechend ihrer 39
58 3 Biochemische Methoden vorhandenen Auxotropien und vorhandenen oder nicht vorhandenen Antibiotikaresistenz gewachsen waren, wurden auf YEPE-Agarplatten ausgestrichen und bei 28 C 4 Tage inkubiert. Vorkultur Sechs Kulturröhrchen mit 10 ml YEPD-Medium wurden von den YEPE-Festkulturen überimpft und 25 h bei 28 C und 220 rpm inkubiert. Eine Probe der gewachsenen Zellen wurde unter sterilen Bedingungen entnommen und unter dem Mikroskop (Zeiss ICM 405, Objektiv Zeiss Planapo 100/1,3 Oel) auf vorhandene Hefezellen untersucht. Für die Mutante YEBTA-c-γ1-b- K174C wurde mit Hilfe einer angeschlossenen UV-Lampe (Zeiss HBO 50W) auf grüne Fluoreszenz geprüft. Waren keine Verunreinigungen ersichtlich, wurden jeweils 3 Kulturröhrchen in 5 l Schikanekolben mit 1,5 l SACC-E-Medium und je 3 ml Aminosäurelösung, entsprechend der vorhandenen Auxotropie, überführt. Das Zellwachstum wurde während der Inkubation (28 C, 200 rpm) spektroskopisch durch Messung der optischen Dichte bei 600 nm verfolgt und parallel unter dem Mikroskop kontrolliert. Der Stamm YRD15 wies nach 25 Stunden eine OD 600 von 3,0 auf. Die Vorkultur der Mutante YEBTA-c-γ1-b-K174C erreichte nach 38 Stunden eine OD 600 von 1,1 und wurde in die Hauptkultur überführt. Hauptkultur In einem 30 l Fermenter wurden zu 26 l des SACC-Mediums und 3 ml Antifoam nach dem Sterilisieren 560 ml Ethanol, je 56 ml der anhand der Auxotrophie benötigten Aminosäuren und 3 l Vorkultur gegeben. Die Kultivierung wurde bei 28 C und einer Rührgeschwindigkeit von 320 rpm durchgeführt und anhand der OD 600 sowie unter dem Mikroskop kontrolliert. Der Stamm YRD15 erreichte nach 22 Stunden eine OD 600 von 5,6 und die Mutante YEBTA-c-γl-b- K174C nach 45 Stunden eine OD 600 von 5,2. Zellernte Mit der Zellernte wurde durch Abkühlen auf 15 C und Zentrifugation der Zellkultur (Beckman Instruments J2-HS, Rotor JLA10.500, rpm, 4 C) direkt begonnen. Die Zellen wurden mit kaltem H 2 O gewaschen, zentrifugiert (Beckman Instruments J2-HS, Rotor JLA10.500, rpm, 4 C) und im Verhältnis 1:1 (w/v) in kaltem STA-Puffer resuspendiert. Die Lagerung erfolgte nach Schockfrostung mit flüssigem Stickstoff in g Portionen bei -80 C. 40
59 3.2 Isolation der H + -ATPsynthase aus Saccharomyces cerevisiae (YF 0 F 1 ) Isolation von YF 0 F 1 aus S. cerevisiae Die Isolation der H + -ATPsynthase aus den Mitochondrien von Saccharomyces cerevisiae wurde wie bei Förster beschrieben durchgeführt [68]. Der schematische Verlauf ist in Abbildung 3.4 dargestellt. SEM-Puffer HEPES-NaOH, ph 7,65 Saccharose 6-Aminohexansäure NaCl MgCl 2 EDTA Lagerung bei 4 C 20 mm 250 mm 5 mm 40 mm 4mM 200 μm Niedrigsalz-Puffer HEPES-NaOH, ph 7,65 Saccharose 6-Aminohexansäure MgCl 2 DDM DTT NaCl 20 mm 250 mm 5 mm 4mM 0,05 % (w/v) 1 mm 100 mm Hochsalz-Puffer HEPES-NaOH, ph 7,65 Saccharose 6-Aminohexansäure MgCl 2 DDM DTT NaCl 20 mm 250 mm 5 mm 4mM 0,05 % (w/v) 1 mm 500 mm Zellaufschluss Die folgenden Schritte wurden bei 4 C durchgeführt. Es wurden 200 g Hefezellmasse auf Eis aufgetaut, 2:1 mit STA-Puffer auf 300 ml Gesamtvolumen verdünnt und mit 300 μl PMSF (2.5 % in Ethanol) versetzt. Jeweils 15 ml der Zellsuspension wurden in 50 ml Greinerröhrchen gegeben, die9-10gmitsta-puffer gewaschene Glasperlen (0,5 mm) enthielten und 30 min auf höchster Stufe geschüttelt (Janke&Kunkel IKA-VIBRAX-VXR mit aufgeschraubter Halterung für 6 Greinerröhrchen). Isolation der Mitochondrien Die Suspension wurde zentrifugiert (Beckman Instruments J2-HS, Rotor JA , 3500 rpm, 30 min, 4 C), das Pellet verworfen und der Überstand erneut zentrifugiert (Beckman Instruments J2-HS, Rotor JA , rpm, 30 min, 4 C). 41
60 3 Biochemische Methoden Herstellung von submitochondrialen Partikeln Das Pellet wurde in 40 ml SEM-Puffer resuspendiert und in 4,5 ml Portionen mit der Mikrospitze (3 mm) auf Eis 3 min beschallt (Branson Sonifier 250, Output: Stufe 2, Duty Cycle: 40 %). Die Suspension wurde 1:2 auf das dreifache Volumen mit SEM-Puffer verdünnt und zentrifugiert (Beckman Instruments J2-HS, Rotor JA-20, rpm, 20 min, 4 C). Der Überstand wurde anschließend in der Ultrazentrifuge mit g zentrifugiert (Beckman Instruments L7, Rotor Ti60, rpm, 40 min, 4 C), das Pellet mit 25 ml SEM-Puffer resuspendiert, tröpfchenweise in flüssigem Stickstoff eingefroren und bei -80 C gelagert. Solubilisierung der Membranproteine Die submitochondrialen Partikel (SMPs) wurden auf Eis aufgetaut und mit SEM-Puffer 1:3 auf 100 ml verdünnt und mit 60 μl PMSF (2,5 % in Ethanol) versetzt. Zu dieser Lösung wurde 1,05 g in SEM-Puffer gelöstes DDM (Endkonzentration 1,4 %) langsam zugetropft und in einem Homogenisator langsam gemischt. Die Mischung wurde 10 min bei Raumtemperatur und 10 min bei 4 C gerührt. Anschließend wurde sie 2:1 auf 150 ml Endvolumen mit SEM-Puffer verdünnt, erneut 10 min bei 4 C gerührt und zentrifugiert (Beckman Instruments L7, Rotor Ti60, rpm, 30 min, 4 C). Anionenaustauschchromatographie HQ20 Das Solubilisat wurde steril filtriert (Whatman, Filtereinheit 0,45 μm, Celluloseacetat, steril) und auf eine Anionenaustauschsäule Poros HQ20 aufgetragen. Die Chromatographie erfolgte an der Amersham ÄKTApurifier Workstation mit den in Tabelle 3.3 aufgeführten Parametern. Die Fraktionen wurden auf Hydrolyseaktivität überprüft (siehe Kapitel 3.9.1), aktive Fraktionen (A6 - A11) vereinigt und in Konzentratoren (Millipore Amicon Ultra-15, Cellulose, MW- CO) auf 1 ml aufkonzentriert. Zur Lagerung bei -80 C wurde die Proteinlösung in flüssigem Stickstoff eingefroren. Gelpermeationschromatographie S-300 Die Proteinlösung wurde auf Eis aufgetaut, auf die Gelpermeationssäule S-300 aufgetragen und die Chromatographie mit den in Tabelle 3.3 aufgeführten Parametern durchgeführt. Bei der Isolierung des Stammes YEBTA-c-γl-b-K174C wurde die Chromatographie im Dunklen durchgeführt. Die Fraktionen wurden auf Hydrolyseaktivität untersucht, aktive Fraktionen mit einer Absorbanz von A 278nm > 0,6 V (Fraktion 41-45), sowie aktive Fraktionen mit einer Absorbanz A 278nm > 0,5 V < 0,6 V (Fraktion 38-40, 46) wurden vereinigt und aufkonzentriert (Millipore Amicon Ultra-15, Cellulose, MWCO). Das Konzentrat wurde in 250 μl Strohhalmen fraktioniert, schockgefroren und unter flüssigem Stickstoff gelagert. 42
61 3.2 Isolation der H + -ATPsynthase aus Saccharomyces cerevisiae (YF 0 F 1 ) Saccharomyces cerevisiae- Zellen Mechanischer Zellaufschluss Zentrifugation Überstand: Mitochondrien und andere Zellorganelle Sediment: Zelltrümmer und Zellkerne Zentrifugation Überstand: Cytosol und kleinere Zellorganellen Sediment: Mitochondrien Beschallung Ultrazentrifugation Überstand Sediment: SMPs Solubilisierung Ultrazentrifugation Überstand: Protein-Detergenz-Komplex Sediment: Membranen Anionenaustauschchromatographie Größenausschlusschromatographie S-300 YF 0 F 1 Abbildung 3.4: Isolation von H + -ATPsynthase aus Saccharomyces cerevisiae: Schematischer Verlauf der Isolation von YF 0 F 1. 43
62 3 Biochemische Methoden Tabelle 3.3: Parameter der Chromatographien für die Isolation von YF0F1. Die Reinigung und Isolation der H + -ATPsynthase erfolgte durch eine Anionenaustauschchromatographie Poros HQ20 und eine weitere Gelpermationschromatographie S-300. Parameter HQ20 S-300 Säulenvolumen (CV) 16 ml 230 ml Säulenmaterial Poros, 20 HQ Sephacryl HR Äquilibrierungspuffer Niedrigsalzpuffer Niedrigsalzpuffer Elutionspuffer Hochsalzpuffer Niedrigsalzpuffer Äquilibrierungsvolumen 80 ml (5 CV) - Probenvolumen 150 ml 1 ml Flussrate 5 ml/min 20 ml/min Größe der Elutionsfraktionen 2 ml 2 ml Detektion UV/VIS (280/488 nm), Leitfähigkeit UV/VIS (278 nm) Gradient Stufengradient: 21 % bzw % (für YEBTA-c-γl-b-K174C) Elutionspuffer 44
63 3.3 Fluoreszenzmarkierung 3.3 Fluoreszenzmarkierung Um Fluoreszenzmessungen an Proteinen durchführen zu können, müssen diese mit Fluoreszenzfarbstoffen markiert werden. Werden zur Markierung von Proteinen kommerziell erhältliche organische Fluoreszenzfarbstoffe mit einer Maleimidkopplungsgruppe verwendet, können diese durch eine Michael-Addition an die Thiolgruppe von Cysteinen kovalent angebunden werden. O O FL N + Protein SH FL N Protein S O O Die Selektivität hängt dabei von der Lage des zu markierenden Cysteins im Enzym und der Struktur des Fluoreszenzfarbstoffes ab. Cysteine an der Proteinoberfläche werden leichter markiert, aber auch Cysteine im Inneren des Proteins können durch Anwendung eines Fluorophors mit einem langen Linker zwischen Farbstoff und Maleimidgruppe markiert werden. Deshalb unterscheiden sich je nach verwendetem Farbstoff die Markierungsbedingungen wie Temperatur, Konzentration des Farbstoffes und Inkubationszeit. Markierung von YF 0 F 1 aus YEBTA-c-γ1-b-K174C mit ATTO565 HEPES-Puffer HEPES-NaOH, ph 7,8 MgCl 2 40 mm 4mM ATTO565-Stammlösung ATTO565 2,37 mm in DMSO Die GFP enthaltenden YF 0 F 1 aus YEBTA-c-γ1-b-K174C wurden mit ATTO565 markiert, wodurch eine doppeltmarkierte Probe erhalten wurde. Es wurden 6,5 μm Enzym und 19,5 μm Fluorophor ATTO565 gemischt und unter Lichtausschluss 2 min auf Eis inkubiert. Nach Ablauf der Inkubationszeit wurde nicht gebundener Fluorophor über zwei nacheinanderfolgende Penefsky-Säulen (Sephadex G50 fine in HEPES-Puffer mit 0,05 % DDM suspendiert, vorzentrifugiert Heraeus Megafuge 1.0R, rpm, 2 min) abgetrennt (Heraeus Megafuge 1.0R, rpm, 3 min). Der Markierungsgrad und die Selektivität wurden durch UV/VIS- Spektroskopie (siehe Kapitel 3.4.1) und SDS-PAGE (siehe Kapitel 3.5) bestimmt. 45
64 3 Biochemische Methoden 3.4 Konzentrationsbestimmungsmethoden Konzentrationsbestimmung mit UV/VIS-Spektroskopie Proteine absorbieren Licht bei einer Wellenlänge von 278 nm. Dies kann zur Konzentrationsbestimmung herangezogen werden. Auch die Konzentration von Fluorophoren kann durch Aufnahme eines Absorptionsspektrums bestimmt werden. Durch Messung des Absorptionsspektrums des markierten Proteins kann somit auch dessen Markierungsgrad bestimmt werden. Die Konzentration kann mit dem Lambert Beer schen Gesetzt berechnet werden (vgl. Gleichung 3.1). c = A ɛ d (3.1) c Konzentration (M) A Absorbanz ɛ molarer Absorptionskoeffizient (M 1 cm 1 ) d Schichtdicke der Küvette = 1 cm Der Markierungsgrad wird mit Gleichung 3.2 berechnet. Markierungsgrad = [Fluorophor] [Protein] 100% = A max(fluorophor) ɛ max(fluorophor) A 278 (Protein) ɛ 278 (Protein) (3.2) mit A 278 (Protein) = A 278 (total) A 278 (Fluorophor) = A 278 (total) ɛ 278(Fluorophor) ɛ max (Fluorophor) A max(fluorophor) Die Messungen wurden am Spektrometer Perkin Elmer Lambda 2 durchgeführt. Zunächst wurde mit dem verwendeten Messpuffer (z.b. Liposomenpuffer) der Untergrund im Messbereich gemessen, dann wurden 2-10 μl der Proteinprobe auf ein Endvolumen von 80 μl mit dem Messpuffer verdünnt und das Absorptionsspektrum aufgenommen. Für reines Protein lag der Messbereich zwischen 240 und 340 nm, bei markiertem Enzym wurde das Spektrum von 240 nm bis 700 nm gemessen. Folgende molare Absorptionskoeffizienten wurden in dieser Arbeit verwendet: 46
65 3.4 Konzentrationsbestimmungsmethoden EF 0 F 1 ɛ 278 = M 1 cm 1 [74] MF 0 F 1 ɛ 278 = M 1 cm 1 [68] GFP ɛ 278 = M 1 cm 1 [125] ɛ 487 = M 1 cm 1 [125] ATTO565 ɛ 278 = M 1 cm 1 [126] ɛ 565 = M 1 cm 1 [126] Konzentrationsbestimmung von Proteinen mit einem Bicinchoninsäure-Test Der Bicinchoninsäure-Test (BCA) ist ein sensitiver und wenig störanfälliger Test, bei dem durch die Anwesenheit von Proteinen Cu 2+ zu Cu + reduziert wird. Dieses bildet mit BCA (siehe Abbildung 3.5) einen violetten Farbkomplex, welcher bei 562 nm spektrometrisch quantitativ über eine Eichgerade bestimmt werden kann. Abbildung 3.5: Strukturformel von Bicinchoninsäure Bild aus Datum: Die Proteinbestimmung wurde nach der Vorschrift des Herstellers (Uptima) durchgeführt. Die Proben wurden 20 min bei 60 C inkubiert und dann die Absorbanz bei 562 nm am UV/VIS- Spektrometer Perkin Elmer Lambda 2 gemessen. Um die Proteinmenge zu bestimmen, wurde parallel eine Kalibrierung durchgeführt. Dazu wurden 6 verschiedene Konzentrationen BSA- Standard (2 mg/ml) im Bereich von 0-20 μg Protein gewählt Konzentrationsbestimmung von Proteinen mit Amidoschwarz Eine weitere Möglichkeit der Proteinbestimmung ist die Methode von Popov et al. [127] durch die Adsorption des Farbstoffs Amidoschwarz (siehe Abbildung 3.6) an das Protein. 47
66 3 Biochemische Methoden Abbildung 3.6: Strukturformel von Amidoschwarz 10B Bild aus Black.svg Datum: Stammlösung Amidoschwarz Amidoschwarz 13 g/l in Säure vor Gebrauch filtrieren, Lagerung RT Säure Essigsäure (99,8 %) Methanol Lagerung RT 1 Teil 9 Teile Färbelösung Stammlösung Amidoschwarz Säure Lagerung RT 1 Teil 49 Teile NaOH (1 M) in H 2 O Lagerung RT Die Messung wurde wie bei Fischer [124] beschrieben durchgeführt. Die Proteinproben wurden mit 1 ml Färbelösung gemischt, 10 min bei Raumtemperaur inkubiert und die Lösung zentrifugiert (Heraeus Biofuge 17RS, Rotor 1243, rpm, 5 min, 4 C). Der Überstand wurde abgegossen, das Pellet in 1 ml Säure gelöst und erneut zentrifugiert (Heraeus Biofuge 17RS, Rotor 1243, rpm, 5 min, 4 C). Dieser Waschschritt wurde nochmals wiederholt und das Pellet anschließend in 1 ml NaOH gelöst. Die Absorption wurde bei 615 nm mit dem Spektrometer Perkin Elmer Lambda 2 gemessen. Die Kalibrierung wurde mit Hilfe von 6 verschiedenen Konzentrationen BSA-Standard (2 mg/ml, 0-20 μg Protein) durchgeführt. Vergleich der Proteinkonzentrationen Beim Vergleich wurde eine Proteinprobe jeweils dreifach mit jeder Bestimmungsmethode gemessen. Für die Umrechnung wurde eine Molmasse von 531 kda für EF 0 F 1 eingesetzt, die Ergebnisse sind in Tabelle 3.4 zusammengefasst. 48
67 3.5 Polyacrylamidgelelektrophorese (SDS-PAGE) Tabelle 3.4: Vergleich der Methoden zur Konzentrationsbestimmung. Bestimmung einer EF 0 F 1 -Probe nach Aufreinigung mittels Chromatographie (vgl. Kapitel mit UV/VIS-Spektroskopie (UV/VIS), Bicinchoninsäure-Test (BCA) und mit Amidoschwarz (Amidoschwarz). UV/VIS BCA Amidoschwarz g/l - 2,4 0,8 μm 2,3 4,5 1,6 Faktor 1,0 2,0 0,7 Daraus ergibt sich für die Proteinbestimmung durch den BCA-Test eine um den Faktor zwei höhere Proteinkonzentration im Vergleich zu der aus der Absorption bei 278 nm gemessenen Proteinkonzentration. Die Bestimmung mit Amidoschwarz lieferte nur 70 % der Proteinkonzentration nach UV/VIS-Bestimmung. Im Folgenden wurden alle Proteinkonzentrationen auf die Bestimmung durch UV/VIS umgerechnet. 3.5 Polyacrylamidgelelektrophorese (SDS-PAGE) Gelelektrophorese Die Reinheit der isolierten Proteine sowie die Markierung der Untereinheiten wurde mit SDS- Gelelektrophorese mit Hilfe eines diskontinuierlichen Lämmli-Gels [128] mit 4 %igem Sammelund 13 %igem Trenngel überprüft. Verwendet wurde die Halterung Mini Protean 3 Cell von BioRad. Gelpuffer Tris-HCl ph 8,45 3 M SDS (w/v) 0,3 % Acrylamidstammlösung Acrylamid/Bisacrylamid 40 % fertige Lösung von Sigma APS-Lösung APS (w/v) 10 % Ethanol-Lösung Ethanol (v/v) 50 % 49
68 3 Biochemische Methoden Probenpuffer Tris-HCl ph mm SDS (w/v) 10 % DTT 5 mm Glycerin (v/v) 50 % BPB (w/v) 0,2 % Anodenpuffer Tris-HCl ph 8,9 200 mm Kathodenpuffer Tris-HCl 100 mm Tricin 100 mm SDS (w/v) 0,1 % Es wurde das Trenngel gegossen, mit Ethanol überschichtet und 30 min bis zur vollständigen Polymerisation abgewartet. Nach Entfernen des Ethanols wurde das Sammelgel aufgetragen und ebenfalls mindestens 30 min gewartet. Das Pipettierschema ist in Tabelle 3.5 aufgeführt. Die Proben wurden mit 5 μl Probenpuffer 20 min bei 90 C inkubiert und mit einer Hamiltonspritze auf das Sammelgel aufgetragen. Die Elektrophorese wurde im Bereich des Sammelgels mit einer Stromstärke von 20 ma, im Bereich des Trenngels mit 40 ma durchgeführt. Bei fluoreszenzmarkierten Proben wurde die Apparatur abgedunkelt. Direkt anschließend konnte die Fluoreszenz unter der UV-Lampe betrachtet werden, oder die Proteinbanden mit Coomassie Blau oder kolloidalem Silber gefärbt werden. Tabelle 3.5: Pipettierschema für ein diskontinuierliches Lämmli-Gel [128] Trenngel 13 % Sammelgel 4 % Acrylamidstammlösung 1800 μl Acrylamidstammlösung 188 μl Gelpuffer 1840 μl Gelpuffer 465 μl Wasser 1880 μl Wasser 1215 μl TEMED 8 μl TEMED 4 μl APS-Lösung 28 μl APS-Lösung 18 μl Färbung mit Coomassie Blau Das Protein im Gel kann durch die Anlagerung von Coomassie Blau an die basischen Seitenketten der Aminosäuren sichtbar gemacht werden, die Nachweisgrenze liegt dabei für das hier verwendete Coomassie Brillant Blue R250 (siehe Abbildung 3.7) bei 0,1 μg Protein [129]. Färbelösung Ethanol (v/v) 25 % Essigsäure (v/v) 10 % Coomassie Blau (w/v) 0,1 % Entfärber Ethanol (v/v) 30 % Essigsäure (v/v) 10 % 50
69 3.5 Polyacrylamidgelelektrophorese (SDS-PAGE) Abbildung 3.7: Strukturformel von Coomassie Brillant Blue R250 Bild aus Brilliant_Blue_R-250.svg Datum: Das aus der Halterung entnommene Gel wurde in Färbelösung gelegt, kurz in der Mikrowelle (20 s, 600 W) erhitzt und 15 min auf dem Schüttler (Janke&Kunkel KS 500, 50 rpm) inkubiert. Anschließend wurde so lange mit Entfärber inkubiert und dieser bei Bedarf gewechselt, bis die Banden die gewünschte Intensität aufwiesen. Das Gel wurde zur Dokumentation fotografiert und in Wasser aufbewahrt Färbung mit kolloidalem Silber Das Gel konnte entweder direkt nach der Elektrophorese oder nach der Färbung mit Coomassie Blau mit kolloidalem Silber angefärbt werden. Die Silberfärbung ist 10- bis 100-fach empfindlicher als die Färbung mit Coomassie Blau [130]. Sensibilisierungslösung Ethanol (v/v) 30 % Natriumacetat 83 mm frisch zugeben: Glutaraldehyd 13 mm Na 2 S 2 O 3 5H 2 O 8 mm Stopbad Na 2 -EDTA 2H 2 O 40 mm Versilberungslösung AgNO 3 frisch zugeben: Formaldehyd Entwickler Na 2 S 2 O 3 5H 2 O frisch zugeben: Formaldehyd 15 mm 5 mm 236 mm 2,5 mm 51
70 3 Biochemische Methoden Wurde das Gel direkt nach der Elektrophorese und nicht nach einer Coomassie-Färbung mit Silber angefärbt, musste es zunächst zur Fixierung eine halbe Stunde in 50 ml Entfärber inkubiert werden. Anschließend wurde es in folgender Reihenfolge mit den entsprechenden Lösungen behandelt und auf dem Schüttler inkubiert (Janke&Kunkel KS 500, 50 rpm): 25 min in 50 ml Sensibilisierungslösung 3 x 5 min in Wasser 20 min in 50 ml Versilberungslösung 2 x 1 min in Wasser 1 min in 50 ml Entwicklerlösung 3-10 min bis zum Erreichen der gewünschten Farbintensität in 50 ml Entwicklerlösung, sofort entnehmen und 30 min in Stopbad Das Gel wurde anschließend zur Dokumentation fotografiert und in Wasser aufbewahrt. 3.6 Darstellung von Dialyseliposomen Die Liposomen wurden nach der Vorschrift von Fischer [124] hergestellt. Beschallungspuffer Tricin-NaOH, ph 8,0 EDTA DTT Cholsäure Desoxycholat Lagerung -20 C 10 mm 0,1 mm 0,5 mm 7,2 g/l 3,6 g/l Dialysepuffer 15-fach Tricin-NaOH, ph 8,0 MgCl 2 EDTA DTT 150 mm 37,5 mm 3 mm 3,75 mm PtdCho-Lösung (25 g/l) Phosphatidylcholin aus Hühnereigelb in Chloroform Lagerung -20 C, lichtgeschützt PtdAc-Lösung (2,5 g/l) Phosphatidsäure aus Hühnereigelb in Chloroform Lagerung -20 C, lichtgeschützt 52
71 3.7 Rekonstitution von F 0 F 1 in Dialyseliposomen Es wurden 6,8 ml PtdCho-Lösung und 3,6 ml PtdAc-Lösung gemischt und das Chloroform unter Stickstoff im Rotationsverdampfer entfernt. Das trockene Lipid wurde in 10 ml Beschallungspuffer gelöst und auf Eiswasser dreimal 30 Sekunden lang beschallt (Branson Sonifier 250, Mikrospitze, Stufe 3, Duty cycle constant). Nach der Beschallung wurde die Lipidlösung langsam bei -30 C über Nacht eingefroren. Das Einfrieren unterstützt die Bildung von homogenen Vesikeln mit einer Lipiddoppelschicht [131] und sollte nicht übersprungen werden. Nach dem Auftauen wird die Dialyse über einen Zeitraum von fünf Stunden bei 30 C durchgeführt. Die daraus erhaltenen Liposomen hatten eine Lipidkonzentration von 16 g/l. Sie wurden schockgefroren und unter flüssigem Stickstoff gelagert. 3.7 Rekonstitution von F 0 F 1 in Dialyseliposomen Die Rekonstitution von F 0 F 1 durchgeführt. in Dialyseliposomen wurde wie bei Fischer [124] beschrieben, Liposomenpuffer Succinat-NaOH, ph 8,0 Tricin KCl NaCl Lagerung -20 C 20 mm 20 mm 0,6 mm 80 mm MgCl 2 -Stammlösung (1 M) MgCl 2 6H 2 OinH 2 O Triton X-100 fertige Lösung von Roche, 10 % (w/v) in H 2 O Lagerung 4 C In einem 200 μl Ansatz wurden 100 μl Dialyseliposomen (8 g/l entspricht einer Konzentration von 48 nm [28]) mit 0,5 μl MgCl 2 und Protein (Endkonzentration 100 nm) gemischt und mit Liposomenpuffer auf 184 μl aufgefüllt. Unter Vortexen wurden 16 μl Triton X-100 zugegeben und der Ansatz 1 h bei Raumtemperatur langsam gerührt. Zum Entfernen des Detergenz wurden ca. 64 mg mit Wasser gewaschene und getrocknete BioBeads zugegeben und erneut langsam 1 h gerührt. Die überstehende Lösung wurde von den BioBeads abgezogen und konnte direkt für Hydrolyse- und Synthesemessungen verwendet werden. 53
72 3 Biochemische Methoden 3.8 Messung der ATP-Synthese Die Synthese von ATP durch die H + -ATPsynthase wurde mittels eines gekoppelten Enzymtests nach der One-Step-Methode [124] gemessen. Die ATP-Konzentration ist proportional zur Lumineszenzintensität des verwendeten Luciferin/Luziferase-Systems (ATP-Monitoring-Kit, Roche). LI Succinat-NaOH, ph 4,7 NaH 2 PO 4 KOH Lagerung -20 C MgCl 2 * ADP* Valinomycin* 20 mm 5 (10) mm 0,6 mm 2,5 (4) mm 0,1 mm 20 μm LII Tricin-NaOH, ph 8,8 NaH 2 PO 4 KOH Lagerung -20 C MgCl 2 * ADP* *frisch zugeben 200 (250) mm 5 (10) mm 160 mm 2,5 (4) mm 0,1 mm Valinomycin (10 mm) in Methanol Lagerung 4 C ATP-Monitoring Kit in Wasser 2-fach konzentriert Lagerung -20 C ATP Standard (15 μm) Dinatriumsalz in Wasser Lagerung -20 C ADP Standard (100 mm) Monokaliumsalz in Wasser Lagerung -20 C Die Werte sind für die ATP-Synthesemessungen mit EF 0 F 1 angegeben, die Werte in Klammern gelten für die mitochondriale H + -ATPsynthase Kalibrierung der ATP-Synthesemessung Die in der ADP-Lösung vorhandene Verunreinigung durch ATP wurde durch Zugabe einer definierten Menge ATP-Standard (5-10 μl) zu einer Lösung aus ATP-Monitoring-Kit (20-30 μl) und LII-Puffer (ad μl) bestimmt. Die ATP-Konzentration des Untergrundsignals wurde daraufhin zur Kalibrierung der ATP-Synthesemessung verwendet. Abbildung 3.8 zeigt eine solche Kalibrierung. Die Lumineszenzintensität wurde mit einem Luminometer BioOrbit 1250 detektiert und über ein Datenerfassungsmodul (National Instruments Ni USB-6009) und mit Hilfe eines LabView11-Programmes digitalisiert. Die Messungen wurden in Küvetten mit 54
73 3.8 Messung der ATP-Synthese 0,03 Lumineszenzintensität / V 0,02 0,01 0,00 H U Nulllinie H E Eichsignal Untergrundsignal Zeit Abbildung 3.8: Kalibrierung der Lumineszenzintensität: Untergrundsignal H U entspricht LII-Puffer+ATP-Monitoring-Kit, Eichsignal H E entspricht Signal nach der Zugabe von ATP-Standard. hoher Lichtdurchlässigkeit durchgeführt (GreinerBio-One Test Tubes 12/55, 4 ml, PS). Aus den Signalhöhen H U und H E konnte mit der Gleichung 3.3 die ATP-Konzentration im LII- Puffer bestimmt werden. Für die Kalibrierung wurde die Eichmessung mindestens dreimal durchgeführt und der Mittelwert gebildet. c LII = H U V 2 c st H E V 1 (3.3) c LII c st H U H E V 1 V 2 ATP-Konzentration im LII-Puffer ATP-Konzentration des Eichsignals Höhe des Untergrundsignals Höhe des Eichsignals Volumen LII Volumen LII + ATP-Monitoring-Kit 55
74 3 Biochemische Methoden ATP-Synthesemessung Nach Einstellung der Gerätenulllinie wurden in einer Küvette (GreinerBio-One) 870 μl LII- Puffer und 30 μl ATP-Monitoring-Kit vorgelegt und das Untergrundsignal aufgezeichnet. LI-Puffer (90 μl) wurde mit 20 μl Proteoliposomen vermischt und 2 Minuten inkubiert. Anschließend wurden 100 μl der Proteoliposomenlösung mit Hilfe einer Hamiltonspritze über ein lichtundurchlässiges Septum vollständig in die Küvette gespritzt. Das erhaltene Signal ist in Abbildung 3.9 gezeigt. Die Auswertung erfolgte mit dem Programm Origin 7.0 durch eine nichtlineare Kurvenanpassung nach Gleichung 3.4. Mit den daraus erhaltenen Parametern wurde die Steigung m zum Zeitpunkt t = 0 nach Gleichung 3.5 berechnet. 0,05 Lumineszenzintensität / V 0,04 0,03 0,02 0,01 0,00 Nulllinie H U m Zugabe der Proteoliposomenlösung Zeit Abbildung 3.9: ATP-Synthesemessung: Untergrundsignal H U entspricht LII-Puffer +ATP- Monitoring-Kit, aus der Anfangssteigung m lässt sich die Geschwindigkeit der ATP-Synthese (turnover) berechnen. f(t) =a e t b + c + dt (3.4) f (t) =m = a b + d (3.5) Aus der Steigung m und dem Untergrundsignal H U (Wechselzahl) der ATP-Synthese berechnet werden. konnte mit Gleichung 3.6 der turnover 56
75 3.8 Messung der ATP-Synthese v = m c LII V 1 H U c F0 F 1 V 2 (3.6) v turnover der ATP-Synthese (s 1 ) m Anfangssteigung der ATP-Synthese (V s 1 ) c LII H U V 1 V 2 c F0 F 1 ATP-Konzentration im LII-Puffer (M) Höhe des Untergrundsignals (V) Volumen LII (μl) Volumen LII + ATP-Monitoring-Kit (μl) Proteinkonzentration im Messansatz (M) Jede Messung wurde dreimal durchgeführt und der Mittelwert, sowie die Standardabweichung gebildet Optimierung der Reaktionsbedingungen bei der Messung der Phosphatabhängigkeit der H + -ATPsynthase Zur Untersuchung der Phosphatabhängigkeit der H + -ATPsynthase sollte der turnover der ATP- Synthese bei verschiedenen Phosphatkonzentrationen (0, 0,1, 0,2, 0,3, 0,5, 1, 2, 5, 10 und 15 mm) und bei verschiedenen ph a -Werten gemessen werden. Da der Reaktionspuffer aus den Puffern LI und LII besteht und die Geschwindigkeit der ATP- Synthese stark von den ph-werten (ph der LI-Lösung und ph der Mischung aus LI und LII) abhängt, wurde der Einfluss des Phosphats auf die Pufferkapazität der Puffer überprüft. Es wurde der ph-wert von LI vor und nach der Zugabe von Proteoliposomen und der ph-wert des LII-Puffers vor und nach einem ph-sprung gemessen. Dazu wurden zu 900 μl LII-Puffer 100 μl LI-Ansatz (90 μl LI-Puffer + 20 μl Proteoliposomen) gegeben. Es konnte keine Änderung des ph-wertes bei den verschiedenen Phosphatkonzentrationen gemessen werden. Zusätzlich sollte der turnover der ATP-Synthese bei verschiedenen ph a -Werten im Bereich von 7,3 bis 8,8 gemessen werden. Durch einen niedrigeren ph a wird insgesamt der ΔpH verringert und der turnover der Synthese wird deutlich kleiner. Um ein gut detektierbares Signal zu erhalten, muss deshalb die Konzentration an aktivem Enzym im Messansatz erhöht werden. Dazu sind verschiedene Möglichkeiten denkbar. Die Erhöhung der Proteinkonzentration im Rekonstitutionsansatz ist begrenzt durch die Vorgabe, dass im Mittel nur 2-3 Enzyme pro Liposom vorhanden sein sollen (entspricht einer Enzymkonzentration von 100 nm) [124]. Bei 57
76 3 Biochemische Methoden einer weiteren Erhöhung des Enzym zu Liposom Verhältnisses nimmt der turnover pro Enzym wieder ab, da in diesem Fall die generierte elektrochemische Potentialdifferenz der Protonen zu schnell abgebaut wird. Eine weitere Möglichkeit bessere Signale zu erhalten, ist die Erhöhung der Proteoliposomenkonzentration im Messansatz. Dies kann entweder durch die Aufkonzentrierung der Proteoliposomenlösung realisiert werden oder indem ein größeres Volumen an Proteoliposomen zugegeben wird. Konzentrierung der Proteoliposomen Um eine höhere Konzentration an aktivem Enzym in Proteoliposomen zu bekommen, wurden 600 μl Proteoliposomen 15 min bei g zentrifugiert (Heraeus Biofuge RS28, Rotor3740, rpm, RT), das Pellet in Liposomenpuffer (siehe Kapitel 3.7) resuspendiert und die Suspension auf ein Endvolumen von 200 μl mit Liposomenpuffer aufgefüllt. Um den Anteil an verbliebenen Proteoliposomen im Überstand zu bestimmen, wurde die optische Dichte bei 600 nm im UV/VIS-Spektrometer gemessen. Diese Messungen ergaben, dass 1/3 der anfänglich vorhandenen Proteoliposomen im Überstand verblieben waren, daraus folgt eine Konzentrierung auf das Doppelte nach der Resuspension des Pellets. Synthesemessungen mit dieser Probe zeigten einen turnover von 2 s 1 im Vergleich zur Standardprobe mit einem turnover von 13 s 1. Bei der Konzentrierung der Proteoliposomen sank die Syntheseaktivität auf 15 % der Ursprungsaktivität ab, damit ist diese Methode ungeeignet. Grund für den Aktivitätsverlust könnte sein, dass bei der Konzentrierung Aggregate aus Liposomen entstehen und die F 1 -Teile bei der Resuspension vom F 0 -Teil abgetrennt werden. Vergrößerung des Volumens von Proteoliposomen im LI-Ansatz Bei der Standardsynthesemessung wurden Proteoliposomen und LI-Puffer im LI-Ansatz im Verhältnis 20:90 μl (einfache Proteoliposomenmenge) gemischt und davon 100 μl in den Reaktionsansatz gegeben (vgl. Kapitel 3.8.2). Um die Menge an aktivem Enzym im Reaktionsansatz zu erhöhen wurden Proteoliposomen und LI-Puffer im Verhältnis 40:70 μl (doppelte Proteoliposomenmenge) und 60:50 μl (dreifache Proteoliposomenmenge) gemischt. Da der LI-Puffer eine kleine Pufferkapazität hat, erhöhte sich durch die Zugabe von größeren Volumen Proteoliposomen (ph = 8) der ph-wert von LI. Um diesem Effekt entgegen zuwirken, musste der ph-wert des LI-Puffers verringert werden. Dies konnte jedoch nicht direkt über die Puffersubstanz berechnet werden, da auch die Liposomen zur Pufferkapazität beitragen. Deshalb wurde der LI-Puffer mit Proteoliposomen titriert. Es wurden 10 LI-Puffer mit ph-werten zwischen 3,3 und 4,7 hergestellt, mit der einfachen, doppelten und dreifachen Proteoliposomenmenge gemischt und der End-pH-Wert bestimmt. 58
77 3.9 Messung der ATP-Hydrolyse Um bei der doppelten Proteoliposomenmenge den gewünschten End-pH-Wert von ph i = 4,9 zu erhalten, musste die LI-Lösung vor der Zugabe der doppelten Proteoliposomenmenge auf ph = 4,4, bei der dreifachen Proteoliposomenmenge auf ph = 4,1 eingestellte werden. Um den Einfluss der Vorgehensweise auf die Enzymaktivität zu überprüfen, wurden die Syntheseraten der dreifachen Proteoliposomenmenge (v =9s 1 ) mit der einfachen (v = 15 s 1 ) verglichen und zeigten nur noch 60 % der Ursprungsaktivität. Der Aktivitätsverlust könnte einerseits durch Denaturierungseffekte aufgrund des niedrigeren phs des LI-Puffers kommen, zum Anderen können wie bei der Aufkonzentrierung durch die höhere Proteoliposomenmenge im LI-Ansatz Liposomenaggregate entstehen. Die Messungen der Phosphatabhängigkeit bei den ph a -Werten von 7,6 und 7,3 wurden mit der dreifachen Menge an Proteoliposomen durchgeführt, wobei der ph-wert des LI-Puffers bei 4,1 und nach Zugabe der Proteoliposomen schließlich bei ph i = 4,9 lag. Für die Bestimmung des K M -Wertes spielt der Absolutwert der Geschwindigkeit keine Rolle, für die Berechnung des turnovers bei ATP-Synthese wurde der Aktivitätsverlust um den Faktor 1,7 berücksichtigt. 3.9 Messung der ATP-Hydrolyse Messung der ATP-Hydrolyse nach der One-step-Methode Die Hydrolyse von ATP durch die H + -ATPsynthase wurde mittels eines gekoppelten Enzymtests nach der One-Step-Methode [124] gemessen. Die Reaktion ist schematisch in Abbildung 3.10 dargestellt. Gemessen wird der Verbrauch von NADH bei einer Wellenlänge von 340 nm. Die Abnahme der Absorbanz wurde mit dem Programm UV WinLab aufgezeichnet. H + EF 0 F 1 H + P i + ADP ATP NADH NAD + Pyruvatkinase Pyruvat Phosphoenolpyruvat Lactatdehydrogenase Lactat Abbildung 3.10: Reaktionsschema des gekoppelten Enzymtests nach der One-Step- Methode zur Messung der Geschwindigkeit der ATP-Hydrolyse durch die H + -ATPsynthase. Der Messpuffer wurde auf 37 C temperiert und die Absorbanz bei 340 nm im UV/VIS- Spektrometer Perkin Elmer Lambda 2 aufgezeichnet. Zu 1 ml Puffer wurde die zu messende Probe gegeben (Endkonzentration Protein c = 3 nm) und die Abnahme der Absorbanz 59
78 3 Biochemische Methoden beobachtet (vgl. Abbildung 3.11). Da pro hydrolysiertem ATP ein NADH verbraucht wurde, konnte aus der Steigung m und dem Absorptionskoeffizienten von NADH der turnover der ATP-Hydrolyse nach Gleichung 3.7 berechnet werden. Messpuffer Tris-HCl, ph 8,0 KCl MgCl 2 PEP ATP NADH Pyruvatkinase Lactatdehydrogenase Lagerung -20 C 100 mm 25 mm 4mM 2,5 mm 2 mm 0,4 mm 18 U/ml 16 U/ml Zugabe F 0 F 1 Absorbanz bei 340 nm m Zeit Abbildung 3.11: ATP-Hydrolysemessung nach der One-step-Methode. Aus der Steigung m wird die Geschwindigkeit der ATP-Hydrolyse (turnover) berechnet. T H = m ɛ 340 d c E (3.7) T H turnover der ATP-Hydrolyse (s 1 ) m Abnahme der Absorbanz bei 340 nm (s 1 ) ɛ Absorptionskoeffizient von NADH bei 340 nm (6220 M 1 cm 1 ) d Dicke der Messküvette (1 cm) Konzentration F 0 F 1 (M) c E 60
79 3.9.2 Messung der mit dem Protonentransport gekoppelten ATP-Hydrolyse anhand der Fluoreszenzlöschung von 9-Amino-6-Chloro-2-methoxyacridin (ACMA) 3.9 Messung der ATP-Hydrolyse Bei der Messung der Hydrolyse nach der One-step-Methode erhält man die Menge an hydrolysiertem ATP. Da auch die F 1 -Teile von dissoziierten H + -ATPsynthasen in der Lage sind, ATP zu hydrolysieren ohne dass ein Protonentransport stattfindet, kann anhand der Geschwindigkeit der ATP-Hydrolyse keine Aussage darüber getroffen werden, ob das Gesamtenzym F 0 F 1 intakt ist. Mit Hilfe der Fluoreszenzlöschung von ACMA kann der Aufbau einer transmembranen ph- Differenz und damit die ATP-Hydrolyse gemessen werden (Theorie siehe Anhang A), die mit einem transmembranen Protonentransport verknüpft ist. Die Messung der ATP-Hydrolyse wurde mit einem ACMA-Test wie bei D Alessandro et al [132] beschrieben durchgeführt. Messpuffer-ACMA Tricin-NaOH, ph 8,0 Succinat MgCl 2 KCl Lagerung 4 C 20 mm 20 mm 2mM 50 mm Valinomycin (10 mm) in Methanol Lagerung -20 C ACMA (1 mm) in H 2 O Lagerung -20 C ATP (100 mm) in H 2 O Lagerung -20 C Nigericin (10 mm) in H 2 O Lagerung -20 C Die Messungen wurden mit dem Fluoreszenzspektrometer (Perkin Elmer LS 45) bei einer Anregungswellenlänge von 418 nm durchgeführt und bei einer Emissionswellenlänge von 482 nm detektiert. Der Messpuffer-ACMA wurde vortemperiert und alle Messungen wurden bei 25 C durchgeführt. In einer Quarzglas-Küvette wurden 975 μl Messpuffer-ACMA vorgelegt und unter Rühren in genau dieser Reihenfolge nacheinander 20 μl Proteoliposomen (2 nm Enzym), 0,5 μl Valinomycin (5 μm) und 4,5 μl ACMA (4,5 μm) zugegeben. Nach Erreichen einer stabilen Basislinie wurde die Hydrolyse durch die Zugabe von 10 μl ATP (1 mm) gestartet. Die Fluoreszenzlöschung von 9-Amino-6-Chloro-2-methoxyacridin (ACMA) wurde mit dem Programm FL WinLab aufgezeichnet (siehe Abbildung A.2). Durch Zugabe von 0,5 μl Nigericin 61
80 3 Biochemische Methoden (5 μm) wird die erzeugte ph-differenz abgebaut und die Anfangsfluoreszenz wieder erreicht. Üblicherweise wird die Anfangsfluoreszenz (nach Zugabe von ACMA) am Ende des Versuches (nach Zugabe von Nigericin) nicht wieder erreicht (siehe Abbildung A.2 Anhang A. In diesem Fall wird die Fluoreszenz, die nach Zugabe von Nigericin erreicht wird, als Anfangsfluoreszenz F 0 bezeichnet. Kalibrierung der ACMA-Fluoreszenzlöschung Da aus der Größe der Fluoreszenzlöschung nicht direkt auf die Größe des ΔpH geschlossen werden kann (siehe Kapitel 5.3 und Anhang A), muss für neue Messbedingungen, wie neue Liposomencharge, andere ACMA-Konzentration oder andere F 0 F 1 -Präparation eine Kalibrierung durchgeführt werden. Zu den Proteoliposomen wurde neben Valinomycin auch Oligomycin (4,5 mg/ml) zugegeben, um eine Fluoreszenzlöschung durch Restaktivität der H + -ATPsynthase ausschließen zu können. Diese Proteoliposomen wurden mit 12 verschiedenen Konzentrationen HCl (4, 8, 12, 16, 20, 23, 27, 31, 35, 38, 42, 46 μm) mindestens eine halbe (4-31 μm) bzw. eine Stunde (35-46 μm) inkubiert und dann der ph-wert bestimmt. Nach Zugabe von ACMA (4 μm) wurde der ph-sprung durch die Zugabe einer äquivalenten Stoffmenge an NaOH (4-46 μm) erzeugt und die Fluoreszenzlöschung aufgezeichnet. Anschließend wurde erneut der ph-wert bestimmt und daraus der ΔpH berechnet. Die erhaltenen Messkurven wurden auf die nach der Zugabe von Nigericin erhaltene Fluoreszenz normiert. Anschließend wurde der zeitliche Verlauf der Fluoreszenzlöschung durch einen exponentiellen Fit mit OriginPro 9G (siehe Gleichung 3.8) bestimmt und durch Extrapolation auf den Zeitpunkt t=0dernaoh-zugabe der Anfangswert der Fluoreszenzlöschung (vergleiche Abbildung A.2) berechnet. y = A 1 exp( x t 1 )+y 0 (3.8) Der ΔpH wurde gegen die relative Fluoreszenzlöschung von 9-Amino-6-Chloro-2-methoxyacridin aufgetragen und die Kurve für die Kalibrierung mit der Gleichung 3.9 [132] erstellt. Q ΔpH = A B Q exp( Q + C Q) (3.9) B Q A,B,C Q=F 0 - F = ΔF Konstanten Fluoreszenzlöschung 62
81 4 Fluoreszenzspektroskopische Methoden 4.1 Messung von Fluoreszenzanregungs- und Fluoreszenzemissionsspektren Die Eigenschaften eines Fluorophors können durch Messungen von Anregungs- und Emissionsspektren untersucht werden. Die Quantenausbeute (QA) und das Spektrum eines freien Fluorophors kann sich durch die Kopplung an ein Protein verändern. Mit Hilfe der Fluoreszenzspektroskopie kann auch ermittelt werden, ob sich ein Fluorophorenpaar für Fluoreszenz-Resonanz- Energie-Transfer-Messungen (FRET) eignet. Dazu müssen sich das Emissionsspektrum des Donorfluorophors und das Anregungsspektrum des Akzeptorfluorophors überlappen. Es wurden Anregungs- und Emissionsspektren der freien Fluorophore, der proteingebundenen Fluorophore, der mit GFP markierten H + -ATPsynthasen und der doppeltmarkierten Proteinprobe (siehe Kapitel 3.3) gemessen. Die Proben wurden in einer Quarzküvette (Hellma, 5mm Lichtweg) auf eine Fluorophorendkonzentration von 50 nm und ein Endvolumen von 300 μl mit HEPES-Puffer (ph 7,8, siehe Kapitel 3.3) verdünnt. Die Spektren wurden mit einem Fluoreszenzspektrometer (Perkin Elmer LS 45) bei 20 C, einer Anregungswellenlänge von 460 nm und einer Emissionswellenlänge von nm gemessen. 4.2 Total-Reflexions-Fluoreszenz-Mikroskopie (TIRF) Bei der TIRF-Mikroskopie wird eine Probe durch ein sogenanntes evaneszentes Feld angeregt. Dieses entsteht an der Grenzfläche im dünneren Medium beim Übergang von einem optisch dichteren in ein optisch dünneres Medium. Der Winkel des eingestrahlten Lichts muss so gewählt sein, dass es zur Totalreflexion kommt [113]. Mit Hilfe der TIRF-Mikroskopie wurden Aufnahmen der Hefezellen des Stammes YEBTA-cγl-b-K174C gemacht, die grün fluoreszierendes Protein (EGFP) exprimierten. Die Messungen wurden an einem konfokalen Aufbau (Aufbau II, Eigenbau Michael Börsch, siehe Abbildung 4.1) mit einem Olympus IX 70 Mikroskop als Kernkomponente und einer angeschlossenen DCC Kamera (Rosperscientific; LN/CCD-1340/400-EB) durchgeführt. Dazu wurden 20 μl Hefezellen 63
82 4 Fluoreszenzspektroskopische Methoden in STA-Puffer (siehe Kapitel 3.3) mit 50 μl Wasser auf einen Objektträger (PlanAPO 60 mm, N. A. 1.45, Olympus) gemischt und mit einem Diodenlaser der Wellenlänge 488 nm (OXX COL-CE Laserdiodenmodul, 50 mw) mit voller Leistung angeregt. Die von der Probe emittierte Strahlung wurde durch einen dichroitischen Spiegel (DCLP 545 nm, AHF) vom Streulicht getrennt, mit der mit flüssigem Stickstoff gekühlten CCD-Kamera aufgenommen und mit dem Programm WinView dargestellt. Probe Dichroitischer Spiegel Klappspiegel CCD Abbildung 4.1: Fotografie des TIRF-Mikroskops mit schematischer Darstellung des Strahlengangs. blau: Anregungslicht der Wellenlänge 488 nm, grün: Fluoreszenz des grün fluoreszierenden Proteins (EGFP). 64
83 4.3 Fluoreszenz-Resonanz-Energie-Transfer-Messungen an Einzelmolekülen (FRET) 4.3 Fluoreszenz-Resonanz-Energie-Transfer-Messungen an Einzelmolekülen (FRET) Alle Messungen an Einzelmolekülen der doppeltmarkierten Probe mit EGFP/ATTO565 (siehe Kapitel 3.3) wurden bei 19 C an einem konfokalen Fluoreszenzmikroskop durchgeführt (schematischer Aufbau siehe Abbildung 4.2). Probe im konfokalen Volumen APD (Donor) Objektiv Sperrfilter APD (Akzeptor) dichroitische Spiegel Filter Klappspiegel Spiegeltreppe Teleskop Glasfaser Laser 476 nm Lochblende CCD Kamera Abbildung 4.2: Schmatischer Aufbau des konfokalen Fluoreszenzmikroskops. Die Anregung mit 476 nm erfolgte mit einem Krypton-Ionen-Laser (Inova 90C, Coherent, 530/476 nm), der auf eine Leistung von 300 μw reguliert wurde. Der Laserstrahl wurde mit einer Glasfaser auf ein Teleskop gerichtet und mit zwei Sammellinsen (f 1 = 40 mm, f 2 = 200 mm) über eine Spiegeltreppe und einen dichroitischen Spiegel (RDC 488 nm, AHF) in die Probe fokussiert. Das emittierte Fluoreszenzlicht wurde über den dichroitischen Spiegel auf eine Lochblende (Durchmesser 85 μm, Owis) fokussiert, um Strahlung außerhalb des Beobachtungsvolumens auszublenden. Das Fluoreszenzlicht des Donors wurde durch einen zweiten dichroitischen Spiegel (DCLP 545 nm, AHF) vom emittierten Licht des Akzeptors getrennt. Die Detektion erfolgte durch Avalanche-Photodioden (APD, SPCM AQR151, EGG). Zum Abtrennen von Licht außerhalb der emittierten Donor- bzw. Akzeptorwellenlänge, befand sich vor der Photodiode des Donorkanals ein Sperrfilter, welcher nur im Bereich nm (525/50 HQ) und vor der Photodiode des Akzeptorkanal ein Sperrfilter, welcher im Bereich 65
84 4 Fluoreszenzspektroskopische Methoden (608/60 HQ) durchlässig war. Die registrierten Signale der APDs wurden mit einer Zeitauflösung von 0,2 ms von einer PC-Karte (TimeHarp 200, PicoQuant) registriert Justage des konfokalen Mikroskops Alexa488-Stammlösung ATTO μm in DMSO Die Justage des Aufbaus erfolgte wie in Anhang D beschrieben. Die Feinjustage wurde an jedem Messtag durchgeführt und mit der Fluoreszenz-Korrelations-Spektroskopie (FCS, siehe Kapitel 1.3.2) überprüft. Dazu wurden 50 μl Immersionswasser auf das Objektiv aufgetragen, die Probenhalterung mit Deckgläschen (Roth, 24mm) eingesetzt und 1 μl einer 1:1000 Verdünnung der Alexa488-Stammlösung (200 nm) mit 89 μl Lipsomenpuffer auf dem Deckgläschen gemischt. Anschließend wurde in die Probe fokussiert und diese mit einer Leistung von 150 μw angeregt. Mit dem Programm SymPhoTime Version 5 wurde die Fluoreszenz 60 Sekunden lang im Donorkanal aufgezeichnet und aus der erhaltenen Spur eine Autokorrelationsfunktion berechnet. Diese wurde mit der Modell-Funktion 4.1 für eine dreidimensionale Diffusion [133] angepasst. G(τ C )=G D (τ C ) G T (τ C ) (4.1) Die Fitfunktion setzt sich aus einem Diffusionsterm (Gleichung 4.2) und einem Term für die Triplettbevölkerung (Gleichung 4.3) zusammen. G D (τ C )= 1 N ( )( ) 1/2 1 1 (4.2) 1+(τ C /τ D ) 1+(ω 0 /z 0 ) 2 (τ C /τ D ) G T (τ C )= [ ( ( τc 1 T 1 exp τ T ))] (4.3) 66
85 4.3 Fluoreszenz-Resonanz-Energie-Transfer-Messungen an Einzelmolekülen (FRET) N τ C τ D T ω 0 z 0 mittlere Anzahl der Fluorophore im konfokalen Volumen Korrelationszeit Diffusionszeit durch das konfokale Volumen Anteil der Fluorophore im Triplettzustand laterale Ausdehnung des Detektionsvolumens axiale Ausdehnung des Detektionsvolumens Der Fit wurde durch Eingabe der Triplett-Lebensdauer τ T mit 3 μs [134] gestartet, die erhaltene Autokorrelationsfunktion und die Ausgleichskurve sind in Abbildung 4.3 dargestellt. normierte Autokorrelation 2,0 1,5 1,0 0,5 gemessene Autokorrelationsfunktion Fit der Modell-Funktion 0, Korrelationszeit / ms Abbildung 4.3: FCS-Kurve einer Alexa488-Probe. Gemessene Autokorrelationsfunktion (schwarz) und Ausgleichskurve der Modell-Funktion einer dreidimensionalen Diffusion (rot) von Alexa488 (Endkonzentration 2 nm) in Liposomenpuffer. Da der Diffusionskoeffizient von Alexa488 (435 μm 2 s 1, [135]) bekannt ist, kann aus Gleichung 4.4 der Radius des Fokus ω 0 und daraus die anderen Parameter z 0 (vgl. Abbildung 4.4), das konfokale Volumen und der Triplettanteil bestimmt werden. D = ω2 0 4τ D (4.4) 67
86 4 Fluoreszenzspektroskopische Methoden D ω 0 τ D Diffusionskoeffizient Radius des Fokus Diffusionszeit z 0 0 Abbildung 4.4: Konfokales Volumen. Elliptischer Fokus (gelb) mit den Radien ω 0 und z 0. In Tabelle 4.1 sind die für die gezeigte Ausgleichskurve erhaltenen Werte angegeben. Die Einzelmolekülmessungen wurden durchgeführt, wenn für das Verhältnis κ = ω 0 /z 0 ein Wert zwischen 5-8 erreicht wurde. Tabelle 4.1: Anpassungsparameter der FCS-Messung aus Abbildung 4.3. Diffusionszeit durch den Fokus (τ D ), Anzahl der Teilchen im Fokus (N), Verhältnis der Radien (κ = ω 0 /z 0 ), Triplettanteil (T) und Triplettlebensdauer (τ T ). τ D N κ T τ T 0,2 ms 0,63 5,8 7 % 0,004 ms Einzelmolekülmessungen an H + -ATPsynthasen Für die Einzelmolekülmessungen wurden 68 nm mit EGFP und ATTO565 doppelt markierte (vgl. Abschnitt 3.3) H + -ATPsynthasen in Liposomen rekonstituiert (vgl. Abschnitt 3.7). Von den Proteoliposomen wurden 0,3 μl (Endkonzentration markiertes Enzym: 0,23 nm) auf dem in der Probenhalterung eingespannten Deckgläschen (Roth, 24mm) mit 89,7 μl Liposomenpuffer gemischt. Auf dem Objektiv wurden 50 μl Immersionswasser aufgebracht, die Probenhalterung mit der Probe eingesetzt und in die Probe fokussiert. Es wurden pro Probe 500 Spuren von je 5 s aufgenommen. Sollten weitere Spuren aufgenommen werden, wurde die gemessene Probe verworfen und erneut frisch angesetzt. 68
87 4.3 Fluoreszenz-Resonanz-Energie-Transfer-Messungen an Einzelmolekülen (FRET) Einzelmolekülmessungen in Gegenwart von Nukleotiden Für Messungen in Gegenwart von Nukleotiden wurde zur Messung unter Hydrolysebedingungen 1 mm ATP zugegeben. Zur Messung unter nichtkatalytischen Bedingungen wurden 0,3 mm AMPPNP zum Messansatz hinzugefügt. Der Messansatz hatte jeweils ein Volumen von 90 μl. Pro Messung wurden 500 Spuren von je 5 s aufgenommen. Einzelmolekülmessungen an mit NaN 3 und ADP inhibierten H + -ATPsynthasen NaN 3 wirkt bei Anwesenheit von ADP als Inhibitor für den F 1 -Teil der H + -ATPsynthasen [136]. Vor den Messungen wurden 5 μl Proteoliposomen (Enzymkonzentration: 68 nm) mit 0,5 μl 100 μm NaN 3 -Lösung und 0,36 μl 1,36 mm ADP-Lösung versetzt und 30 min inkubiert. Anschließend wurde für die Messung 0,4 μl der inkubierten Proteoliposomen mit 89,6 l Liposomenpuffer gemischt und diese wie oben beschrieben durchgeführt. Auswertung der Einzelmolekülmessungen Die Messdateien wurden mit dem Programm Burst Analyzer V 4.39 (Nawid Zarrabi, Universität Stuttgart) eingelesen und ausgewertet. Von allen Bursts, die den in Kapitel aufgeführten Kriterien entsprachen, wurde der mittlere Proximity-Faktor bestimmt. Daraus wurde mit Hilfe des Programms OriginPro 9G die FRET-Effizienz und die Donor-Akzeptor-Abstände berechnet und in einem Histogramm zusammengefasst. Die mittleren Abstände wurden innerhalb der Histogramme durch Anpassung mittels einer Gaußfunktion mit OriginPro 9G ermittelt. 69
88 4 Fluoreszenzspektroskopische Methoden 70
89 Ergebnisse
90
91 5 Die H + -ATPsynthase aus Escherichia coli 5.1 Anzucht von E. coli Die Zellanzucht für E. coli erfolgte wie unter Kapitel beschrieben. Das Zellwachstum des Stammes DK8 (blaue Linie) war deutlich langsamer als das des RA1-Stammes (grüne Linie). Auch die Ausbeute nach Erreichen der gewünschten optischen Dichte von 1,4 lag mit ca. 68 g unter der des RA1-Stammes mit ca. 90 g. In Abbildung 5.1 sind die Latenz- und die exponentielle Phase bis zu einer optischen Dichte von 1,4 der Wachstumskurven im Vergleich dargestellt. 1,6 1,2 OD 578 0,8 0,4 0, Zeit/min Abbildung 5.1: Wachstumskurven der E. coli-zellstämme DK8 und RA1. Wachstumskurve des RA1-Stammes mit dem Plasmid pra100 (grün) im Vergleich zur Wachstumskurve des DK8-Stammes mit dem Plasmid pbwu13 (blau). 73
92 5 Die H + -ATPsynthase aus Escherichia coli 5.2 Isolation der H + -ATPsynthase aus E. coli Isolation durch Ionenaustausch-/Größenausschlusschromatographie Die Isolation von EF 0 F 1 aus dem Stamm pra100/ra1 erfolgte wie in Kapitel beschrieben. Das Elutionsprofil der Größenausschlusschromatografie S-400 im letzten Reinigungsschritt ist in Abbildung 5.2 dargestellt. relative Absorbanz bei 280 nm 1,0 0,8 0,6 0,4 0,2 0, Fraktionsnummer Abbildung 5.2: Elutionsprofil der Größenausschlusschromatografie S-400. Gemessene Absorbanz bei 278 nm der Präparation von EF 0 F 1 aus dem Stamm pra100/ra1, die Absorbanz der Fraktionen lag außerhalb des Detektionsbereichs. Die Fraktionen 20 bis 28 wurden mit einer SDS-Gelelektrophorese (vgl. Kapitel 3.5) auf deren Reinheit überprüft. Das erhaltene Gel ist in Abbildung 5.3 dargestellt. Die Proteinkonzentration in den einzelnen Fraktionen wurde mittels UV/VIS-Spektroskopie (vgl. Kapitel 3.4.1) genau bestimmt, dazu wurde jede Fraktion dreimal gemessen und der Mittelwert gebildet. Die Fraktionen wurden weiter auf ATP-Synthese- und ATP-Hydrolyseaktivität untersucht (vgl. Kapitel 3.8 und 3.9.1). Die erhaltenen Werte (Mittelwerte aus je drei Messungen) sind in Abbildung 5.4 links dargestellt. Die gleichen Bestimmungen wurden auch mit den Proteinfraktionen nach der Konzentrierung durch Ammoniumsulfatfällung durchgeführt und sind in Abbildung 5.4 rechts dargestellt. 74
93 5.2 Isolation der H + -ATPsynthase aus E. coli Abbildung 5.3: SDS-PAGE der Fraktionen der Größenausschlusschromatografie S-400. Gel (mit 13 %igem Trenngel) der Peakfraktionen nach der Präparation von EF 0 F 1 des Stammes pra100/ra1. Zusätzlich wurde ein LMW-Proteinstandard (14,4-97 kda) aufgetragen. Die Färbung erfolgte mit Coomassie Blau. turnover / s Hydrolyse Synthese Konzentration Konzentration / µm turnover / s Hydrolyse Synthese Konzentration Konzentration / µm Fraktionsnummer 0 21K 23K 25K 27K Fraktionsnummer 0 Abbildung 5.4: Vergleich der katalytischen Aktivität der Fraktionen Links: Proteinkonzentration (blau), ATP-Hydrolyseaktivität (rot) und ATP- Syntheseaktivität (grün) der Peakfraktionen nach der Größenausschlusschromatografie S-400 der Präparation von von EF 0 F 1 des Stammes pra1/ra1. Rechts: Proteinkonzentration, ATP-Hydrolyseaktivität und ATP-Syntheseaktivität der Fraktionen nach der Konzentrierung durch Ammoniumsulfatfällung. Anhand der Kurven ist erkennbar, dass die Fraktionen mit syntheseaktivem Enzym zu Beginn des Proteinpeaks eluiert werden. Hingegen zeigen die späteren Fraktionen eine höhere ATP- 75
94 5 Die H + -ATPsynthase aus Escherichia coli Hydrolyseaktivität. Dies bedeutet, dass bei der hier durchgeführte Reinigung Fraktionen mit eine hohe Konzentration an Protein erhalten wird, welches durch den Verlust von Untereinheiten syntheseinaktiv geworden ist aber eine hohe Hydrolyseaktivität besitzen. Vergleicht man die gefundenen Ergebnisse mit der SDS-Page, findet man in den Fraktionen alle Untereinheiten, während in den Fraktionen die Untereinheiten a,b,δ und c nur noch schwach oder gar nicht mehr vorhanden sind. Dort liegen vor allem die Untereinheiten des F 1 -Teils vor. Es zeigt sich auch, dass die Abtrennung von hoch syntheseaktivem Enzym gut gelungen ist, allerdings ist die Ausbeute mit 5,6 mg für Fraktion 22 im Verhältnis zur Gesamtausbeute von 58,3 mg für die Fraktionen in diesem Beispiel gering. Die Ammoniumsulfatfällung hat keinen Einfluss auf die ATP-Syntheseaktivität und kann deshalb gut zur Konzentrierung der Proteinproben angewendet werden Isolation durch Dichtegradientenzentrifugation Die Isolation von EF 0 F 1 aus dem Stamm pbwu13/dk8 erfolgte wie in Kapitel beschrieben. Die erhaltenen Dichtegradienten nach der Zentrifugation und die Verteilung der Fraktionen ist in Abbildung 5.5 dargestellt. Abbildung 5.5: Dichtegradient nach der Zentrifugation und Verteilung der Fraktionen Stufengradient mit 20, 25, 35, 40 und 45 % Saccharose (w/w). Die Skala zeigt die einzelnen Fraktion mit jeweils einem Volumen von 0,5 ml. Die rot markierten Fraktionen zeigten Syntheseaktivität. 76
95 5.2 Isolation der H + -ATPsynthase aus E. coli Ausgewählte Fraktionen des Dichtegradienten wurden mit einer SDS-Gelelektrophorese (vgl. Kapitel 3.5) auf Reinheit überprüft. Das erhaltene Gel ist in Abbildung 5.6 dargestellt C21 a b Abbildung 5.6: SDS-PAGE ausgewählter Fraktionen des Dichtegradienten. Es wurden die Fraktionen 7, 9, 11, 12, 13, 15, 17, 19 und 20 des Dichtegradienten aus Abbildung 5.5 auf ein Gel mit 13 %igem Trenngel aufgetragen. Zum Vergleich wurde die Fraktion C21 der Größenausschlusschromatographie aufgetragen, da sich die Spur am Rand des SDS-Gels befand, zeigten die Banden ein etwas anderes Laufverhalten, die sichtbaren Banden konnten aber den entsprechenden Untereinheiten zugeordnet werden. Die Färbung erfolgte mit Coomassie Blau. Die auf das Gel aufgetragenen Fraktionen wurden rekonstituiert (vgl. Kapitel 3.7) und die Synthese gemessen (siehe Kapitel 3.8). Die Ergebnisse sind in Tabelle 5.1 zusammengefasst. Die Fraktionen 7-13zeigten keine Syntheseaktivität. Beim Vergleich mit dem Coomassie Blau gefärbten Gel ist erkennbar, dass die Untereinheiten der H + -ATPsynthase nur in den Fraktionen vorliegen, dies entspricht auch dem Ergebnis der Synthesemessung. Im Allgemeinen ist die Probe aus der Dichtegradientenzentrifugation im Vergleich zu der Probe aus der Reinigung mittels Größenausschlusschromatographie (vgl. Abbildung 5.3) deutlich stärker mit weiteren Proteinbanden verunreinigt. Tabelle 5.1: Turnover der Synthese ausgewählter Fraktionen des Dichtegradienten. Synthesegeschwindigkeit der Fraktionen 15, 17, 19 und 20 des Dichtegradienten aus Abbildung 5.5. Jede Synthesegeschwindigkeit wurde dreimal gemessen und der Mittelwert mit der Standardabweichung gebildet. Fraktion turnover / s 1 4,9 ± 2,6 22,0 ± 0,3 19,7 ± 2,7 12,7 ± 2,9 77
96 5 Die H + -ATPsynthase aus Escherichia coli Eine genaue Analyse des Dichtegradienten nach der Zentrifugation wurde im Rahmen der Doktorarbeit von S. Fischer [124] durchgeführt. Die Daten sind in Abbildung 5.7 dargestellt. Die Fraktionsgröße des Dichtegradienten betrug 500 μl. Auch hier weisen die Fraktionen mit der höchsten ATP-Hydrolyseaktivität nicht gleichzeitig auch die höchste ATP-Syntheseakitvität auf Synthese Hydrolyse Konzentration 10 8 turnover / s Konzentration / µm Fraktionsnummer Abbildung 5.7: Vergleich der katalytischen Aktivität der Fraktionen Proteinkonzentration (blau), ATP-Hydrolyseaktivität (rot) und ATP- Syntheseaktivität (grün) der Dichtegradientenzentrifugation der Präparation von EF 0 F 1 des Stammes pbwu13/dk8. Die Daten wurden aus der Doktorarbeit von S. Fischer [124] entnommen. Die Größe der Fraktionen des Dichtegradienten betrug 500 μl Fazit Für Messungen der ATP-Synthese kann also für beide Isolationsmethoden nicht direkt von der ATP-Hydrolyseaktivität nach der One-step-Methode (vgl. Kapitel 3.9.1) auf die ATP- Syntheseaktivität geschlossen werden. In beiden Fällen zeigen frühe Fraktionen die höchste ATP- Syntheseaktivität im Vergleich zu den Fraktionen mit der höchsten ATP-Hydrolyseaktivität. Eine bessere Möglichkeit von der ATP-Hydrolyseaktivität auf die ATP-Syntheseaktivität zu schließen, stellt die ACMA-Methode, da dabei die mit dem Protonentransport gekoppelte ATP- Hydrolyse gemessen wird (vgl. Kapitel 3.9.2). 78
97 5.3 Fluoreszenzlöschung von ACMA in Dialyseliposomen 5.3 Fluoreszenzlöschung von ACMA in Dialyseliposomen Bei der protonentransportgekoppelten Hydrolyse von ATP werden Protonen über die Lipiddoppelschicht des Liposoms transportiert und es erhöht sich die Protonenkonzentration im Liposomeninneren. Die H + -Konzentration im Liposomeninneren kann durch die ph-abhängige Verteilung eines Amins zwischen dem Außen- und dem Innenraum bestimmt werden (Theorie siehe Anhang A). Als Amin eignet sich 9-Amino-6-Chloro-2-methoxyacridin (ACMA). Allerdings zeigt ACMA ein nichtideales Verhalten. Eine Restfluoreszenz beruht auf nichtprotoniertem ACMA, welches an die innere Liposomenoberfläche adsorbiert [137]. Deshalb kann der Absolutwert der Protonenkonzentration im inneren Liposomenraum nicht direkt aus dem Quotienten der Fluoreszenzlöschung und der Anfangsfluoreszenz (ΔF/F) berechnet werden. Neben der Adsorption des ACMAs an die innere Lipidschicht beeinflusst auch die Konzentration von ACMA das Fluoreszenzsignal. Durch die Erhöhung der ACMA-Konzentration kommt es zu Dimerisierungen und zu Änderungen der Fluoreszenzlöschung [137]. Um diese Einflüsse zu untersuchen, wurden Messungen bei verschiedenen ACMA-Konzentrationen durchgeführt. In einem Messansatz (1140 μl, ph = 4,2 vgl. Kapitel 3.9.2) wurden 20 μl Proteoliposomen mit verschiedenen Mengen ACMA (1,5-15 μl, 1 mm) versetzt und der Verlauf der Fluoreszenz gemessen. relative Fluoreszenzintensität NaOH Nigericin 13,2 µm 7,0 µm 5,3 µm 3,9 µm 2,6 µm 1,3 µm Zeit / s Abbildung 5.8: Fluoreszenz in Abhängigkeit der ACMA-Konzentration bezogen auf die jeweilige Anfangsfluoreszenz. Erzeugung eines ΔpH durch Zugabe von NaOH (100 μl, 40 mm zu 1160 μl Lösung) bei 6 verschiedenen ACMA-Konzentrationenen (1,3-13,2 μm), Abbau des ΔpH durch Zugabe von Nigericin (blaue Pfeile, 0,5 μl, 10 mm). 79
98 5 Die H + -ATPsynthase aus Escherichia coli Durch Zugabe von 100 μl NaOH (40 mm) wurde ein ΔpH = 3,6 erzeugt und durch Zugabe von 0,5 μl Nigericin (5 μm Endkonzentration) wieder abgebaut. Die Kurven wurden auf die maximale ACMA-Fluoreszenz direkt nach der Zugabe von ACMA normiert (siehe Abbildung 5.8). Bei ACMA-Konzentration < 3,9 μm wurde nach der Zugabe von Nigericin die ursprüngliche Fluoreszenz nicht wieder erreicht. Weitere Untersuchungen zeigten, dass dieser Effekt auch ohne die Anwesenheit von Liposomen zu beobachten ist. Dazu wurde ein Messansatz (1020 μl, ph = 4,2 vgl. Kapitel 3.9.2) mit ACMA versetzt (1,5 8 μl, 1 mm), dann NaOH dazugegeben (100 μl, 40 mm, ph End = 7,8) und die Fluoreszenz gemessen. Auch hier wurde bei kleinen ACMA-Konzentrationen nach Zugabe von NaOH eine Fluoreszenzabnahme beobachtet. Die Ursache dieses Effekts ist unklar. Daher wurde im Folgenden nicht auf die Anfangsfluoreszenz, sondern auf die Endfluoreszenz nach Zugabe des Nigericins normiert. In Abbildung 5.9 sind die Daten aus Abbildung 5.8 mit Normierung auf die Endfluoreszenz dargestellt. NaOH relative Fluoreszenzintensität Nigericin 13,2 µm 7,0 µm 5,3 µm 3,9 µm 2,6 µm 1,3 µm Zeit/s Abbildung 5.9: Fluoreszenz in Abhängigkeit der ACMA-Konzentration bezogen auf die Endfluoreszenz nach Zugabe von Nigericin. Erzeugung eines ΔpH durch Zugabe von NaOH (100 μl, 40 mm zu 1160 μl Lösung) bei 6 verschiedenen ACMA-Konzentrationen (1,3-13,2 μm), Abbau des ΔpH durch Zugabe von Nigericin (blaue Pfeile, 0,5 μl, 10 mm). Es ist erkennbar, dass der Maximalwert der Fluoreszenzlöschung bei einer ACMA-Konzentration von 3,9 μm am größten ist. Bei einer Endkonzentration von 13,2 μm ACMA wurde kein Signal mehr gemessen (siehe Abbildung 5.10 links). Es wurde die Steigung der Fluoreszenzabnahme nach Zugabe von NaOH bestimmt, diese blieb unabhängig von der zugegebenen Menge an NaOH annähernd konstant (siehe Abbildung 5.10 rechts). Dies bedeutet, dass die Äquilibrierung 80
99 5.3 Fluoreszenzlöschung von ACMA in Dialyseliposomen über die Lipiddoppelschicht in der Messzeit gewährleistet ist. Abschließend wurde auch die Geschwindigkeit der Zunahme der Fluoreszenz nach Zugabe von Nigericin bestimmt. Wie in Abbildung 5.10 rechts dargestellt, nimmt diese linear mit der ACMA-Konzentration zu. Sowohl eine Verstärkung des entkoppelnden Effekts als auch eine schnellere Einstellung des Gleichgewichts über die Lipiddoppelschicht könnten Ursache hierfür sein. relative Fluoreszenzlöschung ΔF ACMA-Konzentration / µm v(fluoreszenzabnahme) / s v(fluoreszenzzunahme) / s ACMA-Konzentration / µm Abbildung 5.10: Geschwindigkeit und Größe der Fluoreszenzabnahme bei verschiedenen ACMA-Konzentrationen. Oben: Fluoreszenzlöschung ΔF abhängig von der ACMA-Konzentration. Unten: Geschwindigkeit v(fluoreszenzabnahme) = ΔF der Fluoreszenzabnahme nach Zugabe von dt NaOH (orange) und Geschwindigkeit v(fluoreszenzzunahme) = ΔF der dt Fluoreszenzzunahme nach Zugabe von Nigericin (grün). Eine hohe ACMA-Konzentration wirkt sich also entkoppelnd auf die Lipiddoppelschicht aus, deshalb kann die Menge an ACMA nicht beliebig hoch gewählt werden. Um aber die Einstellung des Gleichgewichts über die Lipidschicht im Rahmen der Messzeit zu gewährleisten, wurde die unter diesen Bedingungen gefundene optimale Konzentration von 4 μm im Messansatz für die weiteren Messungen verwendet. Damit ergibt sich unter diesen Bedingungen ein optimales 0 81
100 5 Die H + -ATPsynthase aus Escherichia coli ACMA zu Lipid-Verhältnis von 1:140. Die Abhängigkeit des ACMA zu Lipid-Verhältnis erklärt auch das Verhalten, das bei früheren Messungen gefunden wurde, in denen die ACMA-Konzentration konstant bei 1,5 μm gehalten und die Proteoliposomenkonzentration im Ansatz variiert wurde (0,08-0,70 mg/ml). Für die Lipid-Konzentration von 0,08 mg/ml und 1,5 μm ACMA (0, mg/ml) ergibt sich somit ein ACMA zu Lipid-Verhältnis von 1:200. Damit liegt das ACMA zu Lipid- Verhältnis bereits über dem hier ermittelten optimalen Verhältnis und es konnte kein größeres Signal erhalten werden, obwohl man mit zunehmender Proteoliposomenmenge eine stärkere Fluoreszenzlöschung erwarten würde Quantitative Auswertung durch Kalibrierung der ACMA-Fluoreszenzlöschung Zur quantitativen Bestimmung der Protonenkonzentration im Liposomeninneren muss aufgrund der Adsorption von ACMA an die Liposomenmembran (hauptsächlich an der Innenseite wegen der erhöhten ACMA-Konzentration) und der Konzentrationsabhängigkeit der Signale von der ACMA-Konzentration eine Kalibrierung durchgeführt werden. Da sowohl die Lipidkonzentration als auch die Permeabilität der Dialyseliposomen nicht immer gleich ist, muss die Kalibrierung (siehe Kapitel 3.9.2) für jede Liposomencharge oder bei Änderung anderer Parameter im Messansatz erneut durchgeführt werden. Insbesondere bei Verwendung anderer Lipide ergeben sich aufgrund der Ladungen der Liposomen andere Kurven. In Abbildung 5.11 sind die Ergebnisse einer solchen Kalibrierung dargestellt, sowie die daraus resultierende ΔpH-abhängige Kurve unter den hier verwendeten Bedingungen. Die Ausgleichskurve der Kalibrierung wurde mit der Gleichung 3.9 berechnet. Die daraus resultierenden Parameter A, B und C sind in Tabelle 5.2 aufgeführt, sowie Werte die in anderen Arbeiten erhalten wurden [132, 138]. Tabelle 5.2: Fitparameter der Kalibrierung der Fluoreszenzlöschung. Vergleich der Parameter A, B und C erhalten aus dem Fit mit OriginPro 9G nach Gleichung 3.9 aus dieser Arbeit, sowie den Arbeiten von Casadio [138] und D Alessandro et al. [132]. Parameter Autor A B C Literatur diese Arbeit 28,5 ± 2 78 ± 4-0,079 ± 0,01 Casadio 6, ,022 [138] D Alessandro et al. 19, ,038 [132] 82
101 5.3 Fluoreszenzlöschung von ACMA in Dialyseliposomen relative Fluoreszenzintensität ΔF F Zeit / s F 0 relative Fluoreszenzintensität ,5 1,8 2,2 2,4 3,1 3,6 4, Zeit / s 4 ΔpH Fluoreszenzlöschung ΔF Abbildung 5.11: Kalibrierung der ACMA-Fluoreszenzlöschung. Oben: Fluoreszenzlöschung ΔF von ACMA nach Zugabe von NaOH durch Extrapolation auf die Zeit der Zugabe von NaOH (siehe Anhang A) Mitte: Fluoreszenzlöschung nach Erzeugung verschiedener ΔpH-Werte durch Zugabe von NaOH Unten: Fluoreszenzlöschung als Funktion von ΔpH und Ausgleichskurve nach Gleichung
102 5 Die H + -ATPsynthase aus Escherichia coli Qualitative Auswertung der ACMA-Fluoreszenzlöschung Häufiger als zur quantitativen Bestimmung von ΔpH wird die Methode der Fluoreszenzlöschung von ACMA bei qualitativen Untersuchungen eingesetzt. Im Folgenden wurden, wie in Anhang C beschrieben, Mitochondrien aus Rinderherz isoliert und daraus submitochondriale Partikel hergestellt. Aus diesen wurden Superkomplexe isoliert und in Liposomen rekonstituiert. Der ACMA-Test wurde verwendet, um die rekonstituierten Superkomplexe anhand der Zugabe verschiedener Substrate und Inhibitoren zu charakterisieren (siehe Abbildung 5.12). Zu 980 μl SEM-Puffer (enthielt 50 mm KCl) wurden 20 μl Proteoliposomen und 0,3 μm 15 mm ACMA (4,5 μm) gegeben und es wurde die Fluoreszenz F gemessen. Der Verlauf ist in Abbildung 5.12 gezeigt. relative Fluoreszenzintensität F F 0 1 ATP 2 P i 3 Oligomycin 4 Decyl-Ubichinon 5 NADH 6 Valinomycin 7 Nigericin Zeit/s Abbildung 5.12: Qualitative Messung der Fluoreszenzlöschung von ACMA am Beispiel der Enzyme der Atmungskette. Veränderung der Fluoreszenz durch die Aktivierung und Inhibierung verschiedener Enzyme der Atmungskette von in Liposomen rekonstituierten Superkomplexen. Start der Hydrolyse von ATP durch die ATPsynthase, 1 2 kompetitive Inhibierung der ATPsynthase durch Phosphat, 3 vollständige Hemmung der ATPsynthase, 4 Aktivierung der Cytochrom-c-Reduktase (Komplex III), 5 Aktivierung der NADH-Dehydrogenase (Komplex I), 6 Erhöhung der K + - Permeabilität der Proteoliposomen, 7 Entkoppelung der Proteoliposomen. Bei Punkt 1 wurden 10 μl 100 mm ATP (1 mm) zugegeben. Dies führte zu einer protonentransportgekoppelten ATP-Hydrolyse und es baute sich eine transmembrane ph-differenz auf, die durch die Fluoreszenzlöschung angezeigt wurde. Die Geschwindigkeit der ATP-Hydrolyse 84
103 5.3 Fluoreszenzlöschung von ACMA in Dialyseliposomen (und damit die Geschwindigkeit des Protonentransports) verringerte sich durch die Zugabe von 5 μl 1 M Natriumphosphat (5 mm, ph 7.9, Punkt 2), das ein kompetitiver Inhibitor der durch MF 0 F 1 katalysierten ATP-Hydrolyse ist. Die Zugabe von 1 μl 10 mg/ml Oligomycin (10 μg/ml, Punkt 3) führte zu einer Hemmung des Protonentransports durch die ATPsynthase und damit zu einer Verringerung des ΔpH. Durch die Zugabe von 1 μl 100 mm Decylubichinon (100 μm) bei Punkt 4 wurde der Elektronentransport der Atmungskette durch den Cytochrom-bc 1 - Komplex (Komplex III) gestartet und der Aufbau der ph-differenz wurde durch die erneute Fluoreszenzlöschung angezeigt. Die ph-differenz wurde durch 1,5 μl 100 mm NADH (150 μm, Punkt 5) noch erhöht, da durch den Umsatz von NADH durch die NADH-Dehydrogenase (Komplex I) weitere Elektronen in die Transportkette eingeleitet wurden. Die Zugabe von 0,5 μl 10 mm Valinomycin (5 μm, Punkt 6) erhöhte die K + -Permeabilität. Dies führte zu einer weiteren kleinen Erhöhung des ΔpH. Vollständig abgebaut wurde die ph-differenz in Punkt 7 durch die Zugabe von 0,5 μl 10 mm Nigericin (5 μm). Die erhaltene Endfluoreszenz F 0 war auch in diesem Versuch etwas geringer als die Ausgangsfluoreszenz F wie in Abbildung 5.8 gezeigt. Diese Untersuchung zeigt die Anwesenheit des Komplexes I, des Komplexes III und der H + -ATPsynthase in den mit Digitonin isolierten Superkomplexen. 85
104 5 Die H + -ATPsynthase aus Escherichia coli 5.4 Bestimmung des K M -Wertes von Phosphat Michaelis-Menten-Kinetik Einfache, enzymatisch katalysierte Reaktionen können mit Hilfe der Michaelis-Menten-Kinetik beschrieben werden. Das Substrat (S) bindet reversibel an das Enzym (E) und bildet mit der Geschwindigkeitskonstanten k 1 den Enzym-Substrat-Komplex (ES). Am Enzym gebunden, erfolgt die Umwandlung in das Produkt (EP). Mit der Geschwindigkeitskonstanten k 2 dissoziiert das Produkt (P) im letzten Schritt der Reaktion irreversibel vom Enzym. E + S k 1 ES EP k 2 E + P (5.1) k 1 Die Bildung und die folgende Dissoziation des Produkts kann zu einem Reaktionsschritt mit der Geschwindigkeitskonstanten k cat zusammengefasst werden; E + S k 1 ES kcat E + P (5.2) k 1 Bei Anwendung der Michaelis-Menten-Kinetik wird die Konzentration des Enzym-Substrat- Komplexes als konstant betrachtet (steady state, d[es] dt = 0). Daraus ergibt sich für die Reaktionsgeschwindigkeit folgender Zusammenhang: v = k cat [E] 0 [S] K M +[S] (5.3) [E] 0 =[E]+[ES] = Gesamtkonzentration Enzym, K M = k 1+k cat k 1, k cat [E] 0 = v max v v max = [S] K M +[S] (5.4) Die Michaelis-Menten-Konstante K M, gibt die Substratkonzentration bei halbmaximaler Reaktionsgeschwindigkeit ( vmax ) an. 2 86
105 5.4 Bestimmung des K M -Wertes von Phosphat Bestimmung der Michaelis-Menten-Konstante von Phosphat Da bei der Katalysereaktion der H + -ATPsynthase verschiedene Substrate und mehrere Bindungsstellen (drei katalytische und drei nichtkatalytische) beteiligt sind, kann die Michaelis- Menten-Kinetik nicht direkt angewendet werden. Werden alle Substratkonzentrationen, außer Phosphat, im Bereich der Sättigung gewählt, kann dessen Verhalten aber näherungsweise durch die Michaelis-Menten-Kinetik beschrieben werden. Im ph-bereich zwischen 7 und 9 kommt Phosphat in verschiedenen Protonierungsstufen vor. Es soll hier untersucht werden, ob diese gleichermaßen ans Enzym binden können oder ob eine einzelne Phosphatspezies als Substrat identifiziert werden kann. Die Dissoziation der Phosphorsäure H 3 PO 4 erfolgt insgesamt in drei Schritten, dabei ist der Anteil der jeweiligen Spezies abhängig vom ph-wert. Die Literaturwerte der Dissoziationskonstanten können Tabelle 5.3 (aus Handbook of Chemistry and Physics) entnommen werden. Sie sind für eine Ionenstärke von I = 0angegeben. Für den in den Experimenten untersuchten ph- Bereich von 7-9 wurde die relevante Dissoziationskonstante K 2 auf die Ionenstärke der Lösung bei der ATP-Synthese (Zusammensetzung siehe Kapitel 3.8) umgerechnet. Die Ionenstärke wurde mit Hilfe des Programms von Krab & van Wezel [139] nach Gleichung 5.5 berechnet. Die Änderungen der Dissoziationskonstanten K 1 und K 3 wurden nicht berücksichtigt. I = 1 2 i z 2 i c i c 0 (5.5) c i = Konzentration des Ions, z i = Ladung des Ions, c 0 = Standardkonzentration Tabelle 5.3: Dissoziationskonstanten der Phosphorsäure. Die Werte wurden aus dem Handbook of Chemistry and Physics entnommen (25 C, I = 0). Die Dissoziationskonstante der H 2 PO 4 -Spezies für die experimentell verwendete Ionenstärke von I = 0,16 wurde nach einer Vorschrift von Krab & van Wezel [139] berechnet. Reaktion Dissoziationskonstante Dissoziationskonstante I = 0 I = 0,16 H 3 PO 4 H 2 PO 4 +H + K 1 =7, H 2 PO 4 HPO 2 4 +H + K 2 =6, K 2 (korr) =1, HPO 2 4 PO 3 4 +H + K 3 =4,
106 5 Die H + -ATPsynthase aus Escherichia coli Da die Messungen bei verschiedenen Phosphatkonzentrationen (0-15 mm) und verschiedenen ph-werten (7,3-8,8) durchgeführt wurden, variierte die Ionenstärke zwischen I = 0,12-0,20. Die folgenden Berechnungen wurden mit einer durchschnittlichen Ionenstärke von 0,16 durchgeführt. Der daraus resultierende Fehler ist im Verhältnis zum Fehler der Messwerte vernachlässigbar. Mit den empirisch ermittelten Funktionskonstanten a = -1,52 und b = 1,96 [140], lässt sich der pk 2 und daraus die Dissoziationskonstante K 2 (korr) nach Gleichung 5.6 und 5.7 berechnen. pk 2 (korr) =pk a I + bi (5.6) K 2 (korr) =10 pk 2(korr) (5.7) Mit Hilfe der so korrigierten Dissoziationskonstante können die Anteile α der einzelnen Phosphatspezies abhängig vom ph-wert nach den vier Gleichungen ermittelt werden. Die Ergebnisse sind in Tabelle 5.4 aufgeführt. α[h 3 PO 4 ]= [H 3PO 4 ] [P i (ges)] = [H + ] 3 [H + ] 3 + K 1 [H + ] 2 + K 1 K 2 (korr)[h + ]+K 1 K 2 (korr)k 3 (5.8) α[h 2 PO 4 ]= [H 2PO 4 ] [P i (ges)] = K 1 [H + ] 2 [H + ] 3 + K 1 [H + ] 2 + K 1 K 2 (korr)[h + ]+K 1 K 2 (korr)k 3 (5.9) α[hpo4 2 ]= [HPO2 4 ] [P i (ges)] = K 1 K 2 (korr)[h + ] (5.10) [H + ] 3 + K 1 [H + ] 2 + K 1 K 2 (korr)[h + ]+K 1 K 2 (korr)k 3 4 ] α[po4 3 ]= [PO3 [P i (ges)] = K 1 K 2 (korr)k 3 (5.11) [H + ] 3 + K 1 [H + ] 2 + K 1 K 2 (korr)[h + ]+K 1 K 2 (korr)k 3 Um die bei der ATP-Synthese an das Enzym gebundene Phosphatspezies zu identifizieren, wurde die Syntheserate (turnover) wie in Kapitel 3.8 beschrieben, als Funktion der Gesamtphos- 88
107 5.4 Bestimmung des K M -Wertes von Phosphat Tabelle 5.4: Anteile der einzelnen Phosphatspezies, für die in dieser Arbeit verwendeten ph-werte ph a H + α[h 3 PO 4 ] α[h 2 PO 4 ] α[hpo 2 4 ] α[po 3 4 ] 7, 3 5, , , , , ,6 2, , , , , ,9 1, , , , , ,2 6, , , , , ,5 3, , , , , ,8 1, , , , , phatkonzentration (0-15 mm) bei sechs verschiedenen ph a gemessen. Der ph i wurde konstant bei ph 4,9 gehalten, jede Messung wurde dreimal wiederholt und die Daten als Funktion der Gesamtphosphatkonzentration aufgetragen (siehe Abbildung 5.13). Mit Hilfe einer nichtlinearen Kurvenanpassung und der vorgegebenen Michaelis-Menten-Kinetik (Gleichung 5.2) wurde mit dem Programm OriginPro 9G die maximale Synthesegeschwindigkeit v max und die Michaelis- Menten-Konstante K M mit den dazugehörigen Fehlern berechnet. Die Ergebnisse sind in Tabelle 5.5 zusammengefasst. Tabelle 5.5: Maximale Synthesegeschwindigkeit v max und Michaelis-Menten- Konstante K M bei verschiedenen ph a. ph a v max /s 1 K M /mm 7,3 8 ±1 0,09 ±0, 04 7,6 37 ±2 0,14 ±0, 04 7,9 8 ±1 0,63 ±0, 25 8,2 36 ±3 0,40 ±0, 10 8,5 46 ±7 1,46 ±0, 50 8,8 103 ±11 1,76 ±0, 05 Die maximale Synthesegeschwindigkeit nahm mit steigendem ph a zu, da dadurch ein größerer ΔpH generiert wurde. Wenn alle Phosphatspezies gleichermaßen an das Enzym binden, sollte der K M -Wert konstant bleiben. Dies ist offenbar nicht der Fall. Deshalb wurde untersucht welche K M -Werte sich ergeben, wenn nur eine Phosphatspezies betrachtet wird. 89
108 5 Die H + -ATPsynthase aus Escherichia coli Abbildung 5.13: ATP-Syntheserate (turnover) als Funktion der Phosphatkonzentration. Die Messungen wurden für 6 verschiedene ph a -Werte bei konstantem ph i -Wert (4,9) durchgeführt. Jede Geschwindigkeit wurde dreimal gemessen und der Mittelwert der Messungen aufgetragen. Die Balken zeigen die Standardabweichung der jeweiligen Messung. Geschwindigkeiten, die in Klammern gesetzt sind, wurden bei der Ausgleichsrechnung nicht berücksichtigt. 90
109 5.4 Bestimmung des K M -Wertes von Phosphat Bestimmung der Michaelis-Menten-Konstante von Dihydrogenphosphat (H 2 PO 4 ) Zum Vergleich der Messreihen bei verschiedenen ph a -Werten wurden die relativen Synthesegeschwindigkeiten verwendet v/v max (vgl. Tabelle 5.5, v entspricht turnover) und gegen die Gesamtphosphatkonzentration aufgetragen (Abbildung 5.14 links). Die K M -Werte zeigen eine Abhängigkeit vom ph a -Wert (siehe Abbildung 5.15 Mitte). Aus der Gesamtphosphatkonzentration wurde die H 2 PO 4 -Konzentration durch Multiplikation mit α[h 2 PO 4 ] (siehe Tabelle 5.4) für den jeweiligen ph a -Wert ermittelt. Abbildung 5.14 rechts zeigt die relativen Synthesegeschwindigkeiten in Abhängigkeit der H 2 PO 4 -Konzentration. Mit diesen Daten wurden ebenfalls nichtlineare Anpassungen durchgeführt und der K M -Wert bestimmt. Diese entsprechen den K M (H 2 PO 4 )-Werten der monoanionischen Phosphatspezies. Wie in Abbildung 5.14 rechts und Abbildung 5.15 unten gezeigt, bleibt der K M (H 2 PO 4 )-Wert annähernd konstant (Werte siehe Tabelle 5.6). Abbildung 5.14: Bestimmung der K M -Werte. Links: Relative Synthesegeschwindigkeit v/v max aufgetragen gegen die Gesamtphosphatkonzentration. Rechts: Relative Synthesegeschwindigkeit aufgetragen gegen die H 2 PO 4 -Konzentration. 91
110 5 Die H + -ATPsynthase aus Escherichia coli Abbildung 5.15: ph a -Abhängigkeit von v max,k M und K M [H 2 PO 4 ]. Oben: Maximale Synthesegeschwindigkeit (Daten aus Abbildung 5.13, v max = max. turnover). Mitte: K M -Werte (Daten aus Abbildung 5.13) und Anteil der H 2 PO 4 - Spezies an der Gesamtphosphatkonzentration (grün, Daten aus Tabelle 5.4). Unten: K M (H 2 PO 4 )-Werte berechnet aus der Abhängigkeit der Synthesegeschwindigkeit von der H 2 PO 4 -Konzentration (Abbildung 5.14 rechts). 92
111 5.4 Bestimmung des K M -Wertes von Phosphat Diskussion Der turnover der ATP-Synthese wurde in Abhängigkeit von der Phosphatkonzentration bei verschiedenen ph a -Werten gemessen. Die erhaltenen Kurven wurden mit einer Michaelis- Menten-Kinetik ausgewertet und die K M -Werte bestimmt. Diese nahmen mit steigendem ph a bei konstantem ph i -Wert (ph 4,9) zu. Aufgrund dieser ph-abhängigkeit wurde vermutet, dass nicht alle Phosphatspezies gleichermaßen an das Enzym binden. In dem untersuchten ph- Bereich (ph 7,3-8,8) liegen die mono- und die dianionische Phosphatspezies anteilsmäßig am stärksten vor. Vergleicht man die Änderung der Anteile in diesem ph-bereich, zeigt es sich, dass die Abnahme von H 2 PO 4 mit der Zunahme des K M -Wertes korreliert. Daraufhin wurde unter Berücksichtigung der Ionenstärke der Anteil der monoanionischen Phosphatspezies ermittelt und die K M -Werte für diese Spezies bestimmt. Der K M (H 2 PO 4 )-Wert hängt nicht vom ph a -Wert ab und man erhält einen Mittelwert von 30 ± 5 μm wie in Abbildung 5.15 unten gezeigt. In den Arbeiten von Fischer [124] und Rudolf [141] wurde ebenfalls die ph- Abhängigkeit des K M -Wertes von Phosphat an der H + -ATPsynthase aus E. coli untersucht. Auch bei diesen Untersuchungen ergab sich eine Abhängigkeit der K M -Werte vom ph a -Wert, wenn die Gesamtphosphatkonzentration verwendet wurde und eine Unabhängigkeit der K M - Werte vom ph a wenn die Abhängigkeit von H 2 PO 4 betrachtet wurde. Allerdings wurden in diesen Arbeiten die Dissoziationskonstanten der Phosphorsäure bei der Ionenstärke I = 0 verwendet, obwohl auch in diesen Arbeiten die Ionenstärke I = 0,16 war. Um die Daten miteinander vergleichen zu können, wurden die Originaldaten von Fischer und Rudolf mit Hilfe der nichtlinearen Kurvenanpassung und der vorgegebenen Michaelis-Menten-Kinetik erneut ausgewertet und die maximalen Synthesegeschwindigkeiten v max, die K M -Werte und die K M (H 2 PO 4 )-Werte bezogen auf I = 0 und auf I = 0,16 mit den jeweiligen Fehlern bestimmt. Diese Ergebnisse sind in Tabelle 5.6 zusammengefasst und zusammen mit den Ergebnissen dieser Arbeit graphisch in Abbildung 5.16 dargestellt. Der Mittelwert K M (H 2 PO 4 ) aus den Daten von Fischer liegt unter den hier erhaltenen Resultaten. Der Mittelwert der Daten von Rudolf entspricht den hier erhaltenen Werten. In der Arbeit von Förster [68] wurde die Phosphatabhängigkeit der H + -ATPsynthase aus Saccharomyces cerevisiae untersucht. Auch hier wurde H 2 PO 4 als die Phosphatspezies identifiziert, die an das Enzym bindet. Die korrigierte Konstante betrug K M (H 2 PO 4 ) = 120 ± 20 μm. Die Messungen wurden abweichend zu denen von E. coli bei einer ADP-Konzentration von 400 μm, einer MgCl 2 -Konzentration von 4 mm durchgeführt und auf eine Ionenkonzentration von 0,14 bezogen. Alle K M -Werte sind in Tabelle 5.6 zusammengefasst. 93
112 5 Die H + -ATPsynthase aus Escherichia coli Tabelle 5.6: K M -Werte und K M (H 2 PO 4 )-Werte bei der Ionenstärke I = 0 und I = 0,16 für die H + -ATPsynthase aus E. coli (EF 0 F 1 ) und S. cerevisiae (YF 0 F 1 ). F 0 F 1 -ATPase Autoren ph a K M -Wert K M (H 2 PO 4 ) K M (H 2 PO 4 ) /mm /μm / μm I=0 I=0,16 EF 0 F 1 Fischer, ,6 0,06 ± 0,01 18 ± 2 11 ± 1 8,8 0,72 ± 0,05 18 ± 1 9 ± 1 Mittelwert 10 ± 1 Rudolf, ,6 0,21 ± 0,05 60 ± 9 35 ± 6 7,9 0,75 ± 0,3 126 ± ± 12 8,3 0,66 ± 0,08 49 ± 3 26 ± 2 8,8 1,01 ± 0,16 25 ± 2 13 ± 1 Mittelwert 36 ± 12 diese Arbeit 7,3 0,09 ± 0,04 25 ± 8 7,6 0,14 ± 0,05 23 ± 5 7,9 0,63 ± 0,3 53 ± 15 8,2 0,40 ± 0,10 20 ± 3 8,5 1,46 ± 0,5 37 ± 5 8,8 1,76 ± 0,05 22 ± 3 Mittelwert 30 ± 5 YF 0 F 1 Förster, 2011 Mittelwert 120 ± 20 94
113 5.4 Bestimmung des K M -Wertes von Phosphat Abbildung 5.16: ph a -Abhängigkeit von v max, K M und K M [H 2 PO 4 ]. Vergleich der Daten aus Abbildung 5.15 mit den Messergebnissen von Fischer [124] (rot) und Rudolf [141] (blau). Oben: Maximale Synthesegeschwindigkeit. Mitte: K M -Werte und Anteil der H 2 PO 4 -Spezies an der Gesamtphosphatkonzentration. Unten: K M (H 2 PO 4 )-Werte berechnet aus der Abhängigkeit der Synthesegeschwindigkeit von der H 2 PO4 -Konzentration. 95
114 5 Die H + -ATPsynthase aus Escherichia coli Aus den Daten der Abbildung 5.16 kann anhand der Michaelis-Menten-Kinetik auch die Geschwindigkeitskonstante der H 2 PO 4 -Bindung (k 1 ) ermittelt werden. Mit der Annahme, dass [S] «K M und k cat»k 1 ergibt sich aus Gleichung 5.4: v = v max[s] K M +[S] v max K M [S] k 1 [S] (5.12) In Abbildung 5.17 ist vmax K M =k 1 in Abhängigkeit von ph a aufgetragen. Man erkennt eine Zunahme, die bei ph a = 8,8 einen Wert von k 1 = M 1 s 1 erreicht. Ist k 1 bekannt, 6,0x10 6 )/M -1 s -1 v max /K M (H 2 PO 4-4,0x10 6 2,0x10 6 ( ) 0,0 7,5 8,0 8,5 9,0 ph a Abbildung 5.17: ph a -Abhängigkeit von v max /K M (H 2 PO 4 )=k 1. Werte in Klammer wurden beim exponentiellen Fit nicht berücksichtigt. kann man die Größen der Geschwindigkeitskonstanten der H 2 PO 4 -Dissoziation k 1 und der Dissoziationskonstanten K D abschätzen. Mit K D = k 1 k 1 erhält man K M = k cat + k 1 k 1 = k cat k 1 + k 1 k 1 = k cat k 1 + K D (5.13) Die hier erhaltenen Werte für k 1,k 1 und K D streuen jedoch so stark, dass keine verläßlichen Aussagen getroffen werden können. Aus den Daten dieser Arbeit wurden auch negative Werte erhalten, sodass auf eine Auswertung verzichtet wurde. In früheren Arbeiten wurden K M -Werte von Phosphat zur Charakterisierung der H + -ATPsynthase (F 0 F 1 ) aus Mitochondrien, Chloroplasten und Bakterien (hauptsächlich E. coli) gemessen und Werte zwischen 0,2-1,6 mm erhalten [ ]. Überraschenderweise konnte jedoch 96
115 5.4 Bestimmung des K M -Wertes von Phosphat bei Untersuchungen der ATP-Hydrolyse keine Produktinhibierung durch Phosphat in diesem Konzentrationsbereich festgestellt werden [146, 147]. K D -Werte von Phosphat wurden unter uni-site Bedingungen an F 1 -Teilen bestimmt, die Werte lagen zwischen 0,1 mm - 1 M [ ]. In der Arbeit von Al-Shawi und Senior [150] wurde vermutet, dass unter diesen Bedingungen H 2 PO 4 an EF 1 bindet. Diese Werte müssten deshalb ebenfalls auf den Anteil von Dihydrogenphosphat korrigiert werden, alle bleiben nach der Korrektur aber trotzdem über den hier erhaltenen Werten, die im μm-bereich lagen. Neuere Einzelmoleküluntersuchungen an immobilisierten TF 1 -Teilen haben gezeigt, dass die K D -Werte für Phosphat von der Position der γ-untereinheit bei der Rotation abhängen [78]. Hier wurde in der 80 Position ein K D von 4,9 mm erhalten und für die 120 Position ein K D zwischen mm abgeschätzt. Dies zeigt, dass die Rotation der γ-untereinheit offenbar zu starken Änderungen der Phosphatbindung führt. Unabhängig von der mechanistischen Interpretation von K D - und K M -Werten zeigen die Ergebnisse dieser Arbeit, dass die Phosphatspezies H 2 PO 4 bei der ATP-Synthese an EF 0 F 1 bindet. Die Untersuchung der ph a -Abhängigkeit der K M -Werte von Phosphat an MF 0 F 1 ergab, dass auch an diesem Enzym vermutlich die Spezies H 2 PO 4 bindet [151]. Zieht man die Ergebnisse von MF 0 F 1 hinzu so ist es wahrscheinlich, dass H 2 PO 4 bei allen H + -ATPsynthasen das eigentliche Substrat für die ATP-Synthese darstellt. 97
116 5 Die H + -ATPsynthase aus Escherichia coli 98
117 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae 6.1 Der S. cerevisiae-stamm YEBTA-c-γ1-b-K174C Bei dem Zellstamm YEBTA-c-γ1-b-K174C [105, 122, 152] handelt es sich um eine EGFP- Fusionprotein-Cysteinmutante von YF 0 F 1. Der in dieser Arbeit verwendete Stamm enthielt das in Abbildung 1.7 rot umrandete Konstrukt. Der Aufbau dieses Zip-Helix-FP-Konstruktes (Molmasse ca. 35 kda, siehe Abbildung 6.1) wird durch die Bezeichnung des Stammes wiedergegeben. TEV-Proteaseschnittstelle TEV- Link Strep-tag II b a c Enhanced grün fluoreszierendes Protein Acoil C-Terminus Bcoil N-Terminus C-Terminus der -Untereinheit Abbildung 6.1: Schematische Darstellung des Konstruktes im Stamm YEBTA-cγ1-b-K174C. Die Reihenfolge im Aufbau ist wie folgt: C-Terminus der γ- Untereinheit des YF 0 F 1 - Bcoil - TEV-Proteaseschnittstelle - Strep-tag II - EGFP - Acoil, das EGFP ist an der Restriktionsschnittstelle c nach der TEV-Sequenz eingebaut (Molmasse ca. 35 kda). 99
118 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae An dem C-Terminus der γ-untereinheit (γ1) des YF 0 F 1 (Y) ist der N-Terminus der Bcoil (B) kovalent gebunden, daran schließt sich zunächst eine TEV-Proteaseschnittstelle und ein Streptag II an. Dieses Modul wird als TEV-Link (T) bezeichnet. Anschließend folgt das enhanced grün fluoreszierende Protein (EGFP, E) und der Acoil (A). Die Bezeichnung c gibt die Position der Insertion des fluoreszierenden Proteins im Bereich des TEV-Links an. Die Auswahl dieses Konsturkts erfolgte in früheren Arbeiten anhand seiner Fluoreszenzeigenschaften, seiner Kultivierbarkeit auf nichtfermentierbarem Substrat und seiner geringen Dissoziations- und Oligomerisierungsneigung [105, 122, 152, 153]. Die Größenabschätzungen wurden aus der Arbeit von J. Petersen [105] entnommen. Aufgrund folgender Kriterien eignet sich das Konstrukt für die Einzelmolekül-FRET-Spektroskopie: Durch die Verwendung zweier unterschiedlicher Helices (Acoil, Bcoil) und deren antiparalleler Anordnung wird der Linker mechanisch stabilisiert. Durch die Insertion des EGFPs an der Positon c erhält man eine abgewinkelte Struktur und FRET-Messungen zwischen dem EGFP und einem Akzeptor an der b-untereinheit sollten möglich sein. Jede der Helices besteht aus 30 Aminosäuren (ca. 3,3 kda), die TEV-Proteaseschnittstelle besteht aus 7 und der Strep-tag II aus 8 Aminosäuren (TEV- Link ca. 1,7 kda). Der Linkerbereich hat eine Größe von ca. 10 Å. Mit einer Größe von ca. 40 Å für das EGFP [89] ergibt sich für das gesamte Konstrukt eine Größe von ca. 50 Å. Um die Möglichkeit zu haben, selektiv eine kovalente Bindung zwischen der Maleimidgruppe eines organischen Fluorophors und YF 0 F 1 für die geplanten FRET-Messungen zu knüpfen, wurde ein Cystein eingeführt. Die Eignung einer entsprechenden Position im Enzym hängt dabei von mehreren Kriterien ab. Um die Selektivität zu erhöhen sollte die Mutation an einer exponierten Stelle im Enzym durchgeführt werden. Als weiteres Kriterium musste beachtet werden, dass die Mutation im Bereich des Stators durchgeführt wurde, da das EGFP sich am Rotor befindet (vgl. Kapitel 1.1), nur so können Rotationsbewegungen in Einzelmolekülexperimenten detektiert werden. Zusätzlich musste sich für die FRET-Messungen die Position im Bereich des Försterradius von Å befinden. Es wurde in der Untereinheit b an der Position 174 das Lysin durch ein Cystein ersetzt [122]. 6.2 Anzucht von S. cerevisiae Die Anzucht der S. cerevisiae Zellen erfolgte wie unter Kapitel beschrieben. Der Wachstumstest auf YMM-Minimalmedium ermöglichte die Kontrolle, dass ausschließlich der Stamm YEBTA-c-γ1-b-K174C angezogen wurde. Durch die Auxotrophie des Stammes YEBTA-c-γl für Leucin und Uracil, konnnte durch Abwesenheit von Histidin der Stamm selektiert werden. Zudem wurde mit der Mutation b-k174c gleichzeitig eine Antibiotikaresistenz für Geneti- 100
119 6.2 Anzucht von S. cerevisiae cin (G418) eingebracht. Durch die Ausplattierung auf YEPE-Medium (enthält Ethanol als nichtfermentierbares Substrat) wurde sichergestellt, dass die H + -ATPsynthasen intakt waren, da diese Hefezellen ihre Energie ausschließlich über die oxidative Phosphorylierung gewinnen können. In Abbildung 6.2 sind die Latenz- und die exponentielle Phase der Wachstumskurven des Stammes YEBTA-c-γ1-b-K174C (blaue Linie) und des Stammes YRD15 (Wildtyp-YF 0 F 1, grüne Linie) im Vergleich dargestellt. Das Zellwachstum des Stammes YEBTA-c-γ1-b-K174C war langsamer als das des YRD15-Stammes. Durch logarithmische Auftragung erhielt man für den Wildtyp eine Verdopplungszeit von 5 Stunden und für das EGFP-Fusionsprotein von 8 Stunden. Dies muss nicht nur an den H + -ATPsynthasen liegen, es können durch die Einführung des Fusionskonstrukts auch Störungen bei der Bildung der Cristae, bei der Teilung der Mitochondrien, oder bei der Weitergabe der mitochondrialen DNA an die Tochterzellen auftreten [62, 154, 155]. Nach dem Erreichen der gewünschten optischen Dichte von ca. 5,5 erhielt man für den YEBTA-c-γ1-b-K174C-Stamm 397 g und für den YRD15-Stamm 373 g Zellsuspension OD Zeit / h Abbildung 6.2: Wachstumskurven der S. cerevisiae-zellstämme YEBTA-c-γl-b- K174C und YRD15. Wachstumskurve des YRD15-Stammes (grün) im Vergleich zur Wachstumskurve des YEBTA-c-γl-b-K174C-Stammes (blau). Von den Hefezellen des Stammes YEBTA-c-γ1-b-K174C wurden mit der CCD-Kamera des konfokalen Mikroskops (vgl. Kapitel 1.3.1) Fotos gemacht. Anschließend wurde sofort die Beleuchtung ausgeschaltet und mittels TIRF-Mikroskopie, wie in Kapitel 4.2 beschrieben, die Fluoreszenz des in den Zellen exprimierten EGFPs fotografiert. Der identische Ausschnitt aus 101
120 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae zwei so aufeinander folgenden Aufnahmen ist in Abbildung 6.3 gezeigt. Im rechten Bild sind die beiden Aufnahmen aufeinander gelegt. Abbildung 6.3: Ausschnitt einer lichtmikroskopischen Aufnahme und einer Aufnahme der Fluoreszenz des Stammes YEBTA-c-γ1-b-K174C. Aufnahme mit einer CCD-Kamera, Links: lichtmikroskopische Aufnahme, Mitte: Aufnahme der Fluoreszenz durch TIRF-Mikroskopie, Rechts: lichtmikroskopische Aufnahme und Aufnahme der Fluoreszenz aufeinander gelegt. In dem Foto der TIRF-Mikroskopie (Foto Mitte) ist die Fluoreszenz in den Mitochondrien, welche sich im Cytosol befinden, gut zu erkennen. Gleichzeitig zeigen sich dunklere Abschnitte in den Zellen, die sich beim Vergleich mit der lichtmikroskopischen Aufnahme als die Bereiche von Zellkern und Vakuole identifizieren lassen. Die leichte Verschiebung zwischen Hefe-Zellen und Fluoreszenz im rechten Bild ist durch die Diffusion der Zellen in der Lösung in der Zeit zwischen den beiden Aufnahmen zu erklären. 6.3 Isolation der H + -ATPsynthase aus S. cerevisiae Die Isolation der H + -ATPsynthasen der Stämme YRD15 und YEBTA-γ1-b-K174C wurde wie in Kapitel beschrieben, durchgeführt. Der Aufschluss der Zellen erfolgte durch Schütteln mit Glasperlen, daraus wurden durch mehrere Zentrifugationsschritte die Mitochondrien gewonnen. Nach der Solubilisierung der Membranproteine, wurden diese durch eine Anionenaustauschund eine Größenausschlusschromatographie getrennt und YF 0 F 1 isoliert. Im Folgenden werden die Elutionsprofile von der Isolation des Stammes YEBTA-c-γ1-b-K174C gezeigt. Bei der Anionenaustauschchromatographie wurde die Absorbanz bei 280 nm und zusätzlich die Absorbanz bei 480 nm gemessen, um die Anwesenheit von EGFP in den eluierten Fraktionen zu detektieren. Abbildung 6.4 zeigt das Elutionsprofil. Nach der Erhöhung der Salzkonzentration von 100 mm NaCl auf 192 mm NaCl (23 % Puffer B, schwarze Linie), wurde ein Anstieg sowohl in der Absorbanz bei 280 nm (rote Linie) als auch in der Absorbanz bei 102
121 6.3 Isolation der H + -ATPsynthase aus S. cerevisiae 480 nm (grüne Linie) detektiert. Nach der Erhöhung der Salzkonzentration auf 220 mm NaCl (30 % Puffer B, zweiter Peak) wurde erneut eine Absorbanz mit jedoch viel geringerer Intensität bei 280 und 480 nm gemessen. Durch die Erhöhung der Salzkonzentration auf 500 mm NaCl (100 % Puffer B) wurde die Chromatographiesäule gereinigt. Es wurden Fraktion von 2 ml gesammelt. 1,0 100 relative Absorbanz 0,5 50 Anteil Puffer B / % 0,0 A5 A10 B Elutionsvolumen / ml Abbildung 6.4: Elutionsprofil der Anionenaustauschchromatographie der Isolation von YF 0 F 1. Exemplarisch ist das Ergebnis für den YEBTA-c-γ1-b-K174C- Stamm gezeigt. Die Kurven zeigen die relative Absorbanz bei 280 nm (rot) und bei 480 nm (grün), sowie den prozentualen Anteil an Puffer B (schwarz). Die Fraktionen wurden sofort unter Lichtausschluss auf Eis gestellt und die Hydrolyseaktivität (vgl. Kapitel 3.9.1) gemessen. Dazu wurden je 5 μl des Eluats in den Hydrolyse-Kit gegeben und die Abnahme der Absorbanz bei 340 nm gemessen. Anschließend wurde zu dem Messansatz Lauryldimethylaminoxid (LDAO) gegeben und kontrolliert, ob eine Aktivierung zu beobachten ist. Diese beruht auf verschiedenen Interaktionen zwischen mitochondrialen H + -ATPsynthasen und LDAO [156]. Sie ist jedoch nicht bei isoliertem F 1 zu beobachten, sodass aus der Aktivierung darauf geschlossen werden kann, ob die Hydrolyseaktivität auf F 0 F 1 beruht. Hydrolyseaktives und aktivierbares Enzym wurde in den Fraktionen A6 - A10 gefunden, die Werte sind in Tabelle 6.1 aufgeführt, zum Vergleich wurde zusätzlich der Wert für die Peakfraktion aus einer Präperation des Wildtyps YRD15 angegeben. Die aktiven Fraktionen wurden vereinigt, das Eluat auf ca. 1 ml konzentriert und auf die Größenausschlusschromatographie aufgetragen. Die Chromatographie wurde im Dunkeln durchgeführt. Das Elutionsprofil der S-300 ist in 103
122 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Tabelle 6.1: Hydrolyse der Fraktionen nach der Anionenaustauschchromatographie bei der Isolation des Stammes YEBTA-c-γ1-b-K174C. Angegeben ist der Umsatz von NADH vor und nach der Zugabe von Lauryldimethylaminoxid (LDAO). Zusätzlich ist der Wert für eine Probe des Wildtyps YRD15 angegeben (grau unterlegt). Fraktion Aktivität ohne LDAO Aktivität mit LDAO Aktivierung / μm min 1 / μm min 1 Faktor A ,7 A ,5 A ,8 A ,3 (2. Peak ) WT-A ,3 Abbildung 6.5 dargestellt. Die Fraktionen wurden unter Lichtausschluss auf Eis gestellt. relative Absorbanz bei 280 nm 1,0 0,5 0, Fraktionsnummer Abbildung 6.5: Elutionsprofil der Größenausschlusschromatographie S-300 der Isolation von YF 0 F 1. Exemplarisch für den YEBTA-c-γ1-b-K174C-Stamm, gezeigt ist die relative Absorbanz bei 280 nm. Grau unterlegt sind hydrolyseaktive Fraktionen, grau schraffierte Fraktionen sind durch LDAO gut aktivierbar. 104
123 6.3 Isolation der H + -ATPsynthase aus S. cerevisiae Mit der One-Step-Methode (vgl. Kapitel 3.9.1) wurde die Hydrolyseaktivität und die Aktivierbarkeit mit LDAO gemessen (Ergebnisse siehe Tabelle 6.2). Von der Peakfraktion 42 wurde Tabelle 6.2: Hydrolyse der Fraktionen nach der Größenausschlusschromatographie S-300 bei der Isolation des Stammes YEBTA-c-γ1-b-K174C. Angegeben ist der Umsatz von NADH vor und nach der Zugabe von Lauryldimethylaminoxid (LDAO). Zusätzlich ist der Wert für eine Probe des Wildtyps YRD15 angegeben (grau unterlegt) Fraktion Volumen Aktivität ohne LDAO Aktivität mit LDAO Aktivierung μl / μm min 1 / μm min 1 Faktor , , , , ,5 WT ,1 mittels UV/VIS-Spektroskopie die Konzentration zu 1,5 μm (ɛ 280nm = M 1 cm 1 ) bestimmt. Für Markierungen und Rekonstitutionen wird eine höhere Proteinkonzentration benötigt, deshalb wurden die hydrolyseaktiven und gut aktivierbaren Fraktionen 41-45, sowie die Randfraktionen und 46 jeweils vereinigt und konzentriert. Von der so erhaltenen Peakfraktion P1 und der Randfraktion R1 wurde die Konzentration des Fusionsproteins, sowie die Konzentration von EGFP (ɛ (478nm) = M 1 cm 1 ) bestimmt (vgl. Tabelle 6.3). Tabelle 6.3: Konzentration der Peak- (P1) und der Randfraktion (R1) des Stammes YEBTA-c-γ1-b-K174C. Die Konzentration des gesamten Fusionsproteins wurde bei 280 nm, die Konzentration des EGFP bei 487 nm mittels UV/VIS-Spektroskopie bestimmt. Daraus wurde der Markierungsgrad für das Fusionsprotein berechnet. Fraktion c[yf 0 F 1 -EGFP] 280nm c[egfp] 487nm Markierungsgrad / μm / μm / % P1 26,0 4,2 16 R1 15,4 1,
124 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Die erhaltenen Spektren, sowie ein Spektrum von reinem EGFP ist in Abbildung 6.6 dargestellt. Nach der genomischen Integration des EGFPs in die H + -ATPsynthase F 0 F 1, wurde ein 1,0 0,15 0,10 relative Absorbanz 0,5 0,05 0, , Wellenlänge / nm Abbildung 6.6: UV/VIS-Spektren von YF 0 F 1 -EGFP-Fusionsprotein und EGFP. Normierte Absorptionsspektren für den YEBTA-c-γ1-b-K174C-Stamm, rote Kurve: Spektrum der Peakfraktion P1 nach der Größenausschlusschromatographie, blaue Kurve: Spektrum der Randfraktion R1, grüne Kurve: Spektrum von reinem EGFP. Der Bereich der Absorption durch EGFP bei 488 nm wurde für die Fraktionen P1 und R1 zusätzlich herausvergrößert. deutlich höherer Markierungsgrad erwartet. Gründe für den geringen Anteil an EGFP im Absorptionsspektrum könnten zum Einen Probleme bei der Expression sein, sodass sich der Chromophor nicht vollständig ausbildet (1. Proteinfaltung, 2. Oxidation des Chromophors). Möglich ist auch, dass das EGFP aus dem Konstrukt im Reinigungsprozess an den integrierten Proteaseschnittstellen abgespalten wird und als Untereinheit des Fusionsproteins verloren geht. Falls die Reinigung des Proteins nicht ausreichend war, ist die Berechnung der Proteinkonzentration von YF 0 F 1 zu hoch. Alle drei Effekte führen zu einer Unterschätzung des Markierungsgrades. Um die Reinheit der Fraktionen zu überprüfen, wurde eine Probe der konzentrierten Lösung nach der Anionenaustauschsäule, eine Probe der Peakfraktion 42 der Größenausschlusschromatographie, sowie Proben der konzentrierten Fraktionen P1 und R1 auf ein SDS-Gel aufgetragen und eine Gelelektrophorese durchgeführt (vgl. Kapitel 3.5). Zum Vergleich wurde eine Probe mit Wildtyp-YF 0 F 1 aufgetragen. Das Gel wurde zunächst mit Coomassie Blau gefärbt (vgl. Kapitel 3.5.2), dann mit kolloidalem Silber (vgl. Kapitel 3.5.3) 106
125 6.3 Isolation der H + -ATPsynthase aus S. cerevisiae und ist in Abbildung 6.7 gezeigt. Beim Vergleich der Spur 1 mit der Spur 5 ist gut zu sehen, dass Abbildung 6.7: SDS-PAGE der Fraktionen der Isolation von YEBTA-c-γ1- b-k174c. Coomassie Blau gefärbtes Gel (13 %iges Trenngel) der konzentrierten Proteinlösung der Anionenaustauschsäule (Spur 1), der Peakfraktion 41 der Größenausschlusschromatographie (Spur 2), der konzentrierten Randfraktion R1 (Spur 3), einer YF 0 F 1 -Wildtyp Probe (Spur 4) und der konzentrierten Peakfraktion P1 (Spur 5). Zusätzlich ist ein LMW- Proteinstandard aufgetragen ( kda, Spur 6) und die konzentrierte Peakfraktion P1 nach der Färbung mit kolloidalem Silber gezeigt (Spur 7). durch die Gelpermeationschromatographie die Reinheit der Probe verbessert wurde. Allerdings sind noch Proteine im Gel zu erkennen, welche nicht der H + -ATPsynthase zugeordnet werden können. Eine exakte Zuordnung der Proteinbanden ist schwierig aufgrund ähnlicher Molmassen von verschiedenen Untereinheiten und damit ähnlichem Laufverhalten im SDS-Gel [68]. Zu erkennen ist jedoch, dass die in Spur 4 gut sichtbare Bande der γ-untereinheit (31 kda) fast vollständig verschwunden ist und dafür eine neue Bande oberhalb der α-untereinheit erscheint, bei der es sich um die Fusionsuntereinheit γ-egfp (ca. 66 kda) handeln kann. Unklar hingegen war, um welches Protein es sich bei der deutlich intensiveren Bande bei ca. 40 kda handelte, deshalb wurde eine Sequenzierung mittels HPLC-MS durchgeführt. 107
126 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Untersuchung unbekannter Proteine mittels HPLC-MS Die Identifizierung wurde von der Arbeitsgruppe Dr. Schlosser (ZBSA, Universität Freiburg) durchgeführt. Zur Vermeidung von Verunreinigungen wurden alle Schritte unter sterilen Bedingungen durchgeführt. Zunächst wurde eine Probe der P1-Fraktion mit Hilfe einer Gradienten- SDS-Gelelektrophorese aufgetrennt und die orangefarben markierten Banden mit einem Skalpell ausgeschnitten (siehe Abbildung 6.8). Anschließend wurden die in den Streifen enthaltenen kda 60 kda 50 kda 40 kda I. II. III. 30 kda Abbildung 6.8: SDS-PAGE der Peakfraktion P1 von YEBTA-c-γ1-b-K174C zur anschließenden Sequenzierung mittels HPLC-MS. Coomassie Blau gefärbtes kommerzielles Gradientengel der konzentrierten Peakfraktion P1 (Spur 2 und 3). Die Proteinbanden, die zur Identifizierung sequenziert wurden, sind orangefarben markiert. Zusätzlich ist ein Proteinstandard aufgetragen (Spur 1). Das Gel wurde in der Arbeitsgruppe Dr. Schlosser angefertigt. Proteine eluiert und mit dem Enzym Trypsin jeweils nach den Aminosäuren Lysin und Arginin gespalten. Die resultierenden Polypeptide wurden durch eine vorgeschaltete HPLC aufgetrennt und in einem Massenspektrometer analysiert. Die erhaltenen Sequenzen wurden mit Hilfe der Proteindatenbank [Swissprot] abgeglichen und so die in den Banden enthaltenen Proteine zugeordnet. Die Bande I enthielt laut Sequenzierung die erwartete γ-tev-link-egfp-untereinheit, zusätzlich wurde in dieser Bande auch die β-untereinheit gefunden. Die Bande II zeigte die β-untereinheit, sowie EGFP. Die dritte intensive Bande III enthielt eindeutig die Untereinheit 2 des Cytochrom bc 1 -Komplexes (Komplex III) und kein EGFP. Dieses Ergebnis stimmt mit der Sequenzierung des Wildtyps von YF 0 F 1 in der Doktorarbeit bei K. Förster überein, in der für die Molmasse von 40,5 kda ebenfalls die Untereinheit 2 des Cytochrom bc 1 -Komplexes identifiziert wurde. 108
127 6.3 Isolation der H + -ATPsynthase aus S. cerevisiae Disskusion Für die Trennung der Untereinheiten zur Sequenzierung wurde ein kommerzielles Gradientengel verwendet (siehe Abbildung 6.8). Es ist auch hier ersichtlich, dass die Peakfraktion P1 noch weitere Proteine enthält. Dies führt bei der Konzentrationsbestimmung zu einer höheren Proteinmenge. Damit ist der Markierungsgrad von EGFP an YF 0 F 1 höher als mit UV/VIS- Spektroskopie gemessen. Bereits im Elutionsprofil der Größenausschlusschromatographie ist erkennbar, dass die Abtrennung der hydrolyseaktiven Fraktionen nicht optimal ist, ein isolierter Peak wäre wünschenswert. Dies könnte durch den Einsatz einer längeren Säule erreicht werden, allerdings erhöht sich damit auch das Elutionsvolumen, was wiederum zu noch geringeren Proteinkonzentrationen in den Fraktionen führt und die Dissoziation des Komplexes begünstigt. Auch die Verwendung eines anderen Säulenmaterials könnte die Trennung verbessern. Durch die Aufkonzentrierung einzelner Fraktionen wäre es ebenfalls möglich, reineres Protein zu erhalten, allerdings würde die Ausbeute sich deutlich verringern und das Aufkonzentrieren von kleinen Volumina in Konzentratoren könnte zu einem höheren Verlust von Untereinheiten und damit zu einem Aktivitätsverlust der isolierten H + -ATPsynthasen führen. Die kovalente Kopplung des Zip-Helix-FP-Konstrukts an die γ-untereinheit scheint stabil, da das Gel sowohl die Anwesenheit der γ-egfp-untereinheit als auch die Abwesenheit der γ- Untereinheit zeigt. In der Untersuchung mit HPLC-MS wurde in der der obersten Bande GFP gefunden. Die Bande entspricht in etwa der Molmasse der γ-untereinheit (31 kda) inklusive des hier verwendeten FP-Konstrukts (ca. 35 kda, vgl. Kapitel 6.1) von ca. 66 kda. Zusätzlich wurde EGFP in der zweiten Bande gefunden, die einer Molmasse von ca. 50 kda entspricht, dies weist möglicherweise auf einen Abbau des Proteins hin. In der dritten, deutlich prominenteren Bande bei ca kda im SDS-Gel wurde keine Übereinstimmung mit der Sequenz von GFP gefunden, d.h. freies GFP ist in der Präparation nicht vorhanden. Warum diese Bande bei der Präparation des YEBTA-c-γ1-b-K174C-Stammes stärker ist, als bei der des Wildtyps YRD15 ist unklar. Eventuell wird durch den Einbau von mehr Cytochrom bc 1 -Komplexen in die Atmungskette, und damit verbunden einem schnelleren Aufbau des Protonengradienten, eine geringere Aktivität der H + -ATPsynthasen ausgeglichen. Aufgrund der Verunreinigungen wird der berechnete Markierungsgrad (vgl. Tabelle 6.3) deutlich geringer als erwartet eingeschätzt. Zusätzlich könnten auch Fehler in der Exprimierung des EGFPs Grund für den geringen, gemessenen Markierungsgrad sein. Sowohl Fehlfaltungen des β-barrels, als auch Fehler in der Chromophorengruppe oder ein Ausbleichen des Chromophors, würden zu einer Veränderung des Absorptionsspektrums führen. Im Folgenden wird mit dem aus der UV/VIS-Spektrometrie bestimmten Markierungsgrad weitergearbeitet, da dieser dem minimalen Markierungsgrad entspricht. 109
128 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae 6.4 Fluoreszenzspektroskopische Charakterisierung Fluoreszenzmarkierung von F 0 b174 Für die Messung von Fluoreszenz-Resonanz-Energie-Transfer (FRET, siehe Kapitel 1.3.3), wurde das EGFP-Fusionsenzym zusätzlich mit dem organischen Fluorophor ATTO565 der Firma ATTO-TEC markiert. Die Markierung erfolgte wie in Kapitel 3.3 beschrieben. Um einen maximalen Markierungsgrad zu erreichen, wurden verschiedene Konzentrationen von YF 0 F 1 zu ATTO565 getestet. Zunächst wurden 6,5 μm Enzym mit 7,8 μm bzw. 19,5 μm Fluorophor gemischt, 2 min unter Lichtausschluss auf Eis inkubiert, freier Fluorophor abgetrennt und die Markierung mittels eines SDS-Gels überprüft (siehe Abbildung 6.9a). In dem Gel ist a) b) c) Abbildung 6.9: SDS-PAGE der mit EGFP/ATTO565 doppelt markierten Probe YEBTA-c-γ1-b-K174C. a) Markierung von 6,5 μm YF 0 F 1 mit 7,8 μm (Spur 1) bzw. 19,5 μm (Spur 2) ATTO565 und unzureichender Abtrennung des nichtgebundenen Fluorophors nach einer Penefsky-Säule. b) Markierung von 6,5 μm YF 0 F 1 mit 32,5 μm ATTO565 (III; Spur 3) und dem zugehörigen Intensitätsplot (Spur 4) im Vergleich zur Markierung mit 19,5 μm (I; Spur 5) ATTO565 und dem Intensitätsplot (Spur 6). Zur Zuordnung der Untereinheiten ist in Spur 7 das Gel mit kolloidalem Silber gefärbt (Intensitätsplot in Spur 8), der freie Fluorophor wurde mit zwei Penefsy- Säulen abgetrennt. c) Markierung von 6,5 μm YF 0 F 1 des Stammes YEBTAc-γ1-b-K174C mit 19,5 μm ATTO565 (I; Spur 10) und dem zugehörigen Intensitätsplot (Spur 9) im Vergleich zur Markierung von 8,5 μm YF 0 F 1 des Stammes YRD15 mit 25,5 μm ATTO565 (IV; Spur 11) und dem zugehörigen Intensitätsplot (Spur 12). Der freie Fluorophor wurde mit zwei Penefsky- Säulen abgetrennt, die Aufnahme wurde mit Hilfe eines UV-Filters gemacht. 110
129 6.4 Fluoreszenzspektroskopische Charakterisierung zu erkennen, dass noch eine größere Menge an freiem Fluorophor in der Probe vorhanden ist. Um den Markierungsgrad zu verbessern, wurden weitere Konzentrationsverhältnisse von YF 0 F 1 und ATTO565 (I:6,5/19,5 μm; II:6,5/26,0 und III:6,5/32,5; siehe Abbildung 6.9b), II ist nicht gezeigt) getestet, gleichzeitig wurde versucht den freien Fluorophor durch eine direkt anschließende zweite Penefsky-Säule vollständig abzutrennen. Da die Zuordnung der Untereinheiten in den SDS-Gelen von YF 0 F 1 nicht eindeutig war, wurde überprüft ob die Markierung an der veränderten Position b174 erfolgte. Dazu wurde sowohl die markierte Doppelmutante (III: YEBTA-c-γ1-b-K174C/ATTO565 = 6,5/19,5 μm) als auch eine gleichermaßen mit ATTO565 markierte Probe des Wildtyp-YF 0 F 1 (IV: YRD15/ATTO565 = 8,5/25,5 μm, zwei Penefsky-Säulen) auf ein SDS-Gel aufgetragen. Dieses Gel ist in Abildung 6.9c gezeigt. Von den Markierungen I - IV wurden zur Bestimmung des Markierungsgrads UV/VIS-Spektren von 2 μl Probe in 78 μl HEPES-Puffer aufgenommen (vgl. Kapitel 3.4.1). Die daraus berechneten Markierungsgrade sind in Tabelle 6.4 zusammengefasst. Tabelle 6.4: Markierungsgrad von YF 0 F 1 des Stammes YEBTA-c-γ1-b-K174C mit ATTO565. Es wurden 6,5 μm YEBTA-c-γ1-b-K174C mit 19,5 μm (I), 26,0 μm (II) und 32,5 μm (III) ATTO565 markiert, sowie 8,5 μm YRD15 mit 25,5 μm ATTO565 (IV). Die Konzentrationen und der Markierungsgrad wurden nach Abtrennung von freiem Fluorophor durch zwei Penefsky-Säulen bestimmt. Probe n[yf 0 F 1 -EGFP] n[atto565] Markierungsgrad / μm / μm / % I 6,3 2,5 40 II 6,2 3,1 50 III 5,6 3,6 64 IV 5,3 0,9 17 Die Abtrennung des noch vorliegenden freien Fluorophors wurde durch eine zweite Penefsky- Säule erreicht. Durch die Zugabe von mehr Fluorophor konnte ein höherer Markierungsgrad erreicht werden, vergleicht man die beiden Intensitätsplots (Abbildung 6.9 Spur 4 und 6) schien eine höhere ATTO565 Konzentration jedoch die Selektivität der Markierung zu verschlechtern. Im Folgenden wurde deshalb eine YF 0 F 1 zu ATTO565 Konzentration von 6,5 und 19,5 μm (I) verwendet. Der Vergleich mit der Markierung einer Wildtyp-YF 0 F 1 Probe aus dem Stamm YRD15 (IV) zeigte deutlich, dass die Markierung an der veränderten Position b174 erfolgte. In dieser Probe war zu erkennen, dass deutlich weniger Banden fluoreszenzmarkiert waren. Dies könnte bedeuten, dass neben der b-untereinheit hauptsächlich die Proteine, welche als 111
130 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Verunreinigung in der Probe vorlagen, markiert wurden, oder die b-untereinheit teilweise abgebaut wurde Syntheseaktivität nach der Markierung Durch die Bindung eines Fluorophors kann die Aktivität des Enzyms verändert werden. Um die Rotationsbewegung der H + -ATPsynthase messen zu können, muss das Enzym noch eine Aktivität vorweisen, deshalb wurde diese überprüft. Dazu wurden zum Vergleich Wildtyp YF 0 F 1, EGFP-YF 0 F 1 -Fusionsprotein und eine mit ATTO565 doppelt markierte Probe rekonstituiert (siehe Kapitel 3.7) und die ATP-Syntheseaktivität (vgl. Kapitel 3.8) bestimmt. Die Werte sind in Tabelle 6.5 aufgeführt. Das EGFP-YF 0 F 1 -Fusionsprotein zeigt nur noch ca. Tabelle 6.5: Syntheseaktivität (turnover) von YF 0 F 1. Es wurde der turnover von Wildtyp-YF 0 F 1 (YRD15), von EGFP-YF 0 F 1 -Fusionsprotein (YEBTA-c-γ1- b-k174c) und von doppelt markiertem EGFP-YF 0 F 1 -Fusionsprotein mit ATTO565 bestimmt. Probe / Stamm turnover /s 1 YF 0 F 1 / YRD15 47 ± 13 EGFP-YF 0 F 1 / YEBTA c-γ1-b-k174c 5 ± 2 ATTO565-EGFP-YF 0 F 1 / YEBTA-c-γ1-b-K174C 6 ± 1 10 % der Syntheseaktivität des Wildtyp-YF 0 F 1. Durch die Markierung mit ATTO565 kam es bei der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1 zu keiner weiteren Abnahme der Syntheseaktivität. 6.5 Bestimmung des Försterradius des Fluorophorenpaars EGFP/ATTO565 Um Aussagen über die Abstände zwischen den beiden Fluorophoren treffen zu können, wird der Försterradius benötigt (siehe Kapitel 1.3.3). Dieser lässt sich mit Gleichung 6.1 berechnen. 112
131 6.5 Bestimmung des Försterradius des Fluorophorenpaars EGFP/ATTO565 R 0 =0, 221 [ κ 2 n 4 Φ D J(λ) ] 1/6 (6.1) R 0 κ 2 Försterradius (Å) Orientierungsfaktor n Brechungsindex des Lösungsmittels Φ D Fluoreszenzquantenausbeute des Donorfluorophors J(λ) Überlappungsintegral (M 1 cm 1 nm 4 ) 0,211 Vorfaktor (Å/[M 1 cm 1 nm 4 ] 1/6 ) Die Fluoreszenzquantenausbeute Φ gibt das Verhältnis zwischen absorbierten und emittierten Photonen wieder. Für das freie EGFP wird bei einem ph = 8 eine Quantenausbeute von 60 % [157] angegeben. Durch Bindung an das Enzym ändert sich die Quantenausbeute der Fluorophore. Um den Effekt zu quantifizieren, wurde das Fluoreszenzspektrum von freiem EGFP, das Fluoreszenzspektrum von EGFP am Fusionsprotein und das Spektrum der doppelt markierten Probe EGFP/ATTO565 gemessen. Für diese Messungen wurde jeweils die gleiche EGFP Konzentration eingestellt (0,05 μm). Die verwendeten Konzentrationen wurden durch Messung der Absorptionsspektren wie in Kapitel beschrieben bestimmt. Der Wert für das Fusionsprotein kann der Tabelle 6.3 (P1) und der Wert für die doppelt markierte Probe der Tabelle 6.4 (I) entnommen werden. Um bei der Integration den Effekt durch ATTO565 vernachlässigen zu können, wurde über den Bereich zwischen 500 und 550 nm integriert und daraus die Änderung der Quantenausbeute bestimmt (siehe Abbildung 6.10 und Tabelle 6.6). Der Unterschied in der Quantenausbeute zwischen der einfach- und der doppelt markierten Probe (EGFP-YF 0 F 1 :ATTO565-EGFP-YF 0 F 1 = -33 %) deutet auf einen Energietransfer von Donor- zu Akzeptorfluorophor hin. Tabelle 6.6: Fluoreszenzquantenausbeute von EGFP. Fluoreszenzquantenausbeute von EGFP, EGFP-YF 0 F 1 und ATTO565-EGFP-YF 0 F 1. Die Werte wurden im Bezug zum Literaturwert für EGFP durch Integration gemessener Fluoreszenzspektren (vgl. Abbildung 6.10) berechnet. Probe / Stamm Fluoreszenzquantenausbeute Literatur EGFP 0,60 [157] EGFP-YF 0 F 1 / YEBTA c-γ1-b-k174c 0,42 ATTO565-EGFP-YF 0 F 1 / YEBTA-c-γ1-b-K174C 0,28 113
132 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae relative Fluoreszenzintensität EGFP EGFP-YF 0 F 1 korrigiert ATTO565-EGFP-YF 0 F 1 korrigiert Wellenlänge / nm Abbildung 6.10: Fluoreszenzspektren zur Bestimmung der Fluoreszenzquantenausbeute. Zur Bestimmung der Fluoreszenzquantenausbeute von gebundenem EGFP wurde das Fluoreszenzspektrum von freiem EGFP mit dem Fluoreszenzspektrum von EGFP-YF 0 F 1 und ATTO565-EGFP-YF 0 F 1 verglichen. Die Anregung erfolgte bei 460 nm in HEPES-Puffer (siehe Kapitel 4.1), die Konzentration des EGFPs betrug 0,05 μm. Die Spektren von EGFP-YF 0 F 1 und ATTO565-EGFP-YF 0 F 1 wurden um den Anteil der Fluoreszenz der Probe korrigiert (siehe Kapitel 6.6.1). Zur Bestimmung des Überlappungsintegrals J(λ) wurde das Emissionsspektrum des EGFPs gemessen. Aufgrund des Geräteaufbaus und der Spaltbreite wurden die Werte bei einer Wellenlänge kleiner 500 nm nicht aufgezeichnet und dieser Wert auf 0 gesetzt. Dies gibt in guter Näherung den realen Verlauf des Spektrums von EGFP wieder [158]. Für das Absorptionsspektrum des ATTO565 wurden die Werte von der Firma ATTO-TEC übernommen [126]; dieses ist für diesen Farbstoff äquivalent zum Anregungsspektrum. Die normierten Spektren sind in Abbildung 6.11 gezeigt. Für das freie EGFP ergab sich durch Integration über F D (λ) (auf Maximum normiertes Fluoreszenzspektrum, Einheit siehe Anhang B): 0 F D (λ) dλ =39, 18 s 1 = F (gesamt) (6.2) Daraus wird die auf die Gesamtfluoreszenz bezogene spektrale Fluoreszenz f D berechnet: f D (λ) = F D (λ) F 0 D (λ) dλ (6.3) 114
133 6.5 Bestimmung des Försterradius des Fluorophorenpaars EGFP/ATTO565 normierte Fluoreszenzintensität normierte Absorbanz 1,0 0,8 0,6 0,4 0,2 0, Wellenlänge / nm Abbildung 6.11: Normiertes Emissionsspektrum des Donorfluorophors EGFP und normiertes Absorptionsspektrum des Akzeptorfluorophors ATTO565. Das Emissionsspektrum von EGFP wurde gemessen (Anregung 460 nm, Emission nm in HEPES-Puffer; siehe Kapitel 4.1), das Absorptionsspektrum von ATTO565 wurde aus dem Datenblatt der Firma ATTO-TEC übernommen [126]. Die Spektren wurden jeweils auf das Maximum normiert. Das auf die Gesamtfluoreszenz bezogene Fluoreszenzspektrum ist in Abbildung 6.12 gezeigt: 0,030 0,025 normiertes Emissionsspektrum des freien EGFP molarer Absorptionskoeffizient des freien ATTO565 1,25x10 5 f D (λ) 0,020 0,015 0,010 0,005 1,00x10 5 7,50x10 4 5,00x10 4 2,50x10 4 ε A (λ) /M -1 cm -1 0,000 0, Wellenlänge / nm Abbildung 6.12: Auf die Gesamtfluoreszenz normiertes Emissionsspektrum des Donorfluorophors EGFP und Spektrum des molaren Absorptionskoeffizienten des Akzeptorfluorophors ATTO
134 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Die Überlappungsfunktion ist in Gleichung 6.4 definiert und durch Einsetzen des auf die Gesamtfluoreszenz normierten Emissionsspektrums und des Spektrums des molaren Absorptionskoeffizienten (siehe Abbildung 6.12) erhält man die wellenlängenabhängige Überlappungsfunktion in Abbildung Überlappungsfunktion = f D (λ) ɛ A (λ) λ 4 (6.4) Überlappungsfunktion / M -1 cm -1 nm 4 6x x x x x x Wellenlänge / nm Abbildung 6.13: Wellenlängenabhängige Überlappungsfunktion des Fluorophorenpaars EGFP/ATTO565. Durch Integration erhält man das Überlappungsintegral J für das hier verwendete Fluorophorenpaar EGFP/ATTO565: J = 0 f D (λ) ɛ A (λ) λ 4 dλ =3, M 1 cm 1 nm 4 (6.5) Mit der Annahme für den Orientierungsfaktor κ 2 = 2/3 (siehe Kapitel 1.3.3), der berechneten Fluoreszenzquantenausbeute für gebundenes EGFP Φ D = 0,42 und einem Brechungsindex der Lösung (Wasser) von n = 1,33 wurde mit Hilfe der Gleichung 6.1 der Försterradius für das hier verwendete Fluorophorenpaar EGFP/ATTO565 berechnet (Einheiten siehe Anhang B). R 0 =0, 221 [ κ 2 n 4 Φ D J(λ) ] 1/6 =0, 221 [ 2 3 1, , 42 3, ] 1/6 =55Å 116
135 6.6 Bestimmung des Korrekturfaktors γ 6.6 Bestimmung des Korrekturfaktors γ Für die Berechnung der FRET-Effizienzen wird bei der Einzemolekülspektroskopie zusätzlich der gerätespezifische Korrekturfaktor γ benötigt (siehe Gleichung 6.6). Dieser berechnet sich aus den Quantenausbeuten der gebundenen Fluorophore, sowie den gerätespezifischen Detektionseffizienzen. γ = Φ A η A Φ D η D (6.6) γ Φ A Φ D η A η D gerätespeziefischer Korrekturfaktor Fluoreszenzquantenausbeute des Akzeptors Fluoreszenzquantenausbeute des Donors Detektionseffizienz des Akzeptorkanals Detektionseffizienz des Donorkanals Zur Berechunung wurde die in Tabelle 6.6 bestimmte Quantenausbeute für den gebundenen Donorfluorophor EGFP (EGFP-YF 0 F 1 ) verwendet, zusätzlich mussten noch die Quantenausbeute des gebundenen Akzeptorfluorophors ATTO565 und die Detektionseffizienzen bestimmt werden Bestimmung der Fluoreszenzquantenausbeute von ATTO565 Die Änderung der Fluoreszenzquantenausbeute von freiem zu gebundenem ATTO565 wurde analog zu der für EGFP bestimmt. Um einen Einfluss von EGFP durch FRET auf die Fluoreszenz von gebundenem ATTO565 zu vermeiden, wurde eine von Kathrin Förster zur Verfügung gestellte Probe der b174-mutante YRD15-b-K174C-γ-C84S/T121C [68] wie in Kapitel 3.3 beschrieben, markiert. Der freie Fluorophor wurde durch zwei Penefsky-Säulen abgetrennt. Die Konzentration des Fluorophors und der Markierungsgrad wurden durch ein Absorptionsspektrum (siehe Kapitel 3.4.1) bei 565 nm bestimmt und die Selektivität der Markierung durch ein SDS-Gel überprüft. In Abbildung 6.14 ist zu erkennen, dass die Markierung bei der b174-mutante YRD15-b-K174C-γ-C84S/T121C analog zu der Probe der doppelt markierten Probe YEBTA-c-γ1-b-K174C selektiv an der b-untereinheit erfolgte. Die zusätzlich eingebrachten Cysteine in der γ-untereinheit wurden nicht markiert. Somit kann die Änderung der Fluoreszenzquantenausbeute von ATTO565 durch Bindung an die b-untereinheit bei der b174-mutante YRD15-b-K174C-γ-C84S/T121C mit der Änderung bei Bindung an die b- Untereinheit im Fusionsprotein des YEBTA-c-γ1-b-K174C-Stammes gleich gesetzt werden. 117
136 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Abbildung 6.14: SDS-PAGE der mit EGFP/ATTO565 doppelt markierten Probe YEBTA-c-γ1-b-K174C und der mit ATTO565 markierten Probe YRD15-b-K174C-γ-C84S/T121C. Markierung von 6,5 μm YF 0 F 1 des Stammes YEBTA-c-γ1-b-K174C mit 19,5 μm ATTO565 (Spur 1, entnommen aus Abbildung 6.9c) im Vergleich zur Markierung von 8,0 μm YF 0 F 1 des Stammes YRD15-b-K174C-γ-C84S/T121C mit 24,0 μm ATTO565 (Spur 3). Zur Zuordnung der Untereinheiten ist in Spur 2 das mit kolloidalem Silber gefärbte Gel aus Abbildung 6.9b gezeigt. Der freie Fluorophor wurde mit zwei Penefsky-Säulen abgetrennt, die Aufnahme wurde mit Hilfe eines UV-Filters gemacht. Anschließend wurden die Fluoreszenzspektren von freiem ATTO565 und dem an YRD15-b- K174C-γ-C84S/T121C gebundenen ATTO565 bei gleicher Konzentration von ATTO565 im Ansatz gemessen (siehe Abbildung 6.15). Im Fluoreszenzspektrum des gebundenen ATTO565 war der Fluoreszenzpeak zu längerer Wellenlänge verbreitert. Auch im Fluoreszenzspektrum des EGFP-Fusionsproteins des YEBTA-c-γ1-b-K174C-Stammes wurde ab 575 nm ein Anstieg der Fluoreszenz gemessen. Vermutlich rührt dies von der Fluoreszenz einer Verunreinigung in der Probe her, möglicherweise handelt es sich um Cytochrome. Diese Fluoreszenz der Verunreinigung wurde wie folgt bestimmt: Vom Spektrum des EGFP-YF 0 F 1 wurde ein auf die maximale Fluoreszenzintensität des EGFP-YF 0 F 1 normiertes EGFP Spektrum substrahiert. Diese Spektren und die Differenz sind in Abbildung 6.16 gezeigt. Da für die Einzelmolekülmessungen die H + -ATPsynthasen zunächst rekonstituiert und dann die Proteoliposomen in sehr geringer Konzentration zugegeben wurden, wirkte sich die Fluoreszenz durch Verunreinigungen in der Probe nicht auf die Einzelmolkülmessungen aus, bzw. diese wurde durch die Bestimmung der Hintergrundfluoreszenz subtrahiert (vgl. Kapitel 6.8). 118
137 6.6 Bestimmung des Korrekturfaktors γ relative Fluoreszenzintensität Wellenlänge / nm ATTO565 ATTO565-YF 0 F 1 Abbildung 6.15: Fluoreszenzspektren von freiem ATTO565 (orange) und an YF 0 F 1 gebundenem ATTO565 (ATTO565-YF 0 F 1 ; blau). Die Anregung erfolgte bei 460 nm in HEPES-Puffer, die Emission wurde im Bereich von nm aufgezeichnet, die Konzentration von ATTO565 betrug 0,164 μm im Ansatz (siehe Kapitel 4.1). relative Fluoreszenzintensität EGFP EGFP-YF 0 F 1 Fluoreszenz der Verunreinigung in der Probe Wellenlänge / nm Abbildung 6.16: Bestimmung der Fluoreszenz der Verunreinigungen in der Probe. Das Fluoreszenzspektrum von EGFP (grün) wurde auf das Maximum des EGFP-YF 0 F 1 Spektrums (schwarz) normiert und anschließend subtrahiert. Die resultierende Differenz zeigt das Spektrum der Verunreinigung in der Probe (rot). Die Anregung erfolgte bei 460 nm in HEPES-Puffer, die Emission wurde im Bereich von nm aufgezeichnet, die Konzentration von EGFP betrug 0,05 μm im Ansatz, die Konzentration von YF 0 F 1 0,31 μm (siehe Kapitel 4.1). 119
138 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Die Abbildung 6.17 zeigt die Fluoreszenzspektren des freien und des um die Fluoreszenz der Verunreinigungen in der Probe korrigierten ATTO565. Durch Integration der Spektren in Abbildung 6.17 über den Bereich von nm und der Fluoreszenzquantenausbeute des freien Fluorophors von Φ = 0,90 [126] wurde die Fluoreszenzquantenausbeute des gebundenen ATTO565 zu Φ A = 0,84 bestimmt. relative Fluoreszenzintensität Wellenlänge / nm ATTO565 ATTO565-YF 0 F 1 korrigiert Abbildung 6.17: Fluoreszenzspektren von freiem ATTO565 (orange; aus Abbildung 6.15) und des um die Fluoreszenz der Verunreinigungen in der Probe korrigierten gebundenen ATTO565 (blau; Differenz der Spektren von ATTO565-YF 0 F 1 Abbildung 6.15 und des Spektrums der Verunreinigung Abbildung 6.16). Die Anregung erfolgte bei 460 nm in HEPES-Puffer, die Emission wurde im Bereich von nm aufgezeichnet, die Konzentration von ATTO565 betrug 0,164 μm im Ansatz (siehe Kapitel 4.1). 120
139 6.6 Bestimmung des Korrekturfaktors γ Bestimmung der Detektionseffizienzen Die Detektionseffizienzen im Akzeptor- und Donorkanal unterscheiden sich durch unterschiedliche, wellenlängenabhängige Transmission und Reflexion der optischen Bauteile im Strahlengang (vgl. Kapitel 4.3). Durch Multiplikation der normierten Spektren (siehe Abbildung 6.18 und 6.19) erhält man nach Gleichung 6.7 die wellenlängenabhängige Detektionseffizienz η(λ). n η(λ) = T n (λ) (6.7) n=0 η(λ) T n (λ) Detektionseffizienz der konfokalen Detektionseinheit normiertes Spektrum der Optik in der Detektionseinheit - Transmissionsspektrum RDC Transmissionsspektrum DCLP 545 (nur Akzeptor) - Reflexionsspektrum DCLP 454 (nur Donor) - Transmissionsspektrum HQ 525/50 (nur Donor) - Transmissionsspektrum HQ 608/60 (nur Akzeptor) - Spektrum der Detektionseffizienz der APD Reflexion / Transmission relative Fluoreszenzintensität 1,0 0,8 0,6 0,4 0,2 0, Wellenlänge / nm RDC 488 (Transmission) DCLP 545 (Reflexion) Emission EGFP Detektionseffizienz APD HQ 525/50 (Transmission) Abbildung 6.18: Normierte Spektren der optischen Bauteile im Strahlengang zur Bestimmung der Detektionseffizienz für den Donor EGFP. Die Spektren für die Strahlenteiler RDC 488 und DCLP 545 wurden gemessen, das Spektrum für den Bandpassfilter 525/50 wurde von der Firma AHF [159] und das Spektrum für die Detektionseffizienz der APD aus der Arbeit von B. Zimmermann [74] übernommen. 121
140 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Transmission der Bauteile relative Fluoreszenzintensität 1,0 0,8 0,6 0,4 0,2 0, Wellenlänge / nm RDC 488 (Transmission) DCLP 545 (Transmission) Emission ATTO565 Detektionseffizienz APD HQ 608/60 (Transmission) Abbildung 6.19: Normierte Spektren der optischen Bauteile im Strahlengang zur Bestimmung der Detektionseffizienz für den Akzeptor ATTO565. Die Spektren für die Strahlenteiler RDC 488 und DCLP 545 wurden gemessen, das Spektrum für den Bandpassfilter 608/60 und das Spektrum für die Detektionseffizienz der APD wurden aus der Arbeit von B. Zimmermann [74] übernommen. Ausgehend von diesen Spektren konnten die Detektionseffizienzen für den Donor- bzw. Akzeptorkanal bei dem jeweiligen Maximum der Fluorophore berechnet werden, die Ergebnisse sind in Tabelle 6.7 aufgeführt. Tabelle 6.7: Detektionseffizienzen für den Donor- und Akzeptorkanal. Die Detektionseffizienz für den Donorkanal wurde bei einer Wellenlänge von 509 nm aus den Spektren der Abbildung 6.18 berechnet, die Detektionseffizienz für den Akzeptorkanal bei einer Wellenlänge von 588 nm aus den Spektren der Abbildung η D η A Detektionseffizienz 0,453 0, Berechung des Korrekturfaktors γ Mit den in Kapitel 6.5 und Kapitel bestimmten Fluoreszenzquantenausbeuten und den in Kapitel berechneten Detektionseffizienzen konnte mit der Gleichung 6.6 der Korrekturfaktor für die FRET-Effizienzen berechnet werden. γ = Φ A η A Φ D η D = 0, 84 0, 601 =2, 65 0, 42 0,
141 6.7 Ensemble Fluoreszenzspektroskopie 6.7 Ensemble Fluoreszenzspektroskopie Test auf Anwesenheit eines aktiven FRET-Systems Bei der Anregung des Donors kann es zusätzlich auch zu einer Anregung des Akzeptors kommen, wenn bei dieser Wellenlänge auch der Akzeptor absorbiert. Das Spektrum einer solchen Direktanregung des Akzeptors ATTO565 ist in Abbildung 6.17 gezeigt. Kommt es zusätzlich zu einer Anregung des Akzeptors durch FRET, sollte sich die Fluoreszenz des Akzeptors ATTO565 im Fluoreszenzspektrum erhöhen. Mit Hilfe dieser Ensemblemessungen kann überprüft werden, ob sich die Proben prinzipiell für Einzelmolekül-FRET-Messungen eignen. Es wurde ein Fluoreszenzspektrum von der mit EGFP und ATTO565 doppelt markierten Probe aufgenommen. Die Durchführung erfolgte wie in Kapitel 4.1 beschrieben. relative Fluoreszenzintensität EGFP-YF 0 F 1 korrigiert ATTO565-YF 0 F 1 korrigiert ATTO565-EGFP-YF 0 F 1 korrigiert Wellenlänge / nm Abbildung 6.20: Normiertes Fluoreszenzemissionsspektrum der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1. Vergleich des Fluoreszenzemissionsspektrums der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1 (magenta) normiert auf das Maximum des Emissionsspektrums von EGFP- YF 0 F 1 (siehe auch Abbildung 6.10; schwarz) und des Fluoreszenzspektrums der mit ATTO565 einfach markierten Probe ATTO565-YF 0 F 1 (siehe auch Abbildung 6.17; blau). Alle Spektren wurden um den Anteil der Fluoreszenz der Verunreinigung in der Probe korrigiert. Die Anregung erfolgte bei 460 nm in HEPES-Puffer, die Emission wurde im Bereich von nm aufgezeichnet, die Konzentration von YF 0 F 1 betrug 0,31 μm, die Konzentration von ATTO565 0,165 μm und von EGFP 0,05 μm im Ansatz. 123
142 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Die Spektren in Abbildung 6.20 wurden um den Anteil an Fluoreszenz durch Verunreinigungen in der Probe korrigiert (siehe Kapitel 6.6.1). Da dieser Anteil bezogen auf das Spektrum des Fusionsproteins EGFP-YF 0 F 1 berechnet wurde (siehe Abbildung 6.16), erfolgte die Normierung aller Spektren auf das Maximum des in der EGFP-YF 0 F 1 -Probe gebundenen EGFPs. Die Fluoreszenzintensität wurde durch Integration des Spektrums berechnet. Dazu wurde von 575 bis 625 nm integriert, so konnte im Spektrum der Einfluss von EGFP bei der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1 im Bereich von ATTO565 vernachlässigt werden. Für die doppelt markierte Probe ergab sich eine um 46 % höhere Fluoreszenzintensität des gebundenen ATTO565 als bei der nur mit ATTO565 einfach markierten Probe aus Abbildung Test auf konformative Änderung durch Zugabe von AMPPNP Beim Spektrum in Abbildung 6.20 können alle Konformationen des Enzyms in statistischer Verteilung vorkommen. Durch die Zugabe von Nukleotiden könnten einzelne Konformationen bevorzugt werden. Ist dies der Fall, könnte sich das Spektrum durch Änderung des FRET ändern. Das nicht hydrolysierbare Nukleotid AMPPNP besetzt die katalytischen Bindungsplätze und inhibiert das Enzym. Vergleicht man die Spektren von gehemmten und nicht gehemmten Enzymen in Ensemblemessungen, kann untersucht werden, ob das Enzym unter diesen Bedingungen eine andere Konformation einnimmt (siehe Abbildung 6.21). Die Messung des Spektrums der doppelt markierten Probe unter Zugabe von AMPPNP (Endkonzentration 1 mm) wurde unter den gleichen Bedingungen wie in Abwesenheit von AMPPNP durchgeführt. Das Spektrum wurde mit dem gleichen Faktor (1,52) wie das Spektrum der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1 in Abbildung 6.20 auf das Maximum des in der EGFP-YF 0 F 1 -Probe gebundenen EGFPs (siehe Abbildung 6.10) normiert, um die Fluoreszenz der Verunreinigung der Probe korrigiert und auf den Wert der Fluoreszenzintensität bei 670 nm der EGFP-YF 0 F 1 -Probe gelegt. Die Fluoreszenzintensität wurde durch Integration im Bereich von 575 bis 625 nm für ATTO565 für die doppelt markierte Probe mit und ohne die Zugabe von AMPPNP berechnet. Die Fluoreszenzintensität war für die doppelt markierte Probe ATTO565-EGFP-YF 0 F 1 in Anwesenheit von AMPPNP um 29 % geringer. Der EGFP-Peak ist im Vergleich zu längerer Wellenlänge verbreitert, was an einer Überlagerung mit dem ATTO565-Peak liegen könnte, da dieser zu kürzerer Wellenlänge verschoben zu sein scheint. 124
143 6.7 Ensemble Fluoreszenzspektroskopie relative Fluoreszenzintensität EGFP-YF 0 F 1 korrigiert ATTO565-EGFP-YF 0 F 1 korrigiert ATTO565-EGFP-YF 0 F 1 korrigiert + AMPPNP Wellenlänge / nm Abbildung 6.21: Normiertes Fluoreszenzemissionsspektrum der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1 nach Zugabe von AMPPNP. Vergleich des Fluoreszenzemissionsspektrums der mit AMPPNP (Endkonzentration 1 mm) gehemmten doppelt markierten Probe (cyan) und der nicht gehemmten doppelt markierten Probe (siehe auch Abbildung 6.20; magenta). Die Spektren wurden auf das Maximum des Emissionsspektrums von EGFP-YF 0 F 1 (siehe auch Abbildung 6.10; schwarz) normiert, um den Anteil der Fluoreszenz der Verunreinigung in der Probe korrigiert und auf den Wert der Fluoreszenzintensität bei 670 nm der EGFP-YF 0 F 1 -Probe gelegt. Die Anregung erfolgte bei 460 nm in HEPES-Puffer, die Emission wurde im Bereich von nm aufgezeichnet, die Konzentration von YF 0 F 1 betrug 0,31 μm, die Konzentration von ATTO565 0,165 μm und von EGFP 0,05 μm im Ansatz Diskussion Vergleicht man die Spektren in Abbildung 6.20 und berechnet den Unterschied der Fluoreszenzintensität im Bereich zwischen 575 und 625 nm, erhält man bei der doppelt markierten Probe ATTO565-EGFP-YF 0 F 1 für das gebundene ATTO565 eine um 46 % höhere Fluoreszenzintensität als durch die Direktanregung gemessen wird. Dies deutet auf einen funktionierenden FRET hin. In Abbildung 6.21 ist zu erkennen, dass sich durch die Zugabe von AMPPNP das Spektrum der doppelt markierten Probe im Vergleich zu der Messung ohne AMPPNP verändert. Die Fluoreszenzintensität von EGFP bleibt durch die Zugabe von AMPPNP gleich, die von ATTO
144 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae wird um 29 % kleiner verglichen zur doppelt markierten Probe ohne AMPPNP. Der ATTO565- Peak zeigt unter diesen Bedingungen eine Verbreiterung und Verschiebung zu niedrigerer Wellenlänge. Da kein antikorreliertes Verhalten vorliegt, kann nicht direkt darauf geschlossen werden, dass durch die Inhibierung mit AMPPNP das Enzym eine Konformation einnimmt, in der der Abstand zwischen Donor und Akzeptor größer ist, als das Mittel aller Abstände ohne Inhibitor, jedoch ändert sich der FRET. Unklar ist woher die Verschiebung zu kürzerer Wellenlänge kommt. Bei Integration über einen gleich großen Bereich unter Berücksichtigung der Verschiebung bei einer relativen Fluoreszenzintensität von 50 von Δλ = 13 nm erhält man nach der Integration über den Bereich von nm für die Fluoreszenzintensität der doppelt markierten Probe in Anwesenheit von AMPPNP einen um 7 % geringeren Wert als ohne AMPPNP. Das würde bedeuten, dass AMPPNP keinen großen Einfluss auf die Konformation der H + -ATPsynthase aus S. cerevisiae hat. 6.8 Einzelmolekül-FRET-Spektroskopie Die Einzelmolekül-Messungen (spfret, single-pair fluorescence resonance energy transfer) wurden an frei in Lösung diffundierenden H + -ATPsynthasen durchgeführt. Dazu wurden mit ATTO565 und EGFP doppelt markierte Enzyme, wie in Kapitel 3.7 beschrieben, in Liposomen rekonstituiert. Die Messungen wurden unter Hydrolysebedingungen wahlweise in Anwesenheit der Nukleotiden ATP oder AMPPNP oder nach der Zugabe von Azid und ADP gestartet (siehe Kapitel 4.3.2). Während der Messzeit diffundierten einzelne Proteoliposomen durch den Beobachtungsfokus, dabei wurde der Donorfluorophor EGFP angeregt. Die Anregung erfolgte mit dem Krypton-Ionen-Laser bei einer Wellenlänge von 476 nm. Es wurde nicht im Absorptionsmaximum des EGFPs bei 488 nm angereregt, da bei dieser Anregungswellenlänge das Maximum der Ramanstreuung des Wassers (3400 cm 1 Band der OH Schwingung) bei ca. 585 nm, im Bereich der Detektionswellenlänge des Akzeptors liegt [160] und dadurch ein hohes Hintergrundsignal gemessen wird. Durch die Anregung bei kürzerer Wellenlänge konnte das Hintergrundsignal reduziert werden. Befand sich während der Diffusion des Proteoliposoms durch den Beobachtungsfokus ein Akzeptorfluorophor in einem für FRET geeigneten Abstand zum Donorfluorophor, kam es zum Energietransfer Auswertung der Einzelmolekülmessungen Die Fluoreszenzintensitäten wurden, wie in Kapitel beschrieben, gemessen und mit dem Programm Burst Analyzer V 4.39 (Nawid Zarrabi, Universität Stuttgart) ausgewertet. 126
145 6.8 Einzelmolekül-FRET-Spektroskopie Korrektur der Fluoreszenzintensitäten Die gemessene Fluoreszenz musste aufgrund der Raman- und Rayleighstreuung, den fluoreszierenden Verunreinigungen in der Probe und eventuell im Puffer, sowie der Dunkelzählrate der APDs um diese Hintergrundfluoreszenz korrigiert werden. Dazu wurde bei jeder Messung (500 Spuren) in jeder 50-sten Spur ein Bereich ohne Burst ausgewählt und für diesen die Zählraten im Donor- sowie im Akzeptorkanal bestimmt, daraus der Mittelwert gebildet und diese Hintergrundzählrate von den Messspuren subtrahiert. Die Zahlenwerte lagen zwischen 1 und 2 khz, wobei der Wert im Donorkanal im allgemeinen etwas höher lag als der im Akzeptorkanal (siehe Abbildung 6.22) I D,I A /khz I D,I A /khz Zeit/ms Zeit/ms Abbildung 6.22: Korrektur der Messspuren um die Hintergrundfluoreszenz. Links: Fluoreszenzintensitäten im Donorkanal (gün) und im Akzeptorkanal (rot) bei Messung des Liposomenpuffers (siehe Kapitel 4.3.2). Rechts: Fluoreszenzintensitäten im Donor- und Akzeptorkanal nach der Korrektur um die Hintergrundfluoreszenz. Die Messspuren mussten des Weiteren um die im Akzeptorkanal detektierten Fluoreszenzphotonen des Donors korrigiert werden (Crosstalk). Die Höhe dieses Anteils hängt von den Spektren und den optischen Eigenschaften der Filter und Spiegel im Aufbau ab. Zur Bestimmung des Crosstalks wurden in 500 korrigierten Messspuren Photonenbursts ausgewählt, deren Fluoreszenz ausschließlich vom Donorfluorophor EGFP stammte (Donor-only) und die gemessene Fluoreszenz im Akzeptorkanal bestimmt. Der Mittelwert ergab, dass ein Anteil von 16 % der Fluoreszenz im Donorkanal im Akzeptorkanal detektiert wurde. Der Akzeptorkanal wurde um diesen Anteil korrigiert (siehe Abbildung 6.23). 127
146 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae I D,I A /khz I D,I A /khz Zeit/ms Zeit/ms Abbildung 6.23: Korrektur der Messspuren um den Crosstalk. Links: Donor-only- Signal vor und rechts nach der Crosstalkkorrektur. Bestimmung der FRET-Effizienz Das verwendete Programm Burst Analyzer V 4.39 errechnet aus den korrigierten Intensitäten für jeden Zeitpunkt der Messspuren die Proximity-Faktoren. P = I A I A + I D (6.8) Um aus diesen Werten die FRET-Effizienz zu bestimmen, müssen die Detektionseffizienzen des Donor- und Akzeptorkanals sowie die Fluoreszenzquantenausbeuten berücksichtigt werden. Diese sind durch den Korrekturfaktor γ aus Kapitel gegeben: E FRET = I A I A + γi D = P P + γ(1 P ) (6.9) E FRET FRET-Effizienz I D Fluoreszenzintensität Donor I A Fluoreszenzintensität Akzeptor γ Korrekturfaktor (siehe Kapitel 6.6.3) = 2,65 P Proximity-Faktor Aus den FRET-Effizienzen können dann mit Hilfe von Gleichung 1.4 und dem in Kapitel 6.5 bestimmten Försterradius von 55 Å die Abstände der Fluorophore berechnet werden. r AD = R 0 ( 1 E FRET 1 ) 1/6 128
147 6.8 Einzelmolekül-FRET-Spektroskopie Auswahlkriterien und Klassifizierung der Photonenbursts Hebt sich innerhalb einer Messspur die Fluoreszenzintensität des Donors und/oder des Akzeptors deutlich von dem Untergrundsignal ab, spricht man von einem Photonenburst. Um in der Auswertung möglichst ausschließlich Signale von einzelnen Molekülen im konfokalen Volumen zu berücksichtigen, wurden für die Auswahl der Bursts sowohl für die Zeitdauer als auch für die Fluoreszenzintensität Kriterien festgelegt. Ein Burst sollte eine Dauer zwischen 10 und 150 ms haben, kürzere Ereignisse rühren von freien Fluorophoren oder von nicht rekonstituierten, markierten Enzymen her. Bei Bursts von einer Länge von mehr als 150 ms kann davon ausgegangen werden, dass es sich entweder um ein Agglomerat von mehreren Liposomen handelt oder, dass das Liposom im Fokus zum Beispiel an der Oberfläche des Objektträgers immobilisiert ist. Des Weiteren wurden nur Bursts berücksichtigt, deren Photonenzahl nach Addition der Intensitäten von Donor- und Akzeptorkanal mindestens 150 betrug und deren maximale Zählrate 200 khz nicht überschritt. Zudem sollte die Fluoreszenzintensität während eines Bursts nicht häufiger als dreimal und nicht länger als 2 ms 5 khz parallel in beide Kanäle unterschreiten. Da, je nach Abstand der zwei Fluorophore zueinander, die FRET-Effizienz variiert, werden zur einfacheren Übersicht die Photonenbursts in verschiedene Klassen unterteilt. Die Einteilung ist in Tabelle 6.8 wiedergegeben. Tabelle 6.8: Klassifizierung der Photonenbursts. Einteilung der Photonenbursts anhand ihres Proximity-Faktors und nach Umrechnung in die FRET-Transfereffizienz, unter Berücksichtigung des Korrekturfaktors des Aufbaus γ = 2,65. Bezeichnung des Photonenbursts Proximity-Faktor FRET-Effizienz Donor-only 0-0,04 0-0,019 Low 0,05-0,39 0,02-0,19 Medium 0,4-0,69 0,2-0,46 High 0,7-1 0,47-1 Während der Dauer eines Photonenbursts kann die Transfereffizienz entweder konstant bleiben oder sich sprunghaft zwischen zwei Stufen ändern. Damit eine Stufe als eigener Zustand gewertet wurde, musste die Dauer mindestens 4 ms und der Unterschied des Proximity-Faktors zweier benachbarter FRET-Zustände mindestens 0,12 betragen. In Abbildung 6.24 ist je ein Beispiel für die Klassifizierung der Photonenbursts gezeigt. In Abbildung 6.25 sind Bursts mit sprunghafter Änderung der FRET-Effizienz dargestellt. 129
148 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae 1,0 FRET-Effizienz 0,5 0,0 60 Low Mittelwert: 0,07 I D,I A /khz ,0 FRET-Effizienz 0,5 0,0 Medium Mittelwert: 0, I D,I A /khz ,0 FRET-Effizienz 0,5 0,0 High Mittelwert: 0,71 60 I D,I A /khz Zeit / ms Abbildung 6.24: Exemplarische Bursts für die einzelnen Klassen. Gezeigt sind von oben nach unten Low, Medium, High. Jedes Diagramm zeigt die FRET-Effizienz (blau), den Mittelwert der FRET-Effizienz (schwarz), die Fluoreszenzintensität des Donors (grün) und des Akzeptors (rot). 130
149 6.8 Einzelmolekül-FRET-Spektroskopie FRET-Effizienz 1,0 0,5 0,0 80 L M H Mittelwert: 0,11 0,27 0,57 I D,I A /khz ,0 FRET-Effizienz 0,5 0,0 M L H Mittelwert: 0,33 0,09 0,61 40 I D,I A /khz Zeit / ms Abbildung 6.25: Exemplarische Bursts für Änderungen in der FRET- Effizienz. Oben: Änderung der Transfereffizienz mit der Sequenz Low (L) Medium (M) High (H), gezeigt mit den Werten 0,11-0,27-0,57. Unten: Änderung der Transfereffizienz mit der Sequenz Medium (M) Low (L) High (H), gezeigt mit den Werten 0,33-0,09-0,61. Die Diagramme zeigen die FRET-Effizienz (blau), den Mittelwert der FRET-Effizienz (schwarz), die Fluoreszenzintensität des Donors (grün) und des Akzeptors (rot). 131
150 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Messung unter Hydrolysebedingungen Die Messungen wurden wie in Kapitel beschrieben in Anwesenheit von 1 mm ATP durchgeführt. Die Anzahl der erhaltenen Bursts und deren Klassifizierung nach der Auswertung sind in Tabelle 6.12 wiedergegeben. Die Auswahl und Klassifizierung der einzelnen Stufen erfolgte nach den Kriterien aus Kapitel und anhand des Proximity-Faktors laut Tabelle 6.8. Diese Werte und wurden zunächst mit dem Programm OriginPro 9G in die Transfereffizienz und anschließend in die Abstände umgerechnet. Die Verteilungen der berechneten Abstände für konstante Photonenbursts bzw. für Photonenbursts bei denen sich die Transfereffizienz sprunghaft ändert sind in Abbildung 6.26 dargestellt. Durch eine Gauss-Anpassung der einzelnen Anzahl der Ereignisse Anzahl der Ereignisse Abstand einstufig / Å Abstand mehrstufig / Å Abbildung 6.26: Histogramme der Abstände zwischen dem Donorfluorophor EGFP und dem Akzeptorfluorophor ATTO565 unter Hydrolysebedingungen. Die Messungen wurden unter Zugabe von 1 mm ATP durchgeführt. Die Abstände wurden in Klassen der Breite 2 Å eingeteilt, die Gauss-Anpassung zur Bestimmung der mittleren Abstände (Ergebnisse siehe Tabelle 6.9) erfolgte mit OriginPro 9G. Links: Histogramm der Abstände der konstanten Bursts. Rechts: Histogramm der Abstände der Bursts mit sich ändernder Transfereffizienz. Populationen konnten für die konstanten Transfereffizienzen vier mittlere Abstände zwischen Donor- und Akzeptorfluorophor bestimmt werden. Für die sich während eines Photonenbursts sprunghaft ändernden Transfereffizienzen wurden drei Populationen ermittelt. Die Werte für die mittleren Abstände sind in Tabelle 6.9 wiedergegeben. Anhand der Einzelmolekülmessungen unter Hydrolysebedingungen konnte die mittlere Verweilzeit für einen FRET-Zustand bestimmt werden. Dazu wurden alle Ereignisse innerhalb dreioder mehrstufiger Bursts, deren Anfang und Ende aufgezeichnet wurde, in dem Histogramm in Abbildung 6.27 erfasst und die mittlere Verweilzeit mit einer monoexponentiellen Anpassung 132
151 6.8 Einzelmolekül-FRET-Spektroskopie Tabelle 6.9: Mittlere Abstände zwischen Donor-und Akzeptorfluorophor unter Hydrolysebedingungen. Die Abstände und ihr Standardfehler zwischen EGFP und ATTO565 wurden durch Gauss-Anpassung (siehe Abbildung 6.26) mit dem Programm OriginPro 9G bestimmt. Klassifizierung r DA /Å mittlerer Abstand mit konstanter Transfereffizienz mittlerer Abstande mit sich ändernder Transfereffizienz 44 ± 1 65 ± 4 77 ± 2 88 ± 5 54 ± 4 62 ± 2 71 ± 3 zu 16 ms bestimmt. Die Verweilzeit in einem Zustand stellt die Zeit dar, die von der H + -ATPsynthase benötigt wird um ein ATP zu hydrolysieren. Daraus ergibt sich für die Hydrolyseaktivität eines Enzyms ein turnover von 63 s Anzahl der Ereignisse Verweilzeit / ms Abbildung 6.27: Histogramm der Verweilzeiten in einem FRET-Zustand während der ATP Hydrolyse. Aus 101 Verweilzeiten (Balkenbreite 1 ms) wurde durch eine monoexponentielle Anpassung (blaue Kurve) die mittlere Verweildauer zu 16 ms bestimmt. Daraus ergibt sich eine Hydrolyseaktivität mit einem turnover von 63 s 1. Durch Bestimmung der mittleren Verweilzeit der konstanten Bursts, konnte die mittlere Aufenthaltsdauer eines Proteoliposoms im konfokalen Fokus bestimmt werden. Dazu wurden alle Verweilzeiten aus den Messungen unter Hydrolysebedingungen in dem Histogramm in 133
152 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Abbildung 6.28 erfasst und mit einer monoexponentiellen Anpassung die mittlere Verweilzeit zu 30 ms bestimmt. 100 Anzahl der Ereignisse Verweilzeit / ms Abbildung 6.28: Histogramm der Verweilzeiten in einem konstanten FRET- Zustand. Aus 1239 Verweilzeiten (Balkenbreite 1 ms) wurde durch eine monoexponentielle Anpassung (blaue Kurve) die mittlere Verweildauer zu 30 ms bestimmt. Dies entspricht der mittleren Aufenthaltsdauer eines Proteoliposoms im konfokalen Fokus FRET-Messungen unter nicht katalytischen Bedingungen (AMPPNP) Die Messungen wurden wie in Kapitel beschrieben in Anwesenheit von 0,3 mm AMPPNP durchgeführt. Das Nukleotid AMPPNP ist ein Analogon zu ATP, welches jedoch nicht hydrolysiert werden kann. Durch die Besetzung der katalytischen Bindungsplätze wird das Enzym kompetitiv inhibiert. Da somit keine Katalyse stattfinden kann, sollte der Abstand zwischen Donor und Akzeptor konstant bleiben und damit auch ausschließlich Bursts mit konstanter Transfereffizienz erhalten werden. Allerdings enthält eine Lösung von AMPPNP immer auch eine Verunreinigung mit ATP, sodass Bursts mit sich ändernder Transfereffizienz vorkommen können. Die Anzahl der erhaltenen Bursts und deren Klassifizierung nach der Auswertung sind in Tabelle 6.12 wiedergegeben. Die erhaltenen Verteilungen der Abstände für konstante Bursts, sowie Bursts mit sich änderender Transfereffizienz sind in Abbildung 6.29 gezeigt. 134
153 6.8 Einzelmolekül-FRET-Spektroskopie Anzahl der Ereignisse Anzahl der Ereignisse Abstand einstufig / Å Abstand mehrstufig / Å Abbildung 6.29: Histogramme der Abstände zwischen dem Donorfluorophor EGFP und dem Akzeptorfluorophor ATTO565 unter nicht katalytischen Bedingungen. Die Messungen wurden unter Zugabe von 0,3 mm AMPPNP durchgeführt. Die Abstände wurden in Klassen der Breite 2 Å eingeteilt und eine Gauss-Anpassung zur Bestimmung der mittleren Abstände (Ergebnisse siehe Tabelle 6.10) durchgeführt. Links: Histogramm der Abstände der konstanten Bursts. Rechts: Histogramm der Abstände der Bursts mit sich ändernder Transfereffizienz. Die Gauss-Anpassung ergab für konstante Bursts vier verschiedene Abstände, für zwei- und mehrstufige Bursts wurden drei Abstände ermittelt. Die Werte sind in Tabelle 6.10 wiedergegeben. Tabelle 6.10: Mittlere Abstände zwischen Donor-und Akzeptorfluorophor unter nicht katalytischen Bedingungen. Die Abstände und ihr Standardfehler zwischen EGFP und ATTO565 wurden durch Gauss-Anpassung (siehe Abbildung 6.29) bestimmt. Klassifizierung r DA /Å mittlerer Abstand mit konstanter Transfereffizienz mittlerer Abstand mit sich ändernder Transfereffizienz 48 ± 3 56 ± 1 63 ± ± 3 90 ± ± 1 63 ± 1 74 ± 1 135
154 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae FRET-Messungen an inhibierten H + -ATPsynthasen (NaN 3 und ADP) Eine weitere Möglichkeit der Inhibierung der H + -ATPsynthase ist die Inkubation mit Azid und ADP. Das Azid-Anion wechselwirkt mit dem β-phosphat des ADP und mit für die Katalyse wichtigen Seitenketten in der ADP bindenden katalytischen Untereinheit β des Enzyms und inhibiert dadurch die ATP-Hydrolyse aber nicht die Synthese [27, 136]. Die Messungen wurden wie in Kapitel beschrieben in Anwesenheit von 10 μm NaN 3 und 100 μm ADP durchgeführt. Die Anzahl der erhaltenen Bursts und deren Klassifizierung nach der Auswertung sind in Tabelle 6.12 wiedergegeben. Die erhaltenen Verteilungen der Abstände für konstante Bursts, sowie Bursts mit sich änderender Transfereffizienz sind in Abbildung 6.30 gezeigt Anzahl der Ereignisse Anzahl der Ereignisse Abstand einstufig / Å Abstand mehrstufig / Å Abbildung 6.30: Histogramme der Abstände zwischen dem Donorfluorophor EGFP und dem Akzeptorfluorophor ATTO565 in inhibierten H + - ATPsynthasen. Die Messungen wurden unter Zugabe von 10 μm NaN 3 und 100 μm ADP durchgeführt. Die Abstände wurden in Klassen der Breite 2 Å eingeteilt und eine Gauss-Anpassung zur Bestimmung der mittleren Abstände (Ergebnisse siehe Tabelle 6.11) durchgeführt. Links: Histogramm der Abstände der konstanten Bursts. Rechts: Histogramm der Abstände der Bursts mit sich ändernder Transfereffizienz. Die Gauss-Anpassung ergab für konstante Bursts drei verschiedene Abstände, für zwei- und mehrstufige Bursts wurden fünf Abstände ermittelt. Die Werte sind in Tabelle 6.10 wiedergegeben. 136
155 6.8 Einzelmolekül-FRET-Spektroskopie Tabelle 6.11: Mittlere Abstände zwischen Donor-und Akzeptorfluorophor bei inhibierten H + -ATPsynthasen. Die Abstände und ihr Standardfehler zwischen EGFP und ATTO565 wurden durch Gauss-Anpassung (siehe Abbildung 6.30) bestimmt. Klassifizierung r DA /Å mittlerer Abstand mit konstanter Transfereffizienz mittlerer Abstand mit sich ändernder Transfereffizienz 46 ± 1 77 ± 2 95 ± ± 1 62 ± 1 71 ± 1 80 ± 1 89 ± 2 Tabelle 6.12: Klassifizierung der erhaltenen Bursts. Anzahl der Ereignisse in den ausgewerteten Messspuren und Unterscheidung der Bursts in konstante und sich ändernde Transfereffizienz (grau unterlegt). Die sich ändernde Transfereffizienz wurde weiter in die Anzahl der erhaltenen FRET-Zustände und in die Reihung der Sequenz unterschieden. Klassifizierung ATP AMPPNP NaN 3 + ADP Anzahl ausgewerteter Spuren Gesamtanzahl der Ereignisse Bursts mit konstanter Transfereffizienz 1232 (62 %) 368 (75 %) 333 (65 %) Bursts mit sich ändernder Transfereffizienz 767 (38 %) 122 (25 %) 179 (35 %) davon sind: Bursts mit zwei FRET- Zuständen Bursts mit drei oder mehr FRET- Zuständen Änderung der Transfereffizienz mit der Sequenz L M H Änderung der Transfereffizienz mit der Sequenz H M L Änderung der Transfereffizienz mit anderer Sequenz 472 (24 %) 74 (15 %) 114 (22 %) 295 (14 %) 48 (10 %) 65 (13 %) 386 (19 %) 70 (14 %) 101 (20 %) 231 (11,5 %) 30 (6 %) 55 (11 %) 150 (7,5 %) 22 (4 %) 23 (4 %) 137
156 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae In den folgenden Betrachtungen wurde zwischen aktiven und inaktiven Enzymen unterschieden. Dabei wurde davon ausgegangen, dass in aktiven Enzymen eine schrittweise Bewegung zu beobachten ist, während in inaktiven Enzymen keine Bewegung auftritt. Dementsprechend zeigen Bursts mit konstanter Transfereffizienz inaktive, Bursts mit sich ändernder Transfereffizienz aktive Enzyme an. 6.9 Vergleich der FRET-Daten mit einem Homologiemodell Zu Beginn der Arbeit wurde davon ausgegangen, dass das EGFP aufgrund des relativ starren Linkers entlang der Molekülachse der γ-untereinheit orientiert ist, da die genaue Position der veränderten Aminosäure in der b-untereinheit, sowie die genaue Lage des OSCP und der b- Untereinheit zum α 3 β 3 -Hexamer unklar war. Das entsprechende Homologiemodell wurde von J. Petersen erstellt [105] und ist in Abbildung 6.31 links gezeigt. Abbildung 6.31: Homologiemodelle des YF 0 F 1 -EGFP-Fusionsproteins. Links: Homologiemodell aus der Arbeit von J. Petersen. [105] Rechts: Auf Basis der FRET-Messungen von J. Petersen neu erstelltes Homologiemodell inklusive der Positionierung des Akzeptorfluorophors ATTO565 (magenta). 138
157 6.9 Vergleich der FRET-Daten mit einem Homologiemodell Die Ergebnisse der Einzelmolekülmessungen zeigen jedoch, dass die Abstände zwischen dem EGFP und ATTO565 an b-k174c signifikant geringer sind, als nach diesem Modell erwartet wird. Die Struktur des Zip-Helix-EGFP-Konstrukts sollte danach deutlich abgewinkelter und damit näher zur Enzymoberfläche, bzw. der b-untereinheit positioniert sein. Auf Basis dieser Daten und der neuen zusätzlichen Strukturdaten der Arbeitsgruppen J. Walker [161] und M.- F. Giraud [162] wurde von J. Petersen ein neues Modell wie folgt erstellt: Es wurde die neue F 1 - Struktur inklusive der b- und der OSCP-Untereinheit [pdb 2wss] über die Struktur von F 0 F 1 aus S. cerevisiae [pdb 2wpd] gelegt, sodass die Überlappung der β-untereinheiten maximal wurde. Anschließend wurden die Koordinaten von b, OSCP und Teile von d in die Datei übernommen. Die Zip-Helix wurde mit dem Programm Coot auf Basis einer Doppelhelix generiert und in eine Geometrie gezwungen, in der Überlappungen mit der Struktur vermieden wurden, sodass die Zipper-Doppelhelix an der OSCP-Untereinheit vorbei passte. Alle nachfolgenden Bilder wurden anhand dieses neuen Modells von J. Petersen erstellt. Im Folgenden sind die α-untereinheiten in grau, die β-untereinheiten in gelb, die γ-untereinheit inklusive der Zipper-Doppelhelix in blau und die b-untereinheit zusammen mit der OSCP-Untereinheit in rot dargestellt. Wie in Abbildung 6.32 zu erkennen ist, läßt die Position des OSCPs nur einen halbmondförmigen Spalt oberhalb des Hohlraums des Hexamers offen. Damit ist die freie Beweglichkeit des EGFPs deutlich eingeschränkt. Bei inaktiven H + -ATPsynthasen sollten entsprechend entlang des Spalts vor allem zwei verschiedene Abstände auftreten. Abbildung 6.32: Homologiemodell für die Positionierung des Zip-Helix-EGFP- Konstrukts unter Berücksichtigung der Untereinheit OSCP. Links: α 3 β 3 -Hexamer. Mitte: α 3 β 3 -Hexamer, OSCP und b-untereinheit. Rechts: F 1 -Teil und mögliche Orientierung des an der γ-untereinheit fusionierten EGFPs. Die α-untereinheiten sind in grau, die β-untereinheiten in gelb, die γ-untereinheit inklusive der Zipper-Doppelhelix in blau und die b- Untereinheit zusammen mit der OSCP-Untereinheit in rot dargestellt. 139
158 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Betrachtet man die Verteilungen in den Histogrammen für die konstanten FRET-Effizienzen (inaktive Enzyme), könnten diese auch als zwei Populationen interpretiert werden. Bei den Histogrammen der H + -ATPsynthasen unter Hydrolysebedingungen mit ATP (Abbildung 6.26) und der mit Azid inhibierten H + -ATPsynthase (Abbildung 6.29) ist dies sehr deutlich zu erkennen. In dem Histogramm der mit AMPPNP inhibierten H + -ATPsynthasen (Abbildung 6.30) können unter Vernachlässigung der Population bei 63 Å ebenfalls zwei Populationen durch eine Gauss-Anpassung ermittelt werden. Die neu berechneten Gauss-Kurven sind in Abbildung 6.33 dargestellt und die aus den Maxima bestimmten mittleren Abstände sind in Tabelle 6.13 aufgeführt. Die zusätzliche Population im Histogramm mit AMPPNP könnte durch eine Überlagerung mit aktiven Enzymen zustande kommen Anzahl der Ereignisse Anzahl der Ereignisse Abstand einstufig / Å Abstand einstufig / Å 30 Anzahl der Ereignisse Abstand einstufig / Å Abbildung 6.33: Gauss-Anpassung der Abstände bei konstanten Bursts unter Annahme von zwei Populationen. Oben-links: Histogramm der Abstände unter Hydrolysebedingungen. Oben-rechts: Histogramm der Abstände an mit NaN 3 + ADP inhibierten H + -ATPsynthasen. Unten: Histogramm der Abstände an mit AMPPNP inhibierten H + -ATPsynthasen. 140
159 6.9 Vergleich der FRET-Daten mit einem Homologiemodell Tabelle 6.13: Mittlere Abstände bei inaktiven H + -ATPsynthasen unter Annahme von zwei Populationen. Die Abstände wurden durch eine Gauss-Anpassung der Histogramme für konstante Bursts bestimmt. Zum Vergleich sind die aus dem Homologiemodell A (siehe Abbildung 6.34) gewonnenen Abstände angegeben. r DA /Å ATP 44 ± 1 76 ± 1 AMPPNP 49 ± 1 77 ± 1 NaN 3 + ADP 46 ± 1 80 ± 2 Homologiemodell A Abbildung 6.34 zeigt die beiden Positionen des EGFPs in einer Seitenansicht und einer Aufsicht. Abbildung 6.34: Homologiemodell A für inaktive H + -ATPsynthasen. Das Homologiemodell zeigt zwei Positionen des EGFPs, bei denen der Donor- Akzeptorabstand jeweils 45 Å bzw. 96 Å hat. Links: Seitenansicht. Die Position des Akzeptors ATTO565 an der Position b-174 wurde durch den magenta eingefärbten Kreis angedeutet. Rechts: Ansicht von oben. Bei aktiven Enzymen erwartet man auf der Grundlage des Rotationsmodells drei Abstände, die den Drehungen der γ-untereinheit von jeweils 120 entsprechen. Tatsächlich findet man auch bei der ATP-Hydrolyse drei verschiedene Abstände (siehe Abbildung 6.26 rechts). Die drei Positionen sind in Abbildung 6.35 dargestellt und die aus den FRET-Experimenten erhaltenen Abstände sind in Tabelle 6.14 aufgeführt. Überraschenderweise wurden auch in Gegenwart von 141
160 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae AMPPNP und in Gegenwart von Azid und ADP mehrstufige Bursts gefunden. Vermutlich deutet dies auf eine nicht vollständige Inhibierung des Enzyms und die Anwesenheit geringer ATP-Verunreinigungen in AMPPNP bzw. ADP hin. Tabelle 6.14: Mittlere Abstände bei aktiven H + -ATPsynthasen. Die Abstände wurden durch eine Gauss-Anpassung der Histogramme für Bursts mit sich ändernder Transfereffizienz bestimmt. Zum Vergleich sind die aus dem Homologiemodell A (siehe Abbildung 6.35) gewonnenen Abstände angegeben. r DA /Å ATP 54 ± 4 62 ± 2 71 ± 3 AMPPNP 55 ± 1 63 ± 1 74 ± 1 NaN 3 + ADP 54 ± 1 62 ± 1 71 ± 1 Homologiemodell A Abbildung 6.35: Homologiemodell A für aktive H + -ATPsynthasen. Das Homologiemodell zeigt zwei Positionen entlang der durch die Untereinheit OSCP halbmondförmigen Öffnung mit einem Abstand von 45 Å und 79 Å zwischen Donor und Akzeptor, sowie einem weiteren Abstand mit 96 Å an der von OSCP abgewandten Seite. Links: Seitenansicht. Rechts: Ansicht von oben. Beim Vergleich der Abstände der aktiven Enzyme mit den beiden Abständen der inaktiven Enzyme (siehe Tabelle 6.13) fällt auf, dass der kleinste Abstand bei den aktiven Enzymen sich um etwa 8 Å und der größte Abstand sich um etwa 6 Å vergrößert hat. Dies könnte daher rühren, dass bei der Rotationsbewegung während der Katalyse das EGFP sich weiter aufrichtet und 142
161 6.9 Vergleich der FRET-Daten mit einem Homologiemodell der Winkel zwischen dem Linker und dem EGFP größer wird. Dies würde zu einem größeren Abstand zwischen EGFP und ATTO565 führen. Bei dem hier angewendeten Modell (Homologiemodell A) wird die Konformationsänderung der α/β-untereinheiten während der Katalyse nicht berücksichtigt. Die Abstände wurden durch die Rotation der γ-untereinheit in 120 -Schritten ermittelt. In Wirklichkeit bewirkt aber die Rotation der γ-untereinheit, dass sich die Konformation aller α/β-untereinheiten ändert. Möchte man die Abstände bestimmen, die bei diesen Konformationsänderungen auftreten, kann man dies geometrisch so machen, dass man die b-untereinheit mit dem ATTO565 an die verschiedenen α/β-untereinheiten anlegt und die Abstände zum festgehaltenen EGFP- Fluorophor bestimmt (Homologiemodell B). In Abbildung 6.36 sind die beiden Modelle gezeigt und in Tabelle 6.15 die daraus resultierenden Abstände einander gegenüber gestellt. Es wird jeweils der kleinste, der mittlere und der größte Abstand zwischen dem EGFP-Fluorophor und ATTO565 in beiden Modellen bestimmt. Abbildung 6.36: Vergleich der beiden Modellvorstellungen. Links: Homologiemodell A, Rotation der γ-untereinheit in 120 -Schritten. Rechts: Homologiemodell B, zur Berücksichtigung der Konformationsänderungen im α 3 β 3 -Hexamer während der Katalyse. Tabelle 6.15: Vergleich der Donor-Akzeptor-Abstände aus den beiden Modellen. Homologiemodell A: Abstände durch Rotation der γ-untereinheit, wenn sich die Konformationen von α/β nicht ändern; Homologiemodell B: Bestimmung der Abstände unter Berücksichtigung der geänderten α/β-konformationen. r DA /Å Homologiemodell A Homologiemodell B
162 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae Die nach beiden Methoden bestimmten Abstände sind sehr ähnlich, unterscheiden sich jedoch von den experimentell gefundenen Abständen (siehe Tabelle 6.14). Betrachtet man jeweils die Differenzen der Abstände im Modell und Experiment so zeigen diese eine Korrelation. Die Unterschiede könnten daher kommen, dass der Winkel zwischen dem Linker und dem EGFP noch nicht richtig gewählt wurde. Vergleichbar wie bereits in Abbildung 6.31 durch Wahl eines geringeren Winkels zwischen dem Linker und EGFP andere Abständen erreicht wurden. Die experimentell ermittelten Daten zeigen eine breite Verteilung der Abstände. Aus dem Homologiemodell geht hervor, dass es während der Rotation zu einer Überlappumg der γ-untereinheit und dem OSCP kommt. Dies könnte zu variablen Positionen, eventuell sogar zur partiellen Entfaltung von γ-egfp führen und ein Grund für die breite Abstandsverteilung sein. Die Auswertung der Sequenz der Transfereffizienzen zeigt für alle drei gemessenen Bedingungen % der Bursts mit einer Reihung Low Medium High, % der Bursts mit einer Reihung High Medium Low und % andere Bursts, wobei andere Bursts Stufenbursts sind, bei denen sich die Transfereffizienz zwischen zwei Stufen hin und her bewegt. Die mittlere Aufenthaltsdauer im konfokalen Fokus war 30 ms. Die mittlere Verweilzeit in einem FRET-Zustand war 16 ms. Dies ist der Grund, dass hauptsächlich zweistufige und nur wenige mehrstufige Bursts beobachtet wurden Ausblick In dieser Arbeit wurde gezeigt, dass die Isolation von aktiven mit EGFP fusionierten H + - ATPsynthasen möglich ist. Diese konnten erfolgreich mit dem organischen Fluorophor ATTO565 der Firma ATTO-TEC markiert und so mit Hilfe von Einzelmolekül-FRET-Spektroskopie Änderungen der FRET-Effizienz bestimmt werden. Aus diesen Transfereffizienzen und durch den Vergleich mit einem Homologiemodell konnten drei Abstände bestimmt werden, die durch eine Rotationsbewegung der γ-egfp-untereinheit erklärt werden können. Da die Messungen unter katalytischen Bedingungen ausschließlich für die Hydrolyse durchgeführt wurden, sollte eine Richtung der Sequenz der Transfereffizienz bevorzugt gemessen werden. Dies war aber nur bedingt der Fall. Durch Messungen unter ATP-Synthesebedingungen könnte diese Frage genauer geklärt werden, da die Rotation unter Synthesebedingungen im Gegensatz zu der Rotation unter Hydrolysebedingungen nicht entkoppelt ist. Das Vorkommen von beiden Rotationsrichtungen in den vorliegenden Messungen könnte auch an einer unvollständigen Solubilisierung der H + -ATPsynthasen liegen. Anhand des SDS-Gels ist 144
163 6.10 Ausblick erkennbar, dass die Probe noch weitere Verunreinigungen enthielt. Eventuell wurden analog zu den in Anhang C hergestellten und rekonstituierten SMPs aus Rinderherz Superkomplexe oder Teile davon solubilisiert und in die Liposomen rekonstituiert. Die gefundene Fluoreszenz der Verunreinigung in der Probe könnte von mit den H + -ATPsynthasen zusammen solubilisierten Cytochromen herstammen. Aufgrund dessen sollte die Solubilisierung und die Trennung der H + -ATPsynthasen aus S. cerevisiae für die Fusionsproteine verbessert werden. Durch den im Zip-Helix-FP-Konstrukt integrierten Strep-tag II könnte dies durch eine entsprechende Affinitätsreinigung versucht werden. Die Messungen wurden an durch den Fokus des konfokalen Mikroskops diffundierenden Liposomen durchgeführt. Die Verweildauer im Fokus war dabei gering und es wurden nur wenige Bursts mit mehr als drei Änderungen in der Transfereffizienz gemessen. Durch Vergrößerung des konfokalen Volumens oder durch die Immobilisierung der Proteoliposomen an einer Glasoder Lipidoberfläche, bzw. durch Immobilisierung mit Hilfe einer optischen Falle könnten die Beobachtungszeiten verlängert und mehr aufeinander folgende Zustände beobachtet werden. Allerdings muss dabei das Bleichverhalten des EGFPs berücksichtigt werden. 145
164 6 Die H + -ATPsynthase aus Saccharomyces cerevisiae 146
165 7 Literaturverzeichnis [1] Peter Mitchell and Jennifer Moyle. Chemiosmotic Hypothesis of Oxidative Phosphorylation. Nature, 213(5072): , Jan [2] S. Karrasch and J. E. Walker. Novel features in the structure of bovine ATP synthase. J Mol Biol, 290(2): , Jul [3] S. Wilkens and R. A. Capaldi. Electron microscopic evidence of two stalks linking the F 1 and F 0 parts of the Escherichia coli ATP synthase. Biochim Biophys Acta, 1365(1-2):93 97, Jun [4] B. Böttcher and P. Gräber. The structure of the H + -ATP synthase from chloroplasts and its subcomplexes as revealed by electron microscopy. Biochim Biophys Acta, 1458(2-3): , May [5] P. D. Boyer. Catalytic site forms and controls in ATP synthase catalysis. Biochim Biophys Acta, 1458(2-3): , May [6] Joachim Weber and Alan E. Senior. ATP synthesis driven by proton transport in F 1 F 0 - ATP synthase. FEBS Lett, 545(1):61 70, Jun [7] J. P. Abrahams, A. G. Leslie, R. Lutter, and J. E. Walker. Structure at 2.8 Å resolution of F 1 -ATPase from bovine heart mitochondria. Nature, 370(6491): , Aug [8] Ian N. Watt, Martin G. Montgomery, Michael J. Runswick, Andrew G W. Leslie, and John E. Walker. Bioenergetic cost of making an adenosine triphosphate molecule in animal mitochondria. Proc Natl Acad Sci U S A, 107(39): , Sep [9] Britta Ballhausen, Karlheinz Altendorf, and Gabriele Deckers-Hebestreit. Constant c 10 ring stoichiometry in the Escherichia coli ATP synthase analyzed by cross-linking. J Bacteriol, 191(7): , Apr [10] Jindrich Symersky, Vijayakanth Pagadala, Daniel Osowski, Alexander Krah, Thomas Meier, José D. Faraldo-Gómez, and David M. Mueller. Structure of the c 10 ring of the yeast mitochondrial ATP synthase in the open conformation. Nat Struct Mol Biol, 19(5):485 91, S1, May
166 7 Literaturverzeichnis [11] Thomas Meier, Jinshu Yu, Thomas Raschle, Fabienne Henzen, Peter Dimroth, and Daniel J. Muller. Structural evidence for a constant c 11 ring stoichiometry in the sodium F-ATP synthase. FEBS J, 272(21): , Nov [12] Wilson C Y. Lau and John L. Rubinstein. Subnanometre-resolution structure of the intact Thermus thermophilus H + -driven ATP synthase. Nature, 481(7380): , Jan [13] H. Seelert, A. Poetsch, N. A. Dencher, A. Engel, H. Stahlberg, and D. J. Müller. Structural biology. Proton-powered turbine of a plant motor. Nature, 405(6785): , May [14] M. E. Girvin and R. H. Fillingame. Hairpin folding of subunit c of F 1 F 0 ATP synthase: 1H distance measurements to nitroxide-derivatized aspartyl-61. Biochemistry, 33(3): , Jan [15] M. E. Girvin, V. K. Rastogi, F. Abildgaard, J. L. Markley, and R. H. Fillingame. Solution structure of the transmembrane H + -transporting subunit c of the F 1 F 0 ATP synthase. Biochemistry, 37(25): , Jun [16] Thomas Meier, Patrick Polzer, Kay Diederichs, Wolfram Welte, and Peter Dimroth. Structure of the rotor ring of F-Type Na + -ATPase from Ilyobacter tartaricus. Science, 308(5722): , Apr [17] Takeshi Murata, Ichiro Yamato, Yoshimi Kakinuma, Andrew G W. Leslie, and John E. Walker. Structure of the rotor of the V-Type Na + -ATPase from Enterococcus hirae. Science, 308(5722): , Apr [18] Oleg Y. Dmitriev and Robert H. Fillingame. The rigid connecting loop stabilizes hairpin folding of the two helices of the ATP synthase subunit c. Protein Sci, 16(10): , Oct [19] Jan Petersen, Kathrin Förster, Paola Turina, and Peter Gräber. Comparison of the H + /ATP ratios of the H + -ATP synthases from yeast and from chloroplast. Proc Natl Acad Sci U S A, 109(28): , Jul [20] R. K. Nakamoto, C. J. Ketchum, and M. K. al Shawi. Rotational coupling in the F 0 F 1 ATP synthase. Annu Rev Biophys Biomol Struct, 28: , [21] D. Stock, A. G. Leslie, and J. E. Walker. Molecular architecture of the rotary motor in ATP synthase. Science, 286(5445): , Nov
167 7 Literaturverzeichnis [22] R. A. Capaldi, B. Schulenberg, J. Murray, and R. Aggeler. Cross-linking and electron microscopy studies of the structure and functioning of the Escherichia coli ATP synthase. J Exp Biol, 203(Pt 1):29 33, Jan [23] Kristi S. Wood and Stanley D. Dunn. Role of the asymmetry of the homodimeric b 2 stator stalk in the interaction with the F 1 sector of Escherichia coli ATP synthase. J Biol Chem, 282(44): , Nov [24] Boris A. Feniouk and Wolfgang Junge. Regulation of the F 0 F 1 -ATP synthase: the conformation of subunit epsilon might be determined by directionality of subunit gamma rotation. FEBS Lett, 579(23): , Sep [25] J. Weber and A. E. Senior. Catalytic mechanism of F 1 -ATPase. Biochim Biophys Acta, 1319(1):19 58, Mar [26] P. D. Boyer. The ATP synthase a splendid molecular machine. Annu Rev Biochem, 66: , [27] D. Bald, T. Amano, E. Muneyuki, B. Pitard, J. L. Rigaud, J. Kruip, T. Hisabori, M. Yoshida, and M. Shibata. ATP synthesis by F 0 F 1 -ATP synthase independent of noncatalytic nucleotide binding sites and insensitive to azide inhibition. J Biol Chem, 273(2): , Jan [28] Verena Catherine Rombach-Riegraf. Untersuchung des F 0 -Motors der H + -ATPsynthase aus Escherichia coli mit Einzelmolekül-Fluoreszenzspektroskopie. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [29] W. Jiang, J. Hermolin, and R. H. Fillingame. The preferred stoichiometry of c subunits in the rotary motor sector of Escherichia coli ATP synthase is 10. Proc Natl Acad Sci U SA, 98(9): , Apr [30] Robert H. Fillingame, Christine M. Angevine, and Oleg Y. Dmitriev. Coupling proton movements to c-ring rotation in F 1 F 0 ATP synthase: aqueous access channels and helix rotations at the a-c interface. Biochim Biophys Acta, 1555(1-3):29 36, Sep [31] S. B. Vik, J. C. Long, T. Wada, and D. Zhang. A model for the structure of subunit a of the Escherichia coli ATP synthase and its role in proton translocation. Biochim Biophys Acta, 1458(2-3): , May [32] F. I. Valiyaveetil and R. H. Fillingame. Transmembrane topography of subunit a in the Escherichia coli F 1 F 0 ATP synthase. J Biol Chem, 273(26): , Jun
168 7 Literaturverzeichnis [33] L. P. Hatch, G. B. Cox, and S. M. Howitt. The essential arginine residue at position 210 in the alpha subunit of the Escherichia coli ATP synthase can be transferred to position 252 with partial retention of activity. J Biol Chem, 270(49): , Dec [34] F. I. Valiyaveetil and R. H. Fillingame. On the role of Arg-210 and Glu-219 of subunit a in proton translocation by the Escherichia coli F 0 F 1 -ATP synthase. J Biol Chem, 272(51): , Dec [35] O. Dmitriev, P. C. Jones, W. Jiang, and R. H. Fillingame. Structure of the membrane domain of subunit b of the Escherichia coli F 0 F 1 ATP synthase. J Biol Chem, 274(22): , May [36] Monika G. Düser, Yumin Bi, Nawid Zarrabi, Stanley D. Dunn, and Michael Börsch. The proton-translocating a subunit of F 0 F 1 -ATP synthase is allocated asymmetrically to the peripheral stalk. J Biol Chem, 283(48): , Nov [37] Paul A. Del Rizzo, Yumin Bi, Stanley D. Dunn, and Brian H. Shilton. The second stalk of Escherichia coli ATP synthase: structure of the isolated dimerization domain. Biochemistry, 41(21): , May [38] Tammy Bohannon Grabar and Brian D. Cain. Integration of b subunits of unequal lengths into F 1 F 0 -ATP synthase. J Biol Chem, 278(37): , Sep [39] Stefan Steigmiller, Michael Börsch, Peter Gräber, and Martina Huber. Distances between the b-subunits in the tether domain of F 0 F 1 -ATP synthase from E. coli. Biochim Biophys Acta, 1708(2): , Jun [40] Stephan Wilkens, Dan Borchardt, Joachim Weber, and Alan E. Senior. Structural characterization of the interaction of the delta and alpha subunits of the Escherichia coli F 1 F 0 -ATP synthase by NMR spectroscopy. Biochemistry, 44(35): , Sep [41] S. D. Dunn and J. Chandler. Characterization of a b 2 δ complex from Escherichia coli ATP synthase. J Biol Chem, 273(15): , Apr [42] T. M. Duncan, V. V. Bulygin, Y. Zhou, M. L. Hutcheon, and R. L. Cross. Rotation of subunits during catalysis by Escherichia coli F 1 -ATPase. Proc Natl Acad Sci U S A, 92(24): , Nov [43] D. Sabbert, S. Engelbrecht, and W. Junge. Functional and idling rotatory motion within F 1 -ATPase. Proc Natl Acad Sci U S A, 94(9): , Apr
169 7 Literaturverzeichnis [44] S. P. Tsunoda, A. J. Rodgers, R. Aggeler, M. C. Wilce, M. Yoshida, and R. A. Capaldi. Large conformational changes of the epsilon subunit in the bacterial F 1 F 0 ATP synthase provide a ratchet action to regulate this rotary motor enzyme. Proc Natl Acad Sci U S A, 98(12): , Jun [45] S. Wilkens and R. A. Capaldi. Solution structure of the epsilon subunit of the F 1 -ATPase from Escherichia coli and interactions of this subunit with beta subunits in the complex. J Biol Chem, 273(41): , Oct [46] Ryota Iino, Tomoe Murakami, Satoshi Iizuka, Yasuyuki Kato-Yamada, Toshiharu Suzuki, and Masasuke Yoshida. Real-time Monitoring of Conformational Dynamics of the ɛ Subunit in F 1 -ATPase. Journal of Biological Chemistry, 280(48): , [47] A. J. Rodgers and M. C. Wilce. Structure of the gamma-epsilon complex of ATP synthase. Nat Struct Biol, 7(11): , Nov [48] Gino Cingolani and Thomas M. Duncan. Structure of the ATP synthase catalytic complex (F 1 ) from Escherichia coli in an autoinhibited conformation. Nat Struct Mol Biol, 18(6): , Jun [49] Bruce Alberts. Molekularbiologie der Zelle 1. Auf. Weinheim. VCH, [50] Lajos Pikó and Lloyd Matsumoto. Number of mitochondria and some properties of mitochondrial DNA in the mouse egg. Developmental Biology, 49(1):1 10, Mar [51] M. W. Gray, G. Burger, and B. F. Lang. Mitochondrial evolution. Science, 283(5407): , Mar [52] T Martin Embley and William Martin. Eukaryotic evolution, changes and challenges. Nature, 440(7084): , Mar [53] William Martin and Michael J. Russell. On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells. Philos Trans R Soc Lond B Biol Sci, 358(1429):59 83; discussion 83 5, Jan [54] William Wickner and Randy Schekman. Protein translocation across biological membranes. Science, 310(5753): , Dec [55] J. Velours and G. Arselin. The Saccharomyces cerevisiae ATP synthase. J Bioenerg Biomembr, 32(4): , Aug
170 7 Literaturverzeichnis [56] R. J. Devenish, M. Prescott, X. Roucou, and P. Nagley. Insights into ATP synthase assembly and function through the molecular genetic manipulation of subunits of the yeast mitochondrial enzyme complex. Biochim Biophys Acta, 1458(2-3): , May [57] I. Arnold, K. Pfeiffer, W. Neupert, R. A. Stuart, and H. Schägger. Yeast mitochondrial F 1 F 0 -ATP synthase exists as a dimer: identification of three dimer-specific subunits. EMBO J, 17(24): , Dec [58] Patrick Paumard, Jacques Vaillier, Bénédicte Coulary, Jacques Schaeffer, Vincent Soubannier, David M. Mueller, Daniel Brèthes, Jean-Paul di Rago, and Jean Velours. The ATP synthase is involved in generating mitochondrial cristae morphology. EMBO J, 21(3): , Feb [59] Marie-France Giraud, Patrick Paumard, Vincent Soubannier, Jacques Vaillier, Geneviève Arselin, Bénédicte Salin, Jacques Schaeffer, Daniel Brèthes, Jean-Paul di Rago, and Jean Velours. Is there a relationship between the supramolecular organization of the mitochondrial ATP synthase and the formation of cristae? Biochim Biophys Acta, 1555(1-3): , Sep [60] Natalya V. Dudkina, Jesco Heinemeyer, Wilko Keegstra, Egbert J. Boekema, and Hans-Peter Braun. Structure of dimeric ATP synthase from mitochondria: an angular association of monomers induces the strong curvature of the inner membrane. FEBS Lett, 579(25): , Oct [61] Vincent Soubannier, Jacques Vaillier, Patrick Paumard, Benedicte Coulary, Jacques Schaeffer, and Jean Velours. In the absence of the first membrane-spanning segment of subunit 4(b), the yeast ATP synthase is functional but does not dimerize or oligomerize. J Biol Chem, 277(12): , Mar [62] Paul D. Gavin, Mark Prescott, Susan E. Luff, and Rodney J. Devenish. Cross-linking ATP synthase complexes in vivo eliminates mitochondrial cristae. J Cell Sci, 117(Pt 11): , May [63] R. G. Hadikusumo, S. Meltzer, W. M. Choo, M. J. Jean-François, A. W. Linnane, and S. Marzuki. The definition of mitochondrial H + ATPase assembly defects in mit- mutants of Saccharomyces cerevisiae with a monoclonal antibody to the enzyme complex as an assembly probe. Biochim Biophys Acta, 933(1): , Mar
171 7 Literaturverzeichnis [64] M. S. Lebowitz and P. L. Pedersen. Protein inhibitor of mitochondrial ATP synthase: relationship of inhibitor structure to ph-dependent regulation. Arch Biochem Biophys, 330(2): , Jun [65] E. Cabezon, P. J. Butler, M. J. Runswick, and J. E. Walker. Modulation of the oligomerization state of the bovine F 1 -ATPase inhibitor protein, IF 1,bypH. J Biol Chem, 275(33): , Aug [66] E. Cabezón, I. Arechaga, P. Jonathan, G. Butler, and J. E. Walker. Dimerization of bovine F 1 -ATPase by binding the inhibitor protein, IF 1. J Biol Chem, 275(37): , Sep [67] P. D. Boyer. A model for conformational coupling of membrane potential and proton translocation to ATP synthesis and to active transport. FEBS Lett, 58(1):1 6, Oct [68] Kathrin Förster. Thermodynamische, kinetische und Einzelmolekülspektroskopische Untersuchungen an der mitochondrialen H + -ATPsyntase aus Saccharomyces cerevisiae. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [69] H. Noji, R. Yasuda, M. Yoshida, and K Kinosita, Jr. Direct observation of the rotation of F 1 -ATPase. Nature, 386(6622): , Mar [70] H. Noji, K. Häsler, W. Junge, K Kinosita, Jr, M. Yoshida, and S. Engelbrecht. Rotation of Escherichia coli F 1 -ATPase. Biochem Biophys Res Commun, 260(3): , Jul [71] Y. Kato-Yamada, H. Noji, R. Yasuda, K Kinosita, Jr, and M. Yoshida. Direct observation of the rotation of epsilon subunit in F 1 -ATPase. J Biol Chem, 273(31): , Jul [72] H. Omote, N. Sambonmatsu, K. Saito, Y. Sambongi, A. Iwamoto-Kihara, T. Yanagida, Y. Wada, and M. Futai. The gamma-subunit rotation and torque generation in F 1 - ATPase from wild-type or uncoupled mutant Escherichia coli. Proc Natl Acad Sci U SA, 96(14): , Jul [73] Manuel Diez, Boris Zimmermann, Michael Börsch, Marcelle König, Enno Schweinberger, Stefan Steigmiller, Rolf Reuter, Suren Felekyan, Volodymyr Kudryavtsev, Claus A M. Seidel, and Peter Gräber. Proton-powered subunit rotation in single membrane-bound F 0 F 1 -ATP synthase. Nat Struct Mol Biol, 11(2): , Feb
172 7 Literaturverzeichnis [74] Boris Zimmermann. Die H + -ATPsynthase aus Escherichia coli: Untersuchung der Bewegung der ɛ-untereinheit während der Katalyse mit Einzelmolekül- Fluoreszenzspektroskopie. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [75] R. Yasuda, H. Noji, M. Yoshida, K Kinosita, Jr, and H. Itoh. Resolution of distinct rotational substeps by submillisecond kinetic analysis of F 1 -ATPase. Nature, 410(6831): , Apr [76] Katsuya Shimabukuro, Ryohei Yasuda, Eiro Muneyuki, Kiyotaka Y. Hara, Kazuhiko Kinosita, Jr, and Masasuke Yoshida. Catalysis and rotation of F 1 motor: cleavage of ATP at the catalytic site occurs in 1 ms before 40 degree substep rotation. Proc Natl Acad Sci U S A, 100(25): , Dec [77] Takayuki Nishizaka, Kazuhiro Oiwa, Hiroyuki Noji, Shigeki Kimura, Eiro Muneyuki, Masasuke Yoshida, and Kazuhiko Kinosita, Jr. Chemomechanical coupling in F 1 -ATPase revealed by simultaneous observation of nucleotide kinetics and rotation. Nat Struct Mol Biol, 11(2): , Feb [78] Kengo Adachi, Kazuhiro Oiwa, Takayuki Nishizaka, Shou Furuike, Hiroyuki Noji, Hiroyasu Itoh, Masasuke Yoshida, and Kazuhiko Kinosita, Jr. Coupling of rotation and catalysis in F 1 -ATPase revealed by single-molecule imaging and manipulation. Cell, 130(2): , Jul [79] Y. Sambongi, Y. Iko, M. Tanabe, H. Omote, A. Iwamoto-Kihara, I. Ueda, T. Yanagida, Y. Wada, and M. Futai. Mechanical rotation of the c subunit oligomer in ATP synthase (F 0 F 1 ): direct observation. Science, 286(5445): , Nov [80] O. Pänke, K. Gumbiowski, W. Junge, and S. Engelbrecht. F-ATPase: specific observation of the rotating c subunit oligomer of EF 0 EF 1. FEBS Lett, 472(1):34 38, Apr [81] A. Baracca, S. Barogi, V. Carelli, G. Lenaz, and G. Solaini. Catalytic activities of mitochondrial ATP synthase in patients with mitochondrial DNA T8993G mutation in the ATPase 6 gene encoding subunit a. J Biol Chem, 275(6): , Feb [82] W. Junge, H. Lill, and S. Engelbrecht. ATP synthase: an electrochemical transducer with rotatory mechanics. Trends Biochem Sci, 22(11): , Nov [83] O. Shimomura, F. H. Johnson, and Y. Saiga. Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. J Cell Comp Physiol, 59: , Jun
173 7 Literaturverzeichnis [84] M. Chalfie, Y. Tu, G. Euskirchen, W. W. Ward, and D. C. Prasher. Green fluorescent protein as a marker for gene expression. Science, 263(5148): , Feb [85] S. Wang and T. Hazelrigg. Implications for bcd mrna localization from spatial distribution of exu protein in Drosophila oogenesis. Nature, 369(6479): , Jun [86] R. Y. Tsien. The green fluorescent protein. Annu Rev Biochem, 67: , [87] M. Ormö, A. B. Cubitt, K. Kallio, L. A. Gross, R. Y. Tsien, and S. J. Remington. Crystal structure of the Aequorea victoria green fluorescent protein. Science, 273(5280): , Sep [88] R. M. Wachter, M. A. Elsliger, K. Kallio, G. T. Hanson, and S. J. Remington. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure, 6(10): , Oct [89] F. Yang, L. G. Moss, and GN Phillips, Jr. The molecular structure of green fluorescent protein. Nat Biotechnol, 14(10): , Oct [90] R. Heim, D. C. Prasher, and R. Y. Tsien. Wavelength mutations and posttranslational autoxidation of green fluorescent protein. Proc Natl Acad Sci U S A, 91(26): , Dec [91] S. Inouye and F. I. Tsuji. Aequorea green fluorescent protein. Expression of the gene and fluorescence characteristics of the recombinant protein. FEBS Lett, 341(2-3): , Mar [92] T. Stearns. Green fluorescent protein. The green revolution. Curr Biol, 5(3): , Mar [93] R. Heim, A. B. Cubitt, and R. Y. Tsien. Improved green fluorescence. Nature, 373(6516): , Feb [94] B. P. Cormack, R. H. Valdivia, and S. Falkow. FACS-optimized mutants of the green fluorescent protein (GFP). Gene, 173(1 Spec No):33 38, [95] S. H. Bokman and W. W. Ward. Renaturation of Aequorea gree-fluorescent protein. Biochem Biophys Res Commun, 101(4): , Aug [96] W. W. Ward and S. H. Bokman. Reversible denaturation of Aequorea green-fluorescent protein: physical separation and characterization of the renatured protein. Biochemistry, 21(19): , Sep
174 7 Literaturverzeichnis [97] Vladislav V. Verkhusha, Irina M. Kuznetsova, Olesia V. Stepanenko, Andrey G. Zaraisky, Michail M. Shavlovsky, Konstantin K. Turoverov, and Vladimir N. Uversky. High stability of Discosoma DsRed as compared to Aequorea EGFP. Biochemistry, 42(26): , Jul [98] William W. Ward, Hugh J. Prentice, Amy F. Roth, Chris W. Cody, and Sue C. Reeves. Spectral pertubations of the aequorea green-fluorescent protein. Photochem Photobiol, 35(6): , Jun [99] D. Yarbrough, R. M. Wachter, K. Kallio, M. V. Matz, and S. J. Remington. Refined crystal structure of DsRed, a red fluorescent protein from coral, at 2.0-Å resolution. Proceedings of the National Academy of Sciences, 98(2): , Jan [100] Mark Prescott, Michael Ling, Travis Beddoe, Aaron J. Oakley, Sophie Dove, Ove Hoegh- Guldberg, Rodney J. Devenish, and Jamie Rossjohn. The 2.2 Å crystal structure of a pocilloporin pigment reveals a nonplanar chromophore conformation. Structure, 11(3): , Mar [101] L. A. Gross, G. S. Baird, R. C. Hoffman, K. K. Baldridge, and R. Y. Tsien. The structure of the chromophore within DsRed, a red fluorescent protein from coral. Proceedings of the National Academy of Sciences, 97(22): , Oct [102] R. Ando, H. Hama, M. Yamamoto-Hino, H. Mizuno, and A. Miyawaki. An optical marker based on the UV-induced green-to-red photoconversion of a fluorescent protein. Proceedings of the National Academy of Sciences, 99(20): , Oct [103] Hideaki Mizuno, Tapas Kumar Mal, Kit I. Tong, Ryoko Ando, Toshiaki Furuta, Mitsuhiko Ikura, and Atsushi Miyawaki. Photo-Induced Peptide Cleavage in the Green-to-Red Conversion of a Fluorescent Protein. Molecular Cell, 12(4): , Oct [104] H. Mizuno, A. Sawano, P. Eli, H. Hama, and A. Miyawaki. Red fluorescent protein from Discosoma as a fusion tag and a partner for fluorescence resonance energy transfer. Biochemistry, 40(8): , Feb [105] Jan Petersen. Fluoreszierende Proteine und gerichtete Fluoreszenzmarkierung der mitochondrialen H + -ATPsynthase aus Saccharomyces cerevisiae. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [106] George Gabriel Stokes. On the change of refrangibility of light. Philosophical Transactions of the Royal Society of London, pages ,
175 7 Literaturverzeichnis [107] Marcel Bruchez, Mario Moronne, Peter Gin, Shimon Weiss, and A Paul Alivisatos. Semiconductor nanocrystals as fluorescent biological labels. Science, 281(5385): , [108] X. Michalet, F. F. Pinaud, L. A. Bentolila, J. M. Tsay, S. Doose, J. J. Li, G. Sundaresan, A. M. Wu, S. S. Gambhir, and S. Weiss. Quantum dots for live cells, in vivo imaging, and diagnostics. Science, 307(5709): , Jan [109] Eva Galvez, Monika Düser, Michael Börsch, Jörg Wrachtrup, and Peter Gräber. Quantum dots for single-pair fluorescence resonance energy transfer in membrane- integrated EF o F 1. Biochem Soc Trans, 36(Pt 5): , Oct [110] Eva María Galvez, Boris Zimmermann, Verena Rombach-Riegraf, Roland Bienert, and Peter Gräber. Fluorescence resonance energy transfer in single enzyme molecules with a quantum dot as donor. Eur Biophys J, 37(8): , Oct [111] A. A. Deniz, M. Dahan, J. R. Grunwell, T. Ha, A. E. Faulhaber, D. S. Chemla, S. Weiss, and P. G. Schultz. Single-pair fluorescence resonance energy transfer on freely diffusing molecules: observation of Förster distance dependence and subpopulations. Proc Natl Acad Sci U S A, 96(7): , Mar [112] JJ Macklin, JK Trautman, TD Harris, and LE Brus. Imaging and time-resolved spectroscopy of single molecules at an interface. Science, 272(5259): , [113] Peter Oswald. Charakterisierung neuer spfret-messmethoden der H + -ATPsynthase aus mittels Immobilisation und Doppellipidbilayer. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [114] Th. Förster. Zwischenmolekulare Energiewanderung und Fluoreszenz. Annalen der Physik, 437(1-2):55 75, [115] Joseph R Lakowicz. Principles of fluorescence spectroscopy. Springer, [116] R. Aggeler, K. Chicas-Cruz, S. X. Cai, J. F. Keana, and R. A. Capaldi. Introduction of reactive cysteine residues in the epsilon subunit of Escherichia coli F 1 -ATPase, modification of these sites with tetrafluorophenyl azide-maleimides, and examination of changes in the binding of the epsilon subunit when different nucleotides are in catalytic sites. Biochemistry, 31(11): , Mar
176 7 Literaturverzeichnis [117] A. Iwamoto, H. Omote, H. Hanada, N. Tomioka, A. Itai, M. Maeda, and M. Futai. Mutations in Ser174 and the glycine-rich sequence (Gly149, Gly150, and Thr156) in the beta subunit of Escherichia coli H + -ATPase. J Biol Chem, 266(25): , Sep [118] Y. Moriyama, A. Iwamoto, H. Hanada, M. Maeda, and M. Futai. One-step purification of Escherichia coli H + -ATPase (F 0 F 1 ) and its reconstitution into liposomes with neurotransmitter transporters. J Biol Chem, 266(33): , Nov [119] D. J. Klionsky, W. S. Brusilow, and R. D. Simoni. In vivo evidence for the role of the epsilon subunit as an inhibitor of the proton-translocating ATPase of Escherichia coli. J Bacteriol, 160(3): , Dec [120] M. Prescott, N. C. Bush, P. Nagley, and R. J. Devenish. Properties of yeast cells depleted of the OSCP subunit of mitochondrial ATP synthase by regulated expression of the ATP5 gene. Biochem Mol Biol Int, 34(4): , Oct [121] A. F. Straffon, M. Prescott, P. Nagley, and R. J. Devenish. The assembly of yeast mitochondrial ATP synthase: subunit depletion in vivo suggests ordered assembly of the stalk subunits b, OSCP and d. Biochim Biophys Acta, 1371(2): , May [122] Sarah Osterwald. Genomische Mutagenese der Untereinheiten OSCP und b der mitochondrialen H + -ATPsynthase aus S. cerevisiae zur selektiven Fluoreszenzmarkierung für Einzelmolekül-FRET-Messungen. Diplomarbeit, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [123] Rolf Reuter. Struktur- und Funktionsuntersuchungen an der ATPsynthase aus Escherichia coli. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [124] Susanne Fischer. Isolation, Rekonstitution und kinetische Charakterisierung der H + - ATPase aus Escherichia coli. Dissertation, Biologisches Institut der Universität Stuttgart, [125] Angela N. Holder, April L. Ellis, Jin Zou, Ning Chen, and Jenny J. Yang. Facilitating chromophore formation of engineered Ca 2+ binding green fluorescent proteins. Arch Biochem Biophys, 486(1):27 34, Jun [126] 158
177 7 Literaturverzeichnis [127] N Popov, M Schmitt, S Schulzeck, and H Matthies. Eine störungsfreie Mikromethode zur Bestimmung des Proteingehaltes in Gewebehomogenaten. Acta Biol. Med. Ger, 34: , [128] U. K. Laemmli. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature, 227(5259): , Aug [129] A Chrambach, RA Reisfeld, Mr Wyckoff, and J Zaccari. A procedure for rapid and sensitive staining of protein fractionated by polyacrylamide gel electrophoresis. Analytical biochemistry, 20(1): , [130] Robert C. Switzer, Carl R. Merril, and Sidney Shifrin. A highly sensitive silver stain for detecting proteins and peptides in polyacrylamide gels. Analytical Biochemistry, 98(1): , Sep [131] L.D. Mayer, M.J. Hope, and P.R. Cullis. Vesicles of variable sizes produced by a rapid extrusion procedure. Biochimica et Biophysica Acta (BBA) - Biomembranes, 858(1): , Jun [132] Manuela D Alessandro, Paola Turina, and B Andrea Melandri. Quantitative evaluation of the intrinsic uncoupling modulated by ADP and P i in the reconstituted ATP synthase of Escherichia coli. Biochim Biophys Acta, 1807(1): , Jan [133] Ana J. García-Sáez and Petra Schwille. Fluorescence correlation spectroscopy for the study of membrane dynamics and protein/lipid interactions. Methods, 46(2): , Oct [134] Silvia Carlotto, Ilaria Fortunati, Camilla Ferrante, Petra Schwille, and Antonino Polimeno. Time correlated fluorescence characterization of an asymmetrically focused flow in a microfluidic device. Microfluidics and nanofluidics, 10(3): , [135] Zdenek Petrásek and Petra Schwille. Precise measurement of diffusion coefficients using scanning fluorescence correlation spectroscopy. Biophys J, 94(4): , Feb [136] Matthew W. Bowler, Martin G. Montgomery, Andrew G W. Leslie, and John E. Walker. How azide inhibits ATP hydrolysis by the F-ATPases. Proc Natl Acad Sci U S A, 103(23): , Jun [137] Thanh Vu Van, Thomas Heinze, J. Buchholz, and Bernd Rumberg. Quantitative Relationship Between 9-Aminoacridine Fluorescence Quenching and Internal ph in Broken Chloroplasts. Progress in Photosynthesis Research, pages ,
178 7 Literaturverzeichnis [138] R. Casadio. Measurements of transmembrane ph differences of low extents in bacterial chromatophores. European Biophysics Journal, 19(4): , [139] K. Krab and J. van Wezel. Improved derivation of phosphate potentials at different temperatures. Biochimica et Biophysica Acta (BBA) - Bioenergetics, 1098(2): , [140] Roger C. Phillips, S. J. Phillips, Philip George, and Robert J. Rutman. Potentiometric Studies of the Secondary Phosphate Ionizations of AMP, ADP, and ATP, and Calculations of Thermodynamic Data for the Hydrolysis Reactions *. Biochemistry, 2(3): , May [141] Bernard Rudolf. Die H + -ATPase aus Escherichia coli, Darstellung fluoreszierender Substratanaloga und kinetische Untersuchungen zur ATP-Synthese des rekonstituierten Enzyms. Dissertation, Fakultät für Chemie, Pharmazie und Geowissenschaften Universität Freiburg im Breisgau, [142] Ingo Grotjohann and Peter Gräber. The H + -ATPase from chloroplasts: effect of different reconstitution procedures on ATP synthesis activity and on phosphate dependence of ATP synthesis. Biochim Biophys Acta, 1556(2-3): , Dec [143] Y. Hatefi, T. Yagi, D. C. Phelps, S. Y. Wong, S. B. Vik, and Y. M. Galante. Substrate binding affinity changes in mitochondrial energy-linked reactions. Proc Natl Acad Sci U SA, 79(6): , Mar [144] C. Kayalar, J. Rosing, and P. D. Boyer. 2,4-Dinitrophenol causes a marked increase in the apparent K M of P i and of ADP for oxidative phosphorylation. Biochem Biophys Res Commun, 72(3): , Oct [145] S. M. Schuster, G. D. Reinhart, and H. A. Lardy. Studies on the kinetic mechanism of oxidative phosphorylation. J Biol Chem, 252(2): , Jan [146] A. D. Vinogradov. Steady-state and pre-steady-state kinetics of the mitochondrial F 1 F 0 ATPase: is ATP synthase a reversible molecular machine? J Exp Biol, 203(Pt 1):41 49, Jan [147] Tatyana V. Zharova and Andrei D. Vinogradov. Energy-linked binding of P i is required for continuous steady-state proton-translocating ATP hydrolysis catalyzed by F 0 F 1 ATP synthase. Biochemistry, 45(48): , Dec
179 7 Literaturverzeichnis [148] Harvey S. Penefsky. P i binding by the F 1 -ATPase of beef heart mitochondria and of the Escherichia coli plasma membrane. FEBS Lett, 579(10): , Apr [149] C. Grubmeyer, R. L. Cross, and H. S. Penefsky. Mechanism of ATP hydrolysis by beef heart mitochondrial ATPase. Rate constants for elementary steps in catalysis at a single site. J Biol Chem, 257(20): , Oct [150] M. K. al Shawi and A. E. Senior. Catalytic sites of Escherichia coli F 1 -ATPase. Characterization of unisite catalysis at varied ph. Biochmistry, 31(3): , Jan [151] Kathrin Förster, Paola Turina, Friedel Drepper, Wolfgang Haehnel, Susanne Fischer, Peter Gräber, and Jan Petersen. Proton transport coupled ATP synthesis by the purified yeast H + -ATP synthase in proteoliposomes. Biochim Biophys Acta, 1797(11): , Nov [152] M. G. Oakley and P. S. Kim. A buried polar interaction can direct the relative orientation of helices in a coiled coil. Biochemistry, 37(36): , Sep [153] Mark Prescott, Szczepan Nowakowski, Paul Gavin, Phillip Nagley, James C Whisstock, and Rodney J Devenish. Subunit γ-green fluorescent protein fusions are functionally incorporated into mitochondrial F 1 F 0 -ATP synthase, arguing against a rigid cap structure at the top of F 1. Journal of Biological Chemistry, 278(1): , [154] M. F. Paul, S. Ackerman, J. Yue, G. Arselin, J. Velours, A. Tzagolof, and S] Ackermann S [corrected to Ackerman. Cloning of the yeast ATP3 gene coding for the gamma-subunit of F 1 and characterization of atp3 mutants. J Biol Chem, 269(42): , Oct [155] J. Lai-Zhang, Y. Xiao, and D. M. Mueller. Epistatic interactions of deletion mutants in the genes encoding the F 1 -ATPase in yeast Saccharomyces cerevisiae. EMBO J, 18(1):58 64, Jan [156] N. Vázquez-Laslop and G. Dreyfus. Mitochondrial H + -ATPase activation by an amine oxide detergent. J Biol Chem, 261(17): , Jun [157] G.H. Patterson, S.M. Knobel, W.D. Sharif, S.R. Kain, and D.W. Piston. Use of the green fluorescent protein and its mutants in quantitative fluorescence microscopy. Biophysical Journal, 73(5): , Nov [158] E. Spiess, F. Bestvater, A. Heckel-Pompey, K. Toth, M. Hacker, G. Stobrawa, T. Feurer, C. Wotzlaw, U. Berchner-Ptannschmidt, T. Porwol, and et al. Two-photon excitation 161
180 7 Literaturverzeichnis and emission spectra of the green fluorescent protein variants ECFP, EGFP and EYFP. J Microsc, 217(3): , Mar [159] [160] J. S. Bartlett, K. J. Voss, S. Sathyendranath, and A. Vodacek. Raman scattering by pure water and seawater. Appl Opt, 37(15): , May [161] D. M. Rees, A. G. W. Leslie, and J. E. Walker. The structure of the membrane extrinsic region of bovine ATP synthase. Proceedings of the National Academy of Sciences, 106(51): , Dec [162] A. Dautant, J. Velours, and M.-F. Giraud. Crystal Structure of the Mg ADP-inhibited State of the Yeast F 1 c 10 -ATP Synthase. Journal of Biological Chemistry, 285(38): , Jul [163] Hagai Rottbnberg, Tilly Grunwald, and Mordhay Avron. Determination of ΔpH in Chloroplasts. 1. Distribution of [14C] Methylamine. European Journal of Biochemistry, 25(1):54 63, Jan [164] S. Schuldiner, H. Rottenberg, and M. Avron. Determination of ph in chloroplasts. 2. Fluorescent amines as a probe for the determination of ph in chloroplasts. Eur J Biochem, 25(1):64 70, Jan [165] Francesco Pallotti and Giorgio Lenaz. Isolation and subfractionation of mitochondria from animal cells and tissue culture lines. Methods Cell Biol, 65:1 35, [166] Archie L Smith. [13] Preparation, properties, and conditions for assay of mitochondria: Slaughterhouse material, small-scale. Methods in enzymology, 10:81 86, [167] A. W. Linnane and D. M. Ziegler. Studies on the mechanism of oxidative phosphorylation. V. The phosphorylating properties of the electron transport particle. Biochim Biophys Acta, 29(3): , Sep [168] Marc Hansen and Archie L Smith. Studies on the mechanism of oxidative phosphorylation: VII. Preparation of a submitochondrial particle (ETPH) which is capable of fully coupled oxidative phosphorylation. Biochimica et Biophysica Acta (BBA)-Specialized Section on Enzymological Subjects, 81(2): , [169] B. Meyer, I. Wittig, E. Trifilieff, M. Karas, and H. Schagger. Identification of Two Proteins Associated with Mammalian ATP Synthase. Molecular & Cellular Proteomics, 6(10): , Jul
181 7 Literaturverzeichnis [170] V. Giorgio, S. von Stockum, M. Antoniel, A. Fabbro, F. Fogolari, M. Forte, G. D. Glick, V. Petronilli, M. Zoratti, I. Szabo, and et al. Dimers of mitochondrial ATP synthase form the permeability transition pore. Proceedings of the National Academy of Sciences, 110(15): , Apr
182 7 Literaturverzeichnis 164
183 Anhang
184
185 A Die H + i Konzentrationsbestimmung mit der Aminmethode Das in Abbildung A.1 gezeigte Liposom befindet sich in einer Lösung, die ein protonierbares Amin (A) enthält, welches sich sowohl im Außenraum (Index a) als auch im Innenraum (Index i) befindet. Das Amin liegt in einem Protonierungsgleichgewicht vor und die Dissoziationskonstante (K) ist innen und außen gleich. K = [A][H+ ] [AH + ] (A.1) [A] a + [H + ] a [AH + ] a [A] i + [H + ] i [AH + ] i Liposomenmembran Abbildung A.1: Protonierungsgleichgewicht und Schema der Verteilung eines Amins zwischen Innen- und Außenraum eines Liposoms. Ist die Protonenkonzentration im Inneren größer als außen, dann bildet sich im Inneren vermehrt AH + i, die Konzentration von A i sinkt und entsprechend wird von außen ungeladenes Amin A nach innen wandern. Lipidmembranen sind im Allgemeinen höher permeabel für ungeladene Stoffe, als für geladene Stoffe. Zur Vereinfachung wird im Weiteren angenommen, dass nur das ungeladene Amin (A) wandert, während die Membran für das protonierte Amin (AH + ) völlig impermeabel ist. Bestimmt man nun die Aminkonzentration innen und außen, kann man daraus 167
Circulardichroismus (CD) und Fluoreszenz - Anwendungen in der Proteinchemie -
 Circulardichroismus (CD) und Fluoreszenz - Anwendungen in der Proteinchemie - Circulardichroismus (CD) - Einführung Circulardichroismus (CD) - Prinzip Circulardichroismus (CD) - Formel Meßsignal beruht
Circulardichroismus (CD) und Fluoreszenz - Anwendungen in der Proteinchemie - Circulardichroismus (CD) - Einführung Circulardichroismus (CD) - Prinzip Circulardichroismus (CD) - Formel Meßsignal beruht
FLUORESZENZSPEKTROSKOPIE. Biophysik Vorlesung Gábor Talián
 FLUORESZENZSPEKTROSKOPIE Biophysik Vorlesung 05.02.2015. Gábor Talián ENTWURF Lumineszenz Fluoreszenzvorgang Fluorimeter Charakterisierung der Fluoreszenz Fluorophore, Biolumineszenz Lumineszenz Elektronenanregung
FLUORESZENZSPEKTROSKOPIE Biophysik Vorlesung 05.02.2015. Gábor Talián ENTWURF Lumineszenz Fluoreszenzvorgang Fluorimeter Charakterisierung der Fluoreszenz Fluorophore, Biolumineszenz Lumineszenz Elektronenanregung
Materialien und Methoden...16
 Inhaltsverzeichnis I. Einleitung...1 1. Mitochondrien - Energielieferanten der eukaryontischen Zelle... 1 2. Die mitochondriale DNA-Replikation... 5 2.1. Transkriptionsabhängige mtdna-replikation... 5
Inhaltsverzeichnis I. Einleitung...1 1. Mitochondrien - Energielieferanten der eukaryontischen Zelle... 1 2. Die mitochondriale DNA-Replikation... 5 2.1. Transkriptionsabhängige mtdna-replikation... 5
Versuch Fluoreszenz-Quenching
 Versuch Fluoreszenz-Quenching Zielstellung: 1.) Aufnahme des UV-Vis-Spektrums eines Fluoreszenzfarbstoffes 2.) Aufnahme der Kennlinie des verwendeten Photon-Counting-Moduls (PCM) im Bereich von 1,9 2,9
Versuch Fluoreszenz-Quenching Zielstellung: 1.) Aufnahme des UV-Vis-Spektrums eines Fluoreszenzfarbstoffes 2.) Aufnahme der Kennlinie des verwendeten Photon-Counting-Moduls (PCM) im Bereich von 1,9 2,9
Fluorescence-Correlation-Spectroscopy (FCS)
 Fluorescence-Correlation-Spectroscopy () 05.03.2012 Überblick 1 2 3 4 5 Fluoreszenz-Korrelations-Spektroskopie: Entwicklung in den 70er Jahren sehr empfindliche Methode ( sehr geringer Konzentrationen)
Fluorescence-Correlation-Spectroscopy () 05.03.2012 Überblick 1 2 3 4 5 Fluoreszenz-Korrelations-Spektroskopie: Entwicklung in den 70er Jahren sehr empfindliche Methode ( sehr geringer Konzentrationen)
Fluoreszenzintensität ist proportional zur Konzentration angeregter Moleküle. Zeitliche Änderung der Konzentration angeregter Moleküle:
 Fluoreszenzintensität ist proportional zur Konzentration angeregter Moleküle Zeitliche Änderung der Konzentration angeregter Moleküle: d[s 1 ] dt = k rad [S 1 ] k ic [S 1 ] k isc [S 1 ]... = 1 [S 1] Integration
Fluoreszenzintensität ist proportional zur Konzentration angeregter Moleküle Zeitliche Änderung der Konzentration angeregter Moleküle: d[s 1 ] dt = k rad [S 1 ] k ic [S 1 ] k isc [S 1 ]... = 1 [S 1] Integration
Marina Fabry LUDWIG-MAXIMILIANS-UNIVERSITÄT
 Self-Assembled Nanoparticle Probes for Recognition and Detection of Biomolecules Dustin J. Maxwell, Jason R. Taylor & Shuming Nie Journal of the American Chemical Society 2002 Marina Fabry LUDWIG-MAXIMILIANS-UNIVERSITÄT
Self-Assembled Nanoparticle Probes for Recognition and Detection of Biomolecules Dustin J. Maxwell, Jason R. Taylor & Shuming Nie Journal of the American Chemical Society 2002 Marina Fabry LUDWIG-MAXIMILIANS-UNIVERSITÄT
Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA
 Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu
Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu
Biologie für Mediziner
 Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Untersuchungen zur Wirkung von Pasteurella multocida Toxin
 Institut fiir Experimentelle und Klinische Pharmakologie und Toxikologie der Albert-Ludwigs-Universitat Freiburg Untersuchungen zur Wirkung von Pasteurella multocida Toxin Dissertation zur Erlangung des
Institut fiir Experimentelle und Klinische Pharmakologie und Toxikologie der Albert-Ludwigs-Universitat Freiburg Untersuchungen zur Wirkung von Pasteurella multocida Toxin Dissertation zur Erlangung des
Enno Schweinberger (Autor) Multidimensionale Einzelmolekül-Fluoreszenz-Spektroskopie von floureszenzmarkierten Biomolekülen
 Enno Schweinberger (Autor) Multidimensionale Einzelmolekül-Fluoreszenz-Spektroskopie von floureszenzmarkierten Biomolekülen https://cuvillier.de/de/shop/publications/3646 Copyright: Cuvillier Verlag, Inhaberin
Enno Schweinberger (Autor) Multidimensionale Einzelmolekül-Fluoreszenz-Spektroskopie von floureszenzmarkierten Biomolekülen https://cuvillier.de/de/shop/publications/3646 Copyright: Cuvillier Verlag, Inhaberin
Abbildungsverzeichnis. Tabellenverzeichnis. Abkürzungsverzeichnis. 1 Einleitung 1
 Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis VI Tabellenverzeichnis VIII Abkürzungsverzeichnis IX 1 Einleitung 1 1.1 Wasser, ein limitierender Faktor für die Landwirtschaft 1 1.2 Anpassungen
Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis VI Tabellenverzeichnis VIII Abkürzungsverzeichnis IX 1 Einleitung 1 1.1 Wasser, ein limitierender Faktor für die Landwirtschaft 1 1.2 Anpassungen
Fluoreszenz-Korrelations- Spektroskopie (FCS) Seminarvortrag Julia Jäger 17.01.2008
 Fluoreszenz-Korrelations- Spektroskopie (FCS) Seminarvortrag Julia Jäger 17.01.2008 Gliederung Grundlagen der FCS Grundlagen der Fluoreszenz FCS Versuchsaufbau und Durchführung Auswertung FCCS Anwendungsbeispiele
Fluoreszenz-Korrelations- Spektroskopie (FCS) Seminarvortrag Julia Jäger 17.01.2008 Gliederung Grundlagen der FCS Grundlagen der Fluoreszenz FCS Versuchsaufbau und Durchführung Auswertung FCCS Anwendungsbeispiele
Johann Wolfgang Goethe-Universität Frankfurt am Main
 Johann Wolfgang Goethe-Universität Frankfurt am Main Fachbereich Biowissenschaften Teilklausur Biochemie Studiengang Biowissenschaften Modul BSc-Biowiss-7 Studiengang Bioinformatik Modul BSc-Bioinf-8.Studiengang
Johann Wolfgang Goethe-Universität Frankfurt am Main Fachbereich Biowissenschaften Teilklausur Biochemie Studiengang Biowissenschaften Modul BSc-Biowiss-7 Studiengang Bioinformatik Modul BSc-Bioinf-8.Studiengang
Aufgabe 5 (Supersekundärstruktur)
 Aufgabe 5 (Supersekundärstruktur) Fragestellung Bei der Untereinheit des Arthropodenhämocyanins aus Limulus polyphemus werden folgende Fragestellungen untersucht: - Welche Supersekundärstrukturen gibt
Aufgabe 5 (Supersekundärstruktur) Fragestellung Bei der Untereinheit des Arthropodenhämocyanins aus Limulus polyphemus werden folgende Fragestellungen untersucht: - Welche Supersekundärstrukturen gibt
Regulation der Glykolyse: Phosphofructokinase
 Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Ringvorlesung B Fluoreszenz und Anwendung in Molekularer Biotechnologie
 Ringvorlesung B Fluoreszenz und Anwendung in Molekularer Biotechnologie Inhalt: Physikalische Grundlagen Eigenschaften von Fluorophoren Ruprecht-Karls-Universität Heidelberg Institut für Pharmazie and
Ringvorlesung B Fluoreszenz und Anwendung in Molekularer Biotechnologie Inhalt: Physikalische Grundlagen Eigenschaften von Fluorophoren Ruprecht-Karls-Universität Heidelberg Institut für Pharmazie and
1 Einleitung Material und Methoden
 1 Einleitung 1 1.1 P-Typ ATPasen 2 1.1.1 Struktur und Funktion von P-Typ ATPasen 4 1.1.2 CPx-ATPasen 7 1.1.2.1 Nukleotidbindungs-/Phosphorylierungsdomäne 8 1.1.2.2 Aktuatordomäne 13 1.1.2.3 Cytoplasmatische
1 Einleitung 1 1.1 P-Typ ATPasen 2 1.1.1 Struktur und Funktion von P-Typ ATPasen 4 1.1.2 CPx-ATPasen 7 1.1.2.1 Nukleotidbindungs-/Phosphorylierungsdomäne 8 1.1.2.2 Aktuatordomäne 13 1.1.2.3 Cytoplasmatische
Abbildung 1: Schematische Darstellung der Domänenorganisation von Survivin
 Abbildung 1: Schematische Darstellung der Domänenorganisation von Survivin Survivin weist eine N-terminale BIR-Domäne (grün) und eine C-terminale α-helix (blau) auf. Das intrinsische nuclear export signal
Abbildung 1: Schematische Darstellung der Domänenorganisation von Survivin Survivin weist eine N-terminale BIR-Domäne (grün) und eine C-terminale α-helix (blau) auf. Das intrinsische nuclear export signal
Praktikum Instrumentelle Bioanalytik Versuch: Fluoreszenzspektroskopie
 Praktikum Instrumentelle Bioanalytik Versuch: Fluoreszenzspektroskopie wichtige Hinweise: Die beiden Versuchsteile werden an zwei unterschiedlichen Tagen im Praktikumssaal durchgeführt. Ein bestandenes
Praktikum Instrumentelle Bioanalytik Versuch: Fluoreszenzspektroskopie wichtige Hinweise: Die beiden Versuchsteile werden an zwei unterschiedlichen Tagen im Praktikumssaal durchgeführt. Ein bestandenes
Wirkungsmechanismen regulatorischer Enzyme
 Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
Biochemie und. chemischer Mechanismus
 Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München Biochemie und chemischer Mechanismus der Thiomethylierung von RNA Dorothea Maria
Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München Biochemie und chemischer Mechanismus der Thiomethylierung von RNA Dorothea Maria
3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung
 3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung Transkriptionsfaktoren erfahren oft eine posttranslationelle Modifikation in Form von Phosphorylierung und werden dadurch in ihrer Aktivität
3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung Transkriptionsfaktoren erfahren oft eine posttranslationelle Modifikation in Form von Phosphorylierung und werden dadurch in ihrer Aktivität
Was bisher geschah 1
 Was bisher geschah 1 Zellatmung (Übersicht) Der Citratcyclus ist die erste Stufe der Zellatmung 2 Citratzyklus Synonyme: Tricarbonsäurezyklus (TCA-Zyklus) Krebszyklus, Zitronensäurezyklus Der Zyklus ist
Was bisher geschah 1 Zellatmung (Übersicht) Der Citratcyclus ist die erste Stufe der Zellatmung 2 Citratzyklus Synonyme: Tricarbonsäurezyklus (TCA-Zyklus) Krebszyklus, Zitronensäurezyklus Der Zyklus ist
Untersuchungen zum Einfluss von Polyalaninsequenzen auf die Bildung amyloider Fibrillen von PABPN1
 Untersuchungen zum Einfluss von Polyalaninsequenzen auf die Bildung amyloider Fibrillen von PABPN1 Dissertation zur Erlangung des akademischen Grades doctor rerum naturalium (Dr. rer. nat.) vorgelegt der
Untersuchungen zum Einfluss von Polyalaninsequenzen auf die Bildung amyloider Fibrillen von PABPN1 Dissertation zur Erlangung des akademischen Grades doctor rerum naturalium (Dr. rer. nat.) vorgelegt der
Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle. in bioinformatisch identifizierten,
 Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle in bioinformatisch identifizierten, Pathogen-responsiven c/s-elementen aus Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften
Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle in bioinformatisch identifizierten, Pathogen-responsiven c/s-elementen aus Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften
Einzelmolekülstudien
 Einzelmolekülstudien Motivation Ensemble-Messung: - Bestimmung eines Mittelwertes des Ensembles (z.b. gemittelte Struktur von 10 13 Proteinmolekülen in einem Proteinkristall) - gute Statistik: verlässlicher
Einzelmolekülstudien Motivation Ensemble-Messung: - Bestimmung eines Mittelwertes des Ensembles (z.b. gemittelte Struktur von 10 13 Proteinmolekülen in einem Proteinkristall) - gute Statistik: verlässlicher
Einzelmolekül. Enzym-Dynamik. Fabian Wolfertstetter
 Einzelmolekül Enzym-Dynamik Fabian Wolfertstetter Bisher: Untersuchung chemischer Reaktionen nur im Ensemble Messung nur der Edukte und Produkte (fast) ohne Details über Zwischenschritte Dazwischen: Black
Einzelmolekül Enzym-Dynamik Fabian Wolfertstetter Bisher: Untersuchung chemischer Reaktionen nur im Ensemble Messung nur der Edukte und Produkte (fast) ohne Details über Zwischenschritte Dazwischen: Black
2. Tertiärstruktur. Lernziele: 1) Verstehen wie Röntgenstrukturanalyse und NMR Spektroskopie gebraucht werden um Proteinstrukturen zu bestimmen.
 2. Tertiärstruktur Lernziele: 1) Verstehen wie Röntgenstrukturanalyse und NMR Spektroskopie gebraucht werden um Proteinstrukturen zu bestimmen. 2) Verstehen warum nicht polare Reste im inneren eines Proteins
2. Tertiärstruktur Lernziele: 1) Verstehen wie Röntgenstrukturanalyse und NMR Spektroskopie gebraucht werden um Proteinstrukturen zu bestimmen. 2) Verstehen warum nicht polare Reste im inneren eines Proteins
FLUORESZENZSPEKTROSKOPIE. Biophysik Vorlesung Gábor Talián
 FLUORESZENZSPEKTROSKOPIE Biophysik Vorlesung Gábor Talián ENTWURF Lumineszenz Fluoreszenzvorgang Fluorimeter Charakterisierung der Fluoreszenz Fluorophore, Biolumineszenz Lumineszenz Elektronenanregung
FLUORESZENZSPEKTROSKOPIE Biophysik Vorlesung Gábor Talián ENTWURF Lumineszenz Fluoreszenzvorgang Fluorimeter Charakterisierung der Fluoreszenz Fluorophore, Biolumineszenz Lumineszenz Elektronenanregung
Mitochondriale Elektronentransportkette (Atmungskette)
 Mitochondriale Elektronentransportkette (Atmungskette) Mitochondriale Elektronentransportkette (Atmungskette) Komplex I und II übetragen Elektronen auf Coenzym Q (Ubichinon) Gekoppelte Elektronen-Protonen
Mitochondriale Elektronentransportkette (Atmungskette) Mitochondriale Elektronentransportkette (Atmungskette) Komplex I und II übetragen Elektronen auf Coenzym Q (Ubichinon) Gekoppelte Elektronen-Protonen
UV/Vis-Spektroskopie oder auch: Elektronenanregungsspektroskopie
 Teil 5 UV/Vis-Spektroskopie oder auch: Elektronenanregungsspektroskopie Dr. Christian Merten, Ruhr-Uni Bochum, WiSe 2018/19 www.ruhr-uni-bochum.de/chirality 1 Komplementärfarben Absorbiert eine Probe gelbes
Teil 5 UV/Vis-Spektroskopie oder auch: Elektronenanregungsspektroskopie Dr. Christian Merten, Ruhr-Uni Bochum, WiSe 2018/19 www.ruhr-uni-bochum.de/chirality 1 Komplementärfarben Absorbiert eine Probe gelbes
UV/Vis-Spektroskopie oder auch: Elektronenanregungsspektroskopie
 Teil 5 UV/Vis-Spektroskopie oder auch: Elektronenanregungsspektroskopie Dr. Christian Merten, Ruhr-Uni Bochum, WiSe 2017/18 www.ruhr-uni-bochum.de/chirality 1 Reminder: MO-Diagramm von O 2 2- MdS-1 UV/Vis-Spektroskopie
Teil 5 UV/Vis-Spektroskopie oder auch: Elektronenanregungsspektroskopie Dr. Christian Merten, Ruhr-Uni Bochum, WiSe 2017/18 www.ruhr-uni-bochum.de/chirality 1 Reminder: MO-Diagramm von O 2 2- MdS-1 UV/Vis-Spektroskopie
Stoffwechsel. Metabolismus (3)
 Vorlesung Zell- und Molekularbiologie Stoffwechsel Metabolismus (3) Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien 1 Glykolyse 1 Glucose und in der Glykolyse daraus gebildete
Vorlesung Zell- und Molekularbiologie Stoffwechsel Metabolismus (3) Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien 1 Glykolyse 1 Glucose und in der Glykolyse daraus gebildete
Z 11 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: 2) ÜBERSICHT ÜBER DEN ENERGIESTOFFWECHSEL
 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE Zusammenfassung Zusammenfassung Kapitel 11 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung.
GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE Zusammenfassung Zusammenfassung Kapitel 11 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung.
ENZYMATISCHE GRUNDLAGEN DER AKTIVIERUNG DES AMIDOXIM- UND ESTER- PRODRUGS XIMELAGATRAN
 ENZYMATISCHE GRUNDLAGEN DER AKTIVIERUNG DES AMIDOXIM- UND ESTER- PRODRUGS XIMELAGATRAN Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Christian-Albrechts-Universität
ENZYMATISCHE GRUNDLAGEN DER AKTIVIERUNG DES AMIDOXIM- UND ESTER- PRODRUGS XIMELAGATRAN Dissertation zur Erlangung des Doktorgrades der Mathematisch-Naturwissenschaftlichen Fakultät der Christian-Albrechts-Universität
Schritt für Schritt Simulation Die Atmungskette
 KENNZEICHEN: KURS - SCHULE - - Schritt für Schritt Simulation Die Atmungskette Inhalt Übersicht über das Mitochondrium und die Mitochondrienmembran S.2 Zeichenerklärung S.3 Stichwortverzeichnis S.4 Leitfaden
KENNZEICHEN: KURS - SCHULE - - Schritt für Schritt Simulation Die Atmungskette Inhalt Übersicht über das Mitochondrium und die Mitochondrienmembran S.2 Zeichenerklärung S.3 Stichwortverzeichnis S.4 Leitfaden
Enzym-Dynamik an einzelnen Molekülen. Paul Käufl
 Enzym-Dynamik an einzelnen Molekülen Paul Käufl Enzym-Dynamik einzelner Moleküle Quelle: (5) 2 Enzym-Dynamik einzelner Moleküle Bis vor ca. 20 Jahren: Chemische Reaktionen (in Lösung) im Wesentlichen nur
Enzym-Dynamik an einzelnen Molekülen Paul Käufl Enzym-Dynamik einzelner Moleküle Quelle: (5) 2 Enzym-Dynamik einzelner Moleküle Bis vor ca. 20 Jahren: Chemische Reaktionen (in Lösung) im Wesentlichen nur
Kapitel 5: 1. Siderophore assistieren dem Transfer welcher der folgenden Makronährstoffe über Membranen? A. Stickstoff B. Phosphor C. Eisen D.
 Kapitel 5: 1 Siderophore assistieren dem Transfer welcher der folgenden Makronährstoffe über Membranen? A. Stickstoff B. Phosphor C. Eisen D. Kalium Kapitel 5: 2 Welcher der folgenden Makronährstoffe ist
Kapitel 5: 1 Siderophore assistieren dem Transfer welcher der folgenden Makronährstoffe über Membranen? A. Stickstoff B. Phosphor C. Eisen D. Kalium Kapitel 5: 2 Welcher der folgenden Makronährstoffe ist
Z11 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: 2) ÜBERSICHT ÜBER DEN ENERGIESTOFFWECHSEL
 GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung. Proteine aus der Nahrung werden
GRUNDLAGEN DER BIOCHEMIE STOFFWECHSELWEGE 1) DIE WICHTIGSTEN STOFFWECHSELWEGE: Fette und Kohlenhydrate aus der Nahrung nutzt der Körper hauptsächlich zur Energiegewinnung. Proteine aus der Nahrung werden
Uni Konstanz - Kristallinität als Schlüsselfunktion
 Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/uni-konstanz-kristallinitaetals-schluesselfunktion/ Uni Konstanz - Kristallinität als Schlüsselfunktion Nachwachsende
Powered by Seiten-Adresse: https://www.gesundheitsindustriebw.de/de/fachbeitrag/aktuell/uni-konstanz-kristallinitaetals-schluesselfunktion/ Uni Konstanz - Kristallinität als Schlüsselfunktion Nachwachsende
Mechanismen der ATP Synthese in Mitochondrien
 Mechanismen der ATP Synthese in Mitochondrien Übersicht Die Bedeutung von ATP Aufbau eines Mitochondriums ATP Synthese: Citratzyklus Atmungskette ATP Synthase Regulation der ATP Synthese Die Bedeutung
Mechanismen der ATP Synthese in Mitochondrien Übersicht Die Bedeutung von ATP Aufbau eines Mitochondriums ATP Synthese: Citratzyklus Atmungskette ATP Synthase Regulation der ATP Synthese Die Bedeutung
Citratzyklus. Biochemie Maria Otto,Bo Mi Ok Kwon Park
 Citratzyklus Biochemie 13.12.2004 Maria Otto,Bo Mi Ok Kwon Park O CH 3 C Acetyl-CoA + H 2 O HO C COO C NADH O C H Citrat Cis-Aconitat H C Malat Citratzyklus HO C H Isocitrat CH H 2 O Fumarat C = O FADH
Citratzyklus Biochemie 13.12.2004 Maria Otto,Bo Mi Ok Kwon Park O CH 3 C Acetyl-CoA + H 2 O HO C COO C NADH O C H Citrat Cis-Aconitat H C Malat Citratzyklus HO C H Isocitrat CH H 2 O Fumarat C = O FADH
- der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen
 Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Die Erwartungswerte von Operatoren sind gegeben durch. (x, t)a (x, t) =h A i
 Die Wahrscheinlichkeit, das System zu einem bestimmten Zeitpunkt in einem bestimmten Zustand anzutreffen, ist durch das Betragsquadrat der Wellenfunktion (x, t) 2 gegeben Die Erwartungswerte von Operatoren
Die Wahrscheinlichkeit, das System zu einem bestimmten Zeitpunkt in einem bestimmten Zustand anzutreffen, ist durch das Betragsquadrat der Wellenfunktion (x, t) 2 gegeben Die Erwartungswerte von Operatoren
TEV Protease, rekombinant (Kat.-Nr )
 GEBRAUCHSANLEITUNG TEV Protease, rekombinant (Kat.-Nr. 36403) SERVA Electrophoresis GmbH - Carl-Benz-Str. 7-69115 Heidelberg Phone +49-6221-138400, Fax +49-6221-1384010 e-mail: info@serva.de -http://www.serva.de
GEBRAUCHSANLEITUNG TEV Protease, rekombinant (Kat.-Nr. 36403) SERVA Electrophoresis GmbH - Carl-Benz-Str. 7-69115 Heidelberg Phone +49-6221-138400, Fax +49-6221-1384010 e-mail: info@serva.de -http://www.serva.de
Fluoreszenzspektroskopie
 Biophysikalisches Praktikum Institut für Biophysik Johann Wolfgang Goethe-Universität Frankfurt am Main Fluoreszenzspektroskopie V. Vogel, D. Schubert, C. Stiebing Stand: Oktober 2011 1 Motivation Die
Biophysikalisches Praktikum Institut für Biophysik Johann Wolfgang Goethe-Universität Frankfurt am Main Fluoreszenzspektroskopie V. Vogel, D. Schubert, C. Stiebing Stand: Oktober 2011 1 Motivation Die
SS Thomas Schrader. der Universität Duisburg-Essen. (Teil 8: Redoxprozesse, Elektrochemie)
 Chemie für Biologen SS 2010 Thomas Schrader Institut t für Organische Chemie der Universität Duisburg-Essen (Teil 8: Redoxprozesse, Elektrochemie) Oxidation und Reduktion Redoxreaktionen: Ein Atom oder
Chemie für Biologen SS 2010 Thomas Schrader Institut t für Organische Chemie der Universität Duisburg-Essen (Teil 8: Redoxprozesse, Elektrochemie) Oxidation und Reduktion Redoxreaktionen: Ein Atom oder
ATTO 565 und ATTO 590
 ATTO 565 und ATTO 590 Allgemeine Informationen ATTO 565 und ATTO 590 sind Fluoreszenzmarker aus der Familie der Rhodamin-Farbstoffe. Diese Farbstoffe besitzen als gemeinsames Strukturelement einen Carboxyphenyl-
ATTO 565 und ATTO 590 Allgemeine Informationen ATTO 565 und ATTO 590 sind Fluoreszenzmarker aus der Familie der Rhodamin-Farbstoffe. Diese Farbstoffe besitzen als gemeinsames Strukturelement einen Carboxyphenyl-
Chemistry Department Cologne University. Photochemie 1 PC 2 SS Chemistry Department Cologne University. Photochemie
 Photochemie 1 PC 2 2016 Photochemie 2 PC 2 2016 1 Wichtige photophysikalische Prozesse 3 PC 2 2016 Der Grundzustand Boltzmann Verteilung: Alle Moleküle sind im elektronischen Grundzustand (0) chwingungsgrundzustand
Photochemie 1 PC 2 2016 Photochemie 2 PC 2 2016 1 Wichtige photophysikalische Prozesse 3 PC 2 2016 Der Grundzustand Boltzmann Verteilung: Alle Moleküle sind im elektronischen Grundzustand (0) chwingungsgrundzustand
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne
 Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Atmung Respiration 1
 Atmung Respiration 1 Atmung Die oxidative Phosphorylierung ist der letzte Schritt einer Serie von Energieumwandlungen, die insgesamt als Zellatmung oder einfach Atmung (Respiration) bezeichnet werden 2
Atmung Respiration 1 Atmung Die oxidative Phosphorylierung ist der letzte Schritt einer Serie von Energieumwandlungen, die insgesamt als Zellatmung oder einfach Atmung (Respiration) bezeichnet werden 2
Die Bedeutung des. mitochondrialen ABC-Transporters ATM3. für die Molybdäncofaktor-Biosynthese. in Arabidopsis thaliana
 Die Bedeutung des mitochondrialen ABC-Transporters ATM3 für die Molybdäncofaktor-Biosynthese in Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften der Technischen Universität Carolo-Wilhelmina
Die Bedeutung des mitochondrialen ABC-Transporters ATM3 für die Molybdäncofaktor-Biosynthese in Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften der Technischen Universität Carolo-Wilhelmina
Georg-August-Universität Göttingen
 Georg-August-Universität Göttingen Abteilung für Allgemeine Mikrobiologie Institut für Mikrobiologie und Genetik Prof. Dr. Jörg Stülke Email:jstuelk@gwdg.de http://wwwuser.gwdg.de/~genmibio/stuelke.html
Georg-August-Universität Göttingen Abteilung für Allgemeine Mikrobiologie Institut für Mikrobiologie und Genetik Prof. Dr. Jörg Stülke Email:jstuelk@gwdg.de http://wwwuser.gwdg.de/~genmibio/stuelke.html
Membranen. U. Albrecht
 Membranen Struktur einer Plasmamembran Moleküle gegeneinander beweglich -> flüssiger Charakter Fluidität abhängig von 1) Lipidzusammensetzung (gesättigt/ungesättigt) 2) Umgebungstemperatur Biologische
Membranen Struktur einer Plasmamembran Moleküle gegeneinander beweglich -> flüssiger Charakter Fluidität abhängig von 1) Lipidzusammensetzung (gesättigt/ungesättigt) 2) Umgebungstemperatur Biologische
SCHWEIZER JUGEND FORSCHT. Chemie und Materialwissenschaften
 SCHWEIZER JUGEND FORSCHT Chemie und Materialwissenschaften Studie einer ultraschnellen Fotochemischen Reaktion mit Laserspektroskopie Gianluca Schmoll Widmer Betreuer: Dr. Sandra Mosquera Vazquez, Dr.
SCHWEIZER JUGEND FORSCHT Chemie und Materialwissenschaften Studie einer ultraschnellen Fotochemischen Reaktion mit Laserspektroskopie Gianluca Schmoll Widmer Betreuer: Dr. Sandra Mosquera Vazquez, Dr.
Entdeckungen unter der Schädeldecke. Jean-Marc Fritschy Institut für Pharmakologie und Toxikologie
 Entdeckungen unter der Schädeldecke Jean-Marc Fritschy Institut für Pharmakologie und Toxikologie Inhalt 1. GFP, das Wunderprotein 2. Die Nervenzellen bei der Arbeit beobachten 3. Nervenzellen mit Licht
Entdeckungen unter der Schädeldecke Jean-Marc Fritschy Institut für Pharmakologie und Toxikologie Inhalt 1. GFP, das Wunderprotein 2. Die Nervenzellen bei der Arbeit beobachten 3. Nervenzellen mit Licht
Antibiotika sind oft Inhibitoren der Genexpression
 Antibiotika sind oft Inhibitoren der Genexpression Inhibitoren der Transkription: Rifampicin, Actinomycin α-amanitin Inhibitoren der Translation: Puromycin, Streptomycin, Tetracycline, Chloramphenicol
Antibiotika sind oft Inhibitoren der Genexpression Inhibitoren der Transkription: Rifampicin, Actinomycin α-amanitin Inhibitoren der Translation: Puromycin, Streptomycin, Tetracycline, Chloramphenicol
INHALTSVERZEICHNIS 1 KURZFASSUNG EINLEITUNG ZIELSETZUNG Inhaltsverzeichnis
 I INHALTSVERZEICHNIS 1 KURZFASSUNG... 1 2 EINLEITUNG... 3 2.1 Die Plasmamembran Vermittler zwischen zwei Welten... 3 2.1.1 Lipide der Plasmamembran... 4 2.1.2 Proteine der Plasmamembran... 7 2.1.3 Modelle
I INHALTSVERZEICHNIS 1 KURZFASSUNG... 1 2 EINLEITUNG... 3 2.1 Die Plasmamembran Vermittler zwischen zwei Welten... 3 2.1.1 Lipide der Plasmamembran... 4 2.1.2 Proteine der Plasmamembran... 7 2.1.3 Modelle
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne
 Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Entstehung der Erde und Lebewesen Entwicklung der Zellforschung Kennzeichen des Lebens Grundbaupläne Kennzeichen einer lebenden Zelle Zellen entstehen aus Zellen jede Zelle hat einen kompletten Satz Erbanlagen
Struktur-Funktionsbeziehungen. NADH-Oxidase aus. Thermus thermophilus
 Struktur-Funktionsbeziehungen der NADH-Oxidase aus Thermus thermophilus Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades eines
Struktur-Funktionsbeziehungen der NADH-Oxidase aus Thermus thermophilus Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades eines
Citratzyklus. Citratzyklus
 Der hat in der Zelle verschiedene Aufgaben. Teilschritte werden z.b. bei manchen Gärungen eingesetzt (Methyl-Malonyl-CoA-Weg). Er ist wichtig zur Bereitstellung verschiedener Vorstufen für Biosynthesen,
Der hat in der Zelle verschiedene Aufgaben. Teilschritte werden z.b. bei manchen Gärungen eingesetzt (Methyl-Malonyl-CoA-Weg). Er ist wichtig zur Bereitstellung verschiedener Vorstufen für Biosynthesen,
Eukaryoten und Prokaryoten
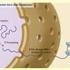 Eukaryoten und Prokaryoten Biochemie Inhalt Zellen Prokaryoten, Eukaryoten Unterschiede und Ähnlichkeiten Zellstrukturen Evolution der Zellen Entwicklung von Mitochondrien und Chloroplasten Angriffsmöglichkeiten
Eukaryoten und Prokaryoten Biochemie Inhalt Zellen Prokaryoten, Eukaryoten Unterschiede und Ähnlichkeiten Zellstrukturen Evolution der Zellen Entwicklung von Mitochondrien und Chloroplasten Angriffsmöglichkeiten
FP III für Biophysiker FLUORESZENZ-KORRELATIONS-SPEKTROSKOPIE
 Physikalisches Institut der Universität Bayreuth -LS für Experimentalphysik IV- FP III für Biophysiker FLUORESZENZ-KORRELATIONS-SPEKTROSKOPIE 1. Einleitung und Aufgabenstellung 2. Theorie 3. Versuchsanordnung
Physikalisches Institut der Universität Bayreuth -LS für Experimentalphysik IV- FP III für Biophysiker FLUORESZENZ-KORRELATIONS-SPEKTROSKOPIE 1. Einleitung und Aufgabenstellung 2. Theorie 3. Versuchsanordnung
NMR - Seite 1. NMR (Kernresonanzspektroskopie) Allgemeines zur Theorie
 NMR - Seite 1 NMR (Kernresonanzspektroskopie) Allgemeines zur Theorie Protonen besitzen ebenso wie Elektronen einen eigenen Spin (Drehung um die eigene Achse).Allerdings gibt es mehrere Möglichkeiten als
NMR - Seite 1 NMR (Kernresonanzspektroskopie) Allgemeines zur Theorie Protonen besitzen ebenso wie Elektronen einen eigenen Spin (Drehung um die eigene Achse).Allerdings gibt es mehrere Möglichkeiten als
Ausbreitung von elektromagnetischer Strahlung
 Ausbreitung von elektromagnetischer Strahlung E! B Der elektrische Feldvektor und der magnetische Feldvektor stehen senkrecht aufeinander Die elektromagentische Welle ist beschrieben durch x x E = E 0
Ausbreitung von elektromagnetischer Strahlung E! B Der elektrische Feldvektor und der magnetische Feldvektor stehen senkrecht aufeinander Die elektromagentische Welle ist beschrieben durch x x E = E 0
Prostaglandinstoffwechsel in Trypanosoma brucei: Klonierung und Charakterisierung der Prostaglandin F 2a Synthase und der PGD 2 induzierte Zelltod
 Prostaglandinstoffwechsel in Trypanosoma brucei: Klonierung und Charakterisierung der Prostaglandin F 2a Synthase und der PGD 2 induzierte Zelltod DISSERTATION der Fakultät für Chemie und Pharmazie der
Prostaglandinstoffwechsel in Trypanosoma brucei: Klonierung und Charakterisierung der Prostaglandin F 2a Synthase und der PGD 2 induzierte Zelltod DISSERTATION der Fakultät für Chemie und Pharmazie der
Versuch 3. Elektrophorese
 Versuch 3 Elektrophorese Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@mustermann.de X X X Prof. Dr. Schäfer Wird benotet?: 1. Einleitung Ziel des ersten Versuches
Versuch 3 Elektrophorese Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@mustermann.de X X X Prof. Dr. Schäfer Wird benotet?: 1. Einleitung Ziel des ersten Versuches
Kapitel 7: Elektronische Spektroskopie
 Kapitel 7: Elektronische Spektroskopie Übersicht: 7.1 Drehimpuls-Kopplungshierarchien in Molekülen: Hundsche Fälle 7.2 Auswahlregeln für rovibronische Übergänge 7.3 Das Franck-Condon-Prinzip 7.4 Zerfall
Kapitel 7: Elektronische Spektroskopie Übersicht: 7.1 Drehimpuls-Kopplungshierarchien in Molekülen: Hundsche Fälle 7.2 Auswahlregeln für rovibronische Übergänge 7.3 Das Franck-Condon-Prinzip 7.4 Zerfall
12. Oxidative Phosphorylierung
 12. Oxidative Phosphorylierung 303 Zweck: Gewinnung von ATP Regeneration von NAD + und FAD Gesamtreaktionen: 3 ADP + 3 P i 3 ATP NADH + 0,5 O 2 + H + NAD + + H-O-H 2 ADP + 2 P i 2 ATP FADH 2 + 0,5 O 2
12. Oxidative Phosphorylierung 303 Zweck: Gewinnung von ATP Regeneration von NAD + und FAD Gesamtreaktionen: 3 ADP + 3 P i 3 ATP NADH + 0,5 O 2 + H + NAD + + H-O-H 2 ADP + 2 P i 2 ATP FADH 2 + 0,5 O 2
In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit
 In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit in der Nucleotidsequenz der DNA verschlüsselt (codiert)
In den Proteinen der Lebewesen treten in der Regel 20 verschiedene Aminosäuren auf. Deren Reihenfolge muss in der Nucleotidsequenz der mrna und damit in der Nucleotidsequenz der DNA verschlüsselt (codiert)
Fluoreszenzspektroskopie
 A 60 Fluoreszenzspektroskopie Aufgabe: Bestimmen Sie das Absorptions- und Fluoreszenzspektrum des Farbstoffs Rhodamin 6G in ethanolischer Lösung. Bestimmen Sie in beiden Spektren jeweils die Wellenlänge
A 60 Fluoreszenzspektroskopie Aufgabe: Bestimmen Sie das Absorptions- und Fluoreszenzspektrum des Farbstoffs Rhodamin 6G in ethanolischer Lösung. Bestimmen Sie in beiden Spektren jeweils die Wellenlänge
9. GV: Atom- und Molekülspektren
 Physik Praktikum I: WS 2005/06 Protokoll zum Praktikum Dienstag, 25.10.05 9. GV: Atom- und Molekülspektren Protokollanten Jörg Mönnich Anton Friesen - Veranstalter Andreas Branding - 1 - Theorie Während
Physik Praktikum I: WS 2005/06 Protokoll zum Praktikum Dienstag, 25.10.05 9. GV: Atom- und Molekülspektren Protokollanten Jörg Mönnich Anton Friesen - Veranstalter Andreas Branding - 1 - Theorie Während
Biochemische UE Alkaline Phosphatase.
 Biochemische UE Alkaline Phosphatase peter.hammerl@sbg.ac.at Alkaline Phosphatase: Katalysiert die Hydrolyse von Phosphorsäure-Estern: O - O - Ser-102 R O P==O O - H 2 O R OH + HO P==O O - ph-optimum im
Biochemische UE Alkaline Phosphatase peter.hammerl@sbg.ac.at Alkaline Phosphatase: Katalysiert die Hydrolyse von Phosphorsäure-Estern: O - O - Ser-102 R O P==O O - H 2 O R OH + HO P==O O - ph-optimum im
Kinetik zusammengesetzter Reaktionen
 Kinetik zusammengesetzter Reaktionen Kap. 23 1 PC 2 SS 2016 Kinetik zusammengesetzter Reaktionen Kettenreaktionen Explosionen Polymerisationen Schrittweise Polymerisation Kettenpolymerisation Homogene
Kinetik zusammengesetzter Reaktionen Kap. 23 1 PC 2 SS 2016 Kinetik zusammengesetzter Reaktionen Kettenreaktionen Explosionen Polymerisationen Schrittweise Polymerisation Kettenpolymerisation Homogene
Fluoreszenzmikroskopie
 Fluoreszenzmikroskopie Fluoreszenz Emission eines Photons beim Übergang eines Elektrons von einem höheren in ein tieferes Energieniveau eines Moleküls nach Absorption von Licht kurzer Wellenlänge (innerhalb
Fluoreszenzmikroskopie Fluoreszenz Emission eines Photons beim Übergang eines Elektrons von einem höheren in ein tieferes Energieniveau eines Moleküls nach Absorption von Licht kurzer Wellenlänge (innerhalb
Fluoreszenz und Excimerenbildung
 Fluoreszenz und Excimerenbildung Fragen und Stichworte zur Vorbereitung: Aufbau und Funktion eines Fluoreszenzspektrometers Spektrale und natürliche Bandbreite Signal-Rausch-Verhältnis Jablonski-Diagramm:
Fluoreszenz und Excimerenbildung Fragen und Stichworte zur Vorbereitung: Aufbau und Funktion eines Fluoreszenzspektrometers Spektrale und natürliche Bandbreite Signal-Rausch-Verhältnis Jablonski-Diagramm:
Mathematik und Naturwissenschaften, Biologie, Biochemie. Biochemie II - Tutorium
 Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 20.10.2016 Alexander Götze 3.Semester Molekulare Biotechnologie a.goetze2207@googlemail.com Mi. 2DS DRU. 68 H Michel
Mathematik und Naturwissenschaften, Biologie, Biochemie Biochemie II - Tutorium Dresden, 20.10.2016 Alexander Götze 3.Semester Molekulare Biotechnologie a.goetze2207@googlemail.com Mi. 2DS DRU. 68 H Michel
Reduction / Oxidation
 Reduction / Oxidation Pyruvat C6H12O 6 Glucose Glycogen Glucose-6-P Glycolyse 2 e - 2 Pyruvat 2 e - 2 Acetyl-CoA 2 CO 2 ATP ADP ATP ADP Citrat-Zyklus oder Tricarbonsäure 4 CO 2 8 e - Zyklus 6 O2 6 H 2
Reduction / Oxidation Pyruvat C6H12O 6 Glucose Glycogen Glucose-6-P Glycolyse 2 e - 2 Pyruvat 2 e - 2 Acetyl-CoA 2 CO 2 ATP ADP ATP ADP Citrat-Zyklus oder Tricarbonsäure 4 CO 2 8 e - Zyklus 6 O2 6 H 2
Herstellung und Charakterisierung von zellpermeablem Interferon-α des Waldmurmeltieres (Marmota monax)
 Aus der Abteilung Innere Medizin II der Medizinischen Universitätsklinik der Albert-Ludwigs-Universität Freiburg im Breisgau Herstellung und Charakterisierung von zellpermeablem Interferon-α des Waldmurmeltieres
Aus der Abteilung Innere Medizin II der Medizinischen Universitätsklinik der Albert-Ludwigs-Universität Freiburg im Breisgau Herstellung und Charakterisierung von zellpermeablem Interferon-α des Waldmurmeltieres
Charakterisierung der PII-P Phosphatase in Cyanobakterien
 Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Einführung in die Biochemie Wirkungsweise von Enzymen
 Wirkungsweise von en Am Aktiven Zentrum kann ein nur in einer ganz bestimmten Orientierung anlegen, wie ein Schlüssel zum Schloss. Dieses Prinzip ist die Ursache der spezifität von en. Dies resultiert
Wirkungsweise von en Am Aktiven Zentrum kann ein nur in einer ganz bestimmten Orientierung anlegen, wie ein Schlüssel zum Schloss. Dieses Prinzip ist die Ursache der spezifität von en. Dies resultiert
2.1 Das prä-mrna Spleißen 3
 I 1. Zusammenfassung 1 2. Einleitung 3 2.1 Das prä-mrna Spleißen 3 2.1.1 Die Konsensussequenzen zweier Klassen von prä-mrna Introns 3 2.1.2 Die katalytischen Transesterifizierungsreaktionen 5 2.2 Das U2-abhängige
I 1. Zusammenfassung 1 2. Einleitung 3 2.1 Das prä-mrna Spleißen 3 2.1.1 Die Konsensussequenzen zweier Klassen von prä-mrna Introns 3 2.1.2 Die katalytischen Transesterifizierungsreaktionen 5 2.2 Das U2-abhängige
Michaelis-Menten-Gleichung
 Physikalisch-Chemische Praktika Michaelis-Menten-Gleichung Versuch K4 1 Aufgabe Experimentelle Bestimmung der Kinetik der Zersetzung von Harnsto durch Urease. 2 Grundlagen Im Bereich der Biochemie spielen
Physikalisch-Chemische Praktika Michaelis-Menten-Gleichung Versuch K4 1 Aufgabe Experimentelle Bestimmung der Kinetik der Zersetzung von Harnsto durch Urease. 2 Grundlagen Im Bereich der Biochemie spielen
Sophia Buhs. Dissertation. Diplom-Biochemikerin
 Beeinflussung Phosphotyrosin-abhängiger Signalwege in humanen unter Einsatz von SH2-Domänen und Phosphatasen in Kombination mit dem TAT-Transduktionssystem Dissertation Zur Erlangung der Würde des Doktors
Beeinflussung Phosphotyrosin-abhängiger Signalwege in humanen unter Einsatz von SH2-Domänen und Phosphatasen in Kombination mit dem TAT-Transduktionssystem Dissertation Zur Erlangung der Würde des Doktors
Physikalische Chemie
 Physikalische Chemie Prüfungstag 03.02.2017 Bitte beachten Sie Erlaubt sind 4 Seiten Zusammenfassung plus ein Periodensystem. Erlaubt ist ein Taschenrechner. Alle Hilfsmittel, die nicht explizit erlaubt
Physikalische Chemie Prüfungstag 03.02.2017 Bitte beachten Sie Erlaubt sind 4 Seiten Zusammenfassung plus ein Periodensystem. Erlaubt ist ein Taschenrechner. Alle Hilfsmittel, die nicht explizit erlaubt
BIOCHEMIE DES PHOSPHATS. Von Ramona Achorbagi und Natali Rabzewitsch
 BIOCHEMIE DES PHOSPHATS Von Ramona Achorbagi und Natali Rabzewitsch ENTDECKUNG DES PHOSPHORS 1669 entdeckte Henning Brand das Phosphor 1833 erste Zündhölzer 1845 fand man den stabileren roten Phosphor
BIOCHEMIE DES PHOSPHATS Von Ramona Achorbagi und Natali Rabzewitsch ENTDECKUNG DES PHOSPHORS 1669 entdeckte Henning Brand das Phosphor 1833 erste Zündhölzer 1845 fand man den stabileren roten Phosphor
Biochemische Übungen
 Dr. Arnulf Hartl Biochemische Übungen Proteine Tag 1: Tag 2: Tag 3: Konzentrieren Denaturierende und native Fällungen Protein Konzentrationsbestimmung Entsalzen Gelchromatographie Dialyse Elektrophorese
Dr. Arnulf Hartl Biochemische Übungen Proteine Tag 1: Tag 2: Tag 3: Konzentrieren Denaturierende und native Fällungen Protein Konzentrationsbestimmung Entsalzen Gelchromatographie Dialyse Elektrophorese
7. Zusammenfassung und Ausblick
 7. Zusammenfassung und Ausblick 7.1 Die Mitglieder der Rag-Familie Die verschiedenen Isoformen der Rag-Familie lagen, sofern sie rekombinant löslich isoliert wurden, als Dimer oder höhere Oligomere vor.
7. Zusammenfassung und Ausblick 7.1 Die Mitglieder der Rag-Familie Die verschiedenen Isoformen der Rag-Familie lagen, sofern sie rekombinant löslich isoliert wurden, als Dimer oder höhere Oligomere vor.
1. Wieso trägt Plastochinon zur Ansäuerung des Thylakoidlumens während der Photosynthese bei (bitte mit Zeichnung)?
 1. Wieso trägt Plastochinon zur Ansäuerung des Thylakoidlumens während der Photosynthese bei (bitte mit Zeichnung)? - Plastochinon nimmt 2 Elektronen auf der Stromaseite von PSII auf, wobei auch 2 H +
1. Wieso trägt Plastochinon zur Ansäuerung des Thylakoidlumens während der Photosynthese bei (bitte mit Zeichnung)? - Plastochinon nimmt 2 Elektronen auf der Stromaseite von PSII auf, wobei auch 2 H +
Die Wege des Kohlenstoffes
 Die Wege des Kohlenstoffes 1. Licht vs. Dunkelreaktionen Lichtgetriebene Reaktionen o Dunkle -Reaktionen laufen nicht im Dunklen ab reduzieren CO 2-Zucker für Pflanzen zum Wachsen und für uns zum Nutzen
Die Wege des Kohlenstoffes 1. Licht vs. Dunkelreaktionen Lichtgetriebene Reaktionen o Dunkle -Reaktionen laufen nicht im Dunklen ab reduzieren CO 2-Zucker für Pflanzen zum Wachsen und für uns zum Nutzen
VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN
 INHALTSVERZEICHNIS I VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VII VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN XIII 1 EINLEITUNG 1 1.1 Osmoadaption halophiler Mikroorganismen 2 1.1.1 Salt-in-cytoplasm
INHALTSVERZEICHNIS I VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VII VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN XIII 1 EINLEITUNG 1 1.1 Osmoadaption halophiler Mikroorganismen 2 1.1.1 Salt-in-cytoplasm
5 Kinetischen Untersuchungen
 5 Kinetischen Untersuchungen 5'-NT wird kinetisch seit mehr als 4 Jahren untersucht. Wie in der Einleitung beschrieben, sind hier besonders die Substratvielfalt und der Gebrauch der Metallionen als Kofaktoren
5 Kinetischen Untersuchungen 5'-NT wird kinetisch seit mehr als 4 Jahren untersucht. Wie in der Einleitung beschrieben, sind hier besonders die Substratvielfalt und der Gebrauch der Metallionen als Kofaktoren
Aus dem Lehrstuhl für Dermatologie Prof. Dr. med. M. Berneburg der Fakultät für Medizin der Universität Regensburg
 Aus dem Lehrstuhl für Dermatologie Prof. Dr. med. M. Berneburg der Fakultät für Medizin der Universität Regensburg UV-B Strahlung induzierte Singulett-Sauerstoff Erzeugung durch angeregte endogene Photosensibilisatoren
Aus dem Lehrstuhl für Dermatologie Prof. Dr. med. M. Berneburg der Fakultät für Medizin der Universität Regensburg UV-B Strahlung induzierte Singulett-Sauerstoff Erzeugung durch angeregte endogene Photosensibilisatoren
Biofunktionalisierung von metallischen Nanopartikeln
 Biofunktionalisierung von metallischen Nanopartikeln zur Entwicklung von Biosensoren Zur Erlangung des akademischen Grades eines DOKTORS DER NATURWISSENSCHAFTEN (Dr. rer. nat.) Fakultät für Chemie und
Biofunktionalisierung von metallischen Nanopartikeln zur Entwicklung von Biosensoren Zur Erlangung des akademischen Grades eines DOKTORS DER NATURWISSENSCHAFTEN (Dr. rer. nat.) Fakultät für Chemie und
Grundlagen der Physiologie
 Grundlagen der Physiologie Abbau eines Zuckermoleküls www.icbm.de/pmbio Lebensweise eines heterotrophen Aerobiers 1 Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien Anabolismus
Grundlagen der Physiologie Abbau eines Zuckermoleküls www.icbm.de/pmbio Lebensweise eines heterotrophen Aerobiers 1 Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien Anabolismus
Weitere Übungsfragen
 1 Strategie bei multiple choice Fragen Wie unterscheidet sich Glucose von Fructose? (2 Punkte) Glucose hat 6 C Atome, Fructose hat nur 5 C Atome. In der Ringform gibt es bei Glucose α und β Anomere, bei
1 Strategie bei multiple choice Fragen Wie unterscheidet sich Glucose von Fructose? (2 Punkte) Glucose hat 6 C Atome, Fructose hat nur 5 C Atome. In der Ringform gibt es bei Glucose α und β Anomere, bei
