Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion 2.0
|
|
|
- Erika Esser
- vor 8 Jahren
- Abrufe
Transkript
1 Züchtungskunde, 83, (4/5) S , 2011, ISSN Verlag Eugen Ulmer, Stuttgart Original Article Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion 2.0 R. Fries 1 und H. Pausch 1 Zusammenfassung Referenzsequenzen der Genome landwirtschaftlicher Nutztierarten stehen erst seit wenigen Jahren zur Verfügung. Sie haben jedoch zu unerwartet schnellen Veränderungen der Tierzuchtmethodik geführt. Die genomische Vorhersage von Zuchtwerten auf der Basis von anonymen Quantitative Trait Loci (QTL) und dichten Markerkarten anstelle (oder in Ergänzung zu) der Nachkommenprüfung ist beim Milchrind bereits weitgehend Realität. Wir bezeichnen diesen genomischen Ansatz zur züchterischen Verbesserung von Nutztieren als genomische Selektion 1.0. Die Sequenziermethodik entwickelt sich rasant weiter und ermöglicht nun die Sequenzierung der Genome einer größeren Anzahl von Tieren. Dadurch ergeben sich neue Möglichkeiten zur systematischen Identifizierung von DNA-Varianten, die der Variation züchterisch wichtiger Merkmale zu Grunde liegen, so genannten Quantitative Trait Nucleotides (QTN). QTN werden vorab für QTL mit einem maßgeblichen Beitrag zur quantitativ-genetischen Variation (very important QTL, viqtl) feststellbar sein. Die explizite Berücksichtigung von QTN im Bereich von viqtl ist die Grundlage der genomischen Selektion 2.0, die vor allem im Hinblick auf die züchterische Verbesserung von Gesundheitsmerkmalen von Bedeutung sein wird. Schlüsselwörter: Genomische Selektion, Genomsequenzierung, Sequenziertechnologie der nächsten Generation, Quantitative Trait Locus, Quantitative Trait Nucleotide Summary Individual genome sequencing of livestock animals en route to genomic selection 2.0 Since reference genome sequences of livestock species have become available few years ago, animal selection methodology is changing at an unexpected rate. Predicting breeding values on the basis of anonymous quantitative trait loci (QTL) tagged by densely spaced markers instead of (or supplementary to) progeny testing is widely applied in dairy cattle. We dub this approach to genomic animal improvement as genomic selection 1.0. The rapid developments of sequencing technologies now enable sequencing of numerous individuals and open new avenues to the systematic identification of DNA variants that affect quantitative traits, so called quantitative trait nucleotides (QTN). QTN will be explored mainly within QTL with large effects (very important QTL, viqtl). The explicit consideration of QTL within viqtl is the main feature of genomic 1 Lehrstuhl für Tierzucht der Technischen Universität München, Liesel-Beckmann-Straße 1, Freising-Weihenstephan, ruedi.fries@tum.de
2 372 R. Fries und H. Pausch selection 2.0, which will be particularly important to improving health traits by means of selection. Keywords: Genomic selection, genome sequencing, next-generation sequencing technology, quantitative trait locus, quantitative trait nucleotide 1 Einleitung Als in den achtziger Jahren des 20. Jahrhunderts die Sequenzierung von Genomen höherer Organismen zum ersten Mal vorgeschlagen wurde, wurden solche Pläne vielfach als Utopie abgetan. Manche warnten vor der Verschwendung von Forschungsgeldern für ein aussichtsloses Großprojekt. Die Entwicklung des ersten Sequenzierautomaten im Jahre 1986 durch Leroy Hood und Kollegen (Smith et al., 1986) und der breite Einsatz dieser Geräte in den neunziger Jahren des letzten Jahrhunderts ließ die Utopie trotz aller Unkenrufe Realität werden. Die Sequenzierung des Humangenoms wurde mit einem Artikel in einer neuen Zeitschrift mit dem Namen Genomics lanciert (Hood et al., 1987). Mit dieser Zeitschrift wurde auch die neue Disziplin Genomik begründet, die laut Editorial der ersten Ausgabe folgendes ist: The new discipline is borne from a marriage of molecular and cell biology with classical genetics and is fostered by computational science (McKusick and Ruddle, 1987). Zuerst wurden die Genome der Bäcker- und Bierhefe, des Fadenwurms und der Fruchtfliege sequenziert. Schon zur Jahrhundertwende (Juni 2000) wurde die Fertigstellung der Entwurfssequenz der ersten Wirbeltierart, nämlich des Menschen, in einer Zeremonie im Weißen Haus der USA bekannt gegeben. Im Jahr 2001 erfolgte die offizielle Publikation der Genomsequenz (International Human Genome Sequencing Consortium, 2001). Im gleichen Jahr wurde, damals kaum beachtet, die genomische Selektion bei Nutztieren vorgeschlagen (Meuwissen et al., 2001). Für die Umsetzung der genombasierten Selektion in die Tierzuchtpraxis mussten zwei weitere Meilensteine erreicht werden: die Verfügbarkeit der Genomsequenz von Nutztieren und die Möglichkeit, aus der Genomsequenzierung resultierende Sequenzvarianten als Single Nucleotide Polymorphisms (SNP) in großer Anzahl bei vielen Tieren typisieren zu können. Eine Entwurfssequenz des Rindes steht seit 2004 zur Verfügung, wurde aber erst 2008 in einer Publikation beschrieben (The Bovine Genome Sequencing and Analysis Consortium, 2008). Technologien zur Hochdurchsatztypisierung von SNP wurden beim Menschen 2005 zum ersten Mal eingesetzt (Klein et al., 2005). Im Folgenden werden wir ausführen, wie ein weiterer Technologiesprung es nun ermöglicht, die Sequenzen von vielen Individuen zu ermitteln und wie sich auf dieser Basis die genomische Selektion von der gegenwärtigen Version 1.0 in eine Version 2.0 entwickeln könnte. 2 Sequenziertechnologie der nächsten Generation Der Technologiesprung, der nun die Sequenzierung von individuellen Genomen in großer Zahl ermöglicht, basiert auf Sequenzierautomaten der nächsten Generation. Damit ist die zweite Generation von Sequenziergeräten gemeint (Tab. 1). Sequenzierautomaten der ersten Generation, die auf der Sequenzierung nach Sanger beruhen, waren entscheidend bei der Erstellung der Referenzsequenzen, d.h. der prototypischen Sequenzen einer ganzen Reihe von Arten. Wegen des relativ geringen Durchsatzes beliefen sich die Kosten der Sequenzierung des Genoms des Menschen auf mehrere Hundert Millionen Dollar. Die Erstellung der Referenzsequenz des Rindergenoms kostete
3 Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion Tab. 1. Generationen der Sequenziertechnologie Generations of sequencing technologies Generation Jahr der Einführung Gerät (Firma) ABI 370 (Applied Biosystems) GS20 (454 Life Sciences, Roche Diagnostics) GA I (Solexa, Illumina) SOLiD 1 (Agencourt, Beckmann, Life Technologies) 3* HeliScope (Helicos Biosciences) PacBio RS (Pacific Biosciences) * Anm.: Einzelmolekül-Sequenzierung etwa 40 Millionen Dollar. Mit den Geräten der zweiten Generation können nun die Kosten der Sequenzierung eines Genoms um mindestens das Tausendfache reduziert werden. Der hohe Durchsatz wird durch eine massive Parallelisierung der Sequenzierreaktion erreicht (siehe z.b. den Übersichtsartikel von Metzker (2010)). Das Ausmaß der Parallelisierung nimmt ständig zu, so dass sich die Kosten pro sequenzierter Base seit 2006 jeweils innerhalb weniger Monate halbieren. Diese Entwicklung ist dramatischer als jene der Halbleitertechnologie, wo sich die Komplexität integrierter Schaltkreise alle Monate verdoppelt (Mooresches Gesetz). In Tabelle 2 ist der Durchsatz für die wichtigsten Sequenziergeräte der zweiten Generation im Vergleich mit einem Gerät der ersten Generation dargestellt. Die erste individuelle Sequenz eines Menschen, nämlich des Genomikpioniers Craig Venter, wurde noch mit Sequenziertechnologie der ersten Generation erstellt (Levy et al., 2007). Bald darauf wurde die Sequenz des Genoms des Nobelpreisträgers James Watson mit Sequenziertechnologie der zweiten Generation ermittelt (Wheeler et al., 2008). Die erste individuelle Genomsequenz eines Nutztieres, nämlich des Fleckviehbullen Vanstein, wurde 2009 publiziert (Eck et al., 2009). Dabei wurden 2,4 Millionen Abweichungen von der Referenzsequenz des Rindes als so genannte Single Nucleotide Polymorphims (SNP) festgestellt. Im Rahmen des 1000-Genom-Projektes wurde die Sequenz von 179 menschlichen Individuen ermittelt (Durbin et al., 2010) und über 15 Millionen SNP identifiziert. Die SNP und kurze Einschübe und Ausfälle (Insertions-/Deletionsvarianten, kurz Indels ) werden durch Angleichung der Sequenzfrag- Tab. 2. Durchsatz verschiedener aktueller Sequenziergeräte Throughput of current sequencing machines Firma Fabrikat ABI ABI 3730* Roche GSFLX ABI SOLiD 4 Illumina HiSeq 2000 Basen/Lauf 70 Kb 400 Mb 300 Gb 600 Gb Leselänge 750 bp 400 bp 75 bp 100 bp Anm.: * Gerät der 1. Generation; Kb: Kilobasen (Tausend Basenpaare); Mb: Megabasen (Millionen Basenpaare; Gb: Gigabasen (Milliarden Basenpaare); bp: Basenpaare
4 374 R. Fries und H. Pausch mente an die Referenzsequenz festgestellt. Diese Fragmente betragen je nach Fabrikat und Version des Sequenziergerätes zwischen dreißig und mehreren hundert Basenpaaren. Ob eine Abweichung von der Referenzsequenz einen echten Polymorphismus darstellt oder als Artefakt verworfen werden muss, hängt von der so genannten Abdeckung ab. Je mehr Sequenzfragmente vorliegen, desto größer ist die Abdeckung. Ab Mai 2011 werden von den Geräten der Firma Illumina pro Spur mehr als 300 Millionen Sequenzfragmente zu 100 Basen produziert, also 30 Milliarden Basen. Bei einer Länge des haploiden Genoms von drei Milliarden Basen ergibt sich eine Abdeckung von mehr als 10 aus den in einer Spur generierten Sequenzfragmenten, d.h. jede Base wird im Durchschnitt 10 gelesen. Mit einer Abdeckung von 7 und mehr können mehr als 98% der SNP richtig festgestellt werden (Aigner et al., 2011). 3 Genomische Vorhersage Version 1.0 Die genomische Selektion beruht auf der Vorhersage des genomischen Zuchtwertes eines Tieres, d.h. der Schätzung der allelischen Zustände von sehr vielen Quantitative Trait Loci (QTL). QTL wurde übrigens vom deutschen Tierzuchtforscher Hermann Geldermann geprägt (Geldermann, 1975). Da die genauen genomischen Koordinaten der QTL nicht bekannt sind, erfolgt diese Vorhersage indirekt über Marker (SNP), die in der Nähe der QTL liegen. Entscheidend für eine möglichst zuverlässige Schätzung der QTL-Allele ist ein hohes Kopplungsungleichgewicht zwischen QTL- und Markerlocus, so dass ein bestimmtes Marker-Allel das entsprechende QTL-Allel mit hoher Sicherheit vorhersagt. Das Kopplungsungleichgewicht zwischen Markern und QTL wird durch die Abstände der Marker sowie von Populationsparametern bestimmt. Wenn eine Population auf relativ wenig Ahnen zurückgeht, welche die gegenwärtige Population vor wenigen Generationen maßgeblich beeinflussten, ist das Kopplungsungleichgewicht von Markern und QTL hoch. Diese Situation trifft für die meisten züchterisch bearbeiteten Rinderpopulationen zu. Die QTL-Allele können in diesem Fall mit relativ wenig Markern zuverlässig vorhergesagt werden. Beim Rind wird die genomische Selektion inzwischen routinemäßig angewendet. Es werden dazu SNP des Bovine SNP50 Genotyping Bead- Chip der Firma Illumina typisiert, wovon etwa in die Analyse eingehen. Die Beziehung von Marker- und QTL-Allelen (genomische Zuchtwertformel) wird bei Bullen ermittelt, für die möglichst sichere, konventionell geschätzte Zuchtwerte vorliegen. Die Sicherheit der genomischen Zuchtwertschätzung hängt von der Anzahl Tiere mit gut geschätzten Zuchtwerten ab, die für die Ermittlung der genomischen Zuchtwertformel herangezogen werden können. Beim Rind werden etwa Bullen benötigt, um Sicherheiten von genomischen Zuchtwerten zu erzielen, die beträchtlich über denjenigen der Pedigree-Zuchtwerte liegen. Dieses Erfordernis kann bei kleineren Populationen kaum erfüllt werden. Eine Übertragung der genomischen Zuchtwertschätzformel von großen Populationen auf kleine Populationen ist jedoch nur bei einer sehr hohen Markerdichte zielführend, denn nur unter dieser Voraussetzung sind die Marker-Allel-QTL-Allelkombinationen über die Populationen hinweg konstant. Inzwischen stehen für das Rind zwei Produkte zur Genotypisierung zur Verfügung, die (BovineHD Genotyping Bead Chip, Illumina) bzw (Axiom TM Genome-Wide BOS 1 Array Plate, Affymetrix) polymorphe Markerpositionen abdecken. Die Markerdichte ist somit um mehr als das Zehnfache höher als für den zur Zeit am häufigsten verwendeten Chip der Firma Illumina mit nominell Markern (BovineSNP50 Genotyping Bead Chip). Zurzeit wird untersucht, ob und in welchem Ausmaß die höhere Markerdichte eine Übertragung der genomischen Zuchtwertformeln von großen auf kleine Populationen ermöglicht. Die genomische Selektion auf der Basis einer hochdichten Markerabdeckung, die neben der Über-
5 Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion tragbarkeit der Schätzformeln auch zu etwas höheren Sicherheiten der genomischen Zuchtwertschätzung führt, könnte man als genomische Selektion 1.5 bezeichnen. 4 Genomische Vorhersage Version 2.0 Bei den erwähnten Ansätzen zur genomischen Zuchtwertschätzung werden die QTL als anonyme Größen betrachtet, die je nach Markerdichte und Populationstruktur mehr oder weniger gut durch die Marker vertreten werden (= genomische Vorhersage 1.0 bis 1.5 ). Das Ziel ist jedoch, möglichst die kausale DNA-Variation von Merkmalsvariation festzustellen. Kausale DNA-Varianten werden auch als so genannte QTN (quantitative trait nucleotides) bezeichnet (Long et al. (1998); Weller and Ron (2011)). Mit der Identifizierung eines QTN im Bereich eines QTL wird der Schleier seiner Anonymität gelüftet. Es wird offenbart, welches Gen, welches Genprodukt und welchen Stoffwechselweg die kausale Variante betrifft. Des Weiteren kann auch die Frequenz der im Hinblick auf ein Merkmal positiven bzw. negativen Variante in jeder interessierenden Population genau ermittelt werden. Kürzlich wurde in den Rassen Jersey und Holstein-Friesain ein QTN für die Körpergröße identifiziert (Karim et al., 2011). Es handelt sich um eine Variante, die sich auf die Regulation mehrerer benachbarter Gene auswirkt, deren Produkte Wachstumsfaktoren beeinflussen. Wir gehen davon aus, dass ein QTL für den paternalen Kalbeverlauf beim Fleckvieh und damit wahrscheinlich für fötales Wachstum ebenfalls im Bereich dieser Gene lokalisiert ist, und dass das für die Körpergröße verantwortliche QTN auch für den Kalbeverlauf beim Fleckvieh verantwortlich ist (Pausch et al., 2011). Es wird nicht möglich sein, für alle QTL, die ein Merkmal beeinflussen, QTN zu identifizieren. Es stellt sich jedoch heraus, dass für die meisten Merkmale einige wenige QTL einen großen Teil der Variation erklären, während sehr viele Loci je nur sehr wenig zur gesamten genetischen Variation beitragen (Abb. 1 und Abb. 2). Wir bezeichnen die QTL mit einem großen Effekt als very important QTL (viqtl). Für viele viqtl wird die Identifizierung der relevanten QTN möglich sein. Hier kommt nun die individuelle Genomsequenzierung ins Spiel. Obwohl die Kosten für die Re-Sequenzierung eines Individuums sich inzwischen nur noch auf wenige Tausend Euro belaufen, ist die Sequenzierung ganzer Populationen keine realistische Option. Durch die Sequenzierung von einflussreichen Ahnentieren einer Population kann die individuelle Sequenz jedes Tieres dieser Population mit einer großen Zuverlässigkeit durch Imputation (Hochrechnung) via hochdichter Genotypisierung ermittelt werden. Wir haben beim Fleckvieh die Sequenz von über Tieren auf der Basis von 23 bei einer durchschnittlich sechsfachen Abdeckung re-sequenzierter Gründertiere imputiert (Aigner et al., 2011). Total wurden mehr als 15 Millionen variable Positionen identifiziert. Durch die Imputation können die Genotypen an diesen Positionen mit einer Sicherheit von mindestens 90% angegeben werden. Man kann davon ausgehen, dass mit den 15 Millionen variablen Position bereits der Großteil der genomischen Variation der Fleckviehrasse und somit auch die QTN der viqtl erfasst werden. In Abbildung 3 ist das Resultat einer Assoziationsstudie im Bereich eines viqtl für den Proteingehalt der Milch dargestellt. Unter den hoch assoziierten Markern dürfte auch die Position vertreten sein, die den QTN ausmacht. Es wird nicht trivial sein, unter mehreren Markern, die sich zueinander in hohem Kopplungsungleichgewicht befinden, die QTN-Position oder die QTN-Positionen auszumachen, zumal es sich abzeichnet, dass für quantitative Variation verantwortliche DNA-Variation eher im regulatorischen als im kodierenden Bereich der Gene lokalisiert ist. Wenn z.b. Enhancer-Regionen betroffen sind, können sich kausale Varianten mehrere zehntausend Basenpaare entfernt vom betroffenen Gen befinden. Es kann auch sein, dass mehrere Positionen
6 376 R. Fries und H. Pausch Abb. 1. Assoziation von Single Nucleotide Polymorphisms (SNP) mit dem Zuchtwert für Eiweiß-Prozent von 3579 nachkommengeprüften Fleckvieh-Bullen. Zur Berücksichtigung der Populationsstratifikation wurde ein gemischtes Modell verwendet, in dem die genomische Verwandtschaft (Van Raden, 2008) berücksichtigt wurde. Offene Symbole repräsentieren genomweit signifikant assoziierte SNP. Association of 654,639 single nucleotide polymorphisms (SNPs) with the estimated breeding values for protein-percentage in 3,579 progeny-tested Fleckvieh bulls. The genomic relationship (Van Raden, 2008) was considered in a mixed model based approach to account for population stratification. Open symbols represent significantly associated SNP on a genome-wide level. für die Variation eines viqtl verantwortlich sind (Bickel et al., 2011). Assoziationsstudien auf der Basis der imputierten Sequenz werden in den meisten Fällen zu einer starken Eingrenzung der QTN-Region(en) eines viqtl führen (siehe auch Abb. 3). Populationsübergreifende Analysen und die vergleichende Genomik werden bei der Identifizierung von QTN eine wichtige Rolle spielen. Mit der Typisierung einiger weniger Kandidaten-QTN kann der viqtl-genotyp jedes Tieres als Diplotyp (= Haplotypen-Genotyp) mit großer Zuverlässigkeit vorhergesagt werden, ohne dass die kausale Rolle der QTN eindeutig nachgewiesen ist. Die explizite Berücksichtigung von Genotypen an viqtl (zusätzlich zu den genomweit ermittelten Zuchtwerten) ist die Grundlage der genomischen Selektion 2.0. Die Genotypen-Information von viqtl für ein bestimmtes Merkmal und ein bestimmtes Tier kann z.b. als Verhältnis der Anzahl positiver Allele zur Gesamtzahl Allele an den viqtl angegeben werden. Die direkte Bestimmung des QTL-Genotypes ermöglicht auch die Identifizierung nicht-additiver Allel-Effekte, indem nicht Zuchtwerte, sondern phänotypische Werte in die Assoziationsstudien einbezogen werden. Die Berücksichtigung pleiotroper Effekte von viqtl kann für die Auflösung unerwünschter Korrelationen von Bedeutung sein. Um effizienter auf einen hohen Eiweißgehalt der Milch bei gleich bleibendem oder abnehmendem Fettgehalt zu züchten, könnte man Tiere mit möglichst vielen viqtl-allelen mit einem stark positiven Effekt auf den Eiweißgehalt aber einem geringen positiven oder sogar negativen Effekt auf den Fettgehalt selektieren. Beim Rind wird die genomische Selektion 2.0 vor allem bei der Remontierung von Kühen und im Hinblick auf Gesundheitsmerkmale von Bedeutung sein. Dadurch, dass die Marker sehr nahe am QTN positioniert sind oder die QTN direkt bekannt sind, ist die Nutzung von viqtl-information nicht auf die Rasse beschränkt, in der ein viqtl initial festgestellt wurde. Es kann
7 Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion Abb. 2. Der Beitrag der einzelnen Chromosomen zur Zuchtwertvarianz von Fleckvieh Bullen in Abhängigkeit der Chromosomenlänge. Schwarzen Nummern stehen für Chromosomen mit identifizierten QTL für Eiweiß-Prozent. Die graue Linie ist die Regressionsgerade mit ß=2, (r 2 = 0,11). Der Anteil der erklärten Zuchtwert-Varianz wurde simultan für alle Chromosomen mit GCTA (Yang et al., 2011) mit folgendem Modell geschätzt: y 30 = g i + e, i = 1 wobei y der Vektor der Zuchtwerte für Eiweiß-Prozent und g i der Vektor der genetischen Effekte des i-ten Chromosoms ist. g i ist normalverteilt 2 N( 0, G i σ gi ), wobei G i die genomische Verwandtschaftsmatrix basierend auf Single Nucleotide Polymorphisms (SNP) des i-ten Chromosoms ist (Van Raden, 2008). The proportion of EBV variance of 3,579 Fleckvieh bulls attributable to a particular chromosome as a function of the chromosome length. Black numbers indicate chromosomes with identified QTL. The grey line is a regression line with slope (r 2 = 0.11). The proportion of EBV variance explained was estimated with the effects of all chromosomes estimated simultaneously using GCTA (Yang et al., 2011) to fit the model: y 30 = g i + e, i = 1 where y is a vector of EBVs for protein percentage, g i is a vector of genetic effects attributable to the i th chromosome. g i is normally distributed 2 N( 0, G i σ gi ), where G i is the genomic relationship matrix built based on single nucleotide polymorphisms (SNP) on the i th chromosome (Van Raden, 2008). sein, dass in einer Rasse ein QTL nicht als viqtl festgestellt werden kann, da die Frequenz des seltenen Allels sehr niedrig ist. Die in einer Rasse gewonnene viqtl-information kann aber zum Aufspüren von Trägern des seltenen Allels in einer anderen Rasse genutzt werden.
8 378 R. Fries und H. Pausch Abb. 3. Detaillierte Übersicht des Eiweiß-Prozent QTL auf Chromosom 6 aus der in Abb. 1 genomweiten Assoziationsstudie. Im Bereich von 87 87,5 Mb lieferte die Sequenzierung von 23 Gründerbullen der Fleckviehrasse insgesamt Single Nucleotide Polymorphisms (SNP), die mit einer Kombination aus Beagle (Browning and Browning, 2009) und MaCH (Li et al., 2010) für insgesamt Fleckvieh Bullen imputiert wurden (A). Mit der 777K-Typisierung werden in diesem Bereich Genotypen für 121 SNP abgefragt (B). Das Kopplungsungleichgewicht (r 2 ) mit dem am stärksten assoziierten SNP wird durch die verschiedenen Grautöne dargestellt. Die Annotation der SNP und Gene basiert auf dem UMD3.1 Assembly des Rindergenoms (Zimin et al., 2009). Detailed view of the protein-percentage QTL on chromosome 6 from the genome-wide association study in Fig. 1. Whole-genome sequencing of 23 founder bulls resulted in the detection of 3,531 SNP, which were imputed for 3,579 Fleckvieh bulls applying Beagle (Browning and Browning, 2009) and MaCH (Li et al., 2010) (A). 777K-genotyping provided 121 SNP within the respective region (B). Different shades of grey indicate the linkage disequilibrium (r 2 ) with the most significantly associated SNP. SNP and genes were annotated based on the UMD3.1 assembly of the bovine genome (Zimin et al., 2009).
9 Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion Schlussfolgerungen und Ausblick Die Genomik hat sich innerhalb weniger Jahre zu einer Schlüsseldisziplin der Tierzuchtforschung entwickelt. Diese Entwicklung kulminiert zunächst in der genomischen Selektion, die auf der Referenzsequenz und davon abgeleiteten Sequenzvarianten sowie Technologien der Hochdurchsatzgenotypisierung basiert. Das Potenzial der genomischen Selektion in ihrer ersten Version wird zurzeit nur beim Milchrind konsequent genutzt. Bei anderen Arten fehlen zum Teil die genomischen Ressourcen und/oder die erwarteten Vorteile sind z.b. wegen der kurzen Generationsdauer nicht so prägnant wie beim Rind. Die relativ kostengünstige Re-Sequenzierung von Individuen eröffnet nun neue Perspektiven für die Weiterentwicklung der genomischen Vorhersage beim Rind und wird bei anderen Tierarten erst die Voraussetzungen für die genomische Selektion schaffen. Nachdem Genomik und quantitative Genetik über viele Jahre rivalisierende Disziplinen darstellten, haben sich die Grenzen der beiden Disziplinen weitgehend aufgelöst. Die genomischen Befunde bestätigen das im Hinblick auf Zuchtfortschritt bei Tieren und Pflanzen sehr erfolgreiche quantitativ-genetische Modell, nämlich dass die allelische Variation an sehr vielen (anonymen) Loci für die quantitativ-genetische Variation verantwortlich ist. Die genomische Selektion in ihrer ersten Implementierung ist mit diesem Modell ohne Abstriche kompatibel, eine Tatsache, welche die hohe Akzeptanz des genomischen Ansatzes in der Tierzucht erklärt. Die Fortschritte in der Genomtechnologie, die in ihrer Dynamik die Entwicklung der Computertechnologie übertreffen, werden es nun ermöglichen, den Schleier der Anonymität über vielen züchterisch wichtigen Genorten zu lüften. Die resultierenden Erkenntnisse werden vor allem für die genetische Verbesserung der Gesundheit von Nutztieren neue Möglichkeiten eröffnen und damit maßgeblich zu einer nachhaltigen, tiergerechten Produktion von Lebensmitteln tierischer Herkunft beitragen. 6 Danksagung Assoziations- und Re-Sequenzierungsdaten, die den Abb. 1 3 zu Grunde liegen, wurden im Rahmen des FUGATO-plus Projektes GenoTrack sowie des Kompetenznetzes Synbreed erarbeitet. Wir bedanken uns beim Deutschen Bundesministerium für Bildung und Forschung für die finanzielle Unterstützung (Förderkennzeichen: D und B). Literatur Aigner, B., S. Jansen, H. Pausch, M. Wysocki, S. Eck, A. Benet-Pagès, T. Wieland, T. Strom, T. Meitinger and R. Fries, (2011): Assessment of the genomic variation in the Fleckvieh breed by re-sequencing at low to medium coverage. In preparation. Bickel, R.D., A. Kopp and S.V. Nuzhdin, (2011): Composite Effects of Polymorphisms near Multiple Regulatory Elements Create a Major-Effect QTL. PLoS Genet 7, e Browning, B.L. and S.R. Browning, (2009): A unified approach to genotype imputation and haplotype-phase inference for large data sets of trios and unrelated individuals. Am. J. Hum. Genet. 84, Durbin, R.M., G.R. Abecasis, D.L. Altshuler, A. Auton, L.D. Brooks, R.M. Durbin, R.A. Gibbs, M.E. Hurles and G.A. McVean, (2010): A map of human genome variation from population-scale sequencing. Nature 467, Eck, S., A. Benet-Pages, K. Flisikowski, T. Meitinger, R. Fries and T. Strom, (2009): Whole genome sequencing of a single Bos taurus animal for SNP discovery. Genome Biology 10, R82.
10 380 R. Fries und H. Pausch Geldermann, H., (1975): Investigations on inheritance of quantitative characters in animals by gene markers. 1. Methods. Theoretical and Applied Genetics 46, Hood, L.E., M.W. Hunkapiller and L.M. Smith, (1987): Automated DNA sequencing and analysis of the human genome. Genomics 1, International Human Genome Sequencing Consortium, (2001): Initial sequencing and analysis of the human genome. Nature 409, Karim, L., H. Takeda, L. Lin, T. Druet, J.A.C. Arias, D. Baurain, N. Cambisano, S.R. Davis, F. Farnir, B. Grisart, B.L. Harris, M.D. Keehan, M.D. Littlejohn, R.J. Spelman, M. Georges and W. Coppieters, (2011): Variants modulating the expression of a chromosome domain encompassing PLAG1 influence bovine stature. Nat. Genet. 43, Klein, R.J., C. Zeiss, E.Y. Chew, J.-Y. Tsai, R.S. Sackler, C. Haynes, A.K. Henning, J.P. SanGiovanni, S.M. Mane, S.T. Mayne, M.B. Bracken, F.L. Ferris, J. Ott, C. Barnstable and J. Hoh., (2005): Complement Factor H Polymorphism in Age-Related Macular Degeneration. Science 308, Levy, S., G. Sutton, P.C. Ng, L. Feuk, A.L. Halpern, B.P. Walenz, N. Axelrod, J. Huang, E.F. Kirkness, G. Denisov, Y. Lin, J.R. MacDonald, A.W.C. Pang, M. Shago, T.B. Stockwell, A. Tsiamouri, V. Bafna, V. Bansal, S.A. Kravitz, D.A. Busam, K.Y. Beeson, T.C. McIntosh, K.A. Remington, J.F. Abril, J. Gill, J. Borman, Y.-H. Rogers, M.E. Frazier, S.W. Scherer, R.L. Strausberg and J.C. Venter, (2007): The Diploid Genome Sequence of an Individual Human. PLoS Biol. 5, e254. Li, Y., C.J. Willer, J. Ding, P. Scheet and G.R. Abecasis, (2010): MaCH: using sequence and genotype data to estimate haplotypes and unobserved genotypes. Genet. Epidemiol. 34, Long, A.D., R.F. Lyman, C.H. Langley and T.F. Mackay, (1998): Two sites in the Delta gene region contribute to naturally occurring variation in bristle number in Drosophila melanogaster. Genetics 149, McKusick, V.A. and F.H. Ruddle, (1987): A new discipline, a new name, a new journal. Genomics 1, 1 2. Metzker, M.L., (2010): Sequencing technologies the next generation. Nat. Rev. Genet. 11, Meuwissen, T.H., B.J. Hayes and M.E. Goddard, (2001): Prediction of total genetic value using genome-wide dense marker maps. Genetics 157, Pausch, H., K. Flisikowski, S. Jung, R. Emmerling, C. Edel, K.-U. Götz and R. Fries, (2011): Genome-wide association study identifies two major loci affecting calving ease and growth-related traits in cattle. Genetics 187, Smith, L.M., J.Z. Sanders, R.J. Kaiser, P. Hughes, C. Dodd, C.R. Connell, C. Heiner, S.B. Kent and L.E. Hood, (1986): Fluorescence detection in automated DNA sequence analysis. Nature 321, The Bovine Genome Sequencing and Analysis Consortium, (2008): The Genome Sequence of Taurine Cattle: A Window to Ruminant Biology and Evolution. Science 342, Van Raden, P.M., (2008): Efficient Methods to Compute Genomic Predictions. J. Dairy Sci. 91, Weller, J.I. and M. Ron, (2011): Invited review: Quantitative trait nucleotide determination in the era of genomic selection. J. Dairy Sci. 94, Wheeler, D.A., M. Srinivasan, M. Egholm, Y. Shen, L. Chen, A. McGuire, W. He, Y.-J. Chen, V. Makhijani, G.T. Roth, X. Gomes, K. Tartaro, F. Niazi, C.L. Turcotte, G.P. Irzyk, J.R. Lupski, C. Chinault, X.-Z. Song, Y. Liu, Y. Yuan, L. Nazareth, X. Qin, D.M. Muzny, M. Margulies, G.M. Weinstock, R.A. Gibbs and J.M. Rothberg, (2008): The complete genome of an individual by massively parallel DNA sequencing. Nature 452,
11 Individuelle Genomsequenzierung von Nutztieren auf dem Weg zur genomischen Selektion Yang, J., S.H. Lee, M.E. Goddard and P.M. Visscher, (2011): GCTA: a tool for genome-wide complex trait analysis. Am. J. Hum. Genet 88, Zimin, A.V., A.L. Delcher, L. Florea, D.R. Kelley, M.C. Schatz, D. Puiu, F. Hanrahan, G. Pertea, C.P. Van Tassell, T.S. Sonstegard, G. Marçais, M. Roberts, P. Subramanian, J.A. Yorke and S.L. Salzberg, (2009): A whole-genome assembly of the domestic cow, Bos taurus. Genome Biol. 10, R42.
Individuelle Genomsequenzen Aussichten für die Tierzucht
 Individuelle Genomsequenzen Aussichten für die Tierzucht Ruedi Fries WZW / Lehrstuhl für Tierzucht Agrarw. Symposium - 1 Auswirkungen der Genomischen Revolution auf die Tierzucht (und Pflanzenzucht) Agrarw.
Individuelle Genomsequenzen Aussichten für die Tierzucht Ruedi Fries WZW / Lehrstuhl für Tierzucht Agrarw. Symposium - 1 Auswirkungen der Genomischen Revolution auf die Tierzucht (und Pflanzenzucht) Agrarw.
Foto: Kate Whitley, www.biotechnologie.de
 Foto: Kate Whitley, www.biotechnologie.de Inhalt o Was kann die genomische ZWS nicht? o QTL: Erfahrungen aus genomweiten Studien o Begriffklärung [Re-]Sequenzierung o Hochdurchsatzsequenzierung technische
Foto: Kate Whitley, www.biotechnologie.de Inhalt o Was kann die genomische ZWS nicht? o QTL: Erfahrungen aus genomweiten Studien o Begriffklärung [Re-]Sequenzierung o Hochdurchsatzsequenzierung technische
Genomische Selektion in der Pflanzenzüchtung
 Genomische Selektion in der Pflanzenzüchtung Chris-CarolinCarolin Schön 30. März 2012 Hybridzüchtung Population 1 Population 2 Kreuzungen 10.000000 Linieni Prüfung 1 Umwelt Kreuzungen 10.000000 Linen 2.000
Genomische Selektion in der Pflanzenzüchtung Chris-CarolinCarolin Schön 30. März 2012 Hybridzüchtung Population 1 Population 2 Kreuzungen 10.000000 Linieni Prüfung 1 Umwelt Kreuzungen 10.000000 Linen 2.000
Neue DNA Sequenzierungstechnologien im Überblick
 Neue DNA Sequenzierungstechnologien im Überblick Dr. Bernd Timmermann Next Generation Sequencing Core Facility Max Planck Institute for Molecular Genetics Berlin, Germany Max-Planck-Gesellschaft 80 Institute
Neue DNA Sequenzierungstechnologien im Überblick Dr. Bernd Timmermann Next Generation Sequencing Core Facility Max Planck Institute for Molecular Genetics Berlin, Germany Max-Planck-Gesellschaft 80 Institute
Voraussetzungen für die Genomische Selektion beim Pferd
 Agrar- und Ernährungswissenschaftliche Fakultät Institut für Tierzucht und Tierhaltung Voraussetzungen für die Genomische Selektion beim Pferd Prof. Dr. Georg Thaller 32. Jahrestagung zur Pferdegesundheit
Agrar- und Ernährungswissenschaftliche Fakultät Institut für Tierzucht und Tierhaltung Voraussetzungen für die Genomische Selektion beim Pferd Prof. Dr. Georg Thaller 32. Jahrestagung zur Pferdegesundheit
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren
 Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
geben. Die Wahrscheinlichkeit von 100% ist hier demnach nur der Gehen wir einmal davon aus, dass die von uns angenommenen
 geben. Die Wahrscheinlichkeit von 100% ist hier demnach nur der Vollständigkeit halber aufgeführt. Gehen wir einmal davon aus, dass die von uns angenommenen 70% im Beispiel exakt berechnet sind. Was würde
geben. Die Wahrscheinlichkeit von 100% ist hier demnach nur der Vollständigkeit halber aufgeführt. Gehen wir einmal davon aus, dass die von uns angenommenen 70% im Beispiel exakt berechnet sind. Was würde
Professionelle Seminare im Bereich MS-Office
 Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
UNIVERSITÄTSKLINIKUM Schleswig-Holstein. Sequenzierung. Norbert Arnold. Dept. Gynecology and Obstetrics Oncology Laboratory
 Sequenzierung Norbert Arnold Neue Sequenziertechnologien (seit 2005) Next Generation Sequenzierung (NGS, Erste Generation) Unterschiedliche Plattformen mit unterschiedlichen Strategien Roche 454 (basierend
Sequenzierung Norbert Arnold Neue Sequenziertechnologien (seit 2005) Next Generation Sequenzierung (NGS, Erste Generation) Unterschiedliche Plattformen mit unterschiedlichen Strategien Roche 454 (basierend
Grundlagen der genomische Selektion - Was sagt ein genomischer Zuchtwert aus?
 Gliederung Seminar des Ausschusses für Genetik der ZAR, Salzburg, 18.03.2010 Grundlagen der genomische Selektion - Was sagt ein genomischer Zuchtwert aus? Versuch einer Begriffserklärung Wie weiß man,
Gliederung Seminar des Ausschusses für Genetik der ZAR, Salzburg, 18.03.2010 Grundlagen der genomische Selektion - Was sagt ein genomischer Zuchtwert aus? Versuch einer Begriffserklärung Wie weiß man,
Kulturelle Evolution 12
 3.3 Kulturelle Evolution Kulturelle Evolution Kulturelle Evolution 12 Seit die Menschen Erfindungen machen wie z.b. das Rad oder den Pflug, haben sie sich im Körperbau kaum mehr verändert. Dafür war einfach
3.3 Kulturelle Evolution Kulturelle Evolution Kulturelle Evolution 12 Seit die Menschen Erfindungen machen wie z.b. das Rad oder den Pflug, haben sie sich im Körperbau kaum mehr verändert. Dafür war einfach
Würfelt man dabei je genau 10 - mal eine 1, 2, 3, 4, 5 und 6, so beträgt die Anzahl. der verschiedenen Reihenfolgen, in denen man dies tun kann, 60!.
 040304 Übung 9a Analysis, Abschnitt 4, Folie 8 Die Wahrscheinlichkeit, dass bei n - maliger Durchführung eines Zufallexperiments ein Ereignis A ( mit Wahrscheinlichkeit p p ( A ) ) für eine beliebige Anzahl
040304 Übung 9a Analysis, Abschnitt 4, Folie 8 Die Wahrscheinlichkeit, dass bei n - maliger Durchführung eines Zufallexperiments ein Ereignis A ( mit Wahrscheinlichkeit p p ( A ) ) für eine beliebige Anzahl
Behörde für Bildung und Sport Abitur 2008 Lehrermaterialien zum Leistungskurs Mathematik
 Abitur 8 II. Insektenpopulation LA/AG In den Tropen legen die Weibchen einer in Deutschland unbekannten Insektenpopulation jedes Jahr kurz vor Beginn der Regenzeit jeweils 9 Eier und sterben bald darauf.
Abitur 8 II. Insektenpopulation LA/AG In den Tropen legen die Weibchen einer in Deutschland unbekannten Insektenpopulation jedes Jahr kurz vor Beginn der Regenzeit jeweils 9 Eier und sterben bald darauf.
Welche Unterschiede gibt es zwischen einem CAPAund einem Audiometrie- Test?
 Welche Unterschiede gibt es zwischen einem CAPAund einem Audiometrie- Test? Auch wenn die Messungsmethoden ähnlich sind, ist das Ziel beider Systeme jedoch ein anderes. Gwenolé NEXER g.nexer@hearin gp
Welche Unterschiede gibt es zwischen einem CAPAund einem Audiometrie- Test? Auch wenn die Messungsmethoden ähnlich sind, ist das Ziel beider Systeme jedoch ein anderes. Gwenolé NEXER g.nexer@hearin gp
ONLINE-AKADEMIE. "Diplomierter NLP Anwender für Schule und Unterricht" Ziele
 ONLINE-AKADEMIE Ziele Wenn man von Menschen hört, die etwas Großartiges in ihrem Leben geleistet haben, erfahren wir oft, dass diese ihr Ziel über Jahre verfolgt haben oder diesen Wunsch schon bereits
ONLINE-AKADEMIE Ziele Wenn man von Menschen hört, die etwas Großartiges in ihrem Leben geleistet haben, erfahren wir oft, dass diese ihr Ziel über Jahre verfolgt haben oder diesen Wunsch schon bereits
Einblicke in das menschliche Erbgut (Genom) am Computer
 Einblicke in das menschliche Erbgut (Genom) am Computer Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de Binger Nacht der Wissenschaft - 16.04.2010 Das menschliche Genom ist entschlüsselt
Einblicke in das menschliche Erbgut (Genom) am Computer Prof. Dr. Antje Krause FH Bingen 06721 / 409 253 akrause@fh-bingen.de Binger Nacht der Wissenschaft - 16.04.2010 Das menschliche Genom ist entschlüsselt
Das große ElterngeldPlus 1x1. Alles über das ElterngeldPlus. Wer kann ElterngeldPlus beantragen? ElterngeldPlus verstehen ein paar einleitende Fakten
 Das große x -4 Alles über das Wer kann beantragen? Generell kann jeder beantragen! Eltern (Mütter UND Väter), die schon während ihrer Elternzeit wieder in Teilzeit arbeiten möchten. Eltern, die während
Das große x -4 Alles über das Wer kann beantragen? Generell kann jeder beantragen! Eltern (Mütter UND Väter), die schon während ihrer Elternzeit wieder in Teilzeit arbeiten möchten. Eltern, die während
Eva Douma: Die Vorteile und Nachteile der Ökonomisierung in der Sozialen Arbeit
 Eva Douma: Die Vorteile und Nachteile der Ökonomisierung in der Sozialen Arbeit Frau Dr. Eva Douma ist Organisations-Beraterin in Frankfurt am Main Das ist eine Zusammen-Fassung des Vortrages: Busines
Eva Douma: Die Vorteile und Nachteile der Ökonomisierung in der Sozialen Arbeit Frau Dr. Eva Douma ist Organisations-Beraterin in Frankfurt am Main Das ist eine Zusammen-Fassung des Vortrages: Busines
Stellen Sie bitte den Cursor in die Spalte B2 und rufen die Funktion Sverweis auf. Es öffnet sich folgendes Dialogfenster
 Es gibt in Excel unter anderem die so genannten Suchfunktionen / Matrixfunktionen Damit können Sie Werte innerhalb eines bestimmten Bereichs suchen. Als Beispiel möchte ich die Funktion Sverweis zeigen.
Es gibt in Excel unter anderem die so genannten Suchfunktionen / Matrixfunktionen Damit können Sie Werte innerhalb eines bestimmten Bereichs suchen. Als Beispiel möchte ich die Funktion Sverweis zeigen.
Zeichen bei Zahlen entschlüsseln
 Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Large Scale SNP Daten beim Pferd: Erste Erfahrungen und Anwendungen
 Fotos: M. Rindlisbacher Large Scale SNP Daten beim Pferd: Erste Erfahrungen und Anwendungen H. Hasler 1, F. Berruex 3, B. Haase 2, C. Flury 1, T. Leeb 2, V. Gerber 4, H. Simianer 5, D. Burger 3, P.A. Poncet
Fotos: M. Rindlisbacher Large Scale SNP Daten beim Pferd: Erste Erfahrungen und Anwendungen H. Hasler 1, F. Berruex 3, B. Haase 2, C. Flury 1, T. Leeb 2, V. Gerber 4, H. Simianer 5, D. Burger 3, P.A. Poncet
Lineare Gleichungssysteme
 Lineare Gleichungssysteme 1 Zwei Gleichungen mit zwei Unbekannten Es kommt häufig vor, dass man nicht mit einer Variablen alleine auskommt, um ein Problem zu lösen. Das folgende Beispiel soll dies verdeutlichen
Lineare Gleichungssysteme 1 Zwei Gleichungen mit zwei Unbekannten Es kommt häufig vor, dass man nicht mit einer Variablen alleine auskommt, um ein Problem zu lösen. Das folgende Beispiel soll dies verdeutlichen
DAS PARETO PRINZIP DER SCHLÜSSEL ZUM ERFOLG
 DAS PARETO PRINZIP DER SCHLÜSSEL ZUM ERFOLG von Urs Schaffer Copyright by Urs Schaffer Schaffer Consulting GmbH Basel www.schaffer-consulting.ch Info@schaffer-consulting.ch Haben Sie gewusst dass... >
DAS PARETO PRINZIP DER SCHLÜSSEL ZUM ERFOLG von Urs Schaffer Copyright by Urs Schaffer Schaffer Consulting GmbH Basel www.schaffer-consulting.ch Info@schaffer-consulting.ch Haben Sie gewusst dass... >
Prozessbewertung und -verbesserung nach ITIL im Kontext des betrieblichen Informationsmanagements. von Stephanie Wilke am 14.08.08
 Prozessbewertung und -verbesserung nach ITIL im Kontext des betrieblichen Informationsmanagements von Stephanie Wilke am 14.08.08 Überblick Einleitung Was ist ITIL? Gegenüberstellung der Prozesse Neuer
Prozessbewertung und -verbesserung nach ITIL im Kontext des betrieblichen Informationsmanagements von Stephanie Wilke am 14.08.08 Überblick Einleitung Was ist ITIL? Gegenüberstellung der Prozesse Neuer
Mind Mapping am PC. für Präsentationen, Vorträge, Selbstmanagement. von Isolde Kommer, Helmut Reinke. 1. Auflage. Hanser München 1999
 Mind Mapping am PC für Präsentationen, Vorträge, Selbstmanagement von Isolde Kommer, Helmut Reinke 1. Auflage Hanser München 1999 Verlag C.H. Beck im Internet: www.beck.de ISBN 978 3 446 21222 0 schnell
Mind Mapping am PC für Präsentationen, Vorträge, Selbstmanagement von Isolde Kommer, Helmut Reinke 1. Auflage Hanser München 1999 Verlag C.H. Beck im Internet: www.beck.de ISBN 978 3 446 21222 0 schnell
«Eine Person ist funktional gesund, wenn sie möglichst kompetent mit einem möglichst gesunden Körper an möglichst normalisierten Lebensbereichen
 18 «Eine Person ist funktional gesund, wenn sie möglichst kompetent mit einem möglichst gesunden Körper an möglichst normalisierten Lebensbereichen teilnimmt und teilhat.» 3Das Konzept der Funktionalen
18 «Eine Person ist funktional gesund, wenn sie möglichst kompetent mit einem möglichst gesunden Körper an möglichst normalisierten Lebensbereichen teilnimmt und teilhat.» 3Das Konzept der Funktionalen
Sequenziertechnologien
 Sequenziertechnologien Folien teilweise von G. Thallinger übernommen 11 Entwicklung der Sequenziertechnologie First Generation 1977 Sanger Sequenzierung (GOLD Standard) Second Generation 2005 454 Sequencing
Sequenziertechnologien Folien teilweise von G. Thallinger übernommen 11 Entwicklung der Sequenziertechnologie First Generation 1977 Sanger Sequenzierung (GOLD Standard) Second Generation 2005 454 Sequencing
Outlook. sysplus.ch outlook - mail-grundlagen Seite 1/8. Mail-Grundlagen. Posteingang
 sysplus.ch outlook - mail-grundlagen Seite 1/8 Outlook Mail-Grundlagen Posteingang Es gibt verschiedene Möglichkeiten, um zum Posteingang zu gelangen. Man kann links im Outlook-Fenster auf die Schaltfläche
sysplus.ch outlook - mail-grundlagen Seite 1/8 Outlook Mail-Grundlagen Posteingang Es gibt verschiedene Möglichkeiten, um zum Posteingang zu gelangen. Man kann links im Outlook-Fenster auf die Schaltfläche
Primzahlen und RSA-Verschlüsselung
 Primzahlen und RSA-Verschlüsselung Michael Fütterer und Jonathan Zachhuber 1 Einiges zu Primzahlen Ein paar Definitionen: Wir bezeichnen mit Z die Menge der positiven und negativen ganzen Zahlen, also
Primzahlen und RSA-Verschlüsselung Michael Fütterer und Jonathan Zachhuber 1 Einiges zu Primzahlen Ein paar Definitionen: Wir bezeichnen mit Z die Menge der positiven und negativen ganzen Zahlen, also
Die Gesellschaftsformen
 Jede Firma - auch eure Schülerfirma - muss sich an bestimmte Spielregeln halten. Dazu gehört auch, dass eine bestimmte Rechtsform für das Unternehmen gewählt wird. Für eure Schülerfirma könnt ihr zwischen
Jede Firma - auch eure Schülerfirma - muss sich an bestimmte Spielregeln halten. Dazu gehört auch, dass eine bestimmte Rechtsform für das Unternehmen gewählt wird. Für eure Schülerfirma könnt ihr zwischen
BIA-Wissensreihe Teil 4. Mind Mapping Methode. Bildungsakademie Sigmaringen
 BIA-Wissensreihe Teil 4 Mind Mapping Methode Bildungsakademie Sigmaringen Inhalt Warum Mind Mapping? Für wen sind Mind Maps sinnvoll? Wie erstellt man Mind Maps? Mind Mapping Software 3 4 5 7 2 1. Warum
BIA-Wissensreihe Teil 4 Mind Mapping Methode Bildungsakademie Sigmaringen Inhalt Warum Mind Mapping? Für wen sind Mind Maps sinnvoll? Wie erstellt man Mind Maps? Mind Mapping Software 3 4 5 7 2 1. Warum
Einfluss der genetischen Architektur auf die empirische Genauigkeit der genomischen Zuchtwertschätzung
 Einfluss der genetischen Architektur auf die empirische Genauigkeit der genomischen Zuchtwertschätzung M. Kramer 1, F. Biscarini 1, B. Bapst 2, C. Stricker 3, H. Simianer 1 1 Department für Nutztierwissenschaften,
Einfluss der genetischen Architektur auf die empirische Genauigkeit der genomischen Zuchtwertschätzung M. Kramer 1, F. Biscarini 1, B. Bapst 2, C. Stricker 3, H. Simianer 1 1 Department für Nutztierwissenschaften,
Das Leitbild vom Verein WIR
 Das Leitbild vom Verein WIR Dieses Zeichen ist ein Gütesiegel. Texte mit diesem Gütesiegel sind leicht verständlich. Leicht Lesen gibt es in drei Stufen. B1: leicht verständlich A2: noch leichter verständlich
Das Leitbild vom Verein WIR Dieses Zeichen ist ein Gütesiegel. Texte mit diesem Gütesiegel sind leicht verständlich. Leicht Lesen gibt es in drei Stufen. B1: leicht verständlich A2: noch leichter verständlich
Einrichten einer Festplatte mit FDISK unter Windows 95/98/98SE/Me
 Einrichten einer Festplatte mit FDISK unter Windows 95/98/98SE/Me Bevor Sie die Platte zum ersten Mal benutzen können, muss sie noch partitioniert und formatiert werden! Vorher zeigt sich die Festplatte
Einrichten einer Festplatte mit FDISK unter Windows 95/98/98SE/Me Bevor Sie die Platte zum ersten Mal benutzen können, muss sie noch partitioniert und formatiert werden! Vorher zeigt sich die Festplatte
Was meinen die Leute eigentlich mit: Grexit?
 Was meinen die Leute eigentlich mit: Grexit? Grexit sind eigentlich 2 Wörter. 1. Griechenland 2. Exit Exit ist ein englisches Wort. Es bedeutet: Ausgang. Aber was haben diese 2 Sachen mit-einander zu tun?
Was meinen die Leute eigentlich mit: Grexit? Grexit sind eigentlich 2 Wörter. 1. Griechenland 2. Exit Exit ist ein englisches Wort. Es bedeutet: Ausgang. Aber was haben diese 2 Sachen mit-einander zu tun?
Welche Lagen können zwei Geraden (im Raum) zueinander haben? Welche Lagen kann eine Gerade bezüglich einer Ebene im Raum einnehmen?
 Welche Lagen können zwei Geraden (im Raum) zueinander haben? Welche Lagen können zwei Ebenen (im Raum) zueinander haben? Welche Lagen kann eine Gerade bezüglich einer Ebene im Raum einnehmen? Wie heiÿt
Welche Lagen können zwei Geraden (im Raum) zueinander haben? Welche Lagen können zwei Ebenen (im Raum) zueinander haben? Welche Lagen kann eine Gerade bezüglich einer Ebene im Raum einnehmen? Wie heiÿt
Welche Bereiche gibt es auf der Internetseite vom Bundes-Aufsichtsamt für Flugsicherung?
 Welche Bereiche gibt es auf der Internetseite vom Bundes-Aufsichtsamt für Flugsicherung? BAF ist die Abkürzung von Bundes-Aufsichtsamt für Flugsicherung. Auf der Internetseite gibt es 4 Haupt-Bereiche:
Welche Bereiche gibt es auf der Internetseite vom Bundes-Aufsichtsamt für Flugsicherung? BAF ist die Abkürzung von Bundes-Aufsichtsamt für Flugsicherung. Auf der Internetseite gibt es 4 Haupt-Bereiche:
Mobile Intranet in Unternehmen
 Mobile Intranet in Unternehmen Ergebnisse einer Umfrage unter Intranet Verantwortlichen aexea GmbH - communication. content. consulting Augustenstraße 15 70178 Stuttgart Tel: 0711 87035490 Mobile Intranet
Mobile Intranet in Unternehmen Ergebnisse einer Umfrage unter Intranet Verantwortlichen aexea GmbH - communication. content. consulting Augustenstraße 15 70178 Stuttgart Tel: 0711 87035490 Mobile Intranet
Persönliches Adressbuch
 Persönliches Adressbuch Persönliches Adressbuch Seite 1 Persönliches Adressbuch Seite 2 Inhaltsverzeichnis 1. WICHTIGE INFORMATIONEN ZUR BEDIENUNG VON CUMULUS 4 2. ALLGEMEINE INFORMATIONEN ZUM PERSÖNLICHEN
Persönliches Adressbuch Persönliches Adressbuch Seite 1 Persönliches Adressbuch Seite 2 Inhaltsverzeichnis 1. WICHTIGE INFORMATIONEN ZUR BEDIENUNG VON CUMULUS 4 2. ALLGEMEINE INFORMATIONEN ZUM PERSÖNLICHEN
Europäischer Fonds für Regionale Entwicklung: EFRE im Bundes-Land Brandenburg vom Jahr 2014 bis für das Jahr 2020 in Leichter Sprache
 Für Ihre Zukunft! Europäischer Fonds für Regionale Entwicklung: EFRE im Bundes-Land Brandenburg vom Jahr 2014 bis für das Jahr 2020 in Leichter Sprache 1 Europäischer Fonds für Regionale Entwicklung: EFRE
Für Ihre Zukunft! Europäischer Fonds für Regionale Entwicklung: EFRE im Bundes-Land Brandenburg vom Jahr 2014 bis für das Jahr 2020 in Leichter Sprache 1 Europäischer Fonds für Regionale Entwicklung: EFRE
Unterrichtsmaterialien in digitaler und in gedruckter Form. Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis
 Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis Das komplette Material finden Sie hier: Download bei School-Scout.de
Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis Das komplette Material finden Sie hier: Download bei School-Scout.de
 extreme Programming (XP) Hermann Götz Sergij Paholchak Agenda Was ist XP? Grundprinzipien Der Entwicklungsprozess Die Projektplanung Praktiken Vorteile und Nachteile Wann macht XP Sinn für ein Projekt?
extreme Programming (XP) Hermann Götz Sergij Paholchak Agenda Was ist XP? Grundprinzipien Der Entwicklungsprozess Die Projektplanung Praktiken Vorteile und Nachteile Wann macht XP Sinn für ein Projekt?
Schritt 1. Anmelden. Klicken Sie auf die Schaltfläche Anmelden
 Schritt 1 Anmelden Klicken Sie auf die Schaltfläche Anmelden Schritt 1 Anmelden Tippen Sie Ihren Benutzernamen und Ihr Passwort ein Tipp: Nutzen Sie die Hilfe Passwort vergessen? wenn Sie sich nicht mehr
Schritt 1 Anmelden Klicken Sie auf die Schaltfläche Anmelden Schritt 1 Anmelden Tippen Sie Ihren Benutzernamen und Ihr Passwort ein Tipp: Nutzen Sie die Hilfe Passwort vergessen? wenn Sie sich nicht mehr
Geld Verdienen im Internet leicht gemacht
 Geld Verdienen im Internet leicht gemacht Hallo, Sie haben sich dieses E-book wahrscheinlich herunter geladen, weil Sie gerne lernen würden wie sie im Internet Geld verdienen können, oder? Denn genau das
Geld Verdienen im Internet leicht gemacht Hallo, Sie haben sich dieses E-book wahrscheinlich herunter geladen, weil Sie gerne lernen würden wie sie im Internet Geld verdienen können, oder? Denn genau das
Statistische Auswertung:
 Statistische Auswertung: Die erhobenen Daten mittels der selbst erstellten Tests (Surfaufgaben) Statistics Punkte aus dem Punkte aus Surftheorietest Punkte aus dem dem und dem Surftheorietest max.14p.
Statistische Auswertung: Die erhobenen Daten mittels der selbst erstellten Tests (Surfaufgaben) Statistics Punkte aus dem Punkte aus Surftheorietest Punkte aus dem dem und dem Surftheorietest max.14p.
Durch Wissen Millionär WerDen... Wer hat zuerst die Million erreicht? spielanleitung Zahl der spieler: alter: redaktion / autor: inhalt:
 Spielanleitung Durch Wissen Millionär werden... Diesen Traum kann man sich in diesem beliebten Quiz-Spiel erfüllen. Ob allein oder in der geselligen Runde dieses Quiz enthält 330 Fragen und 1.320 Multiple-Choice-Antworten.
Spielanleitung Durch Wissen Millionär werden... Diesen Traum kann man sich in diesem beliebten Quiz-Spiel erfüllen. Ob allein oder in der geselligen Runde dieses Quiz enthält 330 Fragen und 1.320 Multiple-Choice-Antworten.
Agile Vorgehensmodelle in der Softwareentwicklung: Scrum
 C A R L V O N O S S I E T Z K Y Agile Vorgehensmodelle in der Softwareentwicklung: Scrum Johannes Diemke Vortrag im Rahmen der Projektgruppe Oldenburger Robot Soccer Team im Wintersemester 2009/2010 Was
C A R L V O N O S S I E T Z K Y Agile Vorgehensmodelle in der Softwareentwicklung: Scrum Johannes Diemke Vortrag im Rahmen der Projektgruppe Oldenburger Robot Soccer Team im Wintersemester 2009/2010 Was
Korrelation. Übungsbeispiel 1. Übungsbeispiel 4. Übungsbeispiel 2. Übungsbeispiel 3. Korrel.dtp Seite 1
 Korrelation Die Korrelationsanalyse zeigt Zusammenhänge auf und macht Vorhersagen möglich Was ist Korrelation? Was sagt die Korrelationszahl aus? Wie geht man vor? Korrelation ist eine eindeutige Beziehung
Korrelation Die Korrelationsanalyse zeigt Zusammenhänge auf und macht Vorhersagen möglich Was ist Korrelation? Was sagt die Korrelationszahl aus? Wie geht man vor? Korrelation ist eine eindeutige Beziehung
Staatssekretär Dr. Günther Horzetzky
 #upj15 #upj15 Staatssekretär Dr. Günther Horzetzky Ministerium für Wirtschaft, Energie, Industrie, Mittelstand und Handwerk des Landes Nordrhein-Westfalen Ministerium für Wirtschaft, Energie, Industrie,
#upj15 #upj15 Staatssekretär Dr. Günther Horzetzky Ministerium für Wirtschaft, Energie, Industrie, Mittelstand und Handwerk des Landes Nordrhein-Westfalen Ministerium für Wirtschaft, Energie, Industrie,
etermin Einbindung in Outlook
 etermin Einbindung in Outlook 1. Einführung Über etermin gebuchte Termine können bei Bedarf auch mit externen Terminkalendern, wie zum Beispiel Outlook, ical oder Google synchronisiert werden. Dieses Dokument
etermin Einbindung in Outlook 1. Einführung Über etermin gebuchte Termine können bei Bedarf auch mit externen Terminkalendern, wie zum Beispiel Outlook, ical oder Google synchronisiert werden. Dieses Dokument
Straße. Tiefgarage. Planung einer Tiefgarageneinfahrt Arbeitsblatt
 Planung einer Tiefgarageneinfahrt Arbeitsblatt Die Problemstellung Du bist Architekt und deine Aufgabe ist die Planung einer Tiefgarageneinfahrt von der Straße bis in die Kelleretage eines neu gebauten
Planung einer Tiefgarageneinfahrt Arbeitsblatt Die Problemstellung Du bist Architekt und deine Aufgabe ist die Planung einer Tiefgarageneinfahrt von der Straße bis in die Kelleretage eines neu gebauten
Windows 8.1. In 5 Minuten Was ist alles neu? Word
 Windows 8.1 In 5 Minuten Was ist alles neu? Word Inhaltsverzeichnis Inhaltsverzeichnis WINDOWS 8.1 IN 5 MINUTEN... 1 1. WINDOWS 8.1 DIE NEUEN FEATURES... 1 2. DIE DESKTOP- UND KACHELOBERFLÄCHE... 2 3.
Windows 8.1 In 5 Minuten Was ist alles neu? Word Inhaltsverzeichnis Inhaltsverzeichnis WINDOWS 8.1 IN 5 MINUTEN... 1 1. WINDOWS 8.1 DIE NEUEN FEATURES... 1 2. DIE DESKTOP- UND KACHELOBERFLÄCHE... 2 3.
Persönliche Zukunftsplanung mit Menschen, denen nicht zugetraut wird, dass sie für sich selbst sprechen können Von Susanne Göbel und Josef Ströbl
 Persönliche Zukunftsplanung mit Menschen, denen nicht zugetraut Von Susanne Göbel und Josef Ströbl Die Ideen der Persönlichen Zukunftsplanung stammen aus Nordamerika. Dort werden Zukunftsplanungen schon
Persönliche Zukunftsplanung mit Menschen, denen nicht zugetraut Von Susanne Göbel und Josef Ströbl Die Ideen der Persönlichen Zukunftsplanung stammen aus Nordamerika. Dort werden Zukunftsplanungen schon
Ebenenmasken Grundlagen
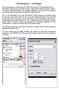 Ebenenmasken Grundlagen Was sind Ebenmasken? Was machen sie? Wofür braucht man sie? Wie funktionieren sie? Ebenmasken sind eines der sinnvollsten Tools in anspruchvollen EBV Programmen (EBV = elektronische
Ebenenmasken Grundlagen Was sind Ebenmasken? Was machen sie? Wofür braucht man sie? Wie funktionieren sie? Ebenmasken sind eines der sinnvollsten Tools in anspruchvollen EBV Programmen (EBV = elektronische
Guide DynDNS und Portforwarding
 Guide DynDNS und Portforwarding Allgemein Um Geräte im lokalen Netzwerk von überall aus über das Internet erreichen zu können, kommt man um die Themen Dynamik DNS (kurz DynDNS) und Portweiterleitung(auch
Guide DynDNS und Portforwarding Allgemein Um Geräte im lokalen Netzwerk von überall aus über das Internet erreichen zu können, kommt man um die Themen Dynamik DNS (kurz DynDNS) und Portweiterleitung(auch
infach Geld FBV Ihr Weg zum finanzellen Erfolg Florian Mock
 infach Ihr Weg zum finanzellen Erfolg Geld Florian Mock FBV Die Grundlagen für finanziellen Erfolg Denn Sie müssten anschließend wieder vom Gehaltskonto Rückzahlungen in Höhe der Entnahmen vornehmen, um
infach Ihr Weg zum finanzellen Erfolg Geld Florian Mock FBV Die Grundlagen für finanziellen Erfolg Denn Sie müssten anschließend wieder vom Gehaltskonto Rückzahlungen in Höhe der Entnahmen vornehmen, um
Berechnung der Erhöhung der Durchschnittsprämien
 Wolfram Fischer Berechnung der Erhöhung der Durchschnittsprämien Oktober 2004 1 Zusammenfassung Zur Berechnung der Durchschnittsprämien wird das gesamte gemeldete Prämienvolumen Zusammenfassung durch die
Wolfram Fischer Berechnung der Erhöhung der Durchschnittsprämien Oktober 2004 1 Zusammenfassung Zur Berechnung der Durchschnittsprämien wird das gesamte gemeldete Prämienvolumen Zusammenfassung durch die
~~ Swing Trading Strategie ~~
 ~~ Swing Trading Strategie ~~ Ebook Copyright by Thomas Kedziora www.forextrade.de Die Rechte des Buches Swing Trading Strategie liegen beim Autor und Herausgeber! -- Seite 1 -- Haftungsausschluss Der
~~ Swing Trading Strategie ~~ Ebook Copyright by Thomas Kedziora www.forextrade.de Die Rechte des Buches Swing Trading Strategie liegen beim Autor und Herausgeber! -- Seite 1 -- Haftungsausschluss Der
Sowohl die Malstreifen als auch die Neperschen Streifen können auch in anderen Stellenwertsystemen verwendet werden.
 Multiplikation Die schriftliche Multiplikation ist etwas schwieriger als die Addition. Zum einen setzt sie das kleine Einmaleins voraus, zum anderen sind die Überträge, die zu merken sind und häufig in
Multiplikation Die schriftliche Multiplikation ist etwas schwieriger als die Addition. Zum einen setzt sie das kleine Einmaleins voraus, zum anderen sind die Überträge, die zu merken sind und häufig in
Agile Enterprise Development. Sind Sie bereit für den nächsten Schritt?
 Agile Enterprise Development Sind Sie bereit für den nächsten Schritt? Steigern Sie noch immer die Wirtschaftlichkeit Ihres Unternehmens alleine durch Kostensenkung? Im Projektportfolio steckt das Potenzial
Agile Enterprise Development Sind Sie bereit für den nächsten Schritt? Steigern Sie noch immer die Wirtschaftlichkeit Ihres Unternehmens alleine durch Kostensenkung? Im Projektportfolio steckt das Potenzial
NEWSLETTER // AUGUST 2015
 NEWSLETTER // AUGUST 2015 Kürzlich ist eine neue Version von SoftwareCentral erschienen, die neue Version enthält eine Reihe von Verbesserungen und neuen Funktionen die das Arbeiten mit SCCM noch einfacher
NEWSLETTER // AUGUST 2015 Kürzlich ist eine neue Version von SoftwareCentral erschienen, die neue Version enthält eine Reihe von Verbesserungen und neuen Funktionen die das Arbeiten mit SCCM noch einfacher
Feiertage in Marvin hinterlegen
 von 6 Goecom GmbH & Co KG Marvin How to's Feiertage in Marvin hinterlegen Feiertage spielen in Marvin an einer Reihe von Stellen eine nicht unerhebliche Rolle. Daher ist es wichtig, zum Einen zu hinterlegen,
von 6 Goecom GmbH & Co KG Marvin How to's Feiertage in Marvin hinterlegen Feiertage spielen in Marvin an einer Reihe von Stellen eine nicht unerhebliche Rolle. Daher ist es wichtig, zum Einen zu hinterlegen,
ALEMÃO. Text 1. Lernen, lernen, lernen
 ALEMÃO Text 1 Lernen, lernen, lernen Der Mai ist für viele deutsche Jugendliche keine schöne Zeit. Denn dann müssen sie in vielen Bundesländern die Abiturprüfungen schreiben. Das heiβt: lernen, lernen,
ALEMÃO Text 1 Lernen, lernen, lernen Der Mai ist für viele deutsche Jugendliche keine schöne Zeit. Denn dann müssen sie in vielen Bundesländern die Abiturprüfungen schreiben. Das heiβt: lernen, lernen,
DER SELBST-CHECK FÜR IHR PROJEKT
 DER SELBST-CHECK FÜR IHR PROJEKT In 30 Fragen und 5 Tipps zum erfolgreichen Projekt! Beantworten Sie die wichtigsten Fragen rund um Ihr Projekt für Ihren Erfolg und für Ihre Unterstützer. IHR LEITFADEN
DER SELBST-CHECK FÜR IHR PROJEKT In 30 Fragen und 5 Tipps zum erfolgreichen Projekt! Beantworten Sie die wichtigsten Fragen rund um Ihr Projekt für Ihren Erfolg und für Ihre Unterstützer. IHR LEITFADEN
Additional Cycle Index (ACIX) Thomas Theuerzeit
 Additional Cycle Index (ACIX) Thomas Theuerzeit Der nachfolgende Artikel über den ACIX stammt vom Entwickler des Indikators Thomas Theuerzeit. Weitere Informationen über Projekte von Thomas Theuerzeit
Additional Cycle Index (ACIX) Thomas Theuerzeit Der nachfolgende Artikel über den ACIX stammt vom Entwickler des Indikators Thomas Theuerzeit. Weitere Informationen über Projekte von Thomas Theuerzeit
Physik & Musik. Stimmgabeln. 1 Auftrag
 Physik & Musik 5 Stimmgabeln 1 Auftrag Physik & Musik Stimmgabeln Seite 1 Stimmgabeln Bearbeitungszeit: 30 Minuten Sozialform: Einzel- oder Partnerarbeit Voraussetzung: Posten 1: "Wie funktioniert ein
Physik & Musik 5 Stimmgabeln 1 Auftrag Physik & Musik Stimmgabeln Seite 1 Stimmgabeln Bearbeitungszeit: 30 Minuten Sozialform: Einzel- oder Partnerarbeit Voraussetzung: Posten 1: "Wie funktioniert ein
Studieren- Erklärungen und Tipps
 Studieren- Erklärungen und Tipps Es gibt Berufe, die man nicht lernen kann, sondern für die man ein Studium machen muss. Das ist zum Beispiel so wenn man Arzt oder Lehrer werden möchte. Hat ihr Kind das
Studieren- Erklärungen und Tipps Es gibt Berufe, die man nicht lernen kann, sondern für die man ein Studium machen muss. Das ist zum Beispiel so wenn man Arzt oder Lehrer werden möchte. Hat ihr Kind das
How to do? Projekte - Zeiterfassung
 How to do? Projekte - Zeiterfassung Stand: Version 4.0.1, 18.03.2009 1. EINLEITUNG...3 2. PROJEKTE UND STAMMDATEN...4 2.1 Projekte... 4 2.2 Projektmitarbeiter... 5 2.3 Tätigkeiten... 6 2.4 Unterprojekte...
How to do? Projekte - Zeiterfassung Stand: Version 4.0.1, 18.03.2009 1. EINLEITUNG...3 2. PROJEKTE UND STAMMDATEN...4 2.1 Projekte... 4 2.2 Projektmitarbeiter... 5 2.3 Tätigkeiten... 6 2.4 Unterprojekte...
Outlook Erstellen einer E-Mail aus einer HTML - Vorlage INHALT
 Outlook Erstellen einer E-Mail aus einer HTML - Vorlage INHALT LADEN DER VORLAGE 2 Öffnen Sie Outlook 2 Klicken Sie auf EXTRAS >> OPTIONEN 2 Im Optionenfeld von Outlook folgend Sie den Schritten 2 Fenster
Outlook Erstellen einer E-Mail aus einer HTML - Vorlage INHALT LADEN DER VORLAGE 2 Öffnen Sie Outlook 2 Klicken Sie auf EXTRAS >> OPTIONEN 2 Im Optionenfeld von Outlook folgend Sie den Schritten 2 Fenster
Serien-eMail mit oder ohne Anhang
 Serien-eMail mit oder ohne Anhang Sie können im WohnungsManager sowohl objektübergreifend als auch in einem Objekt Serien-eMails versenden. Die Serien-eMail ist für SMTP (Short Message Tranfer Protocol)
Serien-eMail mit oder ohne Anhang Sie können im WohnungsManager sowohl objektübergreifend als auch in einem Objekt Serien-eMails versenden. Die Serien-eMail ist für SMTP (Short Message Tranfer Protocol)
Die Bundes-Zentrale für politische Bildung stellt sich vor
 Die Bundes-Zentrale für politische Bildung stellt sich vor Die Bundes-Zentrale für politische Bildung stellt sich vor Deutschland ist ein demokratisches Land. Das heißt: Die Menschen in Deutschland können
Die Bundes-Zentrale für politische Bildung stellt sich vor Die Bundes-Zentrale für politische Bildung stellt sich vor Deutschland ist ein demokratisches Land. Das heißt: Die Menschen in Deutschland können
Die Post hat eine Umfrage gemacht
 Die Post hat eine Umfrage gemacht Bei der Umfrage ging es um das Thema: Inklusion Die Post hat Menschen mit Behinderung und Menschen ohne Behinderung gefragt: Wie zufrieden sie in dieser Gesellschaft sind.
Die Post hat eine Umfrage gemacht Bei der Umfrage ging es um das Thema: Inklusion Die Post hat Menschen mit Behinderung und Menschen ohne Behinderung gefragt: Wie zufrieden sie in dieser Gesellschaft sind.
Pflegende Angehörige Online Ihre Plattform im Internet
 Pflegende Angehörige Online Ihre Plattform im Internet Wissen Wichtiges Wissen rund um Pflege Unterstützung Professionelle Beratung Austausch und Kontakt Erfahrungen & Rat mit anderen Angehörigen austauschen
Pflegende Angehörige Online Ihre Plattform im Internet Wissen Wichtiges Wissen rund um Pflege Unterstützung Professionelle Beratung Austausch und Kontakt Erfahrungen & Rat mit anderen Angehörigen austauschen
Suche schlecht beschriftete Bilder mit Eigenen Abfragen
 Suche schlecht beschriftete Bilder mit Eigenen Abfragen Ist die Bilderdatenbank über einen längeren Zeitraum in Benutzung, so steigt die Wahrscheinlichkeit für schlecht beschriftete Bilder 1. Insbesondere
Suche schlecht beschriftete Bilder mit Eigenen Abfragen Ist die Bilderdatenbank über einen längeren Zeitraum in Benutzung, so steigt die Wahrscheinlichkeit für schlecht beschriftete Bilder 1. Insbesondere
Jede Zahl muss dabei einzeln umgerechnet werden. Beginnen wir also ganz am Anfang mit der Zahl,192.
 Binäres und dezimales Zahlensystem Ziel In diesem ersten Schritt geht es darum, die grundlegende Umrechnung aus dem Dezimalsystem in das Binärsystem zu verstehen. Zusätzlich wird auch die andere Richtung,
Binäres und dezimales Zahlensystem Ziel In diesem ersten Schritt geht es darum, die grundlegende Umrechnung aus dem Dezimalsystem in das Binärsystem zu verstehen. Zusätzlich wird auch die andere Richtung,
Abamsoft Finos im Zusammenspiel mit shop to date von DATA BECKER
 Abamsoft Finos im Zusammenspiel mit shop to date von DATA BECKER Abamsoft Finos in Verbindung mit der Webshopanbindung wurde speziell auf die Shop-Software shop to date von DATA BECKER abgestimmt. Mit
Abamsoft Finos im Zusammenspiel mit shop to date von DATA BECKER Abamsoft Finos in Verbindung mit der Webshopanbindung wurde speziell auf die Shop-Software shop to date von DATA BECKER abgestimmt. Mit
Senkung des technischen Zinssatzes und des Umwandlungssatzes
 Senkung des technischen Zinssatzes und des Umwandlungssatzes Was ist ein Umwandlungssatz? Die PKE führt für jede versicherte Person ein individuelles Konto. Diesem werden die Beiträge, allfällige Einlagen
Senkung des technischen Zinssatzes und des Umwandlungssatzes Was ist ein Umwandlungssatz? Die PKE führt für jede versicherte Person ein individuelles Konto. Diesem werden die Beiträge, allfällige Einlagen
Eigenen Farbverlauf erstellen
 Diese Serie ist an totale Neulinge gerichtet. Neu bei PhotoLine, evtl. sogar komplett neu, was Bildbearbeitung betrifft. So versuche ich, hier alles einfach zu halten. Ich habe sogar PhotoLine ein zweites
Diese Serie ist an totale Neulinge gerichtet. Neu bei PhotoLine, evtl. sogar komplett neu, was Bildbearbeitung betrifft. So versuche ich, hier alles einfach zu halten. Ich habe sogar PhotoLine ein zweites
14. Minimale Schichtdicken von PEEK und PPS im Schlauchreckprozeß und im Rheotensversuch
 14. Minimale Schichtdicken von PEEK und PPS im Schlauchreckprozeß und im Rheotensversuch Analog zu den Untersuchungen an LDPE in Kap. 6 war zu untersuchen, ob auch für die Hochtemperatur-Thermoplaste aus
14. Minimale Schichtdicken von PEEK und PPS im Schlauchreckprozeß und im Rheotensversuch Analog zu den Untersuchungen an LDPE in Kap. 6 war zu untersuchen, ob auch für die Hochtemperatur-Thermoplaste aus
Diese Ansicht erhalten Sie nach der erfolgreichen Anmeldung bei Wordpress.
 Anmeldung http://www.ihredomain.de/wp-admin Dashboard Diese Ansicht erhalten Sie nach der erfolgreichen Anmeldung bei Wordpress. Das Dashboard gibt Ihnen eine kurze Übersicht, z.b. Anzahl der Beiträge,
Anmeldung http://www.ihredomain.de/wp-admin Dashboard Diese Ansicht erhalten Sie nach der erfolgreichen Anmeldung bei Wordpress. Das Dashboard gibt Ihnen eine kurze Übersicht, z.b. Anzahl der Beiträge,
Agentur für Werbung & Internet. Schritt für Schritt: E-Mail-Konfiguration mit Apple Mail
 Agentur für Werbung & Internet Schritt für Schritt: E-Mail-Konfiguration mit Apple Mail E-Mail-Konfiguration mit Apple Mail Inhalt E-Mail-Konto erstellen 3 Auswahl des Servertyp: POP oder IMAP 4 Konfiguration
Agentur für Werbung & Internet Schritt für Schritt: E-Mail-Konfiguration mit Apple Mail E-Mail-Konfiguration mit Apple Mail Inhalt E-Mail-Konto erstellen 3 Auswahl des Servertyp: POP oder IMAP 4 Konfiguration
Es gilt das gesprochene Wort. Anrede
 Sperrfrist: 28. November 2007, 13.00 Uhr Es gilt das gesprochene Wort Statement des Staatssekretärs im Bayerischen Staatsministerium für Unterricht und Kultus, Karl Freller, anlässlich des Pressegesprächs
Sperrfrist: 28. November 2007, 13.00 Uhr Es gilt das gesprochene Wort Statement des Staatssekretärs im Bayerischen Staatsministerium für Unterricht und Kultus, Karl Freller, anlässlich des Pressegesprächs
GLEICH WEIT WEG. Aufgabe. Das ist ein Ausschnitt aus der Tausenderreihe:
 GLEICH WEIT WEG Thema: Sich orientieren und operieren an der Tausenderreihe Klasse: 3. Klasse (Zahlenbuch nach S. 26-27) Dauer: 3-4 Lektionen Material: Tausenderreihe, Arbeitsblatt, evt. Plättchen Bearbeitung:
GLEICH WEIT WEG Thema: Sich orientieren und operieren an der Tausenderreihe Klasse: 3. Klasse (Zahlenbuch nach S. 26-27) Dauer: 3-4 Lektionen Material: Tausenderreihe, Arbeitsblatt, evt. Plättchen Bearbeitung:
Text Formatierung in Excel
 Text Formatierung in Excel Das Aussehen des Textes einer oder mehrerer Zellen kann in Excel über verschiedene Knöpfe beeinflusst werden. Dazu zuerst die betroffenen Zelle(n) anwählen und danach den entsprechenden
Text Formatierung in Excel Das Aussehen des Textes einer oder mehrerer Zellen kann in Excel über verschiedene Knöpfe beeinflusst werden. Dazu zuerst die betroffenen Zelle(n) anwählen und danach den entsprechenden
AZK 1- Freistil. Der Dialog "Arbeitszeitkonten" Grundsätzliches zum Dialog "Arbeitszeitkonten"
 AZK 1- Freistil Nur bei Bedarf werden dafür gekennzeichnete Lohnbestandteile (Stundenzahl und Stundensatz) zwischen dem aktuellen Bruttolohnjournal und dem AZK ausgetauscht. Das Ansparen und das Auszahlen
AZK 1- Freistil Nur bei Bedarf werden dafür gekennzeichnete Lohnbestandteile (Stundenzahl und Stundensatz) zwischen dem aktuellen Bruttolohnjournal und dem AZK ausgetauscht. Das Ansparen und das Auszahlen
Bericht über die Untersuchung zur Erblichkeit von Herzerkrankungen beim PON
 1 Bericht über die Untersuchung zur Erblichkeit von Herzerkrankungen beim PON Einleitung Bei der Rasse PON wurden im APH in der letzten Zeit auffällig viele Herzkrankheiten und Herzveränderungen unterschiedlicher
1 Bericht über die Untersuchung zur Erblichkeit von Herzerkrankungen beim PON Einleitung Bei der Rasse PON wurden im APH in der letzten Zeit auffällig viele Herzkrankheiten und Herzveränderungen unterschiedlicher
Multicheck Schülerumfrage 2013
 Multicheck Schülerumfrage 2013 Die gemeinsame Studie von Multicheck und Forschungsinstitut gfs-zürich Sonderauswertung ICT Berufsbildung Schweiz Auswertung der Fragen der ICT Berufsbildung Schweiz Wir
Multicheck Schülerumfrage 2013 Die gemeinsame Studie von Multicheck und Forschungsinstitut gfs-zürich Sonderauswertung ICT Berufsbildung Schweiz Auswertung der Fragen der ICT Berufsbildung Schweiz Wir
Meet the Germans. Lerntipp zur Schulung der Fertigkeit des Sprechens. Lerntipp und Redemittel zur Präsentation oder einen Vortrag halten
 Meet the Germans Lerntipp zur Schulung der Fertigkeit des Sprechens Lerntipp und Redemittel zur Präsentation oder einen Vortrag halten Handreichungen für die Kursleitung Seite 2, Meet the Germans 2. Lerntipp
Meet the Germans Lerntipp zur Schulung der Fertigkeit des Sprechens Lerntipp und Redemittel zur Präsentation oder einen Vortrag halten Handreichungen für die Kursleitung Seite 2, Meet the Germans 2. Lerntipp
Lichtbrechung an Linsen
 Sammellinsen Lichtbrechung an Linsen Fällt ein paralleles Lichtbündel auf eine Sammellinse, so werden die Lichtstrahlen so gebrochen, dass sie durch einen Brennpunkt der Linse verlaufen. Der Abstand zwischen
Sammellinsen Lichtbrechung an Linsen Fällt ein paralleles Lichtbündel auf eine Sammellinse, so werden die Lichtstrahlen so gebrochen, dass sie durch einen Brennpunkt der Linse verlaufen. Der Abstand zwischen
Nach der Anmeldung im Backend Bereich landen Sie im Kontrollzentrum, welches so aussieht:
 Beiträge erstellen in Joomla Nach der Anmeldung im Backend Bereich landen Sie im Kontrollzentrum, welches so aussieht: Abbildung 1 - Kontrollzentrum Von hier aus kann man zu verschiedene Einstellungen
Beiträge erstellen in Joomla Nach der Anmeldung im Backend Bereich landen Sie im Kontrollzentrum, welches so aussieht: Abbildung 1 - Kontrollzentrum Von hier aus kann man zu verschiedene Einstellungen
4. BEZIEHUNGEN ZWISCHEN TABELLEN
 4. BEZIEHUNGEN ZWISCHEN TABELLEN Zwischen Tabellen können in MS Access Beziehungen bestehen. Durch das Verwenden von Tabellen, die zueinander in Beziehung stehen, können Sie Folgendes erreichen: Die Größe
4. BEZIEHUNGEN ZWISCHEN TABELLEN Zwischen Tabellen können in MS Access Beziehungen bestehen. Durch das Verwenden von Tabellen, die zueinander in Beziehung stehen, können Sie Folgendes erreichen: Die Größe
Die Gleichung A x = a hat für A 0 die eindeutig bestimmte Lösung. Für A=0 und a 0 existiert keine Lösung.
 Lineare Gleichungen mit einer Unbekannten Die Grundform der linearen Gleichung mit einer Unbekannten x lautet A x = a Dabei sind A, a reelle Zahlen. Die Gleichung lösen heißt, alle reellen Zahlen anzugeben,
Lineare Gleichungen mit einer Unbekannten Die Grundform der linearen Gleichung mit einer Unbekannten x lautet A x = a Dabei sind A, a reelle Zahlen. Die Gleichung lösen heißt, alle reellen Zahlen anzugeben,
trivum Multiroom System Konfigurations- Anleitung Erstellen eines RS232 Protokolls am Bespiel eines Marantz SR7005
 trivum Multiroom System Konfigurations- Anleitung Erstellen eines RS232 Protokolls am Bespiel eines Marantz SR7005 2 Inhalt 1. Anleitung zum Einbinden eines über RS232 zu steuernden Devices...3 1.2 Konfiguration
trivum Multiroom System Konfigurations- Anleitung Erstellen eines RS232 Protokolls am Bespiel eines Marantz SR7005 2 Inhalt 1. Anleitung zum Einbinden eines über RS232 zu steuernden Devices...3 1.2 Konfiguration
Energetische Klassen von Gebäuden
 Energetische Klassen von Gebäuden Grundsätzlich gibt es Neubauten und Bestandsgebäude. Diese Definition ist immer aktuell. Aber auch ein heutiger Neubau ist in drei (oder vielleicht erst zehn?) Jahren
Energetische Klassen von Gebäuden Grundsätzlich gibt es Neubauten und Bestandsgebäude. Diese Definition ist immer aktuell. Aber auch ein heutiger Neubau ist in drei (oder vielleicht erst zehn?) Jahren
Kurzanleitung. MEYTON Aufbau einer Internetverbindung. 1 Von 11
 Kurzanleitung MEYTON Aufbau einer Internetverbindung 1 Von 11 Inhaltsverzeichnis Installation eines Internetzugangs...3 Ist mein Router bereits im MEYTON Netzwerk?...3 Start des YAST Programms...4 Auswahl
Kurzanleitung MEYTON Aufbau einer Internetverbindung 1 Von 11 Inhaltsverzeichnis Installation eines Internetzugangs...3 Ist mein Router bereits im MEYTON Netzwerk?...3 Start des YAST Programms...4 Auswahl
Plotten von Linien ( nach Jack Bresenham, 1962 )
 Plotten von Linien ( nach Jack Bresenham, 1962 ) Ac Eine auf dem Bildschirm darzustellende Linie sieht treppenförmig aus, weil der Computer Linien aus einzelnen (meist quadratischen) Bildpunkten, Pixels
Plotten von Linien ( nach Jack Bresenham, 1962 ) Ac Eine auf dem Bildschirm darzustellende Linie sieht treppenförmig aus, weil der Computer Linien aus einzelnen (meist quadratischen) Bildpunkten, Pixels
Anbinden der Visualisierung GILLES TOUCH (VNC)
 Anbinden der Visualisierung GILLES TOUCH (VNC) Seite 1 von 19 Inhalt 1. Ermitteln der internen IP-Adresse... 3 2. Einstellen der IP-Adresse an der Gilles-Touch Regelung... 6 3. Installieren des Fernwartungsprogramms
Anbinden der Visualisierung GILLES TOUCH (VNC) Seite 1 von 19 Inhalt 1. Ermitteln der internen IP-Adresse... 3 2. Einstellen der IP-Adresse an der Gilles-Touch Regelung... 6 3. Installieren des Fernwartungsprogramms
Organische Photovoltaik: Auf dem Weg zum energieautarken Haus. Referat von Dr. Gerhard Felten. Geschäftsleiter Zentralbereich Forschung und
 27. Juni 2007 RF 70602 Organische Photovoltaik: Auf dem Weg zum energieautarken Haus Referat von Dr. Gerhard Felten Geschäftsleiter Zentralbereich Forschung und Vorausentwicklung anlässlich des Starts
27. Juni 2007 RF 70602 Organische Photovoltaik: Auf dem Weg zum energieautarken Haus Referat von Dr. Gerhard Felten Geschäftsleiter Zentralbereich Forschung und Vorausentwicklung anlässlich des Starts
1 Mathematische Grundlagen
 Mathematische Grundlagen - 1-1 Mathematische Grundlagen Der Begriff der Menge ist einer der grundlegenden Begriffe in der Mathematik. Mengen dienen dazu, Dinge oder Objekte zu einer Einheit zusammenzufassen.
Mathematische Grundlagen - 1-1 Mathematische Grundlagen Der Begriff der Menge ist einer der grundlegenden Begriffe in der Mathematik. Mengen dienen dazu, Dinge oder Objekte zu einer Einheit zusammenzufassen.
