Genomsequenzierung für Anfänger
|
|
|
- Edmund Glöckner
- vor 8 Jahren
- Abrufe
Transkript
1 Genomsequenzierung für Anfänger Philipp Pagel 8. November DNA Sequenzierung Heute wird DNA üblicherweise mit der sogenannten Sanger (oder chain-terminationoder Didesoxy-) Methode sequenziert dessen wesentliche Schritte im Folgenden schematisch dargestellt werden. (Biologisch versierte Leser mögen verzeihen, dass hier stark vereinfacht wird.) Zunächst wird ein kleines (ca bp) Stück einzelstängiger DNA hergestellt, dessen Sequenz am Anfang (5 Ende) des kodierenden Strangs der zu sequenzierenden DNA vorkommt der sogenannte Primer. Dieser ist somit komplementär zum korresponierenden Abschnitt des Komplementärstrangs (template Strang) und wird unter geeigneten Bedingungen an diesen binden (hybridisieren): Primer: 5 ACGGCTATGC 3 Nun gibt man eine sogenannte DNA-Polymerase, sowie ein Gemisch der vier Nukleotide (A, T, C und G) hinzu und lässt die Polymerase ihre Arbeit tun. Dabei hängt diese Schritt für Schritt weitere Nukleotide an den Primer an und zwar nicht zufällig, sondern streng nach der Vorlage (dem Template). Dadurch entsteht mit der Zeit ein vollständiger DNA-Strang aus dem Primer: Primer: 5 ACGGCTATGCATGCCGAACGAATTGACTGACCATGGGTAC... 3 Wenn man nun zusätzlich zu den vier normalen Nukleotiden A, T, C und G eine geringe Menge A hinzufügt, welches chemisch so modifiziert ist, dass es nicht verlängert werden kann (Didesoxy-ATP), so kommt es an den Stellen, an denen ein solches A zufällig eingebaut wird, zu einem Kettenabbruch: Primer: 5 ACGGCTATGCATGCCGAACGAA 3 oder 1
2 Primer: 5 ACGGCTATGCATGCCGAACGAATTGACTGACCA 3 oder Primer: 5 ACGGCTATGCA 3 oder... In unserem Reagenzglas befindet sich nun also ein Gemisch von neugebildeten DNA Molekülen, von denen wir wissen, dass sie mit einem A enden. Das Gleiche wiederholen wir in drei weiteren Reagenzgläsern analog mit den übrigen Nukleotiden. So erhalten wir vier separate Reaktionen, die jeweils Kettenabbrüche an je einer der vier Basen enthalten. Diese vier DNA Lösungen werden mittels Gelelektrophorese aufgetrennt. Dabei wandern kurze Moleküle schneller durch das Agarosegel, als längere. Diese Information kann man verwenden, um die Sequenz zu bestimmen. Für unser Beispiel ergäbe sich etwa folgendes Bild: A T C G Laufrichtung ATGCCGAACGAATTGACTGACCA Das kürzeste Molekül auf dem Gel (ganz unten) findet sich in der ersten Spur (Spalte) also ist die erste Base ein A. Das nächstlängere ist in der zweiten Spur: ein T. Es folgen ein G, ein C usw. Wenn wir die Banden nun schrittweise nach oben verfolgen, erhalten wir die gesuchte Basensequenz. Man kann das Ganze noch übersichtlicher gestalten, indem man die DNA Moleküle in jeder der vier Reaktionen mit einem anderen Fluoreszenzfarbstoff markiert, zusammenmischt und dann gemeinsam in einer Spur laufen lässt: 2
3 Laufrichtung A T G CC G A A CG A A T T G A C T G A CCA Heute verwendet man meist Sequenziergeräte, die die obigen Schritte automatisch ablaufen lassen. Die Auftrennung erfolgt dabei in einer Kapillare und ein optischer Sensor registriert die verschiedenfarbigen Banden. Am Ende erhält man Intensitätskurven für die vier Kanäle, sowie eine daraus vom Computer generierte Basensequenz. Hier ein Auschnitt: Quelle: Mit zunehmender Länge, wird das Signal schwächer und somit unzuverlässiger. Daher kann man mit dieser Methode nur ca Basen in einem Read (oder Run) sequenzieren. Um längere DNA-Moleküle zu sequenzieren (die codierende Sequenz für ein durchschnittliches Protein kann leicht mehrere tausend Basen lang sein) benötigt man also mehrere Reads mit unterschiedlichen Primern: Template Primer 1 Primer 2 Primer 3 Primer 4 Dies ist kein Problem, wenn man die passenden Sequenzen für die einzelnen Primer bereits kennt. Sequenziert man jedoch eine unbekannte DNA, so muß man das Ergebnis 3
4 des ersten Runs abwarten, um eine Primersequenz für den nächsten Run zu bestimmen. Dieser Vorgang wird als primer walking bezeichnet und ist durchaus praktikabel, wenn insgesamt nur wenige Reads nötig sind um die gewünschte Sequenz zu bekommen. Im Falle ganzer Genome ist dies aber viel zu umständlich und langwierig. 2 Kleinere Häppchen BAC sequencing Um ein ganzes Genom zu sequenzieren bietet es sich an, die DNA in eine Vielzahl deutlich kürzerer Abschnitte ( Klone, bp) zu zerlegen, die man dann parallel sequenziert und am Ende zusammensetzt (assembliert). Hierzu werden die einzelnen Abschnitte z.b. in sogenannte bacterial artificial chromosomes (BACs) kloniert. Diese zerlegt man weiter in Subklone, die dann mittels Primer-Walking sequenziert werden. Die Reihenfolge der Klone und Subklone wird dabei anhand bekannter Markersequenzen (sequence tags), deren relative Lage zueinander man aus vorher durchgeführten Mapping-Experimenten kennt (z.b. Radiation hybrid maps) bestimmt. Dabei bezeichnet man die aus den einzelnen Reads zusammengesetzten Sequenzen als Contigs, die ihrerseits zu Supercontigs (oder Scaffolds) zusammengefügt werden. Quelle: 3 Whole genome shotgun sequencing Das whole genome shotgun Verfahren hat die Genomsequenzierung revolutioniert. Es verzichtet auf vorheriges Wissen über die Reihenfolge von Klonen. Stattdessen wird die gesamte Genomsequenz in einer großen Zahl zufällig plazierter Reads sequenziert ( shotgun ). Im Anschluß werden die einzelnen Reads anhand ihrer Überlappungen von einem Computerprogramm zusammengesetzt. 4
5 Quelle: Da die Reads zufällig über das Genom verteilt sind, reicht es nicht aus, genau die Anzahl Basen zu sequenzieren, die das Genom enthält. Dabei würden einige Bereiche mehrfach sequenziert werden, andere hingegen garnicht. Aus diesem Grund ist es erforderlich, eine höhere mittlere Abdeckung (coverage) zu erreichen. Doch wie hoch muss diese sein? 3.1 Wieviele Reads? Wie viele Reads benötigt werden hängt in erster Linie von den vorgegebenen Qualitätsansprüchen ab. D.h. Wie groß darf die Wahrscheinlichkeit sein, dass eine Base garnicht, nur einmal, nur zweimal etc. sequenziert wurde? Beispiel: Ein Genom von 5 Mb ( Basen) soll mit der shotgun Methode sequenziert werden. Ein Read habe eine Länge von 500 Basen. Wieviele Reads werden benötigt, damit 99 % der Basen mindestens zweimal sequenziert wurden? Die Wahrscheinlichkeit p, dass eine bestimmte Base von einem zufälligen Read abgedeckt wird ist angenähert p = D.h. einfache Abdeckung entspricht /500 = Reads. Die Wahrscheinlichkeit, dass eine Base bei n Reads genau k mal sequenziert wurde (k Hits) folgt einer Binomialverteilung: 5
6 ( ) n P (k p, n) = p k (1 p) n k k Betrachten wir die Wahrscheinlichkeitsverteilung für eine zunehmende Zahl von Reads: n= reads n= reads n= reads n= reads Wie man sieht, werden bei Reads ca. 37 % der Basen von keinem Read abgedeckt. Bei Reads sind es nur noch ca %, bei ca. 1.8 %. Bei Reads werden nur noch 0.25 % nicht sequenziert und ca. 1.5 % werden nur von einem Read abgedeckt. Unser Ziel war es, dass 99 % der Basen mindestens zweimal sequenziert wurden also maximal 1 % der Basen Null oder einen Hit hatten. Der folgende Graph zeigt die Anzahl der zu erwartenden Basen mit keinem, einem oder weniger als zwei Hits in Anhängigkeit von der Anzahl der Reads: 6
7 Expected hits vs. number of reads [%] % 0 Hits 1 Hit < 2 Hits 0e+00 2e+04 4e+04 6e+04 8e+04 1e+05 n Die blaue Kurve erreicht die gewünschte 1 % Marke bei ca Reads (64744 um genau zu sein); nur noch 0.15 % werden nicht abgedeckt. D.h. wir benötigen mindestens diese Anzahl von Reads um unsere Qualitätsansprüche zu erfüllen. Dies entspricht einer mittleren Abdeckung von 6.5 fach. Bei der angenommenen Genomgröße von 5 Mb sind 0.15 % aber immerhin 7500 unsequenzierte Basen d.h. maximal auch ebensoviele Lücken (Gaps). Zwar kann man die Reihenfolge der entstandenen Contigs, wie schon bei der herkömmlichen Methode beschrieben, meist anhand von Markern festlegen, ein Schließen der Gaps bleibt dennoch wünschenswert. Anstatt nun die Anzahl der Shotgun Reads zu erhöhen ist es aber ökonomischer, die Gaps durch gezielte Sequenzierung zu eliminieren. In der Praxis trifft man zusätzlich auf Regionen, die aus verschiedenen Gründen schwer zu sequenzieren sind und bei keiner noch so großen Anzahl von Shotgun Reads abgedeckt werden. Solche Problemfälle lassen sich (wenn überhaupt) nur durch gezielte Sequenzierung (mit bestimmten Tricks) lösen. 7
DNA-Sequenzierung. Martina Krause
 DNA-Sequenzierung Martina Krause Inhalt Methoden der DNA-Sequenzierung Methode nach Maxam - Gilbert Methode nach Sanger Einleitung Seit 1977 stehen zwei schnell arbeitende Möglichkeiten der Sequenzanalyse
DNA-Sequenzierung Martina Krause Inhalt Methoden der DNA-Sequenzierung Methode nach Maxam - Gilbert Methode nach Sanger Einleitung Seit 1977 stehen zwei schnell arbeitende Möglichkeiten der Sequenzanalyse
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren
 Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
Lineargleichungssysteme: Additions-/ Subtraktionsverfahren W. Kippels 22. Februar 2014 Inhaltsverzeichnis 1 Einleitung 2 2 Lineargleichungssysteme zweiten Grades 2 3 Lineargleichungssysteme höheren als
1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet und
 10. Methoden 1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet und welche Funktion haben diese bei der Sequenzierungsreaktion?
10. Methoden 1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet und welche Funktion haben diese bei der Sequenzierungsreaktion?
Professionelle Seminare im Bereich MS-Office
 Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Der Name BEREICH.VERSCHIEBEN() ist etwas unglücklich gewählt. Man kann mit der Funktion Bereiche zwar verschieben, man kann Bereiche aber auch verkleinern oder vergrößern. Besser wäre es, die Funktion
Würfelt man dabei je genau 10 - mal eine 1, 2, 3, 4, 5 und 6, so beträgt die Anzahl. der verschiedenen Reihenfolgen, in denen man dies tun kann, 60!.
 040304 Übung 9a Analysis, Abschnitt 4, Folie 8 Die Wahrscheinlichkeit, dass bei n - maliger Durchführung eines Zufallexperiments ein Ereignis A ( mit Wahrscheinlichkeit p p ( A ) ) für eine beliebige Anzahl
040304 Übung 9a Analysis, Abschnitt 4, Folie 8 Die Wahrscheinlichkeit, dass bei n - maliger Durchführung eines Zufallexperiments ein Ereignis A ( mit Wahrscheinlichkeit p p ( A ) ) für eine beliebige Anzahl
1 topologisches Sortieren
 Wolfgang Hönig / Andreas Ecke WS 09/0 topologisches Sortieren. Überblick. Solange noch Knoten vorhanden: a) Suche Knoten v, zu dem keine Kante führt (Falls nicht vorhanden keine topologische Sortierung
Wolfgang Hönig / Andreas Ecke WS 09/0 topologisches Sortieren. Überblick. Solange noch Knoten vorhanden: a) Suche Knoten v, zu dem keine Kante führt (Falls nicht vorhanden keine topologische Sortierung
Erstellen von x-y-diagrammen in OpenOffice.calc
 Erstellen von x-y-diagrammen in OpenOffice.calc In dieser kleinen Anleitung geht es nur darum, aus einer bestehenden Tabelle ein x-y-diagramm zu erzeugen. D.h. es müssen in der Tabelle mindestens zwei
Erstellen von x-y-diagrammen in OpenOffice.calc In dieser kleinen Anleitung geht es nur darum, aus einer bestehenden Tabelle ein x-y-diagramm zu erzeugen. D.h. es müssen in der Tabelle mindestens zwei
DNA Sequenzierung. Transkriptionsstart bestimmen PCR
 10. Methoden DNA Sequenzierung Transkriptionsstart bestimmen PCR 1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet
10. Methoden DNA Sequenzierung Transkriptionsstart bestimmen PCR 1. Erklären Sie das Prinzip der Sanger Sequenzierung. Klären Sie dabei folgende Punkte: a) Welche besondere Art von Nukleotiden wird verwendet
Jede Zahl muss dabei einzeln umgerechnet werden. Beginnen wir also ganz am Anfang mit der Zahl,192.
 Binäres und dezimales Zahlensystem Ziel In diesem ersten Schritt geht es darum, die grundlegende Umrechnung aus dem Dezimalsystem in das Binärsystem zu verstehen. Zusätzlich wird auch die andere Richtung,
Binäres und dezimales Zahlensystem Ziel In diesem ersten Schritt geht es darum, die grundlegende Umrechnung aus dem Dezimalsystem in das Binärsystem zu verstehen. Zusätzlich wird auch die andere Richtung,
Repetitionsaufgaben Wurzelgleichungen
 Repetitionsaufgaben Wurzelgleichungen Inhaltsverzeichnis A) Vorbemerkungen B) Lernziele C) Theorie mit Aufgaben D) Aufgaben mit Musterlösungen 4 A) Vorbemerkungen Bitte beachten Sie: Bei Wurzelgleichungen
Repetitionsaufgaben Wurzelgleichungen Inhaltsverzeichnis A) Vorbemerkungen B) Lernziele C) Theorie mit Aufgaben D) Aufgaben mit Musterlösungen 4 A) Vorbemerkungen Bitte beachten Sie: Bei Wurzelgleichungen
Die Übereckperspektive mit zwei Fluchtpunkten
 Perspektive Perspektive mit zwei Fluchtpunkten (S. 1 von 8) / www.kunstbrowser.de Die Übereckperspektive mit zwei Fluchtpunkten Bei dieser Perspektivart wird der rechtwinklige Körper so auf die Grundebene
Perspektive Perspektive mit zwei Fluchtpunkten (S. 1 von 8) / www.kunstbrowser.de Die Übereckperspektive mit zwei Fluchtpunkten Bei dieser Perspektivart wird der rechtwinklige Körper so auf die Grundebene
geben. Die Wahrscheinlichkeit von 100% ist hier demnach nur der Gehen wir einmal davon aus, dass die von uns angenommenen
 geben. Die Wahrscheinlichkeit von 100% ist hier demnach nur der Vollständigkeit halber aufgeführt. Gehen wir einmal davon aus, dass die von uns angenommenen 70% im Beispiel exakt berechnet sind. Was würde
geben. Die Wahrscheinlichkeit von 100% ist hier demnach nur der Vollständigkeit halber aufgeführt. Gehen wir einmal davon aus, dass die von uns angenommenen 70% im Beispiel exakt berechnet sind. Was würde
Dow Jones am 13.06.08 im 1-min Chat
 Dow Jones am 13.06.08 im 1-min Chat Dieser Ausschnitt ist eine Formation: Wechselstäbe am unteren Bollinger Band mit Punkt d über dem 20-er GD nach 3 tieferen Hoch s. Wenn ich einen Ausbruch aus Wechselstäben
Dow Jones am 13.06.08 im 1-min Chat Dieser Ausschnitt ist eine Formation: Wechselstäbe am unteren Bollinger Band mit Punkt d über dem 20-er GD nach 3 tieferen Hoch s. Wenn ich einen Ausbruch aus Wechselstäben
Das große ElterngeldPlus 1x1. Alles über das ElterngeldPlus. Wer kann ElterngeldPlus beantragen? ElterngeldPlus verstehen ein paar einleitende Fakten
 Das große x -4 Alles über das Wer kann beantragen? Generell kann jeder beantragen! Eltern (Mütter UND Väter), die schon während ihrer Elternzeit wieder in Teilzeit arbeiten möchten. Eltern, die während
Das große x -4 Alles über das Wer kann beantragen? Generell kann jeder beantragen! Eltern (Mütter UND Väter), die schon während ihrer Elternzeit wieder in Teilzeit arbeiten möchten. Eltern, die während
Thema: Winkel in der Geometrie:
 Thema: Winkel in der Geometrie: Zuerst ist es wichtig zu wissen, welche Winkel es gibt: - Nullwinkel: 0 - spitzer Winkel: 1-89 (Bild 1) - rechter Winkel: genau 90 (Bild 2) - stumpfer Winkel: 91-179 (Bild
Thema: Winkel in der Geometrie: Zuerst ist es wichtig zu wissen, welche Winkel es gibt: - Nullwinkel: 0 - spitzer Winkel: 1-89 (Bild 1) - rechter Winkel: genau 90 (Bild 2) - stumpfer Winkel: 91-179 (Bild
1. Man schreibe die folgenden Aussagen jeweils in einen normalen Satz um. Zum Beispiel kann man die Aussage:
 Zählen und Zahlbereiche Übungsblatt 1 1. Man schreibe die folgenden Aussagen jeweils in einen normalen Satz um. Zum Beispiel kann man die Aussage: Für alle m, n N gilt m + n = n + m. in den Satz umschreiben:
Zählen und Zahlbereiche Übungsblatt 1 1. Man schreibe die folgenden Aussagen jeweils in einen normalen Satz um. Zum Beispiel kann man die Aussage: Für alle m, n N gilt m + n = n + m. in den Satz umschreiben:
Versuch 8. Plasmid - Isolierung
 Versuch 8 Plasmid - Isolierung Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@quantentunnel.de X X X C. Weindel & M. Schwarz Wird benotet?: Einleitung Ein Plasmid
Versuch 8 Plasmid - Isolierung Protokollant: E-mail: Studiengang: Gruppen-Nr: Semester: Betreuer: Max Mustermann max@quantentunnel.de X X X C. Weindel & M. Schwarz Wird benotet?: Einleitung Ein Plasmid
Kurzanleitung OOVS. Reseller Interface. Allgemein
 Kurzanleitung OOVS Reseller Interface Allgemein Durch die Einführung des neuen Interfaces hat sich für Reseller von Syswebcom etwas geändert. Die Struktur der Kundenverwaltung ist einprägsamer, wenn man
Kurzanleitung OOVS Reseller Interface Allgemein Durch die Einführung des neuen Interfaces hat sich für Reseller von Syswebcom etwas geändert. Die Struktur der Kundenverwaltung ist einprägsamer, wenn man
Austausch- bzw. Übergangsprozesse und Gleichgewichtsverteilungen
 Austausch- bzw. Übergangsrozesse und Gleichgewichtsverteilungen Wir betrachten ein System mit verschiedenen Zuständen, zwischen denen ein Austausch stattfinden kann. Etwa soziale Schichten in einer Gesellschaft:
Austausch- bzw. Übergangsrozesse und Gleichgewichtsverteilungen Wir betrachten ein System mit verschiedenen Zuständen, zwischen denen ein Austausch stattfinden kann. Etwa soziale Schichten in einer Gesellschaft:
Lineare Gleichungssysteme
 Lineare Gleichungssysteme 1 Zwei Gleichungen mit zwei Unbekannten Es kommt häufig vor, dass man nicht mit einer Variablen alleine auskommt, um ein Problem zu lösen. Das folgende Beispiel soll dies verdeutlichen
Lineare Gleichungssysteme 1 Zwei Gleichungen mit zwei Unbekannten Es kommt häufig vor, dass man nicht mit einer Variablen alleine auskommt, um ein Problem zu lösen. Das folgende Beispiel soll dies verdeutlichen
Menü auf zwei Module verteilt (Joomla 3.4.0)
 Menü auf zwei Module verteilt (Joomla 3.4.0) Oft wird bei Joomla das Menü in einem Modul dargestellt, wenn Sie aber z.b. ein horizontales Hauptmenü mit einem vertikalen Untermenü machen möchten, dann finden
Menü auf zwei Module verteilt (Joomla 3.4.0) Oft wird bei Joomla das Menü in einem Modul dargestellt, wenn Sie aber z.b. ein horizontales Hauptmenü mit einem vertikalen Untermenü machen möchten, dann finden
Aufgabe 12 Nach dem Eintippen der Kantenlänge soll die folgende Tabelle den Rauminhalt und die Oberfläche eines Würfels automatisch berechnen.
 Aufgabe 11 Excel hat für alles eine Lösung. So kann das Programm automatisch den größten oder den kleinsten Wert einer Tabelle bestimmen. Wenn man die richtige Funktion kennt, ist das überhaupt kein Problem.
Aufgabe 11 Excel hat für alles eine Lösung. So kann das Programm automatisch den größten oder den kleinsten Wert einer Tabelle bestimmen. Wenn man die richtige Funktion kennt, ist das überhaupt kein Problem.
Die Größe von Flächen vergleichen
 Vertiefen 1 Die Größe von Flächen vergleichen zu Aufgabe 1 Schulbuch, Seite 182 1 Wer hat am meisten Platz? Ordne die Figuren nach ihrem Flächeninhalt. Begründe deine Reihenfolge. 1 2 3 4 zu Aufgabe 2
Vertiefen 1 Die Größe von Flächen vergleichen zu Aufgabe 1 Schulbuch, Seite 182 1 Wer hat am meisten Platz? Ordne die Figuren nach ihrem Flächeninhalt. Begründe deine Reihenfolge. 1 2 3 4 zu Aufgabe 2
5. Bildauflösung ICT-Komp 10
 5. Bildauflösung ICT-Komp 10 Was sind dpi? Das Maß für die Bildauflösung eines Bildes sind dpi. Jeder spricht davon, aber oft weiß man gar nicht genau was das ist. Die Bezeichnung "dpi" ist ein Maß, mit
5. Bildauflösung ICT-Komp 10 Was sind dpi? Das Maß für die Bildauflösung eines Bildes sind dpi. Jeder spricht davon, aber oft weiß man gar nicht genau was das ist. Die Bezeichnung "dpi" ist ein Maß, mit
Was meinen die Leute eigentlich mit: Grexit?
 Was meinen die Leute eigentlich mit: Grexit? Grexit sind eigentlich 2 Wörter. 1. Griechenland 2. Exit Exit ist ein englisches Wort. Es bedeutet: Ausgang. Aber was haben diese 2 Sachen mit-einander zu tun?
Was meinen die Leute eigentlich mit: Grexit? Grexit sind eigentlich 2 Wörter. 1. Griechenland 2. Exit Exit ist ein englisches Wort. Es bedeutet: Ausgang. Aber was haben diese 2 Sachen mit-einander zu tun?
Leichte-Sprache-Bilder
 Leichte-Sprache-Bilder Reinhild Kassing Information - So geht es 1. Bilder gucken 2. anmelden für Probe-Bilder 3. Bilder bestellen 4. Rechnung bezahlen 5. Bilder runterladen 6. neue Bilder vorschlagen
Leichte-Sprache-Bilder Reinhild Kassing Information - So geht es 1. Bilder gucken 2. anmelden für Probe-Bilder 3. Bilder bestellen 4. Rechnung bezahlen 5. Bilder runterladen 6. neue Bilder vorschlagen
Primzahlen und RSA-Verschlüsselung
 Primzahlen und RSA-Verschlüsselung Michael Fütterer und Jonathan Zachhuber 1 Einiges zu Primzahlen Ein paar Definitionen: Wir bezeichnen mit Z die Menge der positiven und negativen ganzen Zahlen, also
Primzahlen und RSA-Verschlüsselung Michael Fütterer und Jonathan Zachhuber 1 Einiges zu Primzahlen Ein paar Definitionen: Wir bezeichnen mit Z die Menge der positiven und negativen ganzen Zahlen, also
Übungsaufgaben Prozentrechnung und / oder Dreisatz
 Übungsaufgaben Prozentrechnung und / oder Dreisatz 1. Bei der Wahl des Universitätssprechers wurden 800 gültige Stimmen abgegeben. Die Stimmen verteilten sich so auf die drei Kandidat/innen: A bekam 300,
Übungsaufgaben Prozentrechnung und / oder Dreisatz 1. Bei der Wahl des Universitätssprechers wurden 800 gültige Stimmen abgegeben. Die Stimmen verteilten sich so auf die drei Kandidat/innen: A bekam 300,
Sequenzierung. Aufklärung der Primärstruktur von DNA. Biotechnik Kurs WS 2006/07
 Sequenzierung Aufklärung der Primärstruktur von DNA Biotechnik Kurs WS 2006/07 AK Engels Angelika Keller Übersicht Geschichtlicher Hintergrund Maxam-Gilbert Sequenzierung Sanger Sequenzierung Neuere Sequenzierungstechnologien
Sequenzierung Aufklärung der Primärstruktur von DNA Biotechnik Kurs WS 2006/07 AK Engels Angelika Keller Übersicht Geschichtlicher Hintergrund Maxam-Gilbert Sequenzierung Sanger Sequenzierung Neuere Sequenzierungstechnologien
Installationsanleitung für das KKL bzw. AGV4000 Interface
 Installationsanleitung für das KKL bzw. AGV4000 Interface Diese Anleitung ist unter Windows XP erstellt worden, ist aber auch übertragbar auf Windows 2000/ Vista / Windows 7. Je nach Einstellungen des
Installationsanleitung für das KKL bzw. AGV4000 Interface Diese Anleitung ist unter Windows XP erstellt worden, ist aber auch übertragbar auf Windows 2000/ Vista / Windows 7. Je nach Einstellungen des
1 Mathematische Grundlagen
 Mathematische Grundlagen - 1-1 Mathematische Grundlagen Der Begriff der Menge ist einer der grundlegenden Begriffe in der Mathematik. Mengen dienen dazu, Dinge oder Objekte zu einer Einheit zusammenzufassen.
Mathematische Grundlagen - 1-1 Mathematische Grundlagen Der Begriff der Menge ist einer der grundlegenden Begriffe in der Mathematik. Mengen dienen dazu, Dinge oder Objekte zu einer Einheit zusammenzufassen.
Erstellen einer Collage. Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu])
![Erstellen einer Collage. Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu]) Erstellen einer Collage. Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu])](/thumbs/29/13170670.jpg) 3.7 Erstellen einer Collage Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu]) Dann Größe des Dokuments festlegen beispielsweise A4 (weitere
3.7 Erstellen einer Collage Zuerst ein leeres Dokument erzeugen, auf dem alle anderen Bilder zusammengefügt werden sollen (über [Datei] > [Neu]) Dann Größe des Dokuments festlegen beispielsweise A4 (weitere
Güte von Tests. die Wahrscheinlichkeit für den Fehler 2. Art bei der Testentscheidung, nämlich. falsch ist. Darauf haben wir bereits im Kapitel über
 Güte von s Grundlegendes zum Konzept der Güte Ableitung der Gütefunktion des Gauss im Einstichprobenproblem Grafische Darstellung der Gütefunktionen des Gauss im Einstichprobenproblem Ableitung der Gütefunktion
Güte von s Grundlegendes zum Konzept der Güte Ableitung der Gütefunktion des Gauss im Einstichprobenproblem Grafische Darstellung der Gütefunktionen des Gauss im Einstichprobenproblem Ableitung der Gütefunktion
Lineare Funktionen. 1 Proportionale Funktionen 3 1.1 Definition... 3 1.2 Eigenschaften... 3. 2 Steigungsdreieck 3
 Lineare Funktionen Inhaltsverzeichnis 1 Proportionale Funktionen 3 1.1 Definition............................... 3 1.2 Eigenschaften............................. 3 2 Steigungsdreieck 3 3 Lineare Funktionen
Lineare Funktionen Inhaltsverzeichnis 1 Proportionale Funktionen 3 1.1 Definition............................... 3 1.2 Eigenschaften............................. 3 2 Steigungsdreieck 3 3 Lineare Funktionen
WORKSHOP für das Programm XnView
 WORKSHOP für das Programm XnView Zur Bearbeitung von Fotos für die Nutzung auf Websites und Online Bildergalerien www.xnview.de STEP 1 ) Bild öffnen und Größe ändern STEP 2 ) Farbmodus prüfen und einstellen
WORKSHOP für das Programm XnView Zur Bearbeitung von Fotos für die Nutzung auf Websites und Online Bildergalerien www.xnview.de STEP 1 ) Bild öffnen und Größe ändern STEP 2 ) Farbmodus prüfen und einstellen
Schrittweise Anleitung zur Erstellung einer Angebotseite 1. In Ihrem Dashboard klicken Sie auf Neu anlegen, um eine neue Seite zu erstellen.
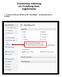 Schrittweise Anleitung zur Erstellung einer Angebotseite 1. In Ihrem Dashboard klicken Sie auf Neu anlegen, um eine neue Seite zu erstellen. Klicken Sie auf Neu anlegen, um Ihre neue Angebotseite zu erstellen..
Schrittweise Anleitung zur Erstellung einer Angebotseite 1. In Ihrem Dashboard klicken Sie auf Neu anlegen, um eine neue Seite zu erstellen. Klicken Sie auf Neu anlegen, um Ihre neue Angebotseite zu erstellen..
icloud nicht neu, aber doch irgendwie anders
 Kapitel 6 In diesem Kapitel zeigen wir Ihnen, welche Dienste die icloud beim Abgleich von Dateien und Informationen anbietet. Sie lernen icloud Drive kennen, den Fotostream, den icloud-schlüsselbund und
Kapitel 6 In diesem Kapitel zeigen wir Ihnen, welche Dienste die icloud beim Abgleich von Dateien und Informationen anbietet. Sie lernen icloud Drive kennen, den Fotostream, den icloud-schlüsselbund und
Überprüfung der digital signierten E-Rechnung
 Überprüfung der digital signierten E-Rechnung Aufgrund des BMF-Erlasses vom Juli 2005 (BMF-010219/0183-IV/9/2005) gelten ab 01.01.2006 nur noch jene elektronischen Rechnungen als vorsteuerabzugspflichtig,
Überprüfung der digital signierten E-Rechnung Aufgrund des BMF-Erlasses vom Juli 2005 (BMF-010219/0183-IV/9/2005) gelten ab 01.01.2006 nur noch jene elektronischen Rechnungen als vorsteuerabzugspflichtig,
Alle Schlüssel-Karten (blaue Rückseite) werden den Schlüssel-Farben nach sortiert und in vier getrennte Stapel mit der Bildseite nach oben gelegt.
 Gentlemen", bitte zur Kasse! Ravensburger Spiele Nr. 01 264 0 Autoren: Wolfgang Kramer und Jürgen P. K. Grunau Grafik: Erhard Dietl Ein Gaunerspiel für 3-6 Gentlemen" ab 10 Jahren Inhalt: 35 Tresor-Karten
Gentlemen", bitte zur Kasse! Ravensburger Spiele Nr. 01 264 0 Autoren: Wolfgang Kramer und Jürgen P. K. Grunau Grafik: Erhard Dietl Ein Gaunerspiel für 3-6 Gentlemen" ab 10 Jahren Inhalt: 35 Tresor-Karten
Anwendungsbeispiele Buchhaltung
 Rechnungen erstellen mit Webling Webling ist ein Produkt der Firma: Inhaltsverzeichnis 1 Rechnungen erstellen mit Webling 1.1 Rechnung erstellen und ausdrucken 1.2 Rechnung mit Einzahlungsschein erstellen
Rechnungen erstellen mit Webling Webling ist ein Produkt der Firma: Inhaltsverzeichnis 1 Rechnungen erstellen mit Webling 1.1 Rechnung erstellen und ausdrucken 1.2 Rechnung mit Einzahlungsschein erstellen
Reporting Services und SharePoint 2010 Teil 1
 Reporting Services und SharePoint 2010 Teil 1 Abstract Bei der Verwendung der Reporting Services in Zusammenhang mit SharePoint 2010 stellt sich immer wieder die Frage bei der Installation: Wo und Wie?
Reporting Services und SharePoint 2010 Teil 1 Abstract Bei der Verwendung der Reporting Services in Zusammenhang mit SharePoint 2010 stellt sich immer wieder die Frage bei der Installation: Wo und Wie?
Hilfe zur Urlaubsplanung und Zeiterfassung
 Hilfe zur Urlaubsplanung und Zeiterfassung Urlaubs- und Arbeitsplanung: Mit der Urlaubs- und Arbeitsplanung kann jeder Mitarbeiter in Coffee seine Zeiten eintragen. Die Eintragung kann mit dem Status anfragen,
Hilfe zur Urlaubsplanung und Zeiterfassung Urlaubs- und Arbeitsplanung: Mit der Urlaubs- und Arbeitsplanung kann jeder Mitarbeiter in Coffee seine Zeiten eintragen. Die Eintragung kann mit dem Status anfragen,
... ... Sicherheitseinstellungen... 2 Pop-up-Fenster erlauben... 3
 Browsereinstellungen Um die Know How! Lernprogramm nutzen zu können, sind bei Bedarf unterschiedliche Einstellungen in Ihren Browsern nötig. Im Folgenden finden Sie die Beschreibung für unterschiedliche
Browsereinstellungen Um die Know How! Lernprogramm nutzen zu können, sind bei Bedarf unterschiedliche Einstellungen in Ihren Browsern nötig. Im Folgenden finden Sie die Beschreibung für unterschiedliche
Sich einen eigenen Blog anzulegen, ist gar nicht so schwer. Es gibt verschiedene Anbieter. www.blogger.com ist einer davon.
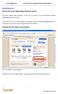 www.blogger.com Sich einen eigenen Blog anzulegen, ist gar nicht so schwer. Es gibt verschiedene Anbieter. www.blogger.com ist einer davon. Sie müssen sich dort nur ein Konto anlegen. Dafür gehen Sie auf
www.blogger.com Sich einen eigenen Blog anzulegen, ist gar nicht so schwer. Es gibt verschiedene Anbieter. www.blogger.com ist einer davon. Sie müssen sich dort nur ein Konto anlegen. Dafür gehen Sie auf
Stellen Sie bitte den Cursor in die Spalte B2 und rufen die Funktion Sverweis auf. Es öffnet sich folgendes Dialogfenster
 Es gibt in Excel unter anderem die so genannten Suchfunktionen / Matrixfunktionen Damit können Sie Werte innerhalb eines bestimmten Bereichs suchen. Als Beispiel möchte ich die Funktion Sverweis zeigen.
Es gibt in Excel unter anderem die so genannten Suchfunktionen / Matrixfunktionen Damit können Sie Werte innerhalb eines bestimmten Bereichs suchen. Als Beispiel möchte ich die Funktion Sverweis zeigen.
3. Verpackungskünstler. Berechnungen am Quader, Umgang mit Termen, räumliche Vorstellung
 Berechnungen am Quader, Umgang mit Termen, räumliche Vorstellung Päckchen, die man verschenken möchte, werden gerne mit Geschenkband verschnürt. Dazu wird das Päckchen auf seine größte Seite gelegt, wie
Berechnungen am Quader, Umgang mit Termen, räumliche Vorstellung Päckchen, die man verschenken möchte, werden gerne mit Geschenkband verschnürt. Dazu wird das Päckchen auf seine größte Seite gelegt, wie
40-Tage-Wunder- Kurs. Umarme, was Du nicht ändern kannst.
 40-Tage-Wunder- Kurs Umarme, was Du nicht ändern kannst. Das sagt Wikipedia: Als Wunder (griechisch thauma) gilt umgangssprachlich ein Ereignis, dessen Zustandekommen man sich nicht erklären kann, so dass
40-Tage-Wunder- Kurs Umarme, was Du nicht ändern kannst. Das sagt Wikipedia: Als Wunder (griechisch thauma) gilt umgangssprachlich ein Ereignis, dessen Zustandekommen man sich nicht erklären kann, so dass
Kugel-Fächer-Modell. 1fach. 3fach. Für die Einzelkugel gibt es 3 Möglichkeiten. 6fach. 3! Möglichkeiten
 Kugel-Fächer-Modell n Kugeln (Rosinen) sollen auf m Fächer (Brötchen) verteilt werden, zunächst 3 Kugeln auf 3 Fächer. 1fach 3fach Für die Einzelkugel gibt es 3 Möglichkeiten } 6fach 3! Möglichkeiten Es
Kugel-Fächer-Modell n Kugeln (Rosinen) sollen auf m Fächer (Brötchen) verteilt werden, zunächst 3 Kugeln auf 3 Fächer. 1fach 3fach Für die Einzelkugel gibt es 3 Möglichkeiten } 6fach 3! Möglichkeiten Es
AGROPLUS Buchhaltung. Daten-Server und Sicherheitskopie. Version vom 21.10.2013b
 AGROPLUS Buchhaltung Daten-Server und Sicherheitskopie Version vom 21.10.2013b 3a) Der Daten-Server Modus und der Tresor Der Daten-Server ist eine Betriebsart welche dem Nutzer eine grosse Flexibilität
AGROPLUS Buchhaltung Daten-Server und Sicherheitskopie Version vom 21.10.2013b 3a) Der Daten-Server Modus und der Tresor Der Daten-Server ist eine Betriebsart welche dem Nutzer eine grosse Flexibilität
Dokumentation für das Spiel Pong
 Dokumentation für das Spiel Pong BwInf - Turnierserver Didaktik der nformatik BWINF KI Wettbewerbs-Plattform Stand: 02.09.2014 Grundlagen In diesem KI-Turnier programmiert ihr einen Schläger für das Retro-Spiel
Dokumentation für das Spiel Pong BwInf - Turnierserver Didaktik der nformatik BWINF KI Wettbewerbs-Plattform Stand: 02.09.2014 Grundlagen In diesem KI-Turnier programmiert ihr einen Schläger für das Retro-Spiel
Wie Sie mit Mastern arbeiten
 Wie Sie mit Mastern arbeiten Was ist ein Master? Einer der großen Vorteile von EDV besteht darin, dass Ihnen der Rechner Arbeit abnimmt. Diesen Vorteil sollten sie nutzen, wo immer es geht. In PowerPoint
Wie Sie mit Mastern arbeiten Was ist ein Master? Einer der großen Vorteile von EDV besteht darin, dass Ihnen der Rechner Arbeit abnimmt. Diesen Vorteil sollten sie nutzen, wo immer es geht. In PowerPoint
lohmeyer White Paper Use Cases II UX+Prozessanalyse
 White Paper Use Cases II Use Cases begleiten uns in der IT seit mehr als 15 Jahren. Nichtsdestotrotz ist es nicht so einfach, Use Cases einfach und verständlich zu schreiben. Dieses White Paper spricht
White Paper Use Cases II Use Cases begleiten uns in der IT seit mehr als 15 Jahren. Nichtsdestotrotz ist es nicht so einfach, Use Cases einfach und verständlich zu schreiben. Dieses White Paper spricht
Suche schlecht beschriftete Bilder mit Eigenen Abfragen
 Suche schlecht beschriftete Bilder mit Eigenen Abfragen Ist die Bilderdatenbank über einen längeren Zeitraum in Benutzung, so steigt die Wahrscheinlichkeit für schlecht beschriftete Bilder 1. Insbesondere
Suche schlecht beschriftete Bilder mit Eigenen Abfragen Ist die Bilderdatenbank über einen längeren Zeitraum in Benutzung, so steigt die Wahrscheinlichkeit für schlecht beschriftete Bilder 1. Insbesondere
(für Grund- und Leistungskurse Mathematik) 26W55DLQHU0DUWLQ(KUHQE UJ*\PQDVLXP)RUFKKHLP
 .RPELQDWRULN (für Grund- und Leistungsurse Mathemati) 6W55DLQHU0DUWLQ(KUHQE UJ*\PQDVLXP)RUFKKHLP Nach dem Studium dieses Sripts sollten folgende Begriffe beannt sein: n-menge, Kreuzprodut, n-tupel Zählprinzip
.RPELQDWRULN (für Grund- und Leistungsurse Mathemati) 6W55DLQHU0DUWLQ(KUHQE UJ*\PQDVLXP)RUFKKHLP Nach dem Studium dieses Sripts sollten folgende Begriffe beannt sein: n-menge, Kreuzprodut, n-tupel Zählprinzip
Tangentengleichung. Wie lautet die Geradengleichung für die Tangente, y T =? Antwort:
 Tangentengleichung Wie Sie wissen, gibt die erste Ableitung einer Funktion deren Steigung an. Betrachtet man eine fest vorgegebene Stelle, gibt f ( ) also die Steigung der Kurve und somit auch die Steigung
Tangentengleichung Wie Sie wissen, gibt die erste Ableitung einer Funktion deren Steigung an. Betrachtet man eine fest vorgegebene Stelle, gibt f ( ) also die Steigung der Kurve und somit auch die Steigung
Was ist clevere Altersvorsorge?
 Was ist clevere Altersvorsorge? Um eine gute Altersvorsorge zu erreichen, ist es clever einen unabhängigen Berater auszuwählen Angestellte bzw. Berater von Banken, Versicherungen, Fondsgesellschaften und
Was ist clevere Altersvorsorge? Um eine gute Altersvorsorge zu erreichen, ist es clever einen unabhängigen Berater auszuwählen Angestellte bzw. Berater von Banken, Versicherungen, Fondsgesellschaften und
Abituraufgabe zur Stochastik, Hessen 2009, Grundkurs (TR)
 Abituraufgabe zur Stochastik, Hessen 2009, Grundkurs (TR) Eine Firma stellt USB-Sticks her. Sie werden in der Fabrik ungeprüft in Packungen zu je 20 Stück verpackt und an Händler ausgeliefert. 1 Ein Händler
Abituraufgabe zur Stochastik, Hessen 2009, Grundkurs (TR) Eine Firma stellt USB-Sticks her. Sie werden in der Fabrik ungeprüft in Packungen zu je 20 Stück verpackt und an Händler ausgeliefert. 1 Ein Händler
Das Prinzip der DNA-Sequenzierung Best.- Nr. 201.3055
 Das Prinzip der DNA-Sequenzierung Best.- Nr. 201.3055 Allgemeine Informationen Prinzip der DNA-Sequenzierung Ziel dieses Versuchs ist einen genauen Überblick über die Verfahrensweise der DNA- Sequenzierung
Das Prinzip der DNA-Sequenzierung Best.- Nr. 201.3055 Allgemeine Informationen Prinzip der DNA-Sequenzierung Ziel dieses Versuchs ist einen genauen Überblick über die Verfahrensweise der DNA- Sequenzierung
Aktivierung der SeKA-Anmeldung
 Aktivierung der SeKA-Anmeldung Bitte, benutzen Sie diese Methode nur, wenn Sie gerade kein Kennwort im SeKA-System erhalten haben. Wenn Sie die Aktivierung vorher schon geschafft, aber das Kennwort vergessen
Aktivierung der SeKA-Anmeldung Bitte, benutzen Sie diese Methode nur, wenn Sie gerade kein Kennwort im SeKA-System erhalten haben. Wenn Sie die Aktivierung vorher schon geschafft, aber das Kennwort vergessen
mysql - Clients MySQL - Abfragen eine serverbasierenden Datenbank
 mysql - Clients MySQL - Abfragen eine serverbasierenden Datenbank In den ersten beiden Abschnitten (rbanken1.pdf und rbanken2.pdf) haben wir uns mit am Ende mysql beschäftigt und kennengelernt, wie man
mysql - Clients MySQL - Abfragen eine serverbasierenden Datenbank In den ersten beiden Abschnitten (rbanken1.pdf und rbanken2.pdf) haben wir uns mit am Ende mysql beschäftigt und kennengelernt, wie man
Kapitalerhöhung - Verbuchung
 Kapitalerhöhung - Verbuchung Beschreibung Eine Kapitalerhöhung ist eine Erhöhung des Aktienkapitals einer Aktiengesellschaft durch Emission von en Aktien. Es gibt unterschiedliche Formen von Kapitalerhöhung.
Kapitalerhöhung - Verbuchung Beschreibung Eine Kapitalerhöhung ist eine Erhöhung des Aktienkapitals einer Aktiengesellschaft durch Emission von en Aktien. Es gibt unterschiedliche Formen von Kapitalerhöhung.
Guide DynDNS und Portforwarding
 Guide DynDNS und Portforwarding Allgemein Um Geräte im lokalen Netzwerk von überall aus über das Internet erreichen zu können, kommt man um die Themen Dynamik DNS (kurz DynDNS) und Portweiterleitung(auch
Guide DynDNS und Portforwarding Allgemein Um Geräte im lokalen Netzwerk von überall aus über das Internet erreichen zu können, kommt man um die Themen Dynamik DNS (kurz DynDNS) und Portweiterleitung(auch
Backup Premium Kurzleitfaden
 Info Memeo Backup Premium bietet viele fortschrittliche automatische Backup-Funktionen und ist großartig für Benutzer von Digitalkameras und für Anwender, die bis zu 50.000 Dateien mit Backups sichern
Info Memeo Backup Premium bietet viele fortschrittliche automatische Backup-Funktionen und ist großartig für Benutzer von Digitalkameras und für Anwender, die bis zu 50.000 Dateien mit Backups sichern
Simplex-Umformung für Dummies
 Simplex-Umformung für Dummies Enthält die Zielfunktion einen negativen Koeffizienten? NEIN Optimale Lösung bereits gefunden JA Finde die Optimale Lösung mit dem Simplex-Verfahren! Wähle die Spalte mit
Simplex-Umformung für Dummies Enthält die Zielfunktion einen negativen Koeffizienten? NEIN Optimale Lösung bereits gefunden JA Finde die Optimale Lösung mit dem Simplex-Verfahren! Wähle die Spalte mit
Eine der Aktien hat immer einen höheren Gewinn als die andere Aktie. Ihre Aufgabe ist es diese auszuwählen.
 Instruktionen am Anfang von Experiment 1 (auf Papier ausgeteilt: grünmarkierte Textstellen zeigen den Instruktionstext in der jeweiligen Bedingung an; Kommentare sind gelb markiert.) Stellen Sie sich vor,
Instruktionen am Anfang von Experiment 1 (auf Papier ausgeteilt: grünmarkierte Textstellen zeigen den Instruktionstext in der jeweiligen Bedingung an; Kommentare sind gelb markiert.) Stellen Sie sich vor,
PowerPoint 2010 Mit Folienmastern arbeiten
 PP.002, Version 1.1 07.04.2015 Kurzanleitung PowerPoint 2010 Mit Folienmastern arbeiten Der Folienmaster ist die Vorlage für sämtliche Folien einer Präsentation. Er bestimmt das Design, die Farben, die
PP.002, Version 1.1 07.04.2015 Kurzanleitung PowerPoint 2010 Mit Folienmastern arbeiten Der Folienmaster ist die Vorlage für sämtliche Folien einer Präsentation. Er bestimmt das Design, die Farben, die
Informationsblatt Induktionsbeweis
 Sommer 015 Informationsblatt Induktionsbeweis 31. März 015 Motivation Die vollständige Induktion ist ein wichtiges Beweisverfahren in der Informatik. Sie wird häufig dazu gebraucht, um mathematische Formeln
Sommer 015 Informationsblatt Induktionsbeweis 31. März 015 Motivation Die vollständige Induktion ist ein wichtiges Beweisverfahren in der Informatik. Sie wird häufig dazu gebraucht, um mathematische Formeln
2.1 Erstellung einer Gutschrift über den vollen Rechnungsbetrag
 1. Einführung Manchmal ist es notwendig, dem Kunden eine Gutschrift zu einer bestimmten Rechnung, über einzelne Positionen oder auch völlig frei von einer Basis-Rechnung zu erstellen. Die verschiedenen
1. Einführung Manchmal ist es notwendig, dem Kunden eine Gutschrift zu einer bestimmten Rechnung, über einzelne Positionen oder auch völlig frei von einer Basis-Rechnung zu erstellen. Die verschiedenen
ONLINE-AKADEMIE. "Diplomierter NLP Anwender für Schule und Unterricht" Ziele
 ONLINE-AKADEMIE Ziele Wenn man von Menschen hört, die etwas Großartiges in ihrem Leben geleistet haben, erfahren wir oft, dass diese ihr Ziel über Jahre verfolgt haben oder diesen Wunsch schon bereits
ONLINE-AKADEMIE Ziele Wenn man von Menschen hört, die etwas Großartiges in ihrem Leben geleistet haben, erfahren wir oft, dass diese ihr Ziel über Jahre verfolgt haben oder diesen Wunsch schon bereits
NINA DEISSLER. Flirten. Wie wirke ich? Was kann ich sagen? Wie spiele ich meine Stärken aus?
 NINA DEISSLER Flirten Wie wirke ich? Was kann ich sagen? Wie spiele ich meine Stärken aus? Die Steinzeit lässt grüßen 19 es sonst zu erklären, dass Männer bei einer Möglichkeit zum One-Night-Stand mit
NINA DEISSLER Flirten Wie wirke ich? Was kann ich sagen? Wie spiele ich meine Stärken aus? Die Steinzeit lässt grüßen 19 es sonst zu erklären, dass Männer bei einer Möglichkeit zum One-Night-Stand mit
Jeopardy and andere Quizformate im bilingualen Sachfachunterricht Tipps zur Erstellung mit Powerpoint
 Bilingual konkret Jeopardy and andere Quizformate im bilingualen Sachfachunterricht Tipps zur Erstellung mit Powerpoint Moderner Unterricht ist ohne die Unterstützung durch Computer und das Internet fast
Bilingual konkret Jeopardy and andere Quizformate im bilingualen Sachfachunterricht Tipps zur Erstellung mit Powerpoint Moderner Unterricht ist ohne die Unterstützung durch Computer und das Internet fast
Internet Kurs. Suchmaschinen
 Internet Kurs Suchmaschinen M. Stalder Internetkurs M. Stalder 1 / 6 Suchmaschinen Suchmaschinen haben sich in letzter Zeit immer mehr zu einem unverzichtbaren Hilfsmittel entwickelt. Das Internet bietet
Internet Kurs Suchmaschinen M. Stalder Internetkurs M. Stalder 1 / 6 Suchmaschinen Suchmaschinen haben sich in letzter Zeit immer mehr zu einem unverzichtbaren Hilfsmittel entwickelt. Das Internet bietet
Datensicherung. Beschreibung der Datensicherung
 Datensicherung Mit dem Datensicherungsprogramm können Sie Ihre persönlichen Daten problemlos Sichern. Es ist möglich eine komplette Datensicherung durchzuführen, aber auch nur die neuen und geänderten
Datensicherung Mit dem Datensicherungsprogramm können Sie Ihre persönlichen Daten problemlos Sichern. Es ist möglich eine komplette Datensicherung durchzuführen, aber auch nur die neuen und geänderten
SJ OFFICE - Update 3.0
 SJ OFFICE - Update 3.0 Das Update auf die vorherige Version 2.0 kostet netto Euro 75,00 für die erste Lizenz. Das Update für weitere Lizenzen kostet jeweils netto Euro 18,75 (25%). inkl. Programmsupport
SJ OFFICE - Update 3.0 Das Update auf die vorherige Version 2.0 kostet netto Euro 75,00 für die erste Lizenz. Das Update für weitere Lizenzen kostet jeweils netto Euro 18,75 (25%). inkl. Programmsupport
DER SELBST-CHECK FÜR IHR PROJEKT
 DER SELBST-CHECK FÜR IHR PROJEKT In 30 Fragen und 5 Tipps zum erfolgreichen Projekt! Beantworten Sie die wichtigsten Fragen rund um Ihr Projekt für Ihren Erfolg und für Ihre Unterstützer. IHR LEITFADEN
DER SELBST-CHECK FÜR IHR PROJEKT In 30 Fragen und 5 Tipps zum erfolgreichen Projekt! Beantworten Sie die wichtigsten Fragen rund um Ihr Projekt für Ihren Erfolg und für Ihre Unterstützer. IHR LEITFADEN
1. Einführung 2. 2. Erstellung einer Teillieferung 2. 3. Erstellung einer Teilrechnung 6
 Inhalt 1. Einführung 2 2. Erstellung einer Teillieferung 2 3. Erstellung einer Teilrechnung 6 4. Erstellung einer Sammellieferung/ Mehrere Aufträge zu einem Lieferschein zusammenfassen 11 5. Besonderheiten
Inhalt 1. Einführung 2 2. Erstellung einer Teillieferung 2 3. Erstellung einer Teilrechnung 6 4. Erstellung einer Sammellieferung/ Mehrere Aufträge zu einem Lieferschein zusammenfassen 11 5. Besonderheiten
Webinar-Partnerprogramm
 Webinar-Partnerprogramm Mit dem Webinar-Partnerprogramm können Sie noch mehr Teilnehmer für Ihre Webinare gewinnen. Nutzen Sie Ihr Netzwerk, um Ihre Webinare noch besser zu vermarkten. Nutzen Sie das Webinar-Partnerprogramm,
Webinar-Partnerprogramm Mit dem Webinar-Partnerprogramm können Sie noch mehr Teilnehmer für Ihre Webinare gewinnen. Nutzen Sie Ihr Netzwerk, um Ihre Webinare noch besser zu vermarkten. Nutzen Sie das Webinar-Partnerprogramm,
Die Erstellung eigener Strukturprofile
 Die Erstellung eigener Strukturprofile Manchmal ist es nötig, eigene Profile zu Erstellen, die man dann mittels Gestellgenerator verbaut. Diese Strukturprofile werden in einer Benutzerbezogenen Bibliothek
Die Erstellung eigener Strukturprofile Manchmal ist es nötig, eigene Profile zu Erstellen, die man dann mittels Gestellgenerator verbaut. Diese Strukturprofile werden in einer Benutzerbezogenen Bibliothek
Lösungsmethoden gewöhnlicher Differentialgleichungen (Dgl.)
 Lösungsmethoden gewöhnlicher Dierentialgleichungen Dgl) Allgemeine und partikuläre Lösung einer gewöhnlichen Dierentialgleichung Eine Dierentialgleichung ist eine Gleichung! Zum Unterschied von den gewöhnlichen
Lösungsmethoden gewöhnlicher Dierentialgleichungen Dgl) Allgemeine und partikuläre Lösung einer gewöhnlichen Dierentialgleichung Eine Dierentialgleichung ist eine Gleichung! Zum Unterschied von den gewöhnlichen
iphone-kontakte zu Exchange übertragen
 iphone-kontakte zu Exchange übertragen Übertragen von iphone-kontakten in ein Exchange Postfach Zunächst muss das iphone an den Rechner, an dem es üblicherweise synchronisiert wird, angeschlossen werden.
iphone-kontakte zu Exchange übertragen Übertragen von iphone-kontakten in ein Exchange Postfach Zunächst muss das iphone an den Rechner, an dem es üblicherweise synchronisiert wird, angeschlossen werden.
Das Leitbild vom Verein WIR
 Das Leitbild vom Verein WIR Dieses Zeichen ist ein Gütesiegel. Texte mit diesem Gütesiegel sind leicht verständlich. Leicht Lesen gibt es in drei Stufen. B1: leicht verständlich A2: noch leichter verständlich
Das Leitbild vom Verein WIR Dieses Zeichen ist ein Gütesiegel. Texte mit diesem Gütesiegel sind leicht verständlich. Leicht Lesen gibt es in drei Stufen. B1: leicht verständlich A2: noch leichter verständlich
ACHTUNG: Es können gpx-dateien und mit dem GP7 aufgezeichnete trc-dateien umgewandelt werden.
 Track in Route umwandeln ACHTUNG: Ein Track kann nur dann in eine Route umgewandelt werden, wenn der Track auf Wegen gefahren wurde. Ein Querfeldein-Track kann nicht in eine Route umgewandelt werden, da
Track in Route umwandeln ACHTUNG: Ein Track kann nur dann in eine Route umgewandelt werden, wenn der Track auf Wegen gefahren wurde. Ein Querfeldein-Track kann nicht in eine Route umgewandelt werden, da
Unterrichtsmaterialien in digitaler und in gedruckter Form. Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis
 Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis Das komplette Material finden Sie hier: Download bei School-Scout.de
Unterrichtsmaterialien in digitaler und in gedruckter Form Auszug aus: Übungsbuch für den Grundkurs mit Tipps und Lösungen: Analysis Das komplette Material finden Sie hier: Download bei School-Scout.de
Gibt es einen Geschmacksunterschied zwischen Coca Cola und Cola Zero?
 Gibt es einen Geschmacksunterschied zwischen Coca Cola und Cola Zero? Manche sagen: Ja, manche sagen: Nein Wie soll man das objektiv feststellen? Kann man Geschmack objektiv messen? - Geschmack ist subjektiv
Gibt es einen Geschmacksunterschied zwischen Coca Cola und Cola Zero? Manche sagen: Ja, manche sagen: Nein Wie soll man das objektiv feststellen? Kann man Geschmack objektiv messen? - Geschmack ist subjektiv
Bewertung des Blattes
 Bewertung des Blattes Es besteht immer die Schwierigkeit, sein Blatt richtig einzuschätzen. Im folgenden werden einige Anhaltspunkte gegeben. Man unterscheidet: Figurenpunkte Verteilungspunkte Längenpunkte
Bewertung des Blattes Es besteht immer die Schwierigkeit, sein Blatt richtig einzuschätzen. Im folgenden werden einige Anhaltspunkte gegeben. Man unterscheidet: Figurenpunkte Verteilungspunkte Längenpunkte
Zeichen bei Zahlen entschlüsseln
 Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
Zeichen bei Zahlen entschlüsseln In diesem Kapitel... Verwendung des Zahlenstrahls Absolut richtige Bestimmung von absoluten Werten Operationen bei Zahlen mit Vorzeichen: Addieren, Subtrahieren, Multiplizieren
IRF2000 Application Note Lösung von IP-Adresskonflikten bei zwei identischen Netzwerken
 Version 2.0 1 Original-Application Note ads-tec GmbH IRF2000 Application Note Lösung von IP-Adresskonflikten bei zwei identischen Netzwerken Stand: 27.10.2014 ads-tec GmbH 2014 IRF2000 2 Inhaltsverzeichnis
Version 2.0 1 Original-Application Note ads-tec GmbH IRF2000 Application Note Lösung von IP-Adresskonflikten bei zwei identischen Netzwerken Stand: 27.10.2014 ads-tec GmbH 2014 IRF2000 2 Inhaltsverzeichnis
SCHRITT 1: Öffnen des Bildes und Auswahl der Option»Drucken«im Menü»Datei«...2. SCHRITT 2: Angeben des Papierformat im Dialog»Drucklayout«...
 Drucken - Druckformat Frage Wie passt man Bilder beim Drucken an bestimmte Papierformate an? Antwort Das Drucken von Bildern ist mit der Druckfunktion von Capture NX sehr einfach. Hier erklären wir, wie
Drucken - Druckformat Frage Wie passt man Bilder beim Drucken an bestimmte Papierformate an? Antwort Das Drucken von Bildern ist mit der Druckfunktion von Capture NX sehr einfach. Hier erklären wir, wie
Outlook. sysplus.ch outlook - mail-grundlagen Seite 1/8. Mail-Grundlagen. Posteingang
 sysplus.ch outlook - mail-grundlagen Seite 1/8 Outlook Mail-Grundlagen Posteingang Es gibt verschiedene Möglichkeiten, um zum Posteingang zu gelangen. Man kann links im Outlook-Fenster auf die Schaltfläche
sysplus.ch outlook - mail-grundlagen Seite 1/8 Outlook Mail-Grundlagen Posteingang Es gibt verschiedene Möglichkeiten, um zum Posteingang zu gelangen. Man kann links im Outlook-Fenster auf die Schaltfläche
SPIELBESCHREIBUNGEN DART RADIKAL SYSTEM
 SPIELBESCHREIBUNGEN DART RADIKAL SYSTEM X01: 301, 501, 701 oder 901 Rundenbegrenzungen: 301 (15); 501 (20); 701 (25); 901 (30) In diesem Spiel beginnen alle Spieler mit 301, 501, 701 oder 901 Punkten.
SPIELBESCHREIBUNGEN DART RADIKAL SYSTEM X01: 301, 501, 701 oder 901 Rundenbegrenzungen: 301 (15); 501 (20); 701 (25); 901 (30) In diesem Spiel beginnen alle Spieler mit 301, 501, 701 oder 901 Punkten.
Schritte 4. Lesetexte 13. Kosten für ein Girokonto vergleichen. 1. Was passt? Ordnen Sie zu.
 Kosten für ein Girokonto vergleichen 1. Was passt? Ordnen Sie zu. a. die Buchung, -en b. die Auszahlung, -en c. der Dauerauftrag, - e d. die Überweisung, -en e. die Filiale, -n f. der Kontoauszug, - e
Kosten für ein Girokonto vergleichen 1. Was passt? Ordnen Sie zu. a. die Buchung, -en b. die Auszahlung, -en c. der Dauerauftrag, - e d. die Überweisung, -en e. die Filiale, -n f. der Kontoauszug, - e
Dieses erste Kreisdiagramm, bezieht sich auf das gesamte Testergebnis der kompletten 182 getesteten Personen. Ergebnis
 Datenanalyse Auswertung Der Kern unseres Projektes liegt ganz klar bei der Fragestellung, ob es möglich ist, Biere von und geschmacklich auseinander halten zu können. Anhand der folgenden Grafiken, sollte
Datenanalyse Auswertung Der Kern unseres Projektes liegt ganz klar bei der Fragestellung, ob es möglich ist, Biere von und geschmacklich auseinander halten zu können. Anhand der folgenden Grafiken, sollte
Kurzanleitung. MEYTON Aufbau einer Internetverbindung. 1 Von 11
 Kurzanleitung MEYTON Aufbau einer Internetverbindung 1 Von 11 Inhaltsverzeichnis Installation eines Internetzugangs...3 Ist mein Router bereits im MEYTON Netzwerk?...3 Start des YAST Programms...4 Auswahl
Kurzanleitung MEYTON Aufbau einer Internetverbindung 1 Von 11 Inhaltsverzeichnis Installation eines Internetzugangs...3 Ist mein Router bereits im MEYTON Netzwerk?...3 Start des YAST Programms...4 Auswahl
3. LINEARE GLEICHUNGSSYSTEME
 176 3. LINEARE GLEICHUNGSSYSTEME 90 Vitamin-C-Gehalt verschiedener Säfte 18,0 mg 35,0 mg 12,5 mg 1. a) 100 ml + 50 ml + 50 ml = 41,75 mg 100 ml 100 ml 100 ml b) : Menge an Kirschsaft in ml y: Menge an
176 3. LINEARE GLEICHUNGSSYSTEME 90 Vitamin-C-Gehalt verschiedener Säfte 18,0 mg 35,0 mg 12,5 mg 1. a) 100 ml + 50 ml + 50 ml = 41,75 mg 100 ml 100 ml 100 ml b) : Menge an Kirschsaft in ml y: Menge an
Erfahrungen mit Hartz IV- Empfängern
 Erfahrungen mit Hartz IV- Empfängern Ausgewählte Ergebnisse einer Befragung von Unternehmen aus den Branchen Gastronomie, Pflege und Handwerk Pressegespräch der Bundesagentur für Arbeit am 12. November
Erfahrungen mit Hartz IV- Empfängern Ausgewählte Ergebnisse einer Befragung von Unternehmen aus den Branchen Gastronomie, Pflege und Handwerk Pressegespräch der Bundesagentur für Arbeit am 12. November
Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr
 Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr Name: Matrikel-Nr.: Code Nummer: Bitte geben Sie Ihre Matrikel-Nr. und Ihren Namen an. Die Code-Nummer erhalten Sie zu Beginn
Klausur zum Modul Molekularbiologie ILS, SS 2010 Freitag 6. August 10:00 Uhr Name: Matrikel-Nr.: Code Nummer: Bitte geben Sie Ihre Matrikel-Nr. und Ihren Namen an. Die Code-Nummer erhalten Sie zu Beginn
Plotten von Linien ( nach Jack Bresenham, 1962 )
 Plotten von Linien ( nach Jack Bresenham, 1962 ) Ac Eine auf dem Bildschirm darzustellende Linie sieht treppenförmig aus, weil der Computer Linien aus einzelnen (meist quadratischen) Bildpunkten, Pixels
Plotten von Linien ( nach Jack Bresenham, 1962 ) Ac Eine auf dem Bildschirm darzustellende Linie sieht treppenförmig aus, weil der Computer Linien aus einzelnen (meist quadratischen) Bildpunkten, Pixels
Wichtige Hinweise zu den neuen Orientierungshilfen der Architekten-/Objektplanerverträge
 Wichtige Hinweise zu den neuen Orientierungshilfen der Architekten-/Objektplanerverträge Ab der Version forma 5.5 handelt es sich bei den Orientierungshilfen der Architekten-/Objektplanerverträge nicht
Wichtige Hinweise zu den neuen Orientierungshilfen der Architekten-/Objektplanerverträge Ab der Version forma 5.5 handelt es sich bei den Orientierungshilfen der Architekten-/Objektplanerverträge nicht
Online-Bestellung Tageskarten für Mitglieder des FC St. Pauli, die nicht im Besitz einer Dauer- oder Saisonkarte sind.
 Online-Bestellung Tageskarten für Mitglieder des FC St. Pauli, die nicht im Besitz einer Dauer- oder Saisonkarte sind. 1. Anmeldung Soweit noch nicht geschehen, muss im Vorfeld (vor Verkaufsstart am 21.07.)
Online-Bestellung Tageskarten für Mitglieder des FC St. Pauli, die nicht im Besitz einer Dauer- oder Saisonkarte sind. 1. Anmeldung Soweit noch nicht geschehen, muss im Vorfeld (vor Verkaufsstart am 21.07.)
