Optimierung des Energiemetabolismus von Synechocystis PCC 6803 für eine zukünftige Photosynthese-basierte Biowasserstoffproduktion
|
|
|
- Viktoria Lange
- vor 5 Jahren
- Abrufe
Transkript
1 Dissertation zur Erlangung des Grades eines Doktor Rerum Naturalium (Dr. rer. nat.) an der Fakultät für Biologie und Biotechnologie Optimierung des Energiemetabolismus von Synechocystis PCC 6803 für eine zukünftige Photosynthese-basierte Biowasserstoffproduktion Optimization of the energy-metabolism of Synechocystis PCC 6803 for a future photosynthesis-based hydrogen production Internationale Graduiertenschule Biowissenschaften Ruhr-Universität-Bochum Lehrstuhl für Biochemie der Pflanzen Vorgelegt von Martin Broekmans aus Kleve Bochum Oktober 2012
2 Referent: Prof. Matthias Rögner Korreferent: Prof. Eckhard Hofmann
3 Teile dieser Arbeit wurden veröffentlicht in: Imashimizu, M; Bernát, G; Sunamura, E; Broekmans, M; Konno, H; Isato, K; Rögner, M; Hisabori, T; Regulation of F0F1-ATPase from Synechocystis sp. PCC 6803 by gamma and epsilon subunits is significant for light/dark adaptation J. Biol. Chem 2011 Konferenzbeiträge in Form von Posterpräsentationen: Broekmans, M; Hagenbucher, J; Rögner, M; Metabolic engineering of Synechocystis PCC 6803 for a photosynthesis based hydrogen production, interne SolarH2-Tagung, Berlin Oktober 2011 Broekmans, M; Hagenbucher,J; Kwon, J-H; Rögner, M; Design of Synechocystis sp. PCC 6803: mutants for a photosynthesis based hydrogen production, Photosynthese-Workshop- Nordwest, Frankfurt Kwon, J.-H; Broekmans, M; Waschewski, N; Bernát, G; Rexroth; S., Rögner, M; Construction of a continuous photobiological reactor system for cyanobacterial H 2 -production, VAAM 2009
4 Inhaltsverzeichnis I Inhaltsverzeichnis I II III Inhaltsverzeichnis... III Zusammenfassung... VIII Summary... IX 1 Einleitung Design eines Modellorganismus für die photobiologische H 2 -Produktion Synechocystis PCC 6803 als Modellorganismus Der Energiemetabolismus von Synechocystis PCC Der respiratorische Elektronentransport Der lineare photosynthetische Elektronentransport Der zyklische Elektronentransport Optimierung des Energiemetabolismus von Synechocystis Phycobilisomen Die Lichtsammelkomplexe der Cyanobakterien Kurzfristige Anpassung an wechselnde Lichtbedingungen Die state transitions Das nicht-photochemische quenching der Cyanobakterien Effekt der Inaktivierung von Proteinen der PS1-Assemblierung Die ATP-Synthase Channelrhodopsin Lichtregulierte Promotoren Ziele der Arbeit Material und Methoden Mikrobiologische Methoden Stämme und Kultivierung von Escherichia coli Kryokulturen III
5 Inhaltsverzeichnis Verwendete Cyanobakterien und ihre Kultivierung Molekularbiologische Methoden Oligonukleotide und Plasmide Agarose-Gelelektrophorese Extraktion von DNA aus Agarosegelen Extraktion von genomischer DNA aus Synechocystis Extraktion von Plasmid-DNA aus E. coli Photometrische Bestimmung von Reinheit und Konzentration der DNA Enzymatische Modifikation von DNA Restriktion Ligation Dephosphorylierung Polymerase Ketten Reaktion (PCR) Transformation von DNA in E. coli Transformation von DNA in Synechocystis Proteinbiochemische Methoden Zellaufschluss und Membrantrennung TCA-Fällung von Proteinen SDS-Gelelektrophorese nach Laemmli Western Blot Bestimmung der Zellparameter und biophysikalische Methoden Optische Dichte und Absorptionsspektren von Zellen Chlorophyllbestimmung nach Porra et al [101] Bestimmung der Zellzahl Tieftemperatur-Fluoreszenzemissionsmessungen bei 77 K Acridin Gelb Fluoreszenzmessungen nach Teuber et al [102] IV
6 Inhaltsverzeichnis Messung der photosynthetischen Sauerstoffentwicklung und der respiratorischen Sauerstoffaufnahme Messung der Chlorophyll a-fluoreszenz Messung von Photoinhibition und Regeneration Messung der photosynthetischen Aktivität zur Bestimmung von Elektronentransportraten P700 Absorptionsänderungsmessung Bestimmung der PS1-Elektronentransportraten nach Klughammer et al. [104] Berechnung der Elektronentransportraten bei verschiedenen Lichtintensitäten nach Eilers und Peeters [105] Messung des photosynthetischen Gasaustausches mittels Gas- Massenspektrometrie (MIMS) Kultivierung von Synechocystis im Flachbettbioreaktor Sterilisation Messungen und Regulierung des ph-wertes Messung der Trübung und des Zellwachstums Beleuchtung, Begasung und Durchmischung Inokulation Probenentnahme und Messung der Produktivität Massenspektrometrische Analyse Ergebnisse Heterologe Expression von Channelrhodopsin-2 in Synechocystis PCC Untersuchung lichtregulierter Promotoren Lichtregulierung von PpsbA3 aus T. elongatus in Synechocystis Lichtregulierung von PhilC Zusammenfassung Entkopplung des photosynthetischen Elektronentransportes und die Erhöhung des PS2/PS1 Verhältnisses in Synechocystis V
7 Inhaltsverzeichnis Erzeugung der verwendeten Synechocystis-Mutanten Veränderung des Pigmentverhältnisses Das PS2/PS1 Verhältnis Aufbau des ph-gradienten an der Thylakoidmembran Zusammenfassung Anpassung an wechselnde Lichtbedingungen Wachstumsverhalten bei verschiedenen Lichtquantitäten Veränderung der Hochlichttoleranz Kurzfristige Adaption state transitions Zusammenfassung Der Elektronentransport in der Thylakoidmembran O 2 -Entwicklung und Respiration Der Elektronentransport am Photosystem Die PS1-Donorseite und der zyklische Elektronentransport Die PS1-Akzeptorseite Aufnahme von Lichtsättigungskurve an Hand der O 2 -Entwicklung Zusammenfassung Der Effekt unterschiedlicher Lichtabsorptionen auf die Photosyntheseleistung Auswirkungen auf die Phycobilisomen Die Auswirkung auf den Pigmentstatus der Zelle Die Effekte auf den photosynthetischen Elektronentransport Aufzeichnung von P-I-Kurven mittels der Nettosauerstoffproduktion Zusammenfassung Die Produktionsraten der photosynthetischen Wasserspaltung Die Raten des Elektronentransports am PS Vergleich der ETR aus O 2 - und Fluoreszenz-Messungen VI
8 Inhaltsverzeichnis Der Elektronentransport mit Methyl Viologen Zusammenfassung Langzeitadaption Kultivierung im Flachbett-Bioreaktor Die ideale Lichtintensität Die ideale Zelldichte Diskussion Die Auswirkung verkleinerter Phycobilisomen auf das PS2/PS1 Verhältnis Die Funktion von Ycf4 bei der Assemblierung von PS Die Entkopplung des Elektronentransportes und die Auswirkung auf die Photoinhibition Die state transitions und der zyklischer ET Erhöhung des Elektronentransportes Veränderte Anforderungen der Kultivierung im Bioreaktor Der Design-Organismus für eine photobiologische H 2 -Produktion Ausblick Abkürzungsverzeichnis Literaturverzeichnis Danksagung Erklärung VII
9 Zusammenfassung II Zusammenfassung Die Optimierung bestehender, natürlicher Systeme bildet die Grundlage für eine biotechnologische Nutzung der photosynthesebasierten H2-Produktion. Dazu soll durch genetic engineering aus dem Cyanobakterium Synechocystis PCC 6803 ein Modellorganismus konstruiert werden, in dem eine O2-tolerante Hydrogenase an einen gesteigerten photosynthetischen Elektronentransport gekoppelt ist. In dieser Arbeit konnte gezeigt werden, dass die Inaktivierung des C-Terminus der Epsilon- Untereinheit der ATP-Synthase ( CԐ) die partielle Entkopplung des Elektronentransportes (ET) von der ATP-Synthese zur Folge hat und mit einer erhöhten photosynthetischen O 2 - Entwicklung einhergeht. Anhand von EPR-Messungen konnte für den WT das PS2/PS1 Verhältnis von 1:10 bestimmt werden. Eine Verkleinerung der Phycobilisomen (PBS) durch den Verlust von Phycocyanin (PC) erhöht dieses in der Olive auf 1:3 und verdreifacht die PS2 Menge. Dadurch wird eine um 80 % verringerte Lichtausnutzung am PS2 ausgeglichen. Eine weitere Verringerung der Lichtausnutzung auf bis zu 90 % durch einen vollständigen Verlust der PBS in der PAL resultiert dagegen in einem Abbau von PS1 und einer Verringerung des PS2/PS1 Verhältnisses auf 1:4. Die Mutante DM stellt die erfolgreiche Kombination aus den Mutanten CԐ und Olive dar. Um eine weitere Erhöhung des PS2/PS1 Verhältnisses zu erhalten, wurde aus der DM Mutante durch zusätzliche Inaktivierung des PS1- Assemblierungsproteins Ycf4, die Mutante TM erstellt. Die Verkleinerung der PBS in Olive und PAL steigert die Hochlichtresistenz deutlich, was in der DM auf Grund geringerer Radikalbildung durch die teilweise Entkopplung des ET noch verstärkt wird. Im Gegensatz dazu resultiert die Inaktivierung von Ycf4 im Verlust der Hochlichtresistenz. In dieser Arbeit wurden erstmals mit Hilfe der Chlorophyll-Fluoreszenz exakte Elektronentransportraten (ETR) für den WT und Mutanten mit veränderten Pigmentverhältnissen berechnet. Diese wurden durch den, in Gasmassenspektrometriemessungen mit 18 O 2 -Isotopen bestimmten, tatsächlichen Gasaustauch bestätigt. Dabei konnte gezeigt werden, dass sowohl der Verlust von PC als auch die partielle Entkopplung des ET die ETR deutlich steigern. Durch die Kombination beider Mutationen werden die ETR in der DM im Vergleich zum WT vervierfacht. Dieser Überschuss an Elektronen kann vom Metabolismus nicht genutzt werden, so dass bis zu 75 % in der Mehler-Reaktion erneut auf O 2 übertragen werden. Eine Nutzung der überschüssigen Elektronen könnte für eine H 2 -Produktion genutzt werden ohne den Metabolismus zu beeinträchtigen. Durch Messungen im Flachbettbioreaktor konnte die hohe Photosyntheseleistung der DM über einen längeren Zeitraum bestätigt werden. Die DM stellt somit den besten Kandidaten für das Design eines Modellorganismus zur photobiologischen H 2 -Produktion dar. VIII
10 Summary III Summary Optimization of existing, natural systems could be the basis for a future photosynthesis based H 2 -production. To achieve this, the photosynthetic electron transport rates in Synechocystis PCC 6803 should be increased by genetic engineering. In a model organism for a future photosynthesis based H 2 -production, hydrogen should be produced in biotechnology amounts by coupling a O 2 -tolerant hydrogenase to high electron transport rates in Synechocystis In this work it could be shown that loss of the c-terminus of ATP-synthase epsilon subunit leads to a partially uncoupling of the electron transport (ET) from rate limiting ATP synthesis. As a result photosynthetic O 2 -evolution is increased. In EPR measurements it was possible to determine the PS2/PS1 ration of 1:10 for the Synechocystis WT. Decreasing the size of the PBS by deleting plastocyanin (PC) leads to a threefold increase of the PS2 content and an increase of the PS2/PS1 ratio to 1:3 in the Olive, which compensate for an 80 % decrease of the antenna cross section. A further decrease of the antenna cross section up to 90 %, due to loss of the total PBS (PAL), rather decrease the PS1 concentration than increasing the PS2 concentration. As result the PS2/PS1 ratio decreased compared to Olive (1:4). A successful combination of CԐ and PC mutations could be achieved in the DM mutant. To achieve a further increase of the PS2/PS1 ratio the mutant TM was constructed by additional deletion of the PS1 assembly protein Ycf4 in the DM. Mutation of the PBS leads to a significant increase of high light resistance, which could be further increased by a reduced production of reactive oxygen species in the DM. In contrast to this deletion of Ycf4 causes a loss of high light resistance. For the first time it was possible to determine exact electron transport rates (ETR) from chlorophyll-fluorescence measurements for WT and mutants with altered pigmentation. Obtained results could be confirmed by measurements of the photosynthetic gas exchange due gas mass spectrometry with oxygen isotopes ( 18 O 2 ). With both methods it could be shown that deletion of PC as well as partially uncoupling of the ET leads to increased ETR. The combination of both mutations in the DM leads to fourfold increase of the ETR compared to the WT. Most of the electrons produced in the DM cannot be used in the metabolism, so that up to 75 % are transferred to O 2 in the Mehler reaction. If these electrons can be redirected to a Hydrogenase, a future H 2 -production would not affect the metabolism. Since high photosynthetic electron transport rates in the DM could be confirmed by long-term measurements of the oxygen evolution in a bioreactor, the DM is the best candidate for the construction of a model organism for a future H 2 production.. IX
11 Einleitung 1 Einleitung In der Photosynthese wird die Energie der Sonne in chemische Energie und organische Moleküle umgewandelt. Dies macht die Photosynthese zur Grundlage für den Großteil des Lebens und damit zu einem der wichtigsten Prozesse auf der Erde. Den Menschen dient die in der Photosynthese produzierte Biomasse nicht nur als Nahrung, sondern sie bildet auch schon immer die Grundlage der Energiegewinnung. Seit der Entdeckung fossiler Brennstoffe als Energiequellen, und der damit verbunden Industrialisierung, haben die Energieansprüche ein Maß erreicht, das durch regenerative Energiequellen nicht gedeckt werden kann. Steigende Bevölkerungszahlen und eine wachsende Wirtschaft in den Entwicklungsländern werden den heutigen Energieverbrauch von etwa 14 TW pro Jahr auf bis zu 30 TW im Jahr 2030 ansteigen lassen [1]. Diese Entwicklung, ausgelöst durch die kostengünstige Energiegewinnung aus fossilen Brennstoffen, ist mit einem Anstieg des jährlichen CO 2 -Ausstoßes auf bis zu 43 Milliarden Tonnen im Jahr 2035 verbunden [1]. Trotz anhaltender Diskussionen gilt es mittlerweile als gesichert, dass der CO 2 -Ausstoß aus der Verbrennung fossiler Rohstoffe zu einem anthropogenen Klimawandel führt. Im Hinblick auf die Energieprognosen ist es daher unerlässlich die bestehenden, CO 2 - neutralen Methoden der Energiegewinnung auszubauen und neue zu entwickeln. Da das Endprodukt seiner Verbrennung CO 2 -neutrales Wasser ist, gilt Molekularer Wasserstoff (H 2 ) als einer der vielversprechendsten Energieträger der Zukunft. Zur Etablierung als allgemein akzeptierten Energieträger müssen jedoch zunächst kostengünstige und klimafreundliche Produktionsverfahren entwickelt werden. Neben vielen rein technischen Ansätzen stellt die Produktion von H 2 in natürlichen und von der Natur inspirierten Systemen eine vielversprechende Alternative dar. Da die jährliche Speicherung von ca. 100 TW Sonnenenergie in Form von Biomasse durch die Photosynthese ein erfolgreiches Beispiel für die Umwandlung von Sonnenenergie in einen Treibstoff ist [2,3], gewinnt die photobiologische H 2 -Produktion zunehmend an Aufmerksamkeit. Diese ist von Grünalgen und Cyanobakterien inspiriert, in denen eine natürliche Kopplung von Photosynthese und H 2 - Produktion über das Enzym Hydrogenase zu beobachten ist [3,4]. 1
12 Einleitung 1.2 Design eines Modellorganismus für die photobiologische H 2 -Produktion Zur Etablierung der photobiologischen H 2 -Produktion muss ein System entwickelt werden, mit dem es möglich ist, H 2 kostengünstig, umweltfreundlich und in großen Mengen zu produzieren. Diese Voraussetzungen sind in den bestehenden, natürlichen Systemen nicht gegeben. Eine vielversprechende Möglichkeit ist das Design eines Modellorganismus für die photobiologische H 2 -Produktion. Dafür müssen, in Abhängigkeit des Metabolismus eines zukünftigen Produktionsstammes, mögliche produktionslimitierende Prozesse evaluiert und optimiert werden. Bei dem in Abbildung 1-1 gezeigten Modellorganismus für eine photobiologische H 2 -Produktion soll die Erzeugung von H 2 durch das Enzym Hydrogenase möglichst direkt an den linearen Elektronentransport der Photosynthese gekoppelt werden. Um einen ökonomisch konkurrenzfähigen Produktionsstamm zu entwickeln, müssen dabei drei Grundvoraussetzungen realisiert werden: Abbildung 1-1: Schematische Darstellung einer Design-Zelle für die photobiologische H 2- Produktion. Photosystem (PS), Hydrogenase (H 2ase). Quelle: Bildarchiv des LS. Der Energiemetabolismus muss so optimiert werden, dass zu jedem Zeitpunkt die größtmögliche Menge von Elektronen aus der Wasserspaltung zur Verfügung gestellt wird. Es muss eine sauerstofftolerante Hydrogenase mit möglichst hoher Aktivität entwickelt werden. Die Reaktion der Hydrogenase muss effizient an den Elektronentransport gekoppelt werden. Hydrogenasen sind Metalloproteine, welche H 2 in Protonen und Elektronen spalten oder umgekehrt diese zu H 2 reduzieren. Nach dem Aufbau ihres aktiven Zentrums können sie in drei Klassen unterteilt werden. Dies sind die Eisen- (Fe), die Nickel-Eisen- (NiFe) und die Eisen-Eisen- (FeFe) Hydrogenasen [5]. Unter diesen besitzen die FeFe-Hydrogenasen aus Grünalgen die höchsten bekannten Aktivitäten von 10 4 s -1 [6,7]. CO- und CN-Liganden im aktiven Zentrum machen Hydrogenasen sensitiv gegen O 2 und H 2. Die Bindung von O 2 führt dabei zu einer reversiblen (NiFe) oder irreversiblen (FeFe) Hemmung [8]. Die Inhibition der H 2 -Produktion durch O 2 erschwert die Kopplung an die oxygene Photosynthese. Ein dadurch 2
13 Einleitung erforderliches genetic engineering zur Erhöhung der O 2 -Toleranz der Hydrogenasen wird besonders in FeFe-Hydrogenasen durch den nicht vollständig verstandenen Mechanismus der Inhibition erschwert. Möglicherweise kann hier die Veränderung des Redoxpotentials eines 4Fe4S-Cluster in Kombination mit der Unterbindung einer möglichen Diffusion von reactive oxygen species (ROS) erfolgversprechend sein [9]. Einen Schritt weiter ist man bei den NiFe-Hydrogenasen. Einige Organismen, wie das Bakterium Ralstonia eutropha, besitzen O 2 -tolerante Hydrogenasen, die Einblicke in den Mechanismus der O 2 -Toleranz erlauben und die Basis für ein genetic engineering von NiFe-Hydrogenasen mit höherer Aktivität bilden [10,11]. Art und Position der Kopplung von Photosynthese und Hydrogenase wird von der gewählten oder konstruierten Hydrogenase bestimmt. Generell gilt, je weniger Reaktionsschritte zwischen Wasserspaltung und H 2 -Produktion liegen, desto verlustfreier ist die Energieübertragung. Eine direkte Kopplung an das PS2 würde also den Idealfall darstellen, ist aber durch ein zu negatives Redoxpotential der bekannten Hydrogenase nicht möglich. Eine Kopplung direkt am PS1 oder über Ferredoxin ist insbesondere bei Hydrogenasen des FeFe-Typs, mit Ferredoxin als natürlichem Elektronendonor, realisierbar. Die Kopplung der NiFe-Hydrogenasen über ihren natürlichen Elektronendonor NADPH würde einen zusätzlichen Reaktionsschritt beinhalten. Denkbar ist hier eine direkte Kopplung an Untereinheiten (UE) des PS1. Die Möglichkeit einer solchen Fusion wurde bereits in vitro für die PsaE-UE gezeigt, jedoch fehlt noch der Nachweis für die Durchführbarkeit in vivo [12]. Zur Optimierung des Energiemetabolismus müssen zunächst ein geeigneter Modellorganismus gefunden und die produktionslimitierenden Prozesse in seinem Metabolismus evaluiert werden. In dieser Arbeit soll, als erster Schritt zu einem Modellorganismus für die H 2 -Produktion, der Energiemetabolismus des Cyanobakteriums Synechocystis PCC 6803 optimiert werden. 1.3 Synechocystis PCC 6803 als Modellorganismus Cyanobakterien waren die ersten Organismen, die in der Lage waren oxygene Photosynthese zu betreiben und erfüllen damit eine grundsätzliche Bedingung für die photobiologische H 2 -Produktion. Sie bilden eine Gruppe von Mikroorganismen, die sich durch eine hohe Diversität auszeichnen, welche sich ebenfalls in ihren Lebensräumen widerspiegelt. Zu diesen zählen nicht nur aquatische Systeme, sondern auch Wüsten, genauso wie arktische Gebiete und Thermalquellen. Diese hohe Diversität hat sich auch auf 3
14 Einleitung den Metabolismus der Bakterien übertragen, so dass viele Vertreter der Cyanobakterien zunehmend als Kandidaten für die biotechnologische Industrie interessant werden [13]. Das in Abbildung 1-2 gezeigte, einzellige, mesophile Cyanobakterium Synechocystis sp. PCC Abbildung 1-2: Elektronenmikroskopaufnahme von Synechocystis PCC Quelle: Bildarchiv des LS (Synechocystis) wurde 1968 aus einem Süßwassersee isoliert und in die Pasteur Culture Collection (PCC) aufgenommen. Auf Grund der Ähnlichkeit seines Photosyntheseapparates zu dem Höherer Pflanzen gehört Synechocystis zu den am meisten untersuchten Cyanobakterien [14] und besitzt entscheidende Eigenschaften, die es zum idealen Kandidaten für das sogenannte metabolic engineering machen. Da es zu den natürlich kompetenten Organismen gehört, kann es Fremd-DNA aufnehmen und über heterologe Rekombination in sein Genom einbauen. Die Fähigkeit zum heterotrophen Wachstum ermöglicht die Mutation und die Inaktivierung von Komplexen der Photosynthese und trägt damit entscheidend zum Erkenntnisgewinn dieser Prozesse bei. Synechocystis ist das erste Cyanobakterium, dessen komplettes Genom sequenziert wurde [15]. Zudem steht eine zunehmende Anzahl an Mutanten-Stämmen zur Verfügung. Die Kombination dieser Eigenschaften machen Synechocystis zum optimalen Modellorganismus für die Optimierung des Energiemetabolismus. 1.4 Der Energiemetabolismus von Synechocystis PCC 6803 Cyanobakterien müssen sich in ihrem natürlichen Lebensraum an permanent wechselnde Umweltbedingungen anpassen. Dies gelingt ihnen durch eine einzigartige Flexibilität ihres Energiemetabolismus, die durch eine effektive Kombination verschiedener metabolischer Reaktionswege bedingt ist. Abbildung 1-3 gibt einen schematischen Überblick über einen Teil der metabolischen Reaktionen in Synechocystis. Wie für Prokaryoten üblich, besitzen die Zellen keine Organellen, verfügen jedoch neben der Zellmembran (CM) über ein zusätzliches, internes Membransystem, die Thylakoidmembran (TYM). In der CM ist eine unvollständige, respiratorische Elektronentransportkette lokalisiert, die sich aus der NAD(P)H- Dehydorgenase-2 (NDH-2), der Succinat-Dehydrogenase (SDH), dem Plastochinon-Pool (PQ-Pool) und den beiden Chinon-Oxidasen Cytochrom-bd-Oxidase (Cyt-bd) und alternative 4
15 Einleitung respiratory terminal oxidase (ARTO) zusammen setzt [16]. Eine aktive Protonentranslokation findet dabei nur in der ARTO statt. Dies deutet auf ein geringes Membranpotential und so auf einen geringen Anteil der CM an der Gesamtrespiration hin. Neben respiratorischen Komplexen finden sich in der CM auch inaktive Komplexe von PS1 und PS2 [17]. Abbildung 1-3: Schematische Übersicht der Elektronentransportwege in Thylakoid- und Zytoplasma-Membran. Eine detaillierte Erläuterung der Abbildung findet sich im Text. Quelle: Abgewandelt aus dem Bildarchiv des LS. In der TYM findet sich sowohl eine respiratorische als auch eine photosynthetische Elektronentransportkette (PET). Die Komponenten der Photosynthese sind das PS2 und das PS1, die der Respiration die NADPH-Dehydrogenase (NDH-1), die Succinate- Dehydrogenase (SDH) und als terminale Oxidase vornehmlich die Cytochrom-aa3-Oxidase (COX). Zusätzlich kommt es, neben der fehlenden räumlichen Trennung, durch die gemeinsame Nutzung des PQ-Pools, des Cytochrom b 6 f-komplexes und des löslichen Elektronenträgers Plastocyanin (Pc), welcher bei Kupfermangel durch Cytochrom c 6 ersetzt wird [18], zur Interaktion beider Elektronentransportketten. Sowohl Respiration als auch 5
16 Einleitung Photosynthese sind mit einer Protonentranslokation in das Thylakoidlumen verbunden. Der entstehende Protonengradient wird von der ATP-Synthase zur Produktion von ATP genutzt. Die Interaktion von Respiration und Photosynthese verleiht Cyanobakterien ein hohes Maß an Flexibilität, muss aber einer strikten Kontrolle unterliegen, um einen Verlust von Elektronen zu vermeiden. Vom PS1 gelangen die Elektronen über Ferredoxin zur FNR, wo sie auf NADP + übertragen werden. Das resultierende NADPH wird im Calvin-Zyklus, zusammen mit ATP, zur CO 2 - Fixierung genutzt. Neben diesem linearen Elektronentransport (ET) der Photosynthese können Elektronen sowohl vom Ferredoxin als auch vom NADPH in verschiedene alternative Elektronentransportwege gespeist werden. Beim Ferredoxin sind dies unter anderem die Stickstoff- und Schwefel-Assimilation (N-, S-Ass.). Elektronen vom NADPH können über Flavodiironproteine (Flv) auf Sauerstoff übertragen werden. Zudem ist NADPH auch am zyklischen ET der Photosynthese beteiligt. In diesem werden Elektronen um das PS1 transportiert und der so gebildete Protonengradient zur ATP-Synthese ohne netto NADPH- Produktion genutzt Der respiratorische Elektronentransport Ein unvollständiger respiratorischer ET in der CM, die vornehmlich von Komponenten ohne aktive Protonentranslokation gebildet wird [16,19-21], deutet darauf hin, dass nur ein geringes Membranpotential gebildet wird. Da die CM zudem nur einen geringen prozentualen Anteil an der Gesamtmembran hat, ist die Respiration hauptsächlich in der TYM lokalisiert. In dieser sind die PQ-reduzierenden Komponenten der Respiration der NDH-1-Komplex und die SDH. Es wird angenommen, dass Elektronen hauptsächlich über die Oxidation von NADPH am NDH-1-Komplex in die Elektronentransportkette gespeist werden [22 24]. Dies wurde aber durch Vermaas et al. in Frage gestellt, welche die SDH als eigentlichen Elektronendonor postulieren [25], so dass der genaue Eintritt von Elektronen in die Respiration einer abschließenden Klärung bedarf. Reduziertes PQ wird am Cyt-b 6 f-komplex oxidiert. Von hier werden die Elektronen über Pc oder Cytochrom c 6, über die terminale Oxidase COX, unter der Bildung von H 2 O auf O 2 übertragen. Der Nachweis weiterer, alternativer Oxidasen lässt vermuten, dass der respiratorische ET eher verzweigt als linear ist. Neben der COX findet sich auch die Cytochrom-bd-Oxidase in der TYM, welche ihre Elektronen direkt von PQ erhält [26]. Zusätzlich wurde die ARTO in der TYM nachgewiesen, jedoch konnte noch nicht abschließend geklärt werden, ob sie Elektronen aus dem PQ-Pool oder von Pc/cyt c 6 erhält [21]. 6
17 Einleitung Zur Vermeidung einer metabolisch sinnlosen Reaktion bei parallelen photosynthetischen und respiratorischen ET muss die Respiration einer strikten Regulation unterliegen. Zum einen kommt es zu einer Inhibition des oxidativen Pentosephosphatwegs (PPW), in dem ATP und NADH aus dem Abbau von Glukose generiert wird. Im Licht kommt es durch Ribulose-1,5- bisphospaht, einem Zwischenprodukt des Calvin-Zyklus, zu einer allosterischen Hemmung der Glukose-6-Phosphat Dehydrogenase, einem Enzym des PPW [27]. Zusätzlich wird eine direkte Konkurrenz von Photosynthese und Respiration um Elektronen durch die, im Vergleich zur COX, signifikant erhöhte Elektronenkapazität von PS1 limitiert [28] Der lineare photosynthetische Elektronentransport Am linearen PET in der TYM sind drei integrale Membranproteine beteiligt. Diese sind, wie in Abbildung 1-4 gezeigt, PS2, PS1 und der Cyt-b 6 f-komplex. Der PET beginnt mit der Abgabe von Anregungsenergie von den Phycobilisomen (PBS) über die Kernantennen, bestehend aus den Chlorophyllproteinen CP43 und CP47, an das Reaktionszentrum von PS2. Von dort wird sie an ein akzessorisches Chlorophyll a an der D1-Untereinheit (Chl aac D1) weitergegeben. Abbildung 1-4: Darstellung des linearen, photosynthetischen Elektronentransportes und der beteiligten Proteinkomplexe mit Kofaktoren in Synechocystis. Quelle: Bildarchiv des LS. 7
18 Einleitung Dieses stellt den primären Elektronendonor der Photosynthese dar. Nach Anregung kommt es zu einer Ladungstrennung zwischen (Chl aac D1), und einem Phaeophytin (Phe), dem primären Elektronenakzeptor, unter Bildung des Radikalpaars Chl + aad1 /Phe - [29]. Phe - reduziert ein fest gebundenes Chinon (Q A ), welches wiederum ein lose gebundenes Chinon (Q B ) reduziert. Chinone sind Zweielektronenüberträger, so dass Q - B gebunden bleibt, bis es nach einer zweiten Ladungstrennung und Aufnahme von zwei Protonen aus dem Zytoplasma zum Plastohydrochinon (PQH 2 ) reduziert wird, dem ersten stabilen Produkt der Photosynthese. Die bei der Ladungstrennung entstehende Elektronenlücke (Chl + aacd1 ) wird durch Aufnahme eines Elektrons vom luminalen Mangancluster geschlossen. Der Mangancluster ist ein Redoxsystem, das, resultierend aus der Spaltung von zwei Molekülen H 2 O, vier Elektronen aufnehmen kann, um diese, über den in der Abbildung 1-4 gezeigten, Tyrosinrest (Y z ) an das Reaktionszentrum abzugeben. In diesem Prozess durchläuft der Mangancluster fünf verschiedene Redoxzustände (S 0 -S 4 ) [30,31]. Das am PS2 freigesetzte PQH 2 diffundiert durch die Thylakoidmembran zur Q 0 -Bindestelle des Cyt-b 6 f-komplexes, wo es unter Abgabe beider Protonen in das Thylakoidlumen zum Semichinon oxidiert wird. Wie in der Abbildung 1-5 gezeigt, wird ein Elektron von PQH2 in einen Reaktionsweg mit hohem Potential abgegeben. Dieser wird durch das Rieske-Eisen-Schwefel-Protein und Cytochrom f gebildet. Von Cytochrom f wird das Elektron an den löslichen Elektronenüberträger Pc im Thylakoidlumen abgegeben [32,33]. Das zweite Elektron von PQH 2 wird in einen Reaktionsweg mit niedrigem Potential, den so genannten Q-Zyklus, abgegeben. In diesem gelangt es von einem Häm b (b L ), mit niedrigem Potential, zu einem zweiten Häm b (b H ) mit hohem Potential. Dieses b H ist elektronisch mit einem Häm c (c i ) gekoppelt, welches möglicherweise die Q i -Bindestelle des Cytb 6 f-komplexes darstellt. Zusammen könnten beide Häme einen Zwei- Elektronen-Übertragungsweg bilden. Über Abbildung 1-5: Elektronentransportwege im Cytochromb 6f-Komplex inklusive Q-Zyklus. Quelle: Archiv LS Biochemie der Pflanzen 8
19 Einleitung diesen würde dann nach Abgabe eines zweiten Elektrons in den Q-Zyklus ein PQ an der Q i - Bindestelle, durch zusätzliche Aufnahme von zwei Protonen aus dem Zytoplasma, zum PQH 2 reduziert werden [32,33]. Der Q-Zyklus erhöht somit die Anzahl der, in das Thylakoidlumen transportierten, Protonen. Wie in Abbildung 1-4 zu sehen, diffundiert das Pc durch das Thylakoidlumen zum PS1 und schließt dort die, durch Lichtanregung entstandene, Elektronenlücke. Am PS1 hat die Lichtanregung zu einer Ladungstrennung zwischen einem Chlorophyll des Reaktionszentrum (A acc ) und einem Chlorophyllmonomer (A 0 ) geführt [34]. Vom entstandenen Radikalpaar A + acc /A - 0 wird das Elektron über Phyllochinon (A 1 ) und die Eisen-Schwefel-Cluster (F X, F A, F B ) auf Ferredoxin übertragen. Die Elektronen des reduzierten Ferredoxins werden von der FNR zur Reduzierung von NADP + zu NADPH genutzt. Die Elektronenlücke am Radikalpaar wird durch Oxidation des sogenannten special pair (P700 P700 + ) und die anschließende Oxidation von Pc geschlossen [34]. Während des PET wird durch die Wasserspaltung am Mangancluster des PS2 und durch die Protonentranslokation am Cyt-b 6 f-komplex ein Protonengradient an der TYM aufgebaut. Dieser wird von der ATP-Synthase, in einem als Photophosphorylierung bezeichneten Prozess, zur Bildung von ATP aus ADP und P i genutzt. NADPH und ATP, die Endprodukte der linearen photosynthetischen Elektronentransportkette, werden im Calvin-Zyklus zur Fixierung von CO 2 genutzt Der zyklische Elektronentransport Der zyklische ET der Photosynthese führt durch Aufbau eines Protonengradienten zu einer ATP-Synthese ohne netto NADPH-Produktion. Hierbei werden Elektronen von der Akzeptorseite des PS1 über den PQ-Pool und/oder den Cyt-b 6 f-komplex zurück zum PS1 transportiert. In Cyanobakterien sind nach aktuellem Forschungsstand mindestens drei verschiedene zyklische Elektronentransportrouten (Abbildung 1-6), mit unterschiedlichen Raten, parallel aktiv. Nach Definition von Hans Matthijs können diese in direkte und indirekte Routen unterteilt werden [35,36]. 9
20 Einleitung Abbildung 1-6: Schematische Darstellung des zyklischen ET in Synechocystis mit indirektem Transport über NDH-1 und direktem Transport über FNR und FQR. Gestrichelte Pfeile deuten hypothetische Transportwege an. Quelle: Archiv LS Biochemie der Pflanzen. Die indirekten Routen sind dabei Teil des respiratorischen ET und bilden den Hauptweg des zyklischen ETs. Hier werden die Elektronen, über die Kofaktoren NADPH oder Succinat, durch die respiratorischen Oxidoreduktasen NDH-1 oder SDH in den PQ-Pool geschleust. Daneben existieren mindestens zwei direkte Elektronentransportwege mit geringerer Kapazität. Bei einem werden die Elektronen von Ferredoxin über die FNR [37], bei dem anderen über die FQR [22, 35] zurück zum PS1 transportiert. Für beide Elektronentransportwege ist noch nicht abschließend geklärt, ob der Wiedereintritt der Elektronen in die Elektronentransportkette über den PQ-Pool oder den Cyt-b 6 f-komplex stattfindet. 1.5 Optimierung des Energiemetabolismus von Synechocystis Die Raten des linearen photosynthetischen ET werden in Synechocystis von folgenden Faktoren negativ beeinflusst: Unausgeglichenes PS2/PS1 Verhältnis Geschwindigkeitslimitierende Reaktion der ATP-Synthase Hohe Raten des zyklischen ET In Cyanobakterien liegt, im Gegensatz zu Höheren Pflanzen, ein unausgeglichenes Verhältnis der beiden Photosysteme vor. Dabei ist die Menge an PS1 (85 ± 5 %) im Vergleich zum PS2 (15 ± 5 %) stark erhöht [38,39]. Der niedrige Anteil des PS2 führt zu einer starken Limitierung der Raten der photosynthetischen Wasserspaltung. Der hohe Anteil an PS1 bewirkt in Synechocystis teilweise hohe Raten des zyklischen ET. Diese reduzieren den PQ- 10
21 Einleitung Pool und limitieren so die Elektronenabnahmen am PS2. Eine Erhöhung des PS2/PS1 Verhältnisses ist daher ein essentieller Schritt zur Steigerung der Wasserspaltungsraten. Durch Belichtung der Zellen mit fernrotem Licht (> 720 nm) oder starkem Weißlicht ( nm) kommt es zum Schutz vor Photoinhibition zu einer Erhöhung des PS2/PS1- Verhältnisses [40]. Neben der lichtinduzierten Erhöhung des PS2/PS1-Verhältnisses konnte zudem gezeigt werden, dass die Inaktivierung von Teilen oder der kompletten PBS zu einer Erhöhung des PS2/PS1-Verhältnisses führt. Vermutlich versuchen die Zellen die verringerte Lichtabsorption durch eine Erhöhung des PS2-Gehalts auszugleichen [34,35]. Durch die verringerte Lichtabsorption kommt es des Weiteren zu einem geringeren Elektronenstau bei hohen Lichtintensitäten. Dies minimiert die Bildung von ROS und damit die Photoinhibition. Eine Entkopplung des ET von der Reaktion der ATP-Synthese soll diesen Effekt noch verstärken. Bei hohen Lichtintensitäten kommt es zu einer Akkumulation von Protonen im Thylakoidlumen. Dies macht die weitere Translokation von Protonen in das Thylakoidlumen thermodynamisch aufwendiger und führt so zu einer Verlangsamung des ET. Insbesondere bei hohen Lichtintensitäten wird der Abbau des Protonengradienten durch die ATP-Synthese zum geschwindigkeitsbestimmenden Schritt. Eine Entkopplung würde, auch bei hohen Lichtintensitäten, hohe Raten des ET zulassen und so zum einen die Raten der Wasserspaltung steigern und zum anderen die Bildung von ROS verringern. Die Raten des zyklischen ET sind von den äußeren Bedingungen beeinflusst und besonders in Stresssituation erhöht. Bei normalen Bedingungen konnte gezeigt werden, dass eine Erhöhung des PS2/PS1-Verhältnisses die Raten des zyklischen-et senkt. Gleiches gilt ebenso für rein photoautotrophes Wachstum [35]. 1.6 Phycobilisomen Die Lichtsammelkomplexe der Cyanobakterien Die PBS sind große Proteinkomplexe, die den meisten Cyanobakterien und Rotalgen als Lichtsammelkomplexe dienen [47]. Aufbau und Zusammensetzung der PBS können in den einzelnen Spezies stark variieren, jedoch bestehen sie immer aus heterodimeren Phycobiliproteinen (αβ), deren Polypeptiduntereinheiten offenkettige Tetrapyrole, so genannte Phycobiline, als chromophore Gruppe kovalent binden. Diese lagern sich zu scheibenförmigen Trimeren (αβ) 3 und Hexameren (αβ) 6 zusammen, welche in ihrem Zentrum durch chromphorlose Linkerpolypetide zusammengehalten werden [48]. In Synechocystis finden sich, wie in Abbildung 1-7 gezeigt, PBS des hemi-discoidalen Typs, welche aus einem dreizylindrigem Kern und sechs stäbchenartigen äußeren Strukturen bestehen [49]. Die 11
22 Einleitung äußeren Stäbchenstrukturen setzen sich aus jeweils drei hexameren Phycocyaninscheiben zusammen. Die Assoziation und Assemblierung wird dabei von den sogenannten rod-linker- Polypeptiden L 10 R, L 33 R und L 35 R unterstützt, so dass eine optimale Lichtweiterleitung gewährleistet ist. Zusätzlich finden sich noch die sogenannten rod-core-linker (L RC ), welche die äußeren Stäbchen mit dem Kern verbinden [50]. Phycocyanin (PC) sammelt den Hauptteil des Lichtes und leitet dies an den PBS-Kern weiter. Abbildung 1-7: Schematische Übersicht des Phycobilisomenaufbaus. APC: Allophycocyanin; PC: Plastocyanin; CpcD: LCM-Linker; FNR: Ferredoxin:NAPPH-Oxidoreduktase. Die Zylinder im Kern bestehen aus jeweils vier Scheiben von trimeren Allophycocyanin (APC) (αβ) 3. Diese sind so angeordnet, dass ihre Längsachsen parallel zueinander liegen, während ihre Ränder zusammen ein Dreieck bilden [44,46,47]. Die Hauptaufgabe des Kerns besteht in der Weiterleitung von Anregungsenergie aus den äußeren Antennen zu den Chlorophyllen der Photosysteme. Dies führt dazu, dass die Strukturen der drei Zylinder sich unterscheiden. Im oberen Zylinder bestehen zwei der Scheiben nur aus APC-Trimeren, während die anderen zusätzlichen den Linker L C gebunden haben. Die unteren beiden Zylinder sind direkt an der Interaktion mit der TYM und den Photosystemen beteiligt und zeigen daher einen veränderten Aufbau. Hier besteht eine Scheibe aus AP (αβ) 3, eine Scheibe trägt zusätzlich den LC-Linker. In einer weiteren Scheibe ist eine α-ue gegen die sogenannte Isoform AP-B ausgetauscht. In der vierten Scheibe ist ein αβ-heteromer durch die in den roten Bereich verschobene Isoform β-18 ersetzt. Zusätzlich besitzt sie an einer α-ue eine Domäne für den Linker L CM [47,48]. L CM ist der Hauptlinker des PBS-Kerns, ohne den es weder zu einer Assemblierung der APC- Trimere, noch zu einer Zusammenlagerung in Zylinder kommt [49,50]. Zudem wird angenommen, dass L CM eine entscheidende Rolle bei der Assoziation der PBS mit der Membran und bei der Interaktion mit den Photosystemen spielt. Die genaue Funktion ist allerdings nicht bekannt. Wie in Abbildung 1-7 gezeigt, sind pro PBS-Komplex ein bis zwei Moleküle FNR über eine L 10 R -ähnlichen Domäne (cpcd in Abb. 1-7) an PC gebunden. Die Bindung der FNR an die 12
23 Einleitung PBS hat vermutlich eine Funktion im linearen photosynthetischen ET. Die Positionierung nahe der Thylakoidmembran verringert Diffusionsdistanzen zwischen FNR und PS1 [51]. 1.7 Kurzfristige Anpassung an wechselnde Lichtbedingungen Da Cyanobakterien in ihren natürlichen Lebensräumen mit permanent wechselnden Lichtbedingungen konfrontiert sind, ist eine rasche Anpassung für sie von entscheidender Bedeutung. Dies beinhaltet sowohl die ideale Lichtausnutzung bei niedrigen Lichtbedingungen, als auch den Schutz vor überschüssiger Anregungsenergie. In beiden Fällen spielen die PBS als Lichtsammelkomplexe eine zentrale Rolle Die state transitions Bei niedrigen Lichtbedingungen muss die von den PBS absorbierte Anregungsenergie so auf die beiden Photosysteme verteilt werden, dass der PET für den Organismus optimal ausbalanciert ist. Bei diesen, state transitions genannten, kurzfristigen Anpassungen wird Anregungsenergie zwischen den beiden Photosystemen verteilt. Eine überschüssige Anregung von PS1 führt zur Weiterleitung von Anregungsenergie an das PS2 ( state 1 ) und umgekehrt ( state 2 ) [52,53]. Eine Beteiligung der Lichtsammelkomplexe an diesem Prozess konnte bereits vor über 40 Jahren in Pflanzen für die light harvesting complexes (LHC), als auch in Rotalgen für die PBS nachgewiesen werden. Trotz der langen Bekanntheit dieses Prozesses sind seine Grundlagen in Cyanobakterien immer noch nicht abschließend geklärt. Zahlreiche Untersuchungen haben durch gezielte Anregung der beiden Photosysteme und den Einsatz von Inhibitoren gezeigt, dass der Redoxzustand des PQ-Pools die state transitions auslöst. Dies geschieht möglicherweise unter Beteiligung des Cyt-b 6 f-komplexes. [54-56]. Der dabei involvierte Mechanismus der Signalweiterleitung ist allerdings noch unbekannt. Zudem ist auch der Mechanismus der state transitions an sich immer noch nicht abschließend geklärt. Durch zahlreiche Untersuchungen haben sich aber drei Modelle etabliert. Spill over - Modell: Direkte Energieübertragung zwischen PS2 und PS1 ohne Beteiligung intermediäre Elektronenüberträger und dem Cyt-b 6 f-komplex. Detachment - Modell: Variation der Menge von PS2-gebundnen PBS Mobile - Modell: Rasche Diffusion von PBS an der Thylakoidoberfläche und Bindung sowohl an PS2, als auch an PS1. 13
24 Einleitung Das Modell des spill over würde durch den Abstand der Photosyteme in der Membran eine Diffusion von PS2 und PS1 voraussetzten. Da mehrere Studien gezeigt haben, dass PS2 in der TYM nicht diffundiert [57,58] und eine Diffusion von PS1-Trimeren auf Grund ihrer Größe unwahrscheinlich ist, hat spill over eher eine geringe Rolle bei den state transitions [59]. Hinweise, dass unter bestimmten Bedingung eine Monomerisierung des PS1 zu einem geringen Anteil von spill over führen kann [60], und die Tatsache, dass in einer Mutante mit ausschließlich monomeren PS1 state transitions beschleunigt werden, lassen dieses Modell aber nicht komplett ausschließen. Besser zur Erklärung der state transitions geeignet sind das detachment - und das mobile - Modell. Zu beiden Modellen gibt es zahlreiche Studien, die entweder das eine oder das andere Modell als wahrscheinlicher annehmen. Für das mobile -Modell spricht, dass die PBS rasch an der Oberfläche der Thylakoidmembran diffundieren können und zudem in der Lage sind sowohl mit dem PS2 als auch mit dem PS1 zu interagieren [61]. Das detachment -Modell wird durch Studien gestärkt, die zeigen, dass eine zusätzliche, verkleinerte PBS-Antenne Energie vornehmlich an das PS1 weiterleitet [62,63]. Zudem scheint die Energieweiterleitung an das PS1 in state 1 und state 2 unverändert zu bleiben, was für eine dauerhafte Bindung der PBS an PS1 spricht [59]. Wie in Abbildung 1-8 gezeigt, setzen sich die state transitions möglicherweise aus einer Kombination der beiden oben aufgeführten Modelle zusammen. Abbildung 1-8: Mögliches Modell der state transitions. Die Diffusion der PBS geht einher mit einer Änderung des Oligomerisierungszustand der Photosysteme. A: state 1 mit PS1-Monomeren. B: state 2 mit PS1-Trimeren (Rögner 2004) Das nicht-photochemische quenching der Cyanobakterien In den letzten Jahren wurde eine Beteiligung der PBS am Schutz gegen überschüssige Anregungsenergie nachgewiesen. El Bissati et al. konnten zeigen, dass starkes, blau-grünes Licht zu einer, quenching genannten, Auslöschung der PS2 Fluoreszenz führt. Dieses ließ sich nicht durch Photoinhibition von PS2 oder state transition erklären, sondern schien von einer thermalen Energieabgabe in den PBS auszugehen [64]. Wilson et al. konnten 14
25 Einleitung nachweisen, dass das lösliche Carotinoidprotein OCP ( orange carotinoid protein ) direkt an dem quenching beteiligt ist [65]. OCP gehört zu den photoaktiven Proteinen und liegt im Dunkeln in seiner stabilen, orangen Form vor. Bei starker Belichtung geht es eine Konformationsänderung zu einer roten Form ein, welche für das quenching verantwortlich ist [66]. Dabei kommt es zu Interaktionen mit den PBS, wodurch hier möglicherweise ebenfalls eine Konformationsänderung ausgelöst wird. Diese Vermutung wird durch die Tatsache bestärkt, dass in vivo ein weiteres Protein, das sogenannte fluorscence recovery protein (frp) zur Wiederherstellung der inaktiven Form des OCP benötigt wird [67]. Das vom OCP induzierte quenching führt zu einer effektiven Verringerung der Antennengröße, welche mit einer stark verringerten Energieweiterleitung von 30 bis 40 % zu den Photosystemen einhergeht [65,67]. 1.8 Effekt der Inaktivierung von Proteinen der PS1-Assemblierung Eine Erhöhung des PS2/PS1 Verhältnisses konnte in Synechocystis nicht nur durch Veränderung der Lichtabsorption, sondern auch durch Eingriffe in die Assemblierung von PS1 erzielt werden. Das PS1 ist einer der drei zentralen Proteinkomplexe im PET. In Membranen von Synechocystis liegt es als Trimer, bestehend aus 36 Proteinuntereinheiten, vor [68]. Die Assemblierung eines solchen Proteinkomplexes und die Integration in die Membran bedürfen der Hilfe weiterer, an der Assemblierung beteiligter, Proteine. In Synechocystis konnte bis jetzt für die drei Proteine Ycf3, Ycf4 und Ycf37 eine Rolle in der Assemblierung von PS1 gezeigt werden. Die Gene für zwei davon, ycf3 und ycf4, kommen in allen bekannten Plastidengenomen von Höheren Pflanzen und Algen vor und konnten auch in Synechocystis nachgewiesen werden. Ein Ausschalten der entsprechenden Gene führt in allen untersuchten Organismen zu verschieden stark ausgeprägten Defekten in der Assemblierung von PS1. Die Inaktivierung von ycf3 verhindert in Chlamydomonas reinhardtii (Chlamydomonas) und Nicotiana tabacum die Assemblierung von funktionellen PS1 und führt zu einem Verlust der Fähigkeit Abbildung 1-9: Modell der Interaktion von Ycf3 und Ycf4 mit PS1 in Synechocystis nach Miao et al. [70] zum photoautotrophen Wachstum [69,71]. Ein vergleichbarer Effekt wurde auch für die 15
26 Einleitung Inaktivierung von ycf4 in Chlamydomonas gezeigt [72]. Dies lässt vermuten das Ycf3 und Ycf4 als Chaperone an der Assemblierung von PS1 beteiligt sind. In Synechocystis führt die Inaktivierung von ycf4 zu einer Reduzierung von assemblierten PS1. Dieses bleibt aber aktiv und die ycf4-mutante zeigt ein mit dem Wildtyp (WT) vergleichbares photoautotrophes Wachstum. Eine Erhöhung des PS2/PS1-Verhältnisses bewirkt zudem gesteigerte Raten des PET [73]. Ycf37 weist eine mit Ycf3 vergleichbare strukturelle Organisation von drei sogenannten tetratrico-peptide Wiederholungen (TPR) auf. Eine Inaktivierung in Synechocystis führt ebenfalls zu einer Erhöhung des PS2/PS1- Verhältnis durch Reduzierung der PS1-Komplexe. Hier konnten aber keine gesteigerten Elektronentransportraten nachgewiesen werden [74]. Wie das Modell in Abbildung 1-9 zeigt, lassen crosslinking -Versuche in Synechocystis eine Interaktion von Ycf3 mit PsaA und von Ycf4 mit PsaC vermuten. 16
27 Einleitung 1.9 Die ATP-Synthase Eine Möglichkeit zur Entkopplung des PET ist eine durch genetic engineering beschleunigte Protonentranslokation durch die ATP-Synthase. Die ATP-Synthase ist ein universelles Membranprotein in der Zellmembran von Bakterien, den Membranen von Mitochondrien und den TYM von Chloroplasten und Cyanobakterien. In Synechocystis nutzt die ATP-Synthase, den von Respiration und Photosynthese gebildeten Proteingradienten an der Thylakoidmembran, zur Synthese von ATP aus ADP und anorganischem Phosphat (P i ), ebenfalls ist ATP-Synthase zur Gegenreaktion, der ATP-Hydrolyse fähig. Der grundlegende Aufbau der ATP- Synthase aus zwei strukturell und funktionell verschiedenen Elementen, F O und F 1, ist in allen Organismen hoch konserviert [75 78]. Wie in Abbildung 1-10 dargestellt, setzt sich der extrinsische F 1 -Teil aus den fünf Untereinheiten α, β, γ, δ und Ԑ in der Komposition α 3 β 3 γδԑ zusammen (in Abbildung: δ gleich I, II) [79]. Der F O -Teil mit der Komposition ab 2 c bildet einen membrandurchspannenden Protonenkanal [80]. F 1 ist verantwortlich für die ATP-Synthese und -Hydrolyse, wobei die katalytisch aktiven Zentren hauptsächlich an den β-ue gebunden sind. Die für die ATP-Synthese benötigte Energie wird aus dem elektrochemischen Potential des Protonengradienten generiert. Dabei führt Bindung und Durchfluss von Protonen durch einen von Abbildung 1-10: Strukturmodell der ATP-Synthase aus Chloroplasten nach Frasch et al. [75]. der C-UE gebildeten Protonenkanal zur Drehung des von der γ- und Ԑ-UE gebildeten Stabs. Dies bewirkt unterschiedliche Bindungseigenschaften der katalytischen Zentren an den β-ue, so dass eine UE ADP und Phosphat bindet, eine zweite das fertige ATP frei gibt, während die Dritte unbesetzt bleibt. Für jedes gebildete ATP gehen γ und Ԑ eine, mit der Protonierung von vier C-UE gekoppelte, 120 Drehung ein [81,82]. Bei einer kompletten Drehung der F 1 -UE 17
28 Einleitung werden also drei Moleküle ATP gebildet und 12 Protonen transportiert. Es wird angenommen, dass ein zweiter an der Peripherie gelegener Stab, gebildet aus der δ-ue von F 1 und den beiden b-ue von F O, für die Stabilisierung des F O F 1 -Komplexes bei der Drehbewegung verantwortlich ist [83]. Zur Verhinderung einer verschwenderischen ATP-Hydrolyse, besonders bei niedrigen Protonengradienten, muss die Funktion der ATP-Synthase strikt reguliert sein. In Bakterien und Chloroplasten ist bekannt, dass die Ԑ-UE bei dieser Regulation eine Rolle spielt [84]. Ein Modell der Struktur der Ԑ-UE ist in Abbildung 1-11 gezeigt. Ihr N-Terminus wird von einem β-faltblatt gebildet, während an ihrem C-Terminus zwei α- Helices zu finden sind. N- und C-Terminus übernehmen in der ATP-Synthase unterschiedliche Funktionen. Der N-Terminus ist in den untersuchten Bakterien essentiell für die Assemblierung des F O F 1 -Komplexes [85 87]. Eine Inaktivierung des αhelicalen C-Terminus hat keine Auswirkung auf die Assemblierung, führt aber zu einer verringerten Kopplu ng von Protonentransport und ATP-Synthese [88 90]. Es wird angenommen, dass Konformationsänderungen der α-helices bei der Regulation der ATP- Synthese und Hydrolyse eine Rolle spielen. Die α-helices können in gefalteter (Abbildung 1-11), oder in entfalteter Konformation auftreten. Je nach Konformation wird die Rotation der ATP-Synthase unterschiedlich beeinflusst. In entfalteter Konformation interagieren positiv geladene Reste mit negativen auf der β-ue und führen so zu einer effektiven Inhibition der ATP-Hydrolyse [84]. Die regulatorische Funktion der Ԑ-UE bei der Kopplung von Protonentransport mit ATP-Synthese und -Hydrolyse machen sie zu dem idealen Kandidaten für ein genetic engineering, um den PET zu entkoppeln. Abbildung 1-11: Strukturmodell der Epsilon-UE der ATP-Synthase (Yagi et al 2007). 18
29 Einleitung 1.10 Channelrhodopsin-2 Eine Entkopplung des PET kann neben dem genetic engineering der ATP-Synthase auch durch die heterologe Expression von Proteinen mit entkoppelnden Eigenschaften erzielt werden. Zu diesen Proteinen gehört das Channelrhodopsin-2 (Chop-2), ein 737 Aminosäure großes Protein aus dem Augenfleck der Grünalge Chlamydomonas. Eine schematische Darstellung der postulierten Struktur ist in der Abbildung 1-12 gezeigt. Der N-Terminus bildet die für Rhodopsine typische Struktur von sieben transmembralen Helices, mit einem zusätzlichen hydrophilen Bereich am C-Terminus, aus. Chop-2 gehört zu den Rhodopsinen des mikrobiellen-typs, welche ein all-trans-retinal als Chromophor binden [92]. In Chlamydomonas fungiert Chop-2 als Photorezeptor in der Phototaxis. Es konnte gezeigt werden, dass Licht der Wellenlänge 460 nm zur Öffnung eines Kationenkanals führt, welcher für Protonen, aber auch eine Reihe zweiwertiger Kationen wie Ba 2+ und Ca 2+ durchlässig ist [91]. Diese Funktion bleibt auch in einer verkürzten Form von 315 Aminosäuren (Chop-2-315) erhalten, welche nur den transmembralen N-Terminus ausbildet. Versuche in Oocyten von Xenopus laevis haben gezeigt, dass Chop-2 nach Belichtung einen effektiven Abbau des Membranpotentials bewirkt [93]. Bei erfolgreicher Expression stellt Chop-2 so ein geeignetes Mittel dar eine Entkopplung des PET von der Reaktion der ATP-Synthase auszulösen Lichtregulierte Promotoren Abbildung 1-12: Schematische Darstellung der Struktur von Chop-2 mit dem Kofaktor all-trans Retinal nach Nagel et al. [91] Im Hinblick auf eine biotechnologische Anwendung eines Organismus ist es von entscheidender Bedeutung, dass es Kontrollmechanismen gibt, die es erlauben, bestimmte Stoffwechselwege an- und auszuschalten. Dies gilt nicht nur explizit, für den in dieser Arbeit vorgestellten Design-Organismus, sondern auch für jede andere biotechnologische Anwendung. Die etwaigen Kontrollmechanismen müssen dabei nicht nur eine strikte Kontrolle erlauben, sondern müssen im Hinblick auf wirtschaftliche Anwendungen auch kostengünstig sein und ohne Zusatz giftiger oder teurer Chemikalien auskommen. 19
30 Einleitung Für Synechocystis ist die Anpassung des Metabolismus an wechselnde Lichtbedingungen essentiell. Die ersten Schritte der Regulation finden hierbei auf der Ebene der Transkription statt, wobei Promotoren eine Schlüsselrolle in der Initiation der RNA-Synthese spielen. In Synechocystis existieren, neben zahlreichen Promotoren mit konstitutiver Lichtaktivität [94,95], ebenfalls Promotoren deren Aktivität von der Lichtintensität beeinflusst wird und so für eine kostengünstige, lichtgesteuerte Expression genutzt werden könnten. Die Untersuchung von lichtregulierten Promotoren in Synechocystis gewinnt zunehmend an Interesse, doch sind bis jetzt noch keine strikt lichtregulierten Promotoren bekannt. Für die Expression eines sognannten high light inducible polypeptide C (HliC) konnte eine Abhängigkeit von der Lichtintensität gezeigt werden. Bei normalen Lichtbedingungen konnte kein HliC nachgewiesen werden, während es bei Hochlicht zu einer raschen Akkumulation von HliC kommt [96]. HliC ist Teil der allgemeinen Stressantwort in Synechocystis, so dass seine Expression nicht nur durch Hochlichtstress, sondern auch durch andere Stressbedingungen induziert wird. HilC ist vermutlich am NPQ der Zelle beteiligt und dient bei Stressbedingungen dem Schutz von PS2. Das D1-Protein ist eine der zentralen UE des PS2 und unterliegt auf Grund rascher Lichtschädigung einem hohen Austausch. In den Genomen vieler photosynthetischer Organismen liegen mehrere Kopien des Gens (psba) vor, welche für gleiche oder ähnliche Kopien von D1 kodieren. In Synechocystis finden sich zwei Kopien (psba2 und 3) im Genom, welche für ein identisches Protein kodieren. Bei niedrigen Lichtbedingungen werden ca. 90 % der D1-Kopien von psba2 und etwa 10% von psba3 gestellt. Bei hohen Lichtintensitäten kommt es zu einer Akkumulation von psba3-transkripten [97]. Der Promotor von psba3 zeigt also eine deutlich höhere Aktivität bei starker Intensität, ist aber auch bei niedriger aktiv und damit nicht strikt reguliert. Durch möglichweise veränderte Promotorregulationen in Synechocystis und Thermosynechococcus elongatus (T. elongatus), könnte eine Expression unter der Kontrolle des psba3-promotors aus T. elongatus eine strikte Lichtregulation in Synechocystis erlauben. 20
31 Einleitung 1.12 Ziele der Arbeit Die Ziele dieser Arbeit stehen im Kontext des Designs eines Systems für eine photosynthesebasierte H 2 -Produktion. Hierbei soll die Kopplung des PET von Synechocystis mit der H 2 -Produktion einer Hydrogenase als Modell für zukünftige, biotechnologisch verwendbare Systeme dienen. Ziel dieser Arbeit war die Charakterisierung des PET zur Evaluierung limitierender Prozesse. Durch gerichtete Mutation sollten diese optimiert und deutliche Steigerungen der photosynthetischen Wasserspaltungsraten erzielt werden. Als grundlegende Annahme galt vor dieser Arbeit, dass sowohl eine Steigerung des PS2/PS1-Verhältnisses durch Verkleinerung der Antennen als auch eine Entkopplung zu deutlichen Steigerungen des PET führen. Ausgehend davon sollten, durch Mutation und heterologe Expression, verschiedene Modelle für eine Entkopplung des PET in Synechocystis getestet werden. Zudem sollte durch Eingriff in die Assemblierung des PS1 der Effekt einer weiteren Steigerung des PS2/PS1- Verhältnisses untersucht werden. Eine Kombination von Entkopplung und erhöhtem PS2/PS1-Verhältnis in Doppel- und Dreifach-Mutanten soll den Elektronentransport weiter steigern. Charakterisierungen von Elektronentransportleistungen und Elektronenverlusten bei unterschiedlichen Lichtqualitäten und quantitäten sollten mögliche Kandidaten für eine photobiologische H 2 -Produktion aufzeigen. Die Eignung dieser Kandidaten sollte anschließend durch Langzeitmessungen im Flachbettbioreaktor verifiziert werden. 21
32 Material und Methoden 2 Material und Methoden 2.2 Mikrobiologische Methoden Stämme und Kultivierung von Escherichia coli Die hier vorgestellten molekularbiologischen Methoden wurden alle in dem Escherichia coli (E. coli) Stamm DH5α durchgeführt. Die Anzucht von E. coli-flüssigkulturen erfolgte in LB- Medium LB-Medium: Hefeextrakt NaCl Trypton 5 g 5 g 10 g mit A.bidest auf 1000 ml auffüllen. Eingestellt auf ph 7,2 Zur Herstellung von Agarplatten wurde vor dem Autoklavieren 1,5 % (w/v) Agar zu dem LB- Medium gegeben. Wenn es erforderlich war, wurden Agarplatten und LB-Medium nach dem Autoklavieren mit entsprechenden Konzentrationen von Antibiotika versetzt. Wurden zwei Antibiotika zum Medium gegeben, wurde die Konzentration eines der beiden Antibiotika auf 50 % reduziert. Die folgenden sterilfiltrierten Antibiotika wurden verwendet: Ampicillin Kanamycin Chloramphenicol 100 µg/ml 50 µg/ml 50 µg/ml Kryokulturen Zur langfristigen Lagerung von Stämmen und Sicherung von Plasmiden wurden E. coli- Kryokulturen hergestellt. Dazu wurden 600 µl einer E. coli Kultur mit 200 µl Glycerin versetzt und, nach einer Inkubation für 30 min auf Eis, bei -80 C gelagert. 22
33 Material und Methoden Verwendete Cyanobakterien und ihre Kultivierung In dieser Arbeit wurden die in Tabelle aufgelisteten Cyanobakterienstämme verwendet. Tabelle 2-1-1: Verwendete Cyanobakterienstämme. Stämme Genotyp Ursprung Synechocystis sp. PCC 6803 Synechocystis sp. Olive Synechocystis sp. PAL Synechocystis sp. CԐ Wildtyp, Glukosetolerant Durch ungerichtete Mutation entstanden. Verlust des Phycocyanin Gene für Phycocyanin und Allophycocyanin inaktiviert ( apce apcab) Deletion des C-Terminus der Epsilon- Untereinheit der ATP-Synthase ( CԐ) Pasteur Culture Collection Stammsammlung des LS Biochemie der Pflanzen, Bochum Zur Verfügung gestellt von Ghada Ajlani und Zoltan Gombos, Ungarn Zur Verfügung gestellt von Toru Hisabori, Tokyo [98] Synechocystis sp. DM Olive mit zusätzlicher Deletion von CԐ Diese Arbeit Synechocystis sp. TM Synechocystis sp. PpsbA3 Synechocystis sp. PhilC Synechocystis sp. PSyR1 Synechocystis sp. PpetF Synechocystis sp. PpetE Synechocystis sp. PnrsABCD Synechocystis sp. Chop-2 DM mit zusätzlicher Deletion des PS1- Assemblierungsproteins Ycf4 (Olive CԐ ycf4) Einbau von Promotor des Gens psba3 aus Thermosynechococcus elongatus zusammen mit GFP Einbau von Promotor des Gens hilc zusammen mit GFP Einbau von Promotor des Gens SyRI zusammen mit GFP Einbau von Promotor des Gens petf zusammen mit GFP Einbau von Promotor des Gens pete zusammen mit GFP Einbau von Promotor des Operons nrsabcd zusammen mit GFP Fusionsprotein aus Chop-2 und GFP mit Promotor des Gens petf. Diese Arbeit Diese Arbeit Diese Arbeit Diese Arbeit Diese Arbeit Diese Arbeit Diese Arbeit Diplomarbeit Jan Hagenbucher 23
34 Material und Methoden Cyanobakterielle Dauerkulturen und frisch von Platte inokulierte Kulturen wurden in Erlenmeyer Kolben bei 30 C und 50 µe Belichtung auf dem Schüttler angezogen. Messkulturen und Vorkulturen für die Anzucht im Fermenter wurden in speziell angefertigten Begasungskolben im 30 C Wasserbad bei 95 µe Belichtung und unter Begasung mit bis zu 5 % CO 2 angereicherter Luft kultiviert. Als Anzuchtmedien wurden sowohl BG-11, als auch Y-BG-11 [99] verwendet. Y-BG-11: NaNO 3 17,5 mm Na 2 MoO 4 2 H 2 O 1,6 μm MgSO 4 7 H 2 O 0,3 mm ZnSO 4 7 H 2 O 0,7 μm Zitronensäure 30 μm CuSO 4 5 H 2 O 0,3 μm K 2 HPO 4 1,75 mm Co(NO 3 ) 2 6 H 2 O 0,16 μm HBO 3 45 μm Na 2 EDTA 2 H 2 O 16 μm MnCl 2 4 H 2 O 9 μm FeCl 3 6 H 2 O 6,0 μm Na 2 CO 3 0,18 mm HEPES/NaOH( ph 7,8) 10 mm BG-11: Spurenelemente: NaNO 3 17,6 mm H 3 BO 346,3 mm MgSO 4 7 H 2 O 304,3 µm MnCl 9,2 mm Ca 2 Cl H 2 O 244,9 µm Na 2 MoO 4 2 H 2 O 1,6 mm K 2 HPO 4 3 H 2 O 175 µm ZnSO 4 7 H 2 O 772,1 µm Na 2 CO µm CuSO 4 5 H 2 O 316,4 µm Eisenammoniumcitrat 57 µm Zitronensäure 31,23 µm EDTA 2,7 µm Spurenelemente HEPES/NaOH (ph 8.2) 0,1 % (v/v) 5 mm Zur Herstellung von BG-11 Agarplatten wurden zu 500 ml BG-11 3 g Na 2 S 2 O 3 gegeben. Außerdem wurden 15 g Difto Bacto Agar in 500 ml H 2 O eingewogen und beide Lösungen getrennt autoklaviert. Vor dem Gießen der Platten wurden die Lösungen vermischt und, wenn erforderlich, mit dem entsprechenden Antibiotikum versetzt. 24
35 Material und Methoden Folgende sterilfiltrierten Antibiotika wurden verwendet: Kanamycin Spectinomycin Chloramphenicol 25 µg/ml 25 µg/ml 25 µg/ml 2.3 Molekularbiologische Methoden Oligonukleotide und Plasmide Tabelle 2-1-2: In der Arbeit verwendete und konstruierte Primer Primerbezeichnung Sequenz 5` 3` Verwendung apcab-down-for ACGTGCTCGAGGTCCTGGATTCCCGTGGGTG Ampilifizierung des upstream -Bereichs von apca apcab-down-rev ACGTGGAGCTCCCCCGCTTCCATTGGCAATT Ampilifizierung des upstream -Bereichs von apca up-apcab-rev ACGTCGAGCTCCATGCCACCACCCAGCTTTG Ampilifizierung des downstream -Bereichs von apcb Up-apcAB-for ATCGTCTCGAGTCCTGGATTCCCGTGGGTGA Ampilifizierung des downstream -Bereichs von apcb seg-apcab-for GGGGAGCTGTTTCTTGATTC Überprüfung der Segregation von apcab seg-apcab-rev GCAACAACTAGTCTCAAGCTTAGG Überprüfung der Segregation von apcab seq-sll1909 ACCCGCATTTCCAAGGATTC Sequenzierung des Plasmids apcab seq-sll1949 CCCGTAACGTAACCGTTGGCG Sequenzierung des Plasmids apcab deltaefor CCGGTCAGGCATTAGATAAACCAC Überprüfung der Segregation von CԐ seq-apca CGCCGCCCTGGGGTTAATTC Sequenzierung des Plasmids apcab piga-np rev CGTATGGTACCACCACCTCAAATTGGGAATTTG Amplifizierung von PnrsABCD zur Konstruktion von pmb1 25
36 Material und Methoden Primerbezeichnung Sequenz 5` 3` Verwendung pr-hlic-for GATCGGATATCGGCGGGTTAGTTTCCAGTGC Amplifizierung von PhilC zur Konstruktion von pmb2 pr-hilc-rev GCTAGGGTACCGGTTAAGAATGTAATTACGAGC TGG Amplifizierung von PhilC zur Konstruktion von pmb2 pr-pete-for GTACTGATATCCAAGGCAAAACCACCGTTATC Amplifizierung von PpetE zur Konstruktion von pmb3 pr-pete-rev GCTAGGGTACCACTTCTTGGCGATTGTATCTAT AGG Amplifizierung von PpetE zur Konstruktion von pmb3 pr-petf-for GATCTGATATCCTGGTCGTCGTCATACATAC Amplifizierung von PpetF zur Konstruktion von pmb4 pr-psba3-for GTGACGATATCCCCTGAGTCTCTGTCTGCGG Amplifizierung von PpsbA3 aus T, elongatus zur Konstruktion von pmb5 ycf4down-for ACTCAGGTACCGACAGTGCAATTAACGTTTATG Amplifizierung des downstream -Bereichs von ycf4 pr-petf-rev GATCGGGTACCAGATGTTTCCTCTTCTCAAA Amplifizierung von PpetF zur Konstruktion von pmb4 ycf4down-rev ACTACGGATCCTTTCTCTAACAATAATTACAACG Amplifizierung des downstream -Bereichs von ycf4 ycf4up-for ACGTAGATATCATTGCCATTCCCGTGCTGTC Amplifizierung des upstream -Bereichs von ycf4 ycf4up-rev ACGTAGTCGACCACCACCACAGCGGCATCAG Amplifizierung des upstream -Bereichs von ycf4 seqsll0228 GGACCCTGCAGTAGAAAATG Sequenzierung Plasmid ycf4 upslr0168 GTTTGTGGGTATTGCCGATG Sequenzierung Promotorbereich in piga seqpucdown CCGGCTCGTATGTTGTGTGG Sequenzierung von puc- Vektoren 26
37 Material und Methoden Tabelle 2-1-3: In der Arbeit verwendete und konstruierte Plasmide Plasmide Charakterisierung Referenz piga Km R, Amp R, GFP, RBS von atpe, slr0168 [95] puc18 Amp R, lacz, rep(pmb1) GeneScript p CԐ Sp R, atpe (258/411bp) [93] pmb1 Km R, Amp R, GFP, PnrsABCD, slr0168 Diese Arbeit pmb2 Km R, Amp R, GFP, PhilC, slr0168 Diese Arbeit pmb3 Km R, Amp R, GFP, PpetE, slr0168 Diese Arbeit pmb4 Km R, Amp R, GFP, PpetF, slr0168 Diese Arbeit pmb5 Km R, Amp R, GFP, PpsbA3, slr0168 Diese Arbeit pmb6 Km R, Amp R, GFP, PsyrI, slr0168 Diese Arbeit p ycf4 Km R,, ycf4 Diese Arbeit p CԐ-KM Km R, atpe (258/411bp) Diese Arbeit p apcab Km R, apcab Diese Arbeit piga-chop2 Km R, Amp R, Chop-2-GFP, PpetF, slr0168 Diplomarbeit Jan Hagenbucher Agarose-Gelelektrophorese Zur Herstellung eines Agarosegels wurde standardmäßig eine Agarosekonzentration von 1 % (w/v) eingesetzt. Dazu wurde die Agarose in einem entsprechenden Volumen TAE-Puffer aufgenommen und geschmolzen, bis sich eine homogene Lösung eingestellt hat. Zum Nachweis von DNA wurden 0,5 µl Ethidiumbromid-Lösung (s.u.) zur Agaroselösung gegeben. Das Gel wurde in einer entsprechenden Vorrichtung gegossen und Taschen mit Hilfe eines Kamms eingefügt. Nach Erstarren des Gels wurde es mit TAE-Puffer bedeckt. Zur Trennung der DNA-Fragmente wurde eine Spannung von 6 V/cm Elektrodendistanz angelegt. Das Ergebnis der Trennung wurde durch Anregung der Ethidiumbromid- Fluoreszenz mit UV-Licht der Wellenlänge von 312 oder 365 nm überprüft. Die Größe der getrennten Fragmente kann dabei durch Vergleich mit einem Standard bestimmt werden (Abbildung 2-1). 27
38 Material und Methoden TAE-Puffer (50x): Ethidiumbromid: TRIS 242 g 10 mg/ml Eisessig EDTA 0,5 M (ph8,0) 57,1 ml 100 ml Aqua bidest. auf 1000 ml Die verwendeten DNA-Proben wurden im Verhältnis 1:6 mit 6-fach DNA-Probenpuffer versetzt. DNA-Probenpuffer (6x): Glycerin Bromphenol Blau Xylenecyanol FF 30 % (v/v) 0,25 % (w/v) 0,25 % (w/v) Abbildung 2-1: GeneRuler 1 kb DNA Ladder. #SM0311 (Fermentas) Extraktion von DNA aus Agarosegelen Zur Extraktion von DNA aus dem Agarosegel wurden Extraktions-Kits der Firmen Machery&Nagel oder Fermentas verwendet. Die Extraktion erfolgte dabei nach Herstellerangaben. Das abschließende Lösen der DNA erfolgte für 10 min mit A.bidest Extraktion von genomischer DNA aus Synechocystis Eine Synechocystis-Kultur wurde bis zu einer OD 750nm von 2 inkubiert. Von dieser Kultur wurden 20 ml sedimentiert (5 min bei 4800 rpm) und in 200 µl TE-Puffer resuspendiert. Zum Aufschluss wurden die Zellen, nach Zugabe von ca. 200 µl Glasperlen (0,1-0,2 mm) und 200 µl Phenol, fünfmal für ca. 10 s auf einem Kleinschüttler (Vortex) aufgeschlossen. Zwischen den Aufschlussschritten wurden die Proben auf Eis gekühlt. Die Proben wurden für 1 min bei g zentrifugiert und die obere, wässrige Phase in ein neues Reaktionsgefäß überführt. Es erfolgte eine Wiederholung der Zugabe von Phenol, gefolgt von starken Mischen und erneuter Zentrifugation. 28
39 Material und Methoden Die obere, wässrige Phase wurde abgenommen und mit 200 µl Chloroform gemischt. Anschließend wurde die Suspension für 1 min bei g zentrifugiert. Die obere, wässrige Phase wurde abgenommen und der Chloroformschritt wiederholt. Zum Fällen der DNA wurde die obere, wässrige Phase mit dem 2,5 fachen Volumen Ethanol (98 %, PA) und einem Zehntel Volumen Natriumacetat (3 M, ph 4,8) gemischt und 5 min auf Eis inkubiert. Die DNA wurde dann für 5 min bei g sedimentiert und das Pellet zweimal mit 75 % (v/v) Ethanol (PA) gewaschen. Nach Trocknen wurde die DNA im gewünschten Volumen A.bidest resuspendiert Extraktion von Plasmid-DNA aus E. coli Die Extraktion von Plasmid-DNA erfolgte nach der Methode der alkalischen Lyse, gefolgt von einer Phenol/Chloroform Extraktion. Im ersten Schritt wurde eine 3 ml ÜN-Kultur mit dem entsprechenden Plasmid bei g (5 min) sedimentiert. Das Pellet wurde in 125 µl GTE- Puffer resuspendiert und die Lösung bei RT für 5 min inkubiert. Danach wurden 300 µl einer NaOH/SDS-Lösung zu den Zellen gegeben und die Lösung für 5 min bei RT inkubiert. Dies führt zum Aufbruch der Zellen und denaturiert die in der Zelle vorhandenen Proteine. Anschließend wurden 225 µl Natriumacetat (3 M; ph 4,8) hinzugegeben, gefolgt von einer erneuten Inkubation für 5 min. Die Proteine in der Lösung fallen aus und werden anschließend für 10 min bei g sedimentiert. Der Überstand wird nun zur Fällung der vorhandenen Plasmid-DNA mit 600 µl Isopropanol für 5 min auf Eis inkubiert. Anschließend wurde die DNA für 5 min bei g sedimentiert und das Pellet in 200 µl TES-Puffer mit 10 µl RNase (10 mg/ml) resuspendiert und für eine Stunde bei 37 C inkubiert, um die vorhandene RNA abzubauen. Nach Zugabe von 200 µl PCI-Lösung, folgte starkes Mischen und eine Zentrifugation für 1 min bei g. Die obere, wässrige Phase wurde mit 400 µl Ethanol (98 %, PA) gemischt, um die vorhandene Plasmid-DNA zu fällen. Diese wurde anschließend durch Zentrifugation (5 min, g) sedimentiert und das Pellet zweimal mit Ethanol (70 % (v/v), PA) gewaschen. Nach Trocknen des Pellets (SpeedVac) wurde die Plasmid-DNA im gewünschten Volumen A.bidest aufgenommen. GTE: NaOH/SDS: Glukose 50 mm NaOH 200 mm TRIS (ph 8,0) 25 mm SDS 1 % (w/v) EDTA 10 mm 29
40 Material und Methoden TES: PCI: TRIS (ph 8,0) 50 mm Phenol 50 % (v/v) EDTA 10 mm Chloroform 49 % (v/v) NaCl 150 mm Isoamylalkohol 1 % (v/v) Photometrische Bestimmung von Reinheit und Konzentration der DNA Zur photometrischen Überprüfung der Reinheit und Konzentration der DNA wurde die Extinktion von 1:10 und 1:100 Verdünnungen der Proben im Photometer (NanoDrop) bei 260 nm und 280 nm gemessen. DNA hat eine maximale Absorption bei 260 nm, wobei eine Extinktion von 1 einer Konzentration von 50 µg/ml doppelsträngiger DNA entspricht. Der Nachweis von Proteinen erfolgt dagegen photometrisch bei 280 nm, da bei dieser Wellenlänge die aromatischen Aminosäuren absorbieren, welche in allen Proteinen vorkommen. Das Verhältnis der Absorptionen bei 260 nm und 280 nm kann daher zur Überprüfung der Reinheit einer DNA-Probe genutzt werden. Hierbei entspricht ein Verhältnis von 1,9 (260 nm/280 nm) einer reinen DNA-Lösung. 2.4 Enzymatische Modifikation von DNA Restriktion Zu analytischen Zwecken wurde die Restriktion in 10 µl Ansätzen durchgeführt, bei präparativen Restriktionen wurde das Volumen auf 50 µl erhöht. Durch analytische Restriktion wurden verwendete Plasmide charakterisiert. Hier wurden 1 bis 2 µl Plasmid eingesetzt. In präparativen Restriktionen wurden DNA-Fragmente für anschließende Ligationen erzeugt. Dabei wurden bis zu 30 µl Plasmid eingesetzt. Die Durchführung der Restriktion erfolgte sowohl bei analytischer als auch bei präparativer Restriktion nach Herstellerangaben. Für die Restriktion wurden in der Regel ca. 5 U des verwendeten Enzyms pro µg DNA eingesetzt und die Restriktion erfolgte bei 37 C für 1 bis 16 h. Bei präparativen Restriktionen erfolgte eine Trennung der entstandenen Fragmente über ein Agarose-Gel, gefolgt von einer Extraktion wie unter beschrieben. 30
41 Material und Methoden Ligation Ligationen wurden in einem Maßstab von 20 µl durchgeführt. Dabei wurden Plasmid und Insert in einem Verhältnis von 1:3 bis 1:5 eingesetzt. Die dafür erforderlichen DNA-Mengen wurden photometrisch oder durch Vergleich der Fluoreszenzstärke im Agarose-Gel bestimmt. Für die Ligation wurde maximal ein Gesamt-DNA Volumen von 10 µl eingesetzt. Zu den Ligationsansätzen wurde 1 U T4-Ligase gegeben und die Ligation erfolgte für 1 h bei 37 C. Zur Kontrolle der Ligation wurde ein zusätzlicher Ansatz ohne Insert erstellt Dephosphorylierung Eine Desphosphorylierung von restringierter Plasmid-DNA erfolgte, um eine Religation von nicht vollständig geschnittenen Plasmidfragmenten zu verhindern. Dies wurde insbesondere dann durchgeführt, wenn es auf Grund ähnlicher Fragmentgrößen nicht möglich war, die bei der Restriktion entstandenen Fragmente über Agarose-Gel Elektrophorese zu trennen. Zur Dephosphorylierung wurde 1 U CIAP ( Calf intestinal alkaline phosphatase ) zu den Restriktionsansätzen gegeben. Anschließend erfolgte eine Inaktivierung des Enzyms für 10 min bei 85 C. Die Ansätze wurden durch Agarose-Gel Elektrophorese und anschließender Gelextraktion isoliert Polymerase Ketten Reaktion (PCR) In dieser Arbeit wurden verschiedene analytische und präparative PCRs durchgeführt. Präparative PCRs erfolgten in der Regel in einem Maßstab von 50 µl und wurden genutzt, um DNA für Klonierungsarbeiten zu erzeugen. Dabei wurden die, für die Klonierung erforderlichen Restriktionsschnittstellen, während der PCR über Primeranhänge an das Fragment angefügt. Analytische PCRs wurden in einem 20 µl Maßstab durchgeführt. In der analytischen PCR wurde die Taq-DNA-Polymerase von Segenetic, für eine präparative PCR die Phusion-DNA- Polymerase der Firma Biotherm verwendet. 31
42 Material und Methoden Typischer Ansatz einer präparativen PCR: DNA ng Primer 1 (5 µm) 5 µl Primer 2 (5 µm) 5 µl dntp-mix (2 µm 5 µl Phusion-Puffer (5x) 10 µl Phusion-Polymerse 0,5 µl A.Bidest auf 50 µl Bei Ansätzen mit blunt end DNA-Fragmenten wurde zusätzlich 2 µl DMSO in den Ansatz gegeben. Typischer Reaktionsverlauf einer präparativen PCR: A: Einleitende Denaturierung 98 C 30 s B: Denaturierung 98 C 5-10 s Anlagerung C s Verlängerung 72 C 15 s pro kb C: Abschließende Verlängerung 72 C 10 min Pause 10 C In Schritt A wird durch eine einleitende Denaturierung der DNA der Doppelstrang aufgebrochen. Die unter Punkt B aufgeführten Reaktionsschritte wurden 29 mal wiederholt. Dabei wurde die Anlagerungstemperatur so gewählt, dass sie ca. 10 C unter der Schmelztemperatur der Primer lag. Die Zeit zur Verlängerung der Oligonukleotide richtete sich nach der erforderlichen Fragmentgröße. Im letzten Schritt C wurde für 10 min eine Verlängerung der Oligonukleotide durchgeführt, um alle begonnenen Fragmente zu vervollständigen. Danach endet die PCR und die Probe wird gekühlt. 32
43 Material und Methoden Transformation von DNA in E. coli Für die Transformation in E. coli wurden zunächst kompetente Zellen erzeugt. Dazu wurde eine 3 ml ÜN-Kultur angeimpft und bei 37 C in einem Schüttler inkubiert. Die ÜN-Kultur wurde dazu genutzt eine 50 ml Kultur zu inokulieren. Nachdem die 50 ml Kultur eine OD 600 von 0,6 bis 0,9 erreicht hatte, wurden die Zellen sedimentiert (10 min, 4800 rpm, 4 C, S4180). Das Pellet wurde in 5 ml einer eiskalten CaCl 2 -Lösung resuspendiert und die Zellen anschließend erneut sedimentiert (10 min, 4800 rpm, 4 C, S4180). Dieser Schritt wurde insgesamt dreimal wiederholt. Zum Schluss wurden die Zellen in 1,25 ml der CaCl 2 -Lösung aufgenommen und in 100 µl Volumina aliquotiert. Bevor die Zellen zur dauerhaften Lagerung bei -80 C eingefroren wurden, wurden sie für 30 min auf Eis inkubiert. Zur Transformation in E. coli wurden maximal 50 ng DNA zu einem 100 µl Aliquot kompetenter DH5α-Zellen gegeben. Die Zellen wurden für 30 min auf Eis inkubiert. Anschließend erfolgte ein Hitzeschock für 2 min bei 42 C. Nach dem Hitzeschock wurde zu den Zellen 1 ml LB-Medium gegeben und diese 30 bis 60 min bei 37 C in einem Schüttler inkubiert. Nach der Inkubation wurden die Zellen auf Agarplatten mit den entsprechenden Antibiotika ausplattiert. Bei der Transformation von Plasmid-DNA wurden die Volumina 10 µl, 100 µl und der Rest des Ansatzes, bei Ligationen jeweils die Hälfte des Transformationsansatzes ausplattiert Transformation von DNA in Synechocystis Synechocystis gehört zu den natürlich kompetenten Organismen, d.h. die Zellen sind in der Lage externe DNA aufzunehmen und in ihr eigens Genom zu integrieren. Zur Transformation wurden 20 ml einer Synechocystis-Kultur mit einer OD 750 > 2 sedimentiert (10 min, 4800 rpm, S4180) und das Pellet in 3 ml frischem BG-11 Medium aufgenommen. Die Zellen wurden in 100 µl Aliquoten aufgeteilt und mit 1 µg DNA (Plasmid oder genomisch) gemischt. Anschließend wurden die Zellen ÜN im Dunkel bei 30 C in einem Schüttler inkubiert und auf Platten mit entsprechenden Antibiotikakonzentrationen ausgestrichen. Zur Erhöhung der Transfromationseffizienz wurde gegebenenfalls Glukose in einer Endkonzentration von 5 µm zum Medium gegeben. Zudem wurden die Zellen in einzelnen 33
44 Material und Methoden Fällen vor der Transformation für zwei Tage in Medium mit EDTA (2 mm) inkubiert, um calciumabhängige Nukleasen in der Zytoplasmamembran zu inaktivieren. 2.5 Proteinbiochemische Methoden Zellaufschluss und Membrantrennung Zum Zellaufschluss von Synechocystis PCC 6803 wurden 300 ml einer Zellkultur sedimentiert und das Pellet nach der Zentrifugation (10 min, rpm, SS34) in entsprechendem Volumen HEN-Puffer zur Einstellung der OD 750nm von 6 aufgenommen. Der Zellaufschluss erfolgte anschließend in der French Press (FA-032) mit zwei Durchgängen von jeweils einer Minute bei psi. Danach wurden nicht aufgeschlossene Zellen und Zelltrümmer durch Zentrifugation sedimentiert (15 min, rpm, SS34). Anschließend wurden lösliche Proteine und die Gesamtmembran in der Ultrazentrifuge getrennt (1h, rpm, Ti70). 10x HEN-Puffer: HEPES/NaOH (ph 7,4) NaCl EDTA 200 mm 50,0 mm 100 mm Für die Membranisolierung aus Synechocystis wurden die Kulturen entweder im 5 l Flachbettbioreaktor oder in Begasungsröhren mit einem Volumen von ca. 350 ml bis zu einer OD 750nm von 2,5 bis 3 angezogen. Die Zellen wurden bei 4 C für 10 min bei 4000 g sedimentiert und in 10 ml HEN-20 (20 % (w/v) Saccharose) aufgenommen. Nach der Zugabe von 0,4 % (w/v) Lysozym wurden die Zellen für 90 min unter konstantem Schütteln im Dunkeln inkubiert. Anschließend wurden die Zellen erneut sedimentiert (10 min, 4000 g, 4 C) und in 10 ml HEN-20 mit 5 µl/ml DNase (10 mg/ml), 5 µm E-64 und 1 mm Pefablock aufgenommen. Nach dem anschließenden Zellaufschluss in der French Press (zwei Durchgänge, 1600 psi) wurde den Zellen 10 µl/ml PMSF und 1 µl/ml TLCK zugegeben. Sollten vor der Membrantrennung auch Proben der löslichen Proteine und der Gesamtmembran genommen werden, wurde die Probe mit HEN-Puffer auf eine Saccharosekonzentration von 10 % (w/v) eingestellt und durch Zentrifugation (10 min, rpm, Ti-70, 4 C) die Gesamtmembran von den löslichen Proteinen getrennt. Für eine weitere Auftrennung der Membranen wurde das Pellet in HEN-Puffer gelöst und mit Saccharose auf eine Konzentration von 50 % (w/v) eingestellt. 34
45 Material und Methoden Erfolgte nur eine Membrantrennung, wurden die Proben direkt nach dem Aufschluss in der French Press auf eine Saccharosekonzentration von 50 % (w/v) eingestellt. Das Lösen der Saccharose erfolgte durch konstantes Schütteln für 30 min bei 4 C. Anschließend erfolgte das Gießen des Saccharosedichtegradienten mit den Schichten: 50, 42, 35, 30, 10 und 0 % (w/v). Dieser wurde für mindestens 12 h bei 4 C zentrifugiert (27000 rpm, SW-28). Danach konnten die entsprechenden Membranfraktionen abgenommen und in HEN-Puffer verdünnt werden. Nach einer erneuten Zentrifugation für 1 h bei rpm (Ti-70, 4 C) wurden die Fraktionen in 1 bis 3 ml HEN-Puffer aufgenommen und bei -20 C gelagert TCA-Fällung von Proteinen Eine TCA-Fällung von Proteinen wurde durchgeführt, um bei der SDS-Gelelektrophorese störende Verunreinigungen zu entfernen oder um eine bestimmte Proteinkonzentration einzustellen, dafür wurde die Bestimmungen der Proteinkonzentrationen mit dem Roti - Quant universal Kit auf 96 Well Mikrotiterplatten durchgeführt. Zur Fällung wurde zu der Proteinlösung ein Viertel des Volumens an 100 % (v/v) Trichloressigsäure (TCA) gegeben, gefolgt von einer Inkubation für 30 min auf Eis. Anschließend wurden die gefällten Proteine sedimentiert (10 min, rpm, 4 C) und der Überstand verworfen. Um den Überstand vollständig zu entfernen, wurde der Zentrifugationsschritt wiederholt und das Pellet anschließend zweimal mit 200 µl 100 % (v/v) Aceton gewaschen (5 min, rpm, 4 C). Nach Trocknen des Pellets (SpeedVac) wurde es in 5 µl HEN-Puffer gelöst und anschließend mit 15 µl SDS-Probenpuffer mit 100 mm NaOH versetzt. Nach einer Inkubation für 20 min bei 37 C auf dem Schüttler zum vollständigen Lösen des Pellets erfolgte eine zweite Inkubation bei 60 C für 30 min. Zum Abschluss wurden die Proben für 1 min zentrifugiert (12000 rpm, 4 C). SDS-Probenpuffer TRIS/HCl (ph 8,0) SDS Glycerin β-mercaptoethanol Bromchlorphenolblau 6,2 mm 3,1 % (w/v) 10 % (w/v) 5 % (w/v) 0,01 % (w/v) 35
46 Material und Methoden SDS-Gelelektrophorese nach Laemmli. Zum Gießen der SDS-Gele wurden Gießstände der Firma BioRad verwendet. Das Trenngel wurde zwischen die Glasscheiben gegossen und während des Polymerisierens mit einer Schicht Isopropanol bedeckt. Nach Aushärten des Trenngels wurde das Isopropanol verworfen und Reste mit einem Filterpapier entfernt. Anschließend wurde das Sammelgel auf das Trenngel gegossen. Zur Erzeugung von Probentaschen wurde ein Gelkamm in das Sammelgel eingeführt. Die Trennung erfolgte in Mini-PROTEAN 3 Kammern der Firma Bio RAD. Für die SDS-Gelelektrophorese verwendete Proben wurden vor dem Auftragen mit TCA gefällt (s ). Zum Größennachweis der Proteinproben auf dem SDS-Gel wurde der PageRuler Prestained Protein Ladder SM0671 verwendet (Abbildung 2-2). Die Gel-Elektrophorese wurde bei einer konstanten Spannung von 100 V durchgeführt. Sammelgelpuffer: Trenngelpuffer: TRIS/HCl (ph 6,8) 1 M TRIS/HCl (ph 8,8) 2 M SDS 0,4 % (w/v) SDS 0,4 % (w/v) Laufpuffer: TRIS/HCl (ph 8,3) Glycin SDS 0,5 M 1,92 M 1 % (w/v) Tabelle 2-1-4: Zusammensetzung für 2 SDS-Gele nach Laemmli Trenngel (12 %) Sammelgel (5 %) Acylamid/Bisacrylamid (37,7:1) 4 ml 1 ml Sammelgelpuffer _ 0,75 ml Trenngelpuffer 3,8 ml _ H 2 O 1,8 ml 4,2 ml APS 20 % 0,04 ml 0,04 ml TEMED 0,008 ml 0,008 ml 36
47 Material und Methoden Abbildung 2-2: PageRuler.. Prestained Protein Ladder SM Western Blot Im Anschluss an die SDS-Gelelektrophorese wurde zum Nachweis des gewünschten Proteins ein Western Blot durchgeführt. Die Western Blots in dieser Arbeit wurden mit dem Semi-dry Verfahren durchgeführt. Das Whatmanpapier wurde vor dem Zusammenbau des Blotes in Transferpuffer getränkt. Zur Äquilibrierung der PVDF-Membran wurde diese erst in Methanol und anschließend in Transferpuffer inkubiert. Der Zusammenbau des Western Blots erfolgte in der Reihenfolge: Whatmanpapier (3x) PVDF-Membran SDS-Gel Whatmanpapier (3x) Der Western Blot wurde bei einer konstanten Stromstärke von 75 ma/gel für eine Stunde durchgeführt. Danach wurde die Membran für eine Stunde oder über Nacht im Kühlschrank in TBST mit 0,5 % Magermilchpuffer geblockt, um unspezifische Proteinbindungen zu vermeiden. Nach Waschen für 5 min in TBST wurde die Membran im entsprechenden Erstantikörper inkubiert. Danach wurde die Membran dreimal in TBST gewaschen (10 min) und eine Stunde im Zweitantiköper inkubiert. Nach der Inkubation im Zweitantikörper wurde die Membran viermal in TBST gewaschen (10 min), gefolgt von einem Waschschritt in AP-Puffer für 10 min. Die Färbung der Membran erfolgte in 10 ml AP-Puffer durch Zugabe von 25 µl NBT und 25 µl BCIP. Nach dem Auftreten der Farbreaktion wurde die Färbung in Wasser gestoppt und die Membran auf Filterpapier getrocknet. 37
48 Material und Methoden Transferpuffer: TBST CAPSO (ph 11,2) 20 mm TRIS/HCl (ph 8,0) 10 mm Methanol 20 % (v/v) NaCl 150 mm Tween 200,05 % (v/v) AP-Puffer: TRIS/HCl (ph 9,5) NaCl MgCl mm 100 mm 5 mm BCIP: NBT: 25 mg/ml in H 2 O 25 mg/ml in 70 % N,N-Dimethylforamid 2.6 Bestimmung der Zellparameter und biophysikalische Methoden Optische Dichte und Absorptionsspektren von Zellen Die optische Dichte von Zellkulturen wurde mit dem Beckmann-DU7400-Photometer gemessen. Die Messung wurde mit 0,5 bis 1 ml der Zellkultur in Küvetten durchgeführt. Die Bestimmung der optischen Dichte erfolgte bei 750 nm, da hier die in der Zelle enthaltenen Pigmente nahezu nicht mehr absorbieren. Parallel dazu wurden auch die Absorptionen bei 680, 625 (Phycobilisomen), 470 (Carotinoide) und 440 nm (Chlorophyll) bestimmt. Überstieg die Absorption der Probe den Wert von Eins, wurde eine Verdünnung der Probe gemessen. Die Absorptionsspektren von Zellen wurden im UV-2450-Photometer (Shimadzu) gemessen. Um den Effekt der Zellstreuung auf die Absorption der Probe zu vermeiden, wurde hier die Absorption bei 750 nm von den gemessenen Absorptionen abgezogen. Die Absorptionsmessung erfolgte im Bereich von 350 bis 750 nm Chlorophyllbestimmung nach Porra et al [101] Zur Bestimmung des Chlorophyllgehalts einer Zellkultur wurde je nach optischer Dichte 200 bis 500 µl Kulturvolumen sedimentiert (5 min, g). Nach Zugabe von 1000 µl Methanol wurden die Zellen für 15 min im Ultraschallbad aufgeschlossen und das Chlorophyll herausgelöst. Anschließend wurden die Zelltrümmer sedimentiert (5 min, g) und der Überstand zur Bestimmung der Chlorophyllkonzentration in eine Messküvette dekantiert. Die Berechnung des Chlorophyllgehalts erfolgte entsprechend Gleichung A nach Porra et al. [101]: 38
49 Material und Methoden A: [ ] ( ) ( ) Bestimmung der Zellzahl Die Zellzahl einer Kultur wurde mit Hilfe des Z2 Coulter particle count und size analyzer der Firma Beckman Coulter bestimmt. Hierbei wird die Zellzahl an Hand der Änderung der elektrischen Impedanz zwischen zwei Elektroden gemessen. Dazu wurden in Abhängigkeit von der OD 750nm 10, 20 oder 30 µl einer Zellkultur in 10 ml Isoton II Lösung verdünnt und in den counter gegeben. Bei der Messung wird zwischen den Elektroden eine Spannung aufgebaut, die so genannte sensing zone. Passieren Zellen diese Zone, ändern sie die Spannung. An Hand des dabei gemessenen elektrischen Signals können die Zellzahl und die Zellgröße bestimmt werden. Die Zellzahl ergab sich danach aus Gleichung B: B: [ ] ( ) Isoton II Lösung: NaCl Na 2 HPO 4 KCl NaH 2 PO 4 Na 2 -EDTA 2 H 2 O NaF 135,69 mm 13,74 mm 5,36 mm 1,58 mm 1,02 mm 7,14 mm mit A.bidest auf 1000 ml auffüllen Tieftemperatur-Fluoreszenzemissionsmessungen bei 77 K Tieftemperatur-Fluoreszenzspektroskopie wurde durchgeführt, um das PS2 zu PS1 Verhältnis in den Zellen zu bestimmen und Informationen über Menge und Position der PBS zu erhalten. Tieftemperatur-Fluoreszenzmessungen wurden mit einem Amincon Bowman Fluoreszenzspektrometer bei einer Bandbreite der Monochromatoren von 4 nm durchgeführt. Für Messungen wurden die Kulturen durch Zentrifugation auf einen Chlorophyllgehalt (s ) von 5 µg/ml konzentriert und in spezielle Glasröhrchen (ᴓ 0,4 mm) überführt. Vor 39
50 Material und Methoden der Messung wurden die Kulturen für mindestens 15 min im Dunkeln inkubiert, um zu gewährleisten, dass die PBS ihre Anregungsenergie an das PS1 übertragen ( state 2 ). Für die Bestimmung des PS2/ PS1 Verhältnisses wurde die Fluoreszenz von Chlorophyll a bei 430 nm angeregt. Nach dreimaligem Messen der Fluoreszenz im Bereich von 650 bis 750 nm (490 bis 750 nm mit Fluoresceine) wurden die Spektren gemittelt und um die Auswirkungen von Messfehlern bei höheren Wellenlängen korrigiert. Zur Bestimmung des PS2/PS1 Verhältnisses wurden die Spektren entweder auf das PS1 Fluoreszenzmaximum bei 725 nm oder auf das Maximum von vor der Messung hinzugegebenem Fluoresceine (5 µm) bei 505 nm normiert. Zur Messung der PBS-Fluoreszenz wurden die Proben bei 580 nm angeregt. Die Aufzeichnung des Emissionsspektrums erfolgte in einem Bereich von 630 bis 750 nm. Zur Untersuchung der state transitions wurden Proben nach der Dunkelinkubation für 10 min in langwelligem Rotlicht ( 710 nm) inkubiert, um den state 2 zu induzieren Acridin Gelb Fluoreszenzmessungen nach Teuber et al [102] Acridine Gelb (AY) Fluoreszenzmessungen wurden durchgeführt, um eine Änderung des ph- Gradienten an der Thylakoidmembran als Folge von Belichtung zu untersuchen. AY ist ein Fluoreszenzfarbstoff, dessen Fluoreszenzeigenschaften von dem ph des ihn umgebenden Mediums beeinflusst wird. Dabei steigt seine Fluoreszenz mit steigendem ph- Wert an. AY akkumuliert im Cytosol von Synechocystis und hat, wenn das umgebende Medium durch Zugabe eines Puffers einen konstanten ph hat, eine stabile ph-abhängige Dunkelfluoreszenz. Nach Anregung der Photosynthese findet ein Protonentransport in das Thylakoidlumen statt. Dies führt zu einem Anstieg des ph-wertes im Cytosol, verbunden mit einem Anstieg der AY Fluoreszenz. Da die Fluoreszenzstärke von AY nicht nur vom ph-wert, sondern auch von der Menge des Farbstoffes selber abhängt, welche durch Anlagerungen an Membran und Dimerisierungseffekten variieren kann, ist dies keine absolute Messung, die es erlaubt einen exakten ph-wert in der Zelle zu bestimmen. Zur Messung der AY Fluoreszenz wurde das Fluorimeter DUAL-PAM 100 (Walz) mit dem dazugehörigen Emitter und Detektor verwendet, welche zur Messung der Fluoreszenz in einem 90 Winkel angebracht wurden. 40
51 Material und Methoden Zur Minimierung des Signal zu Rauschen Verhältnisses wurden die zu messenden Kulturen auf einen Chlorophyllgehalt (s ) von 15 µg/ml eingestellt. Zu einem Milliliter der Kulturen wurden vor der Messung 5 µm AY und 25 µm Tricin-Puffer (ph 8,0) gegeben. Anschließend erfolgte eine Dunkelinkubation von 10 min, um zu gewährleisten, dass AY vollständig in die Zellen aufgenommen wurde und sich der ph-gradient an der Thylakoidmembran auf Dunkelniveau eingestellt hat. Für die Messung wurde zuerst die AY Fluoreszenz für 1 min im Dunkeln gemessen, anschließend wurde die Photosynthese für 5 min mit aktinischem Licht der Intensität von 225 µe angeregt, um den Aufbau des ph-gradienten zu verfolgen Messung der photosynthetischen Sauerstoffentwicklung und der respiratorischen Sauerstoffaufnahme Das Messverfahren der FIBOX beruht auf dem Prinzip der dynamischen Lumineszenzauslöschung. Dabei wird ein Indikatormolekül nach Kollision mit einem Sauerstoffmolekül strahlungslos in den Grundzustand versetzt. Die Aufnahme und Wandlung der Signale der Sauerstoffelektrode erfolgte über die FIBOX-Software. In der Dunkelphase, zu Beginn der Messung, wird eine Sauerstoffabnahme gemessen, welche der Rate der Respiration entspricht. Nach Einschalten der Belichtung wird eine durch die photosynthetische Wasserspaltung bedingte Sauerstoffzunahme beobachtet. Mit der Software Origin wurde die jeweilige Steigung berechnet, welche der Sauerstoffentwicklung, bzw. Abnahme in µmol/l entspricht. Aus der Differenz von Sauerstoffaufnahme und Entwicklung kann die Nettophotosyntheserate bestimmt werden. Zur Bestimmung von ETR wurde die Sauerstoffmessung parallel zu der im Kapitel beschriebenen Fluoreszenzmessungen durchgeführt, wobei Belichtungszeiten und Längen, sowie die Probenvorbereitungen dem dort beschriebenen Protokoll entsprechen. Zur Berechnung von Elektronentransportraten wurde die Nettosauerstoffentwicklung für jede Lichtintensität ermittelt. Nach Gleichung C war es nun möglich für jede Lichtintensität eine ETR zu bestimmen. Die so ermittelten ETR wurden nach der Methode von Eilers und Peeters (s )) gefittet. C: [ ] ( ) 41
52 Material und Methoden Messung der Chlorophyll a-fluoreszenz Durch Absorption eines Photons erreichen Chlorophyllmoleküle einen angeregten elektronischen Zustand. Es gibt für sie vier verschiedene Möglichkeiten wieder in ihren Grundzustand zu gelangen. Die aufgenommene Energie kann elektrochemisch durch Abgabe eines Elektrons in die Elektronentransportkette abgebaut werden. Eine weitere Möglichkeit ist die Abgabe der aufgenommenen Energie in Form von Wärme. Zudem ist es für das Chlorophyllmolekül möglich die aufgenommene Energie an ein zweites Pigment weiterzugeben. Die letzte Möglichkeit ist die Abgabe der Energie in Form von Fluoreszenz [98]. Alle diese Wege stehen in direkter Konkurrenz zueinander. Eine Änderung in einem Weg beeinflusst somit auch alle anderen Wege. Diese Tatsache ermöglicht es an Hand der Messung der Chlorophyllfluoreszenz, auch wenn diese nur einen geringen Anteil von 0,6 bis 3 % hat, Aussagen über die Effizienz der Photosynthese und damit den Status der Zelle zu treffen. Um Aussagen über die photosynthetischen Eigenschaften der gemessenen Kulturen zu treffen, wurden die in der folgenden Tabelle aufgelisteten, gängigen Fluoreszenzparameter bestimmt. Parameter F 0 F M Beschreibung Die minimale Grundfluoreszenz einer dunkeladaptierten Probe. Hier gilt: Alle Reaktionszentren sind offen, das photochemische Auslöschen der Fluoreszenz ist maximal. Die maximale Fluoreszenz einer dunkeladaptierten Probe, angeregt durch einen sättigenden Lichtblitz (ST). Hier gilt: Alle Reaktionszentren sind geschlossen, die Photochemie ist minimal, die Fluoreszenz ist maximal. F M` Maximale Fluoreszenz einer belichteten Probe, vergleichbar mit F M. F Steady state Fluoreszenz. Aktuelle Fluoreszenz einer belichteten Probe, ohne Anregung der maximalen Fluoreszenz durch ST. Die vorgestellten Fluoreszenzparameter wurden mit der gängigen Messroutine der Dual-PAM 100 bestimmt Messung von Photoinhibition und Regeneration Zur Bestimmung der Photoinhibition wurden die Proben auf einen Chlorophyllgehalt von 10 µg/ml (s ) eingestellt und mit 50 µg/ml Chloramphenicol versetzt, um die Proteinbiosynthese zu inhibieren. Nach einer Dunkelinkubation für 10 min wurden F M und F 0 42
53 Material und Methoden der Proben bestimmt und nach Gleichung D die maximale photosynthetische Aktivität (PS2 max ) berechnet. D: ( ) Anschließend wurden die Proben für 25 min mit rotem Licht mit einer Intensität von 1500 µe belichtet. Alle 5 min erfolgte eine erneute Bestimmung von PS2 max zur Verfolgung der Photoinhibition. Anschließend wurden die Zellen zur Entfernung des Chloramphenicols zweimal mit frischem Medium gewaschen und dann für 80 min mit rotem Licht einer Intensität von 50 µe belichtet. In definierten Zeitabständen erfolgte eine erneute Bestimmung von PS2 max, um die Wiederherstellung der photosynthetischen Aktivität zu verfolgen Messung der photosynthetischen Aktivität zur Bestimmung von Elektronentransportraten Als Grundlage für die Berechnung von Elektronentransportraten an Hand der Chlorophyllfluoreszenz wurde die photosynthetische Aktivität (YII) von Proben bei verschiedenen Belichtungsstufen gemessen. Die Bestimmung von YII entspricht der Bestimmung von PS2 max (s ), mit dem Unterschied, dass YII in belichteten Proben bestimmt wird. YII ergibt sich danach aus Gleichung E. E: ( ) Die Messung erfolgte hierbei nach einer voreingestellten Messroutine im Programm des Fluorimeters Dual-PAM 100. Zur Berechnung von Elektronentransportraten wurden Lichtsättigungskurven, so genannte PI-Kurven gemessen. Erfolgten ausschließlich Fluoreszenzmessungen, wurde 1 ml einer Kultur mit einem Chlorophyllgehalt von 10 µg/ml vorbereitet und in die Messküvette gegeben. Erfolgte parallel zur Fluoreszenzmessung auch eine Bestimmung der Sauerstoffentwicklung, wurden 2 ml Kultur vorbereitet und mit der Kultur auch der Sensor der FIBOX (s ) in die Küvette gegeben. Die Messung der Sauerstoffentwicklung wurde sofort gestartet, während für die Fluoreszenzmessung die Proben für 10 min im Dunkeln inkubiert wurden. Nach Start der Fluoreszenzmessung erfolgte eine Bestimmung von PS2 max, gefolgt von der Bestimmung von YII im Anschluss an die Belichtung (0 bis 2000 µe). Für reine Fluoreszenzmessungen hatten die Lichtintensitäten eine Dauer von 30 s, wurde parallel die Sauerstoffentwicklung gemessen, wurde die Dauer auf 1 min erhöht. An Hand der gemessenen 43
54 Material und Methoden Fluoreszenzparameter (YII) und photosynthetic activ radiation (PAR) konnte der Elektronentransport mit folgender Gleichung berechnet werden. F: [ ] Alternative Berechnungen des Elektronentransportes sind im Ergebnissteil beschrieben. Alle ermittelten ETRs wurden nach der Methode von Eilers und Peeters (s ) angenähert P700 Absorptionsänderungsmessung Die Messung der P700 Absorptionsänderung erfolgte mit dem Fluorimeter Dual-PAM 100 (Walz). Das Chlorophyll im Reaktionszentrum von PS1 ist in seiner oxidierten Form im Vergleich zum Reaktionszentrum von PS2 relativ stabil und zeigt eine Absorption im Wellenlängenbereich von 800 bis 840 nm. Dies ermöglicht es die Oxidation und die Reduktion des PS1-Reaktionszentrums an Hand der Änderung der Absorption zu verfolgen. Zu diesem Zweck kann mit der Dual-PAM 100 routinemäßig die Differenz der Absorption von pulsmoduliertem Licht der Wellenlängen 830 und 875 nm gemessen werden. Hierbei dient die Absorption bei 830 nm zur Messung der Änderung der PS1 Oxidation, während die Absorption bei 875 nm zur Bestimmung der generellen Zellabsorption dient. Die Messung der P700 Absorptionsänderung kann dazu genutzt werden die Raten des linearen und des zyklischen, photosynthetischen Elektronentransportes zu messen. Zu diesem Zweck wurden die verwendeten Kulturen auf einen Chlorophyllgehalt von 10 µg/ml (s ) eingestellt und für 10 min dunkeladaptiert. Somit wurde gewährleistet, dass alle Reaktionszentren in reduzierter Form vorliegen. Anschließend wurden durch eine 50 ms lange Belichtung mit 2000 µe alle Reaktionszentren oxidiert. Nach Beendigung des Lichtblitzes erfolgte die erneute Reduktion der PS1 Reaktionszentren durch die in der Elektronentransportkette vorhandenen Elektronen. Die Geschwindigkeit dieser Rück- Reduktion der PS1-Reaktionszentren folgt dabei einer Reaktion pseudo erster Ordnung und kann über eine Exponentialgleichung kinetisch bestimmt werden. Zur Bestimmung der Raten des linearen und zyklischen Elektronentransportes wurde zunächst die Geschwindigkeitskonstante der Rück-Reduktion gemessen und als 100 %-Wert normiert. Anschließend wurde die Messung mit Zugabe von jeweils 10 µm DCMU und 500 µm Methyl Viologen (MV) wiederholt. DCMU inhibiert den Elektronentransport am PS2. Die so bestimmte Geschwindigkeitskonstante kann als Maß der Rück-Reduktion ohne 44
55 Material und Methoden linearen Elektronentransport aufgefasst werden. Somit entspricht sie im Vergleich zum 100 %-Wert dem prozentualen Anteil des zyklischen Elektronentransportes. MV ist ein starker PS1-Elektronenakzeptor, so dass vom PS1 keine Elektronen auf Ferredoxin übertragen und somit in die zyklischen Elektronentransportwege eingebracht werden. Der prozentuale Wert der so ermittelten Geschwindigkeitskonstanten kann also als Maß für die Raten des linearen photosynthetischen Elektronentransports angenommen werden Bestimmung der PS1-Elektronentransportraten nach Klughammer et al. [104] Ähnlich zu der unter Punkt beschriebenen Methode zur Bestimmung der photosynthetischen Aktivität mittels Chlorophyllfluoreszenz können auch aus den P700- Absorptionänderungen Rückschlüsse auf die Elektronentransportraten im PS1 gezogen werden. Aus dem gemessenen Signal, wie es in Abbildung 2-3 gezeigt wird, können dabei Parameter abgelesenen werden, die es erlauben, den Oxidations- und Reduktionszustand des PS1 zu beschreiben. Abbildung 2-3: Bestimmung der Energieumwandlung im PS1 mit einem sättigenden Lichtblitz. P m: maximales P700 Signal; P 0: minimales P700 Signal nach Belichtung mit langwelligem Rotlicht; P: Zwischenzeitliches Signal bei Belichtung; P m`: Maximales Signal bei Belichtung; a: geschlossene P Zentren (donorseitenlimitiert); b: offene P700 Zentren; c: Geschlossene P700 - Zentren (akzeptorseitenlimitiert) [104]. Die in Abbildung 2-3 gezeigten Parameter a, b und c werden wie folgt berechnet: a = P-P 0 ; b = P m -P ; c = P m -P m Aus diesen Parametern können Quantenausbeuten berechnet werden, die denen in der Chlorophyllfluoreszenz Verwendeten sehr ähnlich sind. 45
56 Material und Methoden ( ) ( ) ( ) ( ) ( ) ( ) Mit Y(I): Quantenausbeute von PS1; Y(ND): Quantenausbeute der nicht photochemischen Energieabgabe (Donorseitenlimitierung); Y(NA): Quantenausbeute der nicht photochemischen Energieabgabe (Akzeptorseitenlimitierung). Für die Messungen wurden die Kulturen auf einen Chlorophyllgehalt von 10 µg/ml eingestellt und die Anregung erfolgte mit rotem Licht der Intensitäten von 0 bis 2000 µe Berechnung der Elektronentransportraten bei verschiedenen Lichtintensitäten nach Eilers und Peeters [105] Das von Eilers und Peeters entwickelte, dynamische Modell zur Beschreibung zwischen Lichtintensität und Photosyntheseraten erlaubt es Vorhersagen zur photosynthetischen Aktivität bei jeder Lichtintensität zu treffen. Dazu wurden die gemessenen Lichtsättigungskurven gegen die tatsächlich verwendete photosynthetisch aktive Strahlung (PAR) aufgetragen und die Parameter a, b und c bestimmt. Mit P Max: Maximale photosynthetische Produktivität; I Max: Lichtintensität bei P Max; s: Anfangssteigung der Lichtsättigungskurve. Nach dem Modell von Eilers und Peeters wurden die Parameter in die Gleichung G eingesetzt. G: ( ) Mit LI: Lichtintensität 46
57 Material und Methoden Messung des photosynthetischen Gasaustausches mittels Gas- Massenspektrometrie (MIMS) Die Messungen wurden mit dem Massenspektrometer Prima δb Process MS der Firma Thermo Scientific durchgeführt. Die Trennung in diesem Massenspektrometer erfolgt durch Anlegen eines magnetischen Feldes, das in diesem Fall in einem Winkel von ca. 80 gebogen ist. Die Trennung der, in das Massenspektrometer eingeleiteten Gasionen Abbildung 2-4: Aufbau der Messkammer für die MIMS-Messung. Bei Belichtung wird durch die Spaltung von H 2O 16 O 2 freigesetzt und durch die Mehler-Reaktion 18 O 2 verbraucht. erfolgte dabei nach unterschiedlichen Masse- und Ladungs-Verhältnissen sowie, nach dadurch bedingten unterschiedlich Flugzeiten im Spektrometer. An das Massenspektrometer war die in der Abbildung 2-4 gezeigte Messkammer angeschlossen. Diese konnte ein Kulturvolumen von 1,5 ml aufnehmen. An ihrem Boden befindet sich eine semipermeable Membran, welche die Trennung von Gas und Flüssigkeit erlaubte. Eine an die Messkammer angeschlossene Vakuumpumpe saugte das freigesetzte Gas durch die Membran in das angeschlossene MS. Zur Gewährleistung eines idealen Signal/Rauschen Verhältnisses, wurden die Proben für die Messung auf einen Chlorophyllgehalt von 10 µg/ml eingestellt. Die Zellen wurden dazu in Phosphatpuffer (ph 7,2) aufgenommen. So liegt das, sich in der Lösung befindende, CO 2 nicht als Bicarbonat gebunden vor und es ist möglich Aufnahme und Verbrauch von CO 2 zu messen. Für die Messung des Gasaustausches während der Photosynthese wurden vor der Messung 5 µm des Kohlenstoffisotops 13 C 2 in Form von Bicarbonat und 100 µl des Sauerstoffisotops 18 O zu den Proben gegeben. Für die Messung wurden die Proben nach Zugabe des O 18 so lange inkubiert, bis sich die Menge beider Isotope ( 16 O 2 (Luft) und 18 O 2 ) angeglichen hatte. Anschließend wurde die Messung gestartet. Zunächst erfolgte eine Dunkelinkubation für 5 min, um die Respirationsraten zu bestimmen. Danach wurden die Proben jeweils für 5 min bei unterschiedlichen Intensitäten belichtet. Abschließend erfolgte 47
58 Material und Methoden erneut eine Dunkelinkubation für 5 min. Für die Belichtung der Proben wurde weißes Licht mit den Intensitäten 450 und 1500 µe verwendet. 2.7 Kultivierung von Synechocystis im Flachbettbioreaktor Die Kultivierung im Fermenter wurde mit dem Programm Labview überwacht. Labview wurde ebenfalls dazu benutzt, um alle im Fermenter erhaltenen Messdaten zu speichern und zu verwalten. Das dazu notwendige Labview-Steuerungsprogramm wurde von Jong-Hee Kwon geschrieben Sterilisation Da der Fermenter auf Grund seiner Größe und seiner Materialbeschaffenheit nicht in einem Autoklav über Dampfdruck sterilisiert werden kann, wurden die im Folgenden beschriebenen Methoden der chemischen Sterilisation angewandt. Alle verwendeten Schläuche und Sterilfilter wurden vor dem Einbau in den Fermenter bei 120 C für 30 min autoklaviert. Die verwendeten Schläuche wurden anschließend zusätzlich chemisch sterilisiert. Eine Methode der chemischen Sterilisation ist die Verwendung des Sterilisationsmittels Virkon. Die desinfizierende Wirkung von Virkon beruht auf den Eigenschaften von Peroxiden, organischen Säuren und zwitterionischen Detergenzien. Als Sterilisationslösung wurden 7 l einer 1 % (w/v) Virkonlösung angesetzt und in den Fermenter gegeben. Dabei war es wichtig, dass die Desinfektionslösung ebenfalls in alle angeschlossenen Schläuche, welche am Ende mit Klemmen verschlossen waren, eingefüllt wurde. Nach der Zugabe von Virkon wurde der Fermenter verschlossen und zur Desinfektion ein bis drei Tage inkubiert. Zur Beseitigung aller Rückstände der Virkonlösung wurde der Fermenter anschließend dreimal mit autoklaviertem VE-Wasser gespült. Dazu wurde das VE-Wasser mit einer Schlauchpumpe in den Fermenter gepumpt, bis dieser zu zwei Drittel gefüllt war. Durch Schütteln des Fermenters wurden sich in Anschlüssen befindende Reste von Virkon entfernt und der Fermenter anschließend vollständig gefüllt. Beim zweiten Spülen wurden zusätzlich die Schläuche gespült. Anschließend wurde der Fermenter mit Medium gefüllt und konnte angeimpft werden. Das verwendete VE-Wasser und die Medien wurden zuvor in 20 l Plastikbehältern autoklaviert. Der Luftausgleich erfolgte über angeschlossene Sterilfilter. Virkon wurde ebenfalls genutzt, um sich im Fermenter befindende, gentechnisch veränderte Organismen zu inaktivieren. Dazu wurde Virkon direkt in Pulverform in den Fermenter gegeben und dieser ein bis drei Stunden inkubiert. Alternativ zur Desinfektion mit Virkon 48
59 Spannung [V] ph Material und Methoden wurde der Fermenter mit Perchloressigsäure (PES) desinfiziert [106]. Zur Desinfektion wurden der Fermenter und alle angeschlossenen Schläuche vollständig mit einer 10 % (v/v) PES-Lösung gefüllt. Danach wurde der Fermenter mindestens 24 h mit PES inkubiert. Zur Beseitigung von PES wurde der Fermenter ebenfalls zweimal mit autoklaviertem VE-Wasser gespült. Anschließend wurde der Fermenter mit Medium gefüllt und nochmals für 12 h inkubiert, um alle sich noch im Fermenter befindenden Reste von PES zu beseitigen Messungen und Regulierung des ph-wertes Der ph-wert des Mediums im Fermenter wurde ständig über eine ph-elektrode überwacht und mit dem Programm Labview online aufgezeichnet. Labview wurde ebenfalls genutzt, um den ph-wert des Mediums konstant auf 7 zu halten. A B y = 2,9907x - 1, ,1 0,2 0,3 0,4 LabView-Zeit Spannung [V] Abbildung 2-5: A: Aufzeichnung der Spannung mit der ph-elektrode in Abhängigkeit von verschiedenen ph- Werten. B: Bestimmung der Formel zur Umrechnung von Spannung in den ph-wert. Mit Hilfe der ph-elektrode war es möglich, eine ph-wertabhängige Spannung zu messen und an Labview weiterzugeben. Um diese Spannung einem entsprechenden ph-wert zuzuordnen, wurde die Elektrode vor jedem Einbau in den Fermenter geeicht. Dazu wurde sie in ph-eichlösungen gehalten und die entsprechende Spannung für 2 min aufgezeichnet (Abbildung 2-5 A). Anschließend wurde die Spannung gegen den entsprechenden ph-wert aufgetragen und die Funktion der eingetragenen Ausgleichsgeraden in das Programm Labview eingegeben (Abbildung 2-5 B). Zur Anpassung des ph-wertes des Mediums wurden autoklavierte 0,2 M NaOH- und 0,2 M HCl-Lösungen an den Fermenter angeschlossen. Das Programm Labview, hielt durch Messung des ph-wertes und durch automatische Zufuhr von NaOH und HCl (Steuerung von Schlauchpumpen), den ph-wert konstant. 49
60 LI [µe] Material und Methoden Messung der Trübung und des Zellwachstums Zur Verfolgung des Zellwachstums im Fermenter wurde mit einem Trübungssensor (Trb 8300, Toledo) die Trübung der Kultur online aufgezeichnet. Mit den aufgezeichneten Messdaten war es möglich durch automatische Mediumzufuhr über die Steuerung einer Schlauchpumpe mit LabView eine bestimmte Trübung konstant zu halten. Da die Trübungsmessung durch tote Zellen im Fermenter und durch wechselnde Lichtintensitäten nicht genau ist, wurden zusätzlich offline die optische Dichte (750 und 680 nm) der Kultur (s ) und die Zellzahl (s ) bestimmt Beleuchtung, Begasung und Durchmischung Der verwendete Bioreaktor ist nach dem air lift Prinzip konstruiert. Bei diesem ist ein vom Boden des Fermenters aufsteigender Gasstrom so reguliert, dass er für die Durchmischung der Kultur sorgt. Das einströmende Gas wurde dabei durch sich im Boden des Fermenters befindende Glasfilter geleitet. Eine Kontamination durch die Gaszufuhr wurde durch einen vorgeschalteten Sterilfilter verhindert. Das Gas wurde mit einer konstanten Flussrate von 50 ml/min in den Fermenter geleitet und mit 3 % (v/v) CO 2 angereichert. Der Gasfluss und die Mischung wurden mit einem Gasmischsystem der Firma MKS eingestellt. Die einströmende Luft wurde dafür dem Hausluftsystem entnommen, während das CO 2 aus einer zusätzlich angeschlossenen Gasflasche stammte. Die Beleuchtung der Zellkultur im Flachbettbioreaktor erfolgte mittels selbstgebauter LED- Paneele. Die gekauften LED-Streifen wurden auf Metallsockel montiert, so dass die gesamte Fläche des Bioreaktors beleuchtet werden konnte. Mit Hilfe von Dimmern und eines Spannungsreglers war es möglich, über die ausgegebene Spannung die Lichtstärke der LEDs zu steuern. Dazu musste zuerst eine Eichgerade erstellt werden, um für jede ausgegebene Spannung die entsprechende Lichtstärke zu berechnen. Für die Beleuchtung im Flachbettbioreaktor wurden LEDs mit einem blau-grünen Lichtspektrum (Abbildung 3-23; y = 41,748x - 72,75 R² = 0, Spannung [V] Abbildung 2-6: Eichgerade zur Umrechnung von Spannung in die Lichtintensität [µe]. B) verwendet. 50
61 Material und Methoden Inokulation Der Fermenter wurde mit einer 500 ml Glasflasche, an die nachträglich oben und unten Stutzen angebracht wurden, inokuliert. Der Luftausgleich erfolgte über einen, am oberen Stutzen angebrachten, Sterilfilter. Der zweite Stutzen befand sich kurz über dem Boden der Flasche. An diesen war ein Schlauch zur Probenüberführung angebracht. Die Vorkultur wurde an der Sterilbank in die zuvor autoklavierte Glasflasche überführt. Danach wurde der Schlauch zur Probenüberführung mit Ethanol (98 % (v/v)) desinfiziert und an einer Bunsenbrennerflamme abgeflämmt. Anschließend wurde der Schlauch an der Flamme mit dem Schlauch für die Mediumzufuhr verbunden. Durch den hydrostatischen Druck wurde der Fermenter anschließend inokuliert. Bei neueren Generationen der Fermenter erfolgte die Inokulation mit Hilfe eines autoklavierten Erlenmeyer-Kolbens. In diesen wurde die Kultur ebenfalls an der Sterilbank überführt. Anschließend wurde die Kultur an der Gasflamme über einen sich an der Oberseite des Fermenters befinden Einlass in den Fermenter gegossen. In beiden Fällen wurden zum Animpfen des Fermenters ca. 350 ml einer Kultur mit einer OD 750nm von 4 verwendet Probenentnahme und Messung der Produktivität Für die Probenentnahme aus dem Fermenter wurde beim Aufbau ein Schlauch mit einem Durchmesser von 2 mm mit eingebaut. Mit diesem Schlauch wurde über eine Pipettenspitze (200 µl) eine sterile Einwegspritze (10 ml) verbunden. Der Schlauch wurde während der Kultivierung mit einer Schlauchklemme abgeschlossen, so dass über diesen kein Druckausgleich stattfinden konnte und es nicht zu Kontaminationen kam. Zur Probenentnahme wurde zuerst die Spritze aufgezogen und dann die Schlauchklemme geöffnet, so dass die Probe über den Unterdruck in die Spritze gesogen wurde und es nicht zu einem Rücklauf von nicht steriler Probe aus der Spritze kam. Während der Kultivierung wurde die Rate der photosynthetischen Produktivität online aufgezeichnet. Zu diesem Zweck wurde die O 2 -Produktion im Fermenter über einen BCP-O 2 Sensor und einen vorgeschalteten Gasflussadapter innerhalb des aus dem Fermenters kommenden Gasstroms gemessen. 51
62 Material und Methoden 2.8 Massenspektrometrische Analyse Für die Messungen wurde jeweils eine Gesamtproteinmenge von 5 µg eingesetzt. Die Proben für die Massenspektrometrie wurden auf ein SDS-Gel aufgetragen und in das Trenngel einlaufen gelassen. Nach Coomassie-Färbung wurden die Gelbanden mit den fokussierten Proteine in ca. 1 mm 3 große Stücke geschnitten. Nach Überführung in ein Reaktionsgefäß wurden diese für 20 min in 1000 µl einer 50 mm Ammoniumbicarbonatlösung (ABC) inkubiert. Im nächsten Schritt wurden die Gelstücke bis zur vollständigen Entfärbung in 25 mm ABC in 50 % (v/v) Acetonitril (ACN) gewaschen. Nach einer Inkubation für 5 min in 1000 µl (100 % (v/v)) ACN (100 %) wurden die Gelstücke für 20 min in der SpeedVac getrocknet. Anschließend wurden 50 µl 25 mm Trypsinlösung mit 250 µl 25 mm ABC und 30 µl (100 % (v/v)) Methanol verdünnt. Jeweils 30 µl der Trypsinlösung wurden zu den Gelstücken gegeben. Waren diese nicht vollständig mit Flüssigkeit bedeckt, wurden 25 mm ABC hinzugegeben. Die Ansätze wurden über Nacht bei 37 C inkubiert. Der Überstand des Verdaus wurde in ein zweites Reaktionsgefäß überführt. Die Gelstücke wurden in einer 50 % (v/v) ACN/0,5 % (v/v) Ameisensäure-Lösung aufgenommen und für 15 min im Ultraschallbad inkubiert. Der Überstand wurde mit dem zuvor gesammelten Überstand kombiniert und der Schritt wiederholt. Anschließend wurden die Proben vollständigen in der SpeedVac eingedampft. Die vorbereiteten Proben wurden mit 20 µl Puffer A versetzt und 1 min im Ultraschallbad inkubiert. Anschließend wurden sie im Massenspektrometer (Orbitrap XL, Thermo Scientific) untersucht. Dafür wurden die Peptide zunächst mittels nano HPLC ( High performance liquid chromatography ) auf einer C18 Säule getrennt. Die Elution erfolgte durch einen steigenden Gradienten von Puffer B. Die Peptide wurden dann mittels FT-MS (Fourier Transformation- Massenspektrometrie) analysiert und anschließend durch CID (collision induced dissociation) fragmentiert. Die MS/MS-Spektren wurden zur Analyse aufgezeichnet. Puffer A Puffer B 0, 1 % (v/v) Ameisensäure 80 % (v/v) Acetonitril 0,1% (v/v) Ameisensäure 52
63 Ergebnisse 3 Ergebnisse 3.1 Heterologe Expression von Channelrhodopsin-2 in Synechocystis PCC 6803 Die Expression von Channelrhodopsin-2 (Chop-2) und die Integration in die Thylakoidmembran (TYM) sollten eine Entkopplung des photosynthetischen Elektronentransportes in Synechocystis PCC 6803 ermöglichen. Die Grundlage der Klonierung einer Chop-2-tragenden Synechocystis-Mutante bildete das Plasmid pcdna3,5-chop2 [93]. Auf dem Plasmid ist die, für die Bildung eines Kationenkanals ausreichende, verkürzte Version von Chop-2, bestehend aus den Aminosäuren 2-315, kodiert. Wie in Abbildung 3-1 gezeigt, wurde Chop-2 über die Primer Chop-GFP-for und -rev amplifiziert und nach Restriktion mit Acc65I und XbaI in das Plasmid pmb4 kloniert. In dieses wurde zuvor eine zusätzlich XbaI-Schnittstelle eingefügt. Im fertigen Konstrukt pmb4-chop steht Chop-2 unter der Kontrolle des Promotors PpetF und bildet ein c-terminales Fusionsprotein mit GFP [111]. Durch Transformation in Synechocystis wurde die Mutante Chop-2 erzeugt. Um mögliche Probleme bei der korrekten Faltung von Chop-2 durch die GFP-Fusion zu vermeiden, sah eine alternative Klonierungsstrategie die Erzeugung eines Plasmides (ebenfalls auf der Grundlage von pmb4) mit Chop-2 ohne GFP vor. Das Ausbleiben eines Klonierungserfolges in Escherichia coli (E. coli) deutet auf eine mögliche basale Expression von Chop-2 mit letalen Folgen hin. Die Mutante Chop-2 wurde zur Erlangung eines ausreichenden Kulturvolumens im Flachbettbioreaktor angezogen. Nach Ernte der Kultur erfolgte eine Isolierung von löslichen Proteinen (Cy) und der Gesamtmembran (GM), aus der über einen Sacharosedichtegradienten die TYM von der Zytoplasmamembran (CM) isoliert wurden. 53
64 Ergebnisse Abbildung 3-1: Schematische Darstellung der Klonierung des Plasmids pmb4-chop. Über die Primer Chop-GFPrev und Chop-GFP-for wurde Chop-2 aus dem Plasmid pcdna3.3-chop-2 amplifiziert und die Restriktionsschnittstellen ACC65I und XBAI angefügt. Die Klonierung in das Plasmid pmb4 ergab pmb4-chop-2. 54
65 Ergebnisse Zum Nachweis von Chop-2-GFP wurde 25 µg Protein der isolierten Fraktionen auf ein SDS- Gel aufgetragen. Die Kontrolle der Expression erfolgte an Hand eines GFP-Antikörpers auf dem Western Blot, der in Abbildung 3-2 A dargestellt ist. Das Chop-2-GFP-Fusionsprotein hat eine Größe von ca. 60 kda, ein Nachweis von GFP konnte auf dieser Höhe nicht erfolgen. Der Nachweis von GFP war jedoch, wie in Abbildung 3-2 B gezeigt, in einer Positivkontrolle möglich. Auch auf dieser Höhe konnte in den isolierten Fraktionen kein Nachweis von möglicherweise abgespaltetem GFP erfolgen. Zusätzlich zum Nachweis über einen Western Blot, erfolgte der Nachweis über Fluoreszenzemission bei 77 K, um ausschließen, dass ein Nachweis von GFP durch die Fusion mit Chop-2 verhindert wurde. Die Fluoreszenzspektren der einzelnen Fraktionen sind in der Abbildung 3-3 gezeigt. Die Fluoreszenz von GFP wurde mit UV-Licht bei 395 nm angeregt und hat für GFP ein Maximum bei 510 nm, welches in einem Fusionsprotein aber durch veränderte Proteineigenschaften verschoben sein kann. In den gezeigten Fluoreszenzspektren scheint im Gegensatz zum Western Blot ein Nachweis von GFP möglich zu sein. Im Spektrum der CM (rot) erscheint ein Maximum bei 510 nm mit einer Schulter bei 490 nm. Dieses Maximum konnte auch in der Fraktion der GM und der TYM nachgewiesen werden. In der GM findet sich ein starkes Signal/Rauschen Verhältnis, möglicherweise verursacht durch den geringen Anteil der CM an der GM. In der TYM ist die Fluoreszenz nur schwach zu erkennen und möglicherweise durch eine unvollständige Trennung der Membranen bedingt. Ausgehend von den Fluoreszenzspektren kann ein Einbau von Chop-2-GFP in die CM vermutet werden. Zur Überprüfung dieser Vermutung wurden Proben der CM und TM mittels Massenspektrometrie untersucht (s. 2.8). Trotz erfolgreicher Integration in das Genom von Synechocystis, konnte in keiner der überprüften Proben eine Expression von Chop-2-GFP, GFP oder Chop-2 nachgewiesen werden. Abbildung 3-2: Nachweis der GFP-Expression mittels GFP - Antikörper. Aufgetragen wurde GM, Cy, CM und TYM und eine GFP-Positivkontrolle. A: Proteine der Größe von 60 kda entsprechend dem Channelrhodopsin-GFP-Fusionsproteins. B: Proteine bei 27 kda entsprechend der Größe von GFP. 55
66 Ergebnisse Abbildung 3-3: Fluoreszenznachweis von GFP bei 77 K. Anregung der GFP-Fluoreszenz bei ca. 510 nm erfolgte mit UV-Licht bei 395 nm Untersuchung lichtregulierter Promotoren Als mögliche regulatorischen Elemente der Expression von Chop-2 wurden die lichtregulierten Promotoren PpetF (Ferredoxin), PhilC ( high light inducible polypeptide C ) und PpsbA3 (D1-Untereinheit von PS2 aus Thermosynechococcus elongatus (T. elongatus)) ausgewählt. Hierbei lag ein besonderes Augenmerk auf den Promotoren PhilC und PpsbA3, da diese als mögliche Hochlichtschalter fungieren können, während der Promotor von Ferredoxin schon bei niedrigen Lichtintensitäten aktiv ist [98,107,108]. Als alternative Expressionskontrolle wurden der nickelinduzierbare Promotor PnrsBACD und der kupferinduzierbare Promotor PpetE gewählt [109,110]. In der Abbildung 3-4 ist eine schematischen Darstellung der Klonierungsstrategie in den Vektor piga gezeigt [100]. Die Integration der Promotoren in den promotorlosen Vektor erlaubte die Charakterisierung ihrer Aktivität über die Expression von GFP. Eine homologe Rekombination in eine neutrale Stelle des Genoms von Synechocystis an Position slr0168 erfolgte über Sequenzbereiche des Gens. Als promotortragende und regulatorische Bereiche wurden jeweils, ausgehend vom Start Codon des entsprechenden Gens, 500 bp des upstream -Bereichs für die Klonierung amplifiziert. Die Integration erfolgte über die Restriktionsschnittstellen EcoRV und Acc65I. 56
67 Ergebnisse Abbildung 3-4: Klonierungsschema zur Untersuchung lichtregulierbarer Promotoren. Die verschiedenen Promotorbereiche wurden über PCR amplifiziert und mit einer EcoRV- und einer ACC65I-Restriktionsschnittstelle versehen. Anschließend wurden sie in den promotorlosen Vektor piga vor GFP als Expressionskontrolle kloniert. Durch Transformation in Synechocystis konnten die Mutanten MB1 (PnrsABCD), MB2 (PHilC), MB3 (PpetE), MB4(PpetF) und MB5 (PpsbA3) erzeugt werden Lichtregulierung von PpsbA3 aus T. elongatus in Synechocystis Zur Charakterisierung der Lichtregulierung von PpsbA3 aus T. elongatus in Synechocystis wurde die Synechocystis-Mutante MB5 unter kontinuierlichen Bedingungen in einem Flachbettbiorektor kultiviert. Während des Versuches wurde die Lichtintensität im Fermenter schrittweise um 50 µe erhöht. Die Probenentnahme zur Untersuchung der GFP-Expression erfolgte jeweils nach einer Inkubationszeit von mindestens 4 Stunden. Nach Zellaufbruch und Abbildung 3-5: Western Blot zur Untersuchung der Aktivität von PpsbA3 aus T. elongatus in Synechocystis bei verschiedenen Lichtintensitäten. Der Nachweis erfolgte über die Expression von GFP. Isolierung der löslichen Proteine wurden für jede Probe 25 µg Protein auf ein SDS-Gel 57
68 Ergebnisse aufgetragen. Die Expression von GFP wurde auf einem Western Blot mittels GFP-Antikörper nachgewiesen. Wie in der Abbildung 3-5 zu erkennen, wurde für die Lichtintensitäten von 50 bis 450 µe eine konstante GFP-Expression nachgewiesen. Da hier schon bei geringen Lichtintensitäten eine GFP-Expression nachgewiesen wurde, ist dieser Promotor nicht für eine strikte Hochlichtregulation in Synechocystis geeignet. Jedoch konnte die Funktionalität einer Expressionskontrolle über einen Promotor aus T. elongatus in Synechocystis erfolgreich nachgewiesen werden Lichtregulierung von PhilC Die Kultivierung der Mutante MB2, sowie Probenvorbereitung und Nachweis von GFP erfolgten wie zuvor für MB5 beschrieben (s ). Zusätzlich wurde eine Dunkelprobe nach einer Inkubationszeit von zwölf Stunden genommen, um eine vollständigen GFP- Abbau zu ermöglichen. Der zum Nachweis der GFP-Expression durchgeführte Western Blot ist in der Abbildung 3-6 abgebildeten. Auf dem Western Blot ist in allen Spuren eine Expression von GFP zu erkennen, so dass von einer dauerhaften, basalen Aktivität des hilc-promotors bei den getesteten Lichtintensitäten auszugehen ist. Eine genauere Betrachtung der Ergebnisse zeigt eine gesteigerte Expression von GFP bei 0, 50, 500, 550 und 600 µe. Bei den Lichtintensitäten zwischen 50 und 500 µe ist ein Rückgang von GFP festzustellen. Die Ergebnisse belegen eine erfolgreiche, lichtabhängige Regulation der Aktivität von PhilC. Eine strikte Hochlichtregulation bedarf jedoch weiterer Modifikationen der Promtorregulation Zusammenfassung Abbildung 3-6: Überprüfung der Aktivität von PhilC bei den Lichtintensitäten von µe. In Spur 1 ist jeweils GFP als Positivkontrolle aufgetragen. Nachweis erfolgte über Antikörper gegen GFP. Die Integration von Chop-2 in das Genom von Synechocystis war erfolgreich, jedoch konnte keine Expression des Fusionsproteins Chop-2-GFP nachgewiesen werden. 58
69 Ergebnisse Eine Expressionskontrolle an einer neutralen Stelle des Genoms von Synechocystis über die Promotoren PpsbA3 und PhilC war erfolgreich. Beide Promotoren sind jedoch ihn ihrer ursprünglichen Form nicht als lichtregulatorisches Element geeignet. 3.2 Entkopplung des photosynthetischen Elektronentransportes und die Erhöhung des PS2/PS1 Verhältnisses in Synechocystis Die im Folgenden beschriebenen und erzeugten Synechocystis-Mutanten wurden basierend auf zwei grundlegenden Annahmen ausgewählt: 1. Eine Verkleinerung der Phycobilisomen erhöht das PS2/PS1 Verhältnis 2. Eine Entkopplung steigert die Raten des PET Die Charakterisierung der Synechocystis-Mutanten wurde mit Kulturen durchgeführt, die mit dem am Lehrstuhl gängigen Anzuchtlicht angezogen wurden. Wie in der Abbildung 3-7 zu sehen, ist das Spektrum der Anzuchtlampe in den grünroten Spektralbereich verschoben. Abbildung 3-7: Spektrum des am LS verwendeten Anzuchtlichtes Erzeugung der verwendeten Synechocystis-Mutanten Neben dem Synechocystis Wild Typ (WT) wurden in dieser Arbeit drei Mutanten verwendet, die von anderen Arbeitsgruppen erstellt wurden. In Zwei davon, Olive und PAL, finden sich verkleinerte PBS. In Olive kam es durch ungerichtete Mutation zu einem Verlust von Phycocyanin (PC) [34]. In der PAL wurden durch gerichtete Mutation die Gene für apcab und apce inaktiviert, was zu einem vollständigen Verlust der PBS führt [37]. Die dritte Mutante ( CԐ) wurde in der Arbeitsgruppe von Toru Hisabori erzeugt und soll zu einer Entkopplung des PET führen. Wie in der Abbildung 3-8 gezeigt, wurde an Position 259 des Gens (slr1330) für die Epsilon-Untereinheit (Ԑ-UE) der ATP-Synthase ein stop codon eingefügt. Dies führt zu einer Inaktivierung des α-helicalen C-Terminus der Ԑ-UE [93]. Zur Kombination von Entkopplung und verkleinerter PBS wurde das von T. Hisabori zur Verfügung gestellte Plasmid für die ATP-Synthase-Mutation in die Olive transformiert. Die erfolgreiche Transformation ergab die Doppelmutante Olive- CԐ (DM). Zur Transformation in 59
70 Ergebnisse Abbildung 3-8: Klonierung der Mutante CԐ. An Position 259 bp des Gens für die Ԑ-UE der ATP-Synthase (slr1330) wurde ein stop codon, zusammen mit einer Spectinomycin-Resistenz, eingefügt. Abbildung nach Imashimizu et al. [93]. die PAL wurde die Spectinomycin- gegen eine Kanamycin-Resistenz ausgetauscht. Die Transformation in die PAL ergab jedoch keine Kolonien. Alternativ wurde das Plasmid apcab konstruiert, um in der Mutante DM die beiden für die Allophycocyanin-Untereinheit (APC) der PBS kodierenden Gene (apca (slr1986); apcb (slr2067)) zu inaktivieren. Die erfolgreiche Transformation diese Plasmides in die DM ergab jedoch keine vollständig segregierten Kulturen. Es ist somit davon auszugehen, dass die Inaktivierung von CԐ in der PAL letale Folgen hat. Zur Erhöhung des PS2/PS1-Verhältnis wurde das Plasmid ycf4 erzeugt. Ycf4 ist an der Assemblierung von PS1 beteiligt und eine Inaktivierung führt zu einem erhöhten PS2/PS1- Verhältnis [73,74,112]. Die erfolgreiche Transformation in die DM ergab die Dreifachmutante Olive- CԐ- Ycf4 (TM) Veränderung des Pigmentverhältnisses Wie in der Abbildung 3-9 gezeigt, kommt es durch die verkleinerten PBS zu einer deutlichen Änderung des Absorptionsspektrums. Im WT ist das für Synechocystis typische Absorptionsspektrum mit zwei Maxima für Chl a bei 440 und 680 nm und den zwei Maxima der PBS- und der Carotinoid-Absorption bei 630 und 490 nm zu sehen. Die Kombination dieser Pigmente ergibt die in Abbildung 3-10 gezeigte blaugrüne Färbung des WT. Der Vergleich der Absorptionen von Olive und PAL mit dem WT zeigt zwei wesentliche Änderungen. Zum einen ist das Maximum der Carotinoidabsorption bei 490 nm in beiden Mutanten deutlich erhöht, zum andern ist eine Verringerung des PBS-Maximums bei 630 nm zu beobachten. In der Olive, welche noch APC enthält, fehlt das Maximum bei 630 nm. In der PAL, in der sich keine PBS mehr finden, ist ein zusätzliches Absorptionsminimum bei 650 nm zu erkennen. Wie in Abbildung 3-10 gezeigt, ändert sich dadurch auch der farbliche Phänotyp. Die Olive ist olivgrün, die PAL orange bis gelblich gefärbt. Dies ist in den PBS- Mutanten nicht ausschließlich durch den Anstieg der Carotinoidabsorption, sondern ebenfalls durch die in Tabelle 3-1 gezeigte verringerte Chl a Konzentration zu erklären. Die Chl a 60
71 Ergebnisse Konzentration des WT (1, µg/zelle) ist in der Olive auf 1, µg/zelle gesunken. In der PAL ist eine deutliche Verringerung auf 1, µg/zelle zu beobachten ist. Abbildung 3-9: Absorptionsspektren der untersuchten Synechocystis-Mutanten CԐ, DM, TM, Olive, PAL und dem WT (n = 5). Die Mutation der ATP-Synthase hat keinen Einfluss auf das Pigmentverhältnis der Zelle. Sowohl das Absorptionsspektrum (Abbildung 3-9) als auch der farbliche Phänotyp (Abbildung 3-10) von CԐ und DM sind vergleichbar mit denen von WT und Olive. Entsprechend kommt es zu keiner signifikanten Änderung der zellulären Pigmentkonzentrationen und Zellgrößen (Tabelle 3-1). Die Inaktivierung von Ycf4 resultiert nicht in einem veränderten Absorptionsspektrum, zeigt aber eine deutliche Auswirkung auf den farblichen Phänotyp. Dieser zeigt wieder die blaugrüne Färbung des WT. Zu erklären ist dies möglicherweise durch den in Tabelle 3-1 gezeigten, deutlichen Rückgang der Chl a Konzentration und den damit verbundenen Anstieg des PBS/Chl a Verhältnis. Dies ist für die Färbung der TM verantwortlich, hat jedoch im Absorptionsspektrum durch die Normierung auf das Chl a Maximum (680 nm) keine Auswirkung. 61
72 Ergebnisse Abbildung 3-10: Optischer Phänotyp der charakterisierten Synechocystis-Mutanten und des WT. Aus der Tabelle 3-1 geht hervor, dass die Verkleinerung der PBS ebenfalls eine Auswirkung auf die Zellgröße hat. Der Vergleich der Zellgrößen zeigt einen im Vergleich zum WT verkleinerten Durchmesser der PBS-Mutanten. Dies kann durch ein verringertes Platzbedürfnisse, auf Grund kleinerer PBS, erklärt werden. Aus den in Tabelle 1-3 gezeigten Chl a Konzentrationen können erste Rückschlüsse auf das PS2/PS1 Verhältnis gezogen werden. Das Verhältnis von an PS2 und PS1 gebundenem Chl a ist ca. 1:3. Ein deutlicher Rückgang der Chl a Konzentration spricht daher für einen Rückgang von PS1. Dies zeigt sich durch den Rückgang von Chl a auf 9, µg/zelle in der TM, in der die Assemblierung von PS1 gestört ist. Die in der PAL zu beobachtende, deutliche Verringerung der Chl a Konzentration spricht daher für einen deutlichen Rückgang von PS1. Tabelle 3-1: Zusammenstellung der Verhältnisse von Chl a und PBS pro Zelle sowie der Zellgrößen in den Mutanten und WT (n = 5). Stamm WT CԐ Olive DM TM PAL Chl a/zelle 1, ,3-08 1, , , , [µg] (± 4, ) (± 3, ) (± 4, ) (± 2, ) (± 2, ) (± 2, ) PBS/Zelle 7, (± 2, ) 6, (± 1, ) 6, (± 1, ) 6, (± 2, ) 6, (± 1, ) 5, (± 1, ) Zellgröße 2,03 1,97 1,89 1,9 1,95 1,87 [µm] (± 0,03) (± 0,04) (± 0,1) (± 0,1) (± 0,03) (± 0,01) 62
73 Ergebnisse Das PS2/PS1 Verhältnis Zur Bestimmung des PS2/PS1 Verhältnisses, wurden Tieftemperaturfluoreszenzemissionsund EPR-Messungen durchgeführt. Zudem sollten die Messungen Aufschluss darüber geben, ob eine Veränderung des Verhältnisses aus einer Zu- oder Abnahme von PS2 und PS1 resultiert. Fluoreszenzemissionsmessungen bei 77 K erlauben das Abschätzen des Verhältnisses beider Photosysteme an Hand der Intensität ihrer Fluoreszenz. Der Einfluss verschiedener Faktoren auf die Fluoreszenz und unterschiedliche Chl a-mengen an PS2 und PS1 erlauben allerdings keine absolute Bestimmung der Photosystemmenge. Zur Verfolgung von Zu- und Abnahme der Photosysteme wurden die in der Abbildung 3-11 gezeigten Spektren auf das Emissionsmaximum von 5 µm Fluoresceine (510 nm) normiert. Abbildung 3-11: Fluoreszenzemissionspektrum bei 77 K von ganzen Zellen nach Anregung bei 440 nm. Spektren wurden auf das Fluoreszenzmaximum von Fluoresceine bei 510 nm normiert (n = 5). Nach Anregung der Chlorophyllfluoreszenz bei 430 nm zeigt der WT das für Synechocystis typische Emissionsspektrum mit drei Maxima. Die Maxima bei 685 und 695 nm resultieren aus der Fluoreszenz von PS2, während das dritte Maximum bei 725 nm PS1 zugeordnet 63
74 Ergebnisse wird. Die in den Mutanten zu beobachtenden Emissionsmaxima unterscheiden sich in ihrer Intensität von denen des WT. Zur Übersicht sind die Fluoreszenzintensitäten in Tabelle 3-2 zusammengefasst. Die gezeigten PS2/PS1 Verhältnisse wurden an Hand des PS2 Maximums bei 695 nm berechnet. Dies kann ausschließlich der PS2 Fluoreszenz zugeordnet werden. Die berechneten PS2/PS1-Verhältnisse zeigen eine Erhöhung des Verhältnisses in Abhängigkeit von der PBS-Größe. Das Verhältnis von 0,59 im WT ist in der Olive auf 0,68 und in der PAL auf 1,09 erhöht. In der TM wurde zudem eine deutliche Erhöhung auf 2,25 beobachtete. Die zu beobachtende Zunahme der PS1 Fluoreszenz steht jedoch sowohl in den PBS- Mutanten, als auch in der TM im Widerspruch zu den zuvor gezeigten Chl a Konzentrationen (Tab. 3-1). Zudem widersprechen die gezeigten Ergebnisse für CԐ und DM der Annahme, dass sich das Pigmentverhältnis durch die CԐ-Mutation nicht ändert. Sowohl in der CԐ, im Vergleich zum WT, als auch in der DM, im Vergleich zur Olive, ist ein verringertes PS2/PS1- Verhältnis zu beobachten. Tabelle 3-2: Vergleich der Fluoreszenzemission von PS2 und PS1, sowie der daraus berechneten PS2/PS1- Verhältnisse. Stamm WT CԐ Olive DM TM PAL Fluo.-685 nm Fluo.-725 nm 0,52 0,37 0,65 1,05 2,7 1,5 0,88 1,27 0,95 1,87 1,2 1,37 PS2/PS1 0,59 0,29 0,68 0,57 2,25 1,09 Zur Bestimmung exakter Werte der Mengen von PS2 und PS1 wurden in Kooperation mit Dr. Fikret Mamedov EPR-Messungen durchgeführt. Die Zellen wurden dafür bis zu einer möglichst hohen OD 750 nm wachsen gelassen und anschließend die GM aus ihnen isoliert (s ). Für die Messung des PS2-Anteils wurde das Signal von an der D2-UE des PS2 gebunden Tyrosins (TyrD) verwendet, da es stabiler als das entsprechende Signal des Tyrosins der D1- UE (TyrZ) ist. Zunächst wurden alle Proben für 5 min im Dunklen inkubiert. Anschließend erfolgte eine Belichtung mit starkem Weißlicht zur Oxidation der Probe. Die folgende Inkubation für 5 min im Dunkeln ist ausreichend, um TyrZ jedoch nicht um das stabilere TyrD zu reduzieren. Das Signal von TyrD (TyrDOX) kann so zu Bestimmung des PS2-Gehaltes genutzt werden. Eine weitere Probe wurde für 1 Minute mit 10 mm Ferricyanid inkubiert, um 64
75 Ergebnisse die maximale Oxidation von P700 zu induzieren (P700 + ). Das Signal dieser Probe wurde zur Bestimmung des PS1-Anteils genutzt. Die Ergebnisse in der Tabelle 3-3 zeigen die bereits aus den Fluoreszenzmessungen (Tab. 3-2) vermutete Erhöhung des PS2/PS21 Verhältnis in den PBS-Mutanten. Zudem kann der, an Hand der Chl a Konzentration (Tab. 3-1) angenommener, Rückgang von PS1 bestätigt werden. Den Chl a Konzentrationen entsprechend, ist dieser Rückgang in Olive und DM gering und in der PAL deutlicher ausgeprägt. Der geringe Rückgang von PS1 in der TM widerspricht jedoch der Annahme, dass durch den Eingriff in die Assemblierung geringerer PS1 Mengen erhalten werden. Dies ist jedoch möglicherweise durch Ungenauigkeiten der Messung, auf Grund der geringen Chl-Konzentration der Probe zu erklären. Tabelle 3-3: Aus EPR-Messungen resultierenden Mengen für PS1 (P700 + ) und PS2 (TyrDox) und die daraus resultierenden PS2/PS1 Verhältnisse. Die Ergebnisse sind in Prozent zum WT (100 %) dargestellt. Probe Chl a Konz. (mg/ml) P700 + /Chl [Relativ] TyrDox /Chl [Relativ] PS2 [% vom WT] PS1 [% vom WT] PS2/PS1 WT ,11 DM ,4 Olive ,39 TM ,33 PAL , ,24 Zudem zeigt sich, dass nur der Verlust von PC zu einem deutlichen Anstieg der PS2 Konzentration führt. Die vollständige Verlust der PBS in der PAL resultiert weder in einer zusätzlichen Erhöhung des PS2/PS1 Verhältnisses noch in einem weiteren Anstieg der PS2 Konzentration Aufbau des ph-gradienten an der Thylakoidmembran Die in die Stämme CԐ, DM und TM eingebrachte Mutation der ATP-Synthase soll zu einer Entkopplung des PET von der Reaktion der ATP-Synthese führen. Ist dies der Fall, muss in den Mutanten mit ATP-Synthase-Mutation eine Verringerung des ph-gradienten an der TYM 65
76 Ergebnisse feststellbar sein. Zum Nachweis wurden die in der Abbildung 3-12 gezeigten Acridine Gelb (AY) -Fluoreszenzmessungen durchgeführt (s ). Hierbei konnte die entkoppelnde Wirkung der ATP-Synthase-Mutation bestätigt werden. Sowohl die Fluoreszenz in der CԐ-Mutante im Vergleich zum WT als auch die Fluoreszenz in den beiden Mutanten DM und TM im Vergleich zur Olive sind deutlich herabgesetzt. Es ist also davon auszugehen, dass durch die eingefügte Inaktivierung in der Ԑ-UE der Aufbau des ph-gradienten an der TYM verringert wird. Eine im Vergleich zum WT erhöhte Fluoreszenz in der Olive lässt auf höhere Elektronentransportraten (ETR) schließen, die einen stärkeren Aufbau eines ph-gradienten an der TYM bedingen Zusammenfassung Abbildung 3-12: Acridine Gelb Fluoreszenzmessung von ganzen Zellen (n = 5). Nach Inkubation im Dunkeln wurden die Zellen mit rotem Licht (s. Abb. 3-29) der Intensität von 200 µe belichtet. Durch Kombination von PBS-Verkleinerung und Entkopplung konnte die Mutante DM erzeugt werden. Zudem war die zusätzliche Inaktivierung von ycf4 erfolgreich und ergab die Mutante TM. Die Mutation der ATP-Synthase führt zu einer Entkopplung, die den ph-gradienten an der TYM verringert. Ein Einfluss auf Pigmentstaus und PS2/PS1-Verhältnis ist jedoch nicht feststellbar. Die PBS-Verkleinerung führt zu einer deutlichen Verschiebung des Pigmentstatus und einer Erhöhung des PS2/PS1 Verhältnisses. In den PC-Mutanten Olive, DM und TM resultiert dies aus einer Erhöhung der PS2-Menge, in der PAL aus einer Verringerung von PS1. 66
77 Ergebnisse 3.3 Anpassung an wechselnde Lichtbedingungen Die Entkopplung des Elektronentransportes (ET) soll zwei wesentliche Funktionen erfüllen: Steigerung der Elektronentransportraten (ETR) Verringerung der Bildung von radikalen Sauerstoffspezies (ROS) und der damit verbundenen Photoinhibition. Eine Entkopplung kann aber auch in einer verringerten Produktion von ATP resultieren und stellt damit einen erheblichen Eingriff in den Zellmetabolismus dar. Dies gilt auch für die Verkleinerung der PBS. Diese führen zu einer signifikanten Veränderung der Lichtabsorption (s. Abbildung 3-9), welche möglicherweise ebenfalls die Anpassung an verschiedene Lichtbedingungen beeinflusst Wachstumsverhalten bei verschiedenen Lichtquantitäten Zur Überprüfung der Auswirkungen von Entkopplung und PBS-Verkleinerung auf die Vitalität und Hochlichtresistenz in den Mutanten wurden Wachstumskurven bei Normallicht- (75 µe) und bei Hochlicht-Bedingungen (500 µe) aufgenommen. Dazu wurden die Kulturen bis zu einer optischen Dichte bei 750 nm (OD 750 ) von eins mit 75 µe angezogen. Anschließend wurde die Hälfte der Kulturen ins Hochlicht überführt. Abbildung 3-13: Wachstum von WT, Olive und PAL bei 75 µe und 500 µe. Gemessen wurde die optischen Dichte bei 750 nm. Der Wechsel der Lichtintensität bei OD wird durch grüne Striche markiert. (n = 3). Wie in Abbildung 3-13 gezeigt, hat der Verlust von PC in der Olive bei 75 µe nur einen geringen Einfluss auf das Wachstum der Zellen. Die Zellen erreichen eine mit dem WT vergleichbare maximale OD 750 von ca. 7. Zudem sind die Verdopplungszeiten, wie in 67
78 Ergebnisse Tabelle 3-4 gezeigt, im Vergleich zum WT mit 33 h nur leicht auf 44 h erhöht. Die geringere Lichtabsorption kann somit scheinbar durch die erhöhten PS2-Mengen ausgeglichen werden. Durch den vollständige Verlust der PBS in der PAL ist maximalen OD 750 dagegen auf 6 und die Verdopplungszeiten deutlich auf ca. 59 h verringert. Die PAL kann die verringerte Absorption von Anregungsenergie somit nicht kompensieren. Dass das verringerte Wachstum durch die geringere Lichtabsorption bedingt ist, zeigt das Wachstum bei 500 µe. Durch das erhöhte Energieangebot wird das Wachstum in der Olive leicht, in der PAL dagegen deutlich beschleunigt, so dass hier höhere Verdopplungszeiten als im WT erreicht werden. Im WT zeigen sich dagegen durch eine Verlangsamung des Wachstums bereits geringe Anzeichen von Photoinhibition. Tabelle 3-4: Verdopplungszeiten der exponentiellen Wachstumsphase von WT und Mutanten bei 75 und 500 µe (n = 3). Stamm WT CԐ Olive DM TM PAL Licht [h] [h] [h] [h] [h] [h] 75 µe 33,5 (± 2,45) 44,1 (± 1,72) 43,5 (± 4,92) 88,4 (± 4,32) 118,4 (± 7,47) 58,6 (± 5,4) 500 µe 38,9 (± 5,63) 43,4 (± 3,75) 40,6 (± 4,89) 37,5 (± 1,73) x 24,4 (± 1,28) Die Mutation der ATP-Synthase in der CԐ hat, wie in der Abbildung 3-14 gezeigt, vergleichbar mit dem Verlust von PC, nur einen geringen Einfluss auf das Wachstum, so dass hier, wie in der Olive, Verdopplungszeiten von ca. 44 h (Tab. 3-4) erreicht werden. Die Verlangsamung zeigt jedoch einen erhöhten Energiebedarf der Zellen durch die Entkopplung. Durch diesen kommt es in der Kombination mit dem Verlust von PC in der DM zu deutlich erhöhten Verdopplungszeiten von ca. 88 h. Diese werden durch die zusätzliche Inaktivierung von Ycf4 in der TM nochmals deutlich auf ca. 118 h gesenkt. In der TM kommt es somit zu deutlichen Einschränkungen des Metabolismus. Diese Einschränkungen führen bei 500 µe zu einem Absterben der TM. In der DM kommt es dagegen, wie bereits in der Olive, durch die erhöhte Energiezufuhr zu einer Beschleunigung des Wachstums. Leicht erhöhte Verdopplungszeiten, im Vergleich zur Olive, deuten eine erhöhte Hochlichtresistenz durch die Entkopplung an. Im Vergleich zum WT, leicht erhöhte Verdopplungszeiten in der CԐ, bestätigt dies. 68
79 Ergebnisse Abbildung 3-14: Wachstum von CԐ, DM und TM bei 75 µe und 500 µe. Gemessen wurde die optischen Dichte bei 750 nm. Der Wechsel der Lichtintensität bei OD wird durch grüne Striche markiert. (n = 3) Veränderung der Hochlichttoleranz Zur Überprüfung, ob die veränderten Wachstumseigenschaften nur durch einen veränderten Energiebedarf, oder auch durch eine erhöhte Hochlichtresistenz bedingt sind, wurden zu verschiedenen Zeitpunkten Absorptionsspektren der Hochlichtkulturen (Abb. 3-13) aufgenommen. In der Abbildung 3-15 sind exemplarisch für die Messungen, die Ergebnisse vom WT und den BPS-Mutanten Olive und PAL gezeigt. Generell ist in allen Kulturen eine vergleichbare Anpassung an das Hochlicht zu beobachten. Diese Anpassung besteht aus einem Abbau von Chl a (680 nm) und der PBS (630 nm), sowie eine Akkumulation von Carotinoiden (460 nm). In WT und PAL ist ein Ausbleichen von Chl a bereits nach 20 h, in der Olive erst nach ca. 120 h zu erkennen. Der Anstieg der PS2-Menge in der Olive scheint somit zu einem Anstieg der Hochlichtresistenz und einem verringerten Abbau der Photosysteme zu führen. Die Entkopplung in CԐ und DM führte zu keiner signifikanten Änderung der Spektren, so dass sie keinen Einfluss auf Abbau oder Schädigung der Photosysteme zu haben scheint. Die Inaktivierung Ycf4 führte zum Ausbleichen und Absterben (Abb. 3-14) der Kulturen. Da durch das Wachstum (s ) ein Effekt der Entkopplung auf die Photoinhibition vermutet wurde, der durch die Spektren (Abb. 3-15) nicht bestätigt werden konnte, wurde ebenfalls die Wiederherstellung der PS2-Aktivität gemessen. Die PS2-Aktivität und damit die Photosyntheseleistung ergab sich dabei an Hand der Fluoreszenzparameter aus (F M -F 0 )/F M. Da die Grundfluoreszenz F 0 mit dem PBS/Chl a-gehalt in der Zelle variiert, wurden hier nur Organismen mit gleichem PBS/Chl a-gehalt verglichen. Die Photoinhibition wurde durch 69
80 Ergebnisse Belichtung für 30 min mit 1500 µe starkem Weißlicht initiiert. Die Zugabe von 50 µg/ml Chloramphenicol inhibierte die Proteinbiosynthese und dadurch die Neusynthese von PS2. Die Wiederherstellung der PS2-Aktivität wurde bei moderatem Licht von 50 µe verfolgt. Das Chloramphenicol wurde zuvor aus den Zellen ausgewaschen. Abbildung 3-15: Absorptionsspektren von WT, Olive und PAL. Die Spektren wurden von bei 50 µe angezogenen Kulturen mit einer OD 750nm von eins (rot) und zu den Zeitpunkten t1 = 20h (grün), t2 = 44h (gelb), t3 = 68h (blau), t4 = 124h (pink) und t5 = 184h (cyan) nach Überführung ins Hochlicht mit 500 µe aufgenommen (n = 3). Wie in Abbildung 3-16 zu erkennen, ist die Verringerung der PS2-Aktivität in den ersten 30 min in WT und CԐ sowie in Olive und DM vergleichbar. Die eingefügte Entkopplung zeigt also keinen Effekt beim Schutz von PS2 und seiner Aktivität selbst. Dies erklärt möglicherweise das vergleichbare Ausbleichen der Kulturen (Abb. 3-14). Die Entkopplung beschleunigt jedoch die Wiederherstellung der PS2-Aktivität bei 50 µe. So werden in der CԐ nach 110 min höhere Aktivitäten als im WT gemessen. In der DM ist die Aktivität nach 110 min mit 40 % im Vergleich zur Olive sogar verdoppelt. Die Bildung von ROS wird zwar durch die Entkopplung verringert, dennoch schützt dies nicht das PS2. Möglicherweise kommt es jedoch zu einer geringeren Schädigung von an der Biosynthese von PS2 beteiligten Proteinen. Eine de novo Synthese von PS2 ist somit weniger beeinträchtigt und die Aktivität kann schneller wieder hergestellt werden. 70
81 Ergebnisse Abbildung 3-16: Wiederherstellung der PS2-Aktivität nach Photoinhibition in ganzen Zellen. Photoinhibition wurde mit rotem Licht der Intensität 1500 µe nach Zugabe von Chloramphenicol induziert. Wiederherstellung der PS2- Aktivität wurde nach Auswaschen des Chloramphenicols bei moderatem Licht von 50 µe gemessen. A: Vergleich von WT mit CԐ. B: Vergleich von Olive mit DM. (n = 3) Kurzfristige Adaption state transitions Die zuvor gezeigten Ergebnisse deuten an, dass die Entkopplung und die PBS-Mutationen die Energieanforderungen der Zelle ändern. Zudem haben die PBS einen wesentlichen Einfluss auf die Quantität und Qualität des für die Photosynthese absorbierten Lichtes. Diese geänderten Energieanforderungen haben möglicherweise einen Einfluss auf die state transitions. Bei diesen, ausführlich in Kapitel beschrieben, Anpassungsprozessen werden die wechselnden Energieanforderungen des Organismus durch eine Veränderung der Energieweiterleitung an das PS2 und das PS1 ausgeglichen. Hierbei können die Zellen entweder im state1 oder im state 2 vorliegen. Zur Überprüfung der state transitions wurden 77 k Fluoreszenzemissionsmessungen durchgeführt. Der state 1 der Zellen wurde dabei durch Inkubation mit langwelligem Rotlicht, der state 2 durch Dunkelinkubation induziert. Nach Anregung bei 580 nm wurden die in Abbildung 3-17 gezeigten Spektren erhalten. Im WT ist das Emissionsspektrum nach Anregung der PBS mit den typischen drei Maxima zu sehen. Das erste Maximum bei 650 bis 680 nm resultiert aus der Fluoreszenz von PC und APC. Die beiden weiteren Maxima bei 690 und 725 nm entstammen von PS2 und PS1, an welche die Anregungsenergie von den PBS weitergeleitet wird. Bei einem Vergleich der Dunkelprobe mit der Rotlichtprobe wird deutlich, 71
82 Ergebnisse dass ein Übergang von state 2 zu state 1 mit einer Veränderung der Energieweiterleitung an PS2 und PS1 einhergeht. Abbildung 3-17: Messung der state transitions. State 1 wurde durch Bestrahlung mit langwelligem Rotlicht induziert (rot, gepunktete und gestrichelte Linie). State 2 wurde durch Inkubation im Dunkel induziert (schwarze, durchgehende Linie) (n = 3). Durch das Fehlen von PC ist in den Mutanten nur das APC-Maximum bei 680 nm zu erkennen. In der PAL fehlt die PBS-Emission vollkommen. Trotz verkleinerter oder fehlender PBS sind jedoch sowohl in Olive als auch in der PAL state transitions zu erkennen. Auch wenn dieser Effekt in der PAL nur gering ist, zeigt dies, dass die state transitions nicht ausschließlich von den PBS beeinflusst sind. In der TM ist ein deutlicher Effekt der Inaktivierung von Ycf4 zu erkennen. Die PBS-Emission ist nur sehr gering und es finden keine state transitions statt. Dies kann möglicherweise durch wenige ungebundenen PBS in der Zelle erklärt werden. Einen Grund dafür kann jedoch auch eine herabgesetzte Bindungsaffinität der PBS an PS1, oder ein veränderter Oligomerisierungszustand von diesem sein. 72
83 Ergebnisse Die Entkopplung in CԐ und DM änderte die state transitions nicht und ist daher hier nicht gezeigt Zusammenfassung Die PBS-Verkleinerung führt zu einer deutlichen Verringerung der Wachstumsgeschwindigkeiten, hat aber keinen Einfluss auf die maximale Zelldichte. Höhere Lichtbedingungen resultieren in einer deutlichen Beschleunigung des Wachstums der PBS- Mutanten. Dabei kann in der Olive durch die Erhöhung der PS2-Menge eine verringerte Absorption von Anregungsenergie ausgeglichen und ein Effekt von Photoinhibition verringert werden. Die Entkopplung erhöht den Energiebedarf der Zellen und verringert die Photoinhibition durch erhöhte PS2-Neusynthese. Die Inaktivierung von ycf4 resultiert im Verlust der für die Anpassung an Hochlicht benötigten, funktionalen Flexibilität der Elektronentransportkette. 3.4 Der Elektronentransport in der Thylakoidmembran Die Effekte der Mutationen auf den PET wurden durch die Messungen der O 2 -Entwicklung und der Elektronentransportraten (ETR) untersucht O 2 -Entwicklung und Respiration An Hand der O 2 -Entwicklung und des O 2 -Verbrauches können die Auswirkungen der Mutationen auf den Metabolismus untersucht werden. In Abbildung 3-18 A ist die Nettosauerstoffproduktion nach Belichtungen mit starkem Rotlicht dargestellt. Die Sauerstoffproduktion wurde pro Zelle berechnet, um Fehler durch die stark verschobenen PS2/PS1 Verhältnisse und veränderte Chl a Konzentrationen zu vermeiden. Die Ergebnisse der O 2 -Messungen ohne Zusätze zeigen, dass sowohl die Entkopplung als auch der Verlust von PC die O 2 -Entwicklung erhöhen. Der PC-Verlust resultiert jedoch nur in einer im Vergleich zum WT (7, µmolo 2 /Zelle h) geringen Erhöhung in der Olive (9, µmolo 2 /Zelle h). Dies ist möglicherweise durch die Verwendung von Rotlicht bedingt. Die Entkopplung führt dagegen zu einer deutlicheren Steigerung der O 2 -Entwicklung, so dass sowohl in der CԐ (1, µmolo 2 /Zelle h) als auch in der DM signifikante Steigerungen der O 2 -Produktionsraten beobachtet werden. Die Inaktivierung von Ycf4 und der Verlust der kompletten PBS in der PAL verringern dagegen die O 2 -Entwicklung signifikant. Dass die Entkopplung tatsächlich einen Erhöhung der O 2 -Entwicklung bewirkt, zeigen die Ergebnisse nach Zugabe von 10 mm NH 4 Cl. Durch die so hervorgerufene Entkopplung ist in den Kulturen 73
84 Ergebnisse ohne ATPase-Mutation eine deutliche Steigerung der O 2 -Entwicklung zu beobachten. In den bereits entkoppelten Mutanten ist dagegen eine Abnahme festzustellen. Wie in Abbildung 3-18 B gezeigt, hat die Verkleinerung der PBS einen deutlichen Einfluss auf die Respiration. In der Olive beträgt die Respiration nur noch 75 % des WT und ist signifikant auf µmolo 2 /Zelle h verringert. In der PAL ist eine weitere signifikante Verringerung auf 2, µmolo 2 /Zelle h festzustellen. Hier liegen die Respirationsraten nur noch bei 5 % des WT. Ein Einfluss der Entkopplung kann nur für die DM beobachtet werden, bei der die Respiration auf 2, µmolo 2 /Zelle h verringert ist. Vergleichbar zur O 2 -Entwicklung resultiert die Inaktivierung von Ycf4 auch bei der Respiration in einer signifikanten Verringerung der Raten auf 1, µmolo 2 /Zelle h. Abbildung 3-18: Charakterisierung von WT, CԐ, Olive, DM, TM und PAL mittels Sauerstoffmessungen. A: Messung der Nettosauerstoffproduktionsraten bei Belichtung ohne (grün) und mit Zusatz von 10 mm Nh 4Cl (blau). B: Messung der Respiration an Hand der Sauerstoffabnahme bei Dunkelinkubation (n = 5; t.test α = 0.05) Der Elektronentransport am Photosystem 1 Das PS1 ist ein zentrales Element des PET, das sowohl am linearen, als auch am zyklischen ET beteiligt ist. Untersuchungen der ETR am PS1 können somit Hinweise auf die Photosyntheseleistung eines Organismus geben und sind geeignet mögliche Engpässe, so genannte bottle necks, im ET aufzuzeigen. 74
85 Ergebnisse Die PS1-Donorseite und der zyklische Elektronentransport Wie bereits beschrieben (s ) können aus der Absorptionsänderung von P700 Limitierungen an der Donor- und Akzeptorseite von PS1 berechnet werden. Die in Abbildung 3-19 gezeigte Donorseitenlimitierung (N(D)) wird hierbei prozentual wiedergegeben und kann auf der Skala entsprechend zwischen Null (keine Limitierung) und Eins (maximale Limitierung) variieren. In Teil A der Abbildung 3-19 ist N(D) ohne Zugabe eines Inhibitors gezeigt. Im WT werden maximale Werte für N(D) bei ca. 800 µe erreicht. Olive und TM zeigen mit dem WT vergleichbare Raten von N(D). In der PAL tritt dagegen eine maximale Limitierung schon bei relativ niedrigen Lichtintensitäten von 200 µe auf und ist möglicherweise durch vergleichsweise niedrige Raten des zyklischen ET bedingt. Im Gegensatz dazu tritt sowohl in der CԐ, als auch in der DM unter den gewählten Bedingungen keine maximale Limitierung von N(D) auf. Dabei finden sich in der DM die niedrigsten Raten von N(D). Mögliche Erklärungen für die geringen Limitierungen sind hohe ETR am PS2 und hohe Raten des zyklischen Elektronentransportes. Abbildung 3-19: Messung der Donorseitenlimitierung (N(D)) am PS1 in WT, CԐ, Olive, DM, TM und PAL bei verschiedenen Lichtintensitäten (PAR). Messung ohne (A) und mit Zugabe (B) von 500 µm MV. Berechnung erfolgte an Hand der P700 Absorptionsänderung (s ) von ganzen Zellen (n = 5). Um den Einfluss des zyklischen ETs zu überprüfen, wurden N(D)-Messungen mit Zugabe von 500 µm Methyl Viologen (MV) durchgeführt (Abbildung 3-19 B). MV ist ein starker PS1- Elektronenakzeptor und verhindert eine Abgabe von Elektronen in den zyklischen ET. In der 75
86 Ergebnisse PAL und der Olive treten keine Veränderung von N(D) auf. Dies spricht für niedrige Raten des zyklischen ET in beiden Mutanten. Eine in der Olive später als in der PAL einsetzende maximale Limitierung zeigt somit hohe Raten des ET am PS2 der Olive. In WT, CԐ, DM und TM ist nach Zugabe von MV ein deutlicher Anstieg von N(D) zu beobachten. Die Raten des zyklischen ET sind also höher als in PAL und Olive. Dass in der DM trotz Zugabe von MV keine vollständige Limitierung zu beobachten ist, spricht hier nicht nur für hohe Raten des zyklischen ET, sondern ebenfalls für hohe lineare ETR. Zur genaueren Bestimmung der Raten des zyklischen ET wurden P700 Absorptionsänderungsmessungen durchgeführt. Die Messungen erfolgten dabei ohne Inhibitoren, um den Gesamt-ET zum PS1 zu messen, sowie mit 500 µm DCMU, um die Raten des zyklischen ET zu bestimmen und mit Zugabe von 500 µm MV zur Bestimmung der Raten des linearen ET. In der Abbildung 3-20 sind die Raten für den linearen und den zyklischen ET jeweils in Prozent vom Gesamt-ET dargestellt. Wie schon durch die Ergebnisse in Abbildung 3-19 zu vermuten, sind in Olive und PAL niedrige Raten des zyklischen ET mit ca. 10 % zu beobachten. Im WT liegen die Raten des zyklischen ET bei ca. 20 %. In der CԐ sind die Raten im Vergleich zum WT gesteigert und erreichen hier ca. 25 %. Für die DM konnten die erwarteten hohen Raten von ca. 50 % gezeigt werden. Für die TM war es nicht möglich gesicherte Ergebnisse für die Raten des Gesamt-ET zu messen. Dies war bedingt durch eine schnelle Rückreduktionen von P700 + während der Belichtung, welche möglicherweise durch Probleme an der Akzeptorseite des PS1 aufgrund der ycf4 Inaktivierung zu erklären sind Die PS1-Akzeptorseite Abbildung 3-20: Raten des linearen (grüne Balken) und zyklischen (blaue Balken) ET in Prozent des Gesamt-ET. Berechnet aus der P700 Absorptionsänderung von ganzen Zellen (n = 5). Zusätzlich zu den Limitierungen an der Donorseite von PS1 wurden ebenfalls mögliche Limitierungen an der Akzeptorseite untersucht. So wie bereits für N(D) erklärt (s ), steht Eins für eine maximale und Null für eine minimale Limitierung. 76
87 Ergebnisse Das PS1 kann Elektronen an seiner Akzeptorseite in viele verschiedene Reaktionswege einspeisen, so dass hier nach einer kurzen Belichtungszeit, die benötigt wird, um den Calvin- Zyklus zu aktivieren, keine Limitierungen erwartet werden. Wie in Abbildung 3-21 gezeigt, trifft dies auf WT, Olive, CԐ und PAL zu. Hier ist nur eine geringe Limitierung an der Akzeptorseite zu beobachten, welche bei höheren Lichtintensitäten abnimmt. Anders verhält es sich in der TM und DM. Hier sind im Vergleich relativ hohe Limitierungen von fast 50 % zu beobachten. Eine mögliche Erklärung dafür kann eine entkopplungsbedingte Verringerung der ATP-Konzentration in diesen Kulturen sein. Dass dieser Effekt ausschließlich von der Entkopplung bedingt ist, kann durch die Ergebnisse der CԐ nicht bestätigt werden. Möglicherweise tritt hier, wie bereits für die Respiration festgestellt (s ), ein Effekt erst durch die Kombination von PBS-Verkleinerung und Entkopplung auf. Nicht gezeigte Kontrollmessungen mit Zugabe von 500 µm MV als PS1 Elektronenakzeptor resultierten in allen Kulturen in einer Aufhebung der Limitierung. Abbildung 3-21: Messung der Akzeptorseitenlimitierung (N(A)) am PS1 an Hand von P700 Absorptionsänderung bei verschiedenen Lichtintensitäten (PAR). (n = 5) Aufnahme von Lichtsättigungskurve an Hand der O 2 -Entwicklung Zur Bestimmung der optimalen Lichtintensität und den möglichen photosynthetischen Produktionsraten in WT und den Mutanten wurden Lichtsättigungskurven (P-I-Kurven) aufgenommen. Dazu wurde in der Abbildung 3-22 die, bei jeder Lichtintensität berechnete Nettosauerstoffproduktion (s ) gegen die Lichtintensität aufgetragen. Die gezeigten Sauerstoffproduktionsraten wurden dabei auf die eingesetzte Zellzahl normiert. Für den WT und CԐ sowie Olive und DM konnten vergleichbare O 2 -Produktionsraten ermittelt werden. Im Gegensatz zu den zuvor gezeigten O 2 -Entwicklungen bei kurzer Belichtung (s ) kann hier keine gesteigerte O 2 -Entwicklung durch die Entkopplung gezeigt werden. Zudem zeigen Olive und DM, im Gegensatz zu den zuvor ermittelten 77
88 Ergebnisse Ergebnissen, signifikant verringerte O 2 -Produktionen als der WT. Erst bei hohen Lichtintensitäten ab 1500 µe kommt es zu einem Angleichen der O 2 -Produktion von Olive und DM und der des WT. Bei diesen Lichtintensitäten kommt es in WT und CԐ bereits zur Photoinhibition. Zudem ist eine Verschiebung der optimalen Lichtintensität zu höheren Intensitäten in Olive und DM zu erkennen. Abbildung 3-22: Messung der Nettosauerstoffproduktion von ganzen Zellen im WT, CԐ, Olive, DM, TM und PAL bei verschiedenen Lichtintensitäten (n = 5; t.test α = 0.05). Verwendet wurde das in Abb gezeigte rote Anregungslicht. Die Produktionsraten in der TM zeigen im Vergleich zur DM keine signifikanten Veränderungen. Deutlich wird hier aber eine Verschiebung der optimalen Lichtintensität zu niedrigeren Intensitäten und eine damit verbundene erhöhte Photoinhibition. Dies führt bei Lichtintensitäten ab 1000 µe zu signifikant erniedrigten Produktionsraten im Vergleich zur Olive. Die PAL zeigte nur geringe O 2 -Produktionsraten. Das Maximum der Produktion wurde hier bei der höchsten Lichtintensität von ca µe beobachtet, so dass der Effekt von Photoinhibition nur gering ist. Das erhöhte PS2/PS1 Verhältnis in den PBS-Mutanten sollte die Photosyntheserate steigern. Dies gilt insbesondere für die Olive-Mutanten mit hohen PS2-Konzentrationen. Dass dies nicht der Fall ist, kann durch zwei Faktoren bedingt sein: Verringerte Lichtabsorption durch PBS-Mutation Veränderte Elektronennutzung in den Mutanten 78
89 Ergebnisse Eine verringerte Lichtabsorption könnte ebenfalls die geringe Anfangssteigung der PI-Kurven in den PBS-Mutanten und die geringere Photoinhibition erklären Zusammenfassung Der Verlust von PC führt zu einer leichten Verringerung der O 2 -Entwicklung, der vollständige Verlust der PBS senkt diese dagegen deutlich Durch die Verkleinerung der PBS ist jedoch die Photoinhibition verringert. Durch die Entkopplung kann die O 2 -Produktion nur bei kurzen Belichtungen gesteigert werden, und es sind hohe Raten des zyklischen ET zu beobachten. Die Inaktivierung von ycf4 senkt den PET und die O 2 -Entwicklung deutlich und erhöht die Photoinhibition. 3.5 Der Effekt unterschiedlicher Lichtabsorptionen auf die Photosyntheseleistung Die in den Kapiteln zuvor gezeigten Ergebnisse wurden mit Kulturen erhalten, die mit dem am LS üblichen Anzuchtlicht angezogen wurden (s. Abbildung 3-7). Diese setzt sich hauptsächlich aus grünem und rotem Licht zusammen. Durch die verringerte Absorption von rotem Licht in den PBS-Mutanten (Abb. 3-9) absorbieren der WT und die Mutanten unterschiedliche Lichtmengen und passen so ihren Metabolismus an unterschiedliche Lichtintensitäten an. Zur Überprüfung des Effektes dieser Anpassungen auf die Photosynthese wurden WT und Mutanten mit dem in Abbildung 3-23 gezeigten, blaugrünen LEDs angezogen. Der Vergleich mit den Absorptionsspektren von WT und DM zeigt, dass im Wellenlängenbereich ab 630 nm, in dem die PBS absorbieren, nur geringe Lichtintensitäten ausgestrahlt werden. WT und Mutanten nutzen für die Photosynthese hauptsächlich das Licht im Wellenlängenbereich von 460 nm, bei dem die Absorption von Anregung für alle Kulturen gleich ist. 79
90 Ergebnisse Abbildung 3-23: Vergleich des Spektrums der für die Anzucht verwendeten blaugrünen LEDs mit der Absorption des WT und der DM. Für die Photosynthese kann hier hauptsächlich das Licht im Wellenlängenbereich um ca. 460 nm verwendet werden Auswirkungen auf die Phycobilisomen Die PBS absorbieren Licht im Bereich von 530 bis 650 nm. In diesem Wellenlängenbereich werden von dem verwendeten blaugrünen Licht (Abb. 3-23) nur sehr geringe Intensitäten ausgestrahlt, so dass die PBS nur zu einem geringen Teil zur Absorption beitragen. Da der größte Teil des Lichtes von Chlorophyll absorbiert wird, ist die Ausbildung von PBS nachteilig. Durch die Kultivierung im blauen Licht ist somit ein Rückgang der PBS zu erwarten. Um dies zu überprüfen, wurden Kulturen sowohl mit rotem, (Abb. 3-7) als auch mit blauem Anzuchtlicht (Abb. 3-22) bis zu einer OD 750 nm von 1 angezogen. Die anschließend aufgenommen 77 K-Spektren nach Anregung bei 580 nm sind in Abbildung 3-24 gezeigt. Die Spektren wurden auf das Fluoreszenzmaximum von PC (650 nm) normiert. 80
91 Ergebnisse Abbildung 3-24: Vergleich der Fluoreszenzemission bei 77 K nach Anregung bei 580 nm. Eingesetzt wurden Kulturen die mit rotem (rot) und blauem Anzuchtlicht (blau) angezogen wurden. Spektren wurden auf das Emissionsmaximum von PC bei 650 nm normiert (n = 3). Die mit blauem Licht angezogenen Kulturen zeigen eine deutliche Verringerung der Fluoreszenz. Die Nutzung der Anregungsenergie ist also durch eine Kultivierung mit blauem Licht verschlechtert worden. Diese kann jedoch nicht ausschließlich durch einen Abbau der PBS erklärt werden, da ein vergleichbarer Effekt ebenfalls in der PAL zu beobachten ist. Möglicherweise spielen hier weitere, durch blaues Licht ausgelöste, Effekte der Fluoreszenzauslöschung (NPQ) eine Rolle Die Auswirkung auf den Pigmentstatus der Zelle In den Fluoreszenzemissionsmessungen ist eine deutlich verschlechterte Ausnutzung des Anregungslichtes durch einen Wechsel zu blauem Anzuchtlicht festzustellen. Um zu überprüfen, ob dies durch einen Veränderung der zellulären Pigmentkonzentration erklärt werden kann, sind in der Tabelle 3-5 die Konzentrationen von Chl a und PBS pro Zelle aufgeführt. Ein Vergleich der Chl a-konzentrationen in Tabelle 3-5 und der von Kulturen aus rotem Anzuchtlicht (s. Tabelle 3-1) zeigt eine Zunahme des Chl a-gehalts pro Zelle für die Kulturen 81
92 Ergebnisse aus blauem Anzuchtlicht (außer PAL). Dies deutet darauf hin, dass die Zellen versuchen die verringerte Lichtabsorption der PBS durch einen Anstieg von Chlorophyll auszugleichen. Tabelle 3-5: Zusammenstellung der Verhältnisse von Chl a und PBS pro Zelle sowie der Zellgrößen in Mutanten und WT bei einer Kultivierung mit blaugrünem Licht (n = 5). Stamm WT CԐ Olive DM TM PAL Wert Chl a / Zelle [µg] 2, (± 6, ) 2, (± 2, ) 2, (± 5, ) 1, (± ) 1, (± 2, ) 1, (± ) PBS / Zelle 5, (± 2, ) 6, (±7, ) 7, (± 5, ) (6, ) (± 1, ) 5, (± 3, ) 7, (± 2, ) Zellgröße [µm] 1,9 (± 0,1) 1,84 (± 0,01) 1,94 (± 0,018) 1,9 (± 0,06) 1,8 (± 0,04) 1,9 (± 0,03) In WT und CԐ ist zudem eine Verringerung der PBS-Absorption pro Zelle zu beobachten, die mit einer Verkleinerung der Zellen einhergeht. Die PBS haben also, wie bereits vermutet, (s ) einen Einfluss auf die Zellgröße. In den PBS-Mutanten Olive, DM, TM sind keine Veränderungen der PBS-Absorption und der Zellgröße zu erkennen. Dies zeigt, dass die Lichtabsorption der PBS in diesen Mutanten eine geringere Rolle spielt Die Effekte auf den photosynthetischen Elektronentransport Die Anpassung der Zellen auf der Pigmentebene resultiert in veränderten Lichtabsorptionen der Zellen. Dieser Effekt sollte besonders in WT und CԐ sichtbar sein, da hier ein Abbau der PBS gezeigt werden konnte. Eine Veränderung der Absorption und Weiterleitung von Anregungsenergie resultiert in veränderten Eigenschaften des PET Die Limitierung des ET zum Photosystem 1 und der zyklische Elektronentransport Wie bereits beschrieben (s und ) kann an Hand der P700 Absorptionsänderung die Donorseitenlimitierung N(D) von PS1 ermittelt werden. Diese wird prozentual angegeben und kann auf der Skala entsprechend zwischen Null (keine Limitierung) und Eins (maximale Limitierung) variieren. In der Abbildung 3-25 sind Messungen von N(D) mit Kulturen aus 82
93 Ergebnisse blauem Anzuchtlicht (Abb. 3-23) dargestellt. Zur Oxidation von PS1 wurde rotes Licht der Wellenlänge 660 nm verwendet (s ). Abbildung 3-25: Messung der Donorseitenlimitierung (N(D)) am PS1 in WT, CԐ, Olive, DM, TM und PAL bei verschiedenen Lichtintensitäten (PAR). A: Messung ohne und B: mit Zugabe von 500 µm MV (n = 5). Für Kulturen aus rotem Anzuchtlicht konnten deutliche Unterschiede von N(D) gezeigt werden (s. Abbildung 3-19). Im Gegensatz dazu zeigen mit blauem Licht angezogene Kulturen vergleichbare Donorseitenlimitierungen (Abbildung 3-25 A). In WT und CԐ ist die Donorseite von PS1 schon bei niedrigen Lichtintensitäten von 200 µe zu 100 % limitiert. Dies deutet darauf hin, dass unter diesen Bedingungen nur geringe Raten des zyklischen ET zu erwarten sind. In Olive, DM und TM sind vergleichbare maximale Donorseitenlimitierungen zwischen 400 und 600 µe zu beobachten. Die Anzucht in blauem Licht hat besonders in der DM zu einer beschleunigten Donorseitenlimitierung und damit geringeren Raten des zyklischen ET geführt. Dies wird ebenfalls durch die in Abbildung 3-26 gezeigten, verringerten Raten des zyklischen ETs bestätigt. Die langsame, erst bei sehr hohen Lichtintensitäten maximale, Donorseitenlimitierung in der PAL deutet eine verbesserte Lichtausnutzung an. 83
94 Ergebnisse Die Ergebnisse in Abbildung 3-25 B zeigen, dass die Zugabe von MV in allen Kulturen bei geringen Lichtintensitäten von 200 µe zu einer maximalen Donorseitenlimitierung führt. Dies bestätigt den Einfluss des zyklischen ET auf N(D). Die in der Abbildung 3-25 gezeigten Ergebnisse werden von denen in der Abbildung 3-26 gezeigten Raten für den zyklischen ET bestätigt. In WT und CԐ mit schneller Donorseitenlimitierung wurden nur geringe Raten des zyklischen ET um 10 % des Gesamt-ET gemessen. Ebenso wurde der Anstieg der Raten des zyklischen-et in der PAL nachgewiesen. Die geringen Raten des zyklischen ET in WT und CԐ zeigen, dass dieser stark von der Lichtausnutzung der Kulturen beeinflusst wird. Die verbesserte Energieausnutzung von Kulturen aus rotem Anzuchtlicht ergibt erhöhte Raten des zyklischen ET. In den PBS- Mutanten ist der Effekt eines Wechsel der Lichtqualität geringer, und damit auch die Auswirkung auf den zyklischen ET. Abbildung 3-26: Raten des linearen (blau) und des zyklischen (grün) Elektronentransportes in Prozent vom Gesamt-ET (n = 5) Die Abnahme von Elektronen am Photosystem 1 Wie bereits beschrieben (s und ) kann an Hand der P700 Absorption die Akzeptorseitenlimitierung von PS1 N(A) ermittelt werden. Diese wird prozentual angegeben und variiert zwischen Null (keine Limitierung) und Eins (max. Limitierung). Das PS1 kann Elektronen an seiner Akzeptorseite in viele verschiedene Reaktionswege einspeisen, so dass hier nach einer kurzen Belichtungszeit, die benötigt wird, um den Calvin-Zyklus zu aktivieren, keine Limitierungen erwartet werden. Die in Abbildung 3-27 dargestellten Ergebnisse zeigen für WT, CԐ, Olive und PAL die erwarteten geringen Limitierungen bei niedrigen Lichtintensitäten. In der DM ist die Limitierung 84
95 Ergebnisse Abbildung 3-27: Durch P700 Absorption ermittelte Akzeptorseitenlimitierung (N(A)) am PS1 von ganzen Zellen (n = 5). Die Anregung erfolgte bei 660 nm mit verschiedenen Lichtintensitäten (PAR) (s ). an der Donorseite (wie in mit rotem Licht angezogenen Kulturen (s. Abbildung 3-21)), deutlich erhöht. Im Gegensatz zu den zuvor gezeigten Ergebnissen wird die Limitierung hier jedoch mit zunehmenden Lichtintensitäten aufgehoben. In der TM ist die Limitierung der Akzeptorseite bei niedrigen Lichtintensitäten bis 100 µe maximal, so dass hier bis zum Anlaufen des Calvin-Zyklus erhebliche Probleme bei der Elektronenabgabe am PS1 bestehen. Dies deutet darauf hin, dass alternative, elektronenakzeptierende Prozesse in der TM stark herunterreguliert sind oder Probleme am PS1 selber auftreten Aufzeichnung von P-I-Kurven mittels der Nettosauerstoffproduktion Wie bereits beschrieben (s und 3.4.3) kann die Photosyntheseleistung an Hand der Nettosauerstoffproduktion überprüft werden. Dazu wurden die in Abbildung 3-28 gezeigten Lichtsättigungskurven (PI-Kurven) aufgezeichnet. Dafür wurde die Nettosauerstoffproduktion gegen die jeweils verwendete Lichtintensität aufgetragen. Die Ergebnisse zeigen eine nahezu identische Nettosauerstoffproduktion für den WT, die CԐ und die Olive. Während in der Olive die O 2 -Entwicklung vergleichbar zu der von mit rotem Licht angezogenen Kulturen ist, ist in WT und CԐ ein deutlicher Rückgang zu erkennen. Zudem ist sowohl in WT und CԐ, als auch in der Olive eine Verschiebung der idealen Lichtintensität zu niedrigeren Intensitäten zu erkennen. Die Ergebnisse zeigen zum einen, dass die verringerte Energieausnutzung in WT und CԐ auch die Photosyntheseraten beeinflusst und zum anderen, dass durch die Entkopplung die O 2 -Produktion nicht erhöht wird. Dies wird durch die O 2 -Entwicklung in der DM bestätigt. In der TM und der PAL wurden erneut die geringsten O 2 -Entwicklungen beobachtet. Die verringerte O 2 -Entwicklung in WT und CԐ kann möglicherweise neben einem Abbau der PBS auch durch ein Auslöschen der Anregungsenergie über das Protein OCP erklärt werden. 85
96 Ergebnisse Da die O 2 -Entwicklungen in Olive und PAL nur leicht geändert sind, ist ein PBS-bedingter Effekt wahrscheinlich. Abbildung 3-28: Aufzeichnung von PI-Kurven an Hand der Nettosauerstoffproduktion in ganzen Zellen des WT, CԐ, Olive, DM, TM und PAL. Kulturen wurden in blauem Licht (s.abb B) angezogen. Anregung erfolgte mit verschiedenen Intensitäten von rotem Licht (Abb. 3-29). (n = 5; t.test α = 0.05) Zusammenfassung Ein Wechsel des Anzuchtlichtes von Rot zu Blau resultiert in einer deutlichen Anpassung auf Ebene der Pigmente. Diese wirkt sich insbesondere in WT und CԐ auf die Elektronentransporteigenschaften in der TYM aus und ist in den PBS-Mutanten geringer ausgeprägt. Die verwendete Lichtqualität hat einen entscheidenden Einfluss auf die Photosyntheseleistung. 86
97 Ergebnisse 3.6 Die Produktionsraten der photosynthetischen Wasserspaltung Zur genauen Evaluierung der Prozesse, die zu niedrigeren O 2 -Produktionsraten in den PBS- Mutanten führen, werden die Raten der photosynthetischen Wasserspaltung mit denen der O 2 -Entwicklung verglichen. Die Raten der Wasserspaltung zeigen die tatsächliche Photosyntheseleistung, während durch die O 2 -Entwicklung nur die Elektronen erfasst werden, die nicht in O 2 -verbrauchenden Prozessen genutzt werden. Mit der im Folgenden beschriebenen Methode können an Hand der Chl a Fluoreszenz die am PS2 transportierten Elektronen berechnet werden. Diese ETR entsprechen den Raten der Wasserspaltung. Für die Berechnung der ETR muss jedoch bekannt sein, wie viel Licht pro PS2 genutzt wird. In der Abbildung 3-29 ist das Spektrum des für die Messungen verwendeten Anregungslicht der PAM mit einem Maximum bei ca. 665 nm gezeigt. Da die PBS im Wellenlängenbereich von 665 nm absorbieren, resultiert die Verkleinerung der PBS in einer verringerten Absorption des Anregungslichts. Daraus ergibt sich eine veränderte Energieweiterleitung an das Abbildung 3-29: Spektrum des für die Messungen verwendeten Anregungslicht der PAM mit einem Maximum bei ca. 665 nm. Der Vergleich mit dem Absorptionsspektrum von WT und DM zeigt eine veränderte Lichtausnutzung durch die PBS Verkleinerung. PS2 in den einzelnen Mutanten und dem WT, die für die Berechnung berücksichtigt werden muss. Für die Messungen der Chl a Fluoreszenz müssen die Kulturen auf den gleichen Chlorophyllgehalt normiert werden. Durch die Verschiebung des PS2/PS1 Verhältnisses und die unterschiedliche Chl a Konzentration pro Zellen werden, bei einer Normierung auf Chlorophyll, unterschiedliche Mengen von PS2 für die Messung eingesetzt. Die exakte Berechnung der ETR setzt somit die Klärung grundlegender Faktoren voraus. Diese sind: Lichtabsorption der Kulturen Energieweiterleitung an das PS2 PS2/PS1 Verhältnis, bzw. eingesetzte Menge von PS2 87
98 Ergebnisse Die Raten des Elektronentransports am PS2 Zur Bestimmung der tatsächlich für die Photosynthese genutzten Strahlung wurden in Kooperation mit Ulrich Schreiber Messungen der antenna cross section vorgenommen. Hierbei kann aus der Anfangssteigung von Fluoreszenzinduktionsmessungen, nach einer speziell entwickelten Fit-Routine [108], die Zeitkonstante für die Q A -Reduktion bestimmt werden. Aus dieser Konstante ergibt sich, nach Einsetzen in Formel H, Sigma(II), die sogenannte funktionale PS2 cross section [113,114]. H: ( ) ( ) Mit k(ii): Zeitkonstante für QA-Reduktion; L: Avogadrokonstante; PAR: Photosynthetisch aktive Strahlung Sigma(II) ist dabei spezifisch für die in der Messung verwendete Wellenlänge des Lichtes und ändert sich mit Kultivierungsbedingungen und verwendeter Lichtfarbe. In der Tabelle 3-6 sind die Ergebnisse der Messungen für Sigma(II) in den für die drei Antennengrößen exemplarischen Kulturen WT, Olive und PAL dargestellt. Tabelle 3-6: Sigma(II) für WT, Olive und PAL zur Bestimmung der effektiven Lichtnutzung in den Mutanten mit unterschiedlichen Antennengrößen. Ergebnisse für Licht der Wellenlängen 440 und 665 nm. Sigma(II) Stamm Sigma(II) 665 nm 2 [nm ] Sigma(II) 440 nm 2 [nm ] WT 7 1,05-1,1 Olive 1,4 1,05-1,1 PAL 0,5 1,05-1,1 Für rotes Licht (665 nm), welches in der Regel auch für Messungen der Chlorophyllfluoreszenz genutzt wird, ergeben sich die erwarteten Abstufungen der Energieausnutzung im WT und den PBS-Mutanten. Im WT, mit vollständigen PBS, wird mit einem Wert von 7 nm 2 für Sigma(II) erwartungsgemäß die größte Menge des roten Lichts absorbiert und für die PS2-Anregung genutzt. In der Olive ohne PC wurde ein stark verkleinerter Wert von 1,4 nm 2 und in der PAL ohne PBS der geringste Wert mit 0,5 nm 2 gemessen. Bei blauem Licht (440 nm), welches vornehmlich Chlorophyll anregt, sind keine Unterschiede zwischen WT und Antennenmutanten festzustellen, da hier eine Verkleinerung der PBS keinen Einfluss auf die Lichtabsorption hat. 88
99 Ergebnisse Aus den Werten für Sigma(II) konnten nach Formel I für jede Mutante, entsprechend ihrer PBS-Größe, die Lichtquanten-Absorptionsrate am PS2-Reaktionszentrum [Quanten/(PS2 s)] berechnet werden [113,114] I: ( ) [ ] ( ) Mit Sigma(II)λ: Effektive PS2 cross section [nm 2 ]; L: Avogadrokonstante; PAR: Photosynthetisch aktive Strahlung. Wird PAR(II) in Gleichung J eingesetzt, ist es möglich aus den in Chlorophyllfluoreszenzmessungen ermittelten Werten für die PS2-Aktivität Elektronentransportraten (ETR) in Elektronen/PS2 s zu berechnen [113,114]. In diese ist die veränderte Lichtausnutzung mit einbezogen. J: ( ) [ ] ( ) ( ) ( ) Mit Y(II): Effektive Quantenausbeute am PS2 bei Belichtung; Y(II) max: Maximale effektive Quantenausbeute am PS2 in der Dunkelheit. In der Abbildung 3-30 sind die, aus der Chlorophyllfluoreszenz berechneten, ETRs für den WT und die Mutanten gegen die verwendetet Lichtintensität aufgetragen. Die Betrachtung der ETRs bei niedrigen Lichtintensitäten bis ca. 500 µe zeigt ein mit den O 2 - Lichtsättigungskurven vergleichbares Ergebnis. WT und CԐ zeigen einen raschen Anstieg ihrer ETR, während die Anfangssteigung der Kurven in den PBS-Mutanten verringert ist. Eine verringerte Absorption von Anregungsenergie in den PBS-Mutanten wirkt sich also besonders bei niedrigen Lichtintensitäten aus. Im Gegensatz zu den O 2 -Lichstättigungskurven kann für den ET am PS2 ein positiver Effekt der Entkopplung festgestellt werden. Sowohl die maximalen ETR in der CԐ im Vergleich zum WT, als auch die in der DM im Vergleich zur Olive sind deutlich erhöht. Zu sehen ist auch, dass in den beiden Mutanten Olive und DM erst bei sehr hohen Lichtintensitäten die maximalen ETR erreicht werden. Im direkten Vergleich Olive zu WT und DM zu CԐ sind diese zwar etwas geringer, zeigen aber, dass die Mutanten mit kleineren PBS besser gegen Photoinhibition geschützt sind. Im Gegensatz zu den O 2 -Lichtsättigungskurven (s. Abb. 3-28) konnten für die TM deutlich geringere ETR als in der Olive gezeigt werden. Dies kann ein Hinweis auf einen Verbrauch der am PS2 produzierten Elektronen in anderen Prozessen als der CO 2 -Fixierung sein. 89
100 Ergebnisse In der PAL wurden, wie bereits in den Sauerstoffmessungen gezeigt werden konnte, nur geringe photosynthetische Aktivitäten gemessen. Im Vergleich zu WT und Olive wird der Effekt der PBS-Inaktivierung auf die Photoinhibition deutlich. In der PAL ohne PBS tritt die Lichtsättigung erst bei den höchsten Lichtintensitäten von 2000 µe ein, während diese im WT bei 500 µe und in der Olive bei ca µe eintritt. Abbildung 3-30: Aus Sigma(II) berechnete Elektronentransportraten für den WT und die Mutanten Olive, PAL, DM, TM und CԐ (n = 5) Vergleich der ETR aus O 2 - und Fluoreszenz-Messungen Zur Evaluierung des Elektronenverlusts zwischen PS2 und der CO 2 -Fixierung wurden die Ergebnisse aus O 2 - und Fluoreszenz-Messungen gegenübergestellt. Hierfür mussten die aus der Fluoreszenz ermittelten ETR in mögliche Sauerstoffproduktionsraten umgerechnet werden. Dazu wurde zunächst nach Formel K die Anzahl der Eingesetzten Chl a-moleküle berechnet. K: [ ] Mit A: Chl a-konzentration [mol/ml]; L: Avogadrokonstante. 90
101 Ergebnisse Nach Einsetzen von C und den PS2 zu PS1 Verhältnissen (EPR-Messungen Kapitel 3.2.3) in Gleichung L ergab sich die Menge an PS2 pro ml. L: Mit PS2/PS1 aus EPR Messung; C: Chla pro ml; 0,3 = Chlorophyllverhältnis PS2/PS1; 35 = Chlorophyllmoleküle am PS2. Aus den in Gleichung L berechneten Mengen an PS2 pro ml und den in Gleichung J berechneten ETR pro PS2 konnte nach Gleichung M die Sauerstoffproduktion pro ml berechnet werden. M: ( ) Mit L: Avogadrokonstante; 3600: Umrechnung in h; 4: Transport von 4 Elektronen für ein Molekül O 2. In der Abbildung 3-31 sind die aus den Fluoreszenz- und den O 2 -Messungen resultierenden Lichtsättigungskurven von WT und den PBS-Mutanten Olive und PAL gegenübergestellt. Die hier gezeigten O 2 -Produktionsraten wurden, zum Vergleich mit den Fluoreszenzdaten, nicht auf die Zellzahl, sondern auf die Chlorophyllkonzentration von 10 µg/ml normiert. Abbildung 3-31: Vergleich von theoretischer O 2-Entwicklung berechnet aus der Chlorophyllfluoreszenz (ETR F) und tatsächlicher O 2-Entwicklung aus Sauerstoffmessungen (ETR O) für WT, Olive, und PAL. Anregung der Photosynthese erfolgte mit Licht bei 660 nm (n = 5). Der Vergleich der theoretischen O 2 -Produktionsraten aus den Fluoreszenzdaten (ETR F ) von WT, mit 384 µmol O 2 mgchla -1 h -1 und PAL, mit 468 µmolo 2 mgchla -1 h -1, zeigt, dass die Erhöhung des PS2/PS1 Verhältnis in der PAL nur zu einer geringen Steigerung der ETR beitragen kann. Wird die Erhöhung des PS2/PS1 Verhältnis dagegen, wie in der Olive, durch eine Erhöhung der PS-Menge verursacht, wird der ETR der Photosynthese, im Vergleich zum 91
102 Ergebnisse WT, mit 1443 µmol O 2 mgchla -1 h -1 mehr als verdreifacht. Dies zeigt deutlich, dass nicht die Erhöhung des PS2/PS1 Verhältnis, sondern nur die Erhöhung von PS2 die Photosyntheseraten steigern kann. In der Abbildung 3-32 sind die theoretischen O 2 -Produktionsraten von den Mutanten mit Entkopplung, CԐ, DM und TM, gezeigt. Der ETR ist in der CԐ mit 559 µmol O 2 mgchla -1 h - 1 im, Vergleich zum WT in Abbildung 3-31, erhöht und zeigt, dass die Entkopplung zu einer Steigerung des ETR beiträgt. Diese ist jedoch nicht so stark, wie durch die Erhöhung der PS2-Menge in der Olive. Die in der Olive gemessenen ETR, können jedoch durch die Entkopplung in der DM weiter auf 1931 µmolo 2 mgchla -1 h -1 erhöht werden. Die Inaktivierung von Ycf4 führt, möglicherweise durch die gezeigten Probleme am PS1 (s ), zu einer deutlichen Verringerung der in Olive und DM gemessenen ETR (26 % von ETR in DM). Abbildung 3-32: Vergleich von theoretischer O 2-Entwicklung berechnet aus der Chlorophyllfluoreszenz (ETR F) und tatsächlicher O 2-Entwicklung aus Sauerstoffmessungen (ETR O) für CԐ, DM, und TM. Anregung der Photosynthese erfolgte mit Licht bei 660 nm (n = 5). 92
103 Ergebnisse Die Ergebnisse in Abbildungen 3-31 und 3-32 zeigen deutliche Differenzen zwischen den Abbildung 3-33: Differenz von ETR F und ETR O in Prozent von ETR F. theoretischen und den tatsächlichen O 2 - Entwicklungen. Zur besseren Übersicht sind in der Abbildung 3-33 die Differenzen von ETR F und ETR O in Prozent von ETR F dargestellt. Im WT werden um die 30 % der Elektronen aus der Wasserspaltung in anderen Prozessen als der CO 2 -Fixierung verbraucht. In der CԐ, mit erhöhten Wasserspaltungsraten, ist der Verlust von Elektronen in anderen Prozessen auf 50 % gesteigert. In Olive werden 85 % und in der DM 90 % der Elektronen nicht in der CO 2 -Fixierung verbraucht. In PAL und TM ist ein vergleichbarer Elektronenverlust von ca. 68 % zu beobachten. Die Differenzen zeigen, dass eine Steigerung der photosynthetischen Wasserspaltung in einem Elektronenüberschuss resultiert, welcher vom Zellmetabolismus nicht genutzt werden kann. Die in diesem Abschnitt vorgestellten Ergebnisse zeigen, dass die Olive und die DM die höchsten Wasserspaltungsraten aufweisen. Diese Mutanten scheinen somit das größte Potential für eine H 2 -Produktion zu besitzen Messung der photosynthetischen Wasserspaltung In Zusammenarbeit mit Dr. Pierre Richaud wurden am CEA Cadarache MIMS-Messungen durchgeführt. Wie bereits beschrieben (s ), ermöglicht diese Methode, durch den Vergleich der Zu- und Abnahme der O 2 Isotope 18 O 2 und 16 O 2, die Messung der tatsächlichen Wasserspaltung und des tatsächlichen O 2 -Verbrauches in der Photosynthese. Sie ist damit die einzige Methode, die es erlaubt die, zuvor aus der der Chl a-fluoreszenz ermittelten, theoretischen O 2 -Entwicklungen und Verbräuche zu überprüfen (s ). Für die Messungen wurden die Kulturen mit dem in Abbildung 3-34 gezeigtem, weißem Licht angezogen. Die verwendete Intensität betrug 100 µe. In Abbildung 3-35 und Tabelle 3-7 ist der O 2 -Verbrauch bei Belichtung dargestellt. In den Mutanten Olive, DM, TM und PAL weicht der gemessene O 2 -Verbrauch von den berechneten Elektronenverlusten (s. Abbildung 3-33) ab. Die größten Abweichungen sind bei 450 µe festzustellen. Hier liegt der O 2 -Verbrauch mit 55, 65 und 18 % für Olive, DM und PAL unter dem theoretisch berechneten. In der TM liegt der gemessene O 2 -Verbrauch mit 77 % jedoch 93
104 Ergebnisse deutlich über dem theoretischen. Für die Berechnungen des theoretischen Verbrauchs wurden die maximalen ETR verwendet, die in diesen Mutanten bei Lichtintensitäten von 1500 µe und höher erreicht werden. Der gemessene O 2 -Verbauch bei 1500 µe ist also eher für eine Überprüfung der berechneten Werte geeignet. Der geringe O 2 -Verbrauch von ca. 20 % in der PAL, sowohl bei 450 als auch bei 1500 µe, lässt eine fehlerhafte Berechnung von ETR F vermuten. Die gemessenen Werte bestätigen jedoch die zuvor vermutete, geringe photosynthetische Aktivität in der PAL. Der für Olive und DM gemessene O 2 -Verbrauch bei 1500 µe liegt zwar unter dem berechneten, bestätigt jedoch den, im Vergleich zum WT, erhöhten Elektronenverlust. Zudem wird der gesteigerte O 2 -Verbauch durch die Entkopplung bestätigt. Der Elektronenverlust ist somit, wie bereits theoretisch gezeigt, in der DM am höchsten. Eine erhöhte Elektronenübertragung (55 %) auf O 2 kann ebenfalls für die TM gezeigt werden. Zudem wird die, im Vergleich zu Olive und DM, beschleunigte Photoinhibition durch die Verringerung des Abbildung 3-34: Spektrum des Anzuchtlichtes für die MIMS-Messung. Elektronenverlustes bei Steigerung der Lichtintensität bestätigt. Im WT beträgt der O 2 - Verbrauch bei Belichtung mit 450 µe ca. 40 % der photosynthetischen O 2 -Entwicklung. Eine Erhöhung der Lichtintensität auf 1500 µe senkt den O 2 -Verbrauch auf ca. 30 %. Dies spiegelt die im WT bei diesen Lichtintensitäten beobachteten Effekte der Photoinhibition wider. Die gemessene O 2 -Aufnahme zwischen 30 und 40 % bestätigt die für den WT berechneten Elektronenverluste von ca. 30 % und damit auch die Berechnung von ETR F im WT. 94
105 Ergebnisse Tabelle 3-7: Übertragung von Elektronen auf O 2. Angabe in Prozent von der photosynthetischen O 2-Entwicklung Stamm/ µe WT Olive DM TM PAL ± 1,5 55 ± 7 65 ± 4,2 77 ± 4,5 18 ± ,5 ± 4,5 60 ± 1,7 75 ± 2,7 55 ± 1,7 21 ± 1,4 Abbildung 3-35: Übertragung von Elektronen auf O 2 während der Belichtung. Angabe in Prozent von der photosynthetischen O 2-Entwicklung (n=2) Der Elektronentransport mit Methyl Viologen Die Ergebnisse aus den Messungen der Donor- und Akzeptor-Limitierung (s und ) zeigen, dass sowohl der zyklische ET als auch eine herabgesetzte Elektronenabnahme den ET am PS1 beeinflussen. Hohe Raten des zyklischen ET führen zu einer Reduzierung von PQ und haben so möglicherweise einen Einfluss auf die ETR am PS2, eine verringerte Abnahme von Elektronen am PS1 verlangsamt die komplette Elektronentransportkette. Um die Auswirkungen auf die Elektronentransportleistung des PS2 zu untersuchen, wurden die ETR aus Fluoreszenzmessungen mit und ohne Zugabe von 500 µm MV verglichen. MV ist ein Elektronenakzeptor am PS1 und gewährleitet so eine Elektronenabnahme. Zudem wird die Einspeisung von Elektronen in den zyklischen ET inhibiert. Wie in Abbildung 3-36 gezeigt resultiert die Zugabe von MV im WT, der Olive und der PAL in einer geringen Steigerung der ETR. 95
106 Ergebnisse Abbildung 3-36: Vergleich der an Hand der Chl a Fluoreszenz berechneten ETR (ETR F). Gezeigt sind Messungen mit ganzen Zellen mit und ohne Zugabe von 500 µm MV (n = 5). In Olive und PAL mit niedrigen Raten des zyklischen ET ist nur eine geringe, im WT mit hohen Raten des zyklischen ET eine hohe Steigerung der ETR zu beobachten. In der TM, in der aus den Messungen von N(D) höhere Raten des zyklischen ET vermutet werden können, ist ein deutlicher Anstieg der ETR nach Zugabe von MV zu erkennen. Hier ist aber möglicherweise auch ein Einfluss einer erhöhten Elektronenabnahme am PS1 zu erkennen. Im Gegensatz zu den andern Kulturen verringert die Zugabe von MV zu der CԐ und der DM die ETR. Dieser Effekt ist in der CԐ besonders stark. In der DM führt die Zugabe von MV bei Lichtintensitäten bis ca. 800 µe zu gesteigerten ETR. Bei höheren Intensitäten sind sie jedoch deutlich verringert. Für beide Mutanten wurden zuvor (s. Abbildung 3-20) deutlich erhöhte Raten des zyklischen ET gezeigt. Es wurden also deutliche Steigerungen des ETR erwartet. Dass dies nicht der Fall ist, deutet an, dass die hohen Raten des zyklischen ET möglicherweise eine Schutzfunktion des PS2 darstellen. Diese kann aus der Produktion von ATP bestehen oder durch eine verstärkte Reduzierung des PQ-Pools Anpassungseffekt wie die state transitions hervorrufen. 96
107 Ergebnisse Zusammenfassung Die Berechnung der theoretischen O 2 -Entwicklung zeigt den positiven Effekt der Entkopplung auf den PET. Die Erhöhung des PS2/PS1 Verhältnis durch die Erhöhung der PS2-Menge in der Olive steigert die ETR jedoch wesentlich deutlicher. Die Kombination von Entkopplung und erhöhter PS-Menge in der DM ergibt die höchsten ETR. Der Vergleich mit den Ergebnissen aus MIMS-Messungen mit O 2 -Isotopen bestätigt die Ergebnisse der Chl a Fluoreszenzmessung und zeigt, dass die erhöhte Wasserspaltungsrate nicht zu erhöhten O 2 - Produktionsraten führt. Die Elektronen werden also erneut auf O 2 übertragen. Messungen mit MV legen eine Schutzfunktion des zyklischen ET bei der Photoinhibition nahe. 3.7 Langzeitadaption Kultivierung im Flachbett-Bioreaktor Die in den Kapiteln zuvor gezeigten Ergebnisse stellen eine kurzfristige Charakterisierung der ET-Leistung dar, die das Potential der Mutanten für eine photobiologische H 2 -Produktion zeigt. Für eine industrielle H 2 -Produktion müssen dauerhaft hohe ET-Leistungen erzielt werden. Eine Auswahl geeigneter Mutanten zur Etablierung eines Design-Organismus erfordert daher eine langfristige Charakterisierung des PET. Abbildung 3-37: Spektrum der für die Kultivierung im Fermenter genutzten, blaugrünen LEDs. Die langfristige Charakterisierung erfolgte im Flachbettbioreaktor über die O 2 -Entwicklung der Kulturen bei Belichtung mit blaugrünem Licht. Das Spektrum der verwendeten LEDs ist in der Abbildung 3-37 dargestellt und die Absorptionseigenschaften der Kulturen entsprechen den in Abbildung 3-23 gezeigten. Da die vorherigen Ergebnisse in der DM die höchsten ET- 97
108 Ergebnisse Leistungen gezeigt haben, wurde diese Mutante für die Charakterisierung im Bioreaktor ausgewählt. Die im Folgenden gezeigten Ergebnisse für die DM wurden mit den von Jong- Hee Kwon erarbeiteten Ergebnissen für WT und Olive verglichen Die ideale Lichtintensität Zur Erzielung des höchst möglichen PET wurden die optimalen Lichtbedingungen für die Photosynthese bestimmt. Dazu wurden die Zellen im Flachbett-Bioreaktor kontinuierlich mit einer OD 750 von 0,8 kultiviert. Während der Kultivierung wurde die Lichtintensität in 100 µe- Schritten von 100 µe auf 400 µe erhöht. Bei jeder Lichtintensität erfolgte eine mehrtägige Kultivierung mit paralleler Aufzeichnung der O 2 -Entwicklung. Wie in der Abbildung 3-38 zu sehen, zeigen WT und Olive bei 100 µe und 200 µe vergleichbare O 2 -Entwicklungen. Abbildung 3-38: Bestimmung der optimalen Lichtintensität für den PET in WT, Olive und DM im Flachbettbioreaktor. Eine Steigerung der Lichtintensität auf 300 µe resultiert in der Olive in einer weiteren Steigerung der O 2 -Entwicklung auf 5 e -9 µmol O 2 und im WT auf 4 e -9 µmol O 2 pro Zelle. Die Ausgleichsgerade deutet jedoch eine optimale Lichtintensität zwischen 200 und 300 µe an. Eine Steigerung auf 400 µe stellt sowohl für WT als auch für Olive eine Stressbedingung dar, so dass die gemessenen O 2 -Entwicklungen abfallen. In der DM wurden bei allen Lichtintensitäten höhere O 2 -Entwicklungen als in WT und Olive gemessen. Die größte Differenz wurde bei 100 µe festgestellt, so dass sich ein positiver Effekt der Entkopplung besonders bei niedrigen Lichtintensitäten einzustellen scheint. Bei 200 µe und 300 µe zeigt 98
109 Ergebnisse die gemessene O 2 -Entwicklungen im Vergleich zur Olive nur leicht erhöht Werte. Eine Erhöhung der Lichtintensität auf 400 µe stellt ebenfalls in der DM eine Stresssituation dar, welche auf Dauer zu einem Abfall der O 2 -Entwicklung führt Die ideale Zelldichte Die Freisetzung von Elektronen bei der Photosynthese wird im Fermenter nicht nur von der Lichtintensität, sondern auch von der optischen Dichte und damit der Zellzahl beeinflusst. Zur Bestimmung der OD mit den höchsten Raten des PET wurden so genannte batch - Kultivierungen bei den Lichtintensitäten 100 und 400 µe durchgeführt. Die Abbildung 3-39 zeigt die gemessenen O 2 -Entwicklungen aufgetragen gegen die OD 750. In Abbildungsteil A sind die Ergebnisse für die Kultivierung bei 100 µe zu sehen. In WT wurde die höchste O 2 -Entwicklungen und damit die optimale Zelldichte bei einer OD 750 von ca. eins festgestellt. In der Olive ist die optimale Zelldichte auf eine OD 750 von 1,5 erhöht. Dies ist ein Hinweis auf einen verbesserten Lichteintrag im Fermenter durch die Verkleinerung der PBS. Zusätzlich zur Erhöhung der optimalen OD wurden in der Olive, im Gegensatz zu den zuvor gezeigten Ergebnissen (Abbildung 3-38), bei 100 µe im Vergleich zum WT erhöhte O 2 - Entwicklungen gemessen. Dies zeigt den positiven Effekt der Optimierung der Zelldichten auf die Gesamt-O 2 -Entwicklung im Fermenter. Wie bereits in Abschnitt wurden in der DM bei 100 µe deutlich erhöhte O 2 -Entwicklungen gemessen. Hier ist im Vergleich zum WT eine Verringerung der optimalen Zelldichte auf eine OD 750 von ca. 0,8 zu beobachten, so dass ein gesteigerter Lichtbedarf durch die Entkopplung zu vermuten ist. In Teil B der Abbildung 3-39 sind die bei der Kultivierung mit 400 µe erzielten Ergebnisse zu sehen. Die Erhöhung der Lichtintensität resultiert in allen Kulturen in einer Steigerung der optimalen OD. Dies zeigt, dass ab einer bestimmten Kulturdichte Licht der limitierende Faktor für die O 2 -Entwicklung im Fermenter ist. Im WT ist die optimale OD auf ca. zwei erhöht. Ähnlich den Ergebnissen in Teil A ist in der Olive im Vergleich zum WT eine Erhöhung der optimalen OD auf ca. 2,5 festzustellen. Zudem zeigt die Olive, wie aus den Ergebnissen in Abbildung 3-38 zu erwarten, höhere O 2 -Entwicklungen als der WT. In der DM wurden bei 400 µe die höchsten O 2 -Entwicklungen gemessen. Im Gegensatz zu den bei 100 µe erhaltenden Ergebnissen ist die optimale OD in der DM im Vergleich zur Olive erhöht und liegt bei ca. 3,5. Die beiden getesteten Lichtbedingungen zeigen somit einen veränderten Einfluss auf die O 2 -Entwicklung in Olive und DM. Zur Ermittlung der Ursachen dieses Effektes wurden in der Abbildung 3-39 Teil C und D die O 2-99
110 Ergebnisse Entwicklung im Fermenter mit dem Wachstum von DM verglichen. Sowohl bei 100 µe als auch bei 400 µe wurden die höchsten O 2 -Entwicklungen in der Mitte der exponentiellen Wachstumsphase gemessen. Die Erhöhung der optimalen OD spiegelt also ein verbessertes Wachstum wieder. Dies bestätigt die Vermutung eines erhöhten Energiebedarfs in der DM. Abbildung 3-39: Bestimmung der OD 750 mit den höchsten Raten des PET. A + B: O 2-Entwicklung in Abhängigkeit von der OD bei 100 und 400 µe in WT, Olive und DM. C + D: Vergleich von Wachstumskurve und O 2-Entwicklung in der DM bei 100 µe und 400 µe. Die Korrelation von Wachstum und O 2 -Entwicklung in der DM lässt einen vergleichbaren Zusammenhang für WT und Olive vermuten. In der Abbildung 3-40 wird das Wachstums von WT, Olive und DM bei 100 µe und 400 µe verglichen. 100
111 Ergebnisse Abbildung 3-40: Vergleich des Wachstums von WT, Olive und DM bei 100 und 400 µe an Hand der OD bei 750 nm. In WT und Olive ist bei beiden Lichtintensitäten ein vergleichbares Wachstumsverhalten zu beobachten, was die nur geringen Verschiebungen der optimalen OD in WT und Olive erklärt. Dies spiegelt sich auch in den Verdopplungszeiten von WT und Olive wider (Tabelle 3-8). Dabei liegen die höchsten O 2 -Entwicklungen im WT leicht unterhalb, in der Olive leicht oberhalb der Mitte des exponentiellen Wachstums und deuten einen verbesserten Lichteintrag im Fermenter für die Olive an. Tabelle 3-8: Verdopplungszeiten in der exponentiellen Wachstumsphase von WT, Olive und DM. Stamm WT Olive DM Licht [h] [h] [h] 100 µe 75, ,8 400 µe 45,4 38,2 109,8 Die DM zeigt bei beiden Lichtintensitäten ein deutlich verlangsamtes Wachstum mit Verdopplungszeiten von 140,8 und 109,8 h bei 100 und 400 µe. Dies steht im Widerspruch zu den gemessenen hohen O 2 -Entwicklungen, da diese normalerweise mit hohen Raten der CO 2 -Fixierung und damit mit einem erhöhten Wachstum korrelieren. 101
112 Diskussion 4 Diskussion Heutige Verfahren der photobiologischen Treibstoffproduktion basieren auf der direkten oder indirekten Kopplung an die Biomasseproduktion der Photosynthese. Die Effizienz der Biomasseproduktion und damit solcher Systeme wird jedoch von zahlreichen energieverbrauchenden, metabolischen Prozessen limitiert [115]. Die direkte Kopplung einer Biogasproduktion, wie die H 2 -Produktion, an den Elektronentransport der Photosynthese kann diese Limitierungen umgehen und zu einer deutlichen Steigerung der Produktionseffizienz führen [ ]. Für die photobiologische H 2 -Produktion über Hydrogenasen existieren drei Methoden mit Potential für eine hohe Effizienz [118]. Auf Grund der O 2 -Sensitivität der Hydrogenasen ist eine direkte Kopplung von Photosynthese und H 2 -Produktion im Moment schwierig. Eine Alternative ist die Kopplung der H 2 -Produktion an die N 2 -Fixierung durch Nitrogenasen. Die Ausbildung spezialisierter Zellen, den Heterozysten, erlaubt eine H 2 -Produktion im O 2 -freien Raum [119]. Der hohe, zelluläre Energieaufwand und die geringe Anzahl von Heterozysten macht diese Methode jedoch ineffizient [118]. In einer zweiten Alternative wird die Expression der Hydrogenase durch die Eliminierung von O 2 über Respiration und Fermentation induziert. In Synechocystis kann so über die eigene NiFe-Hydrogenase zu Beginn einer Belichtung H 2 produziert werden [119]. In Algen, wie Chlamydomonas reinhardtii ist bei Schwefelmangel eine kontinuierliche H 2 -Produktion über den Stärkeabbau möglich. Hier wurden in einer Mutante mit erhöhter Stärkeproduktion die bis jetzt höchsten H 2 -Entwicklungen gemessen (3,5 ml h -1 l -1 ) [121]. Bereits vor Beginn dieser Arbeit wurde im Zuge der Projekte Solar-H und Solar-H2 mit dem Design eines Modellorganismus für eine direkte H 2 -Produktion begonnen. Wie in der Abbildung 4-1 zu sehen, sollen zukünftige optimale H 2 -Produktionsraten durch Kopplung einer O 2 -toleranten Hydrogenase an einen optimierten linearen photosynthetischen Elektronentransportes (PET) erzielt werden. Vor dieser Arbeit wurde angenommen, dass drei Faktoren wesentlich zur Optimierung der Raten des PET beitragen: Erhöhung des PS2/PS1-Verhältnisses und der Raten des PET durch Verkleinerung der Antennen Verringerte Photoinhibition und gesteigerte Raten des PET durch Entkopplung Verringerung des zyklischen ET 102
113 Diskussion Abbildung 4-1: Schematische Darstellung eines für die H 2-Produktion optimierten PET. A: ET in Synechocystis. B: Optimierter ET zur H 2-Produktion über eine heterolog exprimierte Hydrogenase. Optimierung wird durch die Erhöhung des PS2/PS1 Verhältnisses, die Verringerung des zyklischen ET und eine Entkopplung erreicht. 4.7 Die Auswirkung verkleinerter Phycobilisomen auf das PS2/PS1 Verhältnis Die Verkleinerung der PBS resultiert nicht nur in einer veränderten Lichtabsorption der Zelle, sondern ebenfalls in einer veränderten Energieweiterleitung zu den Photosystemen. In dieser Arbeit wurde die Energieweiterleitung an das PS2 in WT und den PBS-Mutanten durch die Messung der antenna cross section bestimmt. Wie in Tabelle 4-1 gezeigt, stimmen die dabei ermittelten Werte mit vergleichbaren Ergebnissen in der Literatur überein [133]. Geringe Abweichungen resultieren dabei aus einer Verschiebung der Anregungswellenlänge um 10 nm. Tabelle 4-1: Vergleich der antenna cross section in WT und PBS-Mutanten. Ergebnisse aus dieser Arbeit wurden bei 665 nm bestimmt, die Ergebnisse von Maksimov et al. [133] bei 655 nm. Diese Arbeit Maksimov et al. PBS-Größe [%] [%] APC + PC APC 20 24,6-7,1 9,3 Wie in der Abbildung 4-2 gezeigt, resultiert der Verlust von PC in einer um ca. 75 bis 80 % verringerten Energieweiterleitung zum PS2. Der zusätzliche Verlust von APC verkleinert die 103
114 Diskussion antenna cross section auf 10 % der im WT gemessenen. Die ermittelte signifikante Verringerung der antenna cross section durch den Verlust von PC zeigt, dass PC den Großteil des Lichtes in den PBS absorbiert. Abbildung 4-2: Energieweiterleitung an das PS2 im WT und den PBS-Mutanten Olive und PAL. Verlust von PC in der Olive verringert die Energieweiterleitung um 80 %, das komplette fehlen der PBS in der PAL um 90 %. Dass in blauem Licht (460 nm), für WT und Mutanten, keine Unterschiede der antenna cross section gezeigt werden konnten, macht eine Funktion der erhöhten Carotinoidkonzentration in den PBS-Mutanten als Antennenersatz unwahrscheinlich. Da Carotinoide am Schutz vor exzessiver Anregung durch Auslöschen von Anregungsenergie und dem Abbau der Triplettzustände von O 2 und Chlorophyll beteiligt sind [59,66,134,135], führt die, in den PBS- Mutanten, relativ erhöhte Chlorophyllabsorption möglicherweise zur vermehrten Bildung von Triplettchlorophyll. Die Erhöhung der relativen Chlorophyllabsorption in den PBS-Mutanten verstärkt in erster Linie die Absorption von PS1, da hier dreimal mehr Chl a als an PS2 gebunden ist. Zusammen mit der verringerten Energieweiterleitung an das PS2 resultiert dies in einem stärker oxidierten PQ-Pool, dessen Oxidationszustand das PS2/PS1 Verhältnis reguliert. Dabei gilt: PS1 ist das regulatorische Elemente, die PS2-Menge bleibt eher konstant Die Regulation balanciert Einspeisung und Abnahme von Elektronen in der Elektronentransportkette Eine Akkumulation von PS1 verhindert dabei eine Überreduktion, ein Abbau von PS1 eine zu starke Oxidation des PQ-Pools. Dies steigert die Effizienz des ET und verringert Schäden durch Photoinhibition [ ]. Die verstärkte Oxidation des PQ-Pools in den PBS- Mutanten sollte also zu einem Abbau von PS1 und einem erhöhten PS2/PS1 Verhältnis führen. In der PAL wurde in früheren Studien, durch den relativen Vergleich von PS2 und PS1, ein Anstieg der PS2-Komplexe auf das Drei- bis Fünffache vermutet [40,151,152]. Die 104
115 Diskussion EPR-Messungen in dieser Arbeit zeigen jedoch, dass die relative Bestimmung zu einer Überschätzung der PS-Menge führt. Zwar kommt es in der PAL zu einem Anstieg von PS2, doch ist dieser nur gering im Vergleich zum Abbau von PS1. Die Anpassung in der PAL entspricht somit den zuvor beschriebenen Anpassungen der Zelle an einen oxidierten PQ- Pool [ ]. Im Gegensatz zur PAL kommt es in den PC-Mutanten Olive, DM und TM zu einer fast dreifachen Erhöhung des PS2-Anteils. Möglicherweise ist die Anregung von PS2 und PS1 durch den Verlust von PC ausbalanciert und es kommt vergleichbar zu Höheren Pflanzen zu einem Angleichen der Photosystemmenge. In Höheren Pflanzen führt dies zu einer räumlichen Unterteilung der TYM in Granastapel und Stromalamellen, um so eine dichtere Konzentrierung der Photosysteme in der Membran und eine verbesserte Lichtausnutzung zu ermöglichen [144]. Eine vergleichbare Aufteilung konnte in PC-Mutanten gezeigt werden, in denen, im Gegensatz zum WT, Bereiche in der Membran existieren, die fast ausschließlich PS2 und APC, aber kein PS1 enthalten [143]. Wahrscheinlich dienen die Erhöhung der PS2- Menge und die räumliche Trennung der Photosysteme in der Olive als Ausgleich für die verringerte Lichtabsorption. Die dadurch im Vergleich zum WT fast verdreifachte PS2-Menge in den PC-Mutanten trägt zu einer Steigerung des PET bei. 4.8 Die Funktion von Ycf4 bei der Assemblierung von PS1 Die Inaktivierung von Ycf4 sollte zu einer Erhöhung des PS2/PS1 Verhältnisses beitragen und so die Raten des PET steigern. Die Inaktivierung von Ycf4 führte jedoch zu Problemen bei Elektronenübertragung von PS1 auf Ferredoxin. Dies lässt sich möglicherweise durch einen Verlust von, am ET beteiligten, UE des PS1 erklären. Cross-Linking-Experimente in Synechocystis lassen eine Interaktion von Ycf4 mit der PsaC-UE des PS1 vermuten [69]. Ein Verlust von PsaC ist jedoch unwahrscheinlich, da entsprechende Synechocystis-Mutanten, im Gegensatz zu Ycf4-Mutanten, nicht mehr photoautotroph wachsen [145,146]. In Chlamydomonas wird eine Rolle von Ycf4 bei der Bindung der PsaF-UE an den zuvor gebildeten PS1-Kernkomplex bestehend aus PsaA, PsaB, PsaE, PsaC und PsaD vermutet [112]. PsaF ist in Eukaryoten essentiell für die Bindung von Plastocyanin [147], und seine Inaktivierung unterbindet, vergleichbar mit der Inaktivierung von Ycf4, die Assemblierung von PS1. In Synechocystis hat PsaF dagegen keine Funktion bei der Bindung von Plastocyanin [148], so dass die Inaktivierung, genauso wie das Fehlen von Ycf4, nicht zu einem Ausbleiben der Assemblierung von PS1 führt. Zudem konnte für eine pasf-psaj-mutante von Synechocystis eine verringerte Elektronenabgabe von PS1 an Ferredoxin und, vergleichbar 105
116 Diskussion mit der Mutante ycf4, gesteigerte Raten des linearen ET gezeigt werden [73,149]. Die verringerte Bindung von Ferredoxin am PS1 in der pasf-psaj-mutante erhöhte die Expression des Proteins IsiA [149]. Eine erhöhte Expression von IsiA kann ebenfalls in der TM vermutet werden, da hier eine geringe Verschiebung der Fluoreszenzemission von PS1 in den blauen Bereich und eine hohe Fluoreszenzintensität von PS2 beobachtet wird. Die vergleichbaren Phäntoypen deuten einen Verlust von PsaF durch Inaktivierung von Ycf4 an. Der so verringerte ET am PS1 und die erhöhte Photoinhibition machen die Inaktivierung von Ycf4 zu keinem geeigneten Mittel die ETR zu steigern. 4.9 Die Entkopplung des Elektronentransportes und die Auswirkung auf die Photoinhibition Die Kopplung des PET an eine Protonentranslokalisation in das Thylakoidlumen resultiert im Aufbau eines ph-gradienten. Dieser macht bei hohen Raten des PET die Protonentranslokalisation durch den Cyt-b 6 f-komplex zum geschwindigkeitslimitierenden Schritt des ET [122,123]. In dieser Arbeit wurden zwei Strategien verfolgt, die zu einer effizienten Verringerung des ph-gradienten und so zu gesteigerten ETR führen sollten. Diese Strategien waren: Expression von Channelrhodopsin-2 (Chop-2) Verkürzung der ATP-Synthase Epsilon-Untereinheit Die Expression von Membranproteinen stellt, im Gegensatz zu löslichen Proteinen, für die viele Expressionsstrategien zur Verfügung stehen, eine große Herausforderung dar [124]. Dies liegt in einer komplizierten, aus vielen Zwischenschritten zusammengesetzten, Biogenese von Membranproteinen begründet. Für eukaryotische Membranproteine, wie Chop-2 aus Chlamydomonas, ergeben sich weitere Anforderungen an die Biogenese, die das Ausbleiben der Expression in dieser Arbeit erklären. Für die Biogenese von Chop-2 ist eine N-Glykolisierung essentiell, so dass Störungen oder das Fehlen dieser N-Glykolisierung eine Expression unterbinden [125]. Der Mechanismus der N-Glykolisierung ist jedoch kennzeichnend für Eukaryoten und fehlt in Prokaryoten. Die Expression von Chop-2 setzt somit ein eukaryotisches Expressionssystem voraus und ist in Synechocystis nicht möglich. Für Inaktivierung des C-Terminus der Epsilon-UE der ATP-Synthase in der Mutante CԐ, konnte ein effizienter Abbau des ph-gradienten an der TYM gezeigt werden. Die so eingefügte Entkopplung steigerte nicht nur die Raten des PET, sondern verringerte ebenfalls die Photoinhibition. Als Auslöser der Photoinhibition wird eine Schädigung von PS2 durch die Bildung von ROS angenommen. In dieser Arbeit konnte für die Entkopplung jedoch kein 106
117 Diskussion Einfluss auf die Schädigung, sondern nur auf die Neu-Synthese von PS2 gezeigt werden. Für diese Neu-Synthese von PS2 wird ATP benötigt, welches durch erhöhte Raten des zyklischen ET bereitgestellt wird [128,129]. Zwar wurden in den Mutanten mit Entkopplung ( CԐ, DM) erhöhte Raten des zyklischen ET gezeigt, da jedoch für den WT und die CԐ vergleichbare, zelluläre ATP-Konzentration nachgewiesen wurden, dienen diese nur dem Ausgleich des NADPH/ATP-Verhältnisses [98]. Es ist anzunehmen, dass dies ebenfalls für Olive und DM zutrifft, so dass eine erhöhte Neu-Synthese nicht durch vermehrte ATP Bildung erklärt werden kann. Die Bildung von ROS schädigt nicht nur das PS2, sondern verlangsamt, durch die Schädigung der Proteinbiosynthese, auch seine Neu-Synthese [ ]. Eine beschleunigte Neu-Synthese durch die Entkopplung ist daher auf eine verringerte Schädigung der Proteinbiosynthese zurückzuführen. Durch die verringerte Photoinhibition und die erhöhten ETR trägt eine Entkopplung zu einer Erhöhung der H 2 -Produktion in einem zukünftigen Design-Organismus bei Lichtregulation in Synechocystis Ein möglicher Kandidat für eine lichtregulierte Expression in Synechocystis ist der Promotor von HilC. In Synechocystis liegt HilC bei niedrigen Lichtintensitäten in geringen Mengen vor, bei einem Anstieg der Intensität kommt es jedoch zu einer Akkumulation [96]. Eine Lichtabhängigkeit der Aktivität des Promotors PhilC konnte in dieser Arbeit, an Hand der Expression von GFP, bestätigt werden. Jedoch konnten erhöhte GFP-Mengen nicht nur bei hohen, sondern auch bei niedrigen Intensitäten nachgewiesen werden. Da HilC in Synechocystis Teil der allgemeinen Stressantwort ist, wird die Aktivität seines Promotors neben der Lichtintensität auch durch Stickstoff- und Schwefelmangel sowie niedrige Temperaturen erhöht. Mögliche Stressfaktoren könnten somit eine erhöhte GFP-Menge bei geringen Lichtintensitäten erklären. Können Stressfaktoren ausgeschlossen und die basale Promotoraktivität unterdrückt werden, kann PhilC für eine Lichtregulation genutzt werden. Wie in Abbildung 4-3 dargestellt, könnte dies durch die Expression geringer Mengen antisense -mrna erreicht werden. Die antisens -mrna verhindert die Translation der basal transkribierten mrna, so dass erst die bei hohen Lichtintensitäten verstärkte Transkription von mrna zu einer Expression des Proteins führt. Die Expression von antisense -mrna könnte durch eine auf den Bereich -25 bis +65 bp (relativ vom Startpunkt der Transkription) verkürzte Version von PpetF (Ferredoxin) kontrolliert werden, für die eine nur geringe basale Aktivität gezeigt wurde [94]. 107
118 Diskussion Abbildung 4-3: A: Durch geringe Expression von antisense -mrna wird die Translation von basal exprimierter mrna unterdrückt. B: Die Erhöhte Transkription von mrna übersteigt die antisense -mrna, es kommt zur Expression des Proteins Die state transitions und der zyklischer ET Wie in Kapitel und beschrieben, sind die Mechanismen der state transitions und des zyklischem ET in Cyanobakterien nicht im Detail geklärt. Es ist jedoch bekannt, dass: Beide Prozesse abhängig vom Redoxzustand des PQ-Pools sind Ein reduzierter PQ-Pool die Anregung von PS1 ( state 2 ) erhöht Ein reduzierter PQ-Pool die Raten des zyklischen ET erhöht [37,129,153,154] Eine gemeinschaftliche Funktion beider Prozesse ist daher wahrscheinlich. Dabei fungiert der zyklische ET als Schutzmechanismus des PS2, der möglicherweise die state transitions als weitere Schutzmaßnahme induziert [155]. Für den zyklischen ET wurden in dieser Arbeit drei wesentliche Ergebnisse erzielt: Eine Entkopplung erhöht die Raten des zyklischen ET Die Verkleinerung der PBS senkt die Raten des zyklischen ET Die Raten des zyklischen ET variieren mit der Lichtqualität Die Ergebnisse dieser Arbeit zeigen, dass nicht nur die Lichtqualität, sondern auch die Anregungsweiterleitung an das PS1 einen Einfluss auf die Raten des zyklischen ET haben, und machen so eine Beteiligung der state transitions wahrscheinlich. Ein solcher 108
119 Diskussion Zusammenhang von state transitions und zyklischem ET konnte in Chlamydomonas bereits nachgewiesen werden [156,157]. Dabei kommt es in Chlamydomonas zur Bildung eines am zyklischen ET beteiligten Superkomplex, bei dem beide Lichtsammelantennen (LHC) ihre Anregung an das PS1 weitergeben. Vergleichbar mit Chlamydomonas wird die Bildung von Superkomplexen ebenfalls für Höhere Pflanzen vermutet [ ] und ist, unter Beteiligung von NDH-1 und der FNR, auch für Cyanobakterien anzunehmen. Eine vom Redoxzustand des PQ-Pools abhängige Komplexbildung der respiratorischen Proteine in der TYM von Synechocystis bestärkt diese Vermutung zusätzlich [164]. Im Gegensatz zu den LHCs von Chlamydomonas konnte die Bindung der PBS an PS1 in Synechocystis allerdings bis jetzt nicht gezeigt werden. Analog zu Höheren Pflanzen existiert jedoch in Synechocystis eine zweite Antenne, die Anregung bevorzugt an das PS1 weiterleitet [63,159]. Diese Antenne enthält nur Phycocyanin und fehlt somit in den, in dieser Arbeit verwendeten, PBS-Mutanten. Vergleichbare Raten des zyklischen ET in Olive und PAL, sowie die verringerten Raten im WT, nach Kultivierung im blauen Licht und den dadurch bedingten Abbau von PC [160], machen eine Funktion dieser Antenne am zyklischen ET wahrscheinlich. Wird die Energieweiterleitung an das PS1 nur durch diese zweite Antenne bestimmt, ist die Konsequenz daraus, dass es bei den state transitions nicht zu einer Bindung der PBS am PS1 kommt. Dies würde, wie in der Abbildung 4-4 gezeigt, für das in der Einleitung beschriebene detachment -Modell sprechen (s ), bei dem die state transitions nur durch Ablösen und Binden der PBS an PS2 ausgelöst werden. Das Ausbleiben von state transitions in der TM kann jedoch nicht allein durch dieses Modell erklärt werden. Möglicherweise liegt in der TM ein veränderter Oligomerisierungszustand von PS1 vor, welcher einen zusätzlichen Effekt des spill over verhindert. Eine Rolle des Oligomerisierungszustand der Photosysteme bei den state transitions wird diskutiert [151,152]. Für den zyklischen ET konnte zudem in dieser Arbeit eine Zunahme mit steigenden Lichtintensitäten gezeigt werden, bei denen er verstärkt mit einer eingebrachten Hydrogenase um Elektronen konkurrieren würde. Dies spricht für die Annahme, dass eine Verringerung des zyklischen ET die Raten der H 2 -Produktion steigert [39,116]. Der zyklische ET hat jedoch ebenfalls eine Funktion beim Schutz vor Photoinhibition, da durch ihn das, für die Reparatur von PS2 benötigt, ATP produziert wird [128,129]. Eine erhöhte Photoinhibition in Mutanten mit hohem zyklischen ET nach Zugabe von MV (Inhibitor des zyklischen ET), bestätigt dies. Um hohe H 2 -Produktionsraten zu erreichen, werden hohe Raten des linearen 109
120 Diskussion ET benötigt. Diese können jedoch nur bei geringer Photoinhibition aufrechterhalten werden. Der zyklische ET trägt somit zur Gewährleistung hoher H 2 -Produktionsraten bei. Abbildung 4-4: Ein mögliches Modell zur Beschreibung von state transitions und der Auslösung des zyklischen ET. A: state 1 mit Energieweiterleitung zum PS2 und B: state 2 mit Energieweiteleitung nur an das PS1. Der veränderte Redoxzustand des PQ-Pools resultiert in der Bildung eines am zyklischen ET beteiligten Superkomplex Erhöhung des Elektronentransportes Das Ziel dieser Arbeit war die Steigerung der Raten des PET durch die Erhöhung des PS2/PS1 Verhältnisses und die Entkopplung des ET. Die Auswirkungen der eingebrachten Mutationen auf die Raten des PET können dabei an Hand der O 2 -Entwicklung und der Chl a Fluoreszenz charakterisiert werden. Durch eine Elektronenübertragung in der Photosynthese auf O 2 können durch die O 2 -Entwicklung jedoch keine exakten ETR ermittelt werden. Daher wurde in dieser Arbeit eine von Schreiber et al. entwickelte Methode zur Bestimmung von ETR aus der Chl a Fluoreszenz speziell an die geänderte Pigmentzusammensetzung in den verwendeten Mutanten angepasst [169]. Zur Bestätigung der mit dieser Methode ermittelten Ergebnisse wurde der photosynthetische Gasaustausch über Gas- Massenspektrometriemessungen (MIMS) mit den O 2 -Isotopen 18 O 2 und 16 O 2 gemessen. Die 110
121 Diskussion vorläufigen Ergebnisse aus den Messungen des Gasaustausches über MIMS mit den Mutanten zeigen eine hohe Übereinstimmung mit den Werten aus der Chl a Fluoreszenz. Zudem konnte die für den WT berechnete, theoretische Elektronenübertragung auf O 2 mit dem in der Literatur beschriebenen, tatsächlichen O 2 -Verbrauch von % bestätigt werden [169,171]. Schwächen dieser Methode werden jedoch bei hohen Lichtintensitäten deutlich. Hier kommt es durch die state transitions, die bei diesen Intensitäten zum Schutz des PS2 sattfinden [52], zu einer veränderten Energieweiterleitung an das PS2, die noch nicht berücksichtigt werden kann. Bei hohen Lichtintensitäten führt dies zu einer Unterschätzung der ETR aus der Chl a Fluoreszenz in WT und CԐ. Vergleichbar kommt es in den PBS-Mutanten zu einer Überschätzung der Elektronenverluste. Die in dieser Arbeit gezeigte, drei- bis vierfache Steigerung der ETR in Olive und DM geht mit einer hohen Elektronenübertragung auf O 2 einher und konnte so an Hand der O 2 - Entwicklung nicht gezeigt werden. Diese Übertragung von Elektronen auf O 2 ist in Synechocystis in drei Reaktionen möglich: Respiration Photorespiration Mehler-Reaktion Für die Respiration konnte bereits gezeigt werden, dass sie bei Belichtung nur ca. 6 % des Elektronentransportes ausmacht [170]. Einen vergleichbar geringen Anteil hat die Photorespiration, deren Raten nur bei CO 2 -Mangel und hohen Lichtintensitäten erhöht sind [166]. Wahrscheinlich ist daher eine Übertragung auf O 2 in der Mehler-Reaktion. Kommt es zu einer Überreduktion von Ferredoxin/NADPH, z.b. durch CO 2 -Mangel, können in dieser, in Synechocystis über Flavodiiron 1 und 3 (FLV1+3) bis zu 60 % der photosynthetischen Elektronen auf O 2 übertragen werden [ ]. In der Olive und DM kommt es also, vergleichbar zum CO 2 -Mangel, zu einem Elektronenüberschuss, der vom Metabolismus nicht genutzt werden kann. Wie in der Abbildung 4-5 gezeigt, könnten diese Elektronen, über eine heterolog exprimierte Hydrogenase, zur H 2 -Produktion genutzt werden, ohne den Metabolismus zu beeinträchtigen. 111
122 Diskussion Abbildung 4-5: Umleitung der Elektronen aus der Mehler-Reaktion in die H 2-Produktion. A: Mehler-Reaktion in Synechocystis in der über Flv1+3 bis zu 60 % der photosynthetischen Elektronen verbraucht werden. B: Durch Inaktivierung der Proteine Flv1+3 könnten bis zu 60 % der Elektronen der Photosynthese für eine H 2-Produktion über eine heterolge Hydrogenase verwendet werden Veränderte Anforderungen der Kultivierung im Bioreaktor Ein zukünftiger Designorganismus soll mit der Energie der Sonne in einem Bioreaktor höchstmögliche H 2 -Produktionsraten erzielen. Aus den Anforderungen einer solchen H 2 - Produktion ergeben sich zwei wesentliche Probleme: Die Energie des Sonnenlicht übersteigt die Kapazität der Photosynthese (Photoinhibition) [175] Die Kultivierung hoher Zelldichten führt zu Beschattungseffekten und verringert den Lichteintrag im Fermenter Die Charakterisierungen der ETR haben für die Olive und die DM nicht nur die höchsten Photosyntheseleistungen, sondern ebenfalls die höchste Widerstandsfähigkeit gegen Photoinhibition gezeigt. Dies konnte im Flachbettbioreaktor über Langzeitmessungen der O 2 - Entwicklung bei verschiedenen Lichtintensitäten bestätigt werden. Der erhöhte Schutz vor Photoinhibition durch verkleinerte Antennen bestätigt frühere Studien, die diesen Effekt für Algen und Cyanobakterien gezeigt haben [39,41, ]. Die im Bioreaktor gemessenen, hohen O 2 -Entwicklungen stehen jedoch im Widerspruch zu einer Studie, die eine 112
123 Diskussion Verringerung der O 2 -Entwicklung durch den Verlust von PC in der, mit der Olive vergleichbaren, CK-Mutante zeigt [179]. Dies kann über die verwendete Lichtqualität erklärt werden. Die Kultivierungen der CK wurden in weißem Licht (blau 100 und rot 50 µmol Photonen m -2 s -1 ), die Kultivierungen in dieser Arbeit dagegen in blaugrünem Licht durchgeführt. Der Effekt des Wechsels der Lichtqualität geht aus den Lichtsättigungskurven hervor [174]. In dieser Arbeit konnte für den WT ein Rückgang der O 2 -Entwicklung in blauem Licht gezeigt werden, während die O 2 -Entwicklung in der Olive nahezu unverändert blieb. In blauem Licht kommt es dabei zu einem quenching der Anregungsenergie durch das Protein OCP [64,181], das, durch die verkleinerten Antennen und die erhöhte PS2-Menge, in der Olive geringer als im WT ausgeprägt ist [59]. Die in dieser Arbeit ermittelten, erhöhten O 2 - Entwicklungen in Olive und DM können somit durch das verwendete blaugrüne Licht beeinflusst sein. Wie in Abbildung 4-6 gezeigt, soll die Verkleinerung der PBS, durch verringerte Dissipation der Anregungsenergie, in einem verbesserten Lichteintrag und somit in erhöhten Zelldichten resultieren [184]. Eine Erhöhung der Zelldichten durch verkleinerte PBS konnte in dieser Arbeit jedoch weder durch die Ergebnisse im Bioreaktor, noch durch die aufgenommen Wachstumskurven bestätigt werden. Dies ist in Übereinstimmung mit weiteren Untersuchungen, die eher eine Abnahme der Biomasseproduktion durch verkleinerte PBS zeigen [180]. Abbildung 4-6: Die Reduzierung der Antennengröße verringert die Dissipation von Anregungsenergie und die Beschattung im Bioreaktor. Der erhöhte Lichteintrag soll die Zellzahl und die Photosyntheseleistung steigern. Möglicherweise wiegt hier eine verringerte Absorption von Anregungsenergie, durch verkleinerte PBS, schwerer als ein verringerter Beschattungseffekt, so dass es bei hohen Zelldichten eher zu einem Lichtmangel kommt. Zudem können die Beschattungseffekte durch eine Optimierung der Durchmischung im Bioreaktor vermindert werden. Darüber hinaus wird die Biomasseproduktion entscheidend von den Produktionskosten für das Wachstum beeinflusst [115]. Eine verringerte Biomasseproduktion kann so auch durch die erhöhten Raten der Stickstoff- und Nitratassimilation in den PBS-Mutanten (Diss. Jong-Hee Kwon) 113
124 Diskussion bedingt sein, welche hier möglicherweise ein Überangebot von reduziertem Ferredoxin/NADPH verringern [160]. Durch den hohen Energieaufwand von acht Molekülen Ferredoxin [182] für die Umwandlung von einem Molekül Nitrat in Nitrit und anschließend in Ammonium wird die Biomasseproduktion verringert [183] Der Design-Organismus für eine photobiologische H 2 -Produktion Zur Optimierung des Energiemetabolismus von Synechocystis für eine zukünftige H 2 - Produktion wurden in dieser Arbeit verschiedene Mutanten konstruiert und charakterisiert. Diese Mutanten wurden dabei auf Grund der Annahmen ausgewählt, dass die Verkleinerung der PBS zu einer Erhöhung des PS2/PS1 Verhältnisses führt und diese Erhöhung die ETR steigert. Eine zusätzliche Steigerung sollte durch eine Entkopplung des ET erreicht werden. Tabelle 4-2: Übersicht über die Eigenschaften des Energiemetabolismus der, in dieser Arbeit verwendeten und konstruierten, Mutanten, sowie dem Synechocystis WT. Stämme Charakteristika WT Vollständige PBS; hohe Photoinhibition; Durch niedriges PS2/PS1 Verhältnis (1:10) nur geringe ETR Cε Olive DM TM PAL Vollständige PBS; hohe Photoinhibition; niedriges PS2/PS1 Verhältnis (1:10) ETR konnten durch Entkopplung gesteigert werden Verlust von PC erhöht PS2-Menge auf das dreifache vom WT und damit PS2/PS1 Verhältnis (1:3), geringe Photoinhibition, hohe ETR Verlust von PC erhöht PS2-Menge auf das dreifache vom WT und damit PS2/PS1 Verhältnis (1:3), geringe Photoinhibition, sehr hohe ETR durch zusätzliche Entkopplung Verlust von PC erhöht PS2-Menge auf das dreifache vom WT und damit PS2/PS1 Verhältnis (1:3), hohe Photoinhibition; geringe ETR Verlust der PBS senkt die PS1-Menge, dadurch PS2/PS1 erhöht (1:4), geringe Photoinhibition, geringe ETR Die in dieser Arbeit ermittelten Eigenschaften des Metabolismus, der verwendeten und konstruierten Mutanten, sind in Tabelle 4-2 gezeigt und ergeben drei Faktoren, die zu einer Steigerung des ETR beitragen: Erhöhung der Menge von PS2 Entkopplung des ET Geringe Photoinhibition 114
125 Diskussion Dabei hat die Erhöhung der PS2-Menge das größte Potential für eine Steigerung der ETR. Wie in der Abbildung 4-7 A gezeigt, macht die Verdreifachung der PS-Menge durch Verlust von PC und die weitere Steigerung des ETR durch die Entkopplung die DM zu einem idealen Kandidaten für weitere Optimierungen des Energiemetabolismus. Steigerungen des PET in der DM könnten durch eine weitere Steigerung des PS2-Gehaltes erreicht werden. Da die hohen ETR in der DM zu Problemen an der Akzeptorseite von PS1 führen, könnten die ETR ebenfalls durch eine erhöhte Elektronenabnahme am PS1 gesteigert werden. Eine erhöhte Elektronenabnahme könnte, wie in der Abbildung 4-7 A gezeigt, durch eine Überexpression der Proteine Flv1+3 und dadurch gesteigerte Raten der Mehler-Reaktion erreicht werden. Da dies den Transport von photosynthetischen Elektronen auf O 2 weiter steigert, ist eine Charakterisierung der tatsächlichen Photosyntheseleistung nur an Hand der Chl a Fluoreszenz möglich. Wie in der Abbildung 4-7 B gezeigt, kann dies in Kombination mit der Messung der O 2 -Entwicklung erfolgen. Durch den gezeigten Einfluss der Lichtqualität ist die Verwendung des gewünschten H 2 -Produktionslichtes für die Charakterisierung im Bioreaktor zwingend erforderlich, da nur so die tatsächliche zukünftige Produktionsleistung gezeigt werden kann. Wie in der Abbildung 4-7 B gezeigt, ist dies durch die Verwendung von blauen und roten LEDs möglich. 115
126 Diskussion Abbildung 4-7: A: Design-Organismus für eine photobiologische H 2-Produktion. Die höchsten ETR werden durch eine Kombination von PC-Verlust und Entkopplung erzielt. Eine weitere Erhöhung kann eine gesteigerte Elektronenabnahme am PS1 ergeben. Dies könnte durch eine Überexpression von Flv1+3 und dadurch erhöhte Raten der Mehler-Reaktion erreicht werden. B: Zukünftige Charakterisierungen der ETR im Bioreaktor sollten unter Verwendung von blauem und rotem Licht über die O 2-Entwicklung und die Chl a Fluoreszenz erfolgen. 116
127 Diskussion 4.14 Ausblick Durch den Verlust von PC und die Inaktivierung des C-Terminus der Epsilon-UE der ATP- Synthase wurden in der DM hohe Raten des linearen PET erhalten und damit die Grundlage für weitere Arbeiten an dem Designorganismus für eine photobiologische H 2 -Produktion geschaffen. Für weitere Optimierungen des PET in der Thylakoidmembran müssen zunächst neue Erkenntnisse über Organisation und Regulation gewonnen werden. Erstrebenswert wären neue Erkenntnisse, die es erlauben den PS2-Gehalt der Zelle weiter zu steigern. Gelingt dies nicht durch gerichtete Mutationen, könnte eine Erhöhung des PS2-Gehaltes durch Bestrahlung mit langwelligem Rotlicht möglich sein. Eine mögliche Zusammenlagerung der Komponenten der Elektronentransportkette zu Superkomplexen kann in Synechocystis durch die Verringerung der Diffusionsdistanzen zwischen den einzelnen Komponenten zur Regulation des ET beitragen. Um sich diese Komplexbildung zu Nutze zu machen, müssen jedoch zunächst die grundlegenden Mechanismen der Komplexbildung verstanden werden. Abbildung 4-8: Zukünftige H 2-Produktion in einem Designorganismus. Entkopplung und Verlust von PC führen zu hohen ETR. Nach Inaktivierung der Mehler-Reaktion (Flv) können im Metabolismus nicht verwendete Elektronen für eine H 2-Produktion genutzt werden. Erhöhungen der ETR können jedoch auch durch Prozessoptimierungen erreicht werden. So haben neben dem in dieser Arbeit gezeigtem Einfluss der Lichtqualität auch die Dauer und der Rhythmus der Belichtung einen Effekt auf die Photosynthese. Untersuchungen zum Tag/Nacht-Rhythmus zeigen, dass bei diesem die Tagesphotosyntheseleistung im Vergleich zu einer kontinuierlichen Beleuchtung gesteigert ist. Erste Voruntersuchungen im Bioreaktor 117
Mitochondriale Elektronentransportkette (Atmungskette)
 Mitochondriale Elektronentransportkette (Atmungskette) Mitochondriale Elektronentransportkette (Atmungskette) Komplex I und II übetragen Elektronen auf Coenzym Q (Ubichinon) Gekoppelte Elektronen-Protonen
Mitochondriale Elektronentransportkette (Atmungskette) Mitochondriale Elektronentransportkette (Atmungskette) Komplex I und II übetragen Elektronen auf Coenzym Q (Ubichinon) Gekoppelte Elektronen-Protonen
Photosynthese. Pflanzen nied. Eukaryoten (Rotalgen) Prokaryoten (Cyanobakterien) Cyanobakterium Anabaena. Abb. aus Stryer (5th Ed.
 Photosynthese Pflanzen nied. Eukaryoten (Rotalgen) Prokaryoten (Cyanobakterien) Cyanobakterium Anabaena Chloroplasten Photosynthese Licht CO 2 + H 2 O (CH 2 O) + O 2 Licht Lichtreaktion: 2 H 2 O O 2 +
Photosynthese Pflanzen nied. Eukaryoten (Rotalgen) Prokaryoten (Cyanobakterien) Cyanobakterium Anabaena Chloroplasten Photosynthese Licht CO 2 + H 2 O (CH 2 O) + O 2 Licht Lichtreaktion: 2 H 2 O O 2 +
Evolutionärer Vorteil als Grundlage zum Leben? Von Nadine Körtel Konrad-Adenauer Straße , Hainburg
 Phototrophe Lebensweise Evolutionärer Vorteil als Grundlage zum Leben? Von Nadine Körtel Konrad-Adenauer Straße 50 63512, Hainburg Gliederung Phototrophe Bakterien Endosymbiontentheorie Chloroplasten Pigmente
Phototrophe Lebensweise Evolutionärer Vorteil als Grundlage zum Leben? Von Nadine Körtel Konrad-Adenauer Straße 50 63512, Hainburg Gliederung Phototrophe Bakterien Endosymbiontentheorie Chloroplasten Pigmente
Lichtgetriebener Elektronentransport in der Thylakoidmembran führt zu PMK
 Lichtgetriebener Elektronentransport in der Thylakoidmembran führt zu PMK PMF ensteht an drei Punkten Wasserspaltung im Lumen Transport von Protonen vom Stroma zum Lumen durch Plastochinon Aufnahme eines
Lichtgetriebener Elektronentransport in der Thylakoidmembran führt zu PMK PMF ensteht an drei Punkten Wasserspaltung im Lumen Transport von Protonen vom Stroma zum Lumen durch Plastochinon Aufnahme eines
Der photosynthetische Elektronentransport
 Der photosynthetische Elektronentransport 1. Die Hill-Reaktion Robert Hill zeigte 1938: o in Gegenwart von Elektronenakzeptoren (A) erfolgt Sauerstoffentwicklung an belichteten Thylakoidmembranen o ohne
Der photosynthetische Elektronentransport 1. Die Hill-Reaktion Robert Hill zeigte 1938: o in Gegenwart von Elektronenakzeptoren (A) erfolgt Sauerstoffentwicklung an belichteten Thylakoidmembranen o ohne
Photosynthetischer Elektronen-Transport
 Photosynthetischer Elektronen-Transport Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2013 Rückblick: Geschichte der Photosynthese Cornelius van Niel (1932)
Photosynthetischer Elektronen-Transport Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2013 Rückblick: Geschichte der Photosynthese Cornelius van Niel (1932)
Regulation der Glykolyse: Phosphofructokinase
 Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Regulation der Glykolyse: Phosphofructokinase Abbauwege des Pyruvats Weitere Oxidation (zu CO 2 ) Alkoholische Gärung Pyruvat- Decarboxylase Alkohol- Dehydrogenase NAD + wird bei der Gärung regneriert,
Atmungskette ( Endoxidation) Reaktionen und ATP-Synthase
 Atmungskette ( Endoxidation) Reaktionen und ATP-Synthase Einleitung Aufrechterhaltung von Struktur und Funktion aller Lebensformen hängt von einer ständigen Energiezufuhr ab Höchste Energieausbeute liefert
Atmungskette ( Endoxidation) Reaktionen und ATP-Synthase Einleitung Aufrechterhaltung von Struktur und Funktion aller Lebensformen hängt von einer ständigen Energiezufuhr ab Höchste Energieausbeute liefert
3. Lichtabhängige Reaktionen
 3. Lichtabhängige Reaktionen Problem: Wie wird die elektromagnetische Strahlung des Sonnenlichts in chemisch verwertbare Energie umgewandelt? Um die Lichtenergie chemisch nutzbar zu machen, muss elektromagnetische
3. Lichtabhängige Reaktionen Problem: Wie wird die elektromagnetische Strahlung des Sonnenlichts in chemisch verwertbare Energie umgewandelt? Um die Lichtenergie chemisch nutzbar zu machen, muss elektromagnetische
Abbildungsverzeichnis. Tabellenverzeichnis. Abkürzungsverzeichnis. 1 Einleitung 1
 Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis VI Tabellenverzeichnis VIII Abkürzungsverzeichnis IX 1 Einleitung 1 1.1 Wasser, ein limitierender Faktor für die Landwirtschaft 1 1.2 Anpassungen
Inhaltsverzeichnis Inhaltsverzeichnis I Abbildungsverzeichnis VI Tabellenverzeichnis VIII Abkürzungsverzeichnis IX 1 Einleitung 1 1.1 Wasser, ein limitierender Faktor für die Landwirtschaft 1 1.2 Anpassungen
Die Wege des Kohlenstoffes
 Die Wege des Kohlenstoffes 1. Licht vs. Dunkelreaktionen Lichtgetriebene Reaktionen o Dunkle -Reaktionen laufen nicht im Dunklen ab reduzieren CO 2-Zucker für Pflanzen zum Wachsen und für uns zum Nutzen
Die Wege des Kohlenstoffes 1. Licht vs. Dunkelreaktionen Lichtgetriebene Reaktionen o Dunkle -Reaktionen laufen nicht im Dunklen ab reduzieren CO 2-Zucker für Pflanzen zum Wachsen und für uns zum Nutzen
Regulation der Carotinoidbiosynthese in Nostoc PCC Dissertation Zur Erlangung des Doktorgrades der Naturwissenschaften
 Regulation der Carotinoidbiosynthese in Nostoc PCC 7120 Dissertation Zur Erlangung des Doktorgrades der Naturwissenschaften vorgelegt beim Fachbereich Biowissenschaften der Johann Wolfgang Goethe-Universität
Regulation der Carotinoidbiosynthese in Nostoc PCC 7120 Dissertation Zur Erlangung des Doktorgrades der Naturwissenschaften vorgelegt beim Fachbereich Biowissenschaften der Johann Wolfgang Goethe-Universität
Physiologie der Photosynthese
 Claus Buschmann Karl Grumbach Physiologie der Photosynthese Mit 106 Abbildungen UNIVERSITÄTS, BIBLIOTHEK Springer-Verlag Berlin Heidelberg New York Tokyo 1985 \ Inhaltsverzeichnis 1. Stoffumwandlungen
Claus Buschmann Karl Grumbach Physiologie der Photosynthese Mit 106 Abbildungen UNIVERSITÄTS, BIBLIOTHEK Springer-Verlag Berlin Heidelberg New York Tokyo 1985 \ Inhaltsverzeichnis 1. Stoffumwandlungen
1. Wieso trägt Plastochinon zur Ansäuerung des Thylakoidlumens während der Photosynthese bei (bitte mit Zeichnung)?
 1. Wieso trägt Plastochinon zur Ansäuerung des Thylakoidlumens während der Photosynthese bei (bitte mit Zeichnung)? - Plastochinon nimmt 2 Elektronen auf der Stromaseite von PSII auf, wobei auch 2 H +
1. Wieso trägt Plastochinon zur Ansäuerung des Thylakoidlumens während der Photosynthese bei (bitte mit Zeichnung)? - Plastochinon nimmt 2 Elektronen auf der Stromaseite von PSII auf, wobei auch 2 H +
Von Nadine Ufermann und Marcus Oldekamp
 Von Nadine Ufermann und Marcus Oldekamp Photosynthese: Allgemein und Lichtreaktion Photosysteme: PSI und PSII, Entdeckung und Funktion Mangan und Manganenzyme: Speziell sauerstoffentwickelnder Mn Cluster
Von Nadine Ufermann und Marcus Oldekamp Photosynthese: Allgemein und Lichtreaktion Photosysteme: PSI und PSII, Entdeckung und Funktion Mangan und Manganenzyme: Speziell sauerstoffentwickelnder Mn Cluster
Biologie I: Funktionen des Blattes
 Biologie I: Funktionen des Blattes Chloroplasten und Photosyntheseapparat Prinzipien der Energiegewinnung und Energienutzung Lichtreaktion der Photosynthese Dunkelreaktion der Photosynthese Copyright Hinweis:
Biologie I: Funktionen des Blattes Chloroplasten und Photosyntheseapparat Prinzipien der Energiegewinnung und Energienutzung Lichtreaktion der Photosynthese Dunkelreaktion der Photosynthese Copyright Hinweis:
1a. Welche Pigmente enthält der Lichtsammelkomplex II (LHCII) höherer Pflanzen? (Namen aller Pigmente für volle Punktzahl)
 1a. Welche Pigmente enthält der Lichtsammelkomplex II (LHCII) höherer Pflanzen? (Namen aller Pigmente für volle Punktzahl) 2,5 P. 1b. Wozu dienen Carotinoide in den Proteinkomplexen der Thylakoidmembran?
1a. Welche Pigmente enthält der Lichtsammelkomplex II (LHCII) höherer Pflanzen? (Namen aller Pigmente für volle Punktzahl) 2,5 P. 1b. Wozu dienen Carotinoide in den Proteinkomplexen der Thylakoidmembran?
Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!!
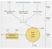 Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!! Indirekte ATP synthese! Protonen können in wässriger Lösung sehr! schnell transportiert werden.! Ionen können biologische Membranen nicht spontan
Glykolyse! Pyruvat-! dehydrogenase! Citronensäure-! Zyklus!! Indirekte ATP synthese! Protonen können in wässriger Lösung sehr! schnell transportiert werden.! Ionen können biologische Membranen nicht spontan
Photosystem=Antenne + Reaktionszentrum
 Lichtsammelkomplexe Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2012 Photosystem=Antenne + Reaktionszentrum Die Antennen absorbieren die Photonen und leiten
Lichtsammelkomplexe Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2012 Photosystem=Antenne + Reaktionszentrum Die Antennen absorbieren die Photonen und leiten
Schritt für Schritt Simulation Die Atmungskette
 KENNZEICHEN: KURS - SCHULE - - Schritt für Schritt Simulation Die Atmungskette Inhalt Übersicht über das Mitochondrium und die Mitochondrienmembran S.2 Zeichenerklärung S.3 Stichwortverzeichnis S.4 Leitfaden
KENNZEICHEN: KURS - SCHULE - - Schritt für Schritt Simulation Die Atmungskette Inhalt Übersicht über das Mitochondrium und die Mitochondrienmembran S.2 Zeichenerklärung S.3 Stichwortverzeichnis S.4 Leitfaden
Einführung in die Biochemie Fotosynthese Autotrophe Assimilation
 Fotosynthese Autotrophe Assimilation Die Fotosynthese läuft in den Chloroplasten ab. Der Chloroplast wird nach außen hin von einer Doppelmembran begrenzt. Im inneren befindet sich das Stroma, Stärkekörner,
Fotosynthese Autotrophe Assimilation Die Fotosynthese läuft in den Chloroplasten ab. Der Chloroplast wird nach außen hin von einer Doppelmembran begrenzt. Im inneren befindet sich das Stroma, Stärkekörner,
Biologie für Mediziner
 Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Biologie für Mediziner - Zellbiologie 1 - Prof. Dr. Reiner Peters Institut für Medizinische Physik und Biophysik/CeNTech Robert-Koch-Strasse 31 Tel. 0251-835 6933, petersr@uni-muenster.de Dr. Martin Kahms
Schrittweise Oxidation und Decarboxylierung von Glucose-6-phosphat zu Ribulose-5- phosphat
 1. Der plastidäre oxidative Pentosephosphatweg Abbau von Hexose unter NADPH+H + -Synthese Schlüsselenzym=Glucose-6-phosphat-Dehydrogenase Glucose-6-phosphat-Dehydrogenase wird durch Thioredoxin im reduzierten
1. Der plastidäre oxidative Pentosephosphatweg Abbau von Hexose unter NADPH+H + -Synthese Schlüsselenzym=Glucose-6-phosphat-Dehydrogenase Glucose-6-phosphat-Dehydrogenase wird durch Thioredoxin im reduzierten
Kohlenstoffdioxid wird in lichtabhängigen Reaktion freigesetzt Sauerstoff wird wie in der Atmung verbraucht. Lichtatmung
 1. Kompartimentierung bei der Photorespiration Kohlenstoffdioxid wird in lichtabhängigen Reaktion freigesetzt Sauerstoff wird wie in der Atmung verbraucht. Lichtatmung Kosten: o Energie, da ATP verbraucht
1. Kompartimentierung bei der Photorespiration Kohlenstoffdioxid wird in lichtabhängigen Reaktion freigesetzt Sauerstoff wird wie in der Atmung verbraucht. Lichtatmung Kosten: o Energie, da ATP verbraucht
Enthalpie. Ob eine Reaktion exergonisch ist, entscheidet die Änderung des Wärmeinhalts (heat content) H = Enthalpie
 Enthalpie Ob eine Reaktion exergonisch ist, entscheidet die Änderung des Wärmeinhalts (heat content) H = Enthalpie C 6 H 12 O 6 + 6O 2 6CO 2 + 6 H 2 O H = -2818 kj/mol - bei exergonischen Reaktionen Entropie
Enthalpie Ob eine Reaktion exergonisch ist, entscheidet die Änderung des Wärmeinhalts (heat content) H = Enthalpie C 6 H 12 O 6 + 6O 2 6CO 2 + 6 H 2 O H = -2818 kj/mol - bei exergonischen Reaktionen Entropie
Atmung Respiration 1
 Atmung Respiration 1 Atmung Die oxidative Phosphorylierung ist der letzte Schritt einer Serie von Energieumwandlungen, die insgesamt als Zellatmung oder einfach Atmung (Respiration) bezeichnet werden 2
Atmung Respiration 1 Atmung Die oxidative Phosphorylierung ist der letzte Schritt einer Serie von Energieumwandlungen, die insgesamt als Zellatmung oder einfach Atmung (Respiration) bezeichnet werden 2
Stoffwechsel. Metabolismus (3)
 Vorlesung Zell- und Molekularbiologie Stoffwechsel Metabolismus (3) Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien 1 Glykolyse 1 Glucose und in der Glykolyse daraus gebildete
Vorlesung Zell- und Molekularbiologie Stoffwechsel Metabolismus (3) Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien 1 Glykolyse 1 Glucose und in der Glykolyse daraus gebildete
Versuchsprotokoll. 1.) Versuch 1d: Bestimmung der photochemischen Aktivität isolierter Chloroplasten mit der Sauerstoffelektrode
 Versuchsprotokoll 1.) Versuch 1d: Bestimmung der photochemischen Aktivität isolierter Chloroplasten mit der Sauerstoffelektrode 1.1. Einleitung: Bei der Isolierung von Chloroplasten erhält man Hauptsächlich
Versuchsprotokoll 1.) Versuch 1d: Bestimmung der photochemischen Aktivität isolierter Chloroplasten mit der Sauerstoffelektrode 1.1. Einleitung: Bei der Isolierung von Chloroplasten erhält man Hauptsächlich
Photosynthetischer Elektronen-Transport
 Photosynthetischer Elektronen-Transport Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2012 Rückblick: Geschichte der Photosynthese Cornelius van Niel (1932)
Photosynthetischer Elektronen-Transport Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2012 Rückblick: Geschichte der Photosynthese Cornelius van Niel (1932)
In den grünen Pflanzenteilen, genauer gesagt in bestimmten Organellen der Pflanzenzellen, den
 A 7 Fotosynthese Stellen sie die Wort- und die Symbolgleichung für den Vorgang der Fotosynthese in grünen Pflanzen auf. Wortgleichung: Symbolgleichung: Vervollständigen Sie den Text. In den grünen Pflanzenteilen,
A 7 Fotosynthese Stellen sie die Wort- und die Symbolgleichung für den Vorgang der Fotosynthese in grünen Pflanzen auf. Wortgleichung: Symbolgleichung: Vervollständigen Sie den Text. In den grünen Pflanzenteilen,
Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA
 Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu
Aufbau eines Assays zur Optimierung einer Polymerase für die Generierung hochgradig fluoreszenz-markierter DNA Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu
Pflanzliche und bakterielle Stoffwechselprozesse
 Pflanzliche und bakterielle Stoffwechselprozesse 4.6 Pflanzliche und bakterielle Stoffwechselprozesse 4.6.1 Photosynthese 4.6.2 Chemosynthese 4.6.3 Calvin-Cyclus Photosynthese Die Photosynthese ist ein
Pflanzliche und bakterielle Stoffwechselprozesse 4.6 Pflanzliche und bakterielle Stoffwechselprozesse 4.6.1 Photosynthese 4.6.2 Chemosynthese 4.6.3 Calvin-Cyclus Photosynthese Die Photosynthese ist ein
Photosystem = Antenne + Reaktionszentrum
 Photosynthetische Reaktionszentren Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2012 Photosystem = Antenne + Reaktionszentrum Licht Die Antennen absorbieren
Photosynthetische Reaktionszentren Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2012 Photosystem = Antenne + Reaktionszentrum Licht Die Antennen absorbieren
Protokoll Versuch 1 d Bestimmung der photochemischen Aktivität isolierter Chloroplasten mit der Sauerstoffelektrode
 Protokoll Versuch 1 d Bestimmung der photochemischen Aktivität isolierter Chloroplasten mit der Sauerstoffelektrode Chloroplasten verlieren bei ihrer Isolation die umhüllende Doppelmembran, die in der
Protokoll Versuch 1 d Bestimmung der photochemischen Aktivität isolierter Chloroplasten mit der Sauerstoffelektrode Chloroplasten verlieren bei ihrer Isolation die umhüllende Doppelmembran, die in der
Struktur-Funktionsbeziehungen. NADH-Oxidase aus. Thermus thermophilus
 Struktur-Funktionsbeziehungen der NADH-Oxidase aus Thermus thermophilus Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades eines
Struktur-Funktionsbeziehungen der NADH-Oxidase aus Thermus thermophilus Von der Naturwissenschaftlichen Fakultät der Technischen Universität Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades eines
1 Einleitung Material und Methoden
 1 Einleitung 1 1.1 P-Typ ATPasen 2 1.1.1 Struktur und Funktion von P-Typ ATPasen 4 1.1.2 CPx-ATPasen 7 1.1.2.1 Nukleotidbindungs-/Phosphorylierungsdomäne 8 1.1.2.2 Aktuatordomäne 13 1.1.2.3 Cytoplasmatische
1 Einleitung 1 1.1 P-Typ ATPasen 2 1.1.1 Struktur und Funktion von P-Typ ATPasen 4 1.1.2 CPx-ATPasen 7 1.1.2.1 Nukleotidbindungs-/Phosphorylierungsdomäne 8 1.1.2.2 Aktuatordomäne 13 1.1.2.3 Cytoplasmatische
Biochemie und. chemischer Mechanismus
 Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München Biochemie und chemischer Mechanismus der Thiomethylierung von RNA Dorothea Maria
Dissertation zur Erlangung des Doktorgrades der Fakultät für Chemie und Pharmazie der Ludwig-Maximilians-Universität München Biochemie und chemischer Mechanismus der Thiomethylierung von RNA Dorothea Maria
VL4: Purpurbakterien - Antennenchlorophylle. Kolonien von Purpurbakterien in Petrischale. Lebende Purpurbakterien
 VL4: Purpurbakterien - Antennenchlorophylle Kolonien von Purpurbakterien in Petrischale Lebende Purpurbakterien VL4: Photosynthetische Einheit in bakteriellen Purpurbakterien PSU (photosynthetic unit)
VL4: Purpurbakterien - Antennenchlorophylle Kolonien von Purpurbakterien in Petrischale Lebende Purpurbakterien VL4: Photosynthetische Einheit in bakteriellen Purpurbakterien PSU (photosynthetic unit)
Photosynthese. Astrobiologie: Panspermie und Terraforming von (Exo-)Planeten WS 13/14. Anna Till
 Astrobiologie: Panspermie und Terraforming von (Exo-)Planeten WS 13/14 Anna Till Einleitung Überblick 1 Photosynthese Allgemeines zur Photosynthese Oxygene Photosyntehese Anoxygene Photosyntehese Verlgeich:
Astrobiologie: Panspermie und Terraforming von (Exo-)Planeten WS 13/14 Anna Till Einleitung Überblick 1 Photosynthese Allgemeines zur Photosynthese Oxygene Photosyntehese Anoxygene Photosyntehese Verlgeich:
Hydrogen from water : Engineering photosynthetic metabolism for a light-powered biohydrogen production
 Hydrogen from water : Engineering photosynthetic metabolism for a light-powered biohydrogen production Matthias Rögner Lehrstuhl für Biochemie der Pflanzen Ruhr-Universität Bochum LEOPOLDINA Recommendation
Hydrogen from water : Engineering photosynthetic metabolism for a light-powered biohydrogen production Matthias Rögner Lehrstuhl für Biochemie der Pflanzen Ruhr-Universität Bochum LEOPOLDINA Recommendation
Neue Enzyme für ein altes Organeil: Kryptische peroxisomale Lokalisationssignale in Pilzen. Dissertation
 Neue Enzyme für ein altes Organeil: Kryptische peroxisomale Lokalisationssignale in Pilzen Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften (Dr. rer. nat.) Dem Fachbereich Biologie der
Neue Enzyme für ein altes Organeil: Kryptische peroxisomale Lokalisationssignale in Pilzen Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften (Dr. rer. nat.) Dem Fachbereich Biologie der
12. Oxidative Phosphorylierung
 12. Oxidative Phosphorylierung 303 Zweck: Gewinnung von ATP Regeneration von NAD + und FAD Gesamtreaktionen: 3 ADP + 3 P i 3 ATP NADH + 0,5 O 2 + H + NAD + + H-O-H 2 ADP + 2 P i 2 ATP FADH 2 + 0,5 O 2
12. Oxidative Phosphorylierung 303 Zweck: Gewinnung von ATP Regeneration von NAD + und FAD Gesamtreaktionen: 3 ADP + 3 P i 3 ATP NADH + 0,5 O 2 + H + NAD + + H-O-H 2 ADP + 2 P i 2 ATP FADH 2 + 0,5 O 2
Manganenzyme und Photosystem II
 Manganenzyme und Photosystem II von Gang Wang & Raimar Broser Steinfurt, den 17. November 2004 Inhaltsangabe 1. Bedeutung der Photosynthese 2. rt und rganelle der Photosynthese 3. Ablauf der Photosynthese
Manganenzyme und Photosystem II von Gang Wang & Raimar Broser Steinfurt, den 17. November 2004 Inhaltsangabe 1. Bedeutung der Photosynthese 2. rt und rganelle der Photosynthese 3. Ablauf der Photosynthese
Grundlagen des Stoffwechsels
 Vorlesung Allgemeine Mikrobiologie Grundlagen des Stoffwechsels www.icbm.de/pmbio Was ist Stoffwechsel? Stoffwechsel fi Chemische Umsetzungen, (Bio-)Chemie fi Umwandlung von Nahrung in Biomasse fi Umwandlung
Vorlesung Allgemeine Mikrobiologie Grundlagen des Stoffwechsels www.icbm.de/pmbio Was ist Stoffwechsel? Stoffwechsel fi Chemische Umsetzungen, (Bio-)Chemie fi Umwandlung von Nahrung in Biomasse fi Umwandlung
B I O T E C H N O L O G I E. Molekulare Biotechnologie Engineering von Biosystemen. Bioprozesstechnik Engineering von Produktionsverfahren
 B I O T E C H N O L O G I E Molekulare Biotechnologie Engineering von Biosystemen Zell-Engineering Stoffwechsel-Engineering Protein-Engineering Integrierte Vernetzung Bioprozesstechnik Engineering von
B I O T E C H N O L O G I E Molekulare Biotechnologie Engineering von Biosystemen Zell-Engineering Stoffwechsel-Engineering Protein-Engineering Integrierte Vernetzung Bioprozesstechnik Engineering von
Struktur, Funktion und Interaktion von Membranproteinkomponenten des cyanobakteriellen Elektronentransportes in Thermosynechococcus elongatus BP-1
 Struktur, Funktion und Interaktion von Membranproteinkomponenten des cyanobakteriellen Elektronentransportes in Thermosynechococcus elongatus BP-1 Dissertation zur Erlangung des Grades eines Doktors der
Struktur, Funktion und Interaktion von Membranproteinkomponenten des cyanobakteriellen Elektronentransportes in Thermosynechococcus elongatus BP-1 Dissertation zur Erlangung des Grades eines Doktors der
Ribulose-1.5-bisphosphatCarboxylase/Oxygenase
 Diss.ETHNr.7610 Der Einfluss des Aktivierungszustandes und der katalytischen Eigenschaften der Ribulose-1.5-bisphosphatCarboxylase/Oxygenase auf die Photosynthese intakter Blätter von Weissklee (Trifolium
Diss.ETHNr.7610 Der Einfluss des Aktivierungszustandes und der katalytischen Eigenschaften der Ribulose-1.5-bisphosphatCarboxylase/Oxygenase auf die Photosynthese intakter Blätter von Weissklee (Trifolium
Citratzyklus. Biochemie Maria Otto,Bo Mi Ok Kwon Park
 Citratzyklus Biochemie 13.12.2004 Maria Otto,Bo Mi Ok Kwon Park O CH 3 C Acetyl-CoA + H 2 O HO C COO C NADH O C H Citrat Cis-Aconitat H C Malat Citratzyklus HO C H Isocitrat CH H 2 O Fumarat C = O FADH
Citratzyklus Biochemie 13.12.2004 Maria Otto,Bo Mi Ok Kwon Park O CH 3 C Acetyl-CoA + H 2 O HO C COO C NADH O C H Citrat Cis-Aconitat H C Malat Citratzyklus HO C H Isocitrat CH H 2 O Fumarat C = O FADH
Oxidative Phosphorylierung
 BICEMIE DER ERÄRUG II Grundzüge des Metabolismus xidative Phosphorylierung 24.04.2012 xidative Phosphorylierung xidative Phosphorylierung -Die Elektronen in AD und in FAD 2 (hohes Übertragungspotential)
BICEMIE DER ERÄRUG II Grundzüge des Metabolismus xidative Phosphorylierung 24.04.2012 xidative Phosphorylierung xidative Phosphorylierung -Die Elektronen in AD und in FAD 2 (hohes Übertragungspotential)
Lichtabsorption. Handout zum Vortrag am REFERAT BIOLOGIE LEISTUNGSKURS. 11 Februar 2007 Verfasst von: Daniela Luxem
 Lichtabsorption Handout zum Vortrag am 13.02.2007 REFERAT BIOLOGIE LEISTUNGSKURS 11 Februar 2007 Verfasst von: Daniela Luxem Lichtabsorption Handout zum Vortrag am 13.02.2007 Ziel dieser Arbeit ist, den
Lichtabsorption Handout zum Vortrag am 13.02.2007 REFERAT BIOLOGIE LEISTUNGSKURS 11 Februar 2007 Verfasst von: Daniela Luxem Lichtabsorption Handout zum Vortrag am 13.02.2007 Ziel dieser Arbeit ist, den
Teil 5: Physiologie der Pflanze
 Teil 5: Physiologie der Pflanze 18. Photosynthese 18.1 Überblick 18.2 Bedeutung und Eigenschaften des Lichtes 18.3 Reaktionssysteme der Photosynthese 18.3.1 Lichtreaktion 18.3.2 Photophosphorylierung 18.3.3
Teil 5: Physiologie der Pflanze 18. Photosynthese 18.1 Überblick 18.2 Bedeutung und Eigenschaften des Lichtes 18.3 Reaktionssysteme der Photosynthese 18.3.1 Lichtreaktion 18.3.2 Photophosphorylierung 18.3.3
Warnung: diese Klausur enthält nur die Fragen von Büchel und Sandmann, die Fragen von Schleiff/Scharf fehlen
 Klausurfragen Pflanzenphysiologie 19 Feb 2009 Warnung: diese Klausur enthält nur die Fragen von Büchel und Sandmann, die Fragen von Schleiff/Scharf fehlen Pkt. 1. a) In welchen der genannten Proteine liegen
Klausurfragen Pflanzenphysiologie 19 Feb 2009 Warnung: diese Klausur enthält nur die Fragen von Büchel und Sandmann, die Fragen von Schleiff/Scharf fehlen Pkt. 1. a) In welchen der genannten Proteine liegen
VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN
 INHALTSVERZEICHNIS I VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VII VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN XIII 1 EINLEITUNG 1 1.1 Osmoadaption halophiler Mikroorganismen 2 1.1.1 Salt-in-cytoplasm
INHALTSVERZEICHNIS I VERZEICHNIS DER ABBILDUNGEN UND TABELLEN VII VERZEICHNIS DER ABKÜRZUNGEN UND TRIVIALNAMEN XIII 1 EINLEITUNG 1 1.1 Osmoadaption halophiler Mikroorganismen 2 1.1.1 Salt-in-cytoplasm
Mechanismen der ATP Synthese in Mitochondrien
 Mechanismen der ATP Synthese in Mitochondrien Übersicht Die Bedeutung von ATP Aufbau eines Mitochondriums ATP Synthese: Citratzyklus Atmungskette ATP Synthase Regulation der ATP Synthese Die Bedeutung
Mechanismen der ATP Synthese in Mitochondrien Übersicht Die Bedeutung von ATP Aufbau eines Mitochondriums ATP Synthese: Citratzyklus Atmungskette ATP Synthase Regulation der ATP Synthese Die Bedeutung
Einführung in die Biochemie Fotosynthese Autotrophe Assimilation
 Fotosynthese Autotrophe Assimilation Die Fotosynthese läuft in den Chloroplasten ab. Der Chloroplast wird nach außen hin von einer Doppelmembran begrenzt. Im inneren befindet sich das Stroma, Stärkekörner,
Fotosynthese Autotrophe Assimilation Die Fotosynthese läuft in den Chloroplasten ab. Der Chloroplast wird nach außen hin von einer Doppelmembran begrenzt. Im inneren befindet sich das Stroma, Stärkekörner,
Charakterisierung der PII-P Phosphatase in Cyanobakterien
 Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
Charakterisierung der PII-P Phosphatase in Cyanobakterien Inaugural-Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften des Fachbereiches Biologie, Chemie und Geowissenschaften der Justus-Liebig-Universität
4. WACHSTUM. 4.1 Messung
 4. WACHSTUM 4. WACHSTUM 4.1 Messung 4. WACHSTUM 4.2 math. Beschreibung expon. Wachstum statische Kultur 4. WACHSTUM 4.3 Wachstumskurve kontinuierliche Kultur 4. WACHSTUM 4.5 Verfahren Chemostat Turbidostat
4. WACHSTUM 4. WACHSTUM 4.1 Messung 4. WACHSTUM 4.2 math. Beschreibung expon. Wachstum statische Kultur 4. WACHSTUM 4.3 Wachstumskurve kontinuierliche Kultur 4. WACHSTUM 4.5 Verfahren Chemostat Turbidostat
Photosynthetische Reaktionszentren
 Photosynthetische Reaktionszentren Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2013 Photosystem = Antenne + Reaktionszentrum Licht Die Antennen absorbieren
Photosynthetische Reaktionszentren Hendrik Küpper, Vorlesungsreihe Einführung in Bau und Funktion der Pflanzen, Sommersemester 2013 Photosystem = Antenne + Reaktionszentrum Licht Die Antennen absorbieren
Grundlagen des Stoffwechsels
 Vorlesung Allgemeine Mikrobiologie Grundlagen des Stoffwechsels www.icbm.de/pmbio Was ist Stoffwechsel? Stoffwechsel fi Chemische Umsetzungen, (Bio-)Chemie fi Umwandlung von Nahrung in Biomasse fi Umwandlung
Vorlesung Allgemeine Mikrobiologie Grundlagen des Stoffwechsels www.icbm.de/pmbio Was ist Stoffwechsel? Stoffwechsel fi Chemische Umsetzungen, (Bio-)Chemie fi Umwandlung von Nahrung in Biomasse fi Umwandlung
- der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen
 Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Die Aufgabe des Citratcyklus ist: - der oxidative Abbau von Acetyl-CoA (und die somit gebildeten Reduktionsäquivalente) - Lieferung von Substraten für verschiedene Synthesen Die Aufgabe des Citratcyklus
Wirkungsmechanismen regulatorischer Enzyme
 Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
Wirkungsmechanismen regulatorischer Enzyme Ein Multienzymsystem ist eine Aufeinanderfolge von Enzymen, bei der das Produkt eines vorstehenden Enzyms das Substrat des nächsten Enzyms wird. Ein regulatorisches
Die Primärreaktion der Photosynthese
 Die Primärreaktion der Photosynthese 1. Energetische Betrachtungen Wie ist es energetisch möglich, dass das so energiearme und stabile Wassermolekül in der Primärreaktion der Photosynthese zu Sauerstoff
Die Primärreaktion der Photosynthese 1. Energetische Betrachtungen Wie ist es energetisch möglich, dass das so energiearme und stabile Wassermolekül in der Primärreaktion der Photosynthese zu Sauerstoff
Biochemische UE Alkaline Phosphatase.
 Biochemische UE Alkaline Phosphatase peter.hammerl@sbg.ac.at Alkaline Phosphatase: Katalysiert die Hydrolyse von Phosphorsäure-Estern: O - O - Ser-102 R O P==O O - H 2 O R OH + HO P==O O - ph-optimum im
Biochemische UE Alkaline Phosphatase peter.hammerl@sbg.ac.at Alkaline Phosphatase: Katalysiert die Hydrolyse von Phosphorsäure-Estern: O - O - Ser-102 R O P==O O - H 2 O R OH + HO P==O O - ph-optimum im
3 Ergebnisse. 3.1 Die Speckling-Domäne von Wt1 umfaßt die Aminosäuren
 3 Ergebnisse 3.1 Die Speckling-Domäne von Wt1 umfaßt die Aminosäuren 76-120 Die Existenz verschiedener Isoformen von WT1 ist unter anderem auf die Verwendung einer für die Aminosäuren KTS kodierenden alternativen
3 Ergebnisse 3.1 Die Speckling-Domäne von Wt1 umfaßt die Aminosäuren 76-120 Die Existenz verschiedener Isoformen von WT1 ist unter anderem auf die Verwendung einer für die Aminosäuren KTS kodierenden alternativen
2. Tertiärstruktur. Lernziele: 1) Verstehen wie Röntgenstrukturanalyse und NMR Spektroskopie gebraucht werden um Proteinstrukturen zu bestimmen.
 2. Tertiärstruktur Lernziele: 1) Verstehen wie Röntgenstrukturanalyse und NMR Spektroskopie gebraucht werden um Proteinstrukturen zu bestimmen. 2) Verstehen warum nicht polare Reste im inneren eines Proteins
2. Tertiärstruktur Lernziele: 1) Verstehen wie Röntgenstrukturanalyse und NMR Spektroskopie gebraucht werden um Proteinstrukturen zu bestimmen. 2) Verstehen warum nicht polare Reste im inneren eines Proteins
Praktikum Biochemie B.Sc. Water Science WS Enzymregulation. Marinja Niggemann, Denise Schäfer
 Praktikum Biochemie B.Sc. Water Science WS 2011 Enzymregulation Marinja Niggemann, Denise Schäfer Regulatorische Strategien 1. Allosterische Wechselwirkung 2. Proteolytische Aktivierung 3. Kovalente Modifikation
Praktikum Biochemie B.Sc. Water Science WS 2011 Enzymregulation Marinja Niggemann, Denise Schäfer Regulatorische Strategien 1. Allosterische Wechselwirkung 2. Proteolytische Aktivierung 3. Kovalente Modifikation
Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen
 Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. des Fachbereichs Biologie, Chemie, Pharmazie der Freien Universität
Posttranskriptionale Veränderungen der Genexpression bei hereditären Erkrankungen Dissertation zur Erlangung des Doktorgrades Dr. rer. nat. des Fachbereichs Biologie, Chemie, Pharmazie der Freien Universität
Lichtreaktionen. und Methoden zu ihrer Untersuchung (I)
 Biophysik der photosynthetischen Lichtreaktionen und Methoden zu ihrer Untersuchung (I) Schema der photosynthetischen Lichtreaktionen Beispiele wichtiger Lichtsammelkomplexe Name des Komplexes Vorkommen
Biophysik der photosynthetischen Lichtreaktionen und Methoden zu ihrer Untersuchung (I) Schema der photosynthetischen Lichtreaktionen Beispiele wichtiger Lichtsammelkomplexe Name des Komplexes Vorkommen
Reduction / Oxidation
 Reduction / Oxidation Pyruvat C6H12O 6 Glucose Glycogen Glucose-6-P Glycolyse 2 e - 2 Pyruvat 2 e - 2 Acetyl-CoA 2 CO 2 ATP ADP ATP ADP Citrat-Zyklus oder Tricarbonsäure 4 CO 2 8 e - Zyklus 6 O2 6 H 2
Reduction / Oxidation Pyruvat C6H12O 6 Glucose Glycogen Glucose-6-P Glycolyse 2 e - 2 Pyruvat 2 e - 2 Acetyl-CoA 2 CO 2 ATP ADP ATP ADP Citrat-Zyklus oder Tricarbonsäure 4 CO 2 8 e - Zyklus 6 O2 6 H 2
40. Vorlesung. Hybridisierung Biologische Moleküle Photosynthese Sehvorgang Selbstorganisation Molekulare Motoren
 Prof. C. von Borczyskowski Physik für CS + SK 40. Vorlesung Hybridisierung 40.1 Biologische Moleküle 40.1.1 Photosynthese 40.1.2 Sehvorgang 40.2 Selbstorganisation 40.3 Molekulare Motoren Verwendete Literatur:
Prof. C. von Borczyskowski Physik für CS + SK 40. Vorlesung Hybridisierung 40.1 Biologische Moleküle 40.1.1 Photosynthese 40.1.2 Sehvorgang 40.2 Selbstorganisation 40.3 Molekulare Motoren Verwendete Literatur:
Rekombinante Antikorperfragmente fur die. Zoonosediagnostik
 Rekombinante Antikorperfragmente fur die Zoonosediagnostik Von der Fakultat fur Lebenswissenschaften der Technischen Universitat Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades eines Doktors
Rekombinante Antikorperfragmente fur die Zoonosediagnostik Von der Fakultat fur Lebenswissenschaften der Technischen Universitat Carolo-Wilhelmina zu Braunschweig zur Erlangung des Grades eines Doktors
Grundlagen der Physiologie
 Grundlagen der Physiologie Abbau eines Zuckermoleküls www.icbm.de/pmbio Lebensweise eines heterotrophen Aerobiers 1 Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien Anabolismus
Grundlagen der Physiologie Abbau eines Zuckermoleküls www.icbm.de/pmbio Lebensweise eines heterotrophen Aerobiers 1 Überblick Stoffwechsel Glykolyse Citratcyklus Chemiosmotische Prinzipien Anabolismus
Marina Fabry LUDWIG-MAXIMILIANS-UNIVERSITÄT
 Self-Assembled Nanoparticle Probes for Recognition and Detection of Biomolecules Dustin J. Maxwell, Jason R. Taylor & Shuming Nie Journal of the American Chemical Society 2002 Marina Fabry LUDWIG-MAXIMILIANS-UNIVERSITÄT
Self-Assembled Nanoparticle Probes for Recognition and Detection of Biomolecules Dustin J. Maxwell, Jason R. Taylor & Shuming Nie Journal of the American Chemical Society 2002 Marina Fabry LUDWIG-MAXIMILIANS-UNIVERSITÄT
Die Bedeutung des. mitochondrialen ABC-Transporters ATM3. für die Molybdäncofaktor-Biosynthese. in Arabidopsis thaliana
 Die Bedeutung des mitochondrialen ABC-Transporters ATM3 für die Molybdäncofaktor-Biosynthese in Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften der Technischen Universität Carolo-Wilhelmina
Die Bedeutung des mitochondrialen ABC-Transporters ATM3 für die Molybdäncofaktor-Biosynthese in Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften der Technischen Universität Carolo-Wilhelmina
Ausarbeitung des Seminars: Co-Kultivierung von Escherichia coli und Saccharomyces cerevisiae zur Paclitaxel-Synthese
 Ausarbeitung des Seminars: Co-Kultivierung von Escherichia coli und Saccharomyces cerevisiae zur Paclitaxel-Synthese Zhou, K., Qiao, K., Edgar, S. & Stephanopoulos, G. Distributing a metabolic pathway
Ausarbeitung des Seminars: Co-Kultivierung von Escherichia coli und Saccharomyces cerevisiae zur Paclitaxel-Synthese Zhou, K., Qiao, K., Edgar, S. & Stephanopoulos, G. Distributing a metabolic pathway
VL3: Struktur und Funktion des Photosystem I. Athina Zouni
 VL3: Struktur und Funktion des Photosystem I Athina Zouni VL3: Plan Einleitung Grundlagen zur Lichtreaktion der Photosynthese Struktur und Funktion des cyanobakteriellen PSI Kristallstruktur des PSI: 2.5
VL3: Struktur und Funktion des Photosystem I Athina Zouni VL3: Plan Einleitung Grundlagen zur Lichtreaktion der Photosynthese Struktur und Funktion des cyanobakteriellen PSI Kristallstruktur des PSI: 2.5
Zellulärer Abbau von Proteinen in Aminosäuren:! Proteine werden in Zellen durch Proteasom-Komplexe in! einzelne Aminosäuren abgebaut.!
 Zellulärer Abbau von Proteinen in Aminosäuren: Proteine werden in Zellen durch Proteasom-Komplexe in einzelne Aminosäuren abgebaut. Abbau von Aminosäuren: Uebersicht über den Aminosäureabbau Als erster
Zellulärer Abbau von Proteinen in Aminosäuren: Proteine werden in Zellen durch Proteasom-Komplexe in einzelne Aminosäuren abgebaut. Abbau von Aminosäuren: Uebersicht über den Aminosäureabbau Als erster
Abschlussklausur zur Vorlesung Biomoleküle II WS 2004/05
 16.02.2005 Abschlussklausur zur Vorlesung Biomoleküle II WS 2004/05 Name: Studienfach: Matrikelnummer: Fachsemester: Hinweise: 1. Bitte tragen Sie Ihren Namen, Matrikelnummer, Studienfach und Semesterzahl
16.02.2005 Abschlussklausur zur Vorlesung Biomoleküle II WS 2004/05 Name: Studienfach: Matrikelnummer: Fachsemester: Hinweise: 1. Bitte tragen Sie Ihren Namen, Matrikelnummer, Studienfach und Semesterzahl
5. STOFFWECHSEL. Energie-Quelle Brennstoffe. 5.1 Übersicht. Red.-Äquivalente. Katabolismus
 Energie-Quelle Brennstoffe 5.1 Übersicht Red.-Äquivalente Katabolismus Kohlenstoff-Quelle Baustoffe CO 2 -Fixierung 5.1 Übersicht Anabolismus 5.1 Übersicht Red.-Äquivalente ATMUNG Oxidative Phosphorylierung
Energie-Quelle Brennstoffe 5.1 Übersicht Red.-Äquivalente Katabolismus Kohlenstoff-Quelle Baustoffe CO 2 -Fixierung 5.1 Übersicht Anabolismus 5.1 Übersicht Red.-Äquivalente ATMUNG Oxidative Phosphorylierung
Ei ne P ä r sentati ti on vo n T om D ro t s e, P t e er Haak k und Patrick Schürmann
 Ei P ä tti T D t P t H k d Eine Präsentation von Tom Droste, Peter Haak und Patrick Schürmann Inhaltsverzeichnis Allgemein: Was ist Stickstofffixierung? Welche Arten der Fixierung gibt es? Welchen Zweck
Ei P ä tti T D t P t H k d Eine Präsentation von Tom Droste, Peter Haak und Patrick Schürmann Inhaltsverzeichnis Allgemein: Was ist Stickstofffixierung? Welche Arten der Fixierung gibt es? Welchen Zweck
Atmungskette inklusive Komplex II
 Atmungskette inklusive Komplex II Energiegewinnung durch oxidative Phosphorylierung GW2014 Das Prinzip der Oxidativen Phosphorylierung 14_01_01_harness_energy.jpg Chemiosmotische Kopplung 2016 1 1) 2)
Atmungskette inklusive Komplex II Energiegewinnung durch oxidative Phosphorylierung GW2014 Das Prinzip der Oxidativen Phosphorylierung 14_01_01_harness_energy.jpg Chemiosmotische Kopplung 2016 1 1) 2)
Zusammenfassung. Abkürzungsverzeichnis
 Zusammenfassung Abkürzungsverzeichnis I IX 1. Einleitung 1 1.1. Glutamatrezeptoren.............................. 1 1.1.1. Modularer Aufbau von ionotropen Glutamatrezeptoren....... 3 1.1.2. AMPA-Rezeptoren...........................
Zusammenfassung Abkürzungsverzeichnis I IX 1. Einleitung 1 1.1. Glutamatrezeptoren.............................. 1 1.1.1. Modularer Aufbau von ionotropen Glutamatrezeptoren....... 3 1.1.2. AMPA-Rezeptoren...........................
Abbildungs- und Tabellenverzeichnis Abkürzungsverzeichnis Genbezeichnung. 1. Einleitung 1
 Abbildungs- und Tabellenverzeichnis Abkürzungsverzeichnis Genbezeichnung VI VIII X 1. Einleitung 1 1.1 Das Plastom 1 1.2 Transkription in Chloroplasten 4 1.2.1 Die plastidäre Transkriptionsmaschinerie
Abbildungs- und Tabellenverzeichnis Abkürzungsverzeichnis Genbezeichnung VI VIII X 1. Einleitung 1 1.1 Das Plastom 1 1.2 Transkription in Chloroplasten 4 1.2.1 Die plastidäre Transkriptionsmaschinerie
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016
 Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 4 (20.06. 24.06.) Regulation der Transkription II, Translation
Biologie I/B: Klassische und molekulare Genetik, molekulare Grundlagen der Entwicklung Theoretische Übungen SS 2016 Fragen für die Übungsstunde 4 (20.06. 24.06.) Regulation der Transkription II, Translation
3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung
 3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung Transkriptionsfaktoren erfahren oft eine posttranslationelle Modifikation in Form von Phosphorylierung und werden dadurch in ihrer Aktivität
3.2 Posttranslationelle Modifikation von Wt1 durch Phosphorylierung Transkriptionsfaktoren erfahren oft eine posttranslationelle Modifikation in Form von Phosphorylierung und werden dadurch in ihrer Aktivität
Photosynthese einer der wichtigsten Stoffwechselprozesse der Pflanzen, beeinflusst maßgeblich unsere Atmosphäre und damit das Gesamtökosystem der Erde
 Photosynthese einer der wichtigsten Stoffwechselprozesse der Pflanzen, beeinflusst maßgeblich unsere Atmosphäre und damit das Gesamtökosystem der Erde 1. Photosynthese- und Zellatmungskreislauf Essentieller
Photosynthese einer der wichtigsten Stoffwechselprozesse der Pflanzen, beeinflusst maßgeblich unsere Atmosphäre und damit das Gesamtökosystem der Erde 1. Photosynthese- und Zellatmungskreislauf Essentieller
Modellkomplexe für das PhotosystemII. Robert Brüninghoff
 Modellkomplexe für das PhotosystemII Robert Brüninghoff 20.05.2014 Überblick Photosynthese Photosystem-II Inhalt Sauerstoffentwickelnder Komplex (OEC) & Modelle 20/05/2014 FH Münster, Bioanorganische Chemie
Modellkomplexe für das PhotosystemII Robert Brüninghoff 20.05.2014 Überblick Photosynthese Photosystem-II Inhalt Sauerstoffentwickelnder Komplex (OEC) & Modelle 20/05/2014 FH Münster, Bioanorganische Chemie
Photosynthese. Stoffwechsel Kontrolle Physiologie. D. W. Lawlor Übersetzung von Merlet Behncke Bearbeitung von Donat-P. Hader
 Photosynthese Stoffwechsel Kontrolle Physiologie D. W. Lawlor Übersetzung von Merlet Behncke Bearbeitung von Donat-P. Hader 98 Abbildungen, 17 Tabellen 1990 Georg Thieme Verlag Stuttgart -New York Inhaltsverzeichnis
Photosynthese Stoffwechsel Kontrolle Physiologie D. W. Lawlor Übersetzung von Merlet Behncke Bearbeitung von Donat-P. Hader 98 Abbildungen, 17 Tabellen 1990 Georg Thieme Verlag Stuttgart -New York Inhaltsverzeichnis
1. Einleitung Biologische Membranen...1
 I Inhaltsverzeichnis 1. Einleitung... 1 1.1 Biologische Membranen...1 1.1.1 Aufbau der Biomembran...1 1.1.2 Funktion der Plasmamembran...3 1.1.3 Membranlipide...4 1.1.4 Cholesterin als Membranbestandteil...5
I Inhaltsverzeichnis 1. Einleitung... 1 1.1 Biologische Membranen...1 1.1.1 Aufbau der Biomembran...1 1.1.2 Funktion der Plasmamembran...3 1.1.3 Membranlipide...4 1.1.4 Cholesterin als Membranbestandteil...5
5 Summary / Zusammenfassung Zusammenfassung
 5 Summary / Zusammenfassung 257 5.2 Zusammenfassung Ziel der vorliegenden Arbeit war es, unser Wissen über die Symbiose verschiedener solarbetriebener Taxa von Nudibranchiern mit Zooxanthellen zu erweitern,
5 Summary / Zusammenfassung 257 5.2 Zusammenfassung Ziel der vorliegenden Arbeit war es, unser Wissen über die Symbiose verschiedener solarbetriebener Taxa von Nudibranchiern mit Zooxanthellen zu erweitern,
Kapitel 5: 1. Siderophore assistieren dem Transfer welcher der folgenden Makronährstoffe über Membranen? A. Stickstoff B. Phosphor C. Eisen D.
 Kapitel 5: 1 Siderophore assistieren dem Transfer welcher der folgenden Makronährstoffe über Membranen? A. Stickstoff B. Phosphor C. Eisen D. Kalium Kapitel 5: 2 Welcher der folgenden Makronährstoffe ist
Kapitel 5: 1 Siderophore assistieren dem Transfer welcher der folgenden Makronährstoffe über Membranen? A. Stickstoff B. Phosphor C. Eisen D. Kalium Kapitel 5: 2 Welcher der folgenden Makronährstoffe ist
Tyrosinkinase- Rezeptoren
 Tyrosinkinase- Rezeptoren für bestimmte Hormone gibt es integrale Membranproteine als Rezeptoren Aufbau und Signaltransduktionsweg unterscheiden sich von denen der G- Protein- gekoppelten Rezeptoren Polypeptide
Tyrosinkinase- Rezeptoren für bestimmte Hormone gibt es integrale Membranproteine als Rezeptoren Aufbau und Signaltransduktionsweg unterscheiden sich von denen der G- Protein- gekoppelten Rezeptoren Polypeptide
Die Primärreaktion der Photosynthese
 Die Primärreaktion der Photosynthese 1. Energetische Betrachtungen Wie ist es energetisch möglich, dass das so energiearme und stabile Wassermolekül in der Primärreaktion der Photosynthese zu Sauerstoff
Die Primärreaktion der Photosynthese 1. Energetische Betrachtungen Wie ist es energetisch möglich, dass das so energiearme und stabile Wassermolekül in der Primärreaktion der Photosynthese zu Sauerstoff
Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle. in bioinformatisch identifizierten,
 Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle in bioinformatisch identifizierten, Pathogen-responsiven c/s-elementen aus Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften
Identifikation einer neuen WRKY-Transkriptionsfaktorbindungsstelle in bioinformatisch identifizierten, Pathogen-responsiven c/s-elementen aus Arabidopsis thaliana Von der Fakultät für Lebenswissenschaften
Enzyme SPF BCH am
 Enzyme Inhaltsverzeichnis Ihr kennt den Aufbau von Proteinen (mit vier Strukturelementen) und kennt die Kräfte, welche den Aufbau und die Funktion von Enzymen bestimmen... 3 Ihr versteht die Einteilung
Enzyme Inhaltsverzeichnis Ihr kennt den Aufbau von Proteinen (mit vier Strukturelementen) und kennt die Kräfte, welche den Aufbau und die Funktion von Enzymen bestimmen... 3 Ihr versteht die Einteilung
Molekulargenetische Charakterisierung von Untereinheiten des Cytochrom b 6 f-komplexes von Cyanobakterien der Gattung Anabaena
 Molekulargenetische Charakterisierung von Untereinheiten des Cytochrom b 6 f-komplexes von Cyanobakterien der Gattung Anabaena Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften (Dr. rer.
Molekulargenetische Charakterisierung von Untereinheiten des Cytochrom b 6 f-komplexes von Cyanobakterien der Gattung Anabaena Dissertation zur Erlangung des Doktorgrades der Naturwissenschaften (Dr. rer.
Stoffwechselbiologie 10 Der Lichtsammelkomplex der Fotosynthese
 Konstruktionsanleitung Die Abbildung zeigt die Wirkung von Lichtquanten auf ein Atom. Licht (Quant; Photon) 1 2 Fluoreszenz 3 4 Oxidationsmittel wird reduziert Werden isolierte Chlorophyllmoleküle im Reagenzglas
Konstruktionsanleitung Die Abbildung zeigt die Wirkung von Lichtquanten auf ein Atom. Licht (Quant; Photon) 1 2 Fluoreszenz 3 4 Oxidationsmittel wird reduziert Werden isolierte Chlorophyllmoleküle im Reagenzglas
Bio-Wasserstoff aus Mikroalgen Ein umweltverträglicher Energieträger der Zukunft?
 Bio-Wasserstoff aus Mikroalgen Ein umweltverträglicher Energieträger der Zukunft? Prof. Dr. Rüdiger chulz-friedrich Botanisches Institut und Botanischer Garten Christian-Albrechts-University Kiel Abt.
Bio-Wasserstoff aus Mikroalgen Ein umweltverträglicher Energieträger der Zukunft? Prof. Dr. Rüdiger chulz-friedrich Botanisches Institut und Botanischer Garten Christian-Albrechts-University Kiel Abt.
WILHELM MENKE. Die Struktur der Chloroplasten ACHIM TREBST. Zum Mechanismus der Photosynthese WESTDE{}TSCHER VERLAG KÖLN UND OPLADEN
 WILHELM MENKE Die Struktur der Chloroplasten ACHIM TREBST Zum Mechanismus der Photosynthese WESTDE{}TSCHER VERLAG KÖLN UND OPLADEN 1967 by Westdeutscher Verlag, Köln und Opladen Gesamtherstellung: Westdeutscher
WILHELM MENKE Die Struktur der Chloroplasten ACHIM TREBST Zum Mechanismus der Photosynthese WESTDE{}TSCHER VERLAG KÖLN UND OPLADEN 1967 by Westdeutscher Verlag, Köln und Opladen Gesamtherstellung: Westdeutscher
